Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Reproducibilidad y precisión mejoradas de la cuantificación de alto rendimiento de los datos de crecimiento bacteriano mediante un lector de microplacas
En este artículo
Resumen
Aquí, se presenta un protocolo de alto rendimiento para medir los datos de crecimiento, incluidas las curvas de crecimiento, la tasa de crecimiento y la tasa máxima de crecimiento. El protocolo se verificó y validó utilizando dos bacterias productoras de biopelícula. Los resultados y el enfoque aplicado en este estudio pueden ampliarse a otros protocolos de alto rendimiento que utilizan lectores de microplacas.
Resumen
Este estudio tuvo como objetivo desarrollar un protocolo repetible, confiable y de alto rendimiento para monitorear el crecimiento bacteriano en placas de 96 pocillos y analizar la tasa máxima de crecimiento. Se determinaron las curvas de crecimiento y las tasas máximas de crecimiento de dos especies bacterianas. Se investigaron cuestiones como (i) la condensación de la tapa, (ii) la corrección de la longitud de la trayectoria, (iii) el tamaño de la inoculación, (iv) el intervalo de tiempo de muestreo y (v) el sesgo espacial. La repetibilidad del protocolo se evaluó con tres réplicas técnicas independientes, con una desviación estándar de 0,03 entre las corridas. Las tasas máximas de crecimiento de Bacillus mycoides y Paenibacillus tundrae fueron (media ± DE) 0,99 h−1 ± 0,03 h−1 y 0,85 h−1 ± 0,025 h−1, respectivamente. Estas bacterias son más difíciles de controlar ópticamente debido a su afinidad por agruparse. Este estudio demuestra la importancia crítica del tamaño de la inoculación, la corrección de la longitud de la trayectoria, el calentamiento de la tapa, los intervalos de tiempo de muestreo y el sesgo espacial de la placa del pocillo para obtener datos confiables, precisos y reproducibles en los lectores de microplacas. El protocolo desarrollado y sus pasos de verificación pueden ampliarse a otros métodos utilizando lectores de microplacas y protocolos de alto rendimiento, lo que reduce los errores innatos de los investigadores y los costes de material.
Introducción
El creciente interés en la manipulación multiómica, incluidos los estudios mecanicistas y metabólicos de las bacterias, enfatiza la importancia de los métodos automatizados y de alto rendimiento, como el registro de datos de crecimiento 1,2. Los datos de crecimiento que comprenden parámetros cinéticos, como las tasas máximas de crecimiento, pueden ayudar a caracterizar las respuestas bacterianas a diferentes condiciones físicas, químicas y antibacterianas. Los datos de la tasa de crecimiento son una variable de respuesta estándar que se utiliza para descubrir posibles vínculos genotipo-fenotipos1 o indicar la seguridad microbiana y la vida útil de los productos alimenticios 3,4. Técnicas como la evolución adaptativa de laboratorio 5,6,7, el cribado de todo el genoma, ciertos ensayos químicos8 y varios cribados genéticos directos9 se basan en las tasas de crecimiento para evaluar los resultados.
Las mediciones de la densidad óptica (DO) de los cultivos bacterianos son un método microbiológico estándar para controlar el crecimiento bacteriano. Las mediciones de OD a menudo se registran a una longitud de onda de 600 nm, dependiendo de la dispersión de la luz y la densidad de celda10,11. La ley de Beer-Lambert explica la dependencia de los valores de OD de la concentración (es decir, la densidad de celdas, el número de celdas), la longitud del camino y el coeficiente de absorción. La geometría y el sistema óptico de un espectrofotómetro influyen en las lecturas de OD11. Los métodos clásicos de medición de OD pueden requerir mucho tiempo y trabajo, y los datos pueden conllevar una variedad de errores humanos. En este protocolo, se utiliza un lector de microplacas para disminuir el tiempo de análisis12,13 y la posibilidad de contaminación biológica. El análisis de alto rendimiento utilizando lectores de microplacas se aplica ampliamente en diferentes áreas de microbiología, como el cribado de bacterias productoras de biopelículas14,15, la inhibición del crecimiento bacteriano16, el crecimiento de células de levadura17, la determinación de la susceptibilidad a los antifúngicos18 y el cribado de toxicidad de los nanomateriales19.
Algunos investigadores han publicado protocolos de tasa de crecimiento bacteriano utilizando un lector de microplacas 12,20,21. Sin embargo, no se ha establecido completamente un protocolo exhaustivo que examine la fiabilidad de los datos recopilados. Se reporta que factores como el tipo de especie 22,23,24 y las cintas de sellado impactan la repetibilidad debido a la inadecuación de la transferencia de oxígeno en una placa de 96 pocillos 25,26. Delaney et al. informaron de grandes grupos de Methylorubrum extorquens (cepa de tipo salvaje) en el medio de crecimiento cuando se utilizó un lector de microplacas, lo que provocó datos de crecimiento extremadamente ruidosos24. El problema se resolvió mediante la eliminación de los genes asociados con la producción de biopelículas24. Debido a la secreción de sustancias poliméricas extracelulares, las bacterias productoras de biopelículas tienen una mayor afinidad para fusionarse y crear grupos de células. Por lo tanto, es más difícil monitorear su crecimiento utilizando técnicas de dispersión de luz (por ejemplo, espectrofotómetros y lectores de microplacas).
Este protocolo tiene como objetivo establecer los pasos para obtener datos reproducibles en un método de alto rendimiento utilizando un lector de microplacas. Se utilizaron Bacillus mycoides y Paenibacillus tundrae debido a su rápido crecimiento y capacidad de producción de biopelículas, que tradicionalmente son desafiantes en enfoques manuales y automatizados. Se investigaron factores como (i) la corrección de la longitud del camino, (ii) la condensación en la tapa, (iii) el tamaño del inóculo, (iv) el intervalo de tiempo de muestreo y (v) el sesgo espacial para evaluar la fiabilidad y reproducibilidad de los datos. Este protocolo presenta los pasos para monitorear con precisión el crecimiento bacteriano y medir las tasas de crecimiento específicas utilizando un lector de microplacas.
Protocolo
NOTA: Todos los pasos de este protocolo deben seguirse en condiciones estériles (es decir, entre dos llamas o una cabina de bioseguridad). Todos los materiales y herramientas se esterilizan en autoclave durante 20 min. Consulte la Tabla de materiales para obtener detalles sobre todos los materiales, equipos y software utilizados en este protocolo. Las manos enguantadas se desinfectan, se mantienen húmedas con desinfectante de manos o solución de alcohol al 70% durante al menos 1 minuto y no se retiran del armario de seguridad después. De lo contrario, se debe repetir el procedimiento de desinfección antes de volver a introducir las manos en el armario de seguridad. PRECAUCIÓN: Asegúrese de que el desinfectante se haya evaporado por completo antes de usar una llama abierta.
NOTA: Se aislaron dos bacterias de la biofiltración de agua potable como se explicó anteriormente27 por su capacidad para producir biopelícula. Se identificaron mediante la secuenciación completa del ARNr 16S y se presentaron al NCBI como Bacillus mycoides (SAMN10518261) y Paenibacillus tundrae (SAMN10452279).
1. Preparación de caldo bacteriano en glicerol
- Pesar 3 g de caldo de soja tríptico (TSB) en polvo y disolverlo en 100 ml de agua destilada. Esteriliza el caldo en autoclave durante 20 minutos y déjalo enfriar a temperatura ambiente. Agregue 50 mL de TSB a un matraz de 50 mL.
- Transfiera las bacterias a placas de agar 27,28. Con un bucle, elija una colonia de bacterias de la placa de agar. Agregue el contenido del bucle en el matraz girando suavemente el bucle. Incubar el matraz durante la noche a 30 °C ± 1 °C con agitación orbital a 150 rpm.
NOTA: La temperatura y la agitación deben ajustarse de acuerdo con el tipo de cepa. - Prepare otro matraz de 50 mL con 47,5 mL de TSB. Transfiera 2,5 mL del cultivo nocturno al matraz. Incubarlo durante 5-6 h para alcanzar OD600 = 0,6 0,05 (para llegar a la fase exponencial media).
- Transfiera el cultivo a un tubo centrífugo esterilizado de 50 mL. Centrifugar el cultivo a 2.200 × g durante 10 min. Decanta el caldo suavemente para evitar perder las células acumuladas en el fondo del tubo.
- Añadir 5 mL de solución salina tamponada con fosfato (PBS) al mismo tubo y centrifugar a 2.200 × g durante 10 min. Repita este paso 3 veces.
- Ajuste el diámetro exterior600 de las células a 0,6 0,05 utilizando una pipeta para valorar una cantidad adecuada de solución de PBS. Registre con precisión la cantidad agregada de PBS para uso futuro.
- Valore el glicerol a la suspensión para alcanzar el 20% v/v. Tape el tubo y el vórtice durante 15 s.
- Transfiera 1,5 mL de la suspensión a crioviales de 2 mL. Coloque los crioviales en una caja adecuada y guárdelos en un congelador a -80 °C.
2. Preparar la cultura de la noche a la mañana.
- Deje que los crioviales con caldo bacteriano se descongelen a temperatura ambiente durante 30 minutos o hasta que el contenido sea líquido.
NOTA: Se recomienda no acelerar el proceso de descongelación. - Prepare un tubo esterilizado con 10 mL de TSB. Transfiera 50 μL de la suspensión original al tubo. Incubar el tubo durante la noche (16-18 h) a 30 °C ± 1 °C con agitación orbital a 150 rpm.
3. Prepara el inóculo.
- Prepare un tubo con 10 mL de TSB esterilizado. Transfiera 500 μL del cultivo nocturno al tubo. Incubar durante 6-8 h para alcanzar OD600 = 0,6 0,05 (o llegar a la fase exponencial media).
- Transfiera el cultivo a un tubo de centrífuga de 15 mL. Centrifugar el tubo a 2.200 × g durante 10 min. Decanta el caldo suavemente.
- Añadir 5 mL de PBS y centrifugar a 2.200 × g durante 10 min. Repita este paso 3 veces.
- Ajuste el diámetro exterior600 de las células a 0,6 0,05 valorando la solución de PBS con una pipeta. Registre con precisión la cantidad agregada de PBS para uso futuro.
4. Transferencia del medio de crecimiento al lector de microplacas
- Elija una placa de pocillo transparente, estéril, de fondo plano con tapa (es decir, placa de 96 pocillos, placa de 6 pocillos, placa de 12 pocillos).
- Mida la corrección de la longitud del camino de la microplaca con agua desionizada antes de ejecutar el protocolo, como se explica en el paso 1.10.
- Dibuje una tabla de referencia basada en la placa de pocillo seleccionada para especificar las posiciones de las muestras antes de cargarlas. Por ejemplo, dibuje una tabla de 8 x 12 para una placa de 96 pocillos.
NOTA: La tabla de referencia evita confusiones durante las etapas de incubación y recolección de resultados. - Coloque los siguientes elementos en una cabina de bioseguridad: una microplaca con tapa, pipeta de 1.000 μL y puntas de pipeta de 200 μL, 1.000 μL y 200 μL, varios crioviales, un vaso de precipitados de 25 mL, el medio de crecimiento y el inóculo.
NOTA: Los elementos mencionados deben transferirse asépticamente a la cabina de bioseguridad. - Añada 10 ml de medio de crecimiento al vaso de precipitados. Transfiera la cantidad calculada de inóculo al vaso de precipitados con el medio de crecimiento.
NOTA: Las proporciones de inóculo en este protocolo son 1% y 5%. Por ejemplo, para lograr una proporción de inóculo del 5% y 1%, agregue 500 μL y 100 μL del inóculo a 9,5 mL y 9,9 mL del medio de crecimiento, respectivamente. - Después de añadir el inóculo, agite suavemente el vaso de precipitados para obtener una distribución uniforme. Dispense 200 μL del medio inoculado en los pocillos designados utilizando la tabla de referencia. Elija al menos tres pocillos para que actúen como controles agregando 200 μL del medio de crecimiento (sin inóculo).
NOTA: Asegúrese de que los pozos de control estén claramente marcados en la tabla de referencia. Si se observa algún crecimiento (contaminación) en estos pozos, se debe repetir el experimento. - Dispense 200 μL de agua desionizada en los pozos de borde donde la tasa de evaporación es mayor. Coloque la tapa suavemente antes de retirar la placa de 96 pocillos del gabinete de seguridad. Coloque la placa de 96 pocillos en el lector de placas con cuidado, asegurándose de que no haya movimientos bruscos.
5. Configuración del lector de microplacas
- Encienda el lector de microplacas y, a continuación, utilice el software integrado para crear el protocolo personalizado. En el Administrador de tareas, abra el protocolo y elija Crear nuevo | Protocolo estándar. Abra Procedimiento y ajuste la configuración de la siguiente manera:
- Ajuste la temperatura a 30 °C (o los valores previstos) mediante Establecer temperatura.
- Haga clic en Incubadora encendida y luego establezca el Gradiente en 1 °C.
NOTA: Para evitar datos poco fiables debido a la condensación en la tapa, asegúrese de que Gradiente esté ajustado a 1 °C. - Elija la opción Precalentar , asegurando una distribución uniforme del calor a lo largo de la placa del pocillo.
- Abra Start Kinetic y elija la duración de incubación deseada utilizando el Tiempo de ejecución (por ejemplo, 12:00:00 o 24:00:00, etc.) y Intervalo entre lecturas para 00:30:00 (representa 30 min) o 01:00:00 (representa 1 h).
NOTA: La lectura de cada placa de 96 pocillos tarda aproximadamente 1 min. - Establezca la configuración de agitación de la siguiente manera: abra Agitar; establezca el modo de vibración en lineal; haga clic en Agitación continua; y ajuste la frecuencia a 567 cpm (o cualquier valor deseado).
- Establezca la configuración de lectura de OD de la siguiente manera: haga clic en Leer | Absorbancia; seleccione Endpoint/Kinetic y Monochromators; establezca la longitud de onda en 600 nm; y haga clic en Validar | Guardar para guardarlo como un nuevo protocolo con el nombre y la fecha adecuados.
6. Registro de datos de crecimiento
- Abrir Administrador de tareas | Leer ahora.
- Elija el protocolo guardado, luego haga clic en Aceptar para comenzar el protocolo.
- Guarde las lecturas para el análisis de datos.
7. Análisis de datos
- Transfiera los resultados de OD600 (datos de crecimiento) a formato de hoja de cálculo. En la sección de resultados , haga clic en la opción de imagen de cada pozo para observar las curvas de crecimiento finales.
- Abra el archivo de la hoja de cálculo y reorganice los datos registrados en función de la tabla de referencia. Reste el valor medio de las lecturas en blanco de las otras lecturas.
NOTA: Los espacios en blanco son las lecturas de OD en el tiempo cero. En este protocolo, se consideran los valores de OD600 para el cálculo. La densidad de celdas se puede utilizar si se dispone de la curva de calibración para OD600 frente al recuento de células.
8. Determinación de la tasa de crecimiento
- Utilice los datos transferidos en la hoja de cálculo creada en el paso 1.6. Utilice el método descrito a continuación y la ecuación (1) para evaluar la tasa de crecimiento.
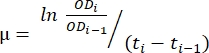 (1)
(1)
donde μ es la tasa de crecimiento, ODi es el OD600 en cada punto de tiempo, ODi-1 es el valor inicial de OD600 en el último punto de tiempo, ti y ti-1 son las diferencias de tiempo entre los dos puntos mencionados (por ejemplo, 0,5 para intervalos de tiempo de 30 minutos, 1 para intervalos de tiempo de 1 h). - Calcule la media y la desviación estándar de cinco tasas de crecimiento sucesivas utilizando el software de análisis de datos deseado. Obsérvese que la media más grande con la desviación estándar más baja es la tasa de crecimiento máxima (μm) 12.
9. Determinación del sesgo espacial
NOTA: Los lectores de microplacas y las placas introducen sesgos en los resultados. Es crucial evaluar los sesgos espaciales de las placas utilizadas en un lector de microplacas específico para garantizar la fiabilidad y la reproducibilidad de los resultados. Para lograrlo, siga estos pasos:
- Repita los pasos 1.2 a 1.6.
- Registre los datos y analícelos de acuerdo con los pasos 1.7 y 1.8 para calcular la tasa de crecimiento de cada pozo.
- Cree un mapa de calor para visualizar los cambios en la tasa de crecimiento en la placa de 96 pocillos.
- Realizar el análisis estadístico (ver las Tablas Suplementarias), asegurándose de que no haya diferencias significativas entre los pozos.
NOTA: Es fundamental utilizar el mismo tipo de bacteria y el mismo tamaño de inóculo en todos los pocillos.
10. Validación de la lectura del DO y factor de corrección de la longitud del trayecto
NOTA: La determinación del factor de corrección de la longitud de ruta es crucial para validar los datos y garantizar la fiabilidad y validez en diferentes dispositivos.
- Utilice los pasos 1.2 y 1.3 para preparar el inóculo de B. mycoides, durante la noche.
- Prepare tres matraces esterilizados de 50 mL y agregue 49,5 mL de TSB fresco a cada matraz. Añadir 500 μL del inóculo a cada matraz en un armario de seguridad de forma aséptica. Añadir 50 mL de TSB no inoculada a un matraz esterilizado, que servirá como control.
- Incubar los matraces a 30 °C y 150 rpm. Cada 30 minutos, tome muestras de cada matraz de forma aséptica y lea y registre el diámetro exterior600 en el espectrofotómetro.
- Agregue 9,9 mL de TSB a un vaso de precipitados de 25 mL. Añadir 100 μl de inóculo al vaso de precipitados y agitar suavemente.
- Añada 200 μL de la mezcla en el vaso de precipitados a los seis pocillos de una columna seleccionada en una placa de 96 pocillos. Añadir 200 μL de TSB no inoculado a tres pocillos como control. Llene los pozos de borde con 200 μL de agua desionizada.
- Coloque la tapa y transfiera suavemente la placa de 96 pocillos al lector de microplacas. Siga los pasos 1.5 y 1.6 para registrar los datos de crecimiento.
- Trace el diámetro exterior600 del lector de microplacas frente al diámetro exterior600 del espectrofotómetro. Tenga en cuenta que la pendiente es el desplazamiento entre los dos conjuntos de lecturas, llamado factor de corrección de longitud de ruta.
NOTA: La incubación de la incubadora y del lector de microplacas debe realizarse simultáneamente el mismo día.
Resultados
Validación de la lectura del diámetro exterior y factor de corrección de la longitud del trayecto
Se tomaron muestras divididas de cultivo de B. mycoides en diferentes puntos de tiempo y se midieron utilizando el lector de microplacas y el espectrofotómetro (Figura 1A). Este paso se llevó a cabo para validar los resultados en diferentes dispositivos. Los datos de OD600 se correlacionaron, pero no coincidieron (
Discusión
Los lectores de microplacas permiten obtener tasas de crecimiento consistentes y repetibles. Esta tecnología minimiza el error humano y permite un muestreo de alto rendimiento. La pequeña cantidad de cultivo requerida por muestra hace que este enfoque sea una alternativa atractiva y de bajo costo a los recuentos de células que utilizan matraces o tubos de ensayo. Los lectores de microplacas permiten un gran tamaño de muestra, lo que aumenta el poder estadístico y, en consecuencia, f...
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Este trabajo fue financiado por el Consejo de Investigación de Ciencias Naturales e Ingeniería (NSERC) / Cátedra de Investigación Industrial de Agua de Halifax en Calidad y Tratamiento del Agua (Subvención No. IRCPJ 349838-16). El equipo de autores también desea agradecer la ayuda de Anita Taylor en la revisión de este artículo.
Materiales
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
Referencias
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados