É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Reprodutibilidade e precisão aprimoradas da quantificação de alto rendimento de dados de crescimento bacteriano usando um leitor de microplacas
Neste Artigo
Resumo
Aqui, um protocolo de alto rendimento é apresentado para medir dados de crescimento, incluindo curvas de crescimento, taxa de crescimento e taxa máxima de crescimento. O protocolo foi verificado e validado usando duas bactérias produtoras de biofilme. Os resultados e a abordagem aplicados neste estudo podem ser expandidos para outros protocolos de alto rendimento usando leitores de microplacas.
Resumo
Este estudo teve como objetivo desenvolver um protocolo repetível, confiável e de alto rendimento para monitorar o crescimento bacteriano em placas de 96 poços e analisar a taxa máxima de crescimento. As curvas de crescimento e as taxas máximas de crescimento de duas espécies bacterianas foram determinadas. Questões incluindo (i) condensação da tampa, (ii) correção do comprimento do caminho, (iii) tamanho da inoculação, (iv) intervalo de tempo de amostragem e (v) viés espacial foram investigados. A repetibilidade do protocolo foi avaliada com três repetições técnicas independentes, com desvio padrão de 0,03 entre as execuções. As taxas máximas de crescimento de Bacillus mycoides e Paenibacillus tundrae foram determinadas em (média ± DP) 0,99 h−1 ± 0,03 h−1 e 0,85 h−1 ± 0,025 h−1, respectivamente. Essas bactérias são mais difíceis de monitorar opticamente devido à sua afinidade para se aglomerar. Este estudo demonstra a importância crítica do tamanho da inoculação, correção do comprimento do caminho, aquecimento da tampa, intervalos de tempo de amostragem e viés espacial da placa do poço para obter dados confiáveis, precisos e reprodutíveis em leitores de microplacas. O protocolo desenvolvido e suas etapas de verificação podem ser expandidos para outros métodos usando leitores de microplacas e protocolos de alto rendimento, reduzindo os erros inatos e os custos de material dos pesquisadores.
Introdução
O desenvolvimento do interesse na manipulação multiômica, incluindo estudos de mecanismos e metabólicos de bactérias, enfatiza a importância de métodos automatizados e de alto rendimento, como o registro de dados de crescimento 1,2. Os dados de crescimento que compreendem parâmetros cinéticos, como taxas máximas de crescimento, podem ajudar a caracterizar as respostas bacterianas a diferentes condições físicas, químicas e antibacterianas. Os dados da taxa de crescimento são uma variável de resposta padrão utilizada para descobrir possíveis ligações genótipo-fenótipos1 ou indicar a segurança microbiana e a vida útil dos produtos alimentícios 3,4. Técnicas como evolução laboratorial adaptativa 5,6,7, triagem de todo o genoma, certos ensaios químicos8 e várias triagens genéticas diretas9 dependem das taxas de crescimento para avaliar os resultados.
As medições de densidade óptica (DO) de culturas bacterianas são um método microbiológico padrão para monitorar o crescimento bacteriano. As medições de DO são frequentemente registradas em um comprimento de onda de 600 nm, contando com o espalhamento de luz e a densidade celular10,11. A lei de Beer-Lambert explica a dependência dos valores de DO na concentração (ou seja, densidade celular, número de células), comprimento do caminho e coeficiente de absortividade. A geometria e o sistema óptico de um espectrofotômetro influenciam as leituras de DO11. Os métodos clássicos de medições de DO podem ser muito demorados e trabalhosos, e os dados podem conter uma variedade de erros humanos. Nesse protocolo, um leitor de microplacas é utilizado para diminuir o tempo doanalista12,13 e a chance de contaminação biológica. A análise de alto rendimento usando leitores de microplacas é amplamente aplicada em diferentes áreas de microbiologia, como triagem de bactérias produtoras de biofilme14,15, inibição do crescimento bacteriano16, crescimento de células de levedura17, determinação da suscetibilidade antifúngica18 e triagem de toxicidade de nanomateriais19.
Alguns pesquisadores publicaram protocolos de taxa de crescimento bacteriano usando um leitor de microplacas 12,20,21. No entanto, um protocolo completo que examine a confiabilidade dos dados coletados não foi totalmente estabelecido. É relatado que fatores como o tipo de espécie 22,23,24 e fitas de vedação afetam a repetibilidade devido à inadequação da transferência de oxigênio em uma placa de 96 poços 25,26. Delaney et al. relataram grandes aglomerados de Methylorubrum extorquens (cepa do tipo selvagem) no meio de crescimento ao usar um leitor de microplacas, o que causou dados de crescimento extremamente ruidosos24. O problema foi resolvido removendo os genes associados à produção de biofilme24. Devido à secreção de substâncias poliméricas extracelulares, as bactérias produtoras de biofilme têm maior afinidade para se aglutinar e criar aglomerados de células. Portanto, é mais desafiador monitorar seu crescimento usando técnicas de espalhamento de luz (por exemplo, espectrofotômetros e leitores de microplacas).
Este protocolo tem como objetivo estabelecer etapas para a obtenção de dados reprodutíveis em um método de alto rendimento utilizando um leitor de microplacas. Bacillus mycoides e Paenibacillus tundrae foram utilizados devido ao seu rápido crescimento e capacidade de produção de biofilme, que são tradicionalmente desafiadores em abordagens manuais e automatizadas. Fatores como (i) correção do comprimento do caminho, (ii) condensação na pálpebra, (iii) tamanho do inóculo, (iv) intervalo de tempo de amostragem e (v) viés espacial foram investigados para avaliar a confiabilidade e reprodutibilidade dos dados. Este protocolo apresenta etapas para monitorar com precisão o crescimento bacteriano e medir taxas de crescimento específicas usando um leitor de microplacas.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: Todas as etapas deste protocolo devem ser seguidas em condições estéreis (ou seja, entre duas chamas ou um gabinete de biossegurança). Todos os materiais e ferramentas são autoclavados por 20 min. Consulte a Tabela de Materiais para obter detalhes sobre todos os materiais, equipamentos e software usados neste protocolo. As mãos enluvadas são desinfetadas, mantidas molhadas com desinfetante para as mãos ou solução de álcool a 70% por pelo menos 1 minuto e não removidas do gabinete de segurança posteriormente. Caso contrário, o procedimento de desinfecção deve ser repetido antes de colocar as mãos de volta no armário de segurança. CUIDADO: Certifique-se de que o desinfetante esteja completamente evaporado antes de usar uma chama aberta.
NOTA: Duas bactérias foram isoladas da biofiltração de água potável, conforme explicado anteriormente27 por sua capacidade de produzir biofilme. Eles foram identificados pelo sequenciamento completo de rRNA 16S e submetidos ao NCBI como Bacillus mycoides (SAMN10518261) e Paenibacillus tundrae (SAMN10452279).
1. Preparando o estoque bacteriano em glicerol
- Pese 3 g de pó de caldo de soja tríptico (TSB) e dissolva-o em 100 mL de água destilada. Autoclave o caldo por 20 min e deixe esfriar até a temperatura ambiente. Adicionar 50 ml de TSB a um balão de 50 ml.
- Transferir as bactérias para placas de ágar27,28. Usando um laço, pegue uma colônia de bactérias da placa de ágar. Adicione o conteúdo do loop ao frasco girando suavemente o loop. Incubar o balão durante a noite a 30 °C ± 1 °C com agitação orbital a 150 rpm.
NOTA: A temperatura e a agitação precisam ser ajustadas de acordo com o tipo de cepa. - Prepare outro balão de 50 mL com 47,5 mL de TSB. Transferir 2,5 ml da cultura durante a noite para o balão. Incube-o por 5-6 h para atingir OD600 = 0,6 0,05 (para chegar à fase exponencial intermediária).
- Transfira a cultura para um tubo de centrífuga esterilizado de 50 mL. Centrifugue a cultura a 2.200 × g por 10 min. Transvase o caldo suavemente para evitar a perda de células coletadas no fundo do tubo.
- Adicione 5 mL de solução salina tamponada com fosfato (PBS) ao mesmo tubo e centrifugue a 2.200 × g por 10 min. Repita esta etapa 3x.
- Ajuste o OD600 das células para 0,6 0,05 usando uma pipeta para titular uma quantidade adequada de solução de PBS. Registre com precisão a quantidade adicional de PBS para uso futuro.
- Titule o glicerol para a suspensão para atingir 20% v/v. Tampe o tubo e o vórtice por 15 s.
- Transfira 1,5 mL da suspensão para 2 mL criogeniais. Coloque os criogenianos em uma caixa apropriada e armazene-os em um freezer a -80 ° C.
2. Prepare a cultura da noite para o dia.
- Deixe os criogeniais com caldo bacteriano descongelarem à temperatura ambiente por 30 min ou até que o conteúdo esteja líquido.
NOTA: Recomenda-se não acelerar o processo de descongelamento. - Prepare um tubo esterilizado com 10 mL de TSB. Transfira 50 μL da suspensão de estoque para o tubo. Incubar o tubo durante a noite (16-18 h) a 30 °C ± 1 °C com agitação orbital a 150 rpm.
3. Prepare o inóculo.
- Prepare um tubo com 10 mL de TSB esterilizado. Transfira 500 μL da cultura noturna para o tubo. Incube-o por 6-8 h para atingir OD600 = 0,6 0,05 (ou chegar à fase exponencial intermediária).
- Transfira a cultura para um tubo de centrífuga de 15 mL. Centrifugue o tubo a 2.200 × g por 10 min. Transvase o caldo delicadamente.
- Adicione 5 mL de PBS e centrifugue a 2.200 × g por 10 min. Repita esta etapa 3x.
- Ajuste o OD600 das células para 0,6 0,05 titulando a solução de PBS usando uma pipeta. Registre com precisão a quantidade adicional de PBS para uso futuro.
4. Transferência do meio de crescimento para o leitor de microplacas
- Escolha uma placa de poço transparente, estéril e de fundo plano com tampa (ou seja, placa de 96 poços, placa de 6 poços, placa de 12 poços).
- Meça a correção do comprimento do caminho para a microplaca usando água DI antes de executar o protocolo, conforme explicado na etapa 1.10.
- Desenhe uma tabela de referência com base na placa de poço selecionada para especificar as posições das amostras antes do carregamento. Por exemplo, desenhe uma tabela de 8 x 12 para uma placa de 96 poços.
NOTA: A tabela de referência evita confusão durante as etapas de incubação e coleta de resultados. - Coloque os seguintes itens em um gabinete de biossegurança: uma microplaca com tampa, pipetador de 1.000 μL e 200 μL, ponteiras de pipeta de 1.000 μL e 200 μL, vários criogeniais, um béquer de 25 mL, o meio de crescimento e o inóculo.
NOTA: Os itens mencionados devem ser transferidos para o gabinete de biossegurança assepticamente. - Adicione 10 ml de meio de crescimento ao copo. Transferir a quantidade calculada de inóculo para o copo com o meio de cultura.
NOTA: As proporções de inóculo neste protocolo são de 1% e 5%. Por exemplo, para atingir uma proporção de inóculo de 5% e 1%, adicione 500 μL e 100 μL do inóculo a 9,5 mL e 9,9 mL do meio de crescimento, respectivamente. - Depois de adicionar o inóculo, agite suavemente o béquer para obter uma distribuição uniforme. Dispensar 200 μL do meio inoculado nos alvéolos designados, utilizando a tabela de referência. Escolha pelo menos três poços para atuar como controles, adicionando 200 μL do meio de crescimento (sem inóculo).
NOTA: Certifique-se de que os poços de controle estejam claramente marcados na tabela de referência. Se algum crescimento (contaminação) for observado nesses poços, o experimento deve ser repetido. - Dispense 200 μL de água DI nos poços de borda onde a taxa de evaporação é maior. Coloque a tampa com cuidado antes de remover a placa de 96 poços do armário de segurança. Coloque a placa de 96 poços no leitor de placas com cuidado, garantindo que não haja movimentos bruscos.
5. Configurações do leitor de microplacas
- Ligue o leitor de microplacas e use o software incorporado para criar o protocolo personalizado. No Gerenciador de Tarefas, abra o protocolo e escolha Criar novo | Protocolo padrão. Abra o Procedimento e ajuste as configurações da seguinte maneira:
- Defina a temperatura para 30 °C (ou os valores pretendidos) utilizando Definir temperatura.
- Clique em Incubadora Ligada e defina o Gradiente para 1 °C.
NOTA: Para evitar dados não confiáveis devido à condensação na tampa, certifique-se de que o gradiente esteja definido para 1 °C. - Escolha a opção Pré-aquecimento , garantindo uma distribuição uniforme do calor ao longo da placa do poço.
- Abra Start Kinetic e escolha a duração de incubação desejada usando o Tempo de execução (por exemplo, 12:00:00 ou 24:00:00, etc.) e Intervalo entre leituras para 00:30:00 (representa 30 min) ou 01:00:00 (representa 1 h).
NOTA: A leitura de cada placa de 96 poços leva aproximadamente 1 min. - Defina as configurações de agitação da seguinte forma: abra Agitar; defina o Modo de agitação como Linear; clique em Agitação contínua; e ajuste a frequência para 567 cpm (ou qualquer valor desejado).
- Defina as configurações de leitura de OD da seguinte forma: clique em Ler | Absorbância; selecione Endpoint/Cinético e Monocromadores; defina o comprimento de onda para 600 nm; e clique em Validar | Save para salvá-lo como um novo protocolo com o nome e a data apropriados.
6. Registro de dados de crescimento
- Abra o Gerenciador de Tarefas | Leia agora.
- Escolha o protocolo salvo e clique em OK para iniciar o protocolo.
- Salve as leituras para análise de dados.
7. Análise de dados
- Transfira os resultados do OD600 (dados de crescimento) para o formato de planilha. Na seção de resultados , clique na opção de imagem de cada poço para observar as curvas finais de crescimento.
- Abra o arquivo de planilha e reorganize os dados gravados com base na tabela de referência. Subtraia o valor médio das leituras em branco das outras leituras.
NOTA: Os espaços em branco são as leituras OD no tempo zero. Neste protocolo, os valores de OD600 são considerados para cálculo. A densidade celular pode ser usada se a curva de calibração para OD600 vs. contagem de células estiver disponível.
8. Determinação da Taxa de Crescimento
- Use os dados transferidos na planilha criada na etapa 1.6. Use o método descrito abaixo e a equação (1) para avaliar a taxa de crescimento.
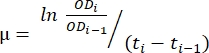 Características (1)
Características (1)
onde μ é a taxa de crescimento, ODi é o OD600 em cada ponto de tempo, ODi-1 é o valor inicial de OD600 no último ponto de tempo, ti e ti-1 são as diferenças de tempo entre os dois pontos mencionados (por exemplo, 0,5 para intervalos de tempo de 30 minutos, 1 para intervalos de tempo de 1 h). - Calcule a média e o desvio padrão de cinco taxas de crescimento sucessivas usando o software de análise de dados desejado. Observe que a maior média com o menor desvio padrão é a taxa máxima de crescimento (μm) 12.
9. Determinando o viés espacial
NOTA: Leitores e placas de microplacas introduzem viés nos resultados. É crucial avaliar os vieses espaciais das placas utilizadas em um leitor de microplacas específico para garantir a confiabilidade e reprodutibilidade dos resultados. Para conseguir isso, use as seguintes etapas:
- Repita as etapas 1.2-1.6.
- Registre os dados e analise de acordo com a etapa 1.7 e a etapa 1.8 para calcular a taxa de crescimento de cada poço.
- Crie um mapa de calor para visualizar as mudanças na taxa de crescimento na placa de 96 poços.
- Realize a análise estatística (consulte as Tabelas Suplementares), garantindo que não haja diferenças significativas entre os poços.
NOTA: É essencial usar o mesmo tipo de bactéria e o mesmo tamanho de inóculo em todos os poços.
10. Validação de leitura OD e fator de correção de comprimento de caminho
NOTA: Determinar o fator de correção do comprimento do caminho é crucial para validar os dados e garantir a confiabilidade e a validade em diferentes dispositivos.
- Use as etapas 1.2 e 1.3 para preparar o inóculo noturno de B. mycoides.
- Preparar três balões esterilizados de 50 ml e adicionar 49,5 ml de TSB fresco a cada balão. Adicionar assepticamente 500 μl do inóculo a cada balão numa cabina de segurança. Adicionar 50 mL de TSB não inoculado a um frasco esterilizado, que servirá como controle.
- Incubar os frascos a 30 °C e 150 rpm. A cada 30 minutos, colher amostras de cada frasco assepticamente e ler e registar OD600 no espectrofotómetro.
- Adicione 9,9 mL de TSB a um béquer de 25 mL. Adicionar 100 μl de inóculo ao copo e agitar suavemente.
- Adicione 200 μL da mistura no béquer aos seis poços em uma coluna selecionada em uma placa de 96 poços. Adicione 200 μL de TSB não inoculado a três poços como controle. Encha os poços de borda com 200 μL de água DI.
- Coloque a tampa e transfira suavemente a placa de 96 poços para o leitor de microplacas. Siga as etapas 1.5 e 1.6 para registrar os dados de crescimento.
- Plotar o OD600 do leitor de microplacas contra o OD600 do espectrofotómetro. Observe que a inclinação é o deslocamento entre os dois conjuntos de leituras, chamado de fator de correção do comprimento do caminho.
NOTA: A incubadora e a incubação do leitor de microplacas devem ser feitas simultaneamente no mesmo dia.
Access restricted. Please log in or start a trial to view this content.
Resultados
Validação de leitura OD e fator de correção de comprimento de caminho
Amostras divididas de cultura de B. mycoides foram coletadas em diferentes momentos e medidas usando o leitor de microplacas e o espectrofotômetro (Figura 1A). Esta etapa foi realizada para validar os resultados em diferentes dispositivos. Os dados do OD600 se correlacionaram, mas não corresponderam (Figura 1B). A ...
Access restricted. Please log in or start a trial to view this content.
Discussão
Os leitores de microplacas permitem obter taxas de crescimento consistentes e repetíveis. Essa tecnologia minimiza o erro humano e permite amostragem de alto rendimento. A pequena quantidade de cultura necessária por amostra torna essa abordagem uma alternativa atraente e de baixo custo para contagens de células usando frascos ou tubos de ensaio. Os leitores de microplacas permitem um grande tamanho de amostra, aumentando o poder estatístico e, posteriormente, facilitando cálculos c...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
Este trabalho foi financiado pelo Conselho de Pesquisa em Ciências Naturais e Engenharia (NSERC) / Halifax Water Industrial Research Chair in Water Quality and Treatment (Grant No. IRCPJ 349838-16). A equipe de autores também gostaria de agradecer a ajuda de Anita Taylor na revisão deste artigo.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
Referências
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935(2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178(2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828(2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , at http://www.thermoscientific.de/eThermo/CMA/PDFs/Product/productPDF_58468.pdf (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437(2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197(2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957(2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235(2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034(2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248(2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064(2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados