Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Reproductibilité et précision améliorées de la quantification à haut débit des données de croissance bactérienne à l’aide d’un lecteur de microplaques
Dans cet article
Résumé
Ici, un protocole à haut débit est présenté pour mesurer les données de croissance, y compris les courbes de croissance, le taux de croissance et le taux de croissance maximal. Le protocole a été vérifié et validé à l’aide de deux bactéries productrices de biofilm. Les résultats et l’approche appliqués dans cette étude peuvent être étendus à d’autres protocoles à haut débit utilisant des lecteurs de microplaques.
Résumé
Cette étude visait à développer un protocole reproductible, fiable et à haut débit pour surveiller la croissance bactérienne dans des plaques à 96 puits et analyser le taux de croissance maximal. Les courbes de croissance et les taux de croissance maximaux de deux espèces bactériennes ont été déterminés. Des problèmes tels que (i) la condensation du couvercle, (ii) la correction de la longueur du trajet, (iii) la taille de l’inoculation, (iv) l’intervalle de temps d’échantillonnage et (v) le biais spatial ont été étudiés. La répétabilité du protocole a été évaluée à l’aide de trois réplications techniques indépendantes, avec un écart-type de 0,03 entre les essais. Les taux de croissance maximaux de Bacillus mycoides et de Paenibacillus tundrae ont été déterminés à 0,99 h−1 ±± 0,03 h−1 et 0,85 h−1 ± 0,025 h−1, respectivement. Ces bactéries sont plus difficiles à surveiller optiquement en raison de leur affinité à s’agglutiner. Cette étude démontre l’importance cruciale de la taille de l’inoculation, de la correction de la longueur du trajet, du réchauffement des couvercles, des intervalles de temps d’échantillonnage et du biais spatial des plaques de puits pour obtenir des données fiables, précises et reproductibles sur les lecteurs de microplaques. Le protocole développé et ses étapes de vérification peuvent être étendus à d’autres méthodes utilisant des lecteurs de microplaques et des protocoles à haut débit, réduisant ainsi les erreurs innées des chercheurs et les coûts des matériaux.
Introduction
L’intérêt croissant pour la manipulation multi-omique, y compris les études du mécanisme et du métabolisme des bactéries, souligne l’importance des méthodes automatisées et à haut débit telles que l’enregistrement des données de croissance 1,2. Les données de croissance comprenant des paramètres cinétiques, tels que les taux de croissance maximaux, peuvent aider à caractériser les réponses bactériennes à différentes conditions physiques, chimiques et antibactériennes. Les données sur le taux de croissance sont une variable de réponse standard utilisée pour découvrir des liens potentiels entre le génotype et les phénotypes1 ou pour indiquer l’innocuité microbienne et la durée de conservation des produits alimentaires 3,4. Des techniques telles que l’évolution adaptative en laboratoire 5,6,7, le criblage à l’échelle du génome, certains tests chimiques8 et divers criblages génétiques précoces9 reposent sur les taux de croissance pour évaluer les résultats.
Les mesures de densité optique (DO) des cultures bactériennes sont une méthode microbiologique standard pour surveiller la croissance bactérienne. Les mesures de DO sont souvent enregistrées à une longueur d’onde de 600 nm, en s’appuyant sur la diffusion de la lumière et la densité cellulaire10,11. La loi de Beer-Lambert explique la dépendance des valeurs de DO vis-à-vis de la concentration (densité cellulaire, nombre de cellules), de la longueur du trajet et du coefficient d’absorptivité. La géométrie et le système optique d’un spectrophotomètre influencent les lectures de DO11. Les méthodes classiques de mesure de la DO peuvent être très chronophages et laborieuses, et les données peuvent comporter diverses erreurs humaines. Dans ce protocole, un lecteur de microplaques est utilisé pour réduire le temps d’analyse12,13 et le risque de contamination biologique. L’analyse à haut débit à l’aide de lecteurs de microplaques est largement appliquée dans différents domaines de la microbiologie, tels que le criblage des bactéries productrices de biofilms14,15, l’inhibition de la croissance bactérienne16, la croissance des cellules de levure17, la détermination de la sensibilité aux antifongiques18 et le dépistage de la toxicité des nanomatériaux19.
Quelques chercheurs ont publié des protocoles de taux de croissance bactérienne à l’aide d’un lecteur de microplaques 12,20,21. Cependant, il n’a pas été entièrement établi qu’il existe un protocole exhaustif permettant d’examiner la fiabilité des données recueillies. Il est rapporté que des facteurs tels que le type d’espèce 22,23,24 et les rubans d’étanchéité ont un impact sur la répétabilité en raison de l’insuffisance du transfert d’oxygène dans une plaque à 96 puits25,26. Delaney et al. ont signalé de grands amas de Methylorubrum extorquens (souche de type sauvage) dans le milieu de croissance lors de l’utilisation d’un lecteur de microplaques, ce qui a provoqué des données de croissance extrêmement bruyantes24. Le problème a été résolu en supprimant les gènes associés à la production de biofilm24. En raison de la sécrétion de substances polymères extracellulaires, les bactéries productrices de biofilm ont une plus grande affinité pour s’unir et créer des amas de cellules. Par conséquent, il est plus difficile de surveiller leur croissance à l’aide de techniques de diffusion de la lumière (p. ex. spectrophotomètres et lecteurs de microplaques).
Ce protocole vise à établir des étapes permettant d’obtenir des données reproductibles dans une méthode à haut débit à l’aide d’un lecteur de microplaques. Bacillus mycoides et Paenibacillus tundrae ont été utilisés en raison de leur croissance rapide et de leur capacité à produire des biofilms, qui sont traditionnellement difficiles dans les approches manuelles et automatisées. Des facteurs tels que (i) la correction de la longueur du trajet, (ii) la condensation sur le couvercle, (iii) la taille de l’inoculum, (iv) l’intervalle de temps d’échantillonnage et (v) le biais spatial ont été étudiés pour évaluer la fiabilité et la reproductibilité des données. Ce protocole présente les étapes à suivre avec précision pour surveiller la croissance bactérienne et mesurer des taux de croissance spécifiques à l’aide d’un lecteur de microplaques.
Protocole
REMARQUE : Toutes les étapes de ce protocole doivent être suivies dans des conditions stériles (c.-à-d. entre deux flammes ou une enceinte de sécurité biologique). Tous les matériaux et outils sont autoclavés pendant 20 min. Consultez la table des matériaux pour plus de détails sur tous les matériaux, équipements et logiciels utilisés dans ce protocole. Les mains gantées sont désinfectées, maintenues humides avec un désinfectant pour les mains ou une solution d’alcool à 70 % pendant au moins 1 min, et ne sont pas retirées de l’enceinte de sécurité par la suite. Sinon, la procédure de désinfection doit être répétée avant de réintroduire les mains dans l’enceinte de sécurité. ATTENTION : Assurez-vous que le désinfectant est complètement évaporé avant d’utiliser une flamme nue.
REMARQUE : Deux bactéries ont été isolées de la biofiltration de l’eau potable, comme expliqué précédemment27 pour leur capacité à produire un biofilm. Ils ont été identifiés par le séquençage complet de l’ARNr 16S et soumis au NCBI sous le nom de Bacillus mycoides (SAMN10518261) et Paenibacillus tundrae (SAMN10452279).
1. Préparation du stock bactérien dans le glycérol
- Pesez 3 g de poudre de bouillon de soja tryptique (TSB) et dissolvez-le dans 100 ml d’eau distillée. Autoclavez le bouillon pendant 20 min et laissez-le refroidir à température ambiante. Ajouter 50 ml de TSB dans une fiole de 50 ml.
- Transférer les bactéries dans des plaques de gélose27,28. À l’aide d’une boucle, prélevez une colonie de bactéries dans la plaque de gélose. Ajoutez le contenu de la boucle dans la fiole en faisant tourner doucement la boucle. Incuber le ballon pendant la nuit à 30 °C ± 1 °C avec agitation orbitale à 150 tr/min.
REMARQUE : La température et l’agitation doivent être ajustées en fonction du type de déformation. - Préparez une autre fiole de 50 mL avec 47,5 mL de TSB. Transférer 2,5 mL de la culture de nuit dans la fiole. Incuber pendant 5-6 h pour atteindre OD600 = 0,6 0,05 (pour arriver à la phase mi-exponentielle).
- Transférez la culture dans un tube à centrifuger stérilisé de 50 ml. Centrifuger la culture à 2 200 × g pendant 10 min. Décantez doucement le bouillon pour éviter de perdre les cellules collectées au fond du tube.
- Ajouter 5 ml de solution saline tamponnée au phosphate (PBS) dans le même tube et centrifuger à 2 200 × g pendant 10 min. Répétez cette étape 3 fois.
- Ajustez la DO600 des cellules à 0,6 0,05 à l’aide d’une pipette pour titrer une quantité adéquate de solution PBS. Enregistrez avec précision la quantité supplémentaire de PBS pour une utilisation future.
- Titrez le glycérol jusqu’à la suspension pour atteindre 20 % v/v. Boucher le tube et le vortex pendant 15 s.
- Transférer 1,5 mL de la suspension dans des flacons cryogéniques de 2 mL. Placez les cryoflacons dans une boîte appropriée et conservez-les dans un congélateur à −80 °C.
2. Préparez la culture pendant la nuit.
- Laissez décongeler les cryoflacons contenant du stock bactérien à température ambiante pendant 30 minutes ou jusqu’à ce que le contenu soit liquide.
REMARQUE : Il est recommandé de ne pas accélérer le processus de décongélation. - Préparez un tube stérilisé avec 10 ml de TSB. Transférez 50 μL de la suspension d’origine dans le tube. Incuber le tube pendant la nuit (16-18 h) à 30 °C ± 1 °C avec agitation orbitale à 150 tr/min.
3. Préparez l’Inoculum.
- Préparez un tube avec 10 ml de TSB stérilisé. Transférez 500 μL de la culture de nuit dans le tube. Incuber pendant 6 à 8 h pour atteindre OD600 = 0,6 0,05 (ou arriver à la phase mi-exponentielle).
- Transférez la culture dans un tube à centrifuger de 15 ml. Centrifugez le tube à 2 200 × g pendant 10 min. Décantez doucement le bouillon.
- Ajouter 5 ml de PBS et centrifuger à 2 200 × g pendant 10 min. Répétez cette étape 3 fois.
- Ajuster la DO600 des cellules à 0,6 0,05 en titrant la solution de PBS à l’aide d’une pipette. Enregistrez avec précision la quantité supplémentaire de PBS pour une utilisation future.
4. Transfert du milieu de croissance vers le lecteur de microplaques
- Choisissez une plaque à puits transparente, stérile et à fond plat avec un couvercle (c’est-à-dire une plaque à 96 puits, une plaque à 6 puits, une plaque à 12 puits).
- Mesurez la correction de la longueur du trajet de la microplaque à l’aide d’eau DI avant d’exécuter le protocole, comme expliqué à l’étape 1.10.
- Dessinez une table de référence basée sur la plaque de puits sélectionnée pour spécifier les positions des échantillons avant le chargement. Par exemple, dessinez une table de 8 x 12 pour une plaque de 96 puits.
REMARQUE : Le tableau de référence permet d’éviter toute confusion lors des étapes d’incubation et de collecte des résultats. - Placez les articles suivants dans une enceinte de biosécurité : une microplaque avec couvercle, une pipette de 1 000 μL et 200 μL, des pointes de pipette de 1 000 μL et 200 μL, plusieurs cryoflacons, un bécher de 25 mL, le milieu de croissance et l’inoculum.
REMARQUE : Les articles mentionnés doivent être transférés dans l’enceinte de biosécurité de manière aseptique. - Ajouter 10 ml de milieu de croissance dans le bécher. Transférez la quantité calculée d’inoculum dans le bécher avec le milieu de croissance.
REMARQUE : Les ratios d’inoculum dans ce protocole sont de 1 % et 5 %. Par exemple, pour obtenir un rapport d’inoculum de 5 % et de 1 %, ajoutez 500 μL et 100 μL de l’inoculum à 9,5 mL et 9,9 mL du milieu de croissance, respectivement. - Après avoir ajouté l’inoculum, secouez doucement le bécher pour obtenir une répartition uniforme. Distribuer 200 μL du milieu inoculé dans les puits désignés à l’aide du tableau de référence. Choisissez au moins trois puits pour agir comme témoins en ajoutant 200 μL du milieu de croissance (sans inoculum).
REMARQUE : Assurez-vous que les puits de contrôle sont clairement indiqués dans le tableau de référence. Si une croissance (contamination) est observée dans ces puits, l’expérience doit être répétée. - Distribuez 200 μL d’eau DI dans les puits de bordure où le taux d’évaporation est plus élevé. Placez doucement le couvercle avant de retirer la plaque à 96 puits de l’armoire de sécurité. Placez soigneusement la plaque à 96 puits dans le lecteur de plaques, en veillant à ce qu’il n’y ait pas de mouvements brusques.
5. Paramètres du lecteur de microplaques
- Allumez le lecteur de microplaques, puis utilisez le logiciel intégré pour créer le protocole personnalisé. Dans le Gestionnaire des tâches, ouvrez le protocole et choisissez Créer un nouveau | Protocole standard. Ouvrez Procédure et ajustez les paramètres comme suit :
- Réglez la température sur 30 °C (ou les valeurs ciblées) à l’aide de la fonction Régler la température.
- Cliquez sur Incubator On , puis réglez le dégradé sur 1 °C.
REMARQUE : Pour éviter des données peu fiables dues à de la condensation sur le couvercle, assurez-vous que le gradient est réglé sur 1 °C. - Choisissez l’option de préchauffage , qui assure une distribution uniforme de la chaleur le long de la plaque de puits.
- Ouvrez Start Kinetic et choisissez la durée d’incubation souhaitée à l’aide de la durée de fonctionnement (par exemple, 12:00:00 ou 24:00:00, etc.) et de l’intervalle entre les lectures pour 00:30:00 (représente 30 min) ou 01:00:00 (représente 1 h).
REMARQUE : La lecture de chaque plaque de 96 puits prend environ 1 min. - Réglez les paramètres de secousse comme suit : ouvrez Shake ; réglez le mode Shake sur Linéaire ; cliquez sur Agitation continue ; et ajustez la fréquence à 567 cpm (ou n’importe quelle valeur souhaitée).
- Réglez les paramètres de lecture OD comme suit : cliquez sur Lecture | Absorbance ; sélectionnez Endpoint/Kinetic et Monochromators ; régler la longueur d’onde à 600 nm ; et cliquez sur Valider | Save pour l’enregistrer en tant que nouveau protocole avec le nom et la date appropriés.
6. Enregistrement des données de croissance
- Ouvrir le Gestionnaire des tâches | Lisez maintenant.
- Choisissez le protocole enregistré, puis cliquez sur OK pour commencer le protocole.
- Enregistrez les relevés pour l’analyse des données.
7. Analyse des données
- Transférez les résultats OD600 (données de croissance) sous forme de tableur. Dans la section des résultats , cliquez sur l’option d’image de chaque puits pour observer les courbes de croissance finales.
- Ouvrez le fichier de la feuille de calcul et réorganisez les données enregistrées en fonction de la table de référence. Soustrayez la valeur moyenne des lectures à blanc des autres lectures.
REMARQUE : Les blancs sont les lectures OD au temps zéro. Dans ce protocole, les valeurs de DO600 sont prises en compte pour le calcul. La densité cellulaire peut être utilisée si la courbe d’étalonnage pour OD600 en fonction du nombre de cellules est disponible.
8. Détermination du taux de croissance
- Utilisez les données transférées dans la feuille de calcul créée à l’étape 1.6. Utilisez la méthode décrite ci-dessous et l’équation (1) pour évaluer le taux de croissance.
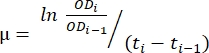 (1)
(1)
où μ est le taux de croissance, ODi est la DO600 à chaque point temporel, ODi-1 est la valeur initiale OD600 au dernier point temporel, ti et ti-1 sont les différences de temps entre les deux points mentionnés (par exemple, 0,5 pour des intervalles de temps de 30 minutes, 1 pour des intervalles de temps de 1 h). - Calculez la moyenne et l’écart-type de cinq taux de croissance successifs à l’aide du logiciel d’analyse de données souhaité. Notez que la moyenne la plus élevée avec l’écart-type le plus faible est le taux de croissance maximal (μm) 12.
9. Déterminer le biais spatial
REMARQUE : Les lecteurs de microplaques et les plaques introduisent un biais dans les résultats. Il est crucial d’évaluer les biais spatiaux des plaques utilisées dans un lecteur de microplaques spécifique afin de garantir la fiabilité et la reproductibilité des résultats. Pour y parvenir, procédez comme suit :
- Répétez les étapes 1.2 à 1.6.
- Enregistrez les données et analysez-les conformément aux étapes 1.7 et 1.8 pour calculer le taux de croissance de chaque puits.
- Créez une carte thermique pour visualiser les changements de taux de croissance dans la plaque à 96 puits.
- Effectuer l’analyse statistique (voir les tableaux supplémentaires), en s’assurant qu’il n’y a pas de différences significatives entre les puits.
REMARQUE : Il est essentiel d’utiliser le même type de bactéries et la même taille d’inoculum dans tous les puits.
10. Validation de la lecture du diamètre extérieur et facteur de correction de la longueur du trajet
REMARQUE : La détermination du facteur de correction de la longueur du chemin est cruciale pour valider les données et garantir la fiabilité et la validité sur différents appareils.
- Utilisez les étapes 1.2 et 1.3 pour préparer l’inoculum de nuit de B. mycoides.
- Préparez trois flacons stérilisés de 50 ml et ajoutez 49,5 ml de BST frais dans chaque flacon. Ajouter 500 μL d’inoculum dans chaque flacon d’une enceinte de sécurité de manière aseptique. Ajouter 50 mL de BST non inoculé dans une fiole stérilisée, qui servira de témoin.
- Incuber les flacons à 30 °C et 150 tr/min. Toutes les 30 min, prélever des échantillons de chaque fiole de manière aseptique et lire et enregistrer la DO600 dans le spectrophotomètre.
- Ajouter 9,9 ml de TSB dans un bécher de 25 ml. Ajouter 100 μL d’inoculum dans le bécher et agiter doucement.
- Ajoutez 200 μL du mélange dans le bécher dans les six puits d’une colonne sélectionnée dans une plaque de 96 puits. Ajouter 200 μL de BST non inoculé dans trois puits à titre de témoin. Remplissez les puits de bord avec 200 μL d’eau DI.
- Placez le couvercle et transférez délicatement la plaque à 96 puits dans le lecteur de microplaques. Suivez les étapes 1.5 et 1.6 pour enregistrer les données de croissance.
- Comparez l’OD600 du lecteur de microplaques à l’OD600 du spectrophotomètre. Notez que la pente est le décalage entre les deux ensembles de lectures, appelé facteur de correction de la longueur du trajet.
REMARQUE : L’incubation de l’incubateur et du lecteur de microplaques doit être effectuée simultanément le même jour.
Résultats
Validation de la lecture OD et facteur de correction de la longueur du trajet
Des échantillons fractionnés de cultures de B. mycoides ont été prélevés à différents moments et mesurés à l’aide du lecteur de microplaques et du spectrophotomètre (figure 1A). Cette étape a été prise pour valider les résultats sur différents appareils. Les données de l’OD600 étaient corrélées, mais ne correspondaient...
Discussion
Les lecteurs de microplaques permettent d’obtenir des taux de croissance constants et reproductibles. Cette technologie minimise l’erreur humaine et permet un échantillonnage à haut débit. La faible quantité de culture requise par échantillon fait de cette approche une alternative attrayante et peu coûteuse au comptage des cellules à l’aide de flacons ou de tubes à essai. Les lecteurs de microplaques permettent d’obtenir une grande taille d’échantillon, ce qui augmente...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Ces travaux ont été financés par la Chaire de recherche industrielle sur la qualité et le traitement de l’eau du Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG) et de la Chaire de recherche industrielle sur l’eau d’Halifax (subvention n°. IRCPJ 349838-16). L’équipe des auteurs tient également à remercier Anita Taylor pour son aide dans la révision de cet article.
matériels
| Name | Company | Catalog Number | Comments |
| Centrifuge | Eppendorf | 5810 R | |
| Centrifuge tubes - 15 mL | ThermoFisher- Scientific | 339650 | Sterile |
| Centriguge tubes - 50 mL | ThermoFisher- Scientific | 339652 | Sterile |
| Disposable inoculating loop , 10 µL | Cole-Parmer | UZ-06231-08 | Sterile |
| Erlenmeyer flasks - 250 mL | Cole-Parmer | UZ-34502-59 | Glass |
| Isopropanol | ThermoFisher- Scientific | 396982500 | ≥99.0 |
| Phosphate Buffer Saline | Sigma-Aldrich | P4417 | |
| Pipett tips 1,000 µL | ThermoFisher- Scientific | UZ-25001-76 | |
| Pipett tips 10 mL | ThermoFisher- Scientific | UZ-25001-83 | |
| Pipett tips 200 µL | ThermoFisher- Scientific | UZ-25001-85 | |
| Pipett tips 5 mL | ThermoFisher- Scientific | UZ-25001-80 | |
| Pipettor 1,000 µL | Cole-Parmer | UZ-07909-11 | |
| Pipettor 10 mL | Cole-Parmer | UZ-07909-15 | |
| Pipettor 200 µL | Cole-Parmer | UZ-07909-09 | |
| Pipettor 5 mL | Cole-Parmer | UZ-07859-30 | |
| Tryptic Soy Broth | Millipore | 22091 | Suitable for microbiology |
Références
- Reuß, D. R., et al. Large-scale reduction of the Bacillus subtilis genome: Consequences for the transcriptional network, resource allocation, and metabolism. Genome Research. 27 (2), 289-299 (2017).
- Sparkes, A., et al. Towards Robot Scientists for autonomous scientific discovery. Automated Experimentation. 2 (1), (2010).
- Zwietering, M. H., Jongenburger, I., Rombouts, F. M., van t Riet, K. Modeling of the bacterial growth curve. Applied and Environmental Microbiology. 56 (6), 1875-1881 (1990).
- Pla, M., Oltra, S., Esteban, M., Andreu, S., Palop, A. Comparison of primary models to predict microbial growth by the plate count and absorbance methods. BioMed Research International. 2015 (6), 1-14 (2015).
- Choe, D., et al. Adaptive laboratory evolution of a genome-reduced Escherichia coli. Nature Communications. 10 (1), 935 (2019).
- Dykhuizen, D. E., Dean, A. M. Enzyme activity and fitness: Evolution in solution. Trends in Ecology and Evolution. 5 (8), 257-262 (1990).
- McDonald, M. J. Microbial experimental evolution - A proving ground for evolutionary theory and a tool for discovery. EMBO Reports. 20 (8), 1-14 (2019).
- Lum, P. Y., et al. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116 (1), 121-137 (2004).
- Cagnon, C., et al. Development of a forward genetic screen to isolate oil mutants in the green microalga Chlamydomonas reinhardtii. Biotechnology for Biofuels. 6 (1), 178 (2013).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Scientific Reports. 6 (1), 38828 (2016).
- Matlock, B. C., Beringer, R. W., Ash, D. L., Page, A. F., Allen, M. W. Differences in Bacterial Optical Density Measurements between Spectrophotometers. Technical Note. ThermoScientific. , (2011).
- Hall, B. G., Acar, H., Nandipati, A., Barlow, M. Growth rates made easy. Molecular Biology and Evolution. 31 (1), 232-238 (2014).
- Rolfe, M. D., et al. Lag phase is a distinct growth phase that prepares bacteria for exponential growth and involves transient metal accumulation. Journal of Bacteriology. 194 (3), 686-701 (2012).
- Djordjevic, D., Wiedmann, M., Mclandsborough, L. A. Microtiter plate assay for assessment of Listeria monocytogenes biofilm formation. Applied and Environmental Microbiology. 68 (6), 2950-2958 (2002).
- O'Toole, G. A. Microtiter dish biofilm formation assay. Journal of Visualized Experiments. (47), e2437 (2011).
- Campbell, J. High-throughput assessment of bacterial growth inhibition by optical density measurements. Current Protocols in Chemical Biology. 3 (3), 1-20 (2012).
- Toussaint, M., Conconi, A. High-throughput and sensitive assay to measure yeast cell growth: A bench protocol for testing genotoxic agents. Nature Protocols. 1 (4), 1922-1928 (2006).
- Goughenour, K. D., Balada-Llasat, J. -. M., Rappleye, C. A. Quantitative microplate-based growth assay for determination of antifungal susceptibility of Histoplasma capsulatum yeasts. Journal of Clinical Microbiology. 53 (10), 3286-3295 (2015).
- Qiu, T. A., et al. Growth-based bacterial viability assay for interference-free and high-throughput toxicity screening of nanomaterials. Analytical Chemistry. 89 (3), 2057-2064 (2017).
- Kurokawa, M., Precise Ying, B. -. W. Precise, high-throughput analysis of bacterial growth. Journal of Visualized Experiments. (127), e56197 (2017).
- Bredit, F., Romick, T. L., Fleming, H. P. A rapid method for determination of bacterial growth kinetics. Journal of Rapid Methods and Automation in Microbiology. 3, 59-68 (1994).
- Delaney, N. F., et al. Development of an optimized medium, strain and high-throughput culturing methods for Methylobacterium extorquens. PLOS ONE. 8 (4), 62957 (2013).
- Haire, T. C., et al. Robust microplate-based methods for culturing and in vivo phenotypic screening of Chlamydomonas reinhardtii. Frontiers in Plant Science. 9, 235 (2018).
- McBirney, S. E., Trinh, K., Wong-Beringer, A., Armani, A. M. Wavelength-normalized spectroscopic analysis of Staphylococcus aureus and Pseudomonas aeruginosa growth rates. Biomedical Optics Express. 7 (10), 4034 (2016).
- Sieben, M., Giese, H., Grosch, J. H., Kauffmann, K., Büchs, J. Permeability of currently available microtiter plate sealing tapes fail to fulfil the requirements for aerobic microbial cultivation. Biotechnology Journal. 11 (12), 1525-1538 (2016).
- Zimmermann, H. F., John, G. T., Trauthwein, H., Dingerdissen, U., Huthmacher, K. Rapid evaluation of oxygen and water permeation through microplate sealing tapes. Biotechnology Progress. 19 (3), 1061-1063 (2003).
- Abkar, L., Gagnon, G. A. Biological responses to P-limitation in indigenous bacteria isolated from drinking water. AWWA Water Science. 3 (5), 1248 (2021).
- Sanders, E. R. Aseptic laboratory techniques: Plating methods. Journal of Visualized Experiments. (63), e3064 (2012).
- Hart, S. F. M., Skelding, D., Waite, A. J., Burton, J. C., Shou, W. High-throughput quantification of microbial birth and death dynamics using fluorescence microscopy. Quantitative Biology. 7 (1), 69-81 (2019).
- Periago, P. M., Abee, T., Wouters, J. A. Analysis of the heat-adaptive response of psychrotrophic Bacillus weihenstephanensis. International Journal of Food Microbiology. 79 (1-2), 17-26 (2002).
- Mira, P., Barlow, M., Meza, J. C., Hall, B. G. Statistical package for growth rates made easy. Molecular Biology and Evolution. 34 (12), 3303-3309 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon