需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用修饰的合成寡核苷酸检测核酸代谢酶
摘要
在这里,使用连接酶、核酸酶和聚合酶的实例提出了一种检测核酸代谢酶的方案。该分析利用荧光标记和未标记的寡核苷酸,这些寡核苷酸可以组合形成模拟 RNA 和/或 DNA 损伤或通路中间体的双链体,从而可以表征酶的行为。
摘要
商业供应商提供的一系列修饰合成寡核苷酸使开发复杂的分析方法来表征核酸代谢酶的不同特性,这些分析可以在任何标准分子生物学实验室中运行。荧光标记的使用使研究人员可以使用标准 PAGE 电泳设备和荧光成像仪使用这些方法,而无需使用放射性材料或需要专为储存和制备放射性材料而设计的实验室,即热实验室。可选添加标准修饰(如磷酸化)可以简化检测设置,而模拟 DNA 损伤或中间体的修饰核苷酸的特异性掺入可用于探测酶行为的特定方面。在这里,展示了使用市售合成寡核苷酸的酶询问 DNA 加工的几个方面的测定的设计和执行。这些包括连接酶连接或核酸酶降解不同 DNA 和 RNA 杂交结构的能力、DNA 连接酶对辅因子的差异使用以及酶 DNA 结合能力的评估。讨论了设计合成核苷酸底物时需要考虑的因素,并提供了一组可用于一系列核酸连接酶、聚合酶和核酸酶测定的基本寡核苷酸。
引言
所有生命形式都需要核酸加工酶来执行基本的生物过程,包括复制、转录和 DNA 修复。这些途径的关键酶功能是聚合酶(产生 RNA/DNA 分子的副本)、连接多核苷酸底物的连接酶、降解多核苷酸底物的核酸酶以及解旋酶和拓扑异构酶(熔化核酸双链体或改变其拓扑结构 1,2,3,4,5,6,7,8,9,10 .此外,其中许多酶为克隆、诊断和高通量测序等应用提供了必要的分子工具 11,12,13,14,15。
这些酶的功能特征、动力学和底物特异性可以使用寡核苷酸退火产生的标记 DNA/RNA 底物来确定。传统上,通过在 5' 链末端引入放射性标记 (32P) 来实现跟踪基材和产品,然后可以通过照相胶片或荧光粉成像系统进行检测16,17。虽然放射性标记底物具有提高实验灵敏度并且不会改变核苷酸化学性质的优势,但使用放射性同位素的潜在健康危害鼓励了非放射性核酸标记的发展,为 DNA 和 RNA 检测提供了更安全的替代方案 18,19,20.其中,荧光检测,包括直接荧光检测、时间分辨荧光和能量转移/荧光猝灭测定,是最通用的 21,22,23,24。广泛的荧光基团阵列可实现 DNA/RNA 底物的不同设计,每个寡核苷酸上具有独特的报告基因25。此外,与放射性同位素相比,荧光团的稳定性使用户能够产生和保存大量荧光标记的 DNA 底物19。这些荧光团标记的底物可以与目标蛋白以及金属和核苷酸辅因子的不同组合一起孵育,以分析结合和/或酶活性。使用带有凝胶成像系统的各种荧光团染料通道可以观察到结合或活性的可视化。由于使用这种技术只能看到荧光标记的寡核苷酸,因此标记的寡核苷酸大小的任何增加或减少都将很容易跟踪。凝胶也可以在之后使用核酸染色染料进行染色,以观察凝胶上存在的所有 DNA 条带。
多核酸连接酶是连接 DNA/RNA 片段的酶,通过在 DNA 的 5' 磷酸化 DNA 末端和 DNA 的 3' OH 之间形成磷酸二酯键来催化断裂的密封。根据其核苷酸底物要求,它们可分为两组。高度保守的 NAD 依赖性连接酶存在于所有细菌中26,而结构多样的 ATP 依赖性酶可以通过生命的所有结构域进行鉴定 8,27。DNA 连接酶在复制过程中的冈崎片段加工中起重要作用,并通过密封自发性缺口和修复后留下的缺口参与各种 DNA 修复途径,例如核苷酸和碱基切除修复 8,10。不同的 DNA 连接酶表现出不同的连接 DNA 断裂不同构象的能力,包括双链断裂、双链断裂、错配和间隙,以及 RNA 和 DNA 杂交体 28,29,30。通过将寡核苷酸与 5' 磷酸盐退火,可以在核酸双链体中生成并列的 5' 和 3' 末端,从而组装各种可连接的底物 31,32,33。最常见的分析方法是通过终点测定形式的尿素 PAGE 进行分离;然而,最近的创新包括使用毛细管凝胶电泳,可实现高通量34、质谱分析35,以及均相分子信标分析,可实现时间分辨监测36。
连接反应的第一步是连接酶被三磷酸腺苷 (ATP) 或烟酰胺腺嘌呤二核苷酸 (NAD) 使连接酶腺苷酸化,从而产生共价酶中间体。反应的第二步是核酸底物在缺口位点 5' 端的腺苷酸化,然后连接核酸缺口链。许多在 大肠杆菌 中重组表达的连接酶以腺苷酸化形式纯化,因此能够在不添加核苷酸辅因子的情况下成功连接核酸。这使得很难确定它们连接核酸需要什么特定类型的核苷酸辅因子。除了描述评估 DNA 连接酶活性的测定外,还提出了一种通过使用未标记底物对酶进行去腺苷酸化来可靠地确定辅因子使用的方法。
核酸酶是一大类多样化的 DNA/RNA 修饰酶和催化 RNA,可裂解核酸之间的磷酸二酯键37。核酸酶的功能是 DNA 复制、修复和 RNA 加工所必需的,可以根据它们对 DNA、RNA 或两者的糖特异性进行分类。核酸内切酶水解 DNA/RNA 链内的磷酸二酯键,而核酸外切酶一次水解 DNA/RNA 链从 DNA 的 3' 或 5' 末端水解一个核苷酸,并且可以从 DNA 的 3' 到 5' 或 5' 到 3' 末端进行水解38。
虽然许多核酸酶蛋白是非特异性的,可能涉及多个过程,但其他核酸酶蛋白对特定序列或 DNA 损伤具有高度特异性 6,39,40。序列特异性核酸酶用于广泛的生物技术应用,例如克隆、诱变和基因组编辑。这些应用中常用的核酸酶是限制性核酸酶41、锌指核酸酶42、转录激活因子样效应核酸酶,以及最近的 RNA 引导的工程化 CRISPR 核酸酶43。最近已鉴定出损伤特异性核酸酶,例如 EndoMS 核酸酶,它通过其错配特异性 RecB 样核酸酶结构域对 DNA 中的错配具有特异性 5,44。核酸酶活性测定过去是作为放射性标记底物的不连续测定进行的;然而,除了其他缺点外,这些缺点还不允许识别被核酸酶蛋白切割的位点,而使用荧光标记的底物时,这是可能的45,46。最近,已经开发了连续核酸酶检测,其工作原理是使用与不同状态的 DNA 相互作用的不同 DNA 染料;例如,与 dsDNA 相互作用时发出的荧光信号高于未结合状态,或与短 RNA 特异性结合47。其他连续核酸酶检测使用 DNA 发夹,5' 端有荧光团基团,3' 端有淬灭基团,因此荧光会随着荧光基团和淬灭基团的分离而降解寡核苷酸48。虽然这些分析允许表征 DNA 降解蛋白质的动力学,但它们需要事先了解酶的功能和底物,并且也仅限于改变 DNA 构象以引起染料结合差异的酶。因此,分离单个核酸酶产物的终点检测仍然需要深入了解由蛋白质活性引起的 DNA 修饰。
这里提出了设计荧光标记的 DNA/RNA 寡核苷酸的详细程序,这些寡核苷酸可以混合和匹配以产生用于测试新型核酸酶、聚合酶和连接酶活性的底物。这组基本寡核苷酸序列的验证简化了实验设计,并有助于经济地分析各种酶功能,而无需购买大量定制底物。提供了使用这些底物运行标准 DNA 加工酶测定的详细程序,使用 DNA 连接酶活性和修饰的示例,描述了用于测定和分析核酸酶和聚合酶的修饰。此外,给出了一种用于高精度测定 DNA 连接酶辅因子特异性的改良测定法,并使用双标记探针来评估多组分连接的组装。最后,讨论了对基本检测形式的修改,使其可用于通过电泳迁移率变化测定 (EMSA) 确定蛋白质-DNA 与相同底物的相互作用。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 寡核苷酸的设计与购买
注:设计单链寡核苷酸以组装并退火成所需的双链体。双链体中的一条或多条链必须带有荧光部分,用于追踪目标酶的寡核苷酸加工。 表 1 提供了一组可以组装用于一系列活动的单链序列的基本集。
- 如下所述,掺入目标酶所需的特定修饰。
- 对于 DNA 连接酶底物(图 1):从三个寡核苷酸中组装最简单的底物:一条 5' 磷酸化供体链 (NL2)、一条 5' FAM 标记的受体链 (NL1) 和一个桥接两者的互补体链 (NL3)。
- 确保在步骤 2 中组装底物预混液之前,提供可连接缺口 5' 末端的链被磷酸化。将其作为 NL2 的修饰订购(如 表 1 所示),或在重悬寡核苷酸后使用 T4 多核苷酸激酶的酶促磷酸化。
- 包括 NL6 和 NL8 的 5'-末端磷酸化,它们包含 图 1A 中描述的双链断裂的互补体(NL6/NL7 和 NL8/NL9),因为它与限制性核酸内切酶产生的天然底物最相似。使用双标记底物确定多部分组装体的相对连接范围(参见步骤 6)。
- 改变补体链以产生错配 (NL10) 和间隙 (NL11)。
注意: 图 1A 描述了简单缺口基板的变化。可以使用其他序列通过改变带下划线的位置来产生更大范围的不匹配或更长的间隙。 - 用 DNA 寡核苷酸代替 RNA 寡核苷酸。
注意: 图 1B 描述了简单缺口基材的变化。通过此处给出的基集的额外组合可以产生更广泛的 DNA/RNA 双链体,例如,包含 RNA 和 DNA 的双链断裂。下面的步骤 6 中给出了这种变化的一个示例,其中使用了双标签策略。
- 对于 DNA 聚合酶底物:组装 表 1 中列出的寡核苷酸 NL1 和 NL3,以得到简单的引物延伸测定。通过将修饰引入 NL1(引物)或 NL3(模板)链来研究聚合酶活性的其他方面。
- 在位置 20 之前将受损的碱基类似物掺入 NL3 寡核苷酸中,以确定绕过模板链上受损损伤的能力。
- 将受损的碱基类似物掺入 NL1 寡核苷酸的第 20 位,以确定延伸受损引物的能力。
- 在双链体中使用 RNL1 或 RNL3 来研究 RNA 引物的延伸或 RNA 模板的使用。
- 对于核酸酶底物(图 2):组装寡核苷酸以获得非详尽的双链和单链底物范围(图 2Ai)以及一系列翻瓣和张开的连接(图 2Aii)和受损的底物(图 2B)。
- 为了探测核糖核酸酶活性,用 RNL1、RNL2 和 RNL3 迭代替换 NL1、NL2 和 NL3。使用其他 RNA 版本的 HJ5 和 HJ6 进一步扩展该集合。
- 使用寡核苷酸 MD5、MD6 和 MD9,它们具有模拟氧化损伤的中心修饰、非碱基修复中间体或脱氨产物(图 2B)。底物将检测该位置的链断裂。用正交荧光团(如 TAMRA)标记补体 NL3 链以检测双链切割(参见步骤 6)。
- 使用补体的正交标记来检测探针(NL5 和 ND9)和补体(MD10 和 NL10)链上错配位点的双链切割。
- 对于 DNA 连接酶底物(图 1):从三个寡核苷酸中组装最简单的底物:一条 5' 磷酸化供体链 (NL2)、一条 5' FAM 标记的受体链 (NL1) 和一个桥接两者的互补体链 (NL3)。
- 从商业供应商处订购包含相关荧光基团和其他修饰的合成寡核苷酸。
注:100 nM 合成规模和合成后的 HPLC 纯化适用于所述分析。
2. 核酸双链体的组装和退火
- 寡核苷酸的重悬和稀释
- 开封前,在台式离心机中将 2 mL 试管中的冻干寡核苷酸全速离心 2-5 分钟,以确保核酸位于试管底部。
- 通过在 TE 缓冲液(10 mM 三(羟甲基)氨基甲烷 (Tris)、1 mM 乙二胺四乙酸 (EDTA)))中重悬寡核苷酸来制备 100 μM 的主储备液。通过全速重复温和涡旋和短暂离心,确保寡核苷酸彻底重悬。
- 用 TE 缓冲液稀释等分试样的原液,制备 10 μM 原液。用超纯水(MQ 水)稀释 10 μM 原液,以制备浓度为 0.5 μM、0.7 μM 或 2.5 μM 的工作原液,如 表 2 所示。
- 组装和退火反应预混液
- 使用工作储备液,使用 表 2 中提供的组合和 表 3 中给出的体积组成反应预混液。对于标准 DNA 连接酶测定和此处描述的大多数其他测定,最终缓冲液组成为 50 mM Tris pH 8.0、50 mM NaCl、10 mM 二硫苏糖醇 (DTT) 和 10 mM Mg 作为二价阳离子。
- 使用加热块或热循环仪在 95 °C 下加热 5 分钟,使寡核苷酸在 PCR 或微量离心管中退火。冷却至室温 30 分钟(体积 <1 mL)至 1 小时(体积 >1 mL)。对于较长的寡核苷酸 (>40 nt),使用热循环仪在 45 分钟内以 95 °C 至 25 °C 的下降斜坡进行缓慢冷却,或将含有退火混合物的试管漂浮在 1 L 沸水烧杯中,并冷却至室温过夜。
- 冷却至室温后,将核苷酸辅因子和其他热敏缓冲液组分添加到预混液中。通过添加酶将最终反应混合物直接用于测定(参见下面的步骤 3)或储存在 -20 °C 以备将来使用。
3. 标准分析设置
- 测定反应的组装和起始
- 将 22.5 μL 目标底物预混液与 2.5 μL DNA 连接酶或其他目标酶混合在 PCR 管中。一式两份或一式三份运行反应,尤其是在对结果进行定量时。
- 在检测样品中加入无蛋白对照(仅缓冲液)。如果需要,此时不包括辅因子控制。
注:酶可以在 -20 °C 下储存在 50% v/v 甘油中,使其能够直接从溶液中移液。确保在添加前将含甘油的酶溶液充分混合,通过移液混合或轻轻涡旋。
- 立即将反应物转移到 25 °C 的 PCR 机器中并孵育 30 分钟。根据酶活性的最佳条件改变温度和持续时间。
- 加入 5 μL 上样染料(95% 甲酰胺、0.5 M 乙二胺四乙酸 (EDTA)、溴酚蓝)淬灭反应,并在 95 °C 下孵育 5 分钟。
4. 分析结果
- 如下所述制备 Tris-硼酸盐-EDTA (TBE) -尿素 PAGE 凝胶。
- 准备 20% 丙烯酰胺、7 M 尿素和 1x TBE 溶液的储备液。对于此处描述的寡核苷酸组,以 29:1 的比例使用丙烯酰胺/双溶液以获得最佳分离度。
- 对于一粒凝胶,将 10 mL 20% 丙烯酰胺和 7 M 尿素溶液与 100 μL APS (10%) 和 3 μL 四甲基乙二胺 (TMED) 混合,然后灌注到凝胶灌注器中。
- 凝胶凝固后,在 45 - 55 °C 的 TBE 尿素凝胶上运行样品。
- 将凝胶在 1x TBE 缓冲液中以 10 mA 每个凝胶预运行30分钟,并进行外部加热。
- 通过使用牧场移液管用 1x TBE 冲洗,去除凝胶孔中多余的尿素。
- 每个反应加载 10 μL,并在外部加热下以 10 mA 运行 1.0-1.5 小时。
- 使用所选荧光团的正确设置在成像仪上可视化凝胶。对于 FAM,请使用在 495/519 nm 处提供激发/发射的滤光片组,该滤光片组在大多数成像仪中作为预设存储。
- 使用带有成像仪的图像处理软件或外部程序(如 ImageJ49,50)量化产品和基材的条带强度,并使用公式计算产品的百分比
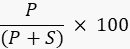
其中 P 是乘积带的积分值, S 是衬底带的积分面积。在 DNA 连接酶反应实例中,产物条带以 40 个核苷酸 (nt) 运行,底物条带以 20 nt 运行。
5. DNA 连接酶的去腺苷酸化以测试辅因子特异性
- 反应预混液的制备
- 如 表 4 所述,制备一组含有 FAM 标记的 NL1 寡核苷酸的主混合物。如 表 4 所述,准备第二组包含无 FAM 标记的 NL1 寡核苷酸。
- 分别将两个 DNA 双链体加热至 95 °C 5 分钟,然后在 25 °C 下冷却 30 分钟至 1 小时。 不要将核苷酸辅因子添加到任一预混液中。
- 脱腺苷酸化反应的组装和启动
- 通过将 10 μL 未标记的预混液与 2.5 μL 连接酶混合,为每种待测辅因子类型/浓度制备单个脱腺苷酸化反应。
- 准备额外的试管作为无辅因子对照和无蛋白质对照(添加 2.5 μL 缓冲液代替酶)。
- 在特定于酶最佳活性的温度下孵育反应 1-2 小时。如果酶仍处于腺苷酸化状态,则可以增加孵育时间。
- 使用辅因子进行连接反应。
- 将 10 μL 标记的主混液和 2.5 μL 所需的核苷酸辅因子(例如 ATP、NAD、ADP 或 GTP)直接添加到去腺苷酸化反应物中(0.1-1 mM 终浓度)。
- 向无核苷酸辅因子对照中加入 2.5 μL 反应缓冲液。
- 在与以前使用的相同时间和温度下孵育反应。如步骤 4 中所述淬灭和可视化。
6. 使用双标记底物进行夹板结扎或多部分组装
- 设计并购买具有荧光部分的寡核苷酸,该部分的激发/发射光谱与已使用的荧光团具有不同。
- 在此处描述的设置中,使用在 3' 端具有 5-羧基四甲基罗丹明 (TAMRA) 的 NL2 (TAMRA) 寡核苷酸(表 1)。
- 如下所述组装预混液。
- 混合步骤 2 中描述的反应组分,包括组装中使用的所有寡核苷酸的等摩尔比,以及缓冲液和二价阳离子。
- 通过在 95 °C 下加热 5 分钟并在 25 °C 下冷却 30 分钟 - 1 小时进行退火。添加辅因子和酶,并按照步骤 3 中的说明孵育。
- 按照步骤 4 中的说明,使用底物中荧光团对的适当通道对样品运行和成像。对于 FAM 和 TAMRA,这些是大多数成像仪上存在的荧光素 (FITC) 和四甲基罗丹明 (TRITC) 通道。
7. 通过电泳迁移率变化测定 (EMSA) 在天然凝胶上评估 DNA 结合
- 如下所述制备 10% 天然 TBE PAGE 凝胶。
- 将 2.5 mL 40% 丙烯酰胺、1 mL 10x TBE、100 μL 10% APS、3 μL TMED 和 6.5 mL MQ 水混合,并在凝胶铸造器中浇铸。
- 如下所述组装结合反应。
- 根据 表 5 组装 EMSA 底物,以便包括 EDTA (10 mM) 并省略金属离子。
- 将 20 μL EMSA 底物预混液与 5 μL 蛋白质混合在 PCR 管中。包括无蛋白对照样品。在 25 °C 下孵育 30 分钟。
- 如下所述,通过非变性电泳进行分析。
- 向样品中加入 5 μL 5x 天然上样染料(100 mM EDTA、0.25% 溴酚蓝、25% v/v 甘油和最多 1 mL 的 MQ 水)。
- 装上制备的凝胶,在 60 V 下运行 2-3 小时,通过水循环冷却,直到染料前沿高于凝胶末端几厘米。
- 如步骤 4 中所述可视化和分析凝胶。
Access restricted. Please log in or start a trial to view this content.
结果
通过 DNA 连接酶进行连接
当在尿素 PAGE 凝胶上观察时,DNA 连接酶活性将导致荧光标记的寡核苷酸的大小增加。在 表 2 中列出的 DNA 和 RNA 连接的底物的情况下,这相当于大小从 20 nt 增加到 40 nt(图 3A)。最佳酶活性可以通过改变条件来确定,例如温度、蛋白质浓度或孵育时间(图 3B)核苷酸辅因子、金属辅因子(
Access restricted. Please log in or start a trial to view this content.
讨论
协议中的关键步骤
寡核苷酸设计和购买:购买用于双链体形成的寡核苷酸时,必须考虑序列设计。建议在订购57 之前,使用寡核苷酸分析仪工具预测核苷酸序列的性质,例如 GC 含量、熔解温度、二级结构和二聚化电位。
核酸双链体的组装和退火:制备 RNA/RNA-DNA 双链体时,应注意使用不含 RNase 的试剂和水或添加 RNAse 抑制剂(只要所研究的酶?...
Access restricted. Please log in or start a trial to view this content.
披露声明
SEG 和 UR 是分销 R2D 连接酶的 ArcticZymes Technologies AS 的员工。AW、ER-S 和 RS 没有相互竞争的利益。
致谢
AW 得到了卢瑟福发现奖学金 (20-UOW-004) 的支持。RS 是新西兰 南极邮政奖学金 的获得者。SG 和 UR 感谢特罗姆瑟大学 - 挪威北极大学化学研究所提供的技术支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
参考文献
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。