È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Utilizzo di oligonucleotidi sintetici modificati per il dosaggio degli enzimi che metabolizzano gli acidi nucleici
In questo articolo
Riepilogo
Qui viene presentato un protocollo per il dosaggio degli enzimi metabolizzanti degli acidi nucleici, utilizzando esempi di enzimi ligasi, nucleasi e polimerasi. Il test utilizza oligonucleotidi marcati in fluorescenza e non marcati che possono essere combinati per formare duplex che imitano i danni all'RNA e/o al DNA o gli intermedi di percorso, consentendo la caratterizzazione del comportamento enzimatico.
Abstract
La disponibilità di una gamma di oligonucleotidi sintetici modificati da fornitori commerciali ha permesso lo sviluppo di saggi sofisticati per caratterizzare diverse proprietà degli enzimi che metabolizzano gli acidi nucleici che possono essere eseguiti in qualsiasi laboratorio di biologia molecolare standard. L'uso di etichette fluorescenti ha reso questi metodi accessibili ai ricercatori con apparecchiature standard per elettroforesi PAGE e un imager abilitato alla fluorescenza, senza utilizzare materiali radioattivi o richiedere un laboratorio progettato per lo stoccaggio e la preparazione di materiali radioattivi, ad esempio un Hot Lab. L'aggiunta opzionale di modifiche standard come la fosforilazione può semplificare la configurazione del saggio, mentre l'incorporazione specifica di nucleotidi modificati che imitano i danni al DNA o gli intermedi può essere utilizzata per sondare aspetti specifici del comportamento enzimatico. Qui vengono dimostrate la progettazione e l'esecuzione di saggi per interrogare diversi aspetti dell'elaborazione del DNA da parte di enzimi utilizzando oligonucleotidi sintetici disponibili in commercio. Questi includono la capacità delle ligasi di unirsi o delle nucleasi di degradare diverse strutture ibride di DNA e RNA, l'uso differenziale del cofattore da parte della DNA ligasi e la valutazione della capacità di legare il DNA degli enzimi. Vengono discussi i fattori da considerare nella progettazione di substrati nucleotidici sintetici e viene fornito un set di base di oligonucleotidi che possono essere utilizzati per una serie di saggi enzimatici di ligasi, polimerasi e nucleasi degli acidi nucleici.
Introduzione
Tutte le forme di vita richiedono enzimi di elaborazione dell'acido nucleico per eseguire processi biologici fondamentali, tra cui la replicazione, la trascrizione e la riparazione del DNA. Le principali funzionalità enzimatiche per queste vie sono le polimerasi, che generano copie di molecole di RNA/DNA, le ligasi che si uniscono ai substrati polinucleotidici, le nucleasi che le degradano, e le elicasi e le topoisomerasi, che fondono duplex di acidi nucleici o ne modificano la topologia 1,2,3,4,5,6,7,8,9,10 . Inoltre, molti di questi enzimi forniscono strumenti molecolari essenziali per applicazioni come il clonaggio, la diagnostica e il sequenziamento ad alto rendimento 11,12,13,14,15.
Le caratteristiche funzionali, la cinetica e le specificità del substrato di questi enzimi possono essere determinate utilizzando substrati di DNA/RNA marcati prodotti mediante ricottura di oligonucleotidi. Il tracciamento dei substrati e dei prodotti è stato tradizionalmente ottenuto introducendo un'etichetta radioattiva (32P) all'estremità del filamento 5', che può quindi essere rilevata da una pellicola fotografica o con un sistema di imaging al fosforo16,17. Mentre i substrati radiomarcati offrono il vantaggio di una maggiore sensibilità sperimentale e non alterano le proprietà chimiche di un nucleotide, i potenziali rischi per la salute derivanti dal lavoro con i radioisotopi hanno incoraggiato lo sviluppo di marcature di acidi nucleici non radioattivi per fornire un'alternativa più sicura per la rilevazione di DNA e RNA 18,19,20. Tra questi, il rilevamento della fluorescenza, incluso il rilevamento diretto della fluorescenza, la fluorescenza risolta nel tempo e i saggi di trasferimento di energia/estinzione della fluorescenza si distinguono come i più versatili 21,22,23,24. L'ampia gamma di fluorofori consente diversi design di substrati di DNA/RNA con reporter unici su ciascun oligonucleotide25. Inoltre, la stabilità dei fluorofori, rispetto ai radioisotopi, consente agli utenti di produrre e conservare quantità significative di substrati di DNA marcati in fluorescenza19. Questi substrati marcati con fluorofori possono essere incubati con la proteina di interesse, insieme a diverse combinazioni di cofattori metallici e nucleotidici, per analizzare il legame e/o l'attività enzimatica. La visualizzazione del legame o dell'attività può essere osservata utilizzando vari canali di coloranti fluorofori con un sistema di imaging su gel. Poiché con questa tecnica saranno visibili solo gli oligonucleotidi marcati in fluorescenza, qualsiasi aumento o diminuzione delle dimensioni dell'oligonucleotide marcato sarà facile da seguire. I gel possono anche essere colorati in seguito, con coloranti coloranti per acidi nucleici per visualizzare tutte le bande di DNA presenti sul gel.
Le ligasi degli acidi polinucleici sono enzimi che uniscono frammenti di DNA/RNA, catalizzando la sigillatura delle rotture mediante la formazione di un legame fosfodiestere tra i terminali del DNA fosforilato 5' e l'OH 3' del DNA. Possono essere divisi in due gruppi in base al loro requisito di substrato nucleotidico. Le ligasi NAD-dipendenti altamente conservate si trovano in tutti i batteri26, mentre gli enzimi ATP-dipendenti, strutturalmente diversi, possono essere identificati in tutti i domini della vita 8,27. Le DNA ligasi svolgono un ruolo importante nell'elaborazione dei frammenti di Okazaki durante la replicazione, oltre ad essere coinvolte in varie vie di riparazione del DNA, come la riparazione per escissione di nucleotidi e basi, attraverso la sigillatura di intaccature spontanee e intaccature che rimangono dopo la riparazione 8,10. Diverse DNA ligasi mostrano diverse capacità di unire diverse conformazioni di rotture del DNA, tra cui intaccature in un duplex, rotture a doppio filamento, disallineamenti e lacune, nonché ibridi di RNA e DNA 28,29,30. Una vasta gamma di substrati legabili può essere assemblata mediante ricottura di oligonucleotidi con un fosfato 5' per generare termini 5' e 3' giustapposti in un duplex di acido nucleico 31,32,33. Il metodo di analisi più comune è la risoluzione mediante urea PAGE in un formato di saggio end-point; Tuttavia, le recenti innovazioni hanno incluso l'uso dell'elettroforesi su gel capillare, che consente un'elevata produttività34, la profilazione spettrometrica di massa35, nonché un saggio beacon molecolare omogeneo, che consente il monitoraggio risolto nel tempo36.
Il primo passo in una reazione di legatura è l'adenilazione dell'enzima ligasi da parte dell'adenosina trifosfato (ATP) o della nicotinammide adenina dinucleotide (NAD), risultando in un intermedio enzimatico covalente. Il secondo passo della reazione è l'adenilazione del substrato dell'acido nucleico all'estremità 5' del sito di nick, che è seguita dalla legatura dei filamenti di nick dell'acido nucleico. Molti enzimi ligasi che sono espressi in modo ricombinante in E. coli sono purificati nella forma adenilata e, quindi, sono in grado di legare con successo gli acidi nucleici senza l'aggiunta di un cofattore nucleotidico. Ciò rende difficile determinare quale particolare tipo di cofattore nucleotidico sono necessari per la legatura degli acidi nucleici. Oltre a descrivere i saggi per valutare l'attività della DNA ligasi, viene presentato anche un metodo per determinare in modo affidabile l'uso del cofattore de-adenilando l'enzima utilizzando substrati non marcati.
Le nucleasi sono un gruppo ampio e diversificato di enzimi che modificano il DNA/RNA e RNA catalitici che scindono i legami fosfodiestere tra gli acidi nucleici37. Le funzionalità dell'enzima nucleasi sono necessarie nella replicazione, riparazione e processazione dell'RNA del DNA e possono essere classificate in base alla loro specificità zuccherina per DNA, RNA o entrambi. Le endonucleasi idrolizzano i legami fosfodiestere all'interno di un filamento di DNA/RNA, mentre le esonucleasi idrolizzano i filamenti di DNA/RNA un nucleotide alla volta dall'estremità 3' o 5' e possono farlo sia dall'estremità 3' a 5' che da 5' a 3' del DNA38.
Mentre molte proteine nucleasi sono aspecifiche e possono essere coinvolte in più processi, altre sono altamente specifiche per una particolare sequenza o danno al DNA 6,39,40. Le nucleasi sequenza-specifiche sono utilizzate in un'ampia gamma di applicazioni biotecnologiche, come la clonazione, la mutagenesi e l'editing del genoma. Le nucleasi popolari per queste applicazioni sono le nucleasi di restrizione41, le nucleasi a dita di zinco42, le nucleasi effettrici simili ad attivatori trascrizionali e, più recentemente, le nucleasi CRISPR ingegnerizzate guidate dall'RNA43. Recentemente sono state identificate nucleasi danno-specifiche, come la nucleasi EndoMS, che ha specificità per i mismatch nel DNA attraverso il suo dominio 5,44 della nucleasi RecB-like mismatch-specific. I saggi di attività nucleasica, storicamente, sono stati eseguiti come saggi discontinui con substrati radiomarcati; Tuttavia, oltre agli altri inconvenienti, questi non consentono l'identificazione del sito che viene tagliato da una proteina nucleasi, cosa possibile quando si utilizzano substrati marcati in fluorescenza 45,46. Più recentemente, sono stati sviluppati saggi di nucleasi continua che funzionano utilizzando diversi coloranti di DNA che interagiscono con il DNA in stati diversi; ad esempio, emettendo un segnale fluorescente più elevato quando interagisce con il dsDNA rispetto al suo stato non legato, o legandosi specificamente a RNA corti47. Altri saggi di nucleasi continua utilizzano forcine di DNA con un gruppo fluoroforo sul 5' e un quencher sull'estremità 3' in modo che la fluorescenza aumenti man mano che l'oligonucleotide viene degradato a causa di una separazione del fluoroforo e del quencher48. Sebbene questi saggi consentano di caratterizzare la cinetica delle proteine che degradano il DNA, richiedono una conoscenza preliminare della funzione e del substrato dell'enzima e sono anche limitati agli enzimi che modificano la conformazione del DNA per causare una differenza nel legame del colorante. Per questo motivo, i saggi endpoint che risolvono i singoli prodotti nucleasici sono ancora auspicabili per fornire informazioni sulle modifiche del DNA causate dall'attività proteica.
Qui, viene presentata una procedura dettagliata per la progettazione di oligonucleotidi DNA/RNA marcati in fluorescenza che possono essere miscelati e abbinati per generare substrati per testare l'attività di nuovi enzimi nucleasi, polimerasi e ligasi. La convalida di questo set di base di sequenze oligonucleotidiche semplifica la progettazione sperimentale e facilita la profilazione economica di un'ampia gamma di funzionalità enzimatiche senza la necessità di acquistare un gran numero di substrati su misura. Viene fornita una procedura dettagliata per l'esecuzione di un saggio enzimatico standard di elaborazione del DNA con questi substrati, utilizzando l'esempio dell'attività della DNA ligasi e vengono descritte le modifiche per il saggio e l'analisi degli enzimi nucleasi e polimerasi. Inoltre, viene fornito un test modificato per determinare la specificità del cofattore dell'enzima DNA ligasi con elevata precisione e vengono utilizzate sonde a doppia marcatura per valutare l'assemblaggio di legature multicomponente. Infine, vengono discusse le modifiche al formato del saggio di base per consentirne l'utilizzo per determinare le interazioni proteina-DNA con gli stessi substrati mediante il saggio di spostamento della mobilità elettroforetica (EMSA).
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Progettazione e acquisto di oligonucleotidi
NOTA: Progettare oligonucleotidi a singolo filamento da assemblare e ricuocere nei duplex desiderati. Uno o più filamenti in un duplex devono avere una porzione fluorescente per tracciare l'elaborazione degli oligonucleotidi da parte dell'enzima di interesse. Nella Tabella 1 viene fornito un set di base di sequenze a singolo filamento che possono essere assemblate per una vasta gamma di attività.
- Incorporare le modifiche specifiche necessarie per l'enzima di interesse come descritto di seguito.
- Per i substrati della DNA ligasi (Figura 1): assemblare il substrato più semplice da tre oligonucleotidi: un filamento donatore fosforilato 5' (NL2), un filamento accettore marcato con FAM 5' (NL1) e un complemento che collega i due (NL3).
- Assicurarsi che i fili che forniscono il termine 5' dell'intaccatura legabile siano fosforilati prima dell'assemblaggio della miscela master del substrato nel passaggio 2. Ordinalo come modifica su NL2 (come indicato nella Tabella 1) o usa la fosforilazione enzimatica con la polinucleotide chinasi T4 dopo aver risospeso gli oligonucleotidi.
- Include la fosforilazione 5'-terminale di NL6 e NL8, che comprendono il complemento di rotture a doppio filamento raffigurate nella Figura 1A (NL6/NL7 e NL8/NL9) in quanto questo assomiglia molto al substrato naturale prodotto da un'endonucleasi di restrizione. Utilizzare un substrato a doppia marcatura per determinare le estensioni relative della legatura per assemblaggi in più parti (vedere il passaggio 6).
- Modificare il filamento del complemento per produrre disallineamenti (NL10) e lacune (NL11).
NOTA: Le variazioni sul substrato intaccato semplice sono illustrate nella Figura 1A. È possibile utilizzare altre sequenze per produrre una gamma ancora più ampia di disallineamenti o spazi più lunghi variando la posizione sottolineata. - Sostituire gli oligonucleotidi del DNA con gli oligonucleotidi dell'RNA.
NOTA: Le variazioni sul substrato semplice intaccato sono illustrate nella Figura 1B. Una gamma più ampia di duplex DNA/RNA può essere generata da combinazioni aggiuntive del set di basi qui fornito per generare, ad esempio, rotture a doppio filamento contenenti sia RNA che DNA. Un esempio di questa variazione è fornito nel passaggio 6 di seguito, in cui viene utilizzata una strategia a doppia etichetta.
- Per i substrati della DNA polimerasi: assemblare gli oligonucleotidi NL1 e NL3 elencati nella Tabella 1 per fornire un semplice saggio di estensione del primer. Studiare ulteriori aspetti dell'attività della polimerasi introducendo modifiche nei filamenti NL1 (primer) o NL3 (modello).
- Incorporare analoghi di base danneggiati nell'oligonucleotide NL3 prima della posizione 20 per determinare la capacità di bypassare le lesioni danneggiate sul filamento stampo.
- Incorporare analoghi di base danneggiati nell'oligonucleotide NL1 in posizione 20 per determinare la capacità di estendere un primer danneggiato.
- Utilizzare RNL1 o RNL3 nel duplex per studiare l'estensione di un primer di RNA o l'uso di un modello di RNA.
- Per i substrati nucleasi (Figura 2): assemblare gli oligonucleotidi per fornire una gamma non esaustiva di substrati a doppio e singolo filamento (Figura 2Ai), nonché una gamma di giunzioni sbattute e allargate (Figura 2Aii) e substrati danneggiati (Figura 2B).
- Per sondare le attività della ribonucleasi, sostituire iterativamente NL1, NL2 e NL3 con RNL1, RNL2 e RNL3. Utilizzare versioni aggiuntive di RNA di HJ5 e HJ6 per espandere ulteriormente questo set.
- Utilizzare oligonucleotidi MD5, MD6 e MD9 che hanno una modifica posizionata centralmente che imita il danno ossidativo, un intermedio di riparazione abasico o un prodotto di deaminazione (Figura 2B). I substrati rileveranno la scissione del filamento in questa posizione. Etichettare il filamento NL3 del complemento con un fluoroforo ortogonale come TAMRA per rilevare la scissione del doppio filamento (vedere il passaggio 6).
- Utilizzare la marcatura ortogonale del complemento per rilevare il taglio a doppio filamento nei siti non corrispondenti sia sulla sonda (NL5 e ND9) che sui filamenti del complemento (MD10 e NL10).
- Per i substrati della DNA ligasi (Figura 1): assemblare il substrato più semplice da tre oligonucleotidi: un filamento donatore fosforilato 5' (NL2), un filamento accettore marcato con FAM 5' (NL1) e un complemento che collega i due (NL3).
- Ordina oligonucleotidi sintetici che incorporano fluorofori pertinenti e altre modifiche da un fornitore commerciale.
NOTA: Per i saggi descritti sono disponibili una scala di sintesi da 100 nM e la purificazione HPLC successiva alla sintesi.
2. Assemblaggio e ricottura di duplex di acidi nucleici
- Risospensione e diluizione di oligonucleotidi
- Prima dell'apertura, centrifugare gli oligonucleotidi liofilizzati nelle loro provette da 2 ml alla massima velocità in una centrifuga da banco per 2-5 minuti per assicurarsi che l'acido nucleico si trovi sul fondo della provetta.
- Preparare un master stock di 100 μM risospendendo gli oligonucleotidi nel tampone TE [10 mM di tris(idrossimetil)amminometano (Tris), 1 mM di acido etilendiamminotetraacetico (EDTA)). Assicurarsi che gli oligonucleotidi siano completamente risospesi mediante vortice delicato ripetuto e breve centrifugazione a piena velocità.
- Preparare un materiale da 10 μM diluendo un'aliquota di stock master con tampone TE. Diluire il brodo da 10 μM con acqua ultrapura (acqua MQ) per preparare materiali di lavoro con concentrazioni di 0,5 μM, 0,7 μM o 2,5 μM come da Tabella 2.
- Assemblaggio e ricottura delle miscele master di reazione
- Utilizzare le scorte di lavoro per la composizione delle miscele madri di reazione utilizzando le combinazioni fornite nella tabella 2 e i volumi indicati nella tabella 3. Per il test standard della DNA ligasi e la maggior parte degli altri test qui descritti, la composizione finale del tampone è 50 mM Tris pH 8.0, 50 mM NaCl, 10 mM Ditiotreitolo (DTT) con 10 mM Mg come catione bivalente.
- Ricuocere gli oligonucleotidi in una provetta per PCR o microcentrifuga riscaldandoli a 95 °C per 5 minuti utilizzando un blocco riscaldante o un termociclatore. Lasciare raffreddare a temperatura ambiente per 30 minuti (volumi <1 mL) a 1 h (volumi >1 mL). Per oligonucleotidi più lunghi (>40 nt), eseguire un raffreddamento più lento utilizzando un termociclatore con una rampa di discesa da 95 °C a 25 °C per 45 minuti, oppure far galleggiare il tubo contenente la miscela di ricottura in un becher da 1 litro di acqua bollente e lasciarlo raffreddare a temperatura ambiente per una notte.
- Aggiungere cofattori nucleotidici e altri componenti tampone sensibili al calore alla miscela master dopo il raffreddamento a temperatura ambiente. Utilizzare la miscela di reazione finale direttamente per il saggio mediante l'aggiunta di enzima (vedere la fase 3 di seguito) o conservare a -20 °C per un uso futuro.
3. Configurazione standard del saggio
- Assemblaggio e avvio della reazione di saggio
- Combinare 22,5 μl della miscela master del substrato di interesse con 2,5 μl della DNA ligasi o di un altro enzima di interesse in una provetta per PCR. Esegui le reazioni in doppio o triplice copia, soprattutto se i risultati saranno quantitati.
- Includere un controllo senza proteine (solo tampone) nei campioni di analisi. A questo punto non includere controlli di cofattori, se necessario.
NOTA: Gli enzimi possono essere conservati a -20 °C in glicerolo al 50% v/v, consentendo loro di essere pipettati direttamente dalla soluzione. Assicurarsi che le soluzioni enzimatiche con glicerolo siano ben miscelate prima dell'aggiunta, pipettando per miscelare o agitando delicatamente.
- Trasferire immediatamente le reazioni in una macchina per PCR a 25 °C e incubare per 30 minuti. Variare la temperatura e la durata a seconda delle condizioni ottimali per l'attività enzimatica.
- Estinguere le reazioni aggiungendo 5 μL di colorante di carico (95% di formammide, acido etilendiamminotetraacetico 0,5 M (EDTA), blu di bromofenolo) e incubare a 95 °C per 5 minuti.
4. Analisi dei risultati dei saggi
- Preparare i gel Tris-Borato-EDTA (TBE)-Urea PAGE come descritto di seguito.
- Preparare una scorta di acrilammide al 20%, 7 M di urea e 1x soluzione di TBE. Per il set di oligonucleotidi qui descritto, utilizzare la soluzione di acrilammide/bis in un rapporto 29:1 per una risoluzione ottimale.
- Per un gel, combinare 10 mL di acrilammide al 20% e una soluzione di urea 7 M con 100 μL di APS (10%) e 3 μL di tetrametiletilendiammina (TMED) e gettare in una rotella di gel.
- Dopo che il gel si è solidificato, eseguire i campioni sul gel di urea TBE a 45 - 55 °C.
- Pre-rotolare il gel in 1x tampone TBE per 30 minuti a 10 mA per gel con riscaldamento esterno.
- Rimuovere l'urea in eccesso nei pozzetti del gel sciacquando con 1x TBE utilizzando una pipetta da pascolo.
- Caricare 10 μL di ciascuna reazione e far funzionare a 10 mA per 1,0-1,5 ore con riscaldamento esterno.
- Visualizza il gel sull'imager con le impostazioni corrette per il fluoroforo scelto. Per la FAM, utilizzare un set di filtri che fornisca eccitazione/emissione a 495/519 nm, che viene memorizzato come preimpostato nella maggior parte degli imager.
- Quantificare l'intensità della banda del prodotto e del substrato utilizzando il software di elaborazione delle immagini con l'imager o un programma esterno come ImageJ49,50 e calcolare la percentuale del prodotto utilizzando la formula
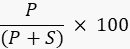
Dove P è il valore integrato della banda del prodotto e S è l'area integrata della banda del substrato. Nel caso della reazione della DNA ligasi, la banda del prodotto funziona a 40 nucleotidi (nt) e la banda del substrato a 20 nt.
5. De-adenilazione della DNA ligasi per testare la specificità del cofattore
- Preparazione di master mix di reazione
- Preparare una serie della miscela master contenente l'oligonucleotide NL1 marcato con FAM come descritto nella Tabella 4. Preparare un secondo set contenente l'oligonucleotide NL1 senza etichetta FAM, come descritto nella Tabella 4.
- Separatamente, riscaldare entrambi i duplex di DNA a 95 °C per 5 minuti e raffreddare per 30 minuti a 1 ora a 25 °C. Non aggiungere cofattore nucleotidico a nessuna delle due miscele master.
- Assemblaggio e inizio della reazione di de-dedenilazione
- Preparare una singola reazione di de-dedenilazione per ogni tipo/concentrazione di cofattore da testare combinando 10 μl della miscela master non marcata con 2,5 μl di enzima ligasi.
- Preparare provette aggiuntive come controllo senza cofattori e senza controllo proteico (2,5 μL di tampone aggiunti al posto dell'enzima).
- Incubare le reazioni a una temperatura specifica per l'attività ottimale dell'enzima, per 1-2 ore. Il tempo di incubazione può essere aumentato se l'enzima è ancora adenilato.
- Esegui la reazione di legatura con il cofattore.
- Aggiungere 10 μl della miscela master marcata e 2,5 μl dei cofattori nucleotidici desiderati (ad es. ATP, NAD, ADP o GTP) direttamente alla reazione dedeninilata (concentrazione finale di 0,1-1 mM).
- Aggiungere 2,5 μl di tampone di reazione al controllo del cofattore senza nucleotidi.
- Incubare le reazioni per lo stesso periodo di tempo e temperatura utilizzati in precedenza. Spegni e visualizza come descritto nel passaggio 4.
6. Utilizzo di substrati a doppia marcatura per la legatura splinted o l'assemblaggio in più parti
- Progettare e acquistare un oligonucleotide con una porzione fluorescente che abbia uno spettro di eccitazione/emissione diverso dal fluoroforo già utilizzato.
- Nella configurazione qui descritta, utilizzare l'oligonucleotide NL2 (TAMRA) con 5-carbossitetrametilrodamina (TAMRA) all'estremità 3' (Tabella 1).
- Assemblare la miscela master come descritto di seguito.
- Combinare i componenti della reazione descritti al punto 2, compresi i rapporti equimolari di tutti gli oligonucleotidi utilizzati nell'assemblaggio, nonché i cationi tampone e bivalenti.
- Ricottura mediante riscaldamento a 95 °C per 5 min e raffreddamento a 25 °C per 30 min - 1 h. Aggiungere il cofattore e l'enzima e incubare come descritto nel passaggio 3.
- Eseguire e visualizzare i campioni come descritto al punto 4 utilizzando i canali appropriati per la coppia di fluorofori nel substrato. Nel caso di FAM e TAMRA, si tratta dei canali della fluoresceina (FITC) e della tetrametilrodamina (TRITC) presenti sulla maggior parte degli imager.
7. Valutazione del legame del DNA mediante Electrophoretic Mobility Shift Assay (EMSA) su gel nativo
- Preparare un gel TBE PAGE nativo al 10% come descritto di seguito.
- Combinare 2,5 mL di acrilammide al 40%, 1 mL di 10x TBE, 100 μL di APS al 10%, 3 μL di TMED e 6,5 mL di acqua MQ e gettare in una rotella di gel.
- Assemblare la reazione di legame come descritto di seguito.
- Assemblare il substrato EMSA secondo la Tabella 5 in modo che l'EDTA (10 mM) sia incluso e gli ioni metallici siano omessi.
- Combinare 20 μl della master mix del substrato EMSA con 5 μl di proteina in una provetta per PCR. Includere un campione di controllo senza proteine. Incubare per 30 minuti a 25 °C.
- Analizza mediante elettroforesi nativa come descritto di seguito.
- Aggiungere ai campioni 5 μl di colorante da carica nativo 5x (100 mM EDTA, 0,25% blu bromofenolo, 25% v/v glicerolo e acqua MQ fino a 1 mL).
- Caricare sul gel preparato e far funzionare a 60 V per 2-3 ore con raffreddamento mediante circolazione dell'acqua fino a quando il fronte del colorante si trova alcuni cm sopra l'estremità del gel.
- Visualizza e analizza i gel come descritto nel passaggio 4.
Access restricted. Please log in or start a trial to view this content.
Risultati
Legatura mediante DNA ligasi
L'attività enzimatica della DNA ligasi si tradurrà in un aumento delle dimensioni dell'oligonucleotide marcato in fluorescenza quando visualizzato su un gel di urea PAGE. Nel caso dei substrati per la legatura del DNA e dell'RNA elencati nella Tabella 2, ciò corrisponde a un raddoppio delle dimensioni da 20 nt a 40 nt (Figura 3A). L'attività enzimatica ottimale può essere determinata modificando condizioni come la tempera...
Access restricted. Please log in or start a trial to view this content.
Discussione
Passaggi critici nel protocollo
Progettazione e acquisto di oligonucleotidi: Quando si acquistano gli oligonucleotidi per la formazione di duplex, è essenziale considerare la progettazione della sequenza. Si consiglia di utilizzare uno strumento di analisi degli oligo per prevedere le proprietà della sequenza nucleotidica, come il contenuto di GC, la temperatura di fusione, la struttura secondaria e il potenziale di dimerizzazione, prima di ordinare57.
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
SEG e UR sono dipendenti di ArcticZymes Technologies AS che distribuisce la R2D ligasi. AW, ER-S e RS non hanno interessi concorrenti.
Riconoscimenti
AW è supportata da una Rutherford Discovery Fellowship (20-UOW-004). RS è il destinatario di una borsa di studio New Zealand Post Antarctic. SG e UR ringraziano l'Istituto Chimico dell'Università di Tromsø - The Norwegian Arctic University per il supporto tecnico.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
Riferimenti
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon