Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Verwendung modifizierter synthetischer Oligonukleotide zum Assay von Nukleinsäure-metabolisierenden Enzymen
In diesem Artikel
Zusammenfassung
Hier wird ein Protokoll zum Assay von Nukleinsäure-metabolisierenden Enzymen vorgestellt, wobei Beispiele von Ligasen-, Nuklease- und Polymerase-Enzymen verwendet werden. Der Assay verwendet fluoreszenzmarkierte und unmarkierte Oligonukleotide, die zu Duplexen kombiniert werden können, die RNA- und/oder DNA-Schäden oder Signalwegzwischenstufen nachahmen, was die Charakterisierung des Enzymverhaltens ermöglicht.
Zusammenfassung
Die Verfügbarkeit einer Reihe von modifizierten synthetischen Oligonukleotiden von kommerziellen Anbietern hat die Entwicklung ausgefeilter Assays zur Charakterisierung verschiedener Eigenschaften von Nukleinsäure-metabolisierenden Enzymen ermöglicht, die in jedem Standardlabor für Molekularbiologie durchgeführt werden können. Die Verwendung von Fluoreszenzmarkierungen hat diese Methoden für Forscher mit Standard-PAGE-Elektrophoresegeräten und einem fluoreszenzfähigen Imager zugänglich gemacht, ohne dass radioaktive Materialien verwendet werden müssen oder ein Labor für die Lagerung und Aufbereitung von radioaktiven Materialien erforderlich ist, d. h. ein Hot Lab. Die optionale Hinzufügung von Standardmodifikationen wie Phosphorylierung kann den Assay-Aufbau vereinfachen, während der spezifische Einbau von modifizierten Nukleotiden, die DNA-Schäden oder Zwischenprodukte nachahmen, verwendet werden kann, um spezifische Aspekte des Enzymverhaltens zu untersuchen. Hier wird das Design und die Durchführung von Assays zur Untersuchung verschiedener Aspekte der DNA-Prozessierung durch Enzyme unter Verwendung kommerziell verfügbarer synthetischer Oligonukleotide demonstriert. Dazu gehören die Fähigkeit von Ligasen, sich zu verbinden, oder von Nukleasen, verschiedene DNA- und RNA-Hybridstrukturen abzubauen, die unterschiedliche Verwendung von Cofaktoren durch die DNA-Ligase und die Bewertung der DNA-Bindungskapazität von Enzymen. Faktoren, die bei der Entwicklung synthetischer Nukleotidsubstrate zu berücksichtigen sind, werden diskutiert, und es wird ein grundlegender Satz von Oligonukleotiden bereitgestellt, die für eine Reihe von Nukleinsäure-Ligasen-, Polymerase- und Nuklease-Enzym-Assays verwendet werden können.
Einleitung
Alle Lebensformen benötigen Nukleinsäure-verarbeitende Enzyme, um grundlegende biologische Prozesse durchzuführen, einschließlich Replikation, Transkription und DNA-Reparatur. Wichtige enzymatische Funktionalitäten für diese Signalwege sind Polymerasen, die Kopien von RNA/DNA-Molekülen erzeugen, Ligasen, die Polynukleotidsubstrate verbinden, Nukleasen, die sie abbauen, sowie Helikasen und Topoisomerasen, die Nukleinsäure-Duplexe schmelzen oder ihre Topologie verändern 1,2,3,4,5,6,7,8,9,10 . Darüber hinaus bieten viele dieser Enzyme wichtige molekulare Werkzeuge für Anwendungen wie Klonierung, Diagnostik und Hochdurchsatz-Sequenzierung 11,12,13,14,15.
Die funktionellen Eigenschaften, die Kinetik und die Substratspezifitäten dieser Enzyme können unter Verwendung von markierten DNA/RNA-Substraten bestimmt werden, die durch Annealing-Oligonukleotide hergestellt werden. Die Verfolgung von Substraten und Produkten wurde traditionell durch Einbringen einer radioaktiven Markierung (32P) entweder am 5'-Strangende erreicht, die dann durch einen fotografischen Film oder mit einem Phosphorbildgebungssystem16,17 detektiert werden kann. Während radioaktiv markierte Substrate den Vorteil einer erhöhten experimentellen Empfindlichkeit bieten und die chemischen Eigenschaften eines Nukleotids nicht verändern, haben die potenziellen Gesundheitsgefahren durch die Arbeit mit Radioisotopen die Entwicklung der nicht-radioaktiven Nukleinsäuremarkierung gefördert, um eine sicherere Alternative für den DNA- und RNA-Nachweis zu bieten 18,19,20. Unter diesen sticht die Fluoreszenzdetektion, einschließlich der direkten Fluoreszenzdetektion, der zeitaufgelösten Fluoreszenz und der Energietransfer-/Fluoreszenzquench-Assays, als die vielseitigsten hervor 21,22,23,24. Das umfangreiche Array an Fluorophoren ermöglicht unterschiedliche Designs von DNA/RNA-Substraten mit einzigartigen Reportern auf jedem Oligonukleotid25. Darüber hinaus ermöglicht die Stabilität von Fluorophoren im Vergleich zu Radioisotopen den Anwendern, signifikante Mengen fluoreszenzmarkierter DNA-Substrate herzustellen und zu konservieren19. Diese Fluorophor-markierten Substrate können mit dem interessierenden Protein zusammen mit verschiedenen Kombinationen von Metall- und Nukleotid-Cofaktoren inkubiert werden, um die Bindungs- und/oder Enzymaktivität zu analysieren. Die Visualisierung der Bindung oder Aktivität kann unter Verwendung verschiedener Fluorophor-Farbstoffkanäle mit einem Gel-Imaging-System beobachtet werden. Da bei dieser Technik nur die fluoreszenzmarkierten Oligonukleotide sichtbar sind, ist jede Zunahme oder Abnahme der Größe des markierten Oligonukleotids leicht zu verfolgen. Gele können auch nachträglich mit Nukleinsäure-Färbefarbstoffen gefärbt werden, um alle auf dem Gel vorhandenen DNA-Banden sichtbar zu machen.
Polynukleinsäure-Ligasen sind Enzyme, die DNA/RNA-Fragmente verbinden und die Versiegelung von Brüchen durch die Bildung einer Phosphodiesterbindung zwischen 5'-phosphorylierten DNA-Termini und dem 3'-OH der DNA katalysieren. Sie können entsprechend ihrer Anforderung an das Nukleotidsubstrat in zwei Gruppen unterteilt werden. Die hochkonservierten NAD-abhängigen Ligasen kommen in allen Bakterienvor 26, während die strukturell vielfältigen ATP-abhängigen Enzyme in allen Lebensbereichen identifiziert werden können 8,27. DNA-Ligasen spielen eine wichtige Rolle bei der Verarbeitung von Okazaki-Fragmenten während der Replikation und sind an verschiedenen DNA-Reparaturwegen beteiligt, wie z. B. der Nukleotid- und Basenexzisionsreparatur, durch die Versiegelung von spontanen Kerben und Kerben, die nach der Reparatur übrig bleiben 8,10. Verschiedene DNA-Ligasen weisen unterschiedliche Fähigkeiten auf, verschiedene Konformationen von DNA-Brüchen zu verbinden, einschließlich Kerben in einem Duplex, doppelsträngige Brüche, Fehlanpassungen und Lücken sowie RNA- und DNA-Hybride 28,29,30. Eine Vielzahl von ligierbaren Substraten kann durch Glühen von Oligonukleotiden mit einem 5'-Phosphat zusammengesetzt werden, um nebeneinander liegende 5'- und 3'-Termini in einem Nukleinsäureduplex 31,32,33 zu erzeugen. Die gebräuchlichste Analysemethode ist die Auflösung durch Harnstoff-PAGE in einem Endpunkt-Assay-Format; Zu den jüngsten Innovationen gehören jedoch die Verwendung der Kapillargelelektrophorese, die einen hohen Durchsatz34 ermöglicht, die massenspektrometrische Profilierung35 sowie ein homogener molekularer Beacon-Assay, der eine zeitaufgelöste Überwachungermöglicht 36.
Der erste Schritt in einer Ligationsreaktion ist die Adenylierung des Ligase-Enzyms durch Adenosintriphosphat (ATP) oder Nicotinamid-Adenindinukleotid (NAD), wodurch ein kovalentes Enzymzwischenprodukt entsteht. Der zweite Schritt in der Reaktion ist die Adenylierung des Nukleinsäuresubstrats am 5'-Ende der Nickstelle, gefolgt von der Ligation der Nukleinsäure-Nick-Stränge. Viele Ligase-Enzyme, die in E. coli rekombinant exprimiert werden, werden in der adenylierten Form gereinigt und sind daher in der Lage, Nukleinsäuren ohne Zugabe eines Nukleotid-Cofaktors erfolgreich zu ligieren. Dies macht es schwierig zu bestimmen, welche Art von Nukleotid-Cofaktor sie für die Ligation von Nukleinsäuren benötigen. Neben der Beschreibung von Assays zur Bewertung der DNA-Ligase-Aktivität wird auch eine Methode zur zuverlässigen Bestimmung des Cofaktor-Einsatzes durch Deadenylierung des Enzyms unter Verwendung unmarkierter Substrate vorgestellt.
Nukleasen sind eine große und vielfältige Gruppe von DNA/RNA-modifizierenden Enzymen und katalytischen RNAs, die die Phosphodiesterbindungen zwischen Nukleinsäuren spalten37. Nuklease-Enzymfunktionalitäten sind für die DNA-Replikation, -Reparatur und -Verarbeitung erforderlich und können nach ihrer Zuckerspezifität für DNA, RNA oder beides klassifiziert werden. Endonukleasen hydrolysieren die Phosphodiesterbindungen innerhalb eines DNA/RNA-Strangs, während Exonukleasen DNA/RNA-Stränge jeweils ein Nukleotid vom 3'- oder 5'-Ende hydrolysieren und dies entweder vom 3'- bis 5'- oder vom 5'- bis 3'-Ende der DNA38 tun können.
Während viele Nukleaseproteine unspezifisch sind und an mehreren Prozessen beteiligt sein können, sind andere hochspezifisch für eine bestimmte Sequenz oder DNA-Schädigung 6,39,40. Sequenzspezifische Nukleasen werden in einer Vielzahl biotechnologischer Anwendungen eingesetzt, wie z. B. Klonierung, Mutagenese und Genome Editing. Beliebte Nukleasen für diese Anwendungen sind Restriktionsnukleasen41, Zinkfinger-Nukleasen42, transkriptionelle Aktivator-ähnliche Effektor-Nukleasen und zuletzt die RNA-gesteuerten technisch hergestellten CRISPR-Nukleasen43. In jüngster Zeit wurden schadensspezifische Nukleasen identifiziert, wie z. B. die EndoMS-Nuklease, die durch ihre Mismatch-spezifische RecB-ähnliche Nukleasedomäne Spezifität für Fehlanpassungen in der DNA aufweist 5,44. Nukleaseaktivitätsassays wurden in der Vergangenheit als diskontinuierliche Assays mit radioaktiv markierten Substraten durchgeführt; Zusätzlich zu ihren anderen Nachteilen erlauben diese jedoch nicht die Identifizierung der Stelle, die von einem Nukleaseprotein durchtrennt wird, was bei Verwendung fluoreszenzmarkierter Substrate möglich ist45,46. In jüngerer Zeit wurden kontinuierliche Nuklease-Assays entwickelt, bei denen verschiedene DNA-Farbstoffe verwendet werden, die mit DNA in unterschiedlichen Zuständen interagieren. zum Beispiel die Emission eines höheren Fluoreszenzsignals bei der Wechselwirkung mit dsDNA als in ihrem ungebundenen Zustand oder die spezifische Bindung an kurze RNAs47. Andere kontinuierliche Nuklease-Assays verwenden DNA-Haarnadeln mit einer Fluorophorgruppe am 5'-Ende und einem Quencher am 3'-Ende, so dass die Fluoreszenz zunimmt, wenn das Oligonukleotid aufgrund einer Trennung des Fluorophors und des Quenchers48 abgebaut wird. Diese Assays ermöglichen es zwar, die Kinetik von DNA-abbauenden Proteinen zu charakterisieren, sie erfordern jedoch Vorkenntnisse über die Funktion und das Substrat des Enzyms und sind auch auf Enzyme beschränkt, die die DNA-Konformation verändern, um einen Unterschied in der Farbstoffbindung zu verursachen. Aus diesem Grund sind Endpunkt-Assays, die einzelne Nukleaseprodukte auflösen, nach wie vor wünschenswert, um Einblicke in DNA-Modifikationen zu geben, die durch Proteinaktivität verursacht werden.
Hier wird ein detailliertes Verfahren für das Design von fluoreszenzmarkierten DNA/RNA-Oligonukleotiden vorgestellt, die gemischt und angepasst werden können, um Substrate zum Testen der Aktivität neuartiger Nuklease-, Polymerase- und Ligaseenzyme zu erzeugen. Die Validierung dieses Basissatzes von Oligonukleotidsequenzen vereinfacht das experimentelle Design und ermöglicht die wirtschaftliche Profilierung einer Vielzahl enzymatischer Funktionalitäten, ohne dass eine große Anzahl maßgeschneiderter Substrate gekauft werden muss. Es wird ein detailliertes Verfahren zum Durchführen eines Standard-DNA-prozessierenden Enzymassays mit diesen Substraten am Beispiel der DNA-Ligaseaktivität bereitgestellt und Modifikationen für das Assaying und die Analyse von Nuklease- und Polymeraseenzymen beschrieben. Darüber hinaus wird ein modifizierter Assay zur Bestimmung der Cofaktor-Spezifität des DNA-Ligase-Enzyms mit hoher Genauigkeit gegeben, und es werden dual-markierte Sonden verwendet, um die Assemblierung von Mehrkomponenten-Ligationen zu bewerten. Schließlich werden Modifikationen am Basis-Assay-Format diskutiert, um es zur Bestimmung von Protein-DNA-Wechselwirkungen mit denselben Substraten durch den electrophoretic mobility shift assay (EMSA) zu ermöglichen.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Entwicklung und Kauf von Oligonukleotiden
HINWEIS: Entwerfen Sie einzelsträngige Oligonukleotide, die zu den gewünschten Duplexen zusammengebaut und geglüht werden sollen. Einer oder mehrere der Stränge in einem Duplex müssen eine fluoreszierende Einheit tragen, um die Oligonukleotidverarbeitung durch das interessierende Enzym zu verfolgen. Ein Basissatz von einzelsträngigen Sequenzen, die für eine Reihe von Aktivitäten assembliert werden können, ist in Tabelle 1 enthalten.
- Integrieren Sie die spezifischen Modifikationen, die für das interessierende Enzym erforderlich sind, wie unten beschrieben.
- Für DNA-Ligase-Substrate (Abbildung 1): Assemblieren Sie das einfachste Substrat aus drei Oligonukleotiden: einem 5' phosphorylierten Donorstrang (NL2), einem 5' FAM-markierten Akzeptorstrang (NL1) und einem Komplement, das die beiden verbindet (NL3).
- Stellen Sie sicher, dass die Stränge, die den 5'-Endpunkt des ligierbaren Kerbens bilden, phosphoryliert sind, bevor Sie den Substrat-Mastermix in Schritt 2 zusammensetzen. Bestellen Sie dies als Modifikation an NL2 (wie in Tabelle 1 angegeben) oder verwenden Sie eine enzymatische Phosphorylierung mit T4-Polynukleotidkinase nach Resuspendierung der Oligonukleotide.
- Eingeschlossen ist die 5'-terminale Phosphorylierung von NL6 und NL8, die das Komplement der in Abbildung 1A dargestellten doppelsträngigen Brüche (NL6/NL7 und NL8/NL9) umfasst, da dies dem natürlichen Substrat, das aus einer Restriktionsendonuklease hergestellt wird, am ehesten ähnelt. Verwenden Sie ein doppelt beschriftetes Substrat, um die relativen Ausdehnungen der Ligation für mehrteilige Baugruppen zu bestimmen (siehe Schritt 6).
- Verändern Sie den Komplementstrang, um Fehlanpassungen (NL10) und Lücken (NL11) zu erzeugen.
HINWEIS: Variationen des einfachen gekerbten Substrats sind in Abbildung 1A dargestellt. Es ist möglich, andere Sequenzen zu verwenden, um eine noch größere Bandbreite an Fehlanpassungen oder längeren Lücken zu erzeugen, indem die unterstrichene Position variiert wird. - Substituieren Sie RNA-Oligonukleotide durch DNA-Oligonukleotide.
HINWEIS: Variationen des einfachen gekerbten Substrats sind in Abbildung 1B dargestellt. Ein breiterer Bereich von DNA/RNA-Duplexen kann durch zusätzliche Kombinationen des hier gegebenen Basissatzes erzeugt werden, um beispielsweise doppelsträngige Brüche zu erzeugen, die sowohl RNA als auch DNA enthalten. Ein Beispiel für diese Variation finden Sie in Schritt 6 unten, in dem eine Dual-Label-Strategie verwendet wird.
- Für DNA-Polymerase-Substrate: Assemblieren Sie die in Tabelle 1 aufgeführten Oligonukleotide NL1 und NL3, um einen einfachen Primer-Verlängerungs-Assay zu erhalten. Untersuchen Sie zusätzliche Aspekte der Polymerase-Aktivität, indem Sie Modifikationen entweder in die Stränge NL1 (Primer) oder NL3 (Template) einführen.
- Beschädigte Basenanaloga vor Position 20 in das NL3-Oligonukleotid einbauen, um die Fähigkeit zu bestimmen, beschädigte Läsionen auf dem Template-Strang zu umgehen.
- Bauen Sie beschädigte Basenanaloga in das NL1-Oligonukleotid an Position 20 ein, um die Fähigkeit zu bestimmen, einen beschädigten Primer zu verlängern.
- Verwenden Sie entweder RNL1 oder RNL3 im Duplex, um die Verlängerung eines RNA-Primers oder die Verwendung eines RNA-Templates zu untersuchen.
- Für Nukleasesubstrate (Abbildung 2): Assemblieren Sie Oligonukleotide, um einen nicht erschöpfenden Bereich von doppel- und einzelsträngigen Substraten (Abbildung 2Ai) sowie einen Bereich von geflatterten und gespreizten Übergängen (Abbildung 2Aii) und beschädigten Substraten (Abbildung 2B) zu erhalten.
- Um die Ribonuklease-Aktivitäten zu untersuchen, ersetzen Sie iterativ NL1, NL2 und NL3 durch RNL1, RNL2 und RNL3. Verwenden Sie zusätzliche RNA-Versionen von HJ5 und HJ6, um diesen Satz weiter zu erweitern.
- Verwenden Sie die Oligonukleotide MD5, MD6 und MD9 mit einer zentral platzierten Modifikation, die oxidative Schäden nachahmt, einem abbasischen Reparaturzwischenprodukt oder einem Desaminierungsprodukt (Abbildung 2B). Die Substrate erkennen die Spaltung des Strangs an dieser Position. Markieren Sie den Komplement-NL3-Strang mit einem orthogonalen Fluorophor wie TAMRA, um eine Doppelstrangspaltung zu erkennen (siehe Schritt 6).
- Verwenden Sie eine orthogonale Markierung des Komplementes, um doppelsträngige Schnitte an den nicht übereinstimmenden Stellen sowohl auf der Sonde (NL5 und ND9) als auch auf den Komplementsträngen (MD10 und NL10) zu erkennen.
- Für DNA-Ligase-Substrate (Abbildung 1): Assemblieren Sie das einfachste Substrat aus drei Oligonukleotiden: einem 5' phosphorylierten Donorstrang (NL2), einem 5' FAM-markierten Akzeptorstrang (NL1) und einem Komplement, das die beiden verbindet (NL3).
- Bestellen Sie synthetische Oligonukleotide mit relevanten Fluorophoren und anderen Modifikationen bei einem kommerziellen Anbieter.
HINWEIS: Für die beschriebenen Assays sind ein Synthesemaßstab von 100 nM und eine HPLC-Aufreinigung im Anschluss an die Synthese geeignet.
2. Assemblieren und Glühen von Nukleinsäure-Duplexen
- Resuspension und Verdünnung von Oligonukleotiden
- Zentrifugieren Sie vor dem Öffnen die lyophilisierten Oligonukleotide in ihren 2-ml-Röhrchen bei voller Geschwindigkeit in einer Tischzentrifuge für 2-5 Minuten, um sicherzustellen, dass sich die Nukleinsäure auf dem Boden des Röhrchens befindet.
- Bereiten Sie einen Masterstock von 100 μM vor, indem Sie die Oligonukleotide in TE-Puffer (10 mM Tris(hydroxymethyl)aminomethan (Tris), 1 mM Ethylendiamintetraessigsäure (EDTA)) resuspendieren. Stellen Sie sicher, dass die Oligonukleotide durch wiederholtes sanftes Vortexen und kurzes Zentrifugieren bei voller Geschwindigkeit gründlich resuspendiert werden.
- Bereiten Sie ein 10-μM-Rohmaterial vor, indem ein Aliquot des Hauptmaterials mit TE-Puffer verdünnt wird. Das 10-μM-Rohmaterial wird mit Reinstwasser (MQ-Wasser) verdünnt, um Arbeitsmaterial mit Konzentrationen von 0,5 μM, 0,7 μM oder 2,5 μM gemäß Tabelle 2 herzustellen.
- Zusammenfügen und Glühen der Reaktions-Mastermixe
- Zur Herstellung der Reaktions-Mastermixe werden die Reaktions-Mastermixe unter Verwendung der in Tabelle 2 angegebenen Kombinationen und der in Tabelle 3 angegebenen Volumina verwendet. Für den Standard-DNA-Ligase-Assay und die meisten anderen hier beschriebenen Assays beträgt die endgültige Pufferzusammensetzung 50 mM Tris pH 8,0, 50 mM NaCl, 10 mM Dithiothreitol (DTT) mit 10 mM Mg als zweiwertigem Kation.
- Die Oligonukleotide werden in einem PCR- oder Mikrozentrifugenröhrchen durch Erhitzen auf 95 °C für 5 Minuten mit einem Heizblock oder Thermocycler geglüht. 30 min (Volumen <1 mL) bis 1 h (Volumen >1 mL) auf Raumtemperatur abkühlen lassen. Bei längeren Oligonukleotiden (>40 nt) ist eine langsamere Abkühlung mit einem Thermocycler mit einer Abwärtsrampe von 95 °C auf 25 °C über einen Zeitraum von 45 min durchzuführen oder das Röhrchen mit der Glühmischung in einem 1-Liter-Becherglas mit kochendem Wasser schwimmen zu lassen und über Nacht auf Raumtemperatur abkühlen zu lassen.
- Fügen Sie dem Mastermix nach dem Abkühlen auf Raumtemperatur Nukleotid-Cofaktoren und andere wärmeempfindliche Pufferkomponenten hinzu. Das endgültige Reaktionsgemisch durch Zugabe von Enzym direkt für den Assay verwenden (siehe Schritt 3 unten) oder für die zukünftige Verwendung bei -20 °C lagern.
3. Standard-Assay-Aufbau
- Assemblierung und Initiierung der Assay-Reaktion
- Kombinieren Sie 22,5 μl des Substrat-Mastermixes von Interesse mit 2,5 μl der DNA-Ligase oder eines anderen Enzyms von Interesse in einem PCR-Röhrchen. Führen Sie Reaktionen doppelt oder dreifach aus, insbesondere wenn die Ergebnisse quantifiziert werden.
- Nehmen Sie eine proteinfreie Kontrolle (nur Puffer) in die Assay-Proben auf. Schließen Sie an dieser Stelle bei Bedarf keine Cofaktor-Steuerelemente ein.
HINWEIS: Enzyme können bei -20 °C in 50 % v/v Glycerin gelagert werden, so dass sie direkt aus der Lösung pipettiert werden können. Stellen Sie sicher, dass die Enzymlösungen mit Glycerin vor der Zugabe gut gemischt werden, entweder durch Pipettieren zum Mischen oder durch sanftes Vortexen.
- Übertragen Sie die Reaktionen sofort bei 25 °C auf eine PCR-Maschine und inkubieren Sie sie 30 Minuten lang. Variieren Sie die Temperatur und Dauer je nach den optimalen Bedingungen für die Enzymaktivität.
- Die Reaktionen werden durch Zugabe von 5 μl Ladefarbstoff (95 % Formamid, 0,5 M Ethylendiamintetraessigsäure (EDTA), Bromphenolblau) abgeschreckt und 5 min lang bei 95 °C inkubiert.
4. Analyse der Untersuchungsergebnisse
- Bereiten Sie Tris-Borat-EDTA (TBE)-Harnstoff-PAGE-Gele wie unten beschrieben vor.
- Bereiten Sie einen Vorrat von 20 % Acrylamid, 7 M Harnstoff und 1x FSME-Lösung vor. Verwenden Sie für das hier beschriebene Oligonukleotid-Set Acrylamid/Bis-Lösung in einem Verhältnis von 29:1, um eine optimale Auflösung zu erzielen.
- Kombinieren Sie für ein Gel 10 ml 20 % Acrylamid und 7 M Harnstofflösung mit 100 μl APS (10 %) und 3 μl Tetramethylethylendiamin (TMED) und gießen Sie es in einen Gelgießer.
- Nachdem das Gel fest geworden ist, lassen Sie die Proben auf dem FSME-Harnstoffgel bei 45 - 55 °C laufen.
- Lassen Sie das Gel in 1x FSME-Puffer für 30 min bei 10 mA pro Gel mit externer Erhitzung vorlaufen.
- Entfernen Sie überschüssigen Harnstoff in den Vertiefungen des Gels, indem Sie mit einer Weidepipette mit 1x FSME spülen.
- Laden Sie 10 μl jeder Reaktion und lassen Sie sie 1,0-1,5 h lang bei 10 mA mit externer Heizung laufen.
- Visualisieren Sie das Gel auf dem Imager mit den richtigen Einstellungen für das ausgewählte Fluorophor. Verwenden Sie für FAM ein Filterset, das Anregung/Emission bei 495/519 nm anzeigt und in den meisten Imagern als Voreinstellung gespeichert ist.
- Quantifizieren Sie die Bandenintensität des Produkts und des Substrats mit Hilfe einer Bildverarbeitungssoftware mit dem Imager oder einem externen Programm wie ImageJ49,50 und berechnen Sie den prozentualen Anteil des Produkts anhand der Formel
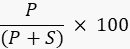
Dabei ist P der integrierte Wert des Produktbandes und S der integrierte Bereich des Substratbandes. Im Fall des Beispiels der DNA-Ligase-Reaktion verläuft die Produktbande bei 40 Nukleotiden (nt) und die Substratbande bei 20 nt.
5. Deadenylierung der DNA-Ligase zur Prüfung der Cofaktor-Spezifität
- Vorbereitung von Reaktions-Mastermixen
- Bereiten Sie einen Satz des Mastermixes vor, der das FAM-markierte NL1-Oligonukleotid enthält, wie in Tabelle 4 beschrieben. Bereiten Sie ein zweites Set vor, das das NL1-Oligonukleotid ohne FAM-Markierung enthält, wie in Tabelle 4 beschrieben.
- Erhitzen Sie beide DNA-Duplexe separat 5 Minuten lang auf 95 °C und kühlte sie 30 Minuten lang auf 1 Stunde bei 25 °C ab. Fügen Sie keinem der beiden Mastermixe einen Nukleotid-Cofaktor hinzu.
- Assemblierung und Initiierung der Deadenylierungsreaktion
- Bereiten Sie eine einzelne Deadenylierungsreaktion für jeden zu testenden Cofaktortyp/jede zu testende Konzentration vor, indem Sie 10 μl des unmarkierten Mastermixes mit 2,5 μl Ligaseenzym kombinieren.
- Bereiten Sie zusätzliche Röhrchen ohne Cofaktorenkontrolle und ohne Proteinkontrolle vor (2,5 μl Puffer anstelle des Enzyms hinzugefügt).
- Die Reaktionen werden 1-2 Stunden lang bei einer Temperatur inkubiert, die für die optimale Aktivität des Enzyms spezifisch ist. Die Inkubationszeit kann verlängert werden, wenn das Enzym noch adenyliert ist.
- Führen Sie die Ligationsreaktion mit dem Cofaktor durch.
- Geben Sie 10 μl des markierten Mastermixes und 2,5 μl der gewünschten Nukleotid-Cofaktoren (z. B. ATP, NAD, ADP oder GTP) direkt zur deadenylierten Reaktion (0,1-1 mM Endkonzentration).
- 2,5 μl Reaktionspuffer zur Kontrolle ohne Nukleotid-Cofaktor hinzufügen.
- Inkubieren Sie die Reaktionen für den gleichen Zeitraum und die gleiche Temperatur wie zuvor verwendet. Quenden und visualisieren Sie, wie in Schritt 4 beschrieben.
6. Verwendung von doppelt markierten Substraten für die geschiente Ligatur oder die mehrteilige Montage
- Entwicklung und Kauf eines Oligonukleotids mit einer fluoreszierenden Einheit, die ein anderes Anregungs-/Emissionsspektrum als das bereits verwendete Fluorophor aufweist.
- Verwenden Sie in dem hier beschriebenen Aufbau das Oligonukleotid NL2 (TAMRA) mit 5-Carboxytetramethylrhodamin (TAMRA) am 3'-Ende (Tabelle 1).
- Stellen Sie den Master-Mix wie unten beschrieben zusammen.
- Kombinieren Sie die Komponenten der in Schritt 2 beschriebenen Reaktion, einschließlich der äquimolaren Verhältnisse aller in der Assemblierung verwendeten Oligonukleotide sowie der Puffer- und zweiwertigen Kationen.
- Glühen durch Erhitzen bei 95 °C für 5 min und Abkühlen bei 25 °C für 30 min - 1 h. Fügen Sie den Cofaktor und das Enzym hinzu und inkubieren Sie wie in Schritt 3 beschrieben.
- Führen Sie die Proben wie in Schritt 4 beschrieben unter Verwendung der entsprechenden Kanäle für das Fluorophorpaar im Substrat durch und bilden Sie sie ab. Im Fall von FAM und TAMRA sind dies die Kanäle Fluorescein (FITC) und Tetramethylrhodamin (TRITC), die auf den meisten Imagern vorhanden sind.
7. Bewertung der DNA-Bindung mittels Electrophoretic Mobility Shift Assay (EMSA) auf nativem Gel
- Bereiten Sie ein 10% natives TBE PAGE Gel wie unten beschrieben vor.
- Kombinieren Sie 2,5 ml 40 % Acrylamid, 1 ml 10x FSME, 100 μl 10 % APS, 3 μl TMED und 6,5 ml MQ-Wasser und gießen Sie es in einen Gelgießer.
- Assemblieren Sie die Bindungsreaktion wie unten beschrieben.
- Montieren Sie das EMSA-Substrat gemäß Tabelle 5 so, dass EDTA (10 mM) enthalten ist und Metallionen weggelassen werden.
- Kombinieren Sie 20 μL des EMSA-Substrat-Mastermixes mit 5 μL des Proteins in einem PCR-Röhrchen. Fügen Sie eine proteinfreie Kontrollprobe hinzu. 30 min bei 25 °C inkubieren.
- Analysieren Sie durch native Elektrophorese, wie unten beschrieben.
- Geben Sie 5 μl 5x nativen Ladefarbstoff (100 mM EDTA, 0,25 % Bromphenolblau, 25 % v/v Glycerin und MQ Wasser bis zu 1 mL) zu den Proben.
- Laden Sie das vorbereitete Gel auf und lassen Sie es bei 60 V für 2-3 h mit Kühlung durch Wasserzirkulation laufen, bis die Farbstofffront einige cm über dem Ende des Gels liegt.
- Visualisieren und analysieren Sie Gele, wie in Schritt 4 beschrieben.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Ligation durch DNA-Ligase
Die enzymatische Aktivität der DNA-Ligase führt zu einer Vergrößerung des fluoreszenzmarkierten Oligonukleotids, wenn es auf einem Harnstoff-PAGE-Gel sichtbar gemacht wird. Bei den in Tabelle 2 aufgeführten Substraten sowohl für die DNA- als auch für die RNA-Ligation entspricht dies einer Verdopplung der Größe von 20 nt auf 40 nt (Abbildung 3A). Die optimale Enzymaktivität kann durch Änderung von Bedingungen wie Temper...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Kritische Schritte im Protokoll
Oligonukleotiddesign und -kauf: Beim Kauf von Oligonukleotiden für die Duplexbildung ist es wichtig, das Sequenzdesign zu berücksichtigen. Es wird empfohlen, vor der Bestellung ein Oligo-Analysewerkzeug zu verwenden, um die Eigenschaften der Nukleotidsequenz wie GC-Gehalt, Schmelztemperatur, Sekundärstruktur und Dimerisierungspotenzial vorherzusagen57.
Assemblierung und Annealing von Nukleinsäure-Duplexen: Bei der ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
SEG und UR sind Mitarbeiter von ArcticZymes Technologies AS, die die R2D-Ligase vertreibt. AW, ER-S und RS haben keine konkurrierenden Interessen.
Danksagungen
AW wird durch ein Rutherford Discovery Fellowship (20-UOW-004) unterstützt. RS ist Empfänger eines neuseeländischen Postantarktis-Stipendiums. SG und UR danken dem Chemischen Institut an der Universität Tromsø - der Norwegischen Arktis-Universität - für die technische Unterstützung.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
Referenzen
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten