É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Usando oligonucleotídeos sintéticos modificados para testar enzimas metabolizadoras de ácidos nucleicos
Neste Artigo
Resumo
Aqui, é apresentado um protocolo para testar enzimas metabolizadoras de ácidos nucleicos, usando exemplos de enzimas ligase, nuclease e polimerase. O ensaio utiliza oligonucleotídeos marcados e não marcados com fluorescência que podem ser combinados para formar duplexes que imitam danos ao RNA e/ou DNA ou intermediários da via, permitindo a caracterização do comportamento enzimático.
Resumo
A disponibilidade de uma variedade de oligonucleotídeos sintéticos modificados de fornecedores comerciais permitiu o desenvolvimento de ensaios sofisticados para caracterizar diversas propriedades de enzimas metabolizadoras de ácidos nucleicos que podem ser executadas em qualquer laboratório de biologia molecular padrão. O uso de rótulos fluorescentes tornou esses métodos acessíveis a pesquisadores com equipamento de eletroforese PAGE padrão e um gerador de imagens habilitado para fluorescência, sem usar materiais radioativos ou exigir um laboratório projetado para o armazenamento e preparação de materiais radioativos, ou seja, um Hot Lab. A adição opcional de modificações padrão, como a fosforilação, pode simplificar a configuração do ensaio, enquanto a incorporação específica de nucleotídeos modificados que imitam danos ao DNA ou intermediários pode ser usada para sondar aspectos específicos do comportamento enzimático. Aqui, o projeto e a execução de ensaios para interrogar vários aspectos do processamento de DNA por enzimas usando oligonucleotídeos sintéticos disponíveis comercialmente são demonstrados. Isso inclui a capacidade das ligases de se unirem ou nucleases de degradar diferentes estruturas híbridas de DNA e RNA, uso diferencial de cofatores pela DNA ligase e avaliação da capacidade de ligação ao DNA das enzimas. Fatores a serem considerados ao projetar substratos de nucleotídeos sintéticos são discutidos e um conjunto básico de oligonucleotídeos que podem ser usados para uma variedade de ensaios de enzimas ligase de ácido nucleico, polimerase e nuclease são fornecidos.
Introdução
Todas as formas de vida requerem enzimas de processamento de ácido nucleico para realizar processos biológicos fundamentais, incluindo replicação, transcrição e reparo de DNA. As principais funcionalidades enzimáticas para essas vias são polimerases, que geram cópias de moléculas de RNA/DNA, ligases que unem substratos polinucleotídeos, nucleases que os degradam e helicases e topoisomerases, que fundem duplex de ácidos nucleicos ou alteram sua topologia 1,2,3,4,5,6,7,8,9,10 . Além disso, muitas dessas enzimas fornecem ferramentas moleculares essenciais para aplicações como clonagem, diagnóstico e sequenciamento de alto rendimento 11,12,13,14,15.
As características funcionais, cinética e especificidades de substrato dessas enzimas podem ser determinadas usando substratos de DNA/RNA marcados produzidos por oligonucleotídeos de recozimento. O rastreamento de substratos e produtos tem sido tradicionalmente alcançado pela introdução de um marcador radioativo (32P) na extremidade da fita 5 ', que pode então ser detectado por filme fotográfico ou com um sistema de imagem de fósforo16 , 17 . Embora os substratos radiomarcados ofereçam o benefício de maior sensibilidade experimental e não alterem as propriedades químicas de um nucleotídeo, os riscos potenciais à saúde decorrentes do trabalho com radioisótopos incentivaram o desenvolvimento da marcação de ácidos nucleicos não radioativos para fornecer uma alternativa mais segura para detecção de DNA e RNA 18,19,20. Dentre estes, a detecção de fluorescência, incluindo detecção direta de fluorescência, fluorescência resolvida no tempo e ensaios de transferência de energia/extinção de fluorescência se destacam como os mais versáteis 21,22,23,24. A extensa gama de fluoróforos permite diferentes designs de substratos de DNA / RNA com repórteres exclusivos em cada oligonucleotídeo25. Além disso, a estabilidade dos fluoróforos, quando comparados aos radioisótopos, permite que os usuários produzam e preservem quantidades significativas de substratos de DNA marcados com fluorescência19. Esses substratos marcados com fluoróforo podem ser incubados com a proteína de interesse, juntamente com diferentes combinações de cofatores de metal e nucleotídeos, para analisar a ligação e/ou a atividade enzimática. A visualização da ligação ou atividade pode ser observada usando vários canais de corante de fluoróforo com um sistema de imagem em gel. Como apenas os oligonucleotídeos marcados com fluorescência serão visíveis usando esta técnica, qualquer aumento ou diminuição no tamanho do oligonucleotídeo marcado será fácil de seguir. Os géis também podem ser corados posteriormente, com corantes de coloração de ácido nucleico para visualizar todas as bandas de DNA presentes no gel.
As ligases de ácidos polinucléicos são enzimas que unem fragmentos de DNA/RNA, catalisando a vedação de quebras pela formação de uma ligação fosfodiéster entre os terminais de DNA fosforilados 5' e o 3' OH do DNA. Eles podem ser divididos em dois grupos de acordo com sua necessidade de substrato de nucleotídeos. As ligases dependentes de NAD altamente conservadas são encontradas em todas as bactérias26, enquanto as enzimas dependentes de ATP estruturalmente diversas podem ser identificadas em todos os domínios da vida 8,27. As DNA ligases desempenham um papel importante no processamento do fragmento de Okazaki durante a replicação, além de estarem envolvidas em várias vias de reparo do DNA, como o reparo por excisão de nucleotídeos e bases, por meio do selamento de cortes espontâneos e cortes que são deixados após o reparo 8,10. Diferentes DNA ligases exibem capacidades variadas para unir diferentes conformações de quebras de DNA, incluindo cortes em um duplex, quebras de fita dupla, incompatibilidades e lacunas, bem como híbridos de RNA e DNA 28,29,30. Uma gama diversificada de substratos ligáveis pode ser montada por recozimento de oligonucleotídeos com um fosfato 5 'para gerar terminais justapostas de 5' e 3' em um duplex de ácido nucleico 31,32,33. O método de análise mais comum é a resolução por ureia PAGE em um formato de ensaio de ponto final; No entanto, inovações recentes incluíram o uso de eletroforese capilar em gel, que permite alto rendimento34, perfil espectrométrico de massa35, bem como um ensaio de farol molecular homogêneo, que permite o monitoramento resolvido no tempo36.
O primeiro passo em uma reação de ligação é a adenilação da enzima ligase por trifosfato de adenosina (ATP) ou dinucleotídeo de nicotinamida adenina (NAD), resultando em um intermediário enzimático covalente. A segunda etapa da reação é a adenilação do substrato de ácido nucleico na extremidade 5 'do local do corte, que é seguida pela ligação das fitas de ácido nucleico. Muitas enzimas ligase que são expressas recombinantemente em E. coli são purificadas na forma adenilada e, portanto, são capazes de ligar com sucesso ácidos nucléicos sem a adição de um cofator de nucleotídeo. Isso torna difícil determinar que tipo específico de cofator de nucleotídeo eles requerem para a ligação de ácidos nucléicos. Além de descrever ensaios para avaliar a atividade da DNA ligase, também é apresentado um método para determinar de forma confiável o uso de cofatores por meio da desadenilação da enzima usando substratos não marcados.
As nucleases são um grupo grande e diversificado de enzimas modificadoras de DNA / RNA e RNAs catalíticos que clivam as ligações fosfodiéster entre os ácidos nucléicos37. As funcionalidades da enzima nuclease são necessárias na replicação, reparo e processamento de RNA do DNA e podem ser classificadas por sua especificidade de açúcar para DNA, RNA ou ambos. As endonucleases hidrolisam as ligações fosfodiéster dentro de uma fita de DNA / RNA, enquanto as exonucleases hidrolisam as fitas de DNA / RNA um nucleotídeo de cada vez a partir da extremidade 3 'ou 5' e podem fazê-lo a partir da extremidade 3 'a 5' ou 5 'a 3' do DNA38.
Embora muitas proteínas de nuclease sejam inespecíficas e possam estar envolvidas em vários processos, outras são altamente específicas para uma sequência específica ou dano ao DNA 6,39,40. As nucleases específicas de sequência são usadas em uma ampla gama de aplicações biotecnológicas, como clonagem, mutagênese e edição de genoma. As nucleases populares para essas aplicações são nucleases de restrição41, nucleases de dedo de zinco42, nucleases efetoras semelhantes a ativadores transcricionais e, mais recentemente, as nucleases CRISPR projetadas guiadas por RNA43. Nucleases específicas de dano foram recentemente identificadas, como a nuclease EndoMS, que tem especificidade para incompatibilidades no DNA por meio de seu domínio de nuclease semelhante a RecB específico para incompatibilidade 5,44. Os ensaios de atividade da nuclease, historicamente, têm sido feitos como ensaios descontínuos com substratos radiomarcados; no entanto, além de suas outras desvantagens, elas não permitem a identificação do local que é cortado por uma proteína nuclease, o que é possível quando se utilizam substratos marcados com fluorescência45,46. Mais recentemente, foram desenvolvidos ensaios de nuclease contínua que funcionam usando diferentes corantes de DNA que interagem com o DNA em diferentes estados; por exemplo, emitindo um sinal fluorescente mais alto ao interagir com o dsDNA do que em seu estado não ligado, ou ligando-se especificamente a RNAs curtos47. Outros ensaios de nuclease contínua usam grampos de cabelo de DNA com um grupo fluoróforo na extremidade 5 'e um atenuador na extremidade 3', de modo que a fluorescência aumenta à medida que o oligonucleotídeo é degradado devido a uma separação do fluoróforo e do atenuador48. Embora esses ensaios permitam caracterizar a cinética das proteínas degradadoras do DNA, eles exigem conhecimento prévio da função e do substrato da enzima e também são limitados a enzimas que alteram a conformação do DNA para causar uma diferença na ligação do corante. Por esse motivo, os ensaios de endpoint que resolvem produtos de nuclease individuais ainda são desejáveis para fornecer informações sobre as modificações do DNA causadas pela atividade da proteína.
Aqui, um procedimento detalhado é apresentado para o projeto de oligonucleotídeos de DNA / RNA marcados com fluorescência que podem ser misturados e combinados para gerar substratos para testar a atividade de novas enzimas nuclease, polimerase e ligase. A validação deste conjunto básico de sequências de oligonucleotídeos simplifica o projeto experimental e facilita o perfil econômico de uma ampla gama de funcionalidades enzimáticas sem a necessidade de comprar um grande número de substratos sob medida. Um procedimento detalhado é fornecido para executar um ensaio enzimático de processamento de DNA padrão com esses substratos, usando o exemplo da atividade da DNA ligase e as modificações para testar e analisar as enzimas nuclease e polimerase são descritas. Além disso, um ensaio modificado para determinar a especificidade do cofator da enzima DNA ligase com alta precisão é fornecido, e sondas duplamente marcadas são usadas para avaliar a montagem de ligações multicomponentes. Finalmente, modificações no formato básico do ensaio são discutidas para permitir que ele seja usado para determinar as interações proteína-DNA com os mesmos substratos pelo ensaio de mudança de mobilidade eletroforética (EMSA).
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Projeto e compra de oligonucleotídeos
NOTA: Projete oligonucleotídeos de fita simples para serem montados e recozidos nos duplex desejados. Uma ou mais fitas de um duplex devem conter uma fração fluorescente para rastrear o processamento de oligonucleotídeos pela enzima de interesse. Um conjunto básico de sequências de fita simples que podem ser montadas para uma variedade de atividades é fornecido na Tabela 1.
- Incorporar as modificações específicas necessárias para a enzima de interesse, conforme descrito abaixo.
- Para substratos de DNA ligase (Figura 1): Monte o substrato mais simples a partir de três oligonucleotídeos: uma fita doadora fosforilada 5' (NL2), uma fita aceptora marcada com 5' FAM (NL1) e um complemento que une as duas (NL3).
- Certifique-se de que os fios que fornecem o terminal 5' do corte ligável sejam fosforilados antes da montagem da mistura mestre do substrato na etapa 2. Encomende isso como uma modificação em NL2 (conforme indicado na Tabela 1) ou use fosforilação enzimática com polinucleotídeo quinase T4 após ressuspender os oligonucleotídeos.
- Inclua a fosforilação 5'-terminal de NL6 e NL8, que compreende o complemento de quebras de fita dupla representadas na Figura 1A (NL6 / NL7 e NL8 / NL9), pois isso se assemelha mais ao substrato natural produzido a partir de uma endonuclease de restrição. Use um substrato de rótulo duplo para determinar as extensões relativas da ligação para montagens de várias partes (consulte a etapa 6).
- Altere a fita complementar para produzir incompatibilidades (NL10) e lacunas (NL11).
NOTA: Variações no substrato cortado simples são representadas na Figura 1A. É possível usar outras sequências para produzir uma gama ainda maior de incompatibilidades ou lacunas mais longas, variando a posição sublinhada. - Substitua oligonucleotídeos de DNA por oligonucleotídeos de RNA.
NOTA: Variações no substrato cortado simples são representadas na Figura 1B. Uma gama mais ampla de duplexes de DNA / RNA pode ser gerada por combinações adicionais do conjunto de bases fornecido aqui para gerar, por exemplo, quebras de fita dupla contendo RNA e DNA. Um exemplo dessa variação é dado na etapa 6 abaixo, onde uma estratégia de rótulo duplo é usada.
- Para substratos de DNA polimerase: Montar os oligonucleotídeos NL1 e NL3 listados na Tabela 1 para obter um ensaio simples de extensão de primer. Investigue aspectos adicionais da atividade da polimerase introduzindo modificações nas fitas NL1 (primer) ou NL3 (molde).
- Incorpore análogos de base danificados no oligonucleotídeo NL3 antes da posição 20 para determinar a capacidade de contornar lesões danificadas na fita molde.
- Incorpore análogos de base danificados no oligonucleotídeo NL1 na posição 20 para determinar a capacidade de estender um primer danificado.
- Use RNL1 ou RNL3 no duplex para investigar a extensão de um primer de RNA ou o uso de um molde de RNA.
- Para substratos de nuclease (Figura 2): Monte oligonucleotídeos para fornecer uma faixa não exaustiva de substratos de fita dupla e simples ( Figura 2 Ai ), bem como uma variedade de junções batidas e abertas ( Figura 2 Aii ) e substratos danificados ( Figura 2B ).
- Para investigar as atividades da ribonuclease, substitua iterativamente NL1, NL2 e NL3 por RNL1, RNL2 e RNL3. Use versões adicionais de RNA de HJ5 e HJ6 para expandir ainda mais esse conjunto.
- Use os oligonucleotídeos MD5, MD6 e MD9 que tenham uma modificação posicionada centralmente que imita o dano oxidativo, um intermediário de reparo abásico ou um produto de desaminação ( Figura 2B ). Os substratos detectarão a clivagem do fio nesta posição. Rotule a fita NL3 do complemento com um fluoróforo ortogonal, como TAMRA, para detectar clivagem de fita dupla (consulte a etapa 6).
- Use a rotulagem ortogonal do complemento para detectar cortes de fita dupla nos locais incompatíveis nas fitas da sonda (NL5 e ND9) e do complemento (MD10 e NL10).
- Para substratos de DNA ligase (Figura 1): Monte o substrato mais simples a partir de três oligonucleotídeos: uma fita doadora fosforilada 5' (NL2), uma fita aceptora marcada com 5' FAM (NL1) e um complemento que une as duas (NL3).
- Solicite oligonucleotídeos sintéticos incorporando fluoróforos relevantes e outras modificações de um fornecedor comercial.
NOTA: Uma escala de síntese de 100 nM e purificação por HPLC subsequente à síntese são adequadas para os ensaios descritos.
2. Montagem e recozimento de duplex de ácido nucleico
- Ressuspensão e diluição de oligonucleotídeos
- Antes de abrir, centrifugue os oligonucleotídeos liofilizados em seus tubos de 2 mL em velocidade máxima em uma centrífuga de bancada por 2-5 min para garantir que o ácido nucléico esteja no fundo do tubo.
- Preparar uma matéria-prima de 100 μM ressuspendendo os oligonucleótidos em tampão TE [tris(hidroximetil)aminometano (Tris) 10 mM, ácido etilenodiaminotetracético (EDTA) 1 mM]. Certifique-se de que os oligonucleotídeos sejam completamente ressuspensos por vórtice suave repetido e centrifugação breve em velocidade máxima.
- Preparar uma matéria-prima de 10 μM diluindo uma alíquota de matéria-prima com tampão TE. Dilua o estoque de 10 μM com água ultrapura (água MQ) para preparar estoques de trabalho com concentrações de 0.5 μM, 0.7 μM ou 2.5 μM conforme a Tabela 2.
- Montagem e recozimento das misturas master de reação
- Utilizar as misturas principais de reacção para compor as combinações indicadas no quadro 2 e os volumes indicados no quadro 3. Para o ensaio de DNA ligase padrão e a maioria dos outros ensaios descritos aqui, a composição final do tampão é 50 mM Tris pH 8,0, 50 mM NaCl, 10 mM Ditiotreitol (DTT) com 10 mM Mg como cátion divalente.
- Recozer os oligonucleótidos num tubo de PCR ou de microcentrífuga aquecendo a 95 °C durante 5 minutos utilizando um bloco de aquecimento ou termociclador. Deixe esfriar até a temperatura ambiente por 30 min (volumes <1 mL) a 1 h (volumes >1 mL). Para oligonucleótidos mais longos (>40 nt), efectuar um arrefecimento mais lento utilizando um termociclador com uma rampa descendente de 95 °C a 25 °C durante 45 min, ou fazer flutuar o tubo que contém a mistura de recozimento num copo de 1 L de água a ferver e deixar arrefecer até à temperatura ambiente durante a noite.
- Adicione cofatores de nucleotídeos e outros componentes tampão sensíveis ao calor à mistura principal após o resfriamento à temperatura ambiente. Utilizar a mistura de reação final directamente para o ensaio por adição de enzima (ver passo 3 abaixo) ou conservar a -20 °C para utilização futura.
3. Configuração padrão do ensaio
- Montagem e início da reação do ensaio
- Combine 22,5 μL da mistura principal de substrato de interesse com 2,5 μL da DNA ligase ou outra enzima de interesse em um tubo de PCR. Execute reações em duplicata ou triplicada, especialmente se os resultados forem quantificados.
- Inclua um controle sem proteína (somente tampão) nas amostras de ensaio. Não inclua controles de cofator neste momento, se necessário.
NOTA: As enzimas podem ser armazenadas a -20 °C em glicerol a 50% v/v, permitindo que sejam pipetadas diretamente da solução. Certifique-se de que as soluções enzimáticas com glicerol estejam bem misturadas antes da adição, seja por pipetagem para misturar ou por vórtice suave.
- Transferir imediatamente as reações para uma máquina de PCR a 25 °C e incubar durante 30 min. Varie a temperatura e a duração dependendo das condições ideais para a atividade enzimática.
- Extinguir as reações adicionando 5 μL de corante de carga (95% de formamida, ácido etilenodiaminotetracético 0,5 M (EDTA), azul de bromofenol) e incubar a 95 °C por 5 min.
4. Análise dos resultados do ensaio
- Prepare os géis Tris-Borato-EDTA (TBE)-Ureia PAGE conforme descrito abaixo.
- Prepare um estoque de acrilamida a 20%, 7 M de ureia e 1x solução de TBE. Para o conjunto de oligonucleotídeos descrito aqui, use solução de acrilamida/bis na proporção de 29:1 para obter a resolução ideal.
- Para um gel, combine 10 mL de acrilamida a 20% e solução de ureia 7 M com 100 μL de APS (10%) e 3 μL de tetrametiletilenodiamina (TMED) e molde em um rodízio de gel.
- Depois que o gel solidificar, execute as amostras no gel de ureia TBE a 45 - 55 °C.
- Pré-execute o gel em 1x tampão TBE por 30 min a 10 mA por gel com aquecimento externo.
- Remova o excesso de uréia nos poços do gel lavando com 1x TBE usando uma pipeta de pastagem.
- Carregue 10 μL de cada reação e execute a 10 mA por 1,0-1,5 h com aquecimento externo.
- Visualize o gel no gerador de imagens com as configurações corretas para o fluoróforo escolhido. Para FAM, use um conjunto de filtros que forneça excitação/emissão a 495/519 nm, que é armazenado como uma predefinição na maioria dos geradores de imagens.
- Quantifique a intensidade da banda do produto e do substrato usando um software de processamento de imagem com o gerador de imagens ou um programa externo, como o ImageJ49,50, e calcule a porcentagem do produto usando a fórmula
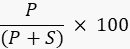
Onde P é o valor integrado da banda do produto e S é a área integrada da banda do substrato. No caso do exemplo da reação de DNA ligase, a banda do produto corre a 40 nucleotídeos (nt) e a banda do substrato a 20 nt.
5. Desadenilação da DNA ligase para testar a especificidade do cofator
- Preparação de master mixes de reação
- Preparar um conjunto de mistura principal contendo o oligonucleótido NL1 marcado com FAM, conforme descrito no quadro 4. Preparar um segundo conjunto contendo o oligonucleótido NL1 sem marcação FAM, conforme descrito no quadro 4.
- Separadamente, aqueça ambos os duplex de DNA a 95 ° C por 5 min e resfrie por 30 min a 1 h a 25 ° C. Não adicione cofator de nucleotídeo a nenhum dos master mixs.
- Montagem e início da reação de desadenilação
- Preparar uma única reacção de desadenilação para cada tipo/concentração de cofactor a ensaiar, combinando 10 μl da mistura principal não marcada com 2,5 μl de enzima ligase.
- Prepare tubos adicionais como controle sem cofator e sem controle de proteína (2,5 μL de tampão adicionado no lugar da enzima).
- Incubar as reações a uma temperatura específica para a atividade ideal da enzima, por 1-2 h. O tempo de incubação pode ser aumentado se a enzima ainda estiver adenilada
- Execute a reação de ligação com o cofator.
- Adicione 10 μL da mistura principal marcada e 2,5 μL dos cofatores de nucleotídeos desejados (por exemplo, ATP, NAD, ADP ou GTP) diretamente à reação desadenilada (concentração final de 0,1-1 mM).
- Adicione 2,5 μL de tampão de reação ao controle de cofator sem nucleotídeo.
- Incube as reações pelo mesmo período de tempo e temperatura usados anteriormente. Resfrie e visualize conforme descrito na etapa 4.
6. Usando substratos duplamente rotulados para ligadura imobilizada ou montagem de várias partes
- Projete e compre um oligonucleotídeo com uma porção fluorescente que tenha um espectro de excitação/emissão diferente do fluoróforo já usado.
- Na configuração descrita aqui, use o oligonucleotídeo NL2 (TAMRA) com 5-carboxitetrametilrodamina (TAMRA) na extremidade 3 '(Tabela 1).
- Monte a mistura master conforme descrito abaixo.
- Combine os componentes da reação descritos na etapa 2, incluindo proporções equimolares de todos os oligonucleotídeos usados na montagem, bem como cátions tampão e divalentes.
- Recozimento por aquecimento a 95 °C por 5 min e resfriamento a 25 °C por 30 min - 1 h. Adicione o cofator e a enzima e incube conforme descrito na etapa 3.
- Execute e crie imagens das amostras conforme descrito na etapa 4 usando os canais apropriados para o par de fluoróforos no substrato. No caso de FAM e TAMRA, esses são os canais de fluoresceína (FITC) e tetrametil rodamina (TRITC) presentes na maioria dos imageadores.
7. Avaliação da ligação ao DNA por Electrophoretic Mobility Shift Assay (EMSA) em gel nativo
- Prepare um gel TBE PAGE nativo a 10% conforme descrito abaixo.
- Combine 2,5 mL de acrilamida a 40%, 1 mL de 10x TBE, 100 μL de APS a 10%, 3 μL de TMED e 6,5 mL de água MQ e molde em um rodízio de gel.
- Monte a reação de ligação conforme descrito abaixo.
- Monte o substrato EMSA de acordo com a Tabela 5 de modo que o EDTA (10 mM) seja incluído e os íons metálicos sejam omitidos.
- Combine 20 μL da mistura principal de substrato EMSA com 5 μL da proteína em um tubo de PCR. Inclua uma amostra de controle sem proteína. Incubar durante 30 min a 25 °C.
- Analise por eletroforese nativa conforme descrito abaixo.
- Adicione 5 μL de corante de carga nativo 5x (EDTA 100 mM, azul de bromofenol a 0,25%, glicerol a 25% v/v e água MQ até 1 mL) às amostras.
- Carregue o gel preparado e deixe funcionar a 60 V por 2-3 h com resfriamento por circulação de água até que a frente do corante esteja alguns centímetros acima da extremidade do gel.
- Visualize e analise os géis conforme descrito na etapa 4.
Access restricted. Please log in or start a trial to view this content.
Resultados
Ligação por DNA ligase
A atividade enzimática da DNA ligase resultará em um aumento no tamanho do oligonucleotídeo marcado com fluorescência quando visualizado em um gel PAGE de ureia. No caso dos substratos para ligação de DNA e RNA listados na Tabela 2, isso corresponde a uma duplicação de tamanho de 20 nt para 40 nt (Figura 3A). A atividade enzimática ideal pode ser determinada alterando as condições, como temperatura, concentração de pr...
Access restricted. Please log in or start a trial to view this content.
Discussão
Etapas críticas no protocolo
Projeto e compra de oligonucleotídeos: Ao comprar os oligonucleotídeos para formação duplex, é essencial considerar o design da sequência. Recomenda-se o uso de uma ferramenta de analisador de oligo para prever as propriedades da sequência de nucleotídeos, como conteúdo de GC, temperatura de fusão, estrutura secundária e potencial de dimerização, antes de solicitar57.
Montagem e recozimento de duplexes de á...
Access restricted. Please log in or start a trial to view this content.
Divulgações
A SEG e a UR são funcionários da ArcticZymes Technologies AS, que distribui a ligase R2D. AW, ER-S e RS não têm interesses concorrentes.
Agradecimentos
AW é apoiado por uma Rutherford Discovery Fellowship (20-UOW-004). RS recebeu uma bolsa de estudos da New Zealand Post Antarctic. A SG e a UR agradecem ao Instituto de Química da Universidade de Tromsø - Universidade Norueguesa do Ártico pelo suporte técnico.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
Referências
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados