Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Methods Article
Uso de oligonucleótidos sintéticos modificados para analizar enzimas metabolizadoras de ácidos nucleicos
En este artículo
Resumen
Aquí, se presenta un protocolo para analizar las enzimas metabolizadoras de ácidos nucleicos, utilizando ejemplos de enzimas ligasa, nucleasa y polimerasa. El ensayo utiliza oligonucleótidos marcados con fluorescencia y sin marcar que pueden combinarse para formar dúplex que imitan el ARN y/o los daños en el ADN o los intermediarios de la vía, lo que permite la caracterización del comportamiento de las enzimas.
Resumen
La disponibilidad de una gama de oligonucleótidos sintéticos modificados de proveedores comerciales ha permitido el desarrollo de ensayos sofisticados para caracterizar diversas propiedades de las enzimas metabolizadoras de ácidos nucleicos que se pueden ejecutar en cualquier laboratorio de biología molecular estándar. El uso de etiquetas fluorescentes ha hecho que estos métodos sean accesibles para los investigadores con un equipo de electroforesis PAGE estándar y un generador de imágenes habilitado para fluorescentes, sin usar materiales radiactivos ni requerir un laboratorio diseñado para el almacenamiento y la preparación de materiales radiactivos, es decir, un laboratorio caliente. La adición opcional de modificaciones estándar, como la fosforilación, puede simplificar la configuración del ensayo, mientras que la incorporación específica de nucleótidos modificados que imitan daños en el ADN o intermedios se puede utilizar para sondear aspectos específicos del comportamiento de las enzimas. Aquí, se demuestra el diseño y la ejecución de ensayos para interrogar varios aspectos del procesamiento del ADN por enzimas utilizando oligonucleótidos sintéticos disponibles comercialmente. Estos incluyen la capacidad de las ligasas para unirse o de las nucleasas para degradar diferentes estructuras híbridas de ADN y ARN, el uso diferencial de cofactores por parte de la ADN ligasa y la evaluación de la capacidad de unión al ADN de las enzimas. Se analizan los factores a tener en cuenta al diseñar sustratos de nucleótidos sintéticos y se proporciona un conjunto básico de oligonucleótidos que se pueden utilizar para una variedad de ensayos de ligasas de ácidos nucleicos, polimerasas y enzimas nucleasas.
Introducción
Todas las formas de vida requieren enzimas de procesamiento de ácidos nucleicos para llevar a cabo procesos biológicos fundamentales, como la replicación, la transcripción y la reparación del ADN. Las funcionalidades enzimáticas clave para estas vías son las polimerasas, que generan copias de moléculas de ARN/ADN, las ligasas que se unen a sustratos de polinucleótidos, las nucleasas que los degradan, y las helicasas y topoisomerasas, que funden dúplex de ácidos nucleicos o cambian su topología 1,2,3,4,5,6,7,8,9,10 . Además, muchas de estas enzimas proporcionan herramientas moleculares esenciales para aplicaciones como la clonación, el diagnóstico y la secuenciación de alto rendimiento 11,12,13,14,15.
Las características funcionales, cinéticas y especificidades del sustrato de estas enzimas se pueden determinar utilizando sustratos de ADN/ARN marcados producidos por oligonucleótidos de recocido. El seguimiento de sustratos y productos se ha logrado tradicionalmente mediante la introducción de una etiqueta radiactiva (32P) en el extremo de la hebra 5', que luego puede ser detectada por película fotográfica o con un sistema de imagen de fósforo16,17. Si bien los sustratos radiomarcados ofrecen el beneficio de una mayor sensibilidad experimental y no alteran las propiedades químicas de un nucleótido, los peligros potenciales para la salud de trabajar con radioisótopos han alentado el desarrollo de marcadores de ácidos nucleicos no radiactivos para proporcionar una alternativa más segura para la detección de ADN y ARN 18,19,20. Entre estos, la detección de fluorescencia, incluida la detección directa de fluorescencia, la fluorescencia resuelta en el tiempo y los ensayos de transferencia de energía/extinción de fluorescencia, se destacan como los más versátiles 21,22,23,24. La amplia gama de fluoróforos permite diferentes diseños de sustratos de ADN/ARN con reporteros únicos en cada oligonucleótido25. Además, la estabilidad de los fluoróforos, en comparación con los radioisótopos, permite a los usuarios producir y preservar cantidades significativas de sustratos de ADN marcados con fluorescencia19. Estos sustratos marcados con fluoróforos se pueden incubar con la proteína de interés, junto con diferentes combinaciones de cofactores metálicos y nucleótidos, para analizar la actividad de unión y/o enzimática. La visualización de la unión o la actividad se puede observar utilizando varios canales de colorante de fluoróforo con un sistema de imágenes en gel. Dado que solo los oligonucleótidos marcados con fluorescencia serán visibles con esta técnica, cualquier aumento o disminución en el tamaño del oligonucleótido marcado será fácil de seguir. Los geles también se pueden teñir después, con tintes de tinción de ácidos nucleicos para visualizar todas las bandas de ADN presentes en el gel.
Las ligasas de ácidos polinucleicos son enzimas que unen fragmentos de ADN/ARN, catalizando el sellado de roturas mediante la formación de un enlace fosfodiéster entre los extremos 5' del ADN fosforilado y el 3' OH del ADN. Se pueden dividir en dos grupos según su requerimiento de sustrato de nucleótidos. Las ligasas dependientes de NAD altamente conservadas se encuentran en todas las bacterias26, mientras que las enzimas dependientes de ATP estructuralmente diversas pueden identificarse en todos los dominios de la vida 8,27. Las ligasas de ADN desempeñan un papel importante en el procesamiento de fragmentos de Okazaki durante la replicación, además de estar involucradas en varias vías de reparación del ADN, como la reparación por escisión de nucleótidos y bases, a través del sellado de muescas espontáneas y muescas que quedan después de la reparación 8,10. Las diferentes ligasas de ADN exhiben capacidades variables para unirse a diferentes conformaciones de roturas de ADN, incluidas muescas en un dúplex, roturas de doble cadena, desajustes y brechas, así como híbridos de ARN y ADN 28,29,30. Se puede ensamblar una amplia gama de sustratos ligables mediante el recocido de oligonucleótidos con un fosfato 5' para generar terminales 5' y 3' yuxtapuestos en un dúplex de ácido nucleico 31,32,33. El método de análisis más común es la resolución por urea PAGE en un formato de ensayo de punto final; Sin embargo, las innovaciones recientes han incluido el uso de la electroforesis en gel capilar, que permite un alto rendimiento34, el perfil de espectrometría de masas35, así como un ensayo de baliza molecular homogénea, que permite el monitoreo resuelto en el tiempo36.
El primer paso en una reacción de ligadura es la adenilación de la enzima ligasa por trifosfato de adenosina (ATP) o dinucleótido de nicotinamida y adenina (NAD), lo que da como resultado un intermediario enzimático covalente. El segundo paso en la reacción es la adenilación del sustrato de ácido nucleico en el extremo 5' del sitio de la muesca, que es seguida por la ligadura de las hebras de ácido nucleico. Muchas enzimas ligasas que se expresan recombinantemente en E. coli se purifican en forma adenilada y, por lo tanto, pueden ligar con éxito ácidos nucleicos sin la adición de un cofactor de nucleótidos. Esto hace que sea difícil determinar qué tipo particular de cofactor de nucleótidos requieren para la ligadura de ácidos nucleicos. Además de describir los ensayos para evaluar la actividad de la ADN ligasa, también se presenta un método para determinar de forma fiable el uso del cofactor mediante la desadenilación de la enzima utilizando sustratos no marcados.
Las nucleasas son un grupo grande y diverso de enzimas modificadoras de ADN/ARN y ARN catalítico que escinden los enlaces fosfodiéster entre los ácidos nucleicos37. Las funcionalidades de la enzima nucleasa son necesarias en la replicación, reparación y procesamiento del ARN del ADN y se pueden clasificar por su especificidad de azúcar para el ADN, el ARN o ambos. Las endonucleasas hidrolizan los enlaces fosfodiéster dentro de una cadena de ADN/ARN, mientras que las exonucleasas hidrolizan las cadenas de ADN/ARN un nucleótido a la vez desde el extremo 3' o 5' y pueden hacerlo desde el extremo 3' a 5' o el extremo 5' a 3' del ADN38.
Mientras que muchas proteínas nucleasas son inespecíficas y pueden estar involucradas en múltiples procesos, otras son altamente específicas para una secuencia particular o daño en el ADN 6,39,40. Las nucleasas de secuencia específica se utilizan en una amplia gama de aplicaciones biotecnológicas, como la clonación, la mutagénesis y la edición del genoma. Las nucleasas populares para estas aplicaciones son las nucleasasde restricción 41, las nucleasas de dedos de zinc42, las nucleasas efectoras de tipo activador transcripcional y, más recientemente, las nucleasas CRISPR43 diseñadas guiadas por ARN. Recientemente se han identificado nucleasas específicas de daño, como la nucleasa EndoMS, que tiene especificidad para las discordancias en el ADN a través de su dominiode nucleasa similar a RecB específico de discordancia 5,44. Los ensayos de actividad nucleasa, históricamente, se han realizado como ensayos discontinuos con sustratos radiomarcados; Sin embargo, además de sus otros inconvenientes, estos no permiten la identificación del sitio que es cortado por una proteína nucleasa, lo que es posible cuando se utilizan sustratos marcados con fluorescencia 45,46. Más recientemente, se han desarrollado ensayos continuos de nucleasas que funcionan mediante el uso de diferentes tintes de ADN que interactúan con el ADN en diferentes estados; por ejemplo, emitiendo una señal fluorescente más alta al interactuar con el ADNd que en su estado no unido, o uniéndose específicamente a ARN cortos47. Otros ensayos de nucleasa continua utilizan horquillas de ADN con un grupo fluoróforo en el extremo 5' y un extintor en el extremo 3' para que la fluorescencia aumente a medida que el oligonucleótido se degrada debido a una separación del fluoróforo y el extintor. Si bien estos ensayos permiten caracterizar la cinética de las proteínas que degradan el ADN, requieren un conocimiento previo de la función y el sustrato de la enzima y también se limitan a las enzimas que cambian la conformación del ADN para causar una diferencia en la unión del colorante. Por esta razón, los ensayos de punto final que resuelven productos individuales de nucleasa siguen siendo deseables para proporcionar información sobre las modificaciones del ADN causadas por la actividad de las proteínas.
Aquí, se presenta un procedimiento detallado para el diseño de oligonucleótidos de ADN/ARN marcados con fluorescencia que se pueden mezclar y combinar para generar sustratos para probar la actividad de nuevas enzimas nucleasa, polimerasa y ligasa. La validación de este conjunto básico de secuencias de oligonucleótidos simplifica el diseño experimental y facilita el perfilado económico de una amplia gama de funcionalidades enzimáticas sin necesidad de comprar un gran número de sustratos a medida. Se proporciona un procedimiento detallado para ejecutar un ensayo estándar de enzimas de procesamiento de ADN con estos sustratos, utilizando el ejemplo de la actividad de la ADN ligasa y se describen las modificaciones para analizar las enzimas nucleasa y polimerasa. Además, se proporciona un ensayo modificado para determinar la especificidad del cofactor de la enzima ADN ligasa con alta precisión, y se utilizan sondas de doble marcado para evaluar el ensamblaje de ligaduras multicomponente. Finalmente, se discuten las modificaciones al formato básico del ensayo para permitir su uso para determinar las interacciones proteína-ADN con los mismos sustratos mediante el ensayo de cambio de movilidad electroforética (EMSA).
Protocolo
1. Diseño y compra de oligonucleótidos
NOTA: Diseñe oligonucleótidos monocatenarios para ensamblarlos y recocerlos en los dúplex deseados. Una o más de las hebras de un dúplex deben tener una fracción fluorescente para rastrear el procesamiento de oligonucleótidos por parte de la enzima de interés. En el Cuadro 1 se proporciona un conjunto básico de secuencias monocatenarias que pueden ensamblarse para una serie de actividades.
- Incorpore las modificaciones específicas necesarias para la enzima de interés como se describe a continuación.
- Para sustratos de ADN ligasa (Figura 1): Ensamble el sustrato más simple a partir de tres oligonucleótidos: una hebra donante fosforilada 5' (NL2), una hebra aceptora marcada con FAM 5' (NL1) y un complemento que une las dos (NL3).
- Asegúrese de que las hebras que proporcionan el extremo 5' de la muesca ligable estén fosforiladas antes del ensamblaje de la mezcla maestra de sustrato en el paso 2. Solicite esto como una modificación en NL2 (como se muestra en la Tabla 1) o use la fosforilación enzimática con la polinucleótido quinasa T4 después de resuspender los oligonucleótidos.
- Incluir la fosforilación 5'-terminal de NL6 y NL8, que comprenden el complemento de las roturas de doble cadena representadas en la Figura 1A (NL6/NL7 y NL8/NL9), ya que es la que más se asemeja al sustrato natural producido a partir de una endonucleasa de restricción. Utilice un sustrato de doble etiqueta para determinar los alcances relativos de la ligadura para ensamblajes de varias partes (consulte el paso 6).
- Alterar la hebra del complemento para producir desajustes (NL10) y espacios (NL11).
NOTA: Las variaciones en el sustrato mellado simple se representan en la Figura 1A. Es posible utilizar otras secuencias para producir una gama aún más amplia de desajustes o espacios más largos variando la posición subrayada. - Oligonucleótidos de ADN sustitutivos de oligonucleótidos de ARN.
NOTA: Las variaciones en el sustrato mellado simple se muestran en la Figura 1B. Se puede generar una gama más amplia de dúplex de ADN/ARN mediante combinaciones adicionales del conjunto de bases que se proporciona aquí para generar, por ejemplo, roturas de doble cadena que contienen ARN y ADN. Un ejemplo de esta variación se da en el paso 6 a continuación, donde se utiliza una estrategia de doble etiqueta.
- Para sustratos de ADN polimerasa: Ensamble los oligonucleótidos NL1 y NL3 enumerados en la Tabla 1 para proporcionar un ensayo simple de extensión de cebador. Investigar aspectos adicionales de la actividad de la polimerasa mediante la introducción de modificaciones en las hebras NL1 (cebador) o NL3 (plantilla).
- Incorporar análogos de bases dañadas en el oligonucleótido NL3 antes de la posición 20 para determinar la capacidad de evitar las lesiones dañadas en la hebra molde.
- Incorpore análogos de base dañados en el oligonucleótido NL1 en la posición 20 para determinar la capacidad de extender un cebador dañado.
- Utilice RNL1 o RNL3 en el dúplex para investigar la extensión de un cebador de ARN o el uso de una plantilla de ARN.
- Para sustratos de nucleasas (Figura 2): Ensamble oligonucleótidos para proporcionar una gama no exhaustiva de sustratos bicatenarios y monocatenarios (Figura 2Ai), así como una gama de uniones aleteadas y separadas (Figura 2Aii) y sustratos dañados (Figura 2B).
- Para sondear las actividades de la ribonucleasa, sustituya iterativamente NL1, NL2 y NL3 por RNL1, RNL2 y RNL3. Utilice versiones adicionales de ARN de HJ5 y HJ6 para ampliar aún más este conjunto.
- Utilice los oligonucleótidos MD5, MD6 y MD9 que tienen una modificación situada en el centro que imita el daño oxidativo, un intermediario de reparación abásico o un producto de desaminación (Figura 2B). Los sustratos detectarán la escisión de la hebra en esta posición. Etiquete la hebra NL3 del complemento con un fluoróforo ortogonal como TAMRA para detectar la escisión de doble hebra (consulte el paso 6).
- Utilice el marcaje ortogonal del complemento para detectar cortes de doble cadena en los sitios no coincidentes en las hebras de la sonda (NL5 y ND9) y del complemento (MD10 y NL10).
- Para sustratos de ADN ligasa (Figura 1): Ensamble el sustrato más simple a partir de tres oligonucleótidos: una hebra donante fosforilada 5' (NL2), una hebra aceptora marcada con FAM 5' (NL1) y un complemento que une las dos (NL3).
- Pida oligonucleótidos sintéticos que incorporen fluoróforos relevantes y otras modificaciones a un proveedor comercial.
NOTA: Una escala de síntesis de 100 nM y la purificación por HPLC posterior a la síntesis son adecuadas para los ensayos descritos.
2. Ensamblaje y recocido de dúplex de ácido nucleico
- Resuspensión y dilución de oligonucleótidos
- Antes de abrir, centrifugar los oligonucleótidos liofilizados en sus tubos de 2 ml a toda velocidad en una centrífuga de sobremesa durante 2-5 minutos para asegurarse de que el ácido nucleico esté en el fondo del tubo.
- Prepare un material maestro de 100 μM resuspendiendo los oligonucleótidos en tampón TE (10 mM de tris(hidroximetil)aminometano (Tris), 1 mM de ácido etilendiaminotetraacético (EDTA)). Asegúrese de que los oligonucleótidos se resuspendan completamente mediante un vórtice suave repetido y una centrifugación breve a toda velocidad.
- Prepare un stock de 10 μM diluyendo una alícuota de material maestro con tampón TE. Diluya el material de 10 μM con agua ultrapura (agua MQ) para preparar material de trabajo con concentraciones de 0,5 μM, 0,7 μM o 2,5 μM según la Tabla 2.
- Montaje y recocido de las mezclas maestras de reacción
- Utilice existencias de trabajo para formar las mezclas maestras de reacción utilizando las combinaciones proporcionadas en la Tabla 2 y los volúmenes indicados en la Tabla 3. Para el ensayo estándar de ADN ligasa y la mayoría de los otros ensayos descritos aquí, la composición final del tampón es 50 mM de Tris pH 8.0, 50 mM de NaCl, 10 mM de ditiotreitol (DTT) con 10 mM de Mg como catión divalente.
- Recocer los oligonucleótidos en un tubo de PCR o microcentrífuga calentándolos a 95 °C durante 5 min utilizando un bloque calefactor o un termociclador. Deje enfriar a temperatura ambiente durante 30 min (volúmenes <1 mL) a 1 h (volúmenes >1 mL). Para oligonucleótidos más largos (>40 nt), realice un enfriamiento más lento utilizando un termociclador con una rampa descendente de 95 °C a 25 °C durante 45 min, o haga flotar el tubo que contiene la mezcla de recocido en un vaso de precipitados de 1 L con agua hirviendo y deje enfriar a temperatura ambiente durante la noche.
- Agregue cofactores de nucleótidos y otros componentes tampón sensibles al calor a la mezcla maestra después de enfriar a temperatura ambiente. Utilice la mezcla de reacción final directamente para el ensayo mediante la adición de enzima (consulte el paso 3 a continuación) o almacene a -20 °C para su uso futuro.
3. Configuración estándar del ensayo
- Montaje e inicio de la reacción del ensayo
- Combine 22,5 μL de la mezcla maestra de sustrato de interés con 2,5 μL de la ADN ligasa u otra enzima de interés en un tubo de PCR. Ejecute reacciones por duplicado o triplicado, especialmente si los resultados se van a cuantificar.
- Incluya un control sin proteínas (solo tampón) en las muestras del ensayo. No incluya controles de cofactor en este punto, si es necesario.
NOTA: Las enzimas pueden almacenarse a -20 °C en glicerol al 50% v/v, lo que permite pipetearlas directamente de la solución. Asegúrese de que las soluciones enzimáticas con glicerol estén bien mezcladas antes de la adición, ya sea pipeteando para mezclar o mediante un vórtice suave.
- Transfiera inmediatamente las reacciones a una máquina de PCR a 25 °C e incube durante 30 min. Variar la temperatura y la duración en función de las condiciones óptimas para la actividad enzimática.
- Apagar las reacciones añadiendo 5 μL de colorante de carga (formamida al 95%, ácido etilendiaminotetraacético (EDTA) 0,5 M, azul de bromofenol) e incubar a 95 °C durante 5 min.
4. Análisis de los resultados de los ensayos
- Prepare los geles de Tris-Borato-EDTA (TBE)-Urea PAGE como se describe a continuación.
- Prepare un caldo de acrilamida al 20%, urea 7 M y 1 solución de TBE. Para el conjunto de oligonucleótidos descrito aquí, utilice la solución de acrilamida/Bis en una proporción de 29:1 para obtener una resolución óptima.
- Para un gel, combine 10 ml de acrilamida al 20% y una solución de urea al 7 M con 100 μl de APS (10 %) y 3 μl de tetrametilendidiamina (TMED) y colóquelo en una fundición de gel.
- Después de que el gel se solidifique, pase las muestras en el gel de urea TBE a 45 - 55 °C.
- Ejecute previamente el gel en 1x tampón TBE durante 30 minutos a 10 mA por gel con calentamiento externo.
- Elimine el exceso de urea en los pocillos del gel enjuagando con 1x TBE con una pipeta de pasto.
- Cargue 10 μL de cada reacción y funcione a 10 mA durante 1,0-1,5 h con calentamiento externo.
- Visualice el gel en el generador de imágenes con la configuración correcta para el fluoróforo elegido. Para FAM, utilice un conjunto de filtros que proporcione excitación/emisión a 495/519 nm, que se almacena como un ajuste preestablecido en la mayoría de los generadores de imágenes.
- Cuantifique la intensidad de la banda del producto y el sustrato utilizando un software de procesamiento de imágenes con el generador de imágenes, o un programa externo como ImageJ49,50 y calcule el porcentaje del producto utilizando la fórmula
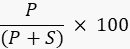
Donde P es el valor integrado de la banda del producto y S es el área integrada de la banda del sustrato. En el caso del ejemplo de la reacción de ADN ligasa, la banda del producto corre a 40 nucleótidos (nt) y la banda del sustrato a 20 nt.
5. Deadenilación de la ADN ligasa para probar la especificidad del cofactor
- Preparación de mezclas maestras de reacción
- Prepare un juego de la mezcla maestra que contenga el oligonucleótido NL1 marcado con FAM, como se describe en la Tabla 4. Prepare un segundo juego que contenga el oligonucleótido NL1 sin marcador FAM, como se describe en la Tabla 4.
- Por separado, caliente ambos dúplex de ADN a 95 °C durante 5 minutos y enfríe de 30 minutos a 1 h a 25 °C. No añada cofactor de nucleótidos a ninguna de las mezclas maestras.
- Montaje e inicio de la reacción de desadenilación
- Prepare una única reacción de desadenilación para cada tipo/concentración de cofactor que se vaya a probar combinando 10 μL de la mezcla maestra no marcada con 2,5 μL de enzima ligasa.
- Prepare tubos adicionales como control sin cofactor y sin control de proteínas (se agregaron 2,5 μL de tampón en lugar de enzima).
- Incubar las reacciones a una temperatura específica para la actividad óptima de la enzima, durante 1-2 h. El tiempo de incubación puede aumentarse si la enzima aún está adenilada.
- Ejecute la reacción de ligadura con el cofactor.
- Añada 10 μL de la mezcla maestra marcada y 2,5 μL de los cofactores de nucleótidos deseados (por ejemplo, ATP, NAD, ADP o GTP) directamente a la reacción desadenilada (concentración final de 0,1-1 mM).
- Añada 2,5 μL de tampón de reacción al control sin cofactor de nucleótidos.
- Incubar las reacciones durante el mismo período de tiempo y temperatura que se utilizaron anteriormente. Apague y visualice como se describe en el paso 4.
6. Uso de sustratos de doble etiqueta para la ligadura entablillada o el ensamblaje de varias piezas
- Diseñar y comprar un oligonucleótido con una fracción fluorescente que tenga un espectro de excitación/emisión diferente al fluoróforo ya utilizado.
- En la configuración descrita aquí, use el oligonucleótido NL2 (TAMRA) que tiene 5-carboxitetrametilrodamina (TAMRA) en el extremo 3' (Tabla 1).
- Ensamble la mezcla maestra como se describe a continuación.
- Combine los componentes de la reacción descrita en el paso 2, incluidas las proporciones equimolares de todos los oligonucleótidos utilizados en el ensamblaje, así como los cationes tampón y divalentes.
- Recocido calentando a 95 °C durante 5 min y enfriando a 25 °C durante 30 min - 1 h. Agregue el cofactor y la enzima e incube como se describe en el paso 3.
- Ejecute y obtenga imágenes de las muestras como se describe en el paso 4 utilizando los canales apropiados para el par de fluoróforos en el sustrato. En el caso de FAM y TAMRA, estos son los canales de fluoresceína (FITC) y tetrametilrodamina (TRITC) presentes en la mayoría de los generadores de imágenes.
7. Evaluación de la unión al ADN mediante el ensayo de cambio de movilidad electroforética (EMSA) en gel nativo
- Prepare un 10% de gel nativo de TBE PAGE como se describe a continuación.
- Combine 2,5 ml de acrilamida al 40 %, 1 ml de 10x TBE, 100 μl de APS al 10 %, 3 μl de TMED y 6,5 ml de agua MQ y colóquelo en una rueda de gel.
- Ensamble la reacción de unión como se describe a continuación.
- Ensamble el sustrato EMSA de acuerdo con la Tabla 5 de modo que se incluya EDTA (10 mM) y se omitan los iones metálicos.
- Combine 20 μL de la mezcla maestra de sustrato de EMSA con 5 μL de la proteína en un tubo de PCR. Incluya una muestra de control sin proteínas. Incubar durante 30 min a 25 °C.
- Analice mediante electroforesis nativa como se describe a continuación.
- Añada 5 μL de colorante de carga nativa 5x (100 mM de EDTA, 0,25 % de azul de bromofenol, 25 % v/v de glicerol y MQ de agua hasta 1 mL) a las muestras.
- Cargue el gel preparado y haga funcionar a 60 V durante 2-3 h con enfriamiento por circulación de agua hasta que el frente del tinte esté unos cm por encima del extremo del gel.
- Visualice y analice los geles como se describe en el paso 4.
Resultados
Ligadura por ADN ligasa
La actividad enzimática de la ADN ligasa dará como resultado un aumento en el tamaño del oligonucleótido marcado con fluorescencia cuando se visualice en un gel de urea PAGE. En el caso de los sustratos para la ligadura de ADN y ARN enumerados en la Tabla 2, esto corresponde a una duplicación de tamaño de 20 nt a 40 nt (Figura 3A). La actividad enzimática óptima puede determinarse cambiando condiciones como la temperatura, ...
Discusión
Pasos críticos en el protocolo
Diseño y compra de oligonucleótidos: Al comprar los oligonucleótidos para la formación de dúplex, es esencial considerar el diseño de secuencias. Se recomienda utilizar una herramienta de análisis de oligonucleótidos para predecir las propiedades de la secuencia de nucleótidos, como el contenido de GC, la temperatura de fusión, la estructura secundaria y el potencial de dimerización, antes de realizar el pedido57.
Divulgaciones
SEG y UR son empleados de ArcticZymes Technologies AS, que distribuye la ligasa R2D. AW, ER-S y RS no tienen intereses contrapuestos.
Agradecimientos
AW cuenta con el apoyo de una beca Rutherford Discovery (20-UOW-004). RS es el beneficiario de una beca postantártica de Nueva Zelanda. SG y UR agradecen al Instituto de Química de la Universidad de Tromsø - la Universidad Ártica Noruega por su apoyo técnico.
Materiales
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
Referencias
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003 (2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009 (2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263 (2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125 (2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391 (2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062 (2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14 (2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2 (2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148 (2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389 (2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097 (2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853 (2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52 (2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817 (2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97, 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711 (2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485 (2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998 (2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62 (2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257 (2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados