Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Nükleik asit metabolize edici enzimleri test etmek için modifiye sentetik oligonükleotidlerin kullanılması
Bu Makalede
Özet
Burada, ligaz, nükleaz ve polimeraz enzimlerinin örnekleri kullanılarak nükleik asit metabolize edici enzimlerin tahlili için bir protokol sunulmaktadır. Test, enzim davranışının karakterizasyonuna izin veren, RNA ve/veya DNA hasarlarını veya yol ara ürünlerini taklit eden dupleksler oluşturmak için birleştirilebilen floresan etiketli ve etiketsiz oligonükleotidleri kullanır.
Özet
Ticari satıcılardan bir dizi modifiye sentetik oligonükleotidin mevcudiyeti, herhangi bir standart moleküler biyoloji laboratuvarında çalıştırılabilen nükleik asit metabolize edici enzimlerin çeşitli özelliklerini karakterize etmek için karmaşık tahlillerin geliştirilmesine izin vermiştir. Floresan etiketlerin kullanılması, bu yöntemleri, radyoaktif malzemeler kullanmadan veya radyoaktif malzemelerin depolanması ve hazırlanması için tasarlanmış bir laboratuvar, yani bir Sıcak Laboratuvar gerektirmeden, standart PAGE elektroforez ekipmanı ve floresan özellikli bir görüntüleyiciye sahip araştırmacılar için erişilebilir hale getirmiştir. Fosforilasyon gibi standart modifikasyonların isteğe bağlı olarak eklenmesi, tahlil kurulumunu basitleştirebilirken, DNA hasarlarını veya ara ürünleri taklit eden modifiye edilmiş nükleotidlerin spesifik olarak dahil edilmesi, enzim davranışının belirli yönlerini araştırmak için kullanılabilir. Burada, ticari olarak temin edilebilen sentetik oligonükleotidler kullanılarak enzimler tarafından DNA işlemenin çeşitli yönlerini sorgulamak için tahlillerin tasarımı ve yürütülmesi gösterilmektedir. Bunlar, ligazların farklı DNA ve RNA hibrit yapılarını bozmak için ligazların birleşme veya nükleazların yeteneğini, DNA ligaz tarafından diferansiyel kofaktör kullanımını ve enzimlerin DNA bağlama kapasitesinin değerlendirilmesini içerir. Sentetik nükleotid substratları tasarlanırken göz önünde bulundurulması gereken faktörler tartışılır ve bir dizi nükleik asit ligaz, polimeraz ve nükleaz enzim tahlili için kullanılabilecek temel bir oligonükleotid seti sağlanır.
Giriş
Tüm yaşam formları, replikasyon, transkripsiyon ve DNA onarımı dahil olmak üzere temel biyolojik süreçleri gerçekleştirmek için nükleik asit işleme enzimlerine ihtiyaç duyar. Bu yollar için temel enzimatik işlevler, RNA/DNA moleküllerinin kopyalarını üreten polimerazlar, polinükleotid substratlarına katılan ligazlar, bunları bozan nükleazlar ve nükleik asit duplekslerini eriten veya topolojilerini değiştiren helikazlar ve topoizomerazlardır 1,2,3,4,5,6,7,8,9,10 . Ek olarak, bu enzimlerin birçoğu klonlama, teşhis ve yüksek verimli dizileme gibi uygulamalar için gerekli moleküler araçları sağlar 11,12,13,14,15.
Bu enzimlerin fonksiyonel özellikleri, kinetiği ve substrat özgüllükleri, oligonükleotidlerin tavlanmasıyla üretilen etiketli DNA/RNA substratları kullanılarak belirlenebilir. Alt tabakaların ve ürünlerin izlenmesi geleneksel olarak, daha sonra fotoğraf filmi veya bir fosfor görüntüleme sistemi16,17 ile tespit edilebilen 5' iplikçik ucuna bir radyoaktif etiket (32P) yerleştirilerek gerçekleştirilmiştir. Radyoaktif işaretli substratlar, artan deneysel hassasiyet avantajı sunarken ve bir nükleotidin kimyasal özelliklerini değiştirmezken, radyoizotoplarla çalışmaktan kaynaklanan potansiyel sağlık tehlikeleri, DNA ve RNA tespiti için daha güvenli bir alternatif sağlamak için radyoaktif olmayan nükleik asit etiketlemesinin geliştirilmesini teşvik etmiştir 18,19,20. Bunlar arasında, doğrudan floresan algılama, zamana bağlı floresan ve enerji transferi/floresan söndürme testleri dahil olmak üzere floresan algılama, en çok yönlü 21,22,23,24 olarak öne çıkıyor. Geniş florofor dizisi, her oligonükleotid25 üzerinde benzersiz raporlayıcılar içeren farklı DNA/RNA substratları tasarımlarını mümkün kılar. Ek olarak, floroforların stabilitesi, radyoizotoplarla karşılaştırıldığında, kullanıcıların önemli miktarlarda floresan etiketli DNA substratları üretmesine ve korumasına olanak tanır19. Bu florofor etiketli substratlar, bağlanma ve/veya enzim aktivitesini analiz etmek için farklı metal ve nükleotid kofaktör kombinasyonları ile birlikte ilgilenilen protein ile inkübe edilebilir. Jel görüntüleme sistemi ile çeşitli florofor boya kanalları kullanılarak bağlanma veya aktivitenin görselleştirilmesi gözlemlenebilir. Bu teknik kullanılarak yalnızca floresan olarak işaretlenmiş oligonükleotidler görülebileceğinden, etiketli oligonükleotidin boyutundaki herhangi bir artış veya azalmayı takip etmek kolay olacaktır. Jeller daha sonra, jel üzerinde bulunan tüm DNA bantlarını görselleştirmek için nükleik asit boyama boyaları ile de boyanabilir.
Poli-nükleik asit ligazları, DNA/RNA fragmanlarını birleştiren, 5' fosforile DNA terminalleri ile DNA'nın 3' OH'si arasında bir fosfodiester bağı oluşturarak kırılmaların sızdırmazlığını katalize eden enzimlerdir. Nükleotid substrat gereksinimlerine göre iki gruba ayrılabilirler. Yüksek oranda korunmuş NAD'ye bağımlı ligazlar tüm bakterilerdebulunur 26 yapısal olarak çeşitli ATP'ye bağımlı enzimler yaşamın tüm alanlarında tanımlanabilir 8,27. DNA ligazları, replikasyon sırasında Okazaki fragmanının işlenmesinde önemli bir rol oynamanın yanı sıra, onarımdan sonra kalan spontan çentiklerin ve çentiklerin sızdırmazlığı yoluyla nükleotid ve baz eksizyon onarımı gibi çeşitli DNA onarım yollarında yer alır 8,10. Farklı DNA ligazları, çift yönlü çentikler, çift sarmallı kırılmalar, uyumsuzluklar ve boşlukların yanı sıra RNA ve DNA hibritleri 28,29,30 dahil olmak üzere farklı DNA kırılma konformasyonlarını birleştirmek için değişen kapasiteler sergiler. Bir nükleik asit dubleks31,32,33'te yan yana 5' ve 3' terminaller oluşturmak için oligonükleotidlerin 5' fosfat ile tavlanmasıyla çok çeşitli ligatlanabilir substratlar birleştirilebilir. En yaygın analiz yöntemi, bir uç nokta tahlil formatında üre PAGE ile çözünürlüktür; Bununla birlikte, son yenilikler arasında, yüksek verime34, kütle spektrometrik profillemeye35 izin veren kılcal jel elektroforezinin yanı sıra zamana bağlı izlemeye36 izin veren homojen bir moleküler işaret testinin kullanımı yer almaktadır.
Bir ligasyon reaksiyonundaki ilk adım, ligaz enziminin adenozin trifosfat (ATP) veya Nikotinamid adenin dinükleotidi (NAD) ile aderilasyonudur ve bu da bir kovalent enzim ara maddesi ile sonuçlanır. Reaksiyondaki ikinci adım, çentik bölgesinin 5' ucundaki nükleik asit substratının adenilasyonudur, bunu nükleik asit çentik ipliklerinin ligasyonu takip eder. E. coli'de rekombinant olarak eksprese edilen birçok ligaz enzimi, adenillenmiş formda saflaştırılır ve bu nedenle, bir nükleotid kofaktörü eklenmeden nükleik asitleri başarılı bir şekilde bağlayabilir. Bu, nükleik asitlerin ligasyonu için hangi belirli tipte nükleotid kofaktörüne ihtiyaç duyduklarını belirlemeyi zorlaştırır. DNA ligaz aktivitesini değerlendirmek için tahlilleri tanımlamaya ek olarak, etiketlenmemiş substratlar kullanarak enzimi dedenilasyon yaparak kofaktör kullanımını güvenilir bir şekilde belirlemek için bir yöntem de sunulmaktadır.
Nükleazlar, nükleik asitler37 arasındaki fosfodiester bağlarını parçalayan geniş ve çeşitli bir DNA/RNA modifiye edici enzimler ve katalitik RNA'lar grubudur. Nükleaz enzim işlevleri, DNA replikasyonu, onarımı ve RNA işlemede gereklidir ve DNA, RNA veya her ikisi için şeker özgüllüklerine göre sınıflandırılabilir. Endonükleazlar, bir DNA/RNA zinciri içindeki fosfodiester bağlarını hidrolize ederken, eksonükleazlar DNA/RNA zincirlerini 3' veya 5' ucundan her seferinde bir nükleotidi hidrolize eder ve bunu DNA'nın 3' ila 5' veya 5' ila 3' ucundan yapabilir38.
Birçok nükleaz proteini spesifik değildir ve birden fazla süreçte yer alabilirken, diğerleri belirli bir dizi veya DNA hasarı için oldukça spesifiktir 6,39,40. Diziye özgü nükleazlar, klonlama, mutajenez ve genom düzenleme gibi çok çeşitli biyoteknolojik uygulamalarda kullanılır. Bu uygulamalar için popüler nükleazlar, kısıtlama nükleazları41, çinko parmak nükleazları42, transkripsiyonel aktivatör benzeri efektör nükleazlar ve en son olarak, RNA güdümlü tasarlanmış CRISPR nükleazları43'tür. Uyumsuzluğa özgü RecB benzeri nükleaz alanı 5,44 aracılığıyla DNA'daki uyumsuzluklar için özgüllüğe sahip olan EndoMS nükleaz gibi hasara özgü nükleazlar yakın zamanda tanımlanmıştır. Nükleaz aktivite deneyleri, tarihsel olarak, radyoaktif işaretli substratlar ile süreksiz deneyler olarak yapılmıştır; Bununla birlikte, diğer dezavantajlarına ek olarak, bunlar, floresan etiketli substratlar45,46 kullanıldığında mümkün olan bir nükleaz proteini tarafından kesilen bölgenin tanımlanmasına izin vermez. Daha yakın zamanlarda, farklı durumlarda DNA ile etkileşime giren farklı DNA boyaları kullanarak çalışan sürekli nükleaz tahlilleri geliştirilmiştir; örneğin, dsDNA ile etkileşime girdiğinde, bağlanmamış durumundan daha yüksek bir floresan sinyali yaymak veya spesifik olarak kısa RNA'larabağlanmak 47. Diğer sürekli nükleaz tahlilleri, 5' ucunda bir florofor grubu ve 3' ucunda bir söndürücü bulunan DNA saç tokaları kullanır, böylece florofor ve söndürücü48'in ayrılması nedeniyle oligonükleotid bozuldukça floresan artar. Bu tahliller, DNA parçalayıcı proteinlerin kinetiğini karakterize etmeye izin verirken, enzimin işlevi ve substratı hakkında önceden bilgi gerektirir ve ayrıca boya bağlanmasında bir farka neden olmak için DNA konformasyonunu değiştiren enzimlerle sınırlıdır. Bu nedenle, tek tek nükleaz ürünlerini çözen son nokta tahlilleri, protein aktivitesinin neden olduğu DNA modifikasyonları hakkında bilgi sağlamak için hala arzu edilir.
Burada, yeni nükleaz, polimeraz ve ligaz enzimlerinin aktivitesini test etmek için substratlar oluşturmak üzere karıştırılabilen ve eşleştirilebilen floresan etiketli DNA/RNA oligonükleotidlerinin tasarımı için ayrıntılı bir prosedür sunulmaktadır. Bu temel oligonükleotid dizileri setinin doğrulanması, deneysel tasarımı basitleştirir ve çok sayıda ısmarlama substrat satın almaya gerek kalmadan çok çeşitli enzimatik işlevlerin ekonomik profilini çıkarmayı kolaylaştırır. DNA ligaz aktivitesi örneği kullanılarak, bu substratlarla standart bir DNA işleme enzim testinin çalıştırılması için ayrıntılı bir prosedür sağlanmıştır ve nükleaz ve polimeraz enzimlerinin tahlil edilmesi ve analiz edilmesi için modifikasyonlar açıklanmaktadır. Ek olarak, DNA ligaz enziminin kofaktör özgüllüğünü yüksek doğrulukla belirlemek için modifiye edilmiş bir test verilir ve çok bileşenli ligasyonların montajını değerlendirmek için çift etiketli problar kullanılır. Son olarak, elektroforetik hareketlilik kaydırma testi (EMSA) ile aynı substratlarla protein-DNA etkileşimlerini belirlemek için kullanılmasına izin vermek için temel tahlil formatındaki modifikasyonlar tartışılmaktadır.
Access restricted. Please log in or start a trial to view this content.
Protokol
1. Oligonükleotidlerin tasarımı ve satın alınması
NOT: İstenilen dublekslere monte edilecek ve tavlanacak tek sarmallı oligonükleotidler tasarlayın. Bir dubleks içindeki ipliklerden biri veya daha fazlası, ilgilenilen enzim tarafından oligonükleotid işlemesini izlemek için bir floresan parçası taşımalıdır. Bir dizi aktivite için bir araya getirilebilen tek sarmallı dizilerin temel bir seti Tablo 1'de verilmiştir.
- Aşağıda tarif edildiği gibi ilgilenilen enzim için gerekli olan spesifik modifikasyonları dahil edin.
- DNA ligaz substratları için (Şekil 1): Üç oligonükleotidden en basit substratı birleştirin: 5' fosforile donör zincir (NL2), 5' FAM etiketli alıcı zincir (NL1) ve ikisini köprüleyen bir tamamlayıcı (NL3).
- Ligatable çentiğin 5' terminalini sağlayan tellerin, 2. adımda substrat ana karışımının montajından önce fosforile edildiğinden emin olun. Bunu NL2 üzerinde bir modifikasyon olarak sipariş edin ( Tablo 1'de verildiği gibi) veya oligonükleotidleri yeniden süspanse ettikten sonra T4 polinükleotid kinaz ile enzimatik fosforilasyon kullanın.
- Şekil 1A'da (NL6 / NL7 ve NL8 / NL9) gösterilen çift sarmallı kırılmaların tamamlayıcısını içeren NL6 ve NL8'in 5'-terminal fosforilasyonunu içerir, çünkü bu, bir kısıtlama endonükleazından üretilen doğal substrata en çok benzer. Çok parçalı montajlar için ligasyonun göreli kapsamlarını belirlemek için çift etiketli bir alt tabaka kullanın (bkz. adım 6).
- Uyumsuzluklar (NL10) ve boşluklar (NL11) oluşturmak için kompleman ipliğini değiştirin.
NOT: Basit çentikli alt tabaka üzerindeki varyasyonlar Şekil 1A'da gösterilmiştir. Altı çizili konumu değiştirerek daha da geniş bir uyumsuzluk aralığı veya daha uzun boşluklar üretmek için diğer dizileri kullanmak mümkündür. - RNA oligonükleotidleri için DNA oligonükleotidlerini değiştirin.
NOT: Basit çentikli alt tabaka üzerindeki varyasyonlar Şekil 1B'de gösterilmiştir. Daha geniş bir DNA/RNA dupleksleri aralığı, örneğin hem RNA hem de DNA içeren çift sarmallı kırılmalar oluşturmak için burada verilen temel setin ek kombinasyonları ile oluşturulabilir. Bu varyasyonun bir örneği, çift etiketli bir stratejinin kullanıldığı aşağıdaki 6. adımda verilmiştir.
- DNA polimeraz substratları için: Basit bir primer-uzatma testi vermek için Tablo 1'de listelenen oligonükleotidler NL1 ve NL3'ü birleştirin. NL1 (primer) veya NL3 (şablon) ipliklerine modifikasyonlar uygulayarak polimeraz aktivitesinin ek yönlerini araştırın.
- Şablon iplikçik üzerindeki hasarlı lezyonları atlama yeteneğini belirlemek için hasarlı baz analoglarını 20. pozisyondan önce NL3 oligonükleotidine dahil edin.
- Hasarlı bir astarı uzatma yeteneğini belirlemek için hasarlı baz analoglarını 1 pozisyonundaki NL20 oligonükleotidine dahil edin.
- Bir RNA primerinin uzantısını veya bir RNA şablonunun kullanımını araştırmak için dubleks içinde RNL1 veya RNL3 kullanın.
- Nükleaz substratları için (Şekil 2): Oligonükleotidleri, kapsamlı olmayan bir çift ve tek sarmallı substrat aralığı (Şekil 2Ai) ve ayrıca bir dizi kanatlı ve yayvan bağlantı noktası (Şekil 2Aii) ve hasarlı substratlar (Şekil 2B) verecek şekilde birleştirin.
- Ribonükleaz aktivitelerini araştırmak için, yinelemeli olarak NL1, NL2 ve NL3'ü RNL1, RNL2 ve RNL3 ile değiştirin. Bu seti daha da genişletmek için HJ5 ve HJ6'nın ek RNA versiyonlarını kullanın.
- Oksidatif hasarı, abazik bir onarım ara ürününü veya bir deaminasyon ürününü taklit eden merkezi olarak yerleştirilmiş bir modifikasyona sahip oligonükleotidler MD5, MD6 ve MD9'u kullanın (Şekil 2B). Alt tabakalar bu konumda ipliğin bölünmesini algılayacaktır. Çift iplikli bölünmeyi tespit etmek için tamamlayıcı NL3 zincirini TAMRA gibi ortogonal bir florofor ile etiketleyin (bkz. adım 6).
- Hem prob (NL5 ve ND9) hem de kompleman (MD10 ve NL10) ipliklerindeki uyumsuz bölgelerde çift sarmallı kesmeyi tespit etmek için tamamlayıcının ortogonal etiketlemesini kullanın.
- DNA ligaz substratları için (Şekil 1): Üç oligonükleotidden en basit substratı birleştirin: 5' fosforile donör zincir (NL2), 5' FAM etiketli alıcı zincir (NL1) ve ikisini köprüleyen bir tamamlayıcı (NL3).
- Ticari bir satıcıdan ilgili floroforları ve diğer modifikasyonları içeren sentetik oligonükleotidler sipariş edin.
NOT: Açıklanan tahliller için 100 nM'lik bir sentez ölçeği ve sentezi takiben HPLC saflaştırması uygundur.
2. Nükleik asit dublekslerinin montajı ve tavlanması
- Oligonükleotidlerin yeniden süspansiyonu ve seyreltilmesi
- Açmadan önce, nükleik asidin tüpün dibinde olduğundan emin olmak için liyofilize oligonükleotidleri 2 mL'lik tüplerinde 2-5 dakika tezgah üstü bir santrifüjde tam hızda santrifüjleyin.
- Oligonükleotidleri TE tamponunda (10 mM tris (hidroksimetil) aminometan (Tris), 1 mM etilendiamintetraasetik asit (EDTA)) içinde yeniden süspanse ederek 100 μM'lik bir ana stok hazırlayın. Oligonükleotidlerin, tekrarlanan yumuşak girdaplama ve tam hızda kısa santrifüjleme ile iyice yeniden süspanse edildiğinden emin olun.
- Ana stoğun bir kısmını TE tamponu ile seyrelterek 10 μM'lik bir stok hazırlayın. Tablo 2'ye göre 0,5 μM, 0,7 μM veya 2,5 μM konsantrasyonlarda çalışma stokları hazırlamak için 10 μM stoğu ultra saf su (MQ su) ile seyreltin.
- Reaksiyon ana karışımlarının montajı ve tavlanması
- Tablo 2'de verilen kombinasyonları ve Tablo 3'te verilen hacimleri kullanarak reaksiyon ana karışımlarını oluşturmak için çalışma stoklarını kullanın. Standart DNA ligaz testi ve burada açıklanan diğer tahlillerin çoğu için, nihai tampon bileşimi, iki değerlikli katyon olarak 10 mM Mg ile 50 mM Tris pH 8.0, 50 mM NaCl, 10 mM Ditiyotreitol'dür (DTT).
- Oligonükleotidleri bir PCR veya mikrosantrifüj tüpünde bir ısıtma bloğu veya termosikleter kullanarak 95 ° C'de 5 dakika ısıtarak tavlayın. 30 dakika (hacim <1 mL) ila 1 saat (hacim >1 mL) oda sıcaklığına soğumaya bırakın. Daha uzun oligonükleotidler (>40 nt) için, 45 dakika boyunca 95 ° C ila 25 ° C arasında bir aşağı rampaya sahip bir termosikler kullanarak daha yavaş soğutma gerçekleştirin veya tavlama karışımı içeren tüpü 1 L'lik bir kaynar su kabında yüzdürün ve gece boyunca oda sıcaklığına soğumaya bırakın.
- Oda sıcaklığına soğuduktan sonra ana karışıma nükleotid kofaktörleri ve diğer ısıya duyarlı tampon bileşenleri ekleyin. Enzim ilavesiyle son reaksiyon karışımını doğrudan test için kullanın (aşağıdaki 3. adıma bakın) veya ileride kullanmak üzere -20 °C'de saklayın.
3. Standart tahlil kurulumu
- Tahlil reaksiyonunun montajı ve başlatılması
- 22.5 μL'lik ilgilenilen substrat ana karışımını 2.5 μL DNA ligaz veya bir PCR tüpünde ilgilenilen diğer enzim ile birleştirin. Reaksiyonları iki veya üç kopya halinde çalıştırın, özellikle de sonuçlar niceliksel olarak belirlenecekse.
- Tahlil numunelerine protein içermeyen bir kontrol (yalnızca tampon) ekleyin. Gerekirse, bu noktada hiçbir kofaktör denetimi eklemeyin.
NOT: Enzimler -20 °C'de %50 v/v gliserol içinde saklanabilir, bu da doğrudan çözeltiden pipetlenmelerine olanak tanır. Gliserol içeren enzim çözeltilerinin, karıştırmak için pipetleme veya hafif girdaplama yoluyla eklemeden önce iyice karıştırıldığından emin olun.
- Reaksiyonları hemen 25 °C'de bir PCR makinesine aktarın ve 30 dakika inkübe edin. Enzim aktivitesi için en uygun koşullara bağlı olarak sıcaklığı ve süreyi değiştirin.
- 5 μL yükleme boyası (%95 formamid, 0.5 M Etilendiamintetraasetik asit (EDTA), bromofenol mavisi) ekleyerek reaksiyonları söndürün ve 95 °C'de 5 dakika inkübe edin.
4. Tahlil sonuçlarının analizi
- Tris-Borat-EDTA (TBE)-Üre PAGE jellerini aşağıda açıklandığı gibi hazırlayın.
- %20 akrilamid, 7 M Üre ve 1x TBE çözeltisinden oluşan bir stok hazırlayın. Burada açıklanan oligonükleotid seti için, optimum çözünürlük için 29:1 oranında Akrilamid/Bis Çözeltisi kullanın.
- Bir jel için, 10 mL %20 akrilamid ve 7 M üre çözeltisini 100 μL APS (%10) ve 3 μL Tetrametiletilendiamin (TMED) ile birleştirin ve bir jel tekerine dökün.
- Jel katılaştıktan sonra, numuneleri TBE üre jeli üzerinde 45 - 55 °C'de çalıştırın.
- Jeli, harici ısıtma ile jel başına 10 mA'da 30 dakika boyunca 1x TBE tamponunda önceden çalıştırın.
- Bir mera pipeti kullanarak 1x TBE ile yıkayarak jelin kuyucuklarındaki fazla üreyi çıkarın.
- Her reaksiyondan 10 μL yükleyin ve harici ısıtma ile 1.0-1.5 saat boyunca 10 mA'da çalıştırın.
- Seçilen florofor için doğru ayarlarla görüntüleyici üzerindeki jeli görselleştirin. FAM için, çoğu görüntüleyicide önceden ayarlanmış olarak saklanan 495/519 nm'de uyarma/emisyon veren bir filtre seti kullanın.
- Görüntüleyici ile görüntü işleme yazılımı veya ImageJ49,50 gibi harici bir program kullanarak ürünün ve alt tabakanın bant yoğunluğunu ölçün ve aşağıdaki formülü kullanarak ürünün yüzdesini hesaplayın:
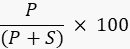
Burada P , ürün bandının entegre değeridir ve S , alt tabaka bandının entegre alanıdır. DNA ligaz reaksiyonu örneğinde, ürün bandı 40 nükleotidde (nt) ve substrat bandı 20 nt'de çalışır.
5. Kofaktör özgüllüğünü test etmek için DNA ligazın de-adenilasyonu
- Reaksiyon ana karışımlarının hazırlanması
- Tablo 4'te tarif edildiği gibi FAM etiketli NL1 oligonükleotidi içeren bir ana karışım seti hazırlayın. Tablo 1'te açıklandığı gibi, FAM etiketi olmayan NL4 oligonükleotidi içeren ikinci bir set hazırlayın.
- Ayrı olarak, her iki DNA dupleksini 5 dakika boyunca 95 °C'ye ısıtın ve 25 °C'de 30 dakika ila 1 saat soğutun. Her iki ana karışıma da nükleotid kofaktörü eklemeyin.
- De-adenilasyon reaksiyonunun montajı ve başlatılması
- Etiketlenmemiş ana karışımın 10 μL'sini 2.5 μL ligaz enzimi ile birleştirerek test edilecek her kofaktör tipi/konsantrasyonu için tek bir de-adenilasyon reaksiyonu hazırlayın.
- Kofaktör kontrolü olmayan ve protein kontrolü olmayan ek tüpler hazırlayın (enzim yerine 2.5 μL tampon eklenir).
- Reaksiyonları, enzimin optimum aktivitesine özgü bir sıcaklıkta 1-2 saat inkübe edin. Enzim hala adenile edilmişse inkübasyon süresi arttırılabilir.
- Ligasyon reaksiyonunu kofaktör ile çalıştırın.
- Etiketli ana karışımdan 10 μL ve istenen nükleotid kofaktörlerinden 2.5 μL ekleyin (ör., ATP, NAD, ADP veya GTP) doğrudan de-adenillenmiş reaksiyona (0.1-1 mM nihai konsantrasyon).
- Nükleotidsiz kofaktör kontrolüne 2.5 μL reaksiyon tamponu ekleyin.
- Reaksiyonları daha önce kullanılanla aynı zaman periyodu ve sıcaklık için inkübe edin. 4. adımda açıklandığı gibi söndürün ve görselleştirin.
6. Splinted ligasyon veya çok parçalı montaj için çift etiketli alt tabakaların kullanılması
- Halihazırda kullanılan florofordan farklı bir uyarma / emisyon spektrumuna sahip floresan parçalı bir oligonükleotid tasarlayın ve satın alın.
- Burada açıklanan kurulumda, 3' ucunda 5-Karboksitetrametilrodamin (TAMRA) bulunan NL2 (TAMRA) oligonükleotid kullanın (Tablo 1).
- Ana karışımı aşağıda açıklandığı gibi birleştirin.
- Montajda kullanılan tüm oligonükleotidlerin eşmolar oranlarının yanı sıra tampon ve iki değerlikli katyonlar da dahil olmak üzere adım 2'de açıklanan reaksiyonun bileşenlerini birleştirin.
- 95 °C'de 5 dakika ısıtarak ve 25 °C'de 30 dakika - 1 saat soğutarak tavlayın. Kofaktör ve enzimi ekleyin ve 3. adımda anlatıldığı gibi inkübe edin.
- Alt tabakadaki florofor çifti için uygun kanalları kullanarak numuneleri 4. adımda açıklandığı gibi çalıştırın ve görüntüleyin. FAM ve TAMRA söz konusu olduğunda, bunlar çoğu görüntüleyicide bulunan Floresein (FITC) ve Tetrametil rodamin (TRITC) kanallarıdır.
7. Doğal jel üzerinde Elektroforetik Mobilite Kaydırma Testi (EMSA) ile DNA bağlanmasının değerlendirilmesi
- Aşağıda açıklandığı gibi %10 doğal bir TBE PAGE jeli hazırlayın.
- 2.5 mL %40 akrilamid, 1 mL 10x TBE, 100 μL %10 APS, 3 μL TMED ve 6.5 mL MQ suyu birleştirin ve bir jel tekeri içine dökün.
- Bağlanma reaksiyonunu aşağıda açıklandığı gibi birleştirin.
- EMSA alt tabakasını Tablo 5'e göre birleştirin, böylece EDTA (10 mM) dahil edilir ve metal iyonları atlanır.
- 20 μL EMSA substrat ana karışımını bir PCR tüpünde 5 μL protein ile birleştirin. Protein içermeyen bir kontrol örneği ekleyin. 25 °C'de 30 dakika inkübe edin.
- Aşağıda açıklandığı gibi yerel elektroforez ile analiz edin.
- Numunelere 5 μL 5x doğal yükleme boyası (100 mM EDTA, %0,25 bromofenol mavisi, %25 v/v gliserol ve 1 mL'ye kadar MQ su) ekleyin.
- Hazırlanan jeli yükleyin ve boya önü jelin ucunun birkaç cm üzerinde olana kadar su sirkülasyonu ile soğutarak 2-3 saat boyunca 60 V'ta çalıştırın.
- Jelleri 4. adımda açıklandığı gibi görselleştirin ve analiz edin.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
DNA ligaz ile ligasyon
DNA ligaz enzimatik aktivitesi, bir üre PAGE jeli üzerinde görselleştirildiğinde floresan olarak işaretlenmiş oligonükleotidin boyutunda bir artışa neden olacaktır. Tablo 2'de listelenen hem DNA hem de RNA ligasyonu için substratlar söz konusu olduğunda, bu, 20 nt'den 40 nt'ye iki katına çıkan bir boyuta karşılık gelir (Şekil 3A). Optimal enzim aktivitesi, sıcaklık, protein konsantrasyonu veya inkübasyon sür...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
Protokoldeki kritik adımlar
Oligonükleotid tasarımı ve satın alma: Dubleks oluşumu için oligonükleotidleri satın alırken, dizi tasarımını dikkate almak önemlidir. 57 siparişinden önce GC içeriği, erime sıcaklığı, ikincil yapı ve dimerizasyon potansiyeli gibi nükleotid dizisinin özelliklerini tahmin etmek için bir oligo analizör aracının kullanılması önerilir.
Nükleik asit duplekslerinin montajı ve tavlanması: RNA/RN...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
SEG ve UR, R2D ligazın dağıtımını yapan ArcticZymes Technologies AS'nin çalışanlarıdır. AW, ER-S ve RS'nin rekabet eden çıkarları yoktur.
Teşekkürler
AW, bir Rutherford Keşif Bursu (20-UOW-004) tarafından desteklenmektedir. RS, Yeni Zelanda Post Antarktika Bursu'nun sahibidir.SG ve UR, teknik destek için Norveç Arktik Üniversitesi olan Tromsø Üniversitesi Kimya Enstitüsü'ne teşekkür eder.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
Referanslar
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır