JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
핵산-대사 효소를 분석하기 위해 변형된 합성 올리고뉴클레오티드를 사용
요약
여기에서는 리가아제(ligase), 뉴클레아제(nuclease) 및 중합효소(polymerase enzymes)의 예를 사용하여 핵산 대사 효소를 분석하기 위한 프로토콜을 제시합니다. 이 분석은 RNA 및/또는 DNA 손상 또는 경로 중간체를 모방하는 듀플렉스를 형성하기 위해 결합할 수 있는 형광 표지 및 표지되지 않은 올리고뉴클레오티드를 사용하여 효소 행동의 특성을 분석할 수 있습니다.
초록
상업 공급업체의 다양한 변형 합성 올리고뉴클레오티드를 사용할 수 있게 됨에 따라 모든 표준 분자 생물학 실험실에서 실행할 수 있는 핵산 대사 효소의 다양한 특성을 특성화할 수 있는 정교한 분석을 개발할 수 있었습니다. 형광 라벨을 사용함으로써 연구자들은 방사성 물질을 사용하거나 방사성 물질의 저장 및 준비를 위해 설계된 실험실(예: Hot Lab)을 필요로 하지 않고 표준 PAGE 전기영동 장비와 형광 지원 이미저를 사용하여 이러한 방법에 액세스할 수 있게 되었습니다. 인산화(phosphorylation)와 같은 표준 변형을 선택적으로 추가하면 분석 설정을 단순화할 수 있으며, DNA 손상 또는 중간체를 모방하는 변형된 뉴클레오티드의 특정 통합을 사용하여 효소 행동의 특정 측면을 조사할 수 있습니다. 여기에서는 상업적으로 이용 가능한 합성 올리고뉴클레오티드를 사용하는 효소에 의한 DNA 처리의 여러 측면을 조사하기 위한 분석의 설계 및 실행을 보여줍니다. 여기에는 서로 다른 DNA 및 RNA 하이브리드 구조를 분해하는 리가아제의 결합 능력 또는 뉴클레아제의 능력, DNA 리가아제에 의한 차별적 보조인자 사용, 효소의 DNA 결합 능력 평가가 포함됩니다. 합성 뉴클레오티드 기질을 설계할 때 고려해야 할 요인에 대해 논의하고, 다양한 핵산 리가아제, 중합효소 및 뉴클레아제 효소 분석에 사용할 수 있는 기본 올리고뉴클레오티드 세트를 제공합니다.
서문
모든 생명체는 복제, 전사 및 DNA 복구를 포함한 기본적인 생물학적 과정을 수행하기 위해 핵산 처리 효소를 필요로 합니다. 이러한 경로에 대한 주요 효소 기능은 RNA/DNA 분자의 복사본을 생성하는 중합효소, 폴리뉴클레오티드 기질을 결합하는 리가아제, 이를 분해하는 뉴클레아제, 핵산 이중체를 녹이거나 토폴로지를 변경하는 헬리카제 및 토포이소머라제입니다 1,2,3,4,5,6,7,8,9,10 . 또한, 이러한 효소의 대부분은 클로닝, 진단 및 고처리량 시퀀싱 11,12,13,14,15와 같은 응용 분야에 필수적인 분자 도구를 제공합니다.
이러한 효소의 기능적 특성, 반응 속도 및 기질 특이성은 올리고뉴클레오티드를 어닐링하여 생성된 표지된 DNA/RNA 기질을 사용하여 결정할 수 있습니다. 기판 및 제품 추적은 전통적으로 5' 가닥 말단에 방사성 라벨(32P)을 도입하여 달성되었으며, 이는 사진 필름 또는 형광체 이미징 시스템(16,17)으로 감지할 수 있습니다. 방사성 표지된 기질은 실험 감도를 높이는 이점을 제공하고 뉴클레오티드의 화학적 특성을 변경하지 않지만, 방사성 동위원소 작업으로 인한 잠재적인 건강 위험으로 인해 DNA 및 RNA 검출을 위한 보다 안전한 대안을 제공하기 위해 비방사성 핵산 표지의 개발이 촉진되었습니다 18,19,20. 이 중 직접 형광 검출, 시간 분해 형광 및 에너지 전달/형광 소멸 분석을 포함한 형광 검출은 가장 다재다능한 것으로 두드러집니다 21,22,23,24. 광범위한 형광단 어레이는 각 올리고뉴클레오티드25에 고유한 리포터를 특징으로 하는 다양한 DNA/RNA 기질 설계를 가능하게 합니다. 또한, 방사성 동위원소와 비교할 때 형광단의 안정성으로 인해 사용자는 상당한 양의 형광 표지된 DNA 기판을 생산하고 보존할 수 있습니다19. 이러한 형광단 표지 기질은 금속 및 뉴클레오티드 보조 인자의 다양한 조합과 함께 관심 단백질과 함께 배양하여 결합 및/또는 효소 활성을 분석할 수 있습니다. 결합 또는 활성의 시각화는 겔 이미징 시스템과 함께 다양한 형광단 염료 채널을 사용하여 관찰할 수 있습니다. 이 기술을 사용하면 형광으로 표지된 올리고뉴클레오티드만 볼 수 있으므로 표지된 올리고뉴클레오티드의 크기 증가 또는 감소를 쉽게 추적할 수 있습니다. 겔은 나중에 핵산 염색 염료를 사용하여 겔에 존재하는 모든 DNA 밴드를 시각화할 수도 있습니다.
폴리핵산 리가아제는 DNA/RNA 단편을 결합하는 효소로, 5' 인산화된 DNA 말단과 DNA의 3' OH 사이에 포스포디에스테르 결합을 형성하여 절단의 밀봉을 촉진합니다. 이들은 뉴클레오티드 기질 요구 사항에 따라 두 그룹으로 나눌 수 있습니다. 고도로 보존된 NAD-의존성 리가아제는 모든 박테리아에서 발견되며(26), 구조적으로 다양한 ATP-의존성 효소는 생명의 모든 영역에서 식별될 수 있다 8,27. DNA 리가아제는 복제 중 오카자키 단편 처리에 중요한 역할을 할 뿐만 아니라 복구 후 남은 자발적인 흠집과 흠집의 밀봉을 통해 뉴클레오티드 및 염기 절제 복구와 같은 다양한 DNA 복구 경로에 관여합니다 8,10. 상이한 DNA 리가아제는 RNA 및 DNA 하이브리드(28,29,30)뿐만 아니라 듀플렉스(duplex)의 흠집(nicks), 이중 가닥 단절(double-stranded breaks), 불일치 및 갭(gaps)을 포함하여 DNA 절단의 다양한 형태에 결합할 수 있는 다양한 능력을 나타낸다(28,29,30). 다양한 범위의 ligatable 기질은 핵산 듀플렉스 31,32,33에서 병치된 5' 및 3' 말단을 생성하기 위해 올리고뉴클레오티드를 5' 인산염으로 어닐링함으로써 조립할 수 있습니다. 가장 일반적인 분석 방법은 end-point assay 형식의 urea PAGE에 의한 분리능입니다. 그러나, 최근의 혁신에는 고처리량34, 질량 분석 프로파일링(35), 그리고 시간 분해 모니터링(36)을 가능하게 하는 균질한 분자 비콘 분석(homogenous molecular beacon assay)을 허용하는 모세관 겔 전기영동(capillary gel electrophoresis)의 사용이 포함되었다.
결찰 반응의 첫 번째 단계는 아데노신 삼인산(ATP) 또는 니코틴아미드 아데닌 디뉴클레오티드(NAD)에 의한 리가아제 효소의 아데닐화로, 공유 효소 중간체를 생성합니다. 반응의 두 번째 단계는 nick site의 5' 말단에서 핵산 기질의 아데닐화(adenylation)이며, 그 후 핵산 nick 가닥의 ligation이 뒤따릅니다. E. coli 에서 재조합 발현되는 많은 리가아제 효소는 아데닐화된 형태로 정제되므로 뉴클레오티드 보조인자를 추가하지 않고도 핵산을 성공적으로 결찰할 수 있습니다. 이로 인해 핵산의 결찰에 필요한 특정 유형의 뉴클레오티드 보조인자를 결정하기 어렵습니다. DNA 리가아제 활성을 평가하기 위한 분석을 설명하는 것 외에도, 표지되지 않은 기질을 사용하여 효소를 탈아데닐화하여 보조 인자 사용을 신뢰성 있게 결정하는 방법도 제시됩니다.
뉴클레아제는 핵산 사이의 포스포디에스테르 결합을 절단하는 DNA/RNA 변형 효소 및 촉매 RNA의 크고 다양한 그룹입니다(37). 뉴클레아제 효소 기능은 DNA 복제, 복구 및 RNA 처리에 필요하며 DNA, RNA 또는 둘 다에 대한 당 특이성에 따라 분류할 수 있습니다. 엔도뉴클레아제는 DNA/RNA 가닥 내의 포스포디에스터 결합을 가수분해하는 반면, 엑소뉴클레아제는 DNA/RNA 가닥을 3' 또는 5' 말단에서 한 번에 하나의 뉴클레오티드를 가수분해하고 DNA의 3'에서 5' 또는 5'에서 3' 말단에서 그렇게 할 수 있습니다(38).
많은 뉴클레아제 단백질이 비특이적이며 여러 과정에 관여할 수 있는 반면, 다른 단백질은 특정 염기서열 또는 DNA 손상에 대해 매우 특이적입니다 6,39,40. 염기서열 특이적 뉴클레아제는 클로닝(cloning), 돌연변이 유발(mutagenesis), 게놈 편집(genome editing)과 같은 광범위한 생명공학 응용 분야에서 사용됩니다. 이러한 응용 분야에서 널리 사용되는 뉴클레아제는 제한 뉴클레아제41, 징크-핑거 뉴클레아제42, 전사 활성제 유사 효과기 뉴클레아제, 그리고 가장 최근에는 RNA 유도 엔지니어링 CRISPR 뉴클레아제43입니다. 최근에는 EndoMS 뉴클레아제와 같은 손상 특이적 뉴클레아제가 확인되었는데, 이 뉴클레아제는 미스매치 특이적 RecB 유사 뉴클레아제 도메인 5,44를 통해 DNA의 미스매치에 대한 특이성을 가지고 있습니다. 뉴클레아제 활성 분석은 역사적으로 방사성 표지된 기질을 사용한 불연속 분석으로 수행되었습니다. 그러나, 그들의 다른 단점 외에도, 이들은 뉴클레아제 단백질에 의해 절단된 부위의 식별을 허용하지 않으며, 이는 형광 표지된 기질(45,46)을 사용할 때 가능하다. 보다 최근에는 서로 다른 상태의 DNA와 상호 작용하는 다양한 DNA 염료를 사용하여 작동하는 연속 뉴클레아제 분석이 개발되었습니다. 예를 들어, dsDNA와 상호 작용할 때 결합되지 않은 상태보다 더 높은 형광 신호를 방출하거나 짧은 RNA에 특이적으로 결합하는것 47. 다른 연속 뉴클레아제 분석은 5'에 형광단이 있고 3' 말단에 소광기가 있는 DNA 헤어핀을 사용하여 형광단과 소광체(48)의 분리로 인해 올리고뉴클레오티드가 분해됨에 따라 형광이 증가합니다. 이러한 분석을 통해 DNA 분해 단백질의 동역학을 특성화할 수 있지만 효소의 기능과 기질에 대한 사전 지식이 필요하며 DNA 구조를 변경하여 염료 결합에 차이를 일으키는 효소로 제한됩니다. 이러한 이유로, 단백질 활성으로 인한 DNA 변형에 대한 통찰력을 제공하기 위해 개별 뉴클레아제 산물을 분해하는 종말점 분석이 여전히 바람직합니다.
여기에서는 새로운 뉴클레아제, 중합효소 및 리가아제 효소의 활성을 테스트하기 위한 기질을 생성하기 위해 혼합 및 일치시킬 수 있는 형광 표지된 DNA/RNA 올리고뉴클레오티드의 설계를 위한 자세한 절차를 제시합니다. 이 기본 올리고뉴클레오티드 염기서열 세트의 검증은 실험 설계를 단순화하고 많은 수의 맞춤형 기질을 구매할 필요 없이 광범위한 효소 기능의 경제적인 프로파일링을 용이하게 합니다. DNA 리가아제 활성의 예를 사용하여 이러한 기질로 표준 DNA 처리 효소 분석을 실행하기 위한 자세한 절차가 제공되며, 뉴클레아제 및 중합효소 효소의 분석 및 분석을 위한 변형이 설명됩니다. 또한 DNA 리가아제 효소의 보조인자 특이성을 높은 정확도로 측정하기 위한 수정된 분석법이 제공되며, 이중 표지 프로브를 사용하여 다중 구성 요소 접합의 조립을 평가합니다. 마지막으로, 전기영동 이동성 이동 분석(EMSA)에 의해 동일한 기질과의 단백질-DNA 상호 작용을 결정하는 데 사용할 수 있도록 기본 분석 형식에 대한 수정에 대해 논의합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 올리고뉴클레오티드의 설계 및 구매
참고: 단일 가닥 올리고뉴클레오티드를 설계하여 원하는 듀플렉스로 조립하고 어닐링합니다. 듀플렉스에 있는 하나 이상의 가닥은 관심 효소에 의한 올리고뉴클레오티드 처리를 추적하기 위해 형광 부분을 가져야 합니다. 다양한 활동을 위해 조립할 수 있는 단일 가닥 서열의 기본 세트가 표 1에 나와 있습니다.
- 아래에 설명된 대로 관심 효소에 필요한 특정 변형을 통합합니다.
- DNA 리가아제 기질의 경우(그림 1): 5' 인산화 공여체 가닥(NL2), 5' FAM 표지 수용체 가닥(NL1) 및 둘을 연결하는 보체(NL3)의 세 가지 올리고뉴클레오티드에서 가장 간단한 기질을 조립합니다.
- ligatable nick의 5' 말단을 제공하는 가닥이 2단계에서 기판 마스터 믹스를 조립하기 전에 인산화되었는지 확인합니다. 이것을 NL2에 대한 변형으로 주문하거나( 표 1에 제시된 대로) 올리고뉴클레오티드를 재현탁한 후 T4 폴리뉴클레오티드 키나아제와 함께 효소 인산화를 사용하십시오.
- 그림 1A(NL6/NL7 및 NL8/NL9)에 표시된 이중 가닥 절단의 보체를 구성하는 NL6 및 NL8의 5'-말단 인산화를 포함하며, 이는 제한 엔도뉴클레아제에서 생성된 자연 기질과 가장 유사합니다. dual-labeled substrate를 사용하여 multi-part assembly에 대한 ligation의 상대적 범위를 결정합니다(단계 6 참조).
- 미스매치(NL10)와 갭(NL11)을 생성하도록 보수 가닥을 변경합니다.
참고: 단순 흠집이 난 기판의 변형은 그림 1A에 표시되어 있습니다. 다른 시퀀스를 사용하여 밑줄이 그어진 위치를 변경하여 훨씬 더 넓은 범위의 불일치 또는 더 긴 간격을 생성할 수 있습니다. - RNA 올리고뉴클레오티드를 DNA 올리고뉴클레오티드로 대체합니다.
참고: 단순 흠집이 있는 기판의 변형은 그림 1B에 표시되어 있습니다. 예를 들어, RNA와 DNA를 모두 포함하는 이중 가닥 절단을 생성하기 위해 여기에 제공된 기본 세트의 추가 조합에 의해 더 넓은 범위의 DNA/RNA 듀플렉스를 생성할 수 있습니다. 이 변형의 예는 아래 6단계에 나와 있으며, 여기서 이중 레이블 전략이 사용됩니다.
- DNA 중합효소 기질의 경우: 표 1 에 나열된 올리고뉴클레오티드 NL1 및 NL3을 조립하여 간단한 프라이머 확장 분석을 제공합니다. NL1(프라이머) 또는 NL3(템플릿) 가닥에 변형을 도입하여 중합효소 활성의 추가 측면을 조사합니다.
- 템플릿 가닥의 손상된 병변을 우회할 수 있는 능력을 결정하기 위해 위치 20 이전에 손상된 염기 유사체를 NL3 올리고뉴클레오티드에 통합합니다.
- 손상된 염기 유사체를 위치 20의 NL1 올리고뉴클레오티드에 통합하여 손상된 프라이머를 확장할 수 있는 능력을 결정합니다.
- 듀플렉스에서 RNL1 또는 RNL3을 사용하여 RNA 프라이머의 확장 또는 RNA 템플릿의 사용을 조사합니다.
- 뉴클레아제 기질의 경우(그림 2): 올리고뉴클레오티드를 조립하여 이중 및 단일 가닥 기질(그림 2Ai)과 다양한 플랩 및 스플레이 접합부(그림 2Aii) 및 손상된 기질(그림 2B)을 제공합니다.
- 리보핵분해효소 활성을 조사하기 위해 NL1, NL2 및 NL3을 RNL1, RNL2 및 RNL3로 반복적으로 대체합니다. HJ5 및 HJ6의 추가 RNA 버전을 사용하여 이 세트를 더욱 확장하십시오.
- 올리고뉴클레오티드 MD5, MD6 및 MD9는 산화 손상, 비염기성 복구 중간체 또는 탈아미네이션 산물을 모방한 중앙에 배치된 변형을 사용합니다(그림 2B). 기판은 이 위치에서 가닥의 절단을 감지합니다. 보체 NL3 가닥에 TAMRA와 같은 직교 형광단을 라벨링하여 이중 가닥 절단을 감지합니다(6단계 참조).
- 보형의 직교 라벨링을 사용하여 프로브(NL5 및 ND9) 및 보체(MD10 및 NL10) 스트랜드 모두의 일치하지 않는 위치에서 이중 가닥 절단을 감지합니다.
- DNA 리가아제 기질의 경우(그림 1): 5' 인산화 공여체 가닥(NL2), 5' FAM 표지 수용체 가닥(NL1) 및 둘을 연결하는 보체(NL3)의 세 가지 올리고뉴클레오티드에서 가장 간단한 기질을 조립합니다.
- 관련 fluorophores 및 기타 변형을 포함하는 합성 올리고뉴클레오티드를 상업 공급업체로부터 주문하십시오.
참고: 100 nM 합성 스케일과 합성 후 HPLC 정제는 설명된 분석에 적합합니다.
2. 핵산 듀플렉스 조립 및 어닐링
- oligonucleotide의 재현탁 및 희석
- 개봉하기 전에 2mL 튜브의 동결건조된 올리고뉴클레오티드를 벤치탑 원심분리기에서 2-5분 동안 최고 속도로 원심분리하여 핵산이 튜브 바닥에 있는지 확인합니다.
- TE 완충액(10mM 트리스(하이드록시메틸)아미노메탄(Tris), 1mM 에틸렌디아민테트라아세트산(EDTA))에 올리고뉴클레오티드를 재현탁하여 100μM의 마스터 스톡을 준비합니다. 올리고뉴클레오티드가 전속력으로 반복적인 부드러운 소용돌이와 짧은 원심분리를 통해 완전히 재현탁되도록 합니다.
- 마스터 스톡의 부분 표본을 TE 버퍼로 희석하여 10μM 스톡을 준비합니다. 10μM 스톡을 초순수(MQ 물)로 희석하여 표 2와 같이 0.5μM, 0.7μM 또는 2.5μM 농도의 작업 스톡을 준비합니다.
- 반응 마스터 믹스 조립 및 어닐링
- 작업 스톡을 사용하여 표 2 에 제공된 조합과 표 3에 제공된 부피를 사용하여 반응 마스터 믹스를 구성합니다. 표준 DNA 리가아제 분석 및 여기에 설명된 대부분의 다른 분석의 경우, 최종 완충액 조성은 50mM Tris pH 8.0, 50mM NaCl, 10mM Dithiothreitol(DTT)이며 10mM Mg을 2가 양이온으로 합니다.
- 가열 블록 또는 열순환기를 사용하여 95°C에서 5분 동안 가열하여 PCR 또는 마이크로 원심분리기 튜브의 올리고뉴클레오티드를 어닐링합니다. 30분(부피 <1mL)에서 1시간(부피 >1mL) 동안 실온으로 냉각합니다. 더 긴 올리고뉴클레오티드(>40 nt)의 경우 95 °C에서 25 °C의 하향 램프가 있는 써모사이클러를 사용하여 45분 동안 천천히 냉각하거나 어닐링 혼합물이 들어 있는 튜브를 끓는 물 1L 비커에 띄우고 밤새 실온으로 냉각시킵니다.
- 뉴클레오티드 보조인자 및 기타 열에 민감한 완충 성분을 실온으로 냉각한 후 마스터 믹스에 추가합니다. 효소를 첨가하여 최종 반응 혼합물을 분석에 직접 사용하거나(아래 3단계 참조) 나중에 사용할 수 있도록 -20°C에서 보관하십시오.
3. 표준 분석 설정
- 분석 반응의 조립 및 시작
- PCR 튜브에서 22.5 μL의 관심 기질 마스터 믹스를 2.5 μL의 DNA 리가아제 또는 기타 관심 효소와 결합합니다. 특히 결과가 정량화되는 경우 중복 또는 삼중 반응을 실행합니다.
- 분석 샘플에 no-protein control(완충액만 해당)을 포함합니다. 필요한 경우 이 시점에서 보조 인자 컨트롤을 포함하지 않습니다.
참고: 효소는 -20°C에서 50% v/v 글리세롤로 보관할 수 있어 용액에서 직접 피펫팅할 수 있습니다. 글리세롤이 함유된 효소 용액이 첨가 전에 잘 혼합되었는지 확인하십시오., 피펫팅을 통해 혼합하거나 부드러운 소용돌이를 통해.
- 즉시 반응을 25°C의 PCR 기계로 옮기고 30분 동안 배양합니다. 효소 활성을 위한 최적의 조건에 따라 온도와 지속 시간을 변경합니다.
- 5μL의 로딩 염료(95% 포름아미드, 0.5M 에틸렌디아민테트라아세트산(EDTA), 브로모페놀 블루)를 첨가하여 반응을 퀀칭하고 95°C에서 5분 동안 배양합니다.
4. 분석 결과 분석
- 아래 설명에 따라 Tris-Borate-EDTA(TBE)-Urea PAGE 겔을 준비합니다.
- 20% 아크릴아마이드, 7M 요소 및 1x TBE 용액을 준비합니다. 여기에 설명된 올리고뉴클레오티드 세트의 경우 최적의 분리능을 위해 Acrylamide/Bis Solution을 29:1 비율로 사용하십시오.
- 겔 1개의 경우 10mL의 20% 아크릴아마이드와 7M 요소 용액을 100μL의 APS(10%) 및 3μL의 테트라메틸에틸렌디아민(TMED)과 결합하고 겔 캐스터에 주조합니다.
- 겔이 응고된 후 45 - 55 °C에서 TBE 요소 겔의 샘플을 실행합니다.
- 외부 가열을 통해 겔당 10mA로 30분 동안 1x TBE 버퍼에서 겔을 사전 실행합니다.
- 목초지 피펫을 사용하여 1x TBE로 세척하여 겔의 웰에서 과도한 요소를 제거합니다.
- 각 반응의 10μL를 로드하고 외부 가열로 1.0-1.5시간 동안 10mA에서 실행합니다.
- 선택한 형광단에 대한 올바른 설정으로 이미저의 겔을 시각화합니다. FAM의 경우 495/519nm에서 여기/방출을 제공하는 필터 세트를 사용하며, 이는 대부분의 이미저에 사전 설정으로 저장됩니다.
- 이미저가 있는 이미지 처리 소프트웨어 또는 ImageJ49,50과 같은 외부 프로그램을 사용하여 제품 및 기판의 밴드 강도를 정량화하고 공식을 사용하여 제품의 백분율을 계산합니다.
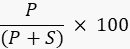
여기서 P 는 제품 밴드의 적분 값이고 S 는 기판 밴드의 적분 면적입니다. DNA 리가아제 반응 예의 경우, 생성물 띠는 40 뉴클레오티드(nt)에서 실행되고 기질 띠는 20 nt에서 실행됩니다.
5. 보조인자 특이성을 테스트하기 위한 DNA 리가아제의 탈아데닐화
- 반응 마스터 믹스의 준비
- 표 4에 기재된 바와 같이 FAM-labeled NL1 oligonucleotide를 함유하는 마스터 믹스 한 세트를 준비한다. 표 4에 설명된 대로 FAM 라벨이 없는 NL1 올리고뉴클레오티드를 포함하는 두 번째 세트를 준비합니다.
- 별도로 두 DNA 듀플렉스를 95°C에서 5분 동안 가열하고 25°C에서 30분에서 1시간 동안 냉각합니다. 두 마스터 믹스 모두에 뉴클레오티드 보조 인자를 추가하지 마십시오.
- 탈아데닐화(de-adenylation) 반응의 조립 및 시작
- 라벨링되지 않은 마스터 믹스 10μL와 리가아제 효소 2.5μL를 결합하여 테스트할 각 보조인자 유형/농도에 대한 단일 탈아데닐화 반응을 준비합니다.
- 비 보조 인자 대조군 및 단백질 대조군이 없는 추가 튜브를 준비합니다(효소 대신 2.5μL의 완충액 추가).
- 효소의 최적 활성에 특정한 온도에서 1-2시간 동안 반응을 배양합니다. 효소가 여전히 아데닐화되어 있는 경우 배양 시간이 증가할 수 있습니다.
- 보조 인자로 결찰 반응을 실행합니다.
- 라벨링된 마스터 믹스 10μL와 원하는 뉴클레오티드 보조인자(예: ATP, NAD, ADP 또는 GTP) 2.5μL를 탈아데닐화 반응(0.1-1mM 최종 농도)에 직접 첨가합니다.
- 2.5μL의 반응 완충액을 뉴클레오티드가 없는 보조인자 대조군에 추가합니다.
- 이전에 사용한 것과 동일한 기간 및 온도 동안 반응을 배양합니다. 4단계에서 설명한 대로 담금질하고 시각화합니다.
6. 부목 결찰 또는 다중 부품 조립을 위해 이중 라벨링 기판 사용
- 이미 사용된 형광단과 다른 excitation/emission 스펙트럼을 가진 형광 부분을 가진 oligonucleotide를 설계하고 구매합니다.
- 여기에 설명된 설정에서는 3' 말단에 5-카르복시테트라메틸로다민(TAMRA)이 있는 NL2(TAMRA) 올리고뉴클레오티드를 사용합니다(표 1).
- 아래 설명에 따라 마스터 믹스를 조립합니다.
- 어셈블리에 사용된 모든 올리고뉴클레오티드의 등몰비와 완충액 및 2가 양이온을 포함하여 2단계에서 설명한 반응 성분을 결합합니다.
- 95 ° C에서 5 분 동안 가열하고 25 ° C에서 30 분 - 1 시간 동안 냉각하여 어닐링합니다. 보조 인자와 효소를 추가하고 3단계에서 설명한 대로 배양합니다.
- 4단계에서 설명한 대로 기판의 fluorophore pair에 적합한 채널을 사용하여 샘플을 실행하고 이미지화합니다. FAM 및 TAMRA의 경우 대부분의 이미저에 존재하는 플루오레세인(FITC) 및 테트라메틸 로다민(TRITC) 채널입니다.
7. 네이티브 겔에 대한 EMSA(Electrophoretic Mobility Shift Assay)에 의한 DNA 결합 평가
- 아래 설명에 따라 10% 천연 TBE PAGE 겔을 준비합니다.
- 40% 아크릴아마이드 2.5mL, 10x TBE 1mL, 10% APS 100μL, TMED 3μL, MQ 물 6.5mL를 겔 캐스터에 넣고 주조합니다.
- 아래 설명된 대로 결합 반응을 조립합니다.
- EDTA(10 mM)를 포함시키고 금속 이온을 생략하도록 표 5 에 따라 EMSA 기판을 조립한다.
- PCR 튜브에서 20μL의 EMSA 기질 마스터 믹스와 5μL의 단백질을 결합합니다. 단백질 대조군이 없는 검체를 포함합니다. 25 °C에서 30분 동안 배양합니다.
- 아래에 설명된 대로 기본 전기영동으로 분석합니다.
- 5x 네이티브 로딩 염료 5μL(100mM EDTA, 0.25% 브로모페놀 블루, 25% v/v 글리세롤 및 최대 1mL의 MQ 물)를 샘플에 추가합니다.
- 준비된 겔에 로드하고 염료 전면이 겔 끝에서 몇 cm 위에 올 때까지 물 순환으로 냉각하면서 60V에서 2-3시간 동안 실행합니다.
- 4단계에서 설명한 대로 겔을 시각화하고 분석합니다.
Access restricted. Please log in or start a trial to view this content.
결과
DNA 리가아제에 의한 결찰
DNA 리가아제 효소 활성은 요소 PAGE 겔에서 시각화할 때 형광 표지된 올리고뉴클레오티드의 크기를 증가시킵니다. 표 2에 나열된 DNA 및 RNA 접합을 위한 기질의 경우, 이는 크기가 20nt에서 40nt로 두 배로 증가하는 것에 해당합니다(그림 3A). 최적의 효소 활성은 온도, 단백질 농도 또는 배양 시간(그림 3B)...
Access restricted. Please log in or start a trial to view this content.
토론
프로토콜의 중요한 단계
올리고뉴클레오티드 설계 및 구매: 이중 형성을 위한 올리고뉴클레오티드를 구매할 때는 염기서열 설계를 고려하는 것이 필수적입니다. 57을 주문하기 전에 올리고 분석기 도구를 사용하여 GC 함량, 용융 온도, 2차 구조 및 이합체화 전위와 같은 뉴클레오티드 서열의 특성을 예측하는 것이 좋습니다.
핵산 듀플렉스의 ?...
Access restricted. Please log in or start a trial to view this content.
공개
SEG와 UR은 R2D 리가아제를 유통하는 ArcticZymes Technologies AS의 직원입니다. AW, ER-S 및 RS는 서로 상충되는 이해관계가 없습니다.
감사의 말
AW는 Rutherford Discovery Fellowship(20-UOW-004)의 지원을 받습니다. RS는 뉴질랜드 남극 포스트 장학금(New Zealand Post Antarctic Scholarship )의 수혜자입니다.SG와 UR은 노르웨이 북극 대학인 트롬쇠 대학교(University of Tromsø)의 화학 연구소(Chemical Institute)의 기술 지원을 인정합니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
참고문헌
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유