Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Использование модифицированных синтетических олигонуклеотидов для анализа ферментов, метаболизирующих нуклеиновые кислоты
В этой статье
Резюме
Здесь представлен протокол анализа ферментов, метаболизирующих нуклеиновые кислоты, с использованием примеров ферментов лигазы, нуклеазы и полимеразы. В анализе используются флуоресцентно меченые и немеченые олигонуклеотиды, которые могут быть объединены для образования дуплексов, имитирующих повреждения РНК и/или ДНК или промежуточные пути, что позволяет охарактеризовать поведение фермента.
Аннотация
Доступность ряда модифицированных синтетических олигонуклеотидов от коммерческих поставщиков позволила разработать сложные анализы для характеристики различных свойств ферментов, метаболизирующих нуклеиновые кислоты, которые могут быть запущены в любой стандартной лаборатории молекулярной биологии. Использование флуоресцентных меток сделало эти методы доступными для исследователей со стандартным оборудованием для электрофореза PAGE и флуоресцентным тепловизором, не используя радиоактивные материалы и не требуя лаборатории, предназначенной для хранения и подготовки радиоактивных материалов, т.е. горячей лаборатории. Необязательное добавление стандартных модификаций, таких как фосфорилирование, может упростить настройку анализа, в то время как специфическое включение модифицированных нуклеотидов, которые имитируют повреждения ДНК или промежуточные продукты, может быть использовано для исследования конкретных аспектов поведения фермента. Здесь демонстрируется разработка и проведение анализов для изучения нескольких аспектов процессинга ДНК ферментами с использованием коммерчески доступных синтетических олигонуклеотидов. К ним относятся способность лигаз присоединяться или нуклеазы разрушать различные гибридные структуры ДНК и РНК, дифференциальное использование кофактора ДНК-лигазой и оценка ДНК-связывающей способности ферментов. Обсуждаются факторы, которые следует учитывать при проектировании синтетических нуклеотидных субстратов, а также приводится базовый набор олигонуклеотидов, которые могут быть использованы для ряда анализов лигазы нуклеиновой кислоты, полимеразы и нуклеазного фермента.
Введение
Все формы жизни нуждаются в ферментах, процессирующих нуклеиновые кислоты, для осуществления фундаментальных биологических процессов, включая репликацию, транскрипцию и репарацию ДНК. Ключевыми ферментативными функциями для этих путей являются полимеразы, которые генерируют копии молекул РНК/ДНК, лигазы, которые присоединяются к полинуклеотидным субстратам, нуклеазы, которые разрушают их, а также геликазы и топоизомеразы, которые расплавляют дуплексы нуклеиновых кислот или изменяют их топологию 1,2,3,4,5,6,7,8,9,10 . Кроме того, многие из этих ферментов обеспечивают необходимые молекулярные инструменты для таких приложений, как клонирование, диагностика и высокопроизводительное секвенирование 11,12,13,14,15.
Функциональные характеристики, кинетика и субстратная специфичность этих ферментов могут быть определены с помощью меченых субстратов ДНК/РНК, полученных путем отжига олигонуклеотидов. Слежение за подложками и продуктами традиционно достигалось путем введения радиоактивной метки (32P) либо на 5'-конце цепи, которая затем может быть обнаружена с помощью фотопленки или с помощью люминофорной системы визуализации16,17. В то время как радиоактивно меченые субстраты обладают повышенной экспериментальной чувствительностью и не изменяют химических свойств нуклеотида, потенциальная опасность для здоровья от работы с радиоизотопами способствовала разработке нерадиоактивных нуклеиновых кислот для обеспечения более безопасной альтернативы для обнаружения ДНК и РНК 18,19,20 . Среди нихнаиболее универсальными являются методы детектирования флуоресценции, в том числе прямое флуоресцентное детектирование, флуоресценция с временным разрешением и анализ переноса энергии/гашения флуоресценции. Обширный массив флуорофоров позволяет создавать различные конструкции субстратов ДНК/РНК с уникальными репортерами на каждом олигонуклеотиде25. Кроме того, стабильность флуорофоров по сравнению с радиоизотопами позволяет пользователям производить и сохранять значительные количества флуоресцентно меченых субстратов ДНК19. Эти меченые флуорофорами субстраты могут быть инкубированы с интересующим белком, а также с различными комбинациями металлических и нуклеотидных кофакторов для анализа связывания и/или активности фермента. Визуализацию связывания или активности можно наблюдать с помощью различных каналов флуорофорных красителей с помощью системы визуализации геля. Поскольку с помощью этого метода будут видны только флуоресцентно меченые олигонуклеотиды, любое увеличение или уменьшение размера меченого олигонуклеотида будет легко проследить. Гели также могут быть окрашены после этого с помощью красителей для окрашивания нуклеиновыми кислотами, чтобы визуализировать все полосы ДНК, присутствующие на геле.
Лигазы полинуклеиновых кислот представляют собой ферменты, которые соединяют фрагменты ДНК/РНК, катализируя герметизацию разрывов путем образования фосфодиэфирной связи между 5'-фосфорилированными концами ДНК и 3'-OH ДНК. Их можно разделить на две группы в зависимости от потребности в нуклеотидном субстрате. Высококонсервативные НАД-зависимые лигазы обнаружены у всех бактерий26, в то время как структурно разнообразные АТФ-зависимые ферменты могут быть идентифицированы во всех областях жизни 8,27. ДНК-лигазы играют важную роль в процессинге фрагментов Оказаки во время репликации, а также участвуют в различных путях репарации ДНК, таких как репарация нуклеотидов и эксцизионная репарация оснований, путем герметизации спонтанных зазубрин и зазубрин, которые остаются после репарации 8,10. Различные ДНК-лигазы демонстрируют различную способность присоединяться к различным конформациям разрывов ДНК, включая зазубрины в дуплексе, двухцепочечные разрывы, несоответствия и разрывы, а также гибриды РНК и ДНК 28,29,30. Разнообразный спектр лигабельных субстратов может быть собран путем отжига олигонуклеотидов с 5'-фосфатом для получения сопоставленных 5' и 3' концов в дуплексе нуклеиновых кислот 31,32,33. Наиболее распространенным методом анализа является разрешение мочевиной PAGE в формате анализа конечной точки; Тем не менее, последние инновации включают использование электрофореза в капиллярном геле, который обеспечивает высокую пропускнуюспособность34, масс-спектрометрическое профилирование35, а также гомогенный молекулярный анализ маяков, который позволяет проводить мониторинг с временным разрешением36.
Первым этапом реакции лигирования является аденилирование фермента лигазы аденозинтрифосфатом (АТФ) или никотинамидадениндинуклеотидом (НАД), в результате чего образуется промежуточный фермент ковалентного типа. Вторым этапом реакции является аденилирование субстрата нуклеиновой кислоты на 5'-конце ника ника, за которым следует лигирование нитей нуклеиновой кислоты. Многие ферменты лигазы, рекомбинантно экспрессируемые в E. coli, очищаются в аденилированной форме и, следовательно, способны успешно лигировать нуклеиновые кислоты без добавления нуклеотидного кофактора. Это затрудняет определение того, какой именно тип нуклеотидного кофактора им требуется для лигирования нуклеиновых кислот. В дополнение к описанию анализов для оценки активности ДНК-лигазы также представлен метод надежного определения использования кофактора путем деаденилирования фермента с использованием немеченых субстратов.
Нуклеазы представляют собой большую и разнообразную группу ферментов, модифицирующих ДНК/РНК, и каталитических РНК, которые расщепляют фосфодиэфирные связи между нуклеиновымикислотами. Функциональные возможности нуклеазных ферментов необходимы для репликации ДНК, репарации и процессинга РНК и могут быть классифицированы по их сахарной специфичности для ДНК, РНК или и того, и другого. Эндонуклеазы гидролизуют фосфодиэфирные связи внутри цепи ДНК/РНК, в то время как экзонуклеазы гидролизуют нити ДНК/РНК по одному нуклеотиду за раз от 3' или 5' конца и могут делать это либо от 3' до 5', либо от 5' до 3' конца ДНК38.
В то время как многие нуклеазные белки неспецифичны и могут участвовать в нескольких процессах, другие обладают высокой специфичностью в отношении определенной последовательности или повреждения ДНК 6,39,40. Последовательно-специфические нуклеазы используются в широком спектре биотехнологических приложений, таких как клонирование, мутагенез и редактирование генома. Популярными нуклеазами для этих применений являются нуклеазы рестрикции41, нуклеазы цинкового пальца42, эффекторные нуклеазы транскрипционного активатора, а в последнее время — РНК-управляемые CRISPR нуклеазы43. Недавно были идентифицированы поврежденные нуклеазы, такие как нуклеаза EndoMS, которая обладает специфичностью к несоответствиям в ДНК через свой специфичный для несоответствия RecB-подобный нуклеазный домен 5,44. Исторически сложилось так, что анализы нуклеазной активности проводились в виде прерывистых анализов с радиоактивно мечеными субстратами; Однако, в дополнение к своим другим недостаткам, они не позволяют идентифицировать сайт, который разрезается нуклеазным белком, что возможно при использовании флуоресцентно меченных субстратов 45,46. В последнее время были разработаны непрерывные нуклеазные анализы, которые работают с использованием различных красителей ДНК, взаимодействующих с ДНК в разных состояниях; например, излучение более высокого флуоресцентного сигнала при взаимодействии с дцДНК, чем в ее несвязанном состоянии, или специфическое связывание с короткими РНК47. В других непрерывных нуклеазных анализах используются шпильки ДНК с флуорофорной группой на 5'-конце и гасителем на 3'-конце, так что флуоресценция увеличивается по мере деградации олигонуклеотида из-за разделения флуорофора и гасителя48. В то время как эти анализы позволяют охарактеризовать кинетику белков, разрушающих ДНК, они требуют предварительных знаний о функции фермента и субстрате, а также ограничены ферментами, которые изменяют конформацию ДНК, вызывая разницу в связывании красителей. По этой причине по-прежнему желательны конечные анализы, которые разрешают отдельные нуклеазные продукты, чтобы дать представление о модификациях ДНК, вызванных активностью белка.
Здесь подробно описана процедура разработки флуоресцентно меченных олигонуклеотидов ДНК/РНК, которые могут быть смешаны и согласованы для получения субстратов для тестирования активности новых ферментов нуклеазы, полимеразы и лигазы. Валидация этого базового набора олигонуклеотидных последовательностей упрощает планирование эксперимента и способствует экономичному профилированию широкого спектра ферментативных функций без необходимости приобретения большого количества специализированных субстратов. Подробно описана процедура проведения стандартного ферментного анализа ДНК с этими субстратами на примере активности ДНК-лигазы и модификаций для анализа и анализа нуклеазных и полимеразных ферментов. Кроме того, приведен модифицированный анализ для определения кофакторной специфичности фермента ДНК-лигазы с высокой точностью, а для оценки сборки многокомпонентных лигаторов используются зонды с двойной меткой. Наконец, обсуждаются модификации базового формата анализа, позволяющие использовать его для определения белок-ДНК-взаимодействий с теми же субстратами с помощью анализа электрофоретического сдвига подвижности (EMSA).
протокол
1. Разработка и закупка олигонуклеотидов
Примечание: Проектирование одноцепочечных олигонуклеотидов для сборки и отжига в желаемые дуплексы. Одна или несколько цепей в дуплексе должны содержать флуоресцентный фрагмент для отслеживания процесса олигонуклеотида интересующим ферментом. Базовый набор одноцепочечных последовательностей, которые могут быть собраны для ряда действий, представлен в таблице 1.
- Включите конкретные модификации, необходимые для интересующего фермента, как описано ниже.
- Для субстратов ДНК-лигазы (рис. 1): Соберите простейший субстрат из трех олигонуклеотидов: 5'-фосфорилированной донорской цепи (NL2), 5'-меченой FAM-меченой акцепторной цепи (NL1) и комплемента, который соединяет их (NL3).
- Убедитесь, что нити, обеспечивающие 5'-конец лигабельного зуба, фосфорилированы перед сборкой основной смеси подложки на этапе 2. Закажите его в качестве модификации на NL2 (как показано в таблице 1) или используйте ферментативное фосфорилирование полинуклеотидкиназой Т4 после ресуспендирования олигонуклеотидов.
- Включают 5'-концевое фосфорилирование NL6 и NL8, которые составляют дополнение двухцепочечных разрывов, изображенных на рисунке 1A (NL6/NL7 и NL8/NL9), поскольку оно наиболее близко напоминает природный субстрат, полученный из эндонуклеазы рестрикции. Используйте подложку с двойной меткой для определения относительных степеней лигирования для сборок, состоящих из нескольких деталей (см. шаг 6).
- Изменяйте цепь комплемента, чтобы получить несоответствия (NL10) и пробелы (NL11).
ПРИМЕЧАНИЕ: Вариации простой подложки с зазубринами изображены на рисунке 1А. Можно использовать другие последовательности для создания еще более широкого диапазона несоответствий или более длинных промежутков путем изменения подчеркнутой позиции. - Замените ДНК-олигонуклеотиды на РНК-олигонуклеотиды.
ПРИМЕЧАНИЕ: Вариации простой подложки с зазубринами изображены на рисунке 1B. Более широкий спектр дуплексов ДНК/РНК может быть получен путем дополнительных комбинаций приведенного здесь базисного набора для получения, например, двухцепочечных разрывов, содержащих как РНК, так и ДНК. Пример этого варианта приведен на шаге 6 ниже, где используется стратегия с двумя метками.
- Для субстратов ДНК-полимеразы: Соберите олигонуклеотиды NL1 и NL3, перечисленные в таблице 1 , чтобы получить простой анализ удлинения праймера. Исследовать дополнительные аспекты активности полимеразы путем внесения модификаций в цепи NL1 (праймер) или NL3 (матрицу).
- Включите поврежденные аналоги основания в олигонуклеотид NL3 до положения 20, чтобы определить способность обхода поврежденных поражений на матричной цепи.
- Включите поврежденные аналоги основания в олигонуклеотид NL1 в положении 20 для определения способности удлинять поврежденный праймер.
- Используйте либо RNL1, либо RNL3 в дуплексе для исследования расширения праймера РНК или использования матрицы РНК.
- Для нуклеазных субстратов (рис. 2): Соберите олигонуклеотиды, чтобы получить неисчерпывающий диапазон двухцепочечных и одноцепочечных субстратов (рис. 2Ai), а также ряд хлопающих и растянутых соединений (рис. 2Aii) и поврежденных субстратов (рис. 2B).
- Чтобы проверить действия рибонуклеазы, итеративно замените NL1, NL2 и NL3 на RNL1, RNL2 и RNL3. Используйте дополнительные версии РНК HJ5 и HJ6, чтобы еще больше расширить этот набор.
- Используйте олигонуклеотиды MD5, MD6 и MD9, которые имеют центрально расположенную модификацию, имитирующую окислительное повреждение, промежуточный продукт репарации аосновных компонентов или продукт дезаминирования (рисунок 2B). Подложки обнаружат спайность пряди в этом положении. Пометьте цепь комплемента NL3 ортогональным флуорофором, таким как TAMRA, чтобы обнаружить двухцепочечное расщепление (см. шаг 6).
- Используйте ортогональную маркировку комплемента для обнаружения двухцепочечного разрезания в несоответствующих участках как на цепи зонда (NL5 и ND9), так и на цепи комплемента (MD10 и NL10).
- Для субстратов ДНК-лигазы (рис. 1): Соберите простейший субстрат из трех олигонуклеотидов: 5'-фосфорилированной донорской цепи (NL2), 5'-меченой FAM-меченой акцепторной цепи (NL1) и комплемента, который соединяет их (NL3).
- Заказывайте синтетические олигонуклеотиды с соответствующими флуорофорами и другими модификациями у коммерческого поставщика.
ПРИМЕЧАНИЕ: Для описанных анализов подходит масштаб синтеза 100 нМ и очистка ВЭЖХ после синтеза.
2. Сборка и отжиг дуплексов нуклеиновых кислот
- Ресуспендирование и разведение олигонуклеотидов
- Перед открытием центрифугируйте лиофилизированные олигонуклеотиды в пробирках объемом 2 мл на полной скорости в настольной центрифуге в течение 2-5 минут, чтобы убедиться, что нуклеиновая кислота находится на дне пробирки.
- Готовят исходное сырье в концентрации 100 мкМ путем ресуспендирования олигонуклеотидов в TE-буфере (10 мМ трис(гидроксиметил)аминометана (Трис), 1 мМ этилендиаминтетрауксусной кислоты (ЭДТА)). Убедитесь, что олигонуклеотиды полностью ресуспендированы путем многократного осторожного вихревания и кратковременного центрифугирования на полной скорости.
- Приготовьте массу 10 μМ, разбавив аликвоту основного материала буфером TE. Массу 10 мкМ разбавляют сверхчистой водой (MQ) для подготовки рабочих масс с концентрациями 0,5 мкМ, 0,7 мкМ или 2,5 мкМ в соответствии с таблицей 2.
- Сборка и отжиг реакционных мастер-смесей
- Используйте рабочие массы для составления реакционных мастер-смесей с использованием комбинаций, приведенных в таблице 2 , и объемов, приведенных в таблице 3. Для стандартного анализа ДНК-лигазы и большинства других описанных здесь анализов конечная буферная композиция представляет собой 50 мМ Tris pH 8,0, 50 мМ NaCl, 10 мМ дитиотреитол (DTT) с 10 мМ Mg в качестве двухвалентного катиона.
- Олигонуклеотиды отжигают в ПЦР или микроцентрифужной пробирке путем нагревания до 95 °C в течение 5 мин с помощью нагревательного блока или термоамплификатора. Дать остыть до комнатной температуры в течение 30 мин (объемы <1 мл) до 1 ч (объемы >1 мл). Для более длинных олигонуклеотидов (>40 нт) проведите более медленное охлаждение с помощью амплификатора с наклоном от 95 °C до 25 °C в течение 45 мин или поместите трубку со смесью для отжига в стакан объемом 1 л кипящей воды и дайте остыть до комнатной температуры в течение ночи.
- Добавьте нуклеотидные кофакторы и другие термочувствительные буферные компоненты в исходную смесь после охлаждения до комнатной температуры. Используйте окончательную реакционную смесь непосредственно для анализа путем добавления фермента (см. шаг 3 ниже) или храните при температуре -20 °C для использования в будущем.
3. Стандартная настройка анализа
- Сборка и инициирование пробирной реакции
- Смешайте 22,5 мкл представляющей интерес мастер-смеси субстрата с 2,5 мкл ДНК-лигазы или другого фермента, представляющего интерес, в ПЦР-пробирке. Запускайте реакции в двух или трех экземплярах, особенно если результаты будут количественно оценены.
- Включите контроль без белка (только буфер) в образцы анализа. При необходимости не включайте элементы управления кофактором на этом этапе.
ПРИМЕЧАНИЕ: Ферменты можно хранить при температуре -20 °C в 50% v/v глицерина, что позволяет их пипетировать непосредственно из раствора. Перед добавлением убедитесь, что ферментные растворы с глицерином хорошо перемешаны, либо с помощью пипетирования для смешивания, либо путем осторожного вортексинга.
- Немедленно перенесите реакции в ПЦР-машину при температуре 25 °C и инкубируйте в течение 30 минут. Варьируйте температуру и продолжительность в зависимости от оптимальных условий активности фермента.
- Погасите реакции, добавив 5 μL загружающего красителя (95% формамид, 0,5 М этилендиаминтетрауксусной кислоты (ЭДТА), бромфеноловый синий) и инкубируйте при 95 °C в течение 5 минут.
4. Анализ результатов анализов
- Приготовьте гели Tris-Borate-EDTA (TBE)-Urea PAGE, как описано ниже.
- Приготовьте запас из 20% акриламида, 7 М мочевины и 1x раствора TBE. Для описанного здесь набора олигонуклеотидов используйте раствор акриламида/Bis в соотношении 29:1 для оптимального разрешения.
- Для одного геля смешайте 10 мл 20% акриламида и 7 М раствора мочевины со 100 мкл APS (10 %) и 3 μл тетраметилэтилендимина (TMED) и смешайте в гелевом кастере.
- После того как гель застынет, прогоните образцы на геле мочевины TBE при температуре 45 - 55 °C.
- Предварительно запустите гель в буфере 1x TBE в течение 30 минут при 10 мА на гель с внешним нагревом.
- Удалите излишки мочевины в лунках геля, промыв 1х КЭ с помощью пастбищной пипетки.
- Загрузите по 10 мкл каждой реакции и запустите при 10 мА в течение 1,0-1,5 ч с внешним обогревом.
- Визуализируйте гель на тепловизоре с правильными настройками для выбранного флуорофора. Для FAM используйте набор фильтров, который дает возбуждение/излучение на длине волны 495/519 нм, который хранится как предустановленный в большинстве тепловизоров.
- Количественно оцените интенсивность полосы пропускания продукта и подложки с помощью программного обеспечения для обработки изображений с помощью имидж-сканера или внешней программы, такой как ImageJ49,50, и рассчитайте процентное содержание продукта по формуле.
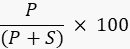
Где P — интегральное значение полосы продукта, а S — интегрированная площадь зоны подложки. В случае примера реакции ДНК-лигазы полоса продукта проходит при 40 нуклеотидах (nt), а полоса субстрата — при 20 nt.
5. Деаденилирование ДНК-лигазы для проверки специфичности кофактора
- Приготовление реакционных мастер-смесей
- Приготовьте один набор мастер-смеси, содержащей меченный FAM олигонуклеотид NL1, как описано в таблице 4. Приготовьте второй набор, содержащий олигонуклеотид NL1 без маркировки FAM, как описано в таблице 4.
- По отдельности нагрейте оба дуплекса ДНК до 95 °C в течение 5 минут и охладите от 30 минут до 1 часа при 25 °C. Не добавляйте нуклеотидный кофактор ни в одну из мастер-смесей.
- Сборка и инициация реакции деаденилирования
- Приготовьте одну реакцию деаденилирования для каждого типа/концентрации кофактора, подлежащего испытанию, путем объединения 10 мкл немеченой мастер-смеси с 2,5 мкл фермента лигазы.
- Приготовьте дополнительные пробирки для контроля без кофактора и без контроля белка (вместо фермента добавлено 2,5 мкл буфера).
- Инкубируйте реакции при температуре, соответствующей оптимальной активности фермента, в течение 1-2 часов. Время инкубации может быть увеличено, если фермент все еще аденилирован.
- Запустите реакцию лигирования с помощью кофактора.
- Добавьте 10 мкл меченой мастер-смеси и 2,5 мкл желаемых нуклеотидных кофакторов (например, АТФ, НАД, АДФ или ГТФ) непосредственно в реакцию деаденилированного (0,1-1 мМ конечная концентрация).
- Добавьте 2,5 мкл реакционного буфера к контролю без нуклеотидного кофактора.
- Инкубируйте реакции в течение того же периода времени и температуры, что и ранее. Закалите и визуализируйте так, как описано в шаге 4.
6. Использование подложек с двойной маркировкой для шинирования лигирования или сборки из нескольких частей
- Спроектируйте и приобретите олигонуклеотид с флуоресцентной группой, который имеет спектр возбуждения/излучения, отличный от уже используемого флуорофора.
- В описанной здесь схеме используют олигонуклеотид NL2 (TAMRA), имеющий 5-карбокситетрамилродамин (TAMRA) на 3'-конце (табл. 1).
- Соберите мастер-микс, как описано ниже.
- Объедините компоненты реакции, описанные на шаге 2, включая эквимолярные отношения всех олигонуклеотидов, используемых в сборке, а также буферные и двухвалентные катионы.
- Отжиг путем нагревания при 95 °C в течение 5 мин и охлаждения при 25 °C в течение 30 мин - 1 ч. Добавьте кофактор и фермент и инкубируйте, как описано в шаге 3.
- Запустите и визуализируйте образцы, как описано в шаге 4, используя соответствующие каналы для пары флуорофоров в субстрате. В случае FAM и TAMRA это каналы флуоресцеина (FITC) и тетраметилродамина (TRITC), присутствующие на большинстве тепловизоров.
7. Оценка связывания ДНК с помощью электрофоретического анализа сдвига подвижности (EMSA) на нативном геле
- Приготовьте 10% нативный гель TBE PAGE, как описано ниже.
- Смешайте 2,5 мл 40% акриламида, 1 мл 10x TBE, 100 мкл 10% APS, 3 мкл TMED и 6,5 мл MQ воды и смешайте в гелевой кастер.
- Соберите реакцию связывания, как описано ниже.
- Соберите подложку EMSA в соответствии с таблицей 5 так, чтобы в нее была включена ЭДТА (10 мМ) и отсутствовали ионы металлов.
- Смешайте 20 мкл мастер-смеси субстрата EMSA с 5 мкл белка в ПЦР-пробирке. Включите контрольный образец без белка. Инкубировать в течение 30 минут при температуре 25 °C.
- Анализ с помощью нативного электрофореза, как описано ниже.
- Добавьте в образцы 5 мкл 5-кратного нативного нагрузочного красителя (100 мМ ЭДТА, 0,25 % бромфенолового синего, 25 % v/v глицерина и MQ воды до 1 мл).
- Нагрузить приготовленный гель и запустить при напряжении 60 В в течение 2-3 ч с охлаждением циркуляцией воды до тех пор, пока фронт красителя не окажется на несколько см выше конца геля.
- Визуализируйте и проанализируйте гели, как описано в шаге 4.
Результаты
Лигирование по ДНК-лигазе
Ферментативная активность ДНК-лигазы приведет к увеличению размера флуоресцентно меченного олигонуклеотида при визуализации на геле мочевины PAGE. В случае субстратов для лигирования ДНК и РНК, перечисленных в таблице 2, это соответствует у...
Обсуждение
Критические шаги в протоколе
Дизайн и покупка олигонуклеотидов: При покупке олигонуклеотидов для образования дуплекса важно учитывать дизайн последовательности. Рекомендуется использовать инструмент олигоанализатора для прогнозирования свойств нуклеотидной последоват...
Раскрытие информации
SEG и UR являются сотрудниками компании ArcticZymes Technologies AS, которая занимается дистрибуцией R2D-лигазы. AW, ER-S и RS не имеют конкурирующих интересов.
Благодарности
AW поддерживается стипендией Rutherford Discovery Fellowship (20-UOW-004). РС является получателем стипендии Новой Зеландии для Постантарктической службы. SG и UR выражают благодарность Химическому институту при Университете Тромсё - Норвежскому арктическому университету за техническую поддержку.
Материалы
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
Ссылки
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003 (2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009 (2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263 (2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125 (2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391 (2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062 (2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14 (2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2 (2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148 (2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389 (2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097 (2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853 (2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52 (2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817 (2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97, 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711 (2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485 (2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998 (2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62 (2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257 (2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены