A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שימוש באוליגונוקלאוטידים סינתטיים שעברו שינוי כדי להעריך אנזימים לחילוף חומרים של חומצות גרעין
In This Article
Summary
כאן מוצג פרוטוקול לבדיקת אנזימים לחילוף חומרים של חומצות גרעין, תוך שימוש בדוגמאות של אנזימי ליגאז, נוקלאז ופולימראז. הבדיקה משתמשת באוליגונוקלאוטידים מסומנים פלואורסצנטית ולא מסומנים שניתן לשלב ליצירת דופלקסים המחקים נזקי RNA ו/או DNA או מתווכי מסלול, ומאפשרים אפיון התנהגות אנזימים.
Abstract
הזמינות של מגוון אוליגונוקלאוטידים סינתטיים מותאמים מספקים מסחריים אפשרה פיתוח של בדיקות מתוחכמות כדי לאפיין תכונות מגוונות של אנזימים מטבוליזם חומצת גרעין שניתן להריץ בכל מעבדה סטנדרטית לביולוגיה מולקולרית. השימוש בתוויות פלואורסצנטיות הפך את השיטות הללו לנגישות לחוקרים עם ציוד אלקטרופורזה סטנדרטי של PAGE ומכשיר הדמיה פלואורסצנטי, ללא שימוש בחומרים רדיואקטיביים וללא צורך במעבדה המיועדת לאחסון והכנה של חומרים רדיואקטיביים, כלומר מעבדת הוט. התוספת האופציונלית של שינויים סטנדרטיים כגון זרחן יכולה לפשט את הגדרת המבחן, בעוד שניתן להשתמש בשילוב ספציפי של נוקלאוטידים שעברו שינוי המחקים נזקי DNA או מתווכים כדי לחקור היבטים ספציפיים של התנהגות אנזימים. כאן, התכנון והביצוע של בדיקות כדי לחקור מספר היבטים של עיבוד DNA על ידי אנזימים באמצעות אוליגונוקלאוטידים סינתטיים זמינים מסחרית מודגמים. אלה כוללים את היכולת של ליגאזות להצטרף או nucleases כדי לפרק מבנים היברידיים שונים של DNA ו- RNA, שימוש בקו-פקטור דיפרנציאלי על ידי ליגאז DNA, והערכה של יכולת קשירת DNA של אנזימים. נדונים גורמים שיש לקחת בחשבון בעת תכנון מצעי נוקלאוטידים סינתטיים, ומסופקת קבוצה בסיסית של אוליגונוקלאוטידים שניתן להשתמש בהם עבור מגוון של חומצות גרעין ליגאז, פולימראז ובדיקות אנזימי נוקלאז.
Introduction
כל צורות החיים דורשות אנזימים לעיבוד חומצות גרעין כדי לבצע תהליכים ביולוגיים בסיסיים, כולל שכפול, שעתוק ותיקון DNA. פונקציות אנזימטיות עיקריות עבור מסלולים אלה הן פולימראזות, אשר מייצרות עותקים של מולקולות RNA/DNA, ליגאזות המצטרפות למצעי פולינוקלאוטידים, נוקלאזות המשפילות אותן, ומנחתים וטופואיזומראזות, אשר ממיסים דופלקסים של חומצות גרעין או משנים את הטופולוגיה שלהם 1,2,3,4,5,6,7,8,9,10 . בנוסף, רבים מהאנזימים הללו מספקים כלים מולקולריים חיוניים ליישומים כגון שיבוט, אבחון וריצוף בתפוקה גבוהה 11,12,13,14,15.
המאפיינים הפונקציונליים, הקינטיקה וייחודיות המצע של אנזימים אלה ניתנים לקביעה באמצעות מצעי DNA/RNA מסומנים המיוצרים על ידי חישול אוליגונוקלאוטידים. מעקב אחר מצעים ומוצרים הושג באופן מסורתי על ידי הצגת תווית רדיואקטיבית (32P) בקצה גדיל 5 ', אשר לאחר מכן ניתן לזהות על ידי סרט צילום או עם מערכת הדמיה זרחן16,17. בעוד שמצעים עם תווית רדיואקטיבית מציעים את היתרון של רגישות ניסויית מוגברת ואינם משנים את התכונות הכימיות של נוקלאוטיד, הסיכונים הבריאותיים הפוטנציאליים מעבודה עם רדיואיזוטופים עודדו את הפיתוח של תיוג חומצות גרעין לא רדיואקטיביות כדי לספק חלופה בטוחה יותר לזיהוי DNA ו- RNA 18,19,20 . בין אלה, זיהוי פלואורסצנטי, כולל זיהוי פלואורסצנטיות ישיר, פלואורסצנטיות שנפתרה בזמן ומבחני העברת אנרגיה/פלואורסצנטיות בולטים כ-21,22,23,24 הרב-תכליתיים ביותר. המערך הנרחב של פלואורופורים מאפשר עיצובים שונים של מצעי DNA/RNA הכוללים כתבים ייחודיים על כל אוליגונוקלאוטיד25. בנוסף, יציבותם של פלואורופורים, בהשוואה לרדיואיזוטופים, מאפשרת למשתמשים לייצר ולשמר כמויות משמעותיות של מצעי DNA המסומנים באופן פלואורסצנטי19. ניתן לדגור על מצעים אלה המסומנים בפלואורופור עם החלבון המעניין, יחד עם שילובים שונים של גורמים משלימים של מתכת ונוקלאוטידים, כדי לנתח את פעילות הקישור או האנזים. הדמיה של קשירה או פעילות ניתן לראות באמצעות תעלות צבע פלואורופור שונות עם מערכת הדמיה ג'ל. מכיוון שרק אוליגונוקלאוטידים המסומנים באופן פלואורסצנטי יהיו גלויים באמצעות טכניקה זו, כל עלייה או ירידה בגודל האוליגונוקלאוטיד המסומן תהיה קלה לעקוב. ג'לים יכולים גם להיות מוכתמים לאחר מכן, עם צבעים מכתימים חומצת גרעין כדי לדמיין את כל רצועות ה- DNA הקיימות על הג'ל.
ליגאזות של חומצות גרעין הן אנזימים המצטרפים למקטעים של DNA/RNA, ומזרזים את אטימת השברים על ידי יצירת קשר פוספודיסטר בין טרמיני DNA פוספורילציה 5' לבין 3' OH של DNA. ניתן לחלק אותם לשתי קבוצות בהתאם לדרישת מצע הנוקלאוטידים שלהם. הליגאזות התלויות ב-NAD שהשתמרו מאוד נמצאות בכל חיידקי26 ואילו את האנזימים תלויי ה-ATP המגוונים מבחינה מבנית ניתן לזהות בכל תחומי החיים 8,27. ליגאזות DNA ממלאות תפקיד חשוב בעיבוד מקטעי אוקזקי במהלך השכפול, כמו גם מעורבות במסלולי תיקון DNA שונים, כגון נוקלאוטיד ותיקון כריתת בסיס, באמצעות איטום ניקים וניקים ספונטניים שנותרים לאחר תיקון 8,10. ליגאזות דנ"א שונות מציגות יכולות שונות להצטרף לקונפורמציות שונות של שברי דנ"א, כולל ניקים בדופלקס, שברים דו-גדיליים, אי-התאמות ורווחים, כמו גם היברידיות RNA ו-DNA 28,29,30. ניתן להרכיב מגוון רחב של מצעים ניתנים לליגה על ידי חישול אוליגונוקלאוטידים עם פוספט 5' כדי ליצור טרמיני 5' ו-3' בדופלקס חומצות גרעין 31,32,33. שיטת הניתוח הנפוצה ביותר היא רזולוציה על ידי אוריאה PAGE בפורמט בדיקת נקודת קצה; עם זאת, חידושים אחרונים כללו שימוש באלקטרופורזה של ג'ל נימי, המאפשר תפוקה גבוהה34, פרופיל ספקטרומטרי מסה35, כמו גם בדיקת משואות מולקולרית הומוגנית, המאפשרת ניטור בזמן36.
הצעד הראשון בתגובת קשירה הוא אדנילציה של אנזים ליגאז על ידי אדנוזין טריפוספט (ATP) או ניקוטינאמיד אדנין דינוקלאוטיד (NAD), וכתוצאה מכך אנזים קוולנטי מתווך. השלב השני בתגובה הוא אדנילציה של מצע חומצת הגרעין בקצה 5' של אתר הניק, ואחריו קשירה של גדילי ניק חומצת הגרעין. אנזימי ליגאז רבים המתבטאים באופן רקומביננטי ב- E. coli מטוהרים בצורה אדנילציה, ולכן מסוגלים לשדר בהצלחה חומצות גרעין ללא תוספת של קו-פקטור נוקלאוטיד. זה מקשה לקבוע איזה סוג מסוים של קו-פקטור נוקלאוטידים הם דורשים עבור קשירת חומצות גרעין. בנוסף לתיאור בדיקות להערכת פעילות ליגאז DNA, מוצגת גם שיטה לקביעת שימוש בקו-פקטור באופן מהימן על ידי דה-אדנילציה של האנזים באמצעות מצעים לא מסומנים.
נוקלאזות הן קבוצה גדולה ומגוונת של אנזימים לשינוי DNA/RNA ורנ"א קטליטי המנתקים את קשרי הפוספודיסטרים בין חומצות גרעין37. תפקודי אנזימי נוקלאז נדרשים בשכפול, תיקון ועיבוד DNA וניתן לסווגם לפי ספציפיות הסוכר שלהם עבור DNA, RNA או שניהם. אנדונוקלאזות מבצעות הידרוליזה של קשרי פוספודיזטר בתוך גדיל דנ"א/רנ"א, בעוד אקסונוקלאזות הידרוליזות של גדילי דנ"א/רנ"א גוועדות נוקלאוטיד אחד בכל פעם מקצה ה-3' או ה-5' או ה-5' עד הקצה ה-5' של הדנ"א38'.
בעוד חלבוני נוקלאז רבים אינם ספציפיים ועשויים להיות מעורבים בתהליכים מרובים, אחרים ספציפיים מאוד עבור רצף מסוים או נזק לדנ"א 6,39,40. נוקלאזות ספציפיות לרצף משמשות במגוון רחב של יישומים ביוטכנולוגיים, כגון שיבוט, מוטגנזה ועריכת גנום. נוקלאזות פופולריות ליישומים אלה הן נוקלאזות הגבלה41, נוקלאזות אצבע אבץ42, נוקלאזות אפקט דמוי מפעיל שעתוק 43, ולאחרונה, נוקלאזות CRISPR מהונדסות מונחות רנ"א43. לאחרונה זוהו נוקלאזות ספציפיות לנזק, כגון נוקלאז EndoMS, שיש לו ספציפיות לאי-התאמות בדנ"א באמצעות תחום הנוקלאז דמוי RecB 5,44 הספציפי לאי-התאמה. מבחני פעילות נוקלאז, מבחינה היסטורית, נעשו כבדיקות לא רציפות עם מצעים מסומנים ברדיו; עם זאת, בנוסף לחסרונות האחרים שלהם, אלה אינם מאפשרים זיהוי של האתר שנחתך על ידי חלבון נוקלאז, אשר אפשרי בעת שימוש במצעים מסומנים פלואורסצנטית45,46. לאחרונה פותחו מבחני נוקלאז רציפים הפועלים באמצעות צבעי דנ"א שונים המקיימים אינטראקציה עם דנ"א במצבים שונים; לדוגמה, פליטת אות פלואורסצנטי גבוה יותר בעת אינטראקציה עם dsDNA מאשר במצבו הלא קשור, או קשירה ספציפית לרנ"א קצר47. בדיקות נוקלאז רציפות אחרות משתמשות בסיכות ראש DNA עם קבוצת פלואורופורים בקצה 5' ומרווה בקצה 3', כך שהפלואורסצנטיות עולה ככל שהאוליגונוקלאוטיד מתפרק עקב הפרדה בין הפלואורופור למרווה48. בעוד שבדיקות אלה מאפשרות לאפיין את הקינטיקה של חלבונים מפרקים DNA, הן דורשות ידע מוקדם על תפקוד האנזים והמצע ומוגבלות גם לאנזימים המשנים את קונפורמציית הדנ"א כדי לגרום להבדל בקישור הצבע. מסיבה זו, מבחני נקודות קצה שפותרים תוצרי נוקלאז בודדים עדיין רצויים כדי לספק תובנה לגבי שינויים בדנ"א הנגרמים על ידי פעילות החלבון.
כאן, מוצג הליך מפורט לתכנון אוליגונוקלאוטידים של DNA/RNA המסומנים באופן פלואורסצנטי שניתן לערבב ולהתאים ליצירת מצעים לבדיקת הפעילות של אנזימי נוקלאז, פולימראז וליגאז חדשים. האימות של קבוצה בסיסית זו של רצפי אוליגונוקלאוטידים מפשט את התכנון הניסיוני ומאפשר פרופיל חסכוני של מגוון רחב של פונקציות אנזימטיות ללא צורך לרכוש מספר רב של מצעים בהתאמה אישית. הליך מפורט מסופק להפעלת בדיקת אנזים סטנדרטית לעיבוד DNA עם מצעים אלה, תוך שימוש בדוגמה של פעילות ליגאז DNA ומתוארים שינויים להערכה וניתוח של אנזימי נוקלאז ופולימראז. בנוסף, ניתנת בדיקה שונה לקביעת ספציפיות הקו-פקטור של אנזים ליגאז ה- DNA בדיוק גבוה, ובדיקות בעלות תווית כפולה משמשות להערכת הרכבה של קשירה מרובת רכיבים. לבסוף, שינויים בפורמט הבדיקה הבסיסית נדונים כדי לאפשר להשתמש בו כדי לקבוע אינטראקציות חלבון-DNA עם אותם מצעים על ידי בדיקת שינוי ניידות אלקטרופורטית (EMSA).
Access restricted. Please log in or start a trial to view this content.
Protocol
1. תכנון ורכישה של אוליגונוקלאוטידים
הערה: תכננו אוליגונוקלאוטידים חד-גדיליים שיורכבו ויחושלו לדופלקסים הרצויים. אחד או יותר מהגדילים בדופלקס חייב לשאת מואטי פלואורסצנטי למעקב אחר עיבוד אוליגונוקלאוטיד על ידי האנזים המעניין. קבוצת בסיס של רצפים חד-גדיליים שניתן להרכיב למגוון פעילויות מוצגת בטבלה 1.
- שלבו את השינויים הספציפיים הדרושים לאנזים המעניין כמתואר להלן.
- עבור מצעי ליגאז דנ"א (איור 1): הרכיבו את המצע הפשוט ביותר משלושה אוליגונוקלאוטידים: גדיל תורם זרחני 5' (NL2), גדיל מקבל 5' FAM (NL1), ומשלים שמגשר בין השניים (NL3).
- ודא שגדילים המספקים את הקצה של 5 אינץ' של הניק הניתן לליגה עוברים פוספורילציה לפני הרכבת תערובת האב של המצע בשלב 2. הזמינו זאת כשינוי ב-NL2 (כפי שניתן בטבלה 1) או השתמשו בזרחן אנזימטי עם פולינוקלאוטיד קינאז T4 לאחר השעיה מחדש של האוליגונוקלאוטידים.
- כללו זרחן 5'-טרמינלי של NL6 ו-NL8, המהווים את המשלים של שברים דו-גדיליים המתוארים באיור 1A (NL6/NL7 ו-NL8/NL9), שכן זה דומה ביותר למצע הטבעי המופק מאנדונוקלאז הגבלה. השתמש במצע בעל תווית כפולה כדי לקבוע את ההיקפים היחסיים של קשירה עבור הרכבות מרובות חלקים (ראה שלב 6).
- שנה את גדיל המשלים כדי ליצור אי-התאמות (NL10) ורווחים (NL11).
הערה: וריאציות על המצע הפשוט מתוארות באיור 1A. ניתן להשתמש ברצפים אחרים כדי ליצור טווח רחב עוד יותר של אי-התאמות או רווחים ארוכים יותר על ידי שינוי המיקום המסומן בקו תחתון. - תחליף אוליגונוקלאוטידים של DNA באוליגונוקלאוטידים של RNA.
הערה: וריאציות על המצע הפשוט מתוארות באיור 1B. טווח רחב יותר של דופלקסים של דנ"א/רנ"א יכול להיווצר על ידי שילובים נוספים של קבוצת הבסיס שניתנה כאן כדי ליצור, למשל, הפסקות דו-גדיליות המכילות גם רנ"א וגם דנ"א. דוגמה לווריאציה זו ניתנת בשלב 6 להלן, שבו נעשה שימוש באסטרטגיית תווית כפולה.
- עבור מצעי DNA פולימראז: הרכיבו את האוליגונוקלאוטידים NL1 ו-NL3 המפורטים בטבלה 1 כדי לתת בדיקת הרחבת פריימר פשוטה. חקור היבטים נוספים של פעילות פולימראז על-ידי הכנסת שינויים לגדילים NL1 (פריימר) או NL3 (תבנית).
- שלב אנלוגים בסיסיים פגומים באוליגונוקלאוטיד NL3 לפני מיקום 20 כדי לקבוע את היכולת לעקוף נגעים פגומים על גדיל התבנית.
- שלב אנלוגים בסיסיים פגומים באוליגונוקלאוטיד NL1 במיקום 20 כדי לקבוע את היכולת להאריך פריימר פגום.
- השתמש ב- RNL1 או ב- RNL3 בדופלקס כדי לחקור את ההרחבה של פריימר RNA או את השימוש בתבנית RNA.
- עבור מצעי נוקלאז (איור 2): הרכיבו אוליגונוקלאוטידים כדי לתת טווח לא ממצה של מצעים כפולים וחד-גדיליים (איור 2Ai), כמו גם טווח של צמתים מנופפים ומשובצים (איור 2Aii) ומצעים פגומים (איור 2B).
- כדי לחקור פעילויות ריבונוקלאז, החלף באופן איטרטיבי את NL1, NL2 ו- NL3 ב- RNL1, RNL2 ו- RNL3. השתמש בגרסאות RNA נוספות של HJ5 ו- HJ6 כדי להרחיב קבוצה זו עוד יותר.
- השתמשו באוליגונוקלאוטידים MD5, MD6 ו-MD9 שיש להם שינוי במיקום מרכזי שמחקה נזק חמצוני, חומר ביניים לתיקון בסיסי או תוצר דה-אמינציה (איור 2B). המצעים יזהו שסע של הגדיל במיקום זה. תייג את גדיל NL3 המשלים עם פלואורופור אורתוגונלי כגון TAMRA כדי לזהות מחשוף דו-גדילי (ראה שלב 6).
- השתמש בתיוג אורתוגונלי של המשלים כדי לזהות חיתוך דו-גדילי באתרים שאינם תואמים הן על גדילי הבדיקה (NL5 ו- ND9) והן על גדילי המשלים (MD10 ו- NL10).
- עבור מצעי ליגאז דנ"א (איור 1): הרכיבו את המצע הפשוט ביותר משלושה אוליגונוקלאוטידים: גדיל תורם זרחני 5' (NL2), גדיל מקבל 5' FAM (NL1), ומשלים שמגשר בין השניים (NL3).
- להזמין אוליגונוקלאוטידים סינתטיים המשלבים פלואורופורים רלוונטיים ושינויים אחרים מספק מסחרי.
הערה: סולם סינתזה של 100 ננומטר וטיהור HPLC לאחר סינתזה מתאימים לבדיקות המתוארות.
2. הרכבה וחישול של דופלקסים של חומצות גרעין
- השעיה ודילול של אוליגונוקלאוטידים
- לפני הפתיחה, צנטריפוגו את האוליגונוקלאוטידים הליופיליים בצינורות 2 מ"ל שלהם במהירות מלאה בצנטריפוגה על ספסל למשך 2-5 דקות כדי להבטיח שחומצת הגרעין נמצאת בתחתית הצינור.
- הכינו מלאי אב של 100 מיקרומטר על ידי השעיה מחדש של האוליגונוקלאוטידים במאגר TE (10 mM tris(hydroxymethyl)aminomethane (Tris), 1 mM ethylenediaminetetraacetic acid (EDTA)). ודא שהאוליגונוקלאוטידים מושהים מחדש ביסודיות על ידי מערבולות עדינות חוזרות ונשנות וצנטריפוגות קצרות במלוא המהירות.
- הכינו מלאי של 10 מיקרומטר על ידי דילול אליציטוט של מלאי מאסטר עם מאגר TE. יש לדלל את מלאי ה-10 מיקרומטר במים אולטרה-טהורים (MQ water) כדי להכין מלאי עבודה בריכוזים של 0.5 מיקרומטר, 0.7 מיקרומטר או 2.5 מיקרומטר לפי טבלה 2.
- הרכבה וחישול של תערובות מאסטר התגובה
- השתמש במניות עבודה כדי להרכיב את תערובות האב לתגובה באמצעות הצירופים המופיעים בטבלה 2 והנפחים המופיעים בטבלה 3. עבור בדיקת ליגאז DNA סטנדרטית ורוב הבדיקות האחרות המתוארות כאן, הרכב החיץ הסופי הוא 50 mM Tris pH 8.0, 50 mM NaCl, 10 mM Dithiothreitol (DTT) עם 10 mM Mg כקטיון דו-ערכי.
- אנאל את האוליגונוקלאוטידים בצינור PCR או מיקרוצנטריפוגה על ידי חימום ב 95 ° C במשך 5 דקות באמצעות בלוק חימום או thermocycler. מצננים לטמפרטורת החדר למשך 30 דקות (נפחים <1 מ"ל) עד שעה אחת (נפחים >1 מ"ל). עבור אוליגונוקלאוטידים ארוכים יותר (>40 nt), בצע קירור איטי יותר באמצעות thermocycler עם רמפה למטה של 95 ° C עד 25 ° C במשך 45 דקות, או לצוף את הצינור המכיל תערובת חישול בכוס 1 L של מים רותחים ולאפשר להתקרר לטמפרטורת החדר במשך הלילה.
- הוסף גורמים משלימים של נוקלאוטידים ורכיבי חיץ רגישים אחרים לחום לתערובת האב לאחר קירור לטמפרטורת החדר. השתמש בתערובת התגובה הסופית ישירות לבדיקה על ידי תוספת של אנזים (ראה שלב 3 להלן) או לאחסן ב -20 ° C לשימוש עתידי.
3. הגדרת בדיקה סטנדרטית
- הרכבה וייזום של תגובת הבדיקה
- שלב 22.5 μL של תערובת האב המצע של עניין עם 2.5 μL של ליגאז DNA או אנזים אחר של עניין בצינור PCR. הפעל תגובות כפולות או משולשות, במיוחד אם התוצאות יכומתו.
- כלול בקרה ללא חלבון (מאגר בלבד) בדגימות הבדיקה. אין לכלול בקרות קו-פקטור בשלב זה, במידת הצורך.
הערה: ניתן לאחסן אנזימים בטמפרטורה של -20°C בגליצרול 50% v/v, מה שמאפשר לגרוף אותם ישירות מתמיסה. ודא שתמיסות אנזימים עם גליצרול מעורבבות היטב לפני התוספת, בין אם על ידי פיפטינג לערבוב או על ידי ערבול עדין.
- מיד להעביר את התגובות למכונת PCR ב 25 ° C ולדגור במשך 30 דקות. שנו את הטמפרטורה ואת משך הזמן בהתאם לתנאים האופטימליים לפעילות האנזים.
- להרוות את התגובות על ידי הוספת 5 μL של צבע העמסה (95% פורממיד, 0.5 M Ethylenediaminetetraacetic חומצה (EDTA), bromophenol כחול) ולדגור על 95 ° C במשך 5 דקות.
4. ניתוח תוצאות הבדיקה
- הכינו ג'לים Tris-Borate-EDTA (TBE)-Urea PAGE כמתואר להלן.
- הכינו מלאי של 20% אקרילאמיד, 7 M Urea ותמיסת TBE 1x. עבור ערכת האוליגונוקלאוטידים המתוארת כאן, השתמש בתמיסת אקרילאמיד/ביס ביחס של 29:1 לקבלת רזולוציה אופטימלית.
- עבור ג'ל אחד, ערבבו 10 מ"ל של 20% אקרילאמיד ותמיסת אוריאה 7M עם 100 μL של APS (10%) ו-3 μL של טטרמתילאתילאנדיאמין (TMED) ויצקו ביציקת ג'ל.
- לאחר שהג'ל מתמצק, הפעל את הדגימות על ג'ל אוריאה TBE ב 45 - 55 מעלות צלזיוס.
- הפעילו מראש את הג'ל במאגר TBE אחד למשך 30 דקות ב-10 mA לג'ל עם חימום חיצוני.
- הסר עודף אוריאה בבארות של ג'ל על ידי שטיפה עם 1x TBE באמצעות פיפטה מרעה.
- טען 10 μL מכל תגובה והפעל ב 10 mA במשך 1.0-1.5 שעות עם חימום חיצוני.
- דמיינו את הג'ל במכונת ההדמיה עם ההגדרות הנכונות עבור הפלואורופור שנבחר. ל-FAM, השתמשו בערכת מסננים המעניקה עירור/פליטה ברזולוציה של 495/519 ננומטר, המאוחסנת כקביעה מוגדרת מראש ברוב מכשירי ההדפוס.
- לכמת את עוצמת הפס של המוצר והמצע באמצעות תוכנת עיבוד תמונה עם מכונת ההדמייה, או תוכנה חיצונית כגון ImageJ49,50 ולחשב את אחוז המוצר באמצעות הנוסחה
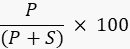
כאשר P הוא הערך המשולב של פס המכפלות, ו - S הוא האזור המשולב של רצועת המצע. במקרה של דוגמת תגובת ליגאז DNA, רצועת המכפלה פועלת ב-40 נוקלאוטידים (nt) ורצועת המצע ב-20 nt.
5. דה-אדנילציה של ליגאז ה-DNA לבדיקת ספציפיות קו-פקטור
- הכנת תערובות מאסטר תגובה
- הכינו ערכה אחת של תערובת האב המכילה את אוליגונוקלאוטיד NL1 המסומן בתווית FAM כמתואר בטבלה 4. הכינו ערכה שנייה המכילה את האוליגונוקלאוטיד NL1 ללא תווית FAM, כמתואר בטבלה 4.
- בנפרד, חממו את שני דופלקסי הדנ"א ל-95°C למשך 5 דקות וקררו למשך 30 דקות עד שעה ב-25°C. אין להוסיף קו-פקטור נוקלאוטידים לאף אחת מתערובות האב.
- הרכבה וייזום של תגובת דה-אדנילציה
- הכינו תגובת דה-אדנילציה יחידה עבור כל סוג / ריכוז קו-פקטור שייבדקו על ידי שילוב של 10 μL של תערובת האב ללא תווית עם 2.5 μL של אנזים ליגאז.
- הכינו צינורות נוספים כבקרה ללא קו-פקטור וללא בקרת חלבונים (2.5 מיקרוליטר של חיץ שנוסף במקום אנזים).
- לדגור על התגובות בטמפרטורה ספציפית לפעילות האופטימלית של האנזים, למשך 1-2 שעות. זמן הדגירה יכול להיות מוגבר אם האנזים הוא עדיין adenylated.
- הפעל את תגובת הקשירה עם הקו-פקטור.
- הוסף 10 μL של תערובת האב המסומנת ו- 2.5 μL של הגורמים משלימים של הנוקלאוטידים הרצויים (לדוגמה, ATP, NAD, ADP או GTP) ישירות לתגובה דה-אדניל (ריכוז סופי של 0.1-1 mM).
- הוסף 2.5 μL של מאגר תגובה לבקרת הקו-פקטור ללא נוקלאוטידים.
- לדגור את התגובות עבור אותו פרק זמן וטמפרטורה כמו בשימוש בעבר. הרוו והציגו באופן חזותי כמתואר בשלב 4.
6. שימוש במצעים בעלי תווית כפולה לקשירת סד או הרכבה מרובת חלקים
- תכננו ורכשו אוליגונוקלאוטיד עם מואטי פלורסנטי בעל ספקטרום עירור / פליטה שונה לפלואורופור שכבר נמצא בשימוש.
- במערך המתואר כאן, השתמש באוליגונוקלאוטיד NL2 (TAMRA) בעל 5-Carboxytetramethylrhodamine (TAMRA) בקצה 3' (טבלה 1).
- הרכיבו את תערובת האב כמתואר להלן.
- שלב את מרכיבי התגובה המתוארים בשלב 2, כולל יחסים שווי ערך של כל האוליגונוקלאוטידים המשמשים בהרכבה, כמו גם קטיונים חוצצים ודיוולנטיים.
- אנאל על ידי חימום ב 95 ° C במשך 5 דקות וקירור ב 25 ° C במשך 30 דקות - 1 שעות. הוסף את הקו-פקטור ואת האנזים ודגור כמתואר בשלב 3.
- הפעל וצלם את הדגימות כמתואר בשלב 4 באמצעות התעלות המתאימות לזוג הפלואורופורים במצע. במקרה של FAM ו-TAMRA, אלה הם תעלות פלואורסצאין (FITC) וטטרמתיל רודאמין (TRITC) הקיימות ברוב מכשירי ההדמיה.
7. הערכה של קשירת DNA על ידי Electrophoretic Mobility Shift Assay (EMSA) על ג'ל טבעי
- הכינו ג'ל TBE PAGE מקורי 10% כמתואר להלן.
- יש לערבב 2.5 מ"ל של 40% אקרילאמיד, 1 מ"ל של 10x TBE, 100 μL של 10% APS, 3 μL של TMED ו-6.5 מ"ל של מי MQ ולצקת ביציקת ג'ל.
- הרכיבו את התגובה המחייבת כמתואר להלן.
- הרכיבו את מצע ה-EMSA לפי טבלה 5 כך שייכלל EDTA (10 מילימטר) ויושמטו יוני מתכת.
- ערבבו 20 μL של תערובת האב של מצע EMSA עם 5 μL של החלבון בצינור PCR. כלול דגימה ללא בקרת חלבון. יש לדגור במשך 30 דקות ב-25°C.
- נתח על ידי אלקטרופורזה טבעית כמתואר להלן.
- הוסף 5 μL של צבע העמסה טבעי 5x (100 mM EDTA, 0.25% ברומופנול כחול, 25% v/v גליצרול, ומי MQ עד 1 מ"ל) לדגימות.
- מעמיסים על הג'ל המוכן ופועלים במתח של 60 וולט למשך 2-3 שעות עם קירור על ידי סירקולציית מים עד שחזית הצבע נמצאת כמה סנטימטרים מעל קצה הג'ל.
- הצג ונתח ג'לים כמתואר בשלב 4.
Access restricted. Please log in or start a trial to view this content.
תוצאות
קשירה על ידי DNA ligase
פעילות אנזימטית של ליגאז DNA תגרום להגדלת גודלו של האוליגונוקלאוטיד המסומן באופן פלואורסצנטי כאשר הוא מוצג על ג'ל PAGE של אוריאה. במקרה של הסובסטרטים עבור קשירת דנ"א ורנ"א המפורטים בטבלה 2, זה מתאים להכפלת הגודל מ-20 nt ל-40 nt (איור 3A). פעילות א?...
Access restricted. Please log in or start a trial to view this content.
Discussion
שלבים קריטיים בפרוטוקול
תכנון ורכישה של אוליגונוקלאוטידים: בעת רכישת אוליגונוקלאוטידים ליצירת דופלקס, חיוני לשקול תכנון רצף. מומלץ להשתמש בכלי אנלייזר אוליגו לחיזוי תכונות רצף הנוקלאוטידים, כגון תכולת GC, טמפרטורת התכה, מבנה משני ופוטנציאל דימריזציה, לפני הזמנת57.<...
Access restricted. Please log in or start a trial to view this content.
Disclosures
SEG ו-UR הם עובדים של ArcticZymes Technologies AS, המפיצה את ליגאז R2D. ל-AW, ER-S ו-RS אין אינטרסים מתחרים.
Acknowledgements
AW נתמך על ידי מלגת Rutherford Discovery Fellowship (20-UOW-004). RS היא זוכת מלגת ניו זילנד פוסט אנטארקטיקה. SG ו- UR מודים למכון הכימי באוניברסיטת טרומסו - האוניברסיטה הארקטית הנורבגית לתמיכה טכנית.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 30% Acrylamide/Bis Solution (29:1) | BioRad | 1610156 | |
| Adenosine triphosphate (ATP) | Many suppliers | ||
| Ammonium persulfate (APS) | Many suppliers | ||
| Benchtop centrifuge | Many suppliers | ||
| Borate | Many suppliers | ||
| Bromophenol blue | Many suppliers | ||
| Dithiothreitol (DTT) | Many suppliers | ||
| Electrophoresis system with circulating water bath | Many suppliers | ||
| Ethylenediaminetetraacetic acid (EDTA) | Many suppliers | ||
| Fluoresnence imager, e.g. iBright FL1000 | Thermo Fisher Scientific | A32752 | |
| Formamide | Many suppliers | ||
| Gel casting system | Many suppliers | ||
| Heating block | Many suppliers | ||
| Magnesium Chloride | Many suppliers | Other metal ions may be preferred depending on the protein studied | |
| Microcentrifuge tubes (1.5 mL) | Many suppliers | ||
| Micropipettes and tips | Many suppliers | 1 mL, 0.2 mL, 0.02 mL, 0.002 mL | |
| Nicotinamide adenine dinucleotide (NAD+) | Many suppliers | ||
| Oligonucleotides | Integrated DNA Technologies | NA | Thermo Fisher, Sigma-Aldrich, Genscript and others also supply these |
| pasture pipette | Many suppliers | ||
| PCR thermocycler | Many suppliers | ||
| PCR tubes | Many suppliers | ||
| RNAse away | ThermoFisher | 7002PK | Only needed when working with RNA oligos |
| RNase AWAY | Merck | 83931-250ML | Surfactant for removal of RNAse contamination on surfaces |
| RNAse-free water | New England Biolabs | B1500L | Only needed when working with RNA oligos |
| Sodium Chloride | Many suppliers | ||
| SUPERase IN RNase inhibitor | Thermo Fisher Scientific | AM2694 | Broad spectrum RNAse inhibitir (protein-based) |
| SYBR Gold | Thermo Fisher Scientific | S11494 | This may be used to post-stain gels and visualise unlabelled oligonucleotides |
| Tetramethylethylenediamine (TMED) | Many suppliers | ||
| Tris, or tris(hydroxymethyl)aminomethane | Many suppliers | ||
| Ultrapure water (Milli-Q) | Merck | ||
| urea | Many suppliers | ||
| Vortex | Many suppliers |
References
- Gao, Y., et al. Structures and operating principles of the replisome. Science. 363 (6429), 7003(2019).
- Yang, W., Gao, Y. Translesion and repair DNA polymerases: Diverse structure and mechanism. Annu Rev Biochem. 87, 239-261 (2018).
- Lohman, T. M., Fazio, N. T. How does a helicase unwind DNA? Insights from RecBCD Helicase. Bioessays. 40 (6), e1800009(2018).
- Ahdash, Z., et al. Mechanistic insight into the assembly of the HerA-NurA helicase-nuclease DNA end resection complex. Nucleic Acids Res. 45 (20), 12025-12038 (2017).
- Wozniak, K. J., Simmons, L. A. Bacterial DNA excision repair pathways. Nat Rev Microbiol. 20 (8), 465-477 (2022).
- Zhang, L., Jiang, D., Wu, M., Yang, Z., Oger, P. M. New insights into DNA repair revealed by NucS endonucleases from hyperthermophilic Archaea. Front Microbiol. 11, 1263(2020).
- Saathoff, J. H., Kashammer, L., Lammens, K., Byrne, R. T., Hopfner, K. P. The bacterial Mre11-Rad50 homolog SbcCD cleaves opposing strands of DNA by two chemically distinct nuclease reactions. Nucleic Acids Res. 46 (21), 11303-11314 (2018).
- Williamson, A., Leiros, H. S. Structural insight into DNA joining: from conserved mechanisms to diverse scaffolds. Nucleic Acids Res. 48 (15), 8225-8242 (2020).
- Caglayan, M. Interplay between DNA polymerases and DNA ligases: Influence on substrate channeling and the fidelity of DNA ligation. J Mol Biol. 431 (11), 2068-2081 (2019).
- Shuman, S. DNA ligases: Progress and prospects. J Biol Chem. 284 (26), 17365-17369 (2009).
- Lohman, G. J., Tabor, S., Nichols, N. M. DNA ligases. Curr Prot Mol Biol. , Chapter 3, Unit 3.14 (2011).
- Rittié, L., Perbal, B. Enzymes used in molecular biology: a useful guide. J Cell Commun Signal. 2 (1-2), 25-45 (2008).
- Chandrasegaran, S., Carroll, D. Origins of programmable nucleases for genome engineering. J Mol Biol. 428 (5), Part b 963-989 (2016).
- Aschenbrenner, J., Marx, A. DNA polymerases and biotechnological applications. Curr Opin Biotechnol. 48, 187-195 (2017).
- Loenen, W. A. M., Dryden, D. T. F., Raleigh, E. A., Wilson, G. G., Murray, N. E. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res. 42 (1), 3-19 (2013).
- Voytas, D., Ke, N. Detection and quantitation of radiolabeled proteins and DNA in gels and blots. Curr Protoc Immunol. , Appendix 3 (A) (2002).
- Phillips, D. H. Detection of DNA modifications by the 32P-postlabelling assay. Mutat Res. 378 (1-2), 1-12 (1997).
- Huang, C., Yu, Y. T. Synthesis and labeling of RNA in vitro. Curr Prot Mol Biol. , Chapter 4, Unit 4.15 (2013).
- Ballal, R., Cheema, A., Ahmad, W., Rosen, E. M., Saha, T. Fluorescent oligonucleotides can serve as suitable alternatives to radiolabeled oligonucleotides. J Biomol Tech. 20 (4), 190-194 (2009).
- Anderson, B. J., Larkin, C., Guja, K., Schildbach, J. F. Using fluorophore-labeled oligonucleotides to measure affinities of protein-DNA interactions. Meth Enzymol. 450, 253-272 (2008).
- Liu, W., et al. Establishment of an accurate and fast detection method using molecular beacons in loop-mediated isothermal amplification assay. Sci Rep. 7 (1), 40125(2017).
- Ma, C., et al. Simultaneous detection of kinase and phosphatase activities of polynucleotide kinase using molecular beacon probes. Anal Biochem. 443 (2), 166-168 (2013).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Meth Mol Biol. 429, 209-224 (2008).
- Yang, C. J., Li, J. J., Tan, W. Using molecular beacons for sensitive fluorescence assays of the enzymatic cleavage of nucleic acids. Meth Mol Biol. 335, 71-81 (2006).
- Nikiforov, T. T., Roman, S. Fluorogenic DNA ligase and base excision repair enzyme assays using substrates labeled with single fluorophores. Anal Biochem. 477, 69-77 (2015).
- Pergolizzi, G., Wagner, G. K., Bowater, R. P. Biochemical and structural characterisation of DNA ligases from bacteria and Archaea. Biosci Rep. 36 (5), 00391(2016).
- Martin, I. V., MacNeill, S. A. ATP-dependent DNA ligases. Genome Biol. 3 (4), REVIEWS3005 (2002).
- Bilotti, K., et al. Mismatch discrimination and sequence bias during end-joining by DNA ligases. Nucleic Acids Res. 50 (8), 4647-4658 (2022).
- Bauer, R. J., et al. Comparative analysis of the end-joining activity of several DNA ligases. PLoS One. 12 (12), e0190062(2017).
- Lohman, G. J. S., Zhang, Y., Zhelkovsky, A. M., Cantor, E. J., Evans, T. C. Efficient DNA ligation in DNA-RNA hybrid helices by Chlorella virus DNA ligase. Nucleic Acids Res. 42 (3), 1831-1844 (2014).
- Bullard, D. R., Bowater, R. P. Direct comparison of nick-joining activity of the nucleic acid ligases from bacteriophage T4. Biochem J. 398 (1), 135-144 (2006).
- Magnet, S., Blanchard, J. S. Mechanistic and kinetic study of the ATP-dependent DNA ligase of Neisseria meningitidis. Biochemistry. 43 (3), 710-717 (2004).
- Williamson, A., Grgic, M., Leiros, H. S. DNA binding with a minimal scaffold: structure-function analysis of Lig E DNA ligases. Nucleic Acids Res. 46 (16), 8616-8629 (2018).
- Lohman, G. J., et al. A high-throughput assay for the comprehensive profiling of DNA ligase fidelity. Nucleic Acids Res. 44 (2), e14(2016).
- Kim, J., Mrksich, M. Profiling the selectivity of DNA ligases in an array format with mass spectrometry. Nucleic Acids Res. 38 (1), e2(2010).
- Tang, Z. W., et al. Real-time monitoring of nucleic acid ligation in homogenous solutions using molecular beacons. Nucleic Acids Res. 31 (23), e148(2003).
- Yang, W. Nucleases: diversity of structure, function and mechanism. Q Rev Biophys. 44 (1), 1-93 (2011).
- Marti, T. M., Fleck, O. DNA repair nucleases. Cell Mol Life Sci. 61 (3), 336-354 (2004).
- Wang, B. B., et al. Review of DNA repair enzymes in bacteria: With a major focus on AddAB and RecBCD. DNA Repair. 118, 103389(2022).
- Pidugu, L. S., et al. Structural insights into the mechanism of base excision by MBD4. J Mol Biol. 433 (15), 167097(2021).
- Roberts, R. J. How restriction enzymes became the workhorses of molecular biology. Proc Natl Acad Sci U S A. 102 (17), 5905-5908 (2005).
- Miller, J. C., et al. An improved zinc-finger nuclease architecture for highly specific genome editing. Nat Biotechnol. 25 (7), 778-785 (2007).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Takemoto, N., Numata, I., Su'etsugu, M., Miyoshi-Akiyama, T. Bacterial EndoMS/NucS acts as a clamp-mediated mismatch endonuclease to prevent asymmetric accumulation of replication errors. Nucleic Acids Res. 46 (12), 6152-6165 (2018).
- Reardon, J. T., Sancar, A. Molecular anatomy of the human excision nuclease assembled at sites of DNA damage. Mol Cell Biol. 22 (16), 5938-5945 (2002).
- Kunkel, T. A., Soni, A. Exonucleolytic proofreading enhances the fidelity of DNA synthesis by chick embryo DNA polymerase-gamma. J Biol Chem. 263 (9), 4450-4459 (1988).
- Sheppard, E. C., Rogers, S., Harmer, N. J., Chahwan, R. A universal fluorescence-based toolkit for real-time quantification of DNA and RNA nuclease activity. Sci Rep. 9 (1), 8853(2019).
- Li, J. J., Geyer, R., Tan, W. Using molecular beacons as a sensitive fluorescence assay for enzymatic cleavage of single-stranded DNA. Nucleic Acids Res. 28 (11), e52(2000).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nat Meth. 9 (7), 671-675 (2012).
- Sharma, J. K., et al. Methods for competitive enrichment and evaluation of superior DNA ligases. Meth Enzymol. 644, 209-225 (2020).
- Rzoska-Smith, E., Stelzer, R., Monterio, M., Cary, S. C., Williamson, A. DNA repair enzymes of the Antarctic Dry Valley metagenome. Front Microbiol. 14, 1156817(2023).
- Williamson, A., Pedersen, H. Recombinant expression and purification of an ATP-dependent DNA ligase from Aliivibrio salmonicida. Protein Expres Purif. 97 (0), 29-36 (2014).
- Akey, D., et al. Crystal structure and nonhomologous end-joining function of the ligase component of Mycobacterium DNA ligase D. J Biol Chem. 281 (19), 13412-13423 (2006).
- Kim, D. J., et al. ATP-dependent DNA ligase from Archaeoglobus fulgidus displays a tightly closed conformation. Acta Crystallogr Sect F Struct Biol Cryst Commun. 65, Pt 6 544-550 (2009).
- Nishida, H., Kiyonari, S., Ishino, Y., Morikawa, K. The closed structure of an archaeal DNA ligase from Pyrococcus furiosus. J Mol Biol. 360 (5), 956-967 (2006).
- Gundesø, S., et al. R2D ligase: Unveiling a novel DNA ligase with surprising DNA-to-RNA ligation activity. Biotechnol J. 19 (3), e2300711(2024).
- Hendling, M., Barišić, I. In silico design of DNA oligonucleotides: Challenges and approaches. Comput Struct Biotechnol J. 17, 1056-1065 (2019).
- Green, M. R., Sambrook, J. How to win the battle with RNase. Cold Spring Harb Prot. 2019 (2), 10.1101/pdb.top101857 (2019).
- Summer, H., Grämer, R., Dröge, P. Denaturing urea polyacrylamide gel electrophoresis (Urea PAGE). J Vis Exp. (32), e1485(2009).
- Smith, D. R. Gel Electrophoresis of DNA. Mol Biometh Handbook. , Humana Press. Totowa, NJ. 17-33 (1998).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp. (63), e3998(2012).
- Rousseau, M., et al. Characterisation and engineering of a thermophilic RNA ligase from Palaeococcus pacificus. Nucleic Acids Res. 52 (7), 3924-3937 (2024).
- Kestemont, D., Herdewijn, P., Renders, M. Enzymatic synthesis of backbone-modified oligonucleotides using T4 DNA ligase. Curr Prot Chem Biol. 11 (2), e62(2019).
- Farell, E. M., Alexandre, G. Bovine serum albumin further enhances the effects of organic solvents on increased yield of polymerase chain reaction of GC-rich templates. BMC Res Notes. 5, 257(2012).
- Nazarenko, I., Pires, R., Lowe, B., Obaidy, M., Rashtchian, A. Effect of primary and secondary structure of oligodeoxyribonucleotides on the fluorescent properties of conjugated dyes. Nucleic Acids Res. 30 (9), 2089-2195 (2002).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved