Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Raumzeitlich kontrollierte nukleare Translokation der Gäste in lebenden Zellen mit Käfig molekulare Leime als Photoactivatable Tags
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt Licht ausgelöste nukleare Translokation der Gäste in lebenden Zellen mit Käfig molekularen Klebstoff-Tags. Diese Methode ist viel versprechend für Website-selektiv-targeting Drug-Delivery.
Zusammenfassung
Der Zellkern ist eines der wichtigsten Organellen als subzelluläre Drug Delivery-Ziel seit Modulation der gen-Replikation und Ausdruck ist wirksam zur Behandlung von verschiedenen Krankheiten. Hier zeigen wir, dass Licht ausgelöste nukleare Translokation der Gäste mit molekularen Klebstoff (CagedKleber-R) Tags, sperrte deren mehrere Guanidinium Ion (Gu+) Anhänger von einer anionischen Photocleavable Gruppe (Butyrat-substituierten geschützt sind Nitroveratryloxycarbonyl; BA SVOC). Gäste, die mit dem Stichwort CagedKleber-R sind berücksichtigt lebenden Zellen über Endozytose und bleiben in Endosomen. Allerdings wird auf Photoirradiation, CagedKleber-R in uncaged molekularen Klebstoff (UncagedKleber-R) tragen mehrere Gu+ Anhänger umgewandelt, die Endosomal entweichen und anschließenden nuklearen Translokation der Gäste erleichtert. Diese Methode ist vielversprechend für Website-selektiv-targeting Drug-Delivery, da tagged Gäste in das Zytoplasma, gefolgt von den Zellkern übergehen können nur dann, wenn Photoirradiated. Im Käfig Kleber-R Tags können makromolekularen Gäste wie Quantenpunkte (QDs) sowie kleine Molekül Gäste liefern. Im Käfig Kleber-R Tags sind uncaged mit nicht nur UV-Licht, sondern auch zwei-Photon Nah-Infrarot (NIR) Licht, das tief in das Gewebe eindringen kann.
Einleitung
Der Zellkern, der genetischen Information trägt, ist eines der wichtigsten Organellen als subzelluläre Drug Delivery-Ziel seit Modulation der gen-Replikation und Ausdruck ist wirksam zur Behandlung von verschiedenen Krankheiten einschließlich Krebs und genetische 1,2,3-Störungen. Für nukleare Lieferung von Medikamenten Konjugation von Peptid Stichwörter wie nukleare Lokalisierung Signale (NLS)4,5,6 allgemein untersucht worden ist. Um unerwünschte Nebenwirkungen zu reduzieren, ist jedoch räumlich-zeitliche Kontrolle der nuklearen Translokation notwendig.
Zuvor wurde Licht ausgelöst Translokation von Proteinen in den Zellkern mit Käfig NLS7,8,9erreicht. NLS wandert in den Zellkern durch die Bindung an zytoplasmatischen Transport Proteine6. In den gemeldeten Methoden sind Gast Proteine mit Käfig NLS direkt in das Zytoplasma durch Mikroinjektion8 integriert oder in den Zielzellen mit einem genetischen Code Ausbau Technik9ausgedrückt. Daher ist eine Methode, die zelluläre Aufnahme und Foto-induzierte nuklearen Translokation erreichen kann vorteilhaft für praktische Anwendungen.
Hier beschreiben wir Licht ausgelöste nukleare Translokation der Gäste in lebenden Zellen mit dendritischen eingesperrte molekularen Klebstoff (CagedKleber-R, Abb. 1) Tags. Wasserlösliche Leime molekulare10,11,12,13,14,15,16,17,18 , 19 , 20 , 21 , 22 , 23 mit mehreren Gu+ -Anhänger sind bereits entwickelt worden, die halten uns fest an Proteine11,12,13,14,15, 16,17, Nukleinsäuren18,19,20, Phospholipid-Membranen-21und Ton Nanosheets22,23 durch die Bildung von zahlreichen Salz Brücken zwischen ihren Gu+ -Anhänger und Oxyanionic Gruppen auf die Ziele. Die Gu+ -Anhänger CagedKleber-r sind durch einen anionischen Photocleavable Gruppe, Butyrat ersetzt Nitroveratryloxycarbonyl (BASVOC) geschützt. Gäste, die mit dem Stichwort CagedKleber-R sind berücksichtigt lebenden Zellen über Endozytose und Aufenthalt in Endosomen (Abbildung 2). Auf Photoirradiation sind die BASVOC Gruppen von CagedKleber-R freistehend ein uncaged molekularen Klebstoff (UncagedKleber-R) tragen mehrere Gu+ Anhänger nachzugeben, dann die Migration von tagged Gast erleichtert in das Zytoplasma, gefolgt von den Zellkern (Abbildung 2). Caged-Kleber-R-Tag kann durch die Einwirkung von UV- oder zwei-Photon Nah-Infrarot (NIR) Licht ohne schwere Phototoxizität uncaged. Wir demonstrieren die raumzeitlich kontrollierte nukleare Lieferung von makromolekularen Gäste sowie kleine Molekül Gäste mit CagedKleber-R Tags Quantenpunkte (QDs) mit einem Fluoreszenzfarbstoff (Nitrobenzoxadiazole; NBD), bzw. als Beispiele.
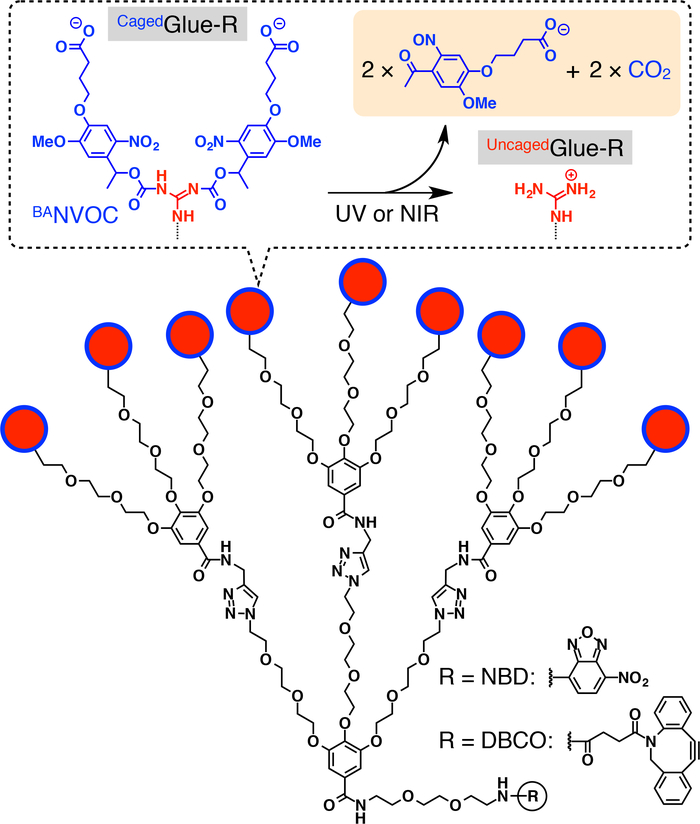
Abbildung 1: Schematische Strukturen der CagedKleber-R. Die 9 Guanidinium Ion (Gu+) Anhänger CagedKleber-r sind von einer Gruppe von Butyrat ersetzt Nitroveratryloxycarbonyl (BASVOC) geschützt. Die BA-SVOC-Gruppen sind durch Bestrahlung mit UV- oder zwei-Photon NIR Licht gespalten. Die fokale Kern CagedKleber-R ist mit Nitrobenzoxadiazole (NBD) oder Dibenzocylooctyne (DBCO) funktionalisiert. Nachdruck mit freundlicher Genehmigung von Referenz-20. Bitte klicken Sie hier für eine größere Version dieser Figur.
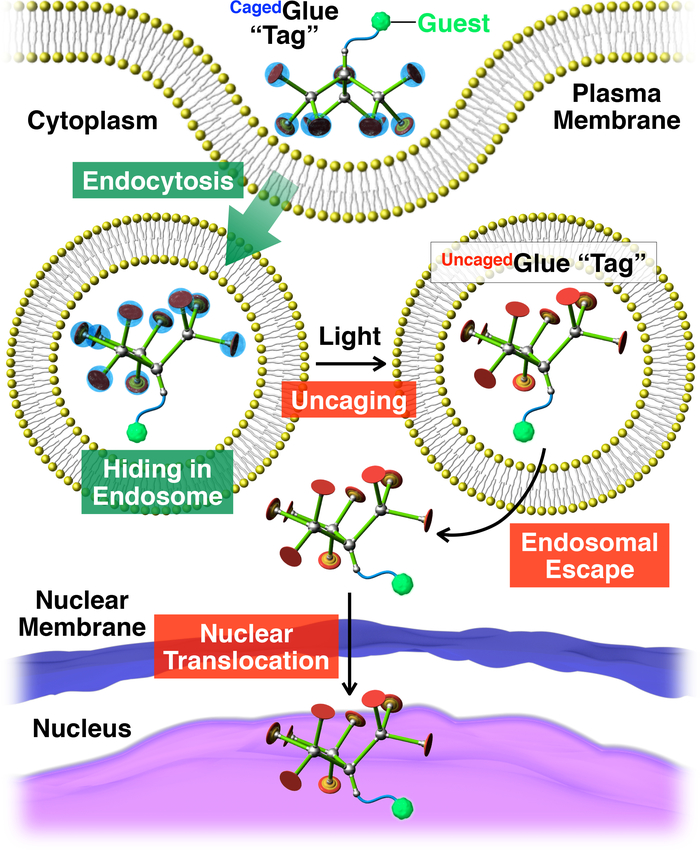
Abbildung 2: Schematische Darstellung der Licht-ausgelösten nuklearen Translokation Gäste konjugiert mit einem CagedKleber-R Tag. Der Gast /CagedKleber-R Konjugat berücksichtigt lebenden Zellen über Endozytose. Auf Photoirradiation ist das CagedKleber-R Tag uncaged die Bezeichnung für einen Uncaged-Kleber-R, weichen, der Endosomal Entweichen von der tagged Gast erleichtern kann. Anschließend wandert der tagged Gast in den Zellkern. Nachdruck mit freundlicher Genehmigung von Referenz-20. Bitte klicken Sie hier für eine größere Version dieser Figur.
Protokoll
1. Vorbereitung der Gäste mit KäfigKleber-R Stichwörter
- CagedKleber-NBD Lösung ansetzen.
- CagedKleber-NBD (Abbildung 1) nach dem Verfahren beschriebenen20zu synthetisieren.
- Bereiten Sie eine Stammlösung CagedKleber-NBD (10 mM) in trockenen Dimethyl Sulfoxid (DMSO).
Hinweis: Bewahren Sie die Stammlösung in Dunkelheit. Die Lösung kann mit wässrigen Puffer oder Zellkulturmedien nach Gebrauch verdünnt werden.
- CagedKleber-QD Lösung ansetzen.
- Synthetisieren CagedKleber-Dibenzocylooctyne (CagedKleber-DBCO) (Abbildung 1) nach den Verfahren zuvor20beschrieben.
- Bereiten Sie eine Stammlösung CagedKleber-DBCO (10 mM) in trockenen DMSO.
- Für die Vorbereitung von CagedKleber-QD zuerst vorbereiten-Azid funktionalisiert QDs (Azid-QD; ( Abbildung 3). 400 µL einer DMF 100 µL einer Dimethyl Formamid (DMF, 125 µM) Lösung von Azid-PEG4-NHS Ester (Abbildung 3) hinzufügen (500 nM) Lösung von Quantenpunkten (QDs) beschichtet mit Amin funktionalisiert PEG (Amin-QD; ( Abbildung 3). Rühren Sie die Mischung für 1 h bei Raumtemperatur.
- Die resultierende Lösung für 24 h gegen 800 mL DMF 3.500 Molekulargewicht Cut-Off (MWCO) eine regenerierte Zellulose-Membran mit Dialyse.
- CagedKleber-DBCO bis 50 µM mit DMF-Stammlösung zu verdünnen. Die Post-Dialyse-Lösung (Abbildung 3) 200 µL der Lösung hinzu und rühren Sie die Mischung für 3 h bei Raumtemperatur.
- Die resultierende Lösung für 24 h gegen 800 mL DMF eine regenerierte Zellulose-Membran (25.000 MWCO) mit Dialyse.
- Verdünnen Sie die resultierende Lösung bis 200 nM mit DMF.
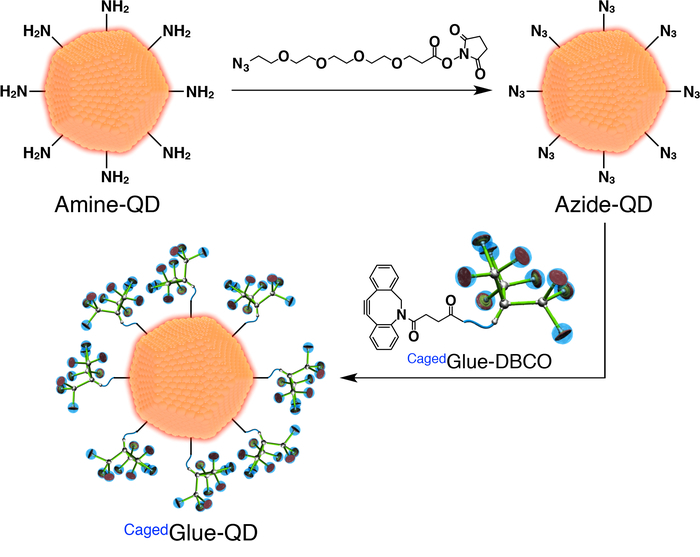
Abbildung 3: Schematische Darstellung der Zubereitung von CagedKleber-QD. Bitte klicken Sie hier für eine größere Version dieser Figur.
2. Vorbereitung des Hep3B Zellprobe für mikroskopische Beobachtungen
- Halten menschliche hepatozellulären Karzinom Hep3B Zellen in Adlers minimal wesentliche Medium (EMEM) mit 10 % fetalen bovine Serum (FBS) bei 37 ° C weniger als 5 % CO2.
- Samen der Zellen am Vortag das Experiment. 5.0 × 103 Hep3B Samenzellen pro Bohrloch eine 8 Kammern Glassubstrat in EMEM (10 % FBS, 200 µL), und inkubieren Sie die Zellprobe bei 37 ° C weniger als 5 % CO2 für 24 h.
- Entfernen Sie das Kulturmedium zu und spülen Sie die Zellprobe mit 100 µL Dulbeccos Phosphat-Puffer Kochsalzlösung (D-PBS) zweimal.
3. Beobachtung der nuklearen Translokation von Small-Molecule Gäste ausgelöst durch UV-Licht
- Liefern die Zellprobe (vorbereitet in Schritt 2.3) mit 200 µL der FBS-freie EMEM mit CagedKleber-NBD (10 µM) und inkubieren Sie die daraus resultierenden Zellprobe bei 37 ° C weniger als 5 % CO2 für 3 h.
Hinweis: Die Zellprobe in FBS-freie EMEM länger als 4 h Inkubation verursacht schwere Zellschäden. - Entfernen Sie das Kulturmedium zu und spülen Sie die Zellprobe mit 100 µL D-PBS zweimal.
- Für die Visualisierung von den Endosomen, liefern die Zellprobe mit 200 µL EMEM (10 % FBS), einen rot-fluoreszierenden Farbstoff enthält (z. B.LysoTracker rot, 100 nM), und die daraus resultierende Zellprobe bei 37 ° C inkubieren unter 5 % CO2 für 20 min. entfernen die Kultur Medium und spülen die Zellprobe mit 100 µL der D-PBS zweimal. Liefern die Zellprobe mit 200 µL EMEM (10 % FBS).
- Setzen Sie für nukleare Translokation von CagedKleber-NBD die Zellprobe mit UV-Licht für 2 min. über eine optische Faser mit einer 100-W Xenon-Lichtquelle mit einem 365 nm Bandpass-Filter ausgestattet. Halten Sie für eine Referenzprobe Zelle ohne UV-Exposition die Zellprobe in Dunkelheit.
Hinweis: Der Deckel der Glassubstrate kann für effiziente UV-Exposition abgenommen werden. Langjähriger Exposition gegenüber UV-Licht kann dazu führen, dass Zytotoxizität zu den Zellen. - Für die Visualisierung der Kerne, dem Kulturmedium 1 µL Hoechst 33342 (1 mg/mL) hinzu, und inkubieren Sie die daraus resultierenden Zellprobe bei 37 ° C weniger als 5 % CO2 10 min. lang.
- Unterliegt die Zellprobe konfokale Laser-scanning-Mikroskopie und erfassen die Mikrographen auf Anregung bei 488 nm (λObs = 500-530 nm), 543 nm (λObs = 565-620 nm), und 710 nm (zwei-Photon; Λ OBS = 390-465 nm) für NBD, rot fluoreszierenden Farbstoff und Hoechst 33342, beziehungsweise.
4. Beobachtung des nuklearen Translokation von Small-Molecule Gäste durch zwei-Photonen-NIR Licht ausgelöst
- Liefern die Zellprobe (vorbereitet in Schritt 2.3) mit 200 µL der FBS-freie EMEM mit CagedKleber-NBD (10 µM) und inkubieren Sie die daraus resultierenden Zellprobe bei 37 ° C weniger als 5 % CO2 für 3 h.
- Entfernen Sie das Kulturmedium zu und spülen Sie die Zellprobe mit 100 µL D-PBS zweimal. Liefern die Zellprobe mit 200 µL EMEM (10 % FBS).
- Unterliegt die Zellprobe konfokale Laser-scanning-Mikroskopie und erfassen die Mikrographen auf Anregung bei 488 nm (λObs = 500-530 nm).
- Für nukleare Translokation von CagedKleber-NBD, die Region, einschließlich der Zelle von Interesse mit einem zwei-Photon Erregung Laser zu bestrahlen (710 nm), als eine Lichtquelle in das Mikroskop für 2 min (30 s × 4) installiert. Beobachten Sie die Translokation, wie unter Punkt 4.3 beschrieben.
5. Beobachtung der nuklearen Translokation der makromolekularen Gäste ausgelöst durch UV-Licht
- Liefern die Zellprobe (vorbereitet in Schritt 2.3) mit 200 µL der FBS-freie EMEM mit CagedKleber-QD (10 nM), und die daraus resultierende Zellprobe bei 37 ° C weniger als 5 % inkubieren CO2 für 3 h.
- Entfernen Sie das Kulturmedium zu und spülen Sie die Zellprobe mit 100 µL D-PBS zweimal. Liefern die Zellprobe mit 200 µL EMEM (10 % FBS).
- Setzen Sie für nukleare Translokation von CagedKleber-QD die Zellprobe mit UV-Licht für 2 min. über eine optische Faser mit einer 100-W Xenon-Lichtquelle mit einem 365 nm Bandpass-Filter ausgestattet. Halten Sie für eine Referenzprobe Zelle ohne UV-Exposition die Zellprobe in Dunkelheit.
- Für die Visualisierung der Kerne, dem Kulturmedium 1 µL Hoechst 33342 (1 mg/mL) hinzu, und inkubieren Sie die daraus resultierenden Zellprobe bei 37 ° C weniger als 5 % CO2 10 min. lang.
- Unterliegt die Zellprobe konfokale Laser-scanning-Mikroskopie und Aufzeichnen der Mikrographen auf Anregung bei 405 nm (λObs = 430-520 nm) und 488 nm (λObs = 625-680 nm) für Hoechst 33342 und QDs, beziehungsweise.
6. Zelle Lebensfähigkeit Assay
- Menschlichen hepatozellulären Karzinom Hep3B Zellen in EMEM pflegen (10 % FBS) bei 37 ° C weniger als 5 % CO2.
- Samen der Zellen am Vortag das Experiment. 5.0 × 103 Hep3B Samenzellen pro Bohrloch einer 96-Well-Kultur-Platte in EMEM (10 % FBS, 200 µL), und inkubieren Sie die Zellprobe bei 37 ° C weniger als 5 % CO2 für 24 h.
- Entfernen Sie das Kulturmedium zu und spülen Sie die Zellprobe mit 100 µL D-PBS zweimal.
- Liefern die Zellprobe mit 200 µL der FBS-freie EMEM mit CagedKleber-NBD (0,1-100 µM) und inkubieren Sie die daraus resultierenden Zellprobe bei 37 ° C weniger als 5 % CO2 für 3 h.
- Setzen Sie die Zellprobe mit UV-Licht für 2 min über eine optische Faser mit einer 100-W Xenon-Lichtquelle mit einem 365 nm Bandpass-Filter ausgestattet. Halten Sie für eine analoge Zellprobe ohne UV-Exposition die Zellprobe in Dunkelheit.
- Das Kulturmedium 10 µL des Reagenz Zelle zählen Kit-8 (10 µL) hinzu und inkubieren Sie die daraus resultierenden Zellprobe bei 37 ° C weniger als 5 % CO2 für 2 h.
- Unterliegen die Zellprobe Absorption Spektroskopie (λ = 450 nm) mit einer Mikrotestplatte Reader.
Ergebnisse
Vor Photoirradiation, Hep3B Zellen inkubiert mit CagedKleber-NBD ausgestellt punctata Fluoreszenzemission von ihrem inneren (λExt = 488 nm; Abbildungen 4A und 4 C, grün). Eine analoge Schliffbild erhielt nach Anregung bei 543 nm für rot fluoreszierenden Farbstoff (Zahlen 4 b und 4 C, rot), darauf hinweist, dass CagedKleber-NBD in den Endosomen lokalisiert. Dem...
Diskussion
Vorausgegangenen Untersuchungen Licht ausgelöst Translokation von Proteinen in den Zellkern sind mit Käfig NLS7,8,9erreicht worden. Wie bereits erwähnt, erfordern diese Methoden zusätzliche Techniken, die NLS-markierten Proteine in das Zytoplasma zu integrieren. Im Gegensatz dazu ermöglicht unser Caged-Kleber-R-Tag nicht nur Foto-induzierte nuklearen Translokation, sondern auch zelluläre Aufnahme der Gäste. Die...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir anerkennen das Zentrum für NanoBio Integration, der University of Tokyo. Diese Arbeit wurde unterstützt durch Beihilfe für junge Wissenschaftler (B) (26810046), K.O. und teilweise unterstützt von Beihilfe für speziell gefördert Forschung (25000005), t.a. r.m. Dank der Research Fellowships der Japan Society für die Förderung der Wissenschaft (JSPS ) für junge Wissenschaftler und das Programm für die führenden Graduate Schools (GPLLI).
Materialien
| Name | Company | Catalog Number | Comments |
| Azide-PEG4-NHS ester | Click Chemistry Tools | AZ103 | |
| Q-dot 655 ITK | Invitrogen | Q21521MP | |
| Regenerated cellulose membrane (MWCO 3,500) | NIPPON Genetics | TOR-3K | |
| Regenerated cellulose membrane (MWCO 25,000) | Harvard Apparatus | 7425-RC25K | |
| Hep3B Cells | ATCC | HB-8064 | |
| 8-chambered glass substrate | Nunc | 155411JP | |
| 96-well culture plate | Nunc | 167008 | |
| Eagle's minimal essential medium (EMEM) | Thermo Fisher Scientific | 10370-021 | |
| Fetal bovine serum (FBS) | GE Healthcare | SH30406.02 | |
| Dulbecco's phosphate buffer saline (D-PBS) | Wako Pure Chemical Industries | 045-29795 | |
| LysoTracker Red | Lonza Walkersville | PA-3015 | |
| Hoechst 33342 | Dojindo | H342 | |
| Cell Counting Kit-8 | Dojindo | CK04 | |
| Confocal laser scanning microscope | Carl-Zeiss | LSM 510 | Equipped with two-photon excitation laser (Mai Tai laser, Spectra-Physics) |
| Confocal laser scanning microscope | Leica | TCS SP8 | |
| Xenon light source | Asahi Spectra | LAX-102 | |
| Microplate reader | Molecular Devices | SpectraMax Paradigm |
Referenzen
- Miller, A. D. Human gene therapy comes of age. Nature. 357, 455-460 (1992).
- Roth, J. A., Cristiano, R. J. Gene Therapy for Cancer: What Have We Done and Where Are We Going. Journal of the National Cancer Institute. 89, 21-39 (1997).
- Verma, I. M., Weitzman, M. D. GENE THERAPY: Twenty-First Century Medicine. Annual Review of Biochemistry. 74, 711-738 (2005).
- Ragin, A. D., Morgan, R. A., Chmielewski, J. Cellular Import Mediated by Nuclear Localization Signal Peptide Sequences. Chemistry & Biology. 9, 943-948 (2002).
- Martin, R. M., et al. Principles of protein targeting to the nucleolus. Nucleus. 6, 314-325 (2015).
- Sun, Y., et al. Factors influencing the nuclear targeting ability of nuclear localization signals. Journal of Drug Targeting. 24, 927-933 (2016).
- Ventura, B. D., Kuhlman, B. Go in! Go out! Inducible control of nuclear localization. Current Opinion in Chemical Biology. 34, 62-71 (2016).
- Watai, Y., Sase, I., Shiono, H., Nakano, Y. Regulation of nuclear import by light-induced activation of caged nuclear localization signal in living cells. FEBS Letters. 488, 39-44 (2001).
- Engelke, H., Chou, C., Uprety, R., Jess, P., Deiters, A. Control of Protein Function through Optochemical Translocation. ACS Synthetic Biology. 3, 731-736 (2014).
- Mogaki, R., Hashim, P. K., Okuro, K., Aida, T. Guanidinium-based "molecular glues" for modulation of biomolecular functions. Chemical Society Reviews. 46, 6480-6491 (2017).
- Okuro, K., Kinbara, K., Tsumoto, K., Ishii, N., Aida, T. Molecular Glues Carrying Multiple Guanidinium Ion Pendants via an Oligoether Spacer: Stabilization of Microtubules against Depolymerization. Journal of the American Chemical Society. 131, 1626-1627 (2009).
- Okuro, K., et al. Adhesion Effects of a Guanidinium Ion Appended Dendritic "Molecular Glue" on the ATP-Driven Sliding Motion of Actomyosin. Angewandte Chemie, International Edition. 48, 3030-3033 (2010).
- Uchida, N., et al. Photoclickable Dendritic Molecular Glue: Noncovalent-to-Covalent Photochemical Transformation of Protein Hybrids. Journal of the American Chemical Society. 135, 4684-4687 (2013).
- Garzoni, M., Okuro, K., Ishii, N., Aida, T., Pavan, G. M. Structure and Shape Effects of Molecular Glue on Supramolecular Tubulin Assemblies. ACS Nano. 8, 904-914 (2014).
- Mogaki, R., Okuro, K., Aida, T. Molecular glues for manipulating enzymes: trypsin inhibition by benzamidine-conjugated molecular glues. Chemical Science. 6, 2802-2805 (2015).
- Okuro, K., Sasaki, M., Aida, T. Boronic Acid-Appended Molecular Glues for ATP-Responsive Activity Modulation of Enzymes. Journal of the American Chemical Society. 138, 5527-5530 (2016).
- Mogaki, R., Okuro, K., Aida, T. Adhesive Photoswitch: Selective Photochemical Modulation of Enzymes under Physiological Conditions. Journal of the American Chemical Society. 139, 10072-10078 (2017).
- Hashim, P. K., Okuro, K., Sasaki, S., Hoashi, Y., Aida, T. Reductively Cleavable Nanocaplets for siRNA Delivery by Template-Assisted Oxidative Polymerization. Journal of the American Chemical Society. 137, 15608-15611 (2015).
- Hatano, J., Okuro, K., Aida, T. Photoinduced Bioorthogonal 1,3-Dipolar Poly-cycloaddition Promoted by Oxyanionic Substrates for Spatiotemporal Operation of Molecular Glues. Angewandte Chemie, International Edition. 55, 193-198 (2016).
- Arisaka, A., Mogaki, R., Okuro, K., Aida, T. Caged Molecular Glues as Photoactivatable Tags for Nuclear Translocation of Guests in Living Cells. Journal of the American Chemical Society. 140, 2687-2692 (2018).
- Suzuki, Y., Okuro, K., Takeuchi, T., Aida, T. Friction-Mediated Dynamic Disordering of Phospholipid Membrane by Mechanical Motions of Photoresponsive Molecular Glue: Activation of Ion Permeation. Journal of the American Chemical Society. 134, 15273-15276 (2012).
- Wang, Q., et al. High-water-content mouldable hydrogels by mixing clay and a dendritic molecular binder. Nature. 463, 339-343 (2010).
- Tamesue, S., et al. Linear versus Dendritic Molecular Binders for Hydrogel Network Formation with Clay Nanosheets: Studies with ABA Triblock Copolyethers Carrying Guanidinium Ion Pendants. Journal of the American Chemical Society. 135, 15650-15655 (2013).
- Mohr, D., Frey, S., Fischer, T., Güttler, T., Görlich, D. Characterisation of the passive permeability barrier of nuclear pore complexes. EMBO Journal. 28, 2541-2553 (2009).
- Best, M. D. Click Chemistry and Bioorthogonal Reactions: Unprecedented Selectivity in the Labeling of Biological Molecules. Biochemistry. 48, 6571-6584 (2009).
- Klán, P., et al. Photoremovable Protecting Groups in Chemistry and Biology: Reaction Mechanisms and Efficacy. Chemical Reviews. 113, 119-191 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten