É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Spatiotemporally controlado translocação Nuclear de convidados em células vivas, usando cola Molecular enjaulada como Photoactivatable Tags
Neste Artigo
Resumo
Este protocolo descreve luz-acionou translocação nuclear de convidados em células vivas, usando tags cola molecular enjaulado. Este método é promissor para a entrega de drogas de nuclear-direcionamento local-seletivo.
Resumo
O núcleo da célula é dentre as organelas mais importantes como um destino de entrega da droga subcellular, desde modulação de replicação de gene e expressão é eficaz no tratamento de várias doenças. Aqui, demonstramos a luz-acionou a translocação nuclear de convidados usando enjaulado tags cola molecular (Cagedcola-R), cujas múltiplas guanidínio pingentes de íon (Gu.+) são protegidos por um grupo de photocleavable aniônicos (butirato-substituídos nitroveratryloxycarbonyl; BA NVOC). Os hóspedes com cola de Caged-R são absorvidos vivos células através de endocitose e permanecem em endossomos. No entanto, após photoirradiation, Cagedcola-R é convertido em uncaged molecular cola (cola-RUncaged) carregando vários pingentes de Gu+ , que facilita a fuga de CDDP e translocação nuclear subsequente dos convidados. Este método é promissor para entrega da droga local-seletiva nuclear multiplataforma, desde que os convidados etiquetados podem migrar para o citoplasma, seguido pelo núcleo da célula apenas quando photoirradiated. Enjaulado Etiquetas de colagem-R podem entregar macromoleculares convidados como pontos quânticos (QDs) bem como convidados da pequeno-molécula. Enjaulado Etiquetas de colagem-R podem ser uncaged com luz UV, não só mas também dois fotões infravermelho próximo (NIR) a luz, que pode penetrar profundamente no tecido.
Introdução
O núcleo da célula, que carrega a informação genética, dentre as mais importantes organelas é como um destino de entrega da droga subcellular, desde modulação de replicação de gene e expressão é eficaz no tratamento de várias doenças, incluindo câncer e genética 1,2,3de distúrbios. Para nuclear entrega de drogas, a conjugação de peptídeo etiquetas tais como um sinal de localização nuclear a4,5,6 tem sido amplamente pesquisada. No entanto, a fim de reduzir os efeitos colaterais indesejados, controle spatiotemporal de translocação nuclear é necessário.
Anteriormente, acionada por luz translocação de proteínas para o núcleo celular foi conseguida usando enjaulado NLS7,8,9. NLS migra para o núcleo da célula por ligação a proteínas de transporte citoplasmáticos6. Nos métodos relatados, proteínas comentários enjaulado NLS do rolamento são diretamente incorporadas no citoplasma por microinjeção8 ou expressos nas células de destino usando um código genético expansão técnica9. Portanto, um método que pode alcançar tanto celular absorção e translocação nuclear induzida por foto é vantajoso para aplicações práticas.
Aqui, descrevemos a luz-acionou a translocação nuclear dos convidados em células vivas, usando tags dendríticas cola molecular enjaulado (Cagedcola-R, Figura 1). Water-soluble colas molecular10,11,12,13,14,15,16,17,18 , 19 , 20 , 21 , 22 , 23 tendo vários pingentes de Gu+ têm sido desenvolvidos anteriormente, que aderem firmemente a proteínas11,12,13,14,15, 16,17, ácidos nucleicos18,19,20, de membranas fosfolipídicas21e argila nanosheets22,23 , através da formação de múltiplas pontes de sal entre seus pingentes Gu+ e oxyanionic grupos dos alvos. O pingentes de Gu+ de Cagedcola-R é protegido por um grupo de photocleavable aniônicos, nitroveratryloxycarbonyl butirato-substituídos (BANVOC). Os hóspedes com cola de Caged-R são absorvidos vivos células através de endocitose e estadia em endossomos (Figura 2). Sobre photoirradiation, os grupos NVOC BAde Cagedcola-R são desanexados para render uma uncaged molecular cola (cola deUncaged-R) carregando vários pingentes de Gu+ , que então facilita a migração do convidado etiquetado para o citoplasma, seguido-se o núcleo da célula (Figura 2). A marca de cola-R Cagedpode ser uncaged pela exposição a UV ou luz de dois fotões infravermelha (NIR) sem grave fototoxicidade. Demonstramos a entrega nuclear controlada spatiotemporally de macromoleculares convidados, bem como convidados da pequeno-molécula com etiquetas de colagem-R Caged, usando pontos quânticos (QDs) e uma tintura fluorescente (nitrobenzoxadiazole; NBD), respectivamente, como exemplos.
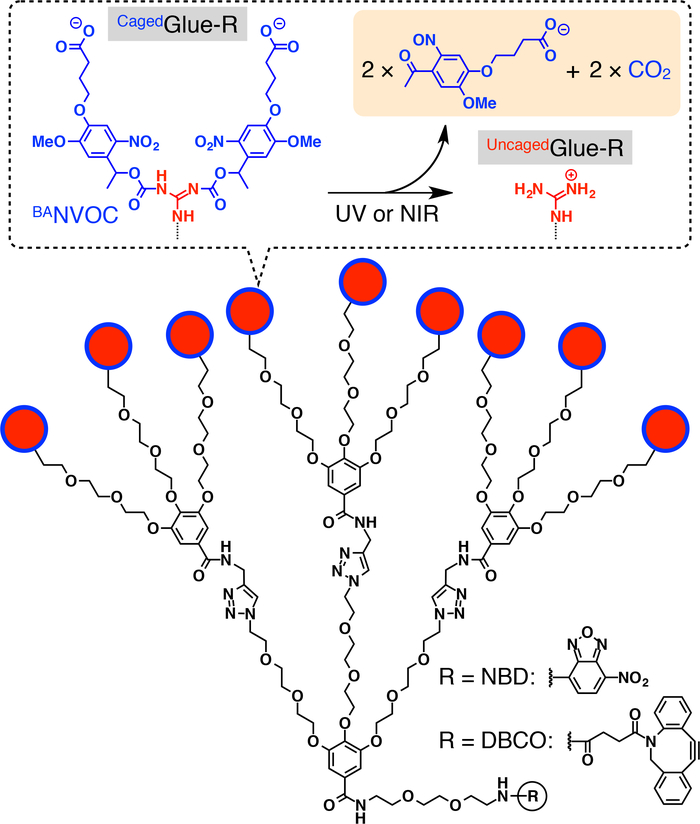
Figura 1: Estruturas esquemáticas do Cagedcola-R. Os 9 pendentes de íon (Gu.+) ultracentrifugation do Cagedcola-R são protegidos por um grupo de nitroveratryloxycarbonyl butirato-substituídos (BANVOC). Os grupos NVOC BAsão clivados por irradiação com UV ou luz NIR dois fotões. O núcleo focal do Cagedcola-R é acrescido com nitrobenzoxadiazole (NBD) ou dibenzocylooctyne (DBCO). Reproduzido com permissão da referência20. Clique aqui para ver uma versão maior desta figura.
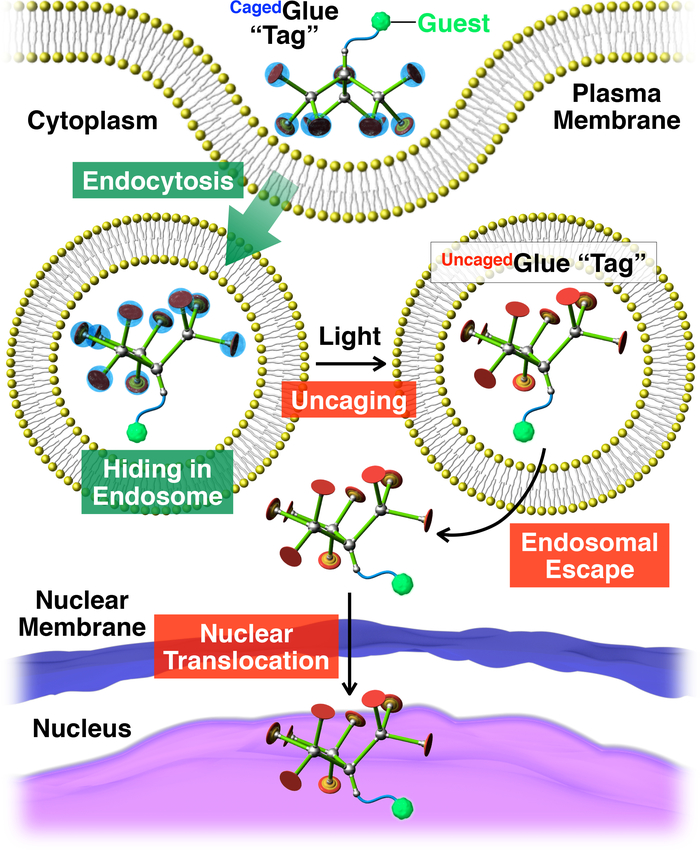
Figura 2: Ilustração esquemática da luz-acionou a translocação nuclear de convidados conjugados com uma marca de cola-R Caged. O convidado / conjugado de colagem-RCagedé retomado em vida células através de endocitose. Após a photoirradiation, a marca de cola-R Cagedé uncaged para produzir uma marca de cola-R Uncaged, que pode facilitar a fuga de CDDP do convidado marcado. Posteriormente, os comentários marcados migra para o núcleo da célula. Reproduzido com permissão da referência20. Clique aqui para ver uma versão maior desta figura.
Protocolo
1. preparação dos convidados com colagem-R enjauladoTags
- Prepare a solução de cola-NBD Caged.
- Sintetiza Cagedcola-NBD (Figura 1) seguindo os procedimentos descritos anteriormente20.
- Prepare uma solução stock de Cagedcola-NBD (10 mM) em seco dimetilsulfóxido (DMSO).
Nota: Guarde a solução-mãe no escuro. A solução pode ser diluída com buffers aquosas ou meios de cultura celular em cima de uso.
- Prepare a solução de cola-QD Caged.
- Sintetizar Cagedcola-dibenzocylooctyne (Cagedcola-DBCO) (Figura 1) seguindo os procedimentos anteriormente descritos20.
- Prepare uma solução stock de Cagedcola-DBCO (10 mM) em DMSO seco.
- Para a preparação do Cagedcola-QD, primeiro preparar acrescida de azida QDs (QD-azida; A Figura 3). Adicionar 100 µ l de solução de azida-PEG4-NHS éster (Figura 3) dimetil formamida (DMF, 125 µM) a 400 µ l de uma DMF (500 nM) solução de pontos quânticos (QDs) revestida com PEG amina acrescida (amina-QD; A Figura 3). Agite a mistura por 1h à temperatura ambiente.
- Dialize a solução resultante para 24h contra 800 mL de DMF usando uma membrana de celulose regenerada com 3.500 de peso molecular de corte (MWCO).
- Dilua a solução-mãe de Cagedcola-DBCO de 50 µM com DMF. Adicionar 200 µ l da solução para a solução de pós-diálise (Figura 3) e agitar a mistura por 3 h à temperatura ambiente.
- Dialize a solução resultante para 24h contra 800 mL de DMF usando uma membrana de celulose regenerada (25.000 MWCO).
- Diluir a solução resultante a 200 nM com DMF.
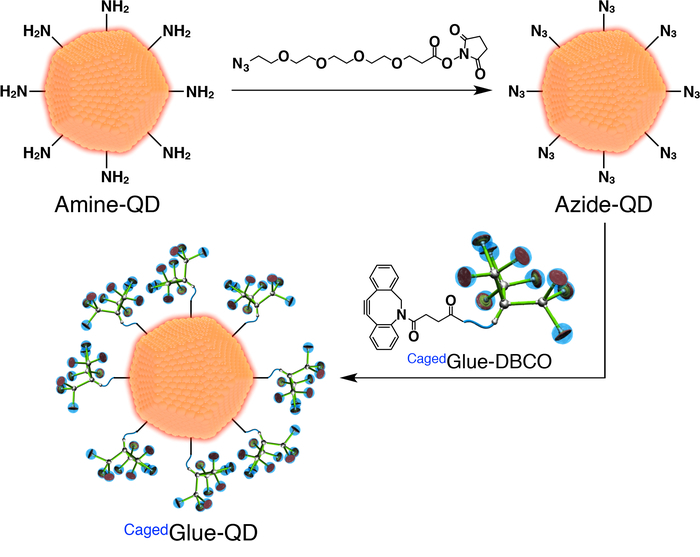
Figura 3: ilustração esquemática da preparação do Cagedcola-QD. Clique aqui para ver uma versão maior desta figura.
2. preparação da amostra de células Hep3B para observações microscópicas
- Manter as células de carcinoma Hep3B hepatocelular humano em essencial meio mínimo do águia (EMEM) contendo 10% de soro fetal bovino (FBS) a 37 ° C, menos de 5% de CO2.
- As células no dia anterior o experimento de semente. Células de3 Hep3B semente 5,0 × 10 por bem de um substrato de vidro 8-septadas no EMEM (10% FBS, 200 µ l) e incubar a amostra de célula a 37 ° C abaixo de 5% de CO2 por 24 h.
- Remover o meio de cultura e enxagúe a amostra de células com 100 µ l de soro de tampão de fosfato de Dulbecco (D-PBS) duas vezes.
3. observação da translocação Nuclear da pequeno-molécula convidados desencadeada pela luz UV
- Fornecer a amostra de células (preparado no passo 2.3) com 200 µ l de EMEM FBS-livre contendo Cagedcola-NBD (10 µM) e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 3 h.
Nota: A incubação da amostra celular no EMEM FBS-livre por mais de 4 h provoca danos celulares graves. - Remover o meio de cultura e enxagúe a amostra de células com 100 µ l de PBS-D duas vezes.
- Para visualização dos endossomos, fornecer a amostra de células com 200 µ l de EMEM (10% FBS) contendo um corante vermelho-fluorescente (por exemplo, LysoTracker vermelho, 100 nM) e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 20 min. remover a cultura médio e enxaguar a amostra de células com 100 µ l de PBS-D duas vezes. Fornecer a amostra de células com 200 µ l de EMEM (10% FBS).
- Para a translocação nuclear do Cagedcola-NBD, expor a amostra de célula à luz por 2 min através de uma fibra óptica usando uma fonte de luz xenon de 100 W, equipada com um filtro de passa-banda 365 nm UV. Para uma amostra de células de referência sem exposição aos raios UV, manter a amostra de células escuras.
Nota: A tampa dos substratos de vidro pode ser retirada para eficiente exposição aos raios UV. Longa exposição à luz UV pode causar citotoxicidade para as células. - Para visualização dos núcleos, adicionar 1 µ l de Hoechst 33342 (1 mg/mL) para o meio de cultura e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 10 min.
- Submeter a amostra de células para a microscopia eletrônica de varredura a laser confocal e gravar as micrografias em cima de excitação em 488 nm (λobs = 500-530 nm), 543 nm (λobs = 565-620 nm) e 710 nm (dois fotões; Λ OBS = 390-465 nm) para NBD e corante vermelho-fluorescente Hoechst 33342, respectivamente.
4. observação da translocação Nuclear de hóspedes pequeno-molécula desencadeada por NIR dois fotões de luz
- Fornecer a amostra de células (preparado no passo 2.3) com 200 µ l de EMEM FBS-livre contendo Cagedcola-NBD (10 µM) e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 3 h.
- Remover o meio de cultura e enxagúe a amostra de células com 100 µ l de PBS-D duas vezes. Fornecer a amostra de células com 200 µ l de EMEM (10% FBS).
- Submeter a amostra de células para a microscopia eletrônica de varredura a laser confocal e gravar as micrografias em cima de excitação em 488 nm (λobs = 500-530 nm).
- Para a translocação nuclear do Cagedcola-NBD, irradiar a região, incluindo a célula de interesse com um laser de excitação de dois fotões (710 nm), instalado como uma fonte de luz no microscópio, por 2 min (30 s × 4). Observe a translocação, conforme descrito no passo 4.3.
5. observação da translocação Nuclear de convidados Macromolecular desencadeada pela luz UV
- Fornecer a amostra de células (preparado no passo 2.3) com 200 µ l de EMEM FBS-livre contendo Cagedcola-QD (10 nM) e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 3 h.
- Remover o meio de cultura e enxagúe a amostra de células com 100 µ l de PBS-D duas vezes. Fornecer a amostra de células com 200 µ l de EMEM (10% FBS).
- Para a translocação nuclear do Cagedcola-QD, expor a amostra de célula à luz por 2 min através de uma fibra óptica usando uma fonte de luz xenon de 100 W, equipada com um filtro de passa-banda 365 nm UV. Para uma amostra de células de referência sem exposição aos raios UV, manter a amostra de células escuras.
- Para visualização dos núcleos, adicionar 1 µ l de Hoechst 33342 (1 mg/mL) para o meio de cultura e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 10 min.
- Submeter a amostra de células para a microscopia eletrônica de varredura a laser confocal e gravar as micrografias sobre excitação em 405 nm (λobs = 430-520 nm) e 488 nm (λobs = 625-680 nm) para Hoechst 33342 e QDs, respectivamente.
6. ensaio da viabilidade celular
- Manter as células de carcinoma Hep3B hepatocelular humano no EMEM (10% FBS) a 37 ° C abaixo de 5% de CO2.
- As células no dia anterior o experimento de semente. Células de3 Hep3B semente 5,0 × 10 por bem de uma placa de 96 poços de cultura no EMEM (10% FBS, 200 µ l) e incubar a amostra de célula a 37 ° C abaixo de 5% de CO2 por 24 h.
- Remover o meio de cultura e enxagúe a amostra de células com 100 µ l de PBS-D duas vezes.
- Fornecer a amostra de células com 200 µ l de EMEM FBS-livre contendo Cagedcola-NBD (0,1-100 µM) e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 3 h.
- Expor a amostra de célula à luz UV para 2 min através de uma fibra óptica usando uma fonte de luz xenon de 100 W, equipada com um filtro de passa-banda 365 nm. Para uma amostra de células análogas sem exposição aos raios UV, manter a amostra de células escuras.
- Adicionar 10 µ l de reagente de célula contando Kit-8 (10 µ l) para o meio de cultura e incubar a amostra resultante de célula a 37 ° C abaixo de 5% de CO2 por 2 h.
- Submeter a amostra de célula a espectroscopia de absorção (λ = 450 nm) usando um leitor de microplacas.
Resultados
Antes de photoirradiation, Hep3B células incubadas com Cagedcola-NBD exibiram a emissão de fluorescência punctate de seu interior (λext = 488 nm; Figuras 4A e 4C, verde). Uma micrografia análoga foi obtida em cima de excitação em 543 nm para corante vermelho-fluorescente (figuras 4B e 4C, vermelho), indica que o Cagedcola-NBD localizada nos endossomos. Con...
Discussão
Inquéritos anteriores de luz-acionou translocação de proteínas para o núcleo da célula foram obtidos usando enjaulado NLS7,8,9. Como mencionado anteriormente, esses métodos exigem técnicas adicionais para incorporar as proteínas NLS-marcados para o citoplasma. Em contraste, a nossa marca de cola-R Cagedpermite não só a translocação nuclear induzida por foto, mas também a captação celular dos convidados...
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Reconhecemos o centro para a integração de NanoBio, da Universidade de Tóquio. Este trabalho foi apoiado por subsídio para jovens cientistas (B) (26810046) K.O. e parcialmente financiado por subsídio para pesquisa especialmente promovido (25000005) T.A. R.M. graças a bolsas de pesquisa de Japão sociedade para a promoção da ciência (JSPS ) para jovens cientistas e o programa para as principais escolas de pós-graduação (GPLLI).
Materiais
| Name | Company | Catalog Number | Comments |
| Azide-PEG4-NHS ester | Click Chemistry Tools | AZ103 | |
| Q-dot 655 ITK | Invitrogen | Q21521MP | |
| Regenerated cellulose membrane (MWCO 3,500) | NIPPON Genetics | TOR-3K | |
| Regenerated cellulose membrane (MWCO 25,000) | Harvard Apparatus | 7425-RC25K | |
| Hep3B Cells | ATCC | HB-8064 | |
| 8-chambered glass substrate | Nunc | 155411JP | |
| 96-well culture plate | Nunc | 167008 | |
| Eagle's minimal essential medium (EMEM) | Thermo Fisher Scientific | 10370-021 | |
| Fetal bovine serum (FBS) | GE Healthcare | SH30406.02 | |
| Dulbecco's phosphate buffer saline (D-PBS) | Wako Pure Chemical Industries | 045-29795 | |
| LysoTracker Red | Lonza Walkersville | PA-3015 | |
| Hoechst 33342 | Dojindo | H342 | |
| Cell Counting Kit-8 | Dojindo | CK04 | |
| Confocal laser scanning microscope | Carl-Zeiss | LSM 510 | Equipped with two-photon excitation laser (Mai Tai laser, Spectra-Physics) |
| Confocal laser scanning microscope | Leica | TCS SP8 | |
| Xenon light source | Asahi Spectra | LAX-102 | |
| Microplate reader | Molecular Devices | SpectraMax Paradigm |
Referências
- Miller, A. D. Human gene therapy comes of age. Nature. 357, 455-460 (1992).
- Roth, J. A., Cristiano, R. J. Gene Therapy for Cancer: What Have We Done and Where Are We Going. Journal of the National Cancer Institute. 89, 21-39 (1997).
- Verma, I. M., Weitzman, M. D. GENE THERAPY: Twenty-First Century Medicine. Annual Review of Biochemistry. 74, 711-738 (2005).
- Ragin, A. D., Morgan, R. A., Chmielewski, J. Cellular Import Mediated by Nuclear Localization Signal Peptide Sequences. Chemistry & Biology. 9, 943-948 (2002).
- Martin, R. M., et al. Principles of protein targeting to the nucleolus. Nucleus. 6, 314-325 (2015).
- Sun, Y., et al. Factors influencing the nuclear targeting ability of nuclear localization signals. Journal of Drug Targeting. 24, 927-933 (2016).
- Ventura, B. D., Kuhlman, B. Go in! Go out! Inducible control of nuclear localization. Current Opinion in Chemical Biology. 34, 62-71 (2016).
- Watai, Y., Sase, I., Shiono, H., Nakano, Y. Regulation of nuclear import by light-induced activation of caged nuclear localization signal in living cells. FEBS Letters. 488, 39-44 (2001).
- Engelke, H., Chou, C., Uprety, R., Jess, P., Deiters, A. Control of Protein Function through Optochemical Translocation. ACS Synthetic Biology. 3, 731-736 (2014).
- Mogaki, R., Hashim, P. K., Okuro, K., Aida, T. Guanidinium-based "molecular glues" for modulation of biomolecular functions. Chemical Society Reviews. 46, 6480-6491 (2017).
- Okuro, K., Kinbara, K., Tsumoto, K., Ishii, N., Aida, T. Molecular Glues Carrying Multiple Guanidinium Ion Pendants via an Oligoether Spacer: Stabilization of Microtubules against Depolymerization. Journal of the American Chemical Society. 131, 1626-1627 (2009).
- Okuro, K., et al. Adhesion Effects of a Guanidinium Ion Appended Dendritic "Molecular Glue" on the ATP-Driven Sliding Motion of Actomyosin. Angewandte Chemie, International Edition. 48, 3030-3033 (2010).
- Uchida, N., et al. Photoclickable Dendritic Molecular Glue: Noncovalent-to-Covalent Photochemical Transformation of Protein Hybrids. Journal of the American Chemical Society. 135, 4684-4687 (2013).
- Garzoni, M., Okuro, K., Ishii, N., Aida, T., Pavan, G. M. Structure and Shape Effects of Molecular Glue on Supramolecular Tubulin Assemblies. ACS Nano. 8, 904-914 (2014).
- Mogaki, R., Okuro, K., Aida, T. Molecular glues for manipulating enzymes: trypsin inhibition by benzamidine-conjugated molecular glues. Chemical Science. 6, 2802-2805 (2015).
- Okuro, K., Sasaki, M., Aida, T. Boronic Acid-Appended Molecular Glues for ATP-Responsive Activity Modulation of Enzymes. Journal of the American Chemical Society. 138, 5527-5530 (2016).
- Mogaki, R., Okuro, K., Aida, T. Adhesive Photoswitch: Selective Photochemical Modulation of Enzymes under Physiological Conditions. Journal of the American Chemical Society. 139, 10072-10078 (2017).
- Hashim, P. K., Okuro, K., Sasaki, S., Hoashi, Y., Aida, T. Reductively Cleavable Nanocaplets for siRNA Delivery by Template-Assisted Oxidative Polymerization. Journal of the American Chemical Society. 137, 15608-15611 (2015).
- Hatano, J., Okuro, K., Aida, T. Photoinduced Bioorthogonal 1,3-Dipolar Poly-cycloaddition Promoted by Oxyanionic Substrates for Spatiotemporal Operation of Molecular Glues. Angewandte Chemie, International Edition. 55, 193-198 (2016).
- Arisaka, A., Mogaki, R., Okuro, K., Aida, T. Caged Molecular Glues as Photoactivatable Tags for Nuclear Translocation of Guests in Living Cells. Journal of the American Chemical Society. 140, 2687-2692 (2018).
- Suzuki, Y., Okuro, K., Takeuchi, T., Aida, T. Friction-Mediated Dynamic Disordering of Phospholipid Membrane by Mechanical Motions of Photoresponsive Molecular Glue: Activation of Ion Permeation. Journal of the American Chemical Society. 134, 15273-15276 (2012).
- Wang, Q., et al. High-water-content mouldable hydrogels by mixing clay and a dendritic molecular binder. Nature. 463, 339-343 (2010).
- Tamesue, S., et al. Linear versus Dendritic Molecular Binders for Hydrogel Network Formation with Clay Nanosheets: Studies with ABA Triblock Copolyethers Carrying Guanidinium Ion Pendants. Journal of the American Chemical Society. 135, 15650-15655 (2013).
- Mohr, D., Frey, S., Fischer, T., Güttler, T., Görlich, D. Characterisation of the passive permeability barrier of nuclear pore complexes. EMBO Journal. 28, 2541-2553 (2009).
- Best, M. D. Click Chemistry and Bioorthogonal Reactions: Unprecedented Selectivity in the Labeling of Biological Molecules. Biochemistry. 48, 6571-6584 (2009).
- Klán, P., et al. Photoremovable Protecting Groups in Chemistry and Biology: Reaction Mechanisms and Efficacy. Chemical Reviews. 113, 119-191 (2013).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados