Method Article
Effiziente und konsistente Generierung von retinalen Pigmentepithel-/Aderhaut-Flatmounts aus dem menschlichen Auge für die histologische Analyse
In diesem Artikel
Zusammenfassung
Wir beschreiben eine Methode, um retinales Pigmentepithel (RPE) effizient von der Netzhaut im menschlichen Auge zu trennen und ganze RPE/Aderhaut-Flatmounts für histologische und morphometrische Analysen des RPE zu generieren.
Zusammenfassung
Das retinale Pigmentepithel (RPE) und die Netzhaut sind funktionell und strukturell verbundene Gewebe, die zusammenarbeiten, um die Lichtwahrnehmung und das Sehen zu regulieren. Proteine auf der apikalen RPE-Oberfläche sind eng mit Proteinen auf der Oberfläche des äußeren Segments des Photorezeptors verbunden, was es schwierig macht, das RPE konsequent von den Photorezeptoren / der Netzhaut zu trennen. Wir haben eine Methode entwickelt, um die Netzhaut effizient vom RPE des menschlichen Auges zu trennen, um vollständige RPE/Aderhaut- und Netzhaut-Flatmounts für die separate zelluläre Analyse der Photorezeptoren und RPE-Zellen zu erzeugen. Eine intravitreale Injektion einer hochosmolaren Lösung von D-Mannitol, einem Zucker, der nicht vom RPE transportiert wird, induzierte die Trennung von RPE und Netzhaut über die gesamte hintere Kammer, ohne die RPE-Zellübergänge zu schädigen. Es wurden keine RPE-Pflaster an der Netzhaut beobachtet. Die Phalloidin-Markierung von Aktin zeigte eine RPE-Formerhaltung und ermöglichte eine morphometrische Analyse des gesamten Epithels. Eine auf künstlicher Intelligenz (KI) basierende Software wurde entwickelt, um die RPE-Zellgrenzen genau zu erkennen und zu segmentieren und 30 verschiedene Formmetriken zu quantifizieren. Diese Präpariermethode ist sehr reproduzierbar und kann leicht auf andere Tiermodelle ausgedehnt werden.
Einleitung
Das retinale Pigmentepithel (RPE) und die neurale Netzhaut sind aufgrund der starken physiologischen Abhängigkeit der Photorezeptoren vom RPE stark miteinander verbunden. Während der Dissektion führt die mechanische Trennung der neuronalen Netzhaut von der RPE zum Zerreißen der RPE-Zellen, wobei die apikalen Teile des RPE an den äußeren Segmenten der retinalen Photorezeptoren gebunden bleiben. Das Ausmaß der RPE-Retina-Adhäsion ist so groß, dass die Menge an Pigment, die nach der Trennung auf der Netzhaut verbleibt, verwendet wird, um die Stärke der Netzhautadhäsion zu quantifizieren1. Insbesondere RPE-Tight Junctions und die Aktinstruktur, die sie verbindet, die sich auf der apikalen Seite befinden, brechen während der mechanischen Trennung ab. Daher führt das Färben von RPE-Flatmounts für Zellgrenzen zu einer fleckigen Monoschicht, in der viele Zellen fehlende Ränder aufweisen. Dieser Effekt wird verstärkt, wenn das Gewebe vor der Dissektion mit Paraformaldehyd (PFA) fixiert wird, da die Proteine vernetzt werden.
Studien zur intravitrealen Wirkstoffabgabe haben gezeigt, dass Injektionen von hyperosmotischen Lösungen in die Hinterkammer eine Netzhautablösung induzieren 2,3. In diesen Experimenten wurden 50 μL verschiedener Lösungen im Bereich von 1.000 mOsm bis 2.400 mOsm, die in den mittleren Glaskörper injiziert wurden, innerhalb von Minuten zu einer Netzhautablösung geführt. Bemerkenswert ist, dass die RPE-Tight Junctions selbst nach langen Expositionen bei Lösungen mit hoher Osmolarität in den transmissionselektronenmikroskopischen Bildern von Kaninchen- und Affenaugen intakt erschienen3. Einer ähnlichen Strategie folgend, injizierten wir eine hyperosmotische Lösung von D-Mannitol in den mittleren Glaskörper, um eine effiziente Netzhautablösung zu induzieren, bevor wir eine RPE-Dissektion durchführten. Da D-Mannitol nicht durch das RPE4 transportiert wird, wird eine hohe intravitreale Konzentration aufrechterhalten, wodurch ein osmotischer Gradient erzeugt wird. Die effiziente Trennung von RPE und Netzhaut über die gesamte hintere Kammer garantiert die Erhaltung der RPE-Zellverbindungen und ermöglicht die Untersuchung der RPE-Morphometrie auf dem gesamten Flatmount. Darüber hinaus haben wir eine auf künstlicher Intelligenz (KI) basierende Software entwickelt, die fluoreszenzmarkierte RPE-Zellgrenzen erkennt und segmentiert, 30 verschiedene Formmetriken quantifiziert und Heatmaps jeder Metrik für die Visualisierung 5,6 erstellt.
Protokoll
Die menschlichen Globen von Leichen wurden vom Advanced Sight Network (Birmingham, AL) bezogen. Arbeiten an Leichengewebe werden vom NIH Institutional Review Board vom Forschungsethikausschuss ausgenommen.
1. Augenglobus Versand
- Nach der Enukleation frische Augenkugeln in einem Behälter verschicken, der 1x mit eiskaltem DPBS mit Ca2+ undMg2+ gefüllt ist.
HINWEIS: Es ist besser, das Auge innerhalb von 24 Stunden nach der Enukleation zu analysieren. Die RPE-Morphologie wird in diesem Zeitfenster nicht verändert.
2. Silikonformvorbereitung
- Schneiden Sie die unteren 20 mm eines Rundbodenrohrs mit einem Durchmesser von 25 mm ab. Legen Sie es auf die Basis eines quadratischen Wiegebootes (81 mm x 81 mm x 25 mm).
- Mischen Sie die beiden Komponenten des Silikon-Elastomer-Kits im Verhältnis 10:1 und achten Sie darauf, keine Luft einzuschließen. Gießen Sie die Mischung in das Wiegeboot, das das kugelförmige Stück des Rundbodenrohrs enthält.
- Das Silikon über Nacht bei Raumtemperatur aushärten. Entfernen Sie das Wiegegefäß und das Rundrohr aus der ausgehärteten Silikonform.
3. RPE-Dissektion
- Reinigen Sie die Sklera von der frischen Augenkugel von Muskeln und Bindegewebe. Befestigen Sie das Auge mit 27 G-Nadeln, die durch die Sklera gefädelt werden, an der Silikonform. Füllen Sie den Hohlraum der Form mit DPBS 1x mit Ca2+ undMg2+.
HINWEIS: Achten Sie darauf, die Augenkammer nicht zu durchbrechen. Die Nadeln sollten nur durch die Sklera gehen. - Verwenden Sie eine 1-ml-Spritze und eine 21-g-Nadel, um ~400 μL 1.700 mOsm D-Mannitol-Lösung in den Glaskörper zu injizieren. Führen Sie die Nadel durch die Pars Plana ein, um eine Punktion der Vorderkammer des Auges zu vermeiden. Lassen Sie das Auge bei Raumtemperatur für ~45 min.
- Schneiden Sie die Vorderkammer auf Höhe der Pars plana mit einer feinen Schere und einer Pinzette auf. Füllen Sie die hintere Augenkammer mit DPBS 1x mit Ca2+ undMg2+. Unter dem Stereomikroskop lokalisieren Sie die Makula (den gelben Fleck auf der Netzhaut).
- Wenn ein chirurgischer Glaskörperschneider zur Verfügung steht, entfernen Sie den Glaskörper und ersetzen Sie ihn durch DPBS 1x mit Ca 2+ und Mg2+. Alternativ können Sie versuchen, den Glaskörper mit einer Pinzette anzuheben und mit einer feinen Schere zu schneiden.
- Achten Sie darauf, die Makularegion zu erhalten, schneiden Sie das Auge in Quadranten: nasal, temporal, superior und inferior. Entfernen Sie die Nadeln, wenn sie im Weg sind.
- Überführen Sie die schmetterlingshintere Augenkammer in eine 100 mm Petrischale, die DPBS 1x mit Ca2+ und Mg2+ enthält. Bevor Sie die Netzhaut entfernen, markieren Sie das Blütenblatt, das die Makula enthält, indem Sie einen kleinen Schnitt (V-förmig) im Ziliarand machen. Heben und schneiden Sie den gesamten Glaskörper, der auf der Netzhaut liegt.
- Heben Sie die Netzhaut vorsichtig von mehreren Seiten mit einem gekrümmten Spatel oder einer Pinzette an, um zu überprüfen, ob sich die Netzhaut vom RPE gelöst hat, und um etwas Flüssigkeit zwischen den beiden Schichten zirkulieren zu lassen.
HINWEIS: Die Netzhaut wird immer noch an der äußersten Peripherie (Ziliarrand) und am Sehnerv befestigt. - Schneiden Sie die Netzhaut von den Ziliarändern in allen Blütenblättern ab und stellen Sie sicher, dass das RPE nicht zerkratzt wird. Legen Sie das Gewebe in 4% PFA und inkubieren Sie für ~ 1 h. 3x mit DPBS waschen, 1x mit Ca2+ undMg2+. Das Gewebe in einen mit DPBS 1x mit Ca2+ undMg2+ gefüllten Behälter überführen und bei 4 °C lagern.
HINWEIS: Zu diesem Zeitpunkt ist die Netzhaut nur am Sehnerv befestigt. Dies ist ein Pausenpunkt in diesem Experiment. - Die Probe wird in eine 100-mm-Petrischale überführt, die DPBS 1x mit Ca2+ undMg2+ enthält. Stanzen Sie den Sehnervenkopf mit einem 1,5-mm-Biopsiestempel aus und entnehmen Sie die Netzhaut. Lagern Sie die neurale Netzhaut in DPBS 1x mit Ca2+ undMg2+ bei 4 °C.
HINWEIS: Bevor Sie den Sehnervenkopf ausstanzen, achten Sie darauf, den Sehnerv so weit wie möglich auf der Skleraseite zu schneiden. Dies erhöht die Präzision des Stempels. Das Ausstanzen des Sehnervs, nachdem der Flatmount in 4% PFA fixiert wurde, reduziert die Schädigung der RPE-Zellen, die sich um den Sehnerv befinden. - Entfernen Sie die Sklera aus dem RPE/Aderhaut, indem Sie die RPE/Aderhautschicht vorsichtig von der Peripherie abheben und die Aderhautgefäße und das Bindegewebe, die sich zwischen der Sklera und dem RPE befinden, mit einer Vannas-Federschere durchtrennen. Sobald die RPE/Aderhaut vollständig von der Sklera getrennt ist, sammeln Sie die RPE/Aderhautschicht. Das Gewebe in einen mit DPBS 1x mit Ca2+ undMg2+ gefüllten Behälter überführen und bei 4°C lagern.
HINWEIS: Zu diesem Zeitpunkt kann das Experiment pausiert werden.
4. Färbung
- Übertragen Sie die RPE/Aderhaut in eine Vertiefung einer 6-Well-Platte. Blockieren und permeabilisieren Sie die Probe in DPBS 1x mit Ca2+ undMg2+ mit 1% Rinderserumalbumin (BSA), 0,5% Tween 20 und 0,5% Triton X-100 für 1 h bei Raumtemperatur.
- Inkubieren Sie die Probe mit Phalloidin, das mit einem 647-Fluorophor konjugiert ist, in einer Verdünnung von 1:250 in DPBS 1x mit Ca 2+ und Mg2 mit 1 % BSA, 0,5 % Tween20 und 0,5 % Triton X-100 für 1 h bei Raumtemperatur. 3x in DPBS 1x mit Ca2+ undMg2+ waschen.
- Übertragen Sie die RPE/Aderhautprobe auf einen 50 mm x 75 mm großen Glasobjektträger und glätten Sie ihn. Schneiden Sie jedes "Blütenblatt" in zwei Teile, um die Probe flacher zu machen. Achten Sie auf die Makula. Zeichne mit einem hydrophoben Stift eine Kontur des Flatmounts.
- Um die Lipofuszin-Autofluoreszenz zu löschen, werden 500 μl der auf 1:20 verdünnten Autofluoreszenzlöschlösung in 70%igem Ethanol zugegeben und bei Raumtemperatur für 2 min inkubiert.
- Gründlich (mindestens 3x) in DPBS 1x mit Ca2+ undMg2+ waschen. Entfernen Sie den DPBS und fügen Sie das Einhängemedium hinzu. Legen Sie ein Deckglas auf das Flatmount und versiegeln Sie es mit Nagellack.
- Fotografieren Sie den Flatmount mit einem Fluoreszenzmikroskop (vorzugsweise mit einem 10-fachen oder 20-fachen Objektiv).
5. REShAPE-Analyse
HINWEIS: Da der KI-basierte Algorithmus von REShAPE mit 10x- und 20x-Bildern trainiert wurde, wird dringend empfohlen, bei der Bildgebung ein 10x- oder 20x-Objektiv zu verwenden . Ist dies nicht der Fall, müssen die Bilder entsprechend skaliert werden.
- Wenn die Bilder mit mehr als einem Fluoreszenzkanal aufgenommen werden, isolieren Sie den Kanal, der zur Erfassung der Zellränder verwendet wird. Exportieren Sie die Bilder als 16-Bit-Graustufen-TIF-Dateien.
HINWEIS: Bilder, die mit der Dateiendung .czi aufgenommen wurden, müssen nicht als TIF-Dateien exportiert werden, aber der Kanal, der die Zellenränder enthält, muss noch isoliert werden. - Installieren Sie die Software auf Windows x64- oder Linux-Plattformen (Centos 7).
HINWEIS: Die Software und die Installationsanweisungen finden Sie unter https://github.com/nih-nei/REShAPE. - Öffnen Sie die Software (Abbildung 1).
- Wählen Sie auf der Registerkarte Verzeichnisse die Eingabe- und Ausgabeordner aus. Wählen Sie im Eingabeverzeichnis den Ordner aus, der die Bilder enthält. Lassen Sie die Software im Ausgabeverzeichnis automatisch den Pfad in " Eingabeverzeichnis/Verarbeitet" ändern. Alternativ können Sie das Ausgabeverzeichnis auch manuell ändern.
HINWEIS: Die Software durchläuft alle Bilder, die im Eingabeordner enthalten sind. - Wählen Sie auf der Registerkarte NN-Segmentierungsoptionen die folgenden Einstellungen für die Bildsegmentierung aus:
- Batchgröße: Geben Sie einen Wert von 20 in das Textfeld als guten Ausgangspunkt ein, senken Sie ihn jedoch, wenn das System keine Grafikprozessoren (GPUs) mehr hat.
HINWEIS: Das Originalbild wird zur Bearbeitung in kleinere Kacheln aufgeteilt. Die Batchgröße gibt an, wie viele Kacheln gleichzeitig verarbeitet werden können. - GPU verwenden?: Wenn das System nicht über genügend GPU-Ressourcen verfügt, aktivieren Sie das Kontrollkästchen Nein.
- Interne Skalierung: Ändern Sie über das Dropdown-Menü die interne Skalierung von "Keine ", wenn die Bilder nicht mit einem 10-fachen oder 20-fachen Objektiv aufgenommen wurden. Skalieren Sie beispielsweise 40-fache Bilder mit der 1/2-Taste auf die halbe Größe. Die im Dropdown-Menü verfügbaren Optionen für die Neuskalierung sind wie folgt: 5x, 2x, Keine, 1/2, 1/5.
HINWEIS: Das maschinelle Lernen wurde mit 10x- und 20x-Bildern trainiert. Es kann sein, dass Zellränder von Bildern, die mit anderen Vergrößerungen aufgenommen wurden, nicht erkannt werden. In diesem Fall werden binäre Bilder vollständig schwarz angezeigt. - Arti-Filter: Verwenden Sie den Artefaktfilter, um sehr helle Partikel (Trümmer oder kleine Teile der Netzhaut oder des Bindegewebes um den Sehnerv herum) zu entfernen, die im Bild vorhanden sein können und die Segmentierung der Zellgrenzen stören können. Standardmäßig wird kein Filter verwendet. Die künstlichen Filteroptionen im Dropdown-Menü lauten wie folgt: Normal, Stark und Schwach.
- Batchgröße: Geben Sie einen Wert von 20 in das Textfeld als guten Ausgangspunkt ein, senken Sie ihn jedoch, wenn das System keine Grafikprozessoren (GPUs) mehr hat.
- Fügen Sie auf der Registerkarte Kachelbildoption einen Wert in das Textfeld ein, um den Grad der Überlappung zwischen den Bildkacheln anzugeben, indem Sie den Parameter " Kacheln auffüllen nach" anpassen. Eine Überlappung von 100 Pixeln funktioniert in der Regel gut.
- Passen Sie auf der Registerkarte "Ausgabegrafikoptionen " die Einstellungen für die Heatmap-Generierung an:
- Kachelbilder rekonstruieren?: Wenn das System nicht über genügend Ressourcen für die Analyse großer Bilder verfügt, aktivieren Sie das Kontrollkästchen Nein , damit die Software die Analyse abschließen kann, indem sie einzelne Kacheln speichert, ohne das gesamte Bild zu rekonstruieren.
- Farbbilder erstellen: Um Heatmaps zu erstellen, wählen Sie " Alle " aus dem Dropdown-Menü.
- Wählen Sie für die Färbung eine der verschiedenen Farbpaletten aus, die im Dropdown-Menü verfügbar sind: Thermisch, Grün, Mpl-Magma, Phase, Feuer, Jet, Cyan Hot.
- Speichern Sie die Bilder unter Bildformat als TIF oder PNG, indem Sie eine der Optionen auf der Registerkarte auswählen.
- Aktivieren Sie in Bezug auf die Funktion "Manuelle Grenzwerte verwenden?" das Kontrollkästchen Nein , damit die Software die in jedem Bild erkannten Minimal- und Maximalwerte verwenden kann. Aktivieren Sie das Kontrollkästchen Ja , um den Wertebereich für jede Heatmap der Formmetrik manuell anzupassen, und klicken Sie auf die Schaltfläche Grenzwerte festlegen , um Bereiche für die einzelnen Parameter auszuwählen, indem Sie Werte in die Textfelder einfügen. Nachdem Sie die gewünschten Werte geändert haben, klicken Sie auf Speichern. Klicken Sie auf Standardwerte laden , um alle Grenzwerte zurückzusetzen.
HINWEIS: Verwenden Sie manuelle Grenzwerte, wenn Heatmaps aus mehreren Bildern verglichen werden müssen, z. B. beim Vergleich der Auswirkungen verschiedener Verbindungen auf die Zellform. Auf diese Weise wird derselbe Wertebereich verwendet. Der Satz der manuell einzugebenden Werte variiert je nach Art der Probe. Es wird empfohlen, einige Iterationen auszuführen, um den optimalen Bereich auszuwählen.
- Wählen Sie auf der Registerkarte Zellengrößenbeschränkungen für die Analyse einen Schwellenwert für die Zellengröße für die Analyse aus:
- Geben Sie unter Niedrigere Zellengröße die Größe der kleinsten Zelle in das Textfeld ein, die in die Analyse einbezogen werden soll.
- Geben Sie unter Obere Zellengröße die Größe der größten Zelle in das Textfeld ein, die in die Analyse einbezogen werden soll.
HINWEIS: Die Einheit der Zellengröße ändert sich von Pixeln zu Mikrometern im Quadrat, abhängig von der Option, die auf der Registerkarte " Automatische Einheitenumrechnung" ausgewählt wurde.
- Wählen Sie auf der Registerkarte Automatische Einheitenumrechnung die bevorzugte Einheit für die Analyse aus:
- Aktivieren Sie unter Pixel in reale Einheiten konvertieren? das Kontrollkästchen Nein , um die Analyse in Pixeleinheiten auszuführen. Aktivieren Sie das Kontrollkästchen Ja , um die Analyse in Mikrometern auszuführen.
- Geben Sie unter Länge des Maßstabsbalkens (Pixel) den Pixelwert in das Textfeld ein.
- Geben Sie unter Länge des Maßstabsbalkens (Mikrometer) den entsprechenden Abstand in Mikrometern in das Textfeld ein.
- Um die Analyse zu starten, klicken Sie auf Go For It.
HINWEIS: Die Software kann auch die Lebensfähigkeit der Zellen messen, wenn die Zellen mit 4',6-Diamidino-2-phenylindol (DAPI) und Propidiumiodid gefärbt sind (Registerkarte CZI-Optionen ), dies gilt jedoch nicht für RPE-Flatmounts.
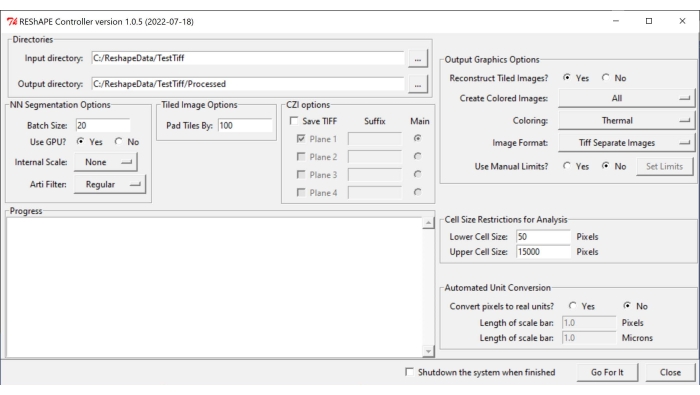
Abbildung 1: Grafische Benutzeroberfläche von REShAPE. Die grafische Benutzeroberfläche verfügt über verschiedene Registerkarten zum Auswählen der Arbeitsverzeichnisse (Registerkarte Verzeichnisse ), zum Ändern der Segmentierungsoptionen (Registerkarten NN-Segmentierungsoptionen und Kachelbildoptionen ), zum Festlegen der Parameter für die Analyse (Zellengrößenbeschränkungen für die Analyse und zur automatischen Einheitenumrechnung ) und für die Heatmap-Generierung (Registerkarte Ausgabegrafikoptionen ). Abkürzung: GUI = grafische Benutzeroberfläche. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergebnisse
Dieses Protokoll führt zu einem einstufigen Bild eines Flatmounts, in dem die Zellposition und 30 Formmetriken für jede korrekt identifizierte RPE-Zelle gemessen werden (Abbildung 2). Ein Ordner mit dem Namen "Processed" wird automatisch im Eingabeverzeichnis generiert. Dieser Ordner enthält vier Unterverzeichnisse mit den Namen "Analyse", "Farbcoded", "Kombinierte Dateien" und "Segmentierte Bilder" sowie einige temporäre Dateien, die während der Analyse generiert werden. Der Ordner "Kombinierte Dateien" enthält eine Tabelle mit allen Formmaßen und eine Tabelle mit den Häufigkeiten der Zellennachbarn aller Dateien kombiniert. Der Ordner "Analyse" enthält eine Tabelle mit allen Formmaßen und eine Tabelle mit den Häufigkeiten der Zellnachbarn für jedes Bild separat. Das Verzeichnis "Segmented Images" enthält die endgültigen binären Masken der RPE-Zellengrenzen. Es kann verwendet werden, um die Qualität der Segmentierung zu bewerten. Das Verzeichnis "Color Coded" enthält Heatmaps für jede Formmessung, um die Formmuster in jedem Bild zu visualisieren. Die Definitionen und Abkürzungen der Formmetrik finden Sie in Tabelle 1.
Manchmal können RPE-Flatmounts Reststücke der Netzhaut enthalten, die nicht sauber entfernt wurden, insbesondere im Bereich des Sehnervs. Die Phalloidin-Färbung der Probe führt zu einem starken Signal von der Netzhaut, was zu Problemen bei der RPE-Zellgrenzsegmentierung führen kann. Einige Kacheln werden vollständig schwarz angezeigt, während die umgebenden Kacheln eine normale Segmentierung aufweisen. Andere helle Objekte, die im Bild vorhanden sein können, verursachen ebenfalls die Erzeugung schwarzer Kacheln (Abbildung 3). In diesen Fällen wird durch die Auswahl einer der Filteroptionen (Schwach, Normal, Stark) im Dropdown-Menü Arti-Filter die Bildung schwarzer Kacheln verhindert.
REShAPE verwendet 8-Bit- oder 16-Bit-Graustufenbilder als Eingabe, aber keine RGB-Bilder. Die Verwendung von RGB-Bildern für die REShAPE-Analyse erzeugt vollständig schwarze Binärbilder. In diesem Fall werden durch die Konvertierung der RGB-Bilder in Graustufen korrekt segmentierte Binärbilder erzeugt (Abbildung 4). In einigen Fällen, in denen die RPE-Ränder nicht korrekt erkannt werden, z. B. wenn die Färbung nicht optimal ist oder wenn die Probe durch einen Kratzer beschädigt ist (Abbildung 5A), können große Zellklumpen als eine einzige sehr große Zelle identifiziert werden (Abbildung 5B). In diesem Fall können große Objekte von der Analyse ausgeschlossen werden, indem der Schwellenwert für die Zellgröße reduziert wird (Abbildung 5C). Dies kann erreicht werden, indem Sie einen niedrigeren Wert in das Textfeld Obere Zellengröße einfügen. Dies führt jedoch zu einer Änderung der Reichweite der Heatmap. Wenn sich ein Forscher dafür entscheidet, ist es auch möglich, den ursprünglichen Heatmap-Bereich (Abbildung 5D) beizubehalten, indem er das Kontrollkästchen Ja in der Funktion "Manuelle Grenzwerte verwenden?" aktiviert. Anschließend muss der Forscher mit der linken Maustaste auf die Schaltfläche Grenzwerte festlegen klicken und die gewünschten Werte in die Textfelder einfügen, um die manuellen Grenzwerte festzulegen.
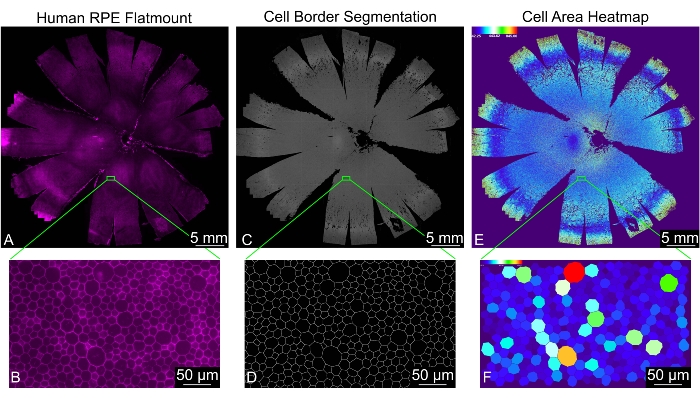
Abbildung 2: Vollständige morphometrische Analyse einer gesamten menschlichen RPE-Monoschicht. (A) Eine Ansicht mit geringer Vergrößerung eines gesamten menschlichen RPE/Aderhaut-Flatmounts (Magenta: Phalloidin). (B) Eine vergrößerte Ansicht von Phalloidin-gefärbten RPE-Zellen. (C) REShAPE-generierte Segmentierung der RPE-Zellgrenzen für ein gesamtes menschliches RPE/Aderhaut-Flatmount und (D) die entsprechende vergrößerte Ansicht. (E) Eine softwaregenerierte Heatmap, die die Zellfläche der einzelnen RPE-Zellen im gesamten menschlichen Flatmount veranschaulicht. Die thermische Skala in der oberen linken Ecke zeigt den Bereich der verwendeten Werte an. (F) Die entsprechende vergrößerte Ansicht, in der einzelne RPE-Zellen nach Fläche gefärbt angezeigt werden. Maßstabsbalken = (B,D,F) 50 μm, (A,C,E) 5 mm. Abkürzung: RPE = retinales Pigmentepithel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
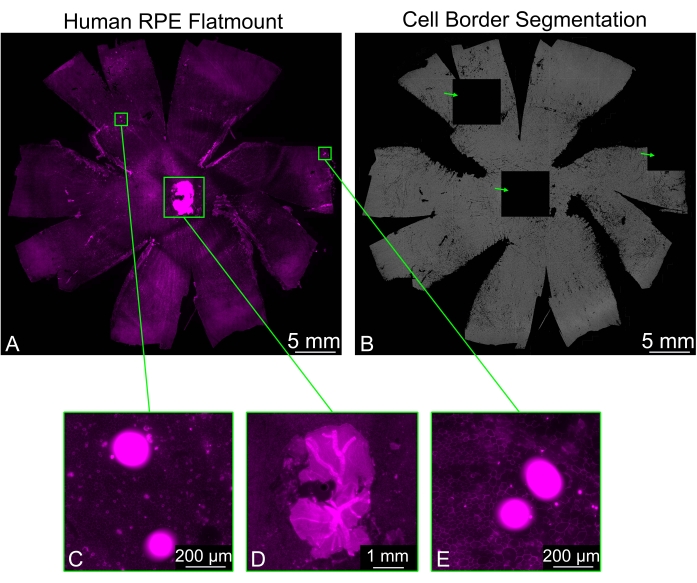
Abbildung 3: Filterung von hellen Artefakten. (A) Ein menschliches RPE-Flatmount, das auf Zellränder gefärbt ist (Magenta: Phalloidin), kann helle Bereiche (grüne Rechtecke darstellen), die die Segmentierung stören. (B) Die RPE-Zellenrandsegmentierung des gesamten Flatmounts enthält drei komplett schwarze Kacheln (grüne Pfeile), die hellen Fluoreszenzbereichen entsprechen. (C,E) Zwei der schwarzen Kacheln entsprechen Bereichen, die helle Punkte enthalten, bei denen es sich möglicherweise um Trümmer handelt. (D) Eine der schwarzen Kacheln wurde durch ein Stück neuronaler Netzhaut um den Sehnerv herum erzeugt, das nicht korrekt entfernt wurde. Die Teile der neuronalen Netzhaut sind wesentlich heller als die RPE-Schicht und behindern die Zellsegmentierung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Eingabebild erforderlich. Die RPE-Zellen, die für die Zellränder gefärbt wurden, wurden als (A) RGB- oder (B) Graustufen-16-Bit-Bilder für die REShAPE-Analyse gespeichert. (C) Das Ergebnis der RBG-Bildanalyse ist ein schwarzes Binärbild, (D) während die Analyse des Graustufenbildes eine korrekt segmentierte Binärdatei der Zellenränder erzeugt. REShAPE kann nur 8-Bit- oder 16-Bit-Graustufenbilder analysieren. Maßstabsbalken = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: Suboptimale Ergebnisse . (A) Ein Bild eines Teils der RPE-Monoschicht, in dem die mit Phalloidin gefärbten Zellen versehentlich zerkratzt wurden. (B) Eine Heatmap von RPE-Zellen, die nach der Dimension der Zellfläche eingefärbt sind. Ein großer Schwellenwert für die obere Zellengröße schließt große Objekte in die Analyse ein. (C) Eine Heatmap der Zellfläche, in der ein kleinerer Schwellenwert für die obere Zellengröße gewählt wurde, um große Objekte von der Analyse auszuschließen. (D) Eine Heatmap für den Zellbereich, in der ein kleinerer Schwellenwert für die obere Zellengröße gewählt und manuelle Grenzwerte festgelegt wurden, um den ursprünglich verwendeten Heatmap-Bereich beizubehalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 1: REShAPE-Parameter. Die Tabelle enthält die Definition der einzelnen Parameter und die Abkürzungen, die in den Rohtabellenkalkulationen ("_Data.csv"-Dateien) und für die Heatmaps verwendet werden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Die konsequente und effiziente Trennung von humanem RPE und Netzhaut kann mit diesem Protokoll erreicht werden. Diese Methode ermöglicht die Untersuchung regionaler Unterschiede in der RPE-Form über die gesamte menschliche Netzhaut5. Ein entscheidender Schritt im Protokoll ist die physische Trennung von RPE und Netzhaut. Wenn sich die beiden Gewebe in einigen Bereichen nicht vollständig lösen, sollte man die Netzhaut vorsichtig anheben, um sicherzustellen, dass das Gewebe nicht bricht. Die REShAPE-Analyse von großen Flatmounts kann den Einsatz von Systemen mit beträchtlichen RAM-Ressourcen erfordern. In diesem Fall kann die Reassemblierung des gesamten Bildes deaktiviert werden, damit die Software die Analyse trotz fehlender Verarbeitungsressourcen erfolgreich abschließen kann.
Die Haupteinschränkung bei der Verwendung von REShAPE zur Segmentierung menschlicher RPE-Flatmounts besteht darin, dass der KI-Algorithmus hauptsächlich auf Bildern von induzierten pluripotenten Stammzellen-abgeleiteten RPE trainiert wurde. Infolgedessen ist die Segmentierung von menschlichen RPE-Flatmounts weniger genau. RPE-Zellen von gealterten Spendern enthalten eine große Menge an Lipofuszin7, und das breite Spektrum seiner Autofluoreszenz stört die Segmentierung der Zellgrenzen. In Zukunft werden mehr Bilder von RPE-Flatmounts verwendet, um die Segmentierung der Zellgrenzen in dieser Art von Probe zu verbessern. Trotz dieser Einschränkung wurde REShAPE speziell darauf trainiert, RPE-Zellgrenzen zu erkennen und zu segmentieren und schneidet besser ab als andere bestehende Methoden, wie z.B. die Segmentierung von RPE-Zellen Voronoi8 und CellProfiler9 .
Darüber hinaus bietet REShAPE im Vergleich zur manuellen Segmentierung 10 den Vorteil, große Bilder schnell zu analysieren (~130.000 Pixel x130.000 Pixel wurden getestet). Zusammenfassend lässt sich sagen, dass diese Präpariermethode sehr reproduzierbar ist und leicht auf andere Tiermodelle ausgedehnt werden kann. Darüber hinaus kann die Software verwendet werden, um die RPE-Form in Augen-Flatmounts oder in Zellkulturmodellen zu untersuchen, um die Wirkung bestimmter Behandlungen zu untersuchen. Schließlich macht die Vielseitigkeit von REShAPE es breit anwendbar für die Analyse anderer Arten von Epithelzellen.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Wir danken dem Histologiekern des National Eye Institute (NEI) für den Einsatz des Zeiss Axio Scan.Z1. Wir danken auch den Spendern, ihren Familien, dem Advancing Sight Network und dem Lions Eye Institute für ihre Großzügigkeit. Diese Arbeit wurde durch NEI IRP-Mittel (Förderkennzeichen ZIA EY000533-04) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Biopsy punch 1.5 mm | Acuderm Inc. | P1525 | |
| Bovine albumin | MP Biomedicals | 160069 | |
| Coverglass 50 x 75 mm, #1.5 thickness | Brain Research Laboratories | 5075-1.5D | |
| Curved spatula | Katena | K3-6600 | |
| D-Mannitol | Sigma | M9546 | |
| DPBS 1x with Ca2+ and Mg2+ | Gibco | 14040-133 | |
| Fine Scissors | Fine Science Tools | 14558-11 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Forceps - Dumont #5 | Fine Science Tools | 11252-23 | |
| Microscope slides 50 x 75 x 1.2 mm | Brain Research Laboratories | 5075 | |
| Needles 21 G x 1-1/2" hypodermic | Becton Dickinson (BD) | 305167 | |
| Needles 27 G x 1-1/4" hypodermic | Becton Dickinson (BD) | 305136 | |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Petri dish 100 mm | Corning | 430167 | |
| Phalloidin-iFluor 647 | Abcam | ab176759 | |
| Razor blades | PAL (Personna) | 62-0177 | |
| Round bottom tubes 50 mL | Newegg | 9SIA4SR9M88854 | |
| Silicon Elastomer Kit | Dow Corning Corporation | 4019862 | |
| Square weighing boat (81 mm x 81 mm x 25 mm) | Sigma | W2876 | |
| Surgical Vitrectomy System | BD Visitrec | 585100 | optional |
| Syringe 1 mL | Becton Dickinson (BD) | 309659 | |
| Triton X-100 | Sigma | T9284 | |
| TrueBlack | Biotium | 23007 | autofluorescence quencher |
| Tween 20 | Affymetrix | 20605 | |
| Vannas Spring Scissors - 3 mm cutting edge | Fine Science Tools | 15000-10 |
Referenzen
- Endo, E. G., Yao, X. Y., Marmor, M. F. Pigment adherence as a measure of retinal adhesion: Dependence on temperature. Investigative Ophthalmology and Visual Science. 29 (9), 1390-1396 (1988).
- Marmor, M. F. Retinal detachment from hyperosmotic intravitreal injection. Investigative Ophthalmology and Visual Science. 18 (12), 1237-1244 (1979).
- Marmor, M. F., Martin, L. J., Tharpe, S. Osmotically induced retinal detachment in the rabbit and primate. Electron microscopy of the pigment epithelium. Investigative Ophthalmology and Visual Science. 19 (9), 1016-1029 (1980).
- Ban, Y., Rizzolo, L. A culture model of development reveals multiple properties of RPE tight junctions. Molecular Vision. 3, 18 (1997).
- Ortolan, D., et al. Single-cell-resolution map of human retinal pigment epithelium helps discover subpopulations with differential disease sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 119 (19), 2117553119 (2022).
- Schaub, N. J., et al. Deep learning predicts function of live retinal pigment epithelium from quantitative microscopy. Journal of Clinical Investigation. 130 (2), 1010-1023 (2020).
- Beatty, S., Koh, H. -. H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Liu, Z., Kocaoglu, O. P., Miller, D. T. 3D imaging of retinal pigment epithelial cells in the living human retina. Investigative Ophthalmology and Visual Science. 57 (9), 533-543 (2016).
- Bhatia, S. K., et al. Analysis of RPE morphometry in human eyes. Molecular Vision. 22, 898-916 (2016).
- vonder Emde, L., et al. Histologic cell shape descriptors for the retinal pigment epithelium in age-related macular degeneration: A comparison to unaffected eyes. Translational Vision Science & Technology. 11 (8), 19 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten