Method Article
Génération efficace et cohérente d’épithélium/platures choroïdes pigmentaires rétiniens à partir d’yeux humains pour l’analyse histologique
Dans cet article
Résumé
Nous décrivons une méthode pour séparer efficacement l’épithélium pigmentaire rétinien (EPR) de la rétine dans les yeux humains et générer des supports plats RPE / choroïdes entiers pour les analyses histologiques et morphométriques de l’EPR.
Résumé
L’épithélium pigmentaire rétinien (EPR) et la rétine sont des tissus fonctionnellement et structurellement connectés qui travaillent ensemble pour réguler la perception de la lumière et la vision. Les protéines de la surface apicale de l’EPR sont étroitement associées aux protéines de la surface du segment externe du photorécepteur, ce qui rend difficile la séparation cohérente de l’EPR des photorécepteurs/rétine. Nous avons développé une méthode pour séparer efficacement la rétine de l’EPR des yeux humains afin de générer des supports plats complets de l’EPR/choroïde et de la rétine pour une analyse cellulaire séparée des photorécepteurs et des cellules EPR. Une injection intravitréenne d’une solution à haute osmolarité de D-mannitol, un sucre non transporté par l’EPR, a induit la séparation de l’EPR et de la rétine sur toute la chambre postérieure sans endommager les jonctions cellulaires de l’EPR. Aucun patch EPR n’a été observé attaché à la rétine. Le marquage de l’actine à la phalloïdine a montré la préservation de la forme de l’EPR et a permis une analyse morphométrique de l’épithélium entier. Un logiciel basé sur l’intelligence artificielle (IA) a été développé pour reconnaître et segmenter avec précision les frontières cellulaires RPE et quantifier 30 mesures de forme différentes. Cette méthode de dissection est hautement reproductible et peut être facilement étendue à d’autres modèles animaux.
Introduction
L’épithélium pigmentaire rétinien (EPR) et la rétine neurale sont fortement interconnectés l’un avec l’autre en raison de la forte dépendance physiologique des photorécepteurs à l’EPR. Pendant la dissection, la séparation mécanique de la rétine neurale de l’EPR provoque la déchirure des cellules EPR, les parties apicales de l’EPR restant attachées aux segments externes des photorécepteurs rétiniens. L’étendue de l’adhérence RPE-rétinienne est si grande que la quantité de pigment restant sur la rétine après la séparation est utilisée pour quantifier la force de l’adhérence rétinienne1. Plus précisément, les jonctions serrées RPE et la structure d’actine qui les relie, qui sont situées du côté apical, se rompent lors de la séparation mécanique. Par conséquent, la coloration des supports plats RPE pour les bordures de cellule entraîne une monocouche inégale dans laquelle de nombreuses cellules ont des bordures manquantes. Cet effet est exacerbé lorsque le tissu est fixé avec du paraformaldéhyde (PFA) avant la dissection, car les protéines deviennent réticulées.
Des études sur l’administration intravitréenne de médicaments ont montré que les injections de solutions hyperosmotiques dans la chambre postérieure induisent un décollement de la rétine 2,3. Dans ces expériences, 50 μL de solutions différentes, allant de 1 000 mOsm à 2 400 mOsm, injectés dans le vitré moyen ont provoqué un décollement de la rétine en quelques minutes. Notamment, même après de longues expositions à des solutions à haute osmolarité, les jonctions serrées RPE sont apparues intactes dans les images microscopiques électroniques de transmission des yeux de lapin et de singe3. Suivant une stratégie similaire, nous avons injecté dans le vitré moyen une solution hyperosmotique de D-mannitol pour induire un décollement de la rétine efficace avant d’effectuer une dissection RPE. Comme le D-mannitol n’est pas transporté par l’EPR4, une concentration intravitréenne élevée est maintenue, générant un gradient osmotique. La séparation efficace de l’EPR et de la rétine sur toute la chambre postérieure garantit la préservation des jonctions cellulaires de l’EPR et permet l’étude de la morphométrie de l’EPR sur l’ensemble du support plat. En outre, nous avons développé un logiciel basé sur l’intelligence artificielle (IA) qui reconnaît et segmente les bordures de cellules RPE marquées par fluorescence, quantifie 30 métriques de forme différentes et produit des cartes thermiques de chaque métrique pour la visualisation 5,6.
Protocole
Les globes humains de cadavres ont été obtenus auprès de l’Advanced Sight Network (Birmingham, AL). Les travaux effectués sur des tissus de cadavres sont exemptés par le NIH Institutional Review Board du comité d’éthique de la recherche.
1. Envoi de globe oculaire
- Après l’énucléation, expédiez les globes oculaires frais dans un récipient rempli de DPBS 1x glacé avec Ca 2+ et Mg2+.
REMARQUE: Il est préférable de disséquer l’œil dans les 24 heures suivant l’énucléation. La morphologie de l’EPR n’est pas modifiée pendant cette fenêtre temporelle.
2. Préparation du moule en silicone
- Couper le fond de 20 mm d’un tube à fond rond de 25 mm de diamètre. Placez-le à la base d’un bateau de pesage carré (81 mm x 81 mm x 25 mm).
- Mélangez les deux composants du kit en élastomère de silicium à un rapport de 10: 1, en faisant attention à ne pas emprisonner l’air. Verser le mélange dans le bateau de pesée contenant la pièce sphérique du tube à fond rond.
- Durcir le silicone à température ambiante pendant la nuit. Retirez le bateau de pesage et le tube à fond rond du moule en silicone durci.
3. Dissection RPE
- Nettoyez la sclérotique du globe oculaire frais des muscles et du tissu conjonctif. Fixez l’œil au moule en silicone à l’aide d’aiguilles de 27 G enfilées dans la sclérotique. Remplissez la cavité du moule avec DPBS 1x avec Ca 2+ et Mg2+.
REMARQUE: Faites attention à ne pas percer la chambre oculaire. Les aiguilles ne doivent passer que par la sclérotique. - Utilisez une seringue de 1 mL et une aiguille de 21 G pour injecter ~400 μL de solution de D-mannitol de 1 700 mOsm dans le vitré. Insérez l’aiguille à travers la pars plana pour éviter de percer la chambre antérieure de l’œil. Laissez l’œil à température ambiante pendant ~45 min.
- Coupez la chambre antérieure au niveau de la pars plana à l’aide d’une paire de ciseaux fins et de pinces. Remplissez la cavité oculaire postérieure avec DPBS 1x avec Ca 2+ et Mg2+. Sous le stéréomicroscope, localisez la macula (la tache jaune sur la rétine).
- Si un coupe-vitré chirurgical est disponible, retirez le vitré et remplacez-le par DPBS 1x avec Ca 2+ et Mg2+. Sinon, essayez de soulever le vitré avec une pince et coupez-le avec des ciseaux fins.
- En faisant attention à préserver la région maculaire, coupez l’œil en quadrants: nasal, temporal, supérieur et inférieur. Retirez les aiguilles si elles sont sur le chemin.
- Transférer la chambre postérieure papillon de l’œil dans une boîte de Petri de 100 mm contenant du DPBS 1x avec Ca 2+ et Mg2+. Avant de retirer la rétine, marquez le pétale qui contient la macula en faisant une petite coupe (en forme de V) dans la marge ciliaire. Soulevez et coupez tout le vitré qui repose sur la rétine.
- Soulevez doucement la rétine de plusieurs côtés à l’aide d’une spatule incurvée ou d’une paire de pinces pour vérifier si la rétine s’est détachée de l’EPR et laisser circuler un peu de liquide entre les deux couches.
REMARQUE: La rétine sera toujours attachée à la périphérie (marges ciliaires) et au nerf optique. - Coupez la rétine des marges ciliaires dans tous les pétales, en veillant à ne pas rayer l’EPR. Placer le tissu dans 4% PFA et incuber pendant ~1 h. Lavez 3x avec DPBS 1x avec Ca 2+ et Mg2+. Transférer le mouchoir dans un récipient rempli de DPBS 1x avec Ca 2+ et Mg2+ et le conserver à 4 °C.
REMARQUE: À ce stade, la rétine n’est attachée qu’au niveau du nerf optique. C’est un point de pause dans cette expérience. - Transférer l’échantillon dans une boîte de Petri de 100 mm contenant du DPBS 1x avec Ca 2+ et Mg2+. Poinçonner la tête du nerf optique avec un poinçon de biopsie de 1,5 mm et recueillir la rétine. Conserver la rétine neurale dans DPBS 1x avec Ca 2+ et Mg2+ à 4 °C.
REMARQUE: Avant de poinçonner la tête du nerf optique, assurez-vous de couper le nerf optique autant que possible du côté scléral. Cela augmentera la précision du poinçon. Le poinçonnage du nerf optique après la fixation du support plat dans 4% de PFA réduit les dommages aux cellules EPR situées autour du nerf optique. - Retirez la sclérotique de l’EPR/choroïde en soulevant doucement la couche EPR/choroïde de la périphérie et en coupant les vaisseaux choroïdiens et le tissu conjonctif qui se trouvent entre la sclérotique et l’EPR avec une paire de ciseaux à ressort Vannas. Une fois que l’EPR/choroïde est complètement séparée de la sclérotique, prélever la couche EPR/choroïde. Transférer le mouchoir dans un récipient rempli de DPBS 1x avec Ca 2+ et Mg2+ et le conserver à 4°C.
Remarque : À ce stade, l’expérience peut être suspendue.
4. Coloration
- Transférer l’EPR/choroïde dans un puits d’une plaque à 6 puits. Bloquer et perméabiliser l’échantillon dans la DPBS 1x avec du Ca 2+ et du Mg 2+ avec 1 % d’albumine sérique bovine (BSA), 0,5 % de Tween20 et 0,5 % de Triton X-100 pendant 1 h à température ambiante.
- Incuber l’échantillon avec de la phalloïdine conjuguée à un fluorophore 647 à une dilution de 1:250 dans du DPBS 1x avec du Ca 2+ et du Mg 2+ avec 1 % de BSA, 0,5 % de Tween20 et 0,5 % de Triton X-100 pendant 1 h à température ambiante. Laver 3x dans DPBS 1x avec Ca 2+ et Mg2+.
- Transférer l’échantillon EPR/choroïde sur une lame de verre de 50 mm x 75 mm et l’aplatir. Coupez chaque « pétale » en deux pour rendre l’échantillon plus plat. Faites attention à la macula. Dessinez un contour du support plat avec un stylo hydrophobe.
- Pour éteindre l’autofluorescence de la lipofuscine, ajouter 500 μL de la solution d’autofluorescence diluée à 1:20 dans de l’éthanol à 70% et incuber à température ambiante pendant 2 min.
- Laver abondamment (au moins 3x) dans DPBS 1x avec Ca 2+ et Mg2+. Retirez le DPBS et ajoutez le support de montage. Placez un verre de couverture sur le support plat et scellez avec du vernis à ongles.
- Imagez le support plat avec un microscope à fluorescence (de préférence en utilisant un objectif 10x ou 20x).
5. Analyse REShAPE
REMARQUE: Comme l’algorithme basé sur l’IA REShAPE a été formé sur des images 10x et 20x, il est donc fortement recommandé d’utiliser un objectif 10x ou 20x lors de l’imagerie. Sinon, les images devront être redimensionnées en conséquence.
- Si les images sont acquises avec plus d’un canal fluorescent, isoler le canal utilisé pour acquérir les bordures cellulaires. Exportez les images sous forme de fichiers TIF en niveaux de gris 16 bits.
REMARQUE: Les images acquises avec l’extension de fichier .czi n’ont pas besoin d’être exportées en tant que fichiers TIF, mais le canal contenant les bordures de cellule doit toujours être isolé. - Installez le logiciel sur les plates-formes Windows x64 ou Linux (Centos 7).
REMARQUE: Le logiciel et les instructions d’installation peuvent être trouvés à https://github.com/nih-nei/REShAPE. - Ouvrez le logiciel (Figure 1).
- Dans l’onglet Répertoires , sélectionnez les dossiers d’entrée et de sortie. Dans le répertoire d’entrée, sélectionnez le dossier qui contient les images. Dans le répertoire de sortie, laissez le logiciel changer automatiquement le chemin en « répertoire d’entrée/Traité ». Vous pouvez également modifier le répertoire de sortie manuellement.
REMARQUE: Le logiciel itérera à travers toutes les images contenues dans le dossier d’entrée. - Dans l’onglet Options de segmentation NN, choisissez les paramètres suivants pour la segmentation des images :
- Taille du lot : saisissez une valeur de 20 dans la zone de texte comme bon point de départ, mais abaissez-la si le système manque d’unités de traitement graphique (GPU).
Remarque : L’image d’origine est divisée en vignettes plus petites pour le traitement. La taille du lot spécifie le nombre de tuiles pouvant être traitées simultanément. - Utiliser le GPU?: Si le système ne dispose pas de suffisamment de ressources GPU, cochez la case Non.
- Échelle interne : à l’aide du menu déroulant, modifiez la mise à l’échelle interne de Aucune dans les cas où les images n’ont pas été acquises avec un objectif 10x ou 20x. Par exemple, réduisez la taille des images 40x à la moitié de la taille à l’aide du bouton 1/2 . Les options de redimensionnement disponibles dans le menu déroulant sont les suivantes : 5x, 2x, Aucune, 1/2, 1/5.
REMARQUE: L’apprentissage automatique a été formé avec des images 10x et 20x. Il peut ne pas reconnaître les bordures cellulaires à partir d’images prises à d’autres grossissements. Dans ce cas, les images binaires apparaîtront complètement noires. - Filtre Arti : Utilisez l’onglet du filtre d’artefact pour éliminer les particules très brillantes (débris ou petites parties de la rétine ou du tissu conjonctif autour du nerf optique) qui peuvent être présentes dans l’image et peuvent interférer avec la segmentation de la frontière cellulaire. Aucun filtre n’est utilisé par défaut. Les options de filtre artificiel dans le menu déroulant sont les suivantes : Régulier, Fort et Faible.
- Taille du lot : saisissez une valeur de 20 dans la zone de texte comme bon point de départ, mais abaissez-la si le système manque d’unités de traitement graphique (GPU).
- Dans l’onglet Option d’image en mosaïque , insérez une valeur dans la zone de texte pour spécifier le degré de chevauchement entre les vignettes d’image en ajustant le paramètre Mosaïque de bloc-notes par . Un chevauchement de 100 pixels fonctionne généralement bien.
- Dans l’onglet Options graphiques de sortie , réglez les paramètres de génération de carte thermique :
- Reconstruct Tiled Images?: si le système ne dispose pas de suffisamment de ressources pour l’analyse de grandes images, cochez la case Non pour permettre au logiciel de terminer l’analyse en enregistrant des tuiles individuelles sans reconstruire l’image entière.
- Créer des images couleur : Pour générer des cartes thermiques, sélectionnez Tout dans le menu déroulant.
- Pour Coloration, choisissez l’une des différentes palettes de couleurs disponibles dans le menu déroulant: Thermique, Vert, Mpl-magma, Phase, Feu, Jet, Cyan Chaud.
- Pour Format d’image, enregistrez les images au format TIF ou PNG en sélectionnant l’une des options de l’onglet.
- En ce qui concerne la fonctionnalité Utiliser les limites manuelles? , cochez la case Non pour permettre au logiciel d’utiliser les valeurs minimales et maximales détectées dans chaque image. Cochez la case Oui pour ajuster manuellement la plage de valeurs pour chaque carte thermique métrique de forme, puis cliquez sur le bouton Définir les limites pour choisir des plages pour les paramètres individuels en insérant des valeurs dans les zones de texte. Après avoir modifié les valeurs d’intérêt, cliquez sur Enregistrer. Cliquez sur Load Defaults pour réinitialiser toutes les limites.
REMARQUE: Utilisez des limites manuelles si les cartes thermiques de plusieurs images doivent être comparées, par exemple lors de la comparaison des effets de différents composés sur la forme des cellules. De cette façon, la même plage de valeurs est utilisée. L’ensemble des valeurs à saisir manuellement varie en fonction du type d’échantillon. Il est recommandé d’exécuter quelques itérations pour choisir la plage optimale.
- Dans l’onglet Restrictions de taille de cellule pour l’analyse , sélectionnez un seuil de taille de cellule pour l’analyse :
- Dans Taille inférieure de la cellule, insérez dans la zone de texte la taille de la plus petite cellule à inclure dans l’analyse.
- Dans Taille de la cellule supérieure, insérez dans la zone de texte la taille de la plus grande cellule à inclure dans l’analyse.
REMARQUE : l’unité de taille de cellule passe de pixels à micromètres carrés en fonction de l’option choisie dans l’onglet Conversion d’unité automatisée .
- Dans l’onglet Conversion d’unités automatisée , choisissez l’unité préférée pour l’analyse :
- Dans Convertir des pixels en unités réelles ?, cochez la case Non pour exécuter l’analyse en unités de pixels. Cochez la case Oui pour exécuter l’analyse en micromètres.
- Dans Longueur de la barre d’échelle (pixels), entrez la valeur en pixels dans la zone de texte.
- Dans Longueur de la barre d’échelle (microns), entrez dans la zone de texte la distance correspondante en micromètres.
- Pour lancer l’analyse, appuyez sur Go For It.
REMARQUE: Le logiciel peut également mesurer la viabilité cellulaire lorsque les cellules sont colorées avec du 4',6-diamidino-2-phénylindole (DAPI) et de l’iodure de propidium (onglet Options CZI ), mais cela ne s’applique pas aux supports plats RPE.
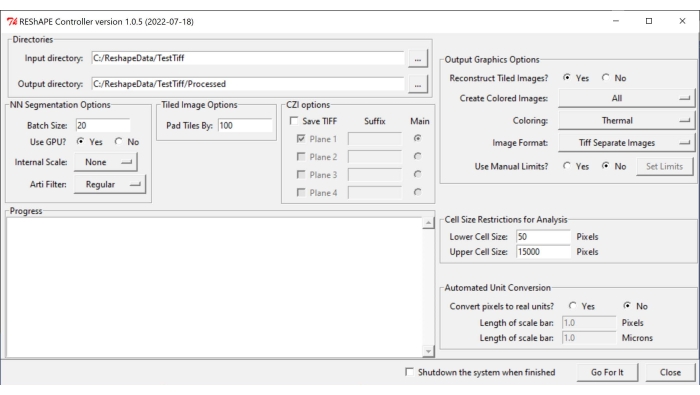
Figure 1 : Interface utilisateur graphique REShAPE. L’interface graphique comporte différents onglets pour sélectionner les répertoires de travail (onglet Répertoires), modifier les options de segmentation (onglets Options de segmentation NN et Options d’image en mosaïque), spécifier les paramètres d’analyse (onglets Restrictions de taille de cellule pour l’analyse et Conversion automatisée d’unités) et pour la génération de cartes thermiques (onglet Options graphiques de sortie). Abréviation : GUI = interface utilisateur graphique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Ce protocole permet d’obtenir une image à plan unique d’un montage plat, où l’emplacement de la cellule et 30 mesures de forme sont mesurés pour chaque cellule RPE correctement identifiée (Figure 2). Un dossier nommé « Traité » est automatiquement généré dans le répertoire d’entrée. Ce dossier contient quatre sous-répertoires, nommés « Analyse », « Code couleur », « Fichiers combinés » et « Images segmentées », ainsi que des fichiers temporaires générés au cours de l’analyse. Le dossier « Fichiers combinés » contient une feuille de calcul avec toutes les mesures de forme et une feuille de calcul avec les fréquences du nombre de voisins de cellules de tous les fichiers combinés. Le dossier « Analyse » contient une feuille de calcul avec toutes les mesures de forme et une feuille de calcul avec les fréquences du nombre de cellules voisines pour chaque image séparément. Le répertoire « Images segmentées » contient les masques binaires finaux des bordures des cellules RPE ; Il peut être utilisé pour évaluer la qualité de la segmentation. Le répertoire « Color Coded » contient des cartes thermiques pour chaque mesure de forme afin de visualiser les motifs de forme dans chaque image. Les définitions et abréviations des métriques de forme se trouvent dans le tableau 1.
Parfois, les supports plats RPE peuvent contenir des morceaux résiduels de rétine qui n’ont pas été enlevés proprement, en particulier autour du nerf optique. La coloration phalloïdine de l’échantillon entraîne un signal fort provenant de la rétine, ce qui peut causer des problèmes de segmentation de la frontière cellulaire RPE. Certaines tuiles apparaîtront complètement noires, tandis que les tuiles environnantes afficheront une segmentation normale. D’autres objets lumineux qui peuvent être présents dans l’image provoqueront également la génération de tuiles noires (Figure 3). Dans ces cas, choisir l’une des options de filtrage (Faible, Régulier, Fort) disponibles dans le menu déroulant Filtre Arti empêchera la formation de tuiles noires.
REShAPE prend des images en niveaux de gris 8 bits ou 16 bits en entrée, mais pas des images RVB. L’utilisation d’images RVB pour l’analyse REShAPE produira des images binaires entièrement noires. Si cela se produit, la conversion des images RVB en niveaux de gris produira des images binaires correctement segmentées (Figure 4). Dans certains cas où les bordures de l’EPR ne sont pas reconnues correctement, par exemple si la coloration n’est pas optimale ou si l’échantillon est endommagé par une égratignure (figure 5A), de gros amas de cellules peuvent être identifiés comme une seule très grande cellule (figure 5B). Dans ce cas, les objets volumineux peuvent être exclus de l’analyse en réduisant le seuil de taille de cellule (Figure 5C). Pour ce faire, vous pouvez insérer une valeur inférieure dans la zone de texte Taille de cellule supérieure . Cependant, cela entraînera une modification de la portée de la carte thermique. Si un chercheur choisit de le faire, il est également possible de conserver la plage de carte thermique d’origine (Figure 5D) en cochant la case Oui dans la fonction Utiliser les limites manuelles. Par la suite, le chercheur doit cliquer avec le bouton gauche de la souris sur le bouton Définir les limites et insérer les valeurs souhaitées dans les zones de texte pour spécifier les limites manuelles.
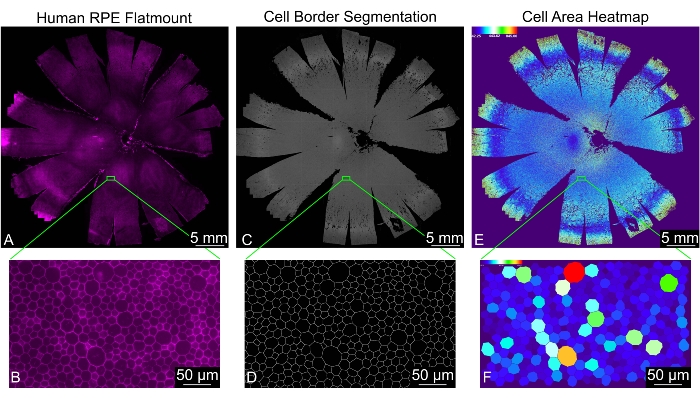
Figure 2 : Analyse morphométrique complète d’une monocouche humaine entière d’EPR. (A) Vue à faible grossissement d’un montage plat humain EPR/choroïde entier (magenta : phalloïdine). (B) Une vue zoomée des cellules RPE colorées à la phalloïdine. (C) segmentation générée par REShAPE des bordures de cellule RPE pour un montage plat RPE/choroïde humain entier et (D) la vue zoomée correspondante. (E) Une carte thermique générée par logiciel illustrant la surface cellulaire des cellules individuelles de l’EPR dans l’ensemble du montage plat humain. L’échelle thermique dans le coin supérieur gauche indique la plage de valeurs utilisées. (F) La vue agrandie correspondante montrant les cellules RPE individuelles colorées par zone. Barres d’échelle = (B,D,F) 50 μm, (A,C,E) 5 mm. Abréviation : EPR = épithélium pigmentaire rétinien. Veuillez cliquer ici pour voir une version agrandie de cette figure.
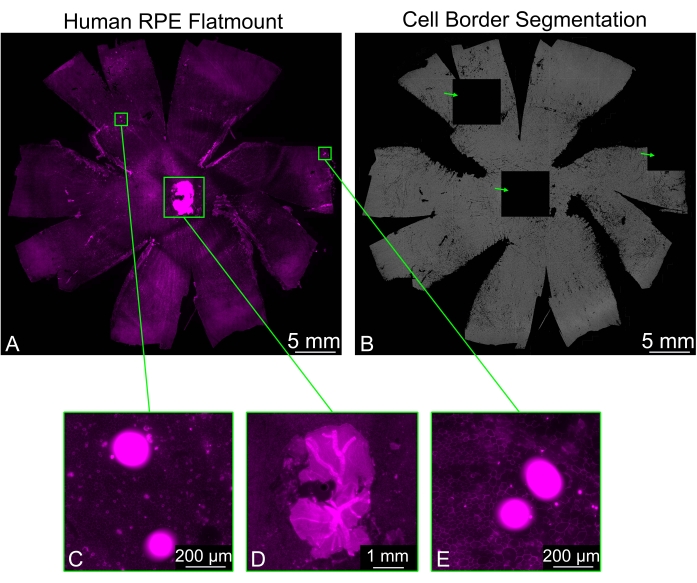
Figure 3 : Filtrage des artefacts lumineux. (A) Un montage plat humain RPE coloré pour les bordures cellulaires (magenta: phalloïdine) peut présenter des zones lumineuses (rectangles verts) qui interfèrent avec la segmentation. (B) La segmentation de la frontière cellulaire RPE de l’ensemble du montage plat contient trois tuiles complètement noires (flèches vertes) correspondant à des régions lumineuses de fluorescence. (C,E) Deux des tuiles noires correspondent à des zones contenant des points lumineux, qui sont peut-être des débris. (D) L’une des tuiles noires a été générée par un morceau de rétine neurale autour du nerf optique qui n’a pas été correctement retiré. Les morceaux de la rétine neurale sont considérablement plus brillants que la couche RPE et entravent la segmentation cellulaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Image d’entrée requise. Les cellules RPE colorées pour les bordures de cellules ont été enregistrées sous forme d’images (A) RVB ou (B) en niveaux de gris 16 bits pour l’analyse REShAPE. (C) La sortie de l’analyse de l’image RBG est une image binaire noire, (D) tandis que l’analyse de l’image en niveaux de gris produit une binaire correctement segmentée des bordures de cellule. REShAPE ne peut analyser que des images en niveaux de gris 8 bits ou 16 bits. Barres d’échelle = 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Résultats sous-optimaux. (A) Image d’une partie de la monocouche RPE où les cellules colorées à la phalloïdine ont été accidentellement rayées. (B) Une carte thermique des cellules RPE colorées par la dimension de la surface cellulaire. Un seuil de grande taille de cellule supérieure inclut les objets volumineux dans l’analyse. (C) Une carte thermique de la surface de la cellule dans laquelle un seuil de taille de cellule supérieure plus petit a été choisi pour exclure les gros objets de l’analyse. (D) Une carte thermique de la surface de la cellule dans laquelle un seuil de taille de cellule supérieur plus petit a été choisi et des limites manuelles ont été fixées pour maintenir la plage de carte thermique utilisée à l’origine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Paramètres REShAPE. Le tableau indique la définition de chaque paramètre et les abréviations utilisées dans les feuilles de calcul brutes (fichiers « _Data.csv ») et pour les cartes thermiques. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
La séparation cohérente et efficace de l’EPR humaine et des rétines peut être réalisée à l’aide de ce protocole. Cette méthode permet d’étudier les différences régionales de forme de l’EPR sur l’ensemble de la rétine humaine5. Une étape cruciale du protocole est la séparation physique de l’EPR et de la rétine. Si les deux tissus ne sont pas complètement détachés dans certaines zones, il faut soulever doucement la rétine, en veillant à ne pas casser les tissus. L’analyse REShAPE de grands supports plats peut nécessiter l’utilisation de systèmes dotés de ressources RAM considérables. Dans ce cas, le réassemblage de l’image entière peut être désactivé pour permettre au logiciel de terminer l’analyse avec succès malgré un manque de ressources de traitement.
La principale limitation de l’utilisation de REShAPE pour segmenter les montages plats humains en EPR est que l’algorithme d’IA a été principalement formé sur des images d’EPR induites dérivées de cellules souches pluripotentes. En conséquence, la segmentation des supports plats RPE humains est moins précise. Les cellules EPR de donneurs âgés contiennent une grande quantité de lipofuscine7, et le large spectre de son autofluorescence interfère avec la segmentation des frontières cellulaires. À l’avenir, davantage d’images de montages plats RPE seront utilisées pour améliorer la segmentation des frontières cellulaires dans ce type d’échantillon. Malgré cette limitation, REShAPE a été spécialement formé pour reconnaître et segmenter les frontières des cellules RPE et fonctionne mieux que d’autres méthodes existantes, telles que la segmentation Voronoi8 et CellProfiler9 des cellules RPE.
De plus, par rapport à la segmentation manuelle 10, REShAPE offre l’avantage d’analyser rapidement les grandes images (~130 000 pixels x130 000 pixels ont été testés). En conclusion, cette méthode de dissection est hautement reproductible et peut être facilement étendue à d’autres modèles animaux. En outre, le logiciel peut être utilisé pour étudier la forme de l’EPR dans des montures plates oculaires ou dans des modèles de culture cellulaire pour examiner l’effet de certains traitements. Enfin, la polyvalence de REShAPE le rend largement applicable à l’analyse d’autres types de cellules épithéliales.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions le noyau d’histologie du National Eye Institute (NEI) pour l’utilisation du Zeiss Axio Scan.Z1. Nous remercions également les donateurs, leurs familles, le Réseau Advancing Sight et le Lions Eye Institute pour leur générosité. Ce travail a été financé par les fonds du PRI NEI (numéro de subvention ZIA EY000533-04).
matériels
| Name | Company | Catalog Number | Comments |
| Biopsy punch 1.5 mm | Acuderm Inc. | P1525 | |
| Bovine albumin | MP Biomedicals | 160069 | |
| Coverglass 50 x 75 mm, #1.5 thickness | Brain Research Laboratories | 5075-1.5D | |
| Curved spatula | Katena | K3-6600 | |
| D-Mannitol | Sigma | M9546 | |
| DPBS 1x with Ca2+ and Mg2+ | Gibco | 14040-133 | |
| Fine Scissors | Fine Science Tools | 14558-11 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Forceps - Dumont #5 | Fine Science Tools | 11252-23 | |
| Microscope slides 50 x 75 x 1.2 mm | Brain Research Laboratories | 5075 | |
| Needles 21 G x 1-1/2" hypodermic | Becton Dickinson (BD) | 305167 | |
| Needles 27 G x 1-1/4" hypodermic | Becton Dickinson (BD) | 305136 | |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Petri dish 100 mm | Corning | 430167 | |
| Phalloidin-iFluor 647 | Abcam | ab176759 | |
| Razor blades | PAL (Personna) | 62-0177 | |
| Round bottom tubes 50 mL | Newegg | 9SIA4SR9M88854 | |
| Silicon Elastomer Kit | Dow Corning Corporation | 4019862 | |
| Square weighing boat (81 mm x 81 mm x 25 mm) | Sigma | W2876 | |
| Surgical Vitrectomy System | BD Visitrec | 585100 | optional |
| Syringe 1 mL | Becton Dickinson (BD) | 309659 | |
| Triton X-100 | Sigma | T9284 | |
| TrueBlack | Biotium | 23007 | autofluorescence quencher |
| Tween 20 | Affymetrix | 20605 | |
| Vannas Spring Scissors - 3 mm cutting edge | Fine Science Tools | 15000-10 |
Références
- Endo, E. G., Yao, X. Y., Marmor, M. F. Pigment adherence as a measure of retinal adhesion: Dependence on temperature. Investigative Ophthalmology and Visual Science. 29 (9), 1390-1396 (1988).
- Marmor, M. F. Retinal detachment from hyperosmotic intravitreal injection. Investigative Ophthalmology and Visual Science. 18 (12), 1237-1244 (1979).
- Marmor, M. F., Martin, L. J., Tharpe, S. Osmotically induced retinal detachment in the rabbit and primate. Electron microscopy of the pigment epithelium. Investigative Ophthalmology and Visual Science. 19 (9), 1016-1029 (1980).
- Ban, Y., Rizzolo, L. A culture model of development reveals multiple properties of RPE tight junctions. Molecular Vision. 3, 18(1997).
- Ortolan, D., et al. Single-cell-resolution map of human retinal pigment epithelium helps discover subpopulations with differential disease sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 119 (19), 2117553119(2022).
- Schaub, N. J., et al. Deep learning predicts function of live retinal pigment epithelium from quantitative microscopy. Journal of Clinical Investigation. 130 (2), 1010-1023 (2020).
- Beatty, S., Koh, H. -H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Liu, Z., Kocaoglu, O. P., Miller, D. T. 3D imaging of retinal pigment epithelial cells in the living human retina. Investigative Ophthalmology and Visual Science. 57 (9), 533-543 (2016).
- Bhatia, S. K., et al. Analysis of RPE morphometry in human eyes. Molecular Vision. 22, 898-916 (2016).
- vonder Emde, L., et al. Histologic cell shape descriptors for the retinal pigment epithelium in age-related macular degeneration: A comparison to unaffected eyes. Translational Vision Science & Technology. 11 (8), 19(2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon