Method Article
Geração eficiente e consistente de epitélio/coroide de pigmento da retina a partir de olhos humanos para análise histológica
Neste Artigo
Resumo
Descrevemos um método para separar eficientemente o epitélio pigmentar da retina (EPR) da retina em olhos humanos e gerar montagens planas inteiras de EPR/coroide para análises histológicas e morfométricas da EPR.
Resumo
O epitélio pigmentar da retina (EPR) e a retina são tecidos funcional e estruturalmente conectados que trabalham juntos para regular a percepção da luz e a visão. As proteínas na superfície apical do EPR estão fortemente associadas a proteínas na superfície do segmento externo do fotorreceptor, dificultando a separação consistente do EPR dos fotorreceptores/retina. Desenvolvemos um método para separar eficientemente a retina do PSE dos olhos humanos para gerar montagens planas completas de PSE/coroide e retina para análise celular separada dos fotorreceptores e células RPE. Uma injeção intravítrea de uma solução de alta osmolaridade de D-manitol, um açúcar não transportado pelo PSE, induziu a separação do RPE e da retina em toda a câmara posterior sem causar danos às junções celulares do RPE. Não foram observados adesivos de PSE ligados à retina. A marcação faloidina da actina mostrou preservação da forma do PSE e permitiu a análise morfométrica de todo o epitélio. Um software baseado em inteligência artificial (IA) foi desenvolvido para reconhecer e segmentar com precisão as bordas celulares RPE e quantificar 30 métricas de forma diferentes. Este método de dissecção é altamente reprodutível e pode ser facilmente estendido a outros modelos animais.
Introdução
O epitélio pigmentar da retina (EPR) e a retina neural estão fortemente interligados entre si devido à forte dependência fisiológica dos fotorreceptores no EPR. Durante a dissecção, a separação mecânica da retina neural do EPR causa o rompimento das células do EPR, com as porções apicais do EPR permanecendo ligadas aos segmentos externos dos fotorreceptores da retina. A extensão da adesão RPE-retiniana é tão grande que a quantidade de pigmento remanescente na retina após a separação é usada para quantificar a força da adesão retiniana1. Especificamente, as junções apertadas do PSE e a estrutura de actina que as conecta, que estão localizadas no lado apical, se rompem durante a separação mecânica. Portanto, a coloração de montagens planas de RPE para bordas celulares resulta em uma monocamada irregular na qual muitas células têm bordas ausentes. Este efeito é exacerbado quando o tecido é fixado com paraformaldeído (PFA) antes da dissecção, à medida que as proteínas se tornam reticuladas.
Estudos sobre a administração intravítrea de fármacos têm demonstrado que injeções de soluções hiperosmóticas na câmara posterior induzem o descolamento de retina 2,3. Nesses experimentos, 50 μL de diferentes soluções, variando de 1.000 mOsm a 2.400 mOsm, injetadas no vítreo médio, causaram descolamento de retina em poucos minutos. Notavelmente, mesmo após longas exposições a soluções de alta osmolaridade, as junções apertadas do PSE apareceram intactas nas imagens microscópicas eletrônicas de transmissão de olhos de coelho e macaco3. Seguindo uma estratégia semelhante, injetamos no vítreo médio uma solução hiperosmótica de D-manitol para induzir um descolamento de retina eficiente antes de realizar a dissecção do PSE. Como o D-manitol não é transportado pelo PSE4, uma alta concentração intravítrea é mantida, gerando um gradiente osmótico. A separação eficiente do PSE e da retina em toda a câmara posterior garante a preservação das junções celulares do PSE e permite o estudo da morfometria do RPE em toda a montagem plana. Além disso, desenvolvemos um software baseado em inteligência artificial (IA) que reconhece e segmenta bordas celulares de RPE marcadas fluorescentemente, quantifica 30 métricas de formas diferentes e produz mapas de calor de cada métrica para visualização 5,6.
Protocolo
Globos humanos cadáveres foram obtidos da Advanced Sight Network (Birmingham, AL). O trabalho realizado em tecido de cadáveres é isento pelo Comitê de Ética em Pesquisa do NIH Institutional Review Board.
1. Envio do globo ocular
- Após a enucleação, envie globos oculares frescos em um contêiner cheio de DPBS 1x gelado com Ca 2+ e Mg2+.
NOTA: É melhor dissecar o olho dentro de 24 h após a enucleação. A morfologia do PSE não é alterada durante esta janela de tempo.
2. Preparação do molde de silicone
- Corte o fundo de 20 mm de um tubo de fundo redondo de 25 mm de diâmetro. Coloque-o na base de um barco de pesagem quadrado (81 mm x 81 mm x 25 mm).
- Misture os dois componentes do Kit Elastômero de Silício na proporção de 10:1, prestando atenção para não prender o ar. Despeje a mistura no barco de pesagem contendo o pedaço esférico do tubo de fundo redondo.
- Cure o silicone à temperatura ambiente durante a noite. Remova o barco de pesagem e o tubo de fundo redondo do molde de silicone curado.
3. Dissecção de PSE
- Limpe a esclera do globo ocular fresco dos músculos e do tecido conjuntivo. Prenda o olho ao molde de silicone usando agulhas 27 G rosqueadas através da esclera. Preencha a cavidade do molde com DPBS 1x com Ca 2+ e Mg2+.
NOTA: Preste atenção para não violar a câmara dos olhos. As agulhas só devem passar pela esclera. - Use uma seringa de 1 mL e uma agulha de 21 G para injetar ~400 μL de solução de D-manitol de 1.700 mOsm no vítreo. Insira a agulha através da pars plana para evitar perfurar a câmara anterior do olho. Deixe o olho à temperatura ambiente por ~45 min.
- Abra a câmara anterior ao nível da pars plana usando um par de tesouras finas e pinças. Encha a câmara ocular posterior com DPBS 1x com Ca 2+ e Mg2+. Sob o estereomicroscópio, localize a mácula (a mancha amarela na retina).
- Se um cortador vítreo cirúrgico estiver disponível, remova o vítreo e substitua-o por DPBS 1x com Ca 2+ e Mg2+. Alternativamente, tente levantar o vítreo com fórceps e corte-o com uma tesoura fina.
- Prestando atenção à preservação da região macular, corte o olho em quadrantes: nasal, temporal, superior e inferior. Remova as agulhas se elas estiverem no caminho.
- Transfira a câmara posterior do olho com borboleta para uma placa de Petri de 100 mm contendo DPBS 1x com Ca 2+ e Mg2+. Antes de remover a retina, marque a pétala que contém a mácula fazendo um pequeno corte (em forma de V) na margem ciliar. Levante e corte todo o vítreo que se deposita na retina.
- Levante suavemente a retina de vários lados com uma espátula curva ou um par de pinças para verificar se a retina se descolou do PSE e para deixar algum fluido circular entre as duas camadas.
NOTA: A retina ainda estará ligada na periferia (margens ciliares) e no nervo óptico. - Corte a retina das margens ciliares em todas as pétalas, garantindo não arranhar o PSE. Coloque o tecido em 4% de PFA e incube por ~1 h. Lave 3x com DPBS 1x com Ca 2+ e Mg2+. Transfira o tecido para um recipiente cheio de DPBS 1x com Ca 2+ e Mg2+ e guarde-o a 4 °C.
NOTA: Neste ponto, a retina só está ligada no nervo óptico. Este é um ponto de pausa neste experimento. - Transfira a amostra para uma placa de Petri de 100 mm contendo DPBS 1x com Ca 2+ e Mg2+. Soque a cabeça do nervo óptico com um soco de biópsia de 1,5 mm e colete a retina. Armazenar a retina neural em DPBS 1x com Ca 2+ e Mg2+ a 4 °C.
NOTA: Antes de perfurar a cabeça do nervo óptico, certifique-se de cortar o nervo óptico, tanto quanto possível no lado escleral. Isso aumentará a precisão do soco. Perfurar o nervo óptico depois que a montagem plana é fixada em 4% de PFA reduz o dano às células RPE localizadas ao redor do nervo óptico. - Remova a esclera do PSE/coroide levantando suavemente a camada RPE/coroide da periferia e cortando os vasos coroidais e o tecido conjuntivo que estão entre a esclera e o PSE com um par de tesouras de mola Vannas. Uma vez que o PSE/coroide esteja completamente separado da esclera, colete a camada RPE/coroide. Transfira o tecido para um recipiente cheio de DPBS 1x com Ca 2+ e Mg2+ e armazene-o a 4°C.
Observação : neste momento, o experimento pode ser pausado.
4. Coloração
- Transfira o PSE/coroide para um poço de uma placa de 6 poços. Bloquear e permeabilizar a amostra em DPBS 1x com Ca 2+ e Mg 2+ com albumina sérica bovina (ASC) a 1%, Tween20 a 0,5% e Triton X-100 a 0,5% por 1 h à temperatura ambiente.
- Incubar a amostra com faloidina conjugada com fluoróforo 647 em diluição 1:250 em DPBS 1x com Ca 2+ e Mg 2+ com 1% de BSA, 0,5% de Tween20 e 0,5% de Triton X-100 por 1 h à temperatura ambiente. Lave 3x em DPBS 1x com Ca 2+ e Mg2+.
- Transfira a amostra de RPE/coroide para uma lâmina de vidro de 50 mm x 75 mm e achate-a. Corte cada "pétala" em duas para tornar a amostra mais plana. Preste atenção à mácula. Desenhe um contorno do flat mount com uma caneta hidrofóbica.
- Para extinguir a autofluorescência da lipofuscina, adicionar 500 μL da solução de quencher de autofluorescência diluída a 1:20 em etanol a 70% e incubar à temperatura ambiente por 2 min.
- Lave bem (pelo menos 3x) em DPBS 1x com Ca 2+ e Mg2+. Remova o DPBS e adicione o meio de montagem. Coloque um copo de cobertura no suporte plano e sele com esmalte.
- Fotografe o plano com um microscópio de fluorescência (de preferência usando uma objetiva de 10x ou 20x).
5. Análise REShAPE
NOTA: Como o algoritmo baseado em IA REShAPE foi treinado em imagens de 10x e 20x, é, portanto, altamente recomendável usar um objetivo de 10x ou 20x ao fazer imagens. Caso contrário, as imagens precisarão ser redimensionadas de acordo.
- Se as imagens forem adquiridas com mais de um canal fluorescente, isole o canal usado para adquirir as bordas celulares. Exporte as imagens como arquivos TIF em escala de cinza de 16 bits.
Observação : imagens adquiridas com a extensão de arquivo .czi não precisam ser exportadas como arquivos TIF, mas o canal que contém as bordas de célula ainda precisa ser isolado. - Instale o software em plataformas Windows x64 ou Linux (Centos 7).
NOTA: O software e as instruções de instalação podem ser encontrados em https://github.com/nih-nei/REShAPE. - Abra o software (Figura 1).
- Na guia Diretórios , selecione as pastas de entrada e saída. No diretório de entrada, selecione a pasta que contém as imagens. No diretório de saída, deixe o software alterar automaticamente o caminho para " diretório de entrada/processado". Como alternativa, altere o diretório de saída manualmente.
NOTA: O software irá iterar através de todas as imagens contidas na pasta de entrada. - Na guia Opções de Segmentação NN , escolha as seguintes configurações para segmentação de imagem:
- Tamanho do lote: digite um valor de 20 na caixa de texto como um bom ponto de partida, mas diminua-o se o sistema ficar sem unidades de processamento gráfico (GPUs).
Observação : A imagem original é dividida em blocos menores para processamento. O tamanho do lote especifica quantos blocos podem ser processados de uma só vez. - Usar GPU?: Se o sistema não tiver recursos de GPU suficientes, marque a caixa Não.
- Escala interna: Usando o menu suspenso, altere o dimensionamento interno de Nenhum nos casos em que as imagens não foram adquiridas com um objetivo de 10x ou 20x. Por exemplo, reduza as imagens em 40x para metade do tamanho usando o botão 1/2 . As opções de reescalonamento disponíveis no menu suspenso são as seguintes: 5x, 2x, Nenhum, 1/2, 1/5.
NOTA: O aprendizado de máquina foi treinado com imagens de 10x e 20x. Ele pode não reconhecer bordas celulares de imagens tiradas em outras ampliações. Nesse caso, as imagens binárias aparecerão completamente pretas. - Filtro Arti: Use a aba do filtro de artefatos para remover partículas muito brilhantes (detritos ou pequenas partes da retina ou tecido conjuntivo ao redor do nervo óptico) que podem estar presentes na imagem e podem interferir na segmentação da borda celular. Nenhum filtro é usado por padrão. As opções de filtro artificial no menu suspenso são as seguintes: Regular, Forte e Fraco.
- Tamanho do lote: digite um valor de 20 na caixa de texto como um bom ponto de partida, mas diminua-o se o sistema ficar sem unidades de processamento gráfico (GPUs).
- Na guia Opção de Imagem Lado a Lado, insira um valor na caixa de texto para especificar a quantidade de sobreposição entre os blocos de imagem ajustando o parâmetro Bloco de Blocos Por . Uma sobreposição de 100 pixels geralmente funciona bem.
- Na guia Opções de Gráfico de Saída , ajuste as configurações para geração de mapa de calor:
- Reconstruct Tiled Images?: se o sistema não tiver recursos suficientes para a análise de imagens grandes, marque a caixa Não para permitir que o software conclua a análise salvando blocos individuais sem reconstruir a imagem inteira.
- Criar imagens coloridas: Para gerar mapas de calor, selecione Todos no menu suspenso.
- Para Colorir, escolha qualquer uma das diferentes paletas de cores disponíveis no menu suspenso: Térmica, Verde, Mpl-magma, Fase, Fogo, Jato, Quente Ciano.
- Para Formato de Imagem, salve as imagens como TIF ou PNG selecionando uma das opções na guia.
- Com relação ao recurso Usar limites manuais? , marque a caixa Não para permitir que o software use os valores mínimo e máximo detectados em cada imagem. Marque a caixa Sim para ajustar manualmente o intervalo de valores para cada mapa de calor métrico de forma e clique no botão Definir limites para escolher intervalos para os parâmetros individuais, inserindo valores nas caixas de texto. Depois de alterar os valores de interesse, clique em Salvar. Clique em Carregar padrões para redefinir todos os limites.
NOTA: Use limites manuais se os mapas de calor de várias imagens precisarem ser comparados, como ao comparar os efeitos de diferentes compostos na forma da célula. Dessa forma, o mesmo intervalo de valores é usado. O conjunto de valores a serem inseridos manualmente varia dependendo do tipo de amostra. Recomenda-se executar algumas iterações para escolher o intervalo ideal.
- Na guia Restrições de Tamanho da Célula para Análise , selecione um limite de tamanho de célula para a análise:
- Em Tamanho Inferior da Célula, insira na caixa de texto o tamanho da menor célula a ser incluída na análise.
- Em Tamanho da Célula Superior, insira na caixa de texto o tamanho da maior célula a ser incluída na análise.
NOTA: A unidade de tamanho da célula muda de pixels para micrômetros ao quadrado, dependendo da opção escolhida na guia Conversão Automatizada de Unidades .
- Na guia Conversão Automatizada de Unidades , escolha a unidade preferida para a análise:
- Em Converter pixels em unidades reais?, marque a caixa Não para executar a análise em unidades de pixels. Marque a caixa Sim para executar a análise em micrômetros.
- Em Comprimento da barra de escala (Pixels), insira o valor do pixel na caixa de texto.
- Em Comprimento da barra de escala (Mícrons), insira na caixa de texto a distância correspondente em micrômetros.
- Para iniciar a análise, pressione Go For It.
NOTA: O software também pode medir a viabilidade celular quando as células são coradas com 4',6-diamidino-2-fenilindol (DAPI) e iodeto de propídio (guia Opções CZI ), mas isso não se aplica a montagens planas de RPE.
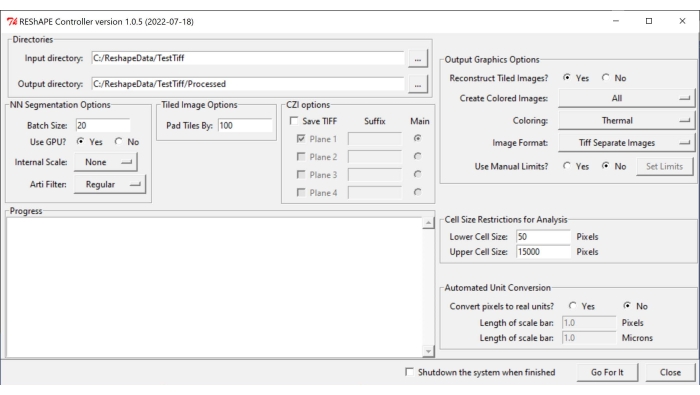
Figura 1: Interface gráfica do usuário do REShAPE. A GUI tem diferentes guias para selecionar os diretórios de trabalho (guia Diretórios ), modificar as opções de segmentação (guias Opções de Segmentação NN e Opções de Imagem em Bloco ), especificar os parâmetros para análise (guias Restrições de Tamanho da Célula para Análise e Conversão Automatizada de Unidades ) e para geração de mapa de calor (guia Opções de Gráficos de Saída ). Abreviação: GUI = interface gráfica do usuário. Por favor, clique aqui para ver uma versão maior desta figura.
Resultados
Esse protocolo resulta em uma imagem de plano único de uma montagem plana, onde a localização da célula e as métricas de 30 formas são medidas para cada célula RPE corretamente identificada (Figura 2). Uma pasta chamada "Processado" é gerada automaticamente dentro do diretório de entrada. Essa pasta contém quatro subdiretórios, chamados "Análise", "Codificado por cores", "Arquivos combinados" e "Imagens segmentadas", e alguns arquivos temporários gerados durante a análise. A pasta "Arquivos Combinados" contém uma planilha com todas as medidas de forma e uma planilha com as frequências das contagens de vizinhos de célula de todos os arquivos combinados. A pasta "Análise" contém uma planilha com todas as medidas de forma e uma planilha com as frequências das contagens do vizinho de célula para cada imagem separadamente. O diretório "Imagens segmentadas" contém as máscaras binárias finais das bordas da célula RPE; ele pode ser usado para avaliar a qualidade da segmentação. O diretório "Color Coded" contém mapas de calor para cada medição de forma para visualizar os padrões de forma em cada imagem. As definições e abreviaturas da métrica de forma podem ser encontradas na Tabela 1.
Às vezes, os flatmounts de PSE podem conter pedaços residuais de retina que não foram removidos de forma limpa, especialmente ao redor do nervo óptico. A coloração com faloidina da amostra resulta em um forte sinal vindo da retina, e isso pode causar problemas para a segmentação da borda celular do PSE. Alguns azulejos aparecerão completamente pretos, enquanto os azulejos ao redor mostrarão segmentação normal. Outros objetos brilhantes que podem estar presentes na imagem também causarão a geração de azulejos pretos (Figura 3). Nesses casos, escolher uma das opções de filtragem (Fraco, Regular, Forte) disponível no menu suspenso Filtro de ArteArti impedirá a formação de blocos pretos.
O REShAPE usa imagens em escala de cinza de 8 bits ou 16 bits como entrada, mas não imagens RGB. O uso de imagens RGB para a análise REShAPE produzirá imagens binárias inteiramente pretas. Se isso ocorrer, a conversão das imagens RGB em escala de cinza produzirá imagens binárias segmentadas corretamente (Figura 4). Em algumas ocasiões em que as bordas do PSE não são reconhecidas corretamente, por exemplo, se a coloração não for ótima ou se a amostra for danificada por um arranhão (Figura 5A), grandes aglomerados de células podem ser identificados como uma única célula muito grande (Figura 5B). Nesse caso, objetos grandes podem ser excluídos da análise reduzindo o limite de tamanho da célula (Figura 5C). Isso pode ser feito inserindo um valor mais baixo na caixa de texto Tamanho da Célula Superior . No entanto, isso resultará em uma alteração no intervalo do mapa de calor. Se um pesquisador optar por fazer isso, também é possível manter o intervalo original do mapa de calor (Figura 5D) marcando a caixa Sim no recurso Usar limites manuais ?. Posteriormente, o pesquisador deve clicar com o botão esquerdo do mouse no botão Definir Limites e inserir os valores desejados nas caixas de texto para especificar os limites manuais.
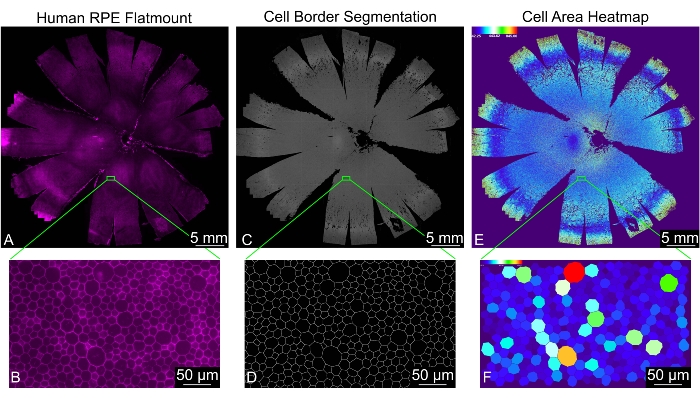
Figura 2: Análise morfométrica completa de uma monocamada inteira de EPR humana. (A) Uma visão de baixa ampliação de um EPR humano inteiro / montagem plana coroide (magenta: faloidina). (B) Uma visão ampliada de células de EPR coradas com faloidina. (C) Segmentação gerada por REShAPE das bordas celulares de RPE para um conjunto plano de RPE/coroide humano e (D) a visualização ampliada correspondente. (E) Um mapa de calor gerado por software ilustrando a área celular das células RPE individuais em todo o flat mount humano. A escala térmica no canto superior esquerdo mostra o intervalo de valores usados. (F) A visualização ampliada correspondente mostrando células RPE individuais coloridas por área. Barras de escala = (B,D,F) 50 μm, (A,C,E) 5 mm. Abreviação: RPE = epitélio pigmentar da retina. Por favor, clique aqui para ver uma versão maior desta figura.
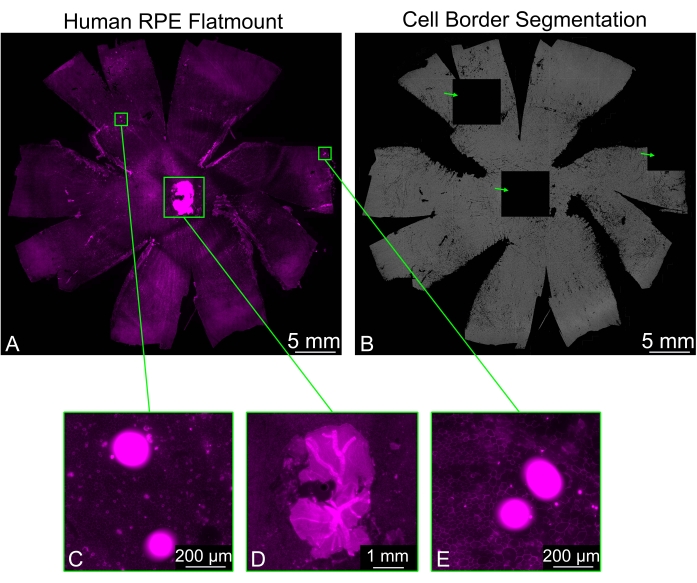
Figura 3: Filtragem de artefatos brilhantes. (A) Um PPE humano de montagem plana corado para bordas celulares (magenta: faloidina) pode apresentar áreas brilhantes (retângulos verdes) que interferem na segmentação. (B) A segmentação da borda da célula RPE de todo o flat mount contém três telhas completamente pretas (setas verdes) correspondentes a regiões brilhantes de fluorescência. (C,E) Dois dos azulejos pretos correspondem a áreas contendo pontos brilhantes, que são possivelmente alguns detritos. (D) Uma das telhas pretas foi gerada por um pedaço de retina neural ao redor do nervo óptico que não foi removido corretamente. Os pedaços de retina neural são consideravelmente mais brilhantes do que a camada RPE e dificultam a segmentação celular. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 4: Requisito da imagem de entrada. As células RPE coradas para bordas celulares foram salvas como (A) RGB ou como (B) imagens em escala de cinza de 16 bits para análise REShAPE. (C) A saída da análise de imagem RBG é uma imagem binária preta, (D) enquanto a análise da imagem em escala de cinza produz uma binária corretamente segmentada das bordas celulares. O REShAPE só pode analisar imagens em escala de cinza de 8 bits ou 16 bits. Barras de escala = 50 μm. Clique aqui para ver uma versão maior desta figura.

Figura 5: Resultados subótimos . (A) Uma imagem de uma porção da monocamada de EPR onde as células coradas com faloidina foram acidentalmente riscadas. (B) Um mapa de calor de células RPE coloridas pela dimensão da área celular. Um grande limite de tamanho de célula superior inclui objetos grandes na análise. (C) Um mapa de calor da área celular no qual um limite menor de tamanho de célula superior foi escolhido para excluir objetos grandes da análise. (D) Um mapa de calor de área celular no qual um limite de tamanho de célula superior menor foi escolhido e limites manuais foram definidos para manter a faixa de mapa de calor usada originalmente. Por favor, clique aqui para ver uma versão maior desta figura.
Tabela 1: Parâmetros REShAPE. A tabela informa a definição de cada parâmetro e as abreviaturas utilizadas nas planilhas brutas (arquivos "_Data.csv") e para os heatmaps. Por favor, clique aqui para baixar esta Tabela.
Discussão
A separação consistente e eficiente de PSE humano e retinas pode ser alcançada usando este protocolo. Esse método permite o estudo das diferenças regionais na forma da EPR em retinas humanas inteiras5. Um passo crucial no protocolo é a separação física do EPR e da retina. Se os dois tecidos não estiverem completamente destacados em algumas áreas, deve-se levantar suavemente a retina, garantindo não quebrar os tecidos. A análise REShAPE de grandes montagens planas pode exigir o uso de sistemas com recursos de RAM consideráveis. Nesse caso, a remontagem de toda a imagem pode ser desativada para permitir que o software conclua com êxito a análise, apesar da falta de recursos de processamento.
A principal limitação do uso do REShAPE para segmentar montagens planas de RPE humanas é que o algoritmo de IA foi treinado principalmente em imagens de PSE derivadas de células-tronco pluripotentes induzidas. Como consequência, a segmentação de montagens planas de RPE humano é menos precisa. As células PSE de doadores idosos contêm uma grande quantidade de lipofuscina7, e o amplo espectro de sua autofluorescência interfere na segmentação da borda celular. No futuro, mais imagens de montagens planas de RPE serão usadas para melhorar a segmentação da borda celular nesse tipo de amostra. Apesar dessa limitação, o REShAPE foi especificamente treinado para reconhecer e segmentar as bordas celulares do PSE e tem um desempenho melhor do que outros métodos existentes, como a segmentação de células RPE Voronoi8 e CellProfiler9 .
Além disso, em comparação com a segmentação manual 10, o REShAPE oferece a vantagem de analisar imagens grandes rapidamente (~ 130.000 pixels x130.000 pixels foram testados). Em conclusão, este método de dissecção é altamente reprodutível e pode ser facilmente estendido a outros modelos animais. Além disso, o software pode ser usado para estudar a forma de PSE em montagens planas oculares ou em modelos de cultura celular para examinar o efeito de certos tratamentos. Finalmente, a versatilidade do REShAPE o torna amplamente aplicável para a análise de outros tipos de células epiteliais.
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Agradecemos ao núcleo de histologia do National Eye Institute (NEI) pelo uso do Zeiss Axio Scan.Z1. Também agradecemos aos doadores, suas famílias, a Advancing Sight Network e o Lions Eye Institute por sua generosidade. Este trabalho foi apoiado por fundos NEI IRP (número de subvenção ZIA EY000533-04).
Materiais
| Name | Company | Catalog Number | Comments |
| Biopsy punch 1.5 mm | Acuderm Inc. | P1525 | |
| Bovine albumin | MP Biomedicals | 160069 | |
| Coverglass 50 x 75 mm, #1.5 thickness | Brain Research Laboratories | 5075-1.5D | |
| Curved spatula | Katena | K3-6600 | |
| D-Mannitol | Sigma | M9546 | |
| DPBS 1x with Ca2+ and Mg2+ | Gibco | 14040-133 | |
| Fine Scissors | Fine Science Tools | 14558-11 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Forceps - Dumont #5 | Fine Science Tools | 11252-23 | |
| Microscope slides 50 x 75 x 1.2 mm | Brain Research Laboratories | 5075 | |
| Needles 21 G x 1-1/2" hypodermic | Becton Dickinson (BD) | 305167 | |
| Needles 27 G x 1-1/4" hypodermic | Becton Dickinson (BD) | 305136 | |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Petri dish 100 mm | Corning | 430167 | |
| Phalloidin-iFluor 647 | Abcam | ab176759 | |
| Razor blades | PAL (Personna) | 62-0177 | |
| Round bottom tubes 50 mL | Newegg | 9SIA4SR9M88854 | |
| Silicon Elastomer Kit | Dow Corning Corporation | 4019862 | |
| Square weighing boat (81 mm x 81 mm x 25 mm) | Sigma | W2876 | |
| Surgical Vitrectomy System | BD Visitrec | 585100 | optional |
| Syringe 1 mL | Becton Dickinson (BD) | 309659 | |
| Triton X-100 | Sigma | T9284 | |
| TrueBlack | Biotium | 23007 | autofluorescence quencher |
| Tween 20 | Affymetrix | 20605 | |
| Vannas Spring Scissors - 3 mm cutting edge | Fine Science Tools | 15000-10 |
Referências
- Endo, E. G., Yao, X. Y., Marmor, M. F. Pigment adherence as a measure of retinal adhesion: Dependence on temperature. Investigative Ophthalmology and Visual Science. 29 (9), 1390-1396 (1988).
- Marmor, M. F. Retinal detachment from hyperosmotic intravitreal injection. Investigative Ophthalmology and Visual Science. 18 (12), 1237-1244 (1979).
- Marmor, M. F., Martin, L. J., Tharpe, S. Osmotically induced retinal detachment in the rabbit and primate. Electron microscopy of the pigment epithelium. Investigative Ophthalmology and Visual Science. 19 (9), 1016-1029 (1980).
- Ban, Y., Rizzolo, L. A culture model of development reveals multiple properties of RPE tight junctions. Molecular Vision. 3, 18(1997).
- Ortolan, D., et al. Single-cell-resolution map of human retinal pigment epithelium helps discover subpopulations with differential disease sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 119 (19), 2117553119(2022).
- Schaub, N. J., et al. Deep learning predicts function of live retinal pigment epithelium from quantitative microscopy. Journal of Clinical Investigation. 130 (2), 1010-1023 (2020).
- Beatty, S., Koh, H. -H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Liu, Z., Kocaoglu, O. P., Miller, D. T. 3D imaging of retinal pigment epithelial cells in the living human retina. Investigative Ophthalmology and Visual Science. 57 (9), 533-543 (2016).
- Bhatia, S. K., et al. Analysis of RPE morphometry in human eyes. Molecular Vision. 22, 898-916 (2016).
- vonder Emde, L., et al. Histologic cell shape descriptors for the retinal pigment epithelium in age-related macular degeneration: A comparison to unaffected eyes. Translational Vision Science & Technology. 11 (8), 19(2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados