Method Article
Generazione efficiente e coerente di epitelio/coroide pigmentato retinico Flatmounts da occhi umani per analisi istologiche
In questo articolo
Riepilogo
Descriviamo un metodo per separare in modo efficiente l'epitelio pigmentato retinico (RPE) dalla retina negli occhi umani e generare interi supporti piatti RPE / coroidi per analisi istologiche e morfometriche dell'RPE.
Abstract
L'epitelio pigmentato retinico (RPE) e la retina sono tessuti funzionalmente e strutturalmente collegati che lavorano insieme per regolare la percezione della luce e la visione. Le proteine sulla superficie apicale dell'RPE sono strettamente associate alle proteine sulla superficie del segmento esterno del fotorecettore, rendendo difficile separare costantemente l'RPE dai fotorecettori / retina. Abbiamo sviluppato un metodo per separare in modo efficiente la retina dall'RPE degli occhi umani per generare RPE / coroide e retina completi flatmount per l'analisi cellulare separata dei fotorecettori e delle cellule RPE. Un'iniezione intravitreale di una soluzione ad alta osmolarità di D-mannitolo, uno zucchero non trasportato dall'RPE, ha indotto la separazione dell'RPE e della retina attraverso l'intera camera posteriore senza causare danni alle giunzioni cellulari RPE. Non sono stati osservati cerotti RPE attaccati alla retina. La marcatura con falloidina dell'actina ha mostrato la conservazione della forma RPE e ha permesso l'analisi morfometrica dell'intero epitelio. È stato sviluppato un software basato sull'intelligenza artificiale (AI) per riconoscere e segmentare con precisione i bordi delle celle RPE e quantificare 30 diverse metriche di forma. Questo metodo di dissezione è altamente riproducibile e può essere facilmente esteso ad altri modelli animali.
Introduzione
L'epitelio pigmentato retinico (RPE) e la retina neurale sono fortemente interconnessi tra loro a causa della forte dipendenza fisiologica dei fotorecettori dall'RPE. Durante la dissezione, la separazione meccanica della retina neurale dall'RPE provoca la lacerazione delle cellule RPE, con le porzioni apicali dell'RPE che rimangono attaccate ai segmenti esterni dei fotorecettori retinici. L'estensione dell'adesione RPE-retinica è così grande che la quantità di pigmento rimanente sulla retina dopo la separazione viene utilizzata per quantificare la forza dell'adesione retinica1. In particolare, le giunzioni strette RPE e la struttura di actina che le collega, che si trovano sul lato apicale, si staccano durante la separazione meccanica. Pertanto, la colorazione dei supporti piatti RPE per i bordi delle celle si traduce in un monostrato irregolare in cui molte celle hanno bordi mancanti. Questo effetto è esacerbato quando il tessuto viene fissato con paraformaldeide (PFA) prima della dissezione, poiché le proteine diventano reticolate.
Studi sulla somministrazione intravitreale di farmaci hanno dimostrato che le iniezioni di soluzioni iperosmotiche nella camera posteriore inducono il distacco della retina 2,3. In questi esperimenti, 50 μL di soluzioni diverse, che vanno da 1.000 mOsm a 2.400 mOsm, iniettati nel vitreo medio hanno causato il distacco della retina in pochi minuti. In particolare, anche dopo lunghe esposizioni a soluzioni ad alta osmolarità, le giunzioni strette RPE apparivano intatte nelle immagini microscopiche elettroniche di trasmissione di entrambi gli occhi di coniglio e scimmia3. Seguendo una strategia simile, abbiamo iniettato nel vitreo medio una soluzione iperosmotica di D-mannitolo per indurre un distacco di retina efficiente prima di eseguire la dissezione RPE. Poiché il D-mannitolo non viene trasportato dall'RPE4, viene mantenuta un'elevata concentrazione intravitreale, generando un gradiente osmotico. L'efficiente separazione di RPE e retina su tutta la camera posteriore garantisce la conservazione delle giunzioni cellulari RPE e consente lo studio della morfometria RPE sull'intero flatmount. Inoltre, abbiamo sviluppato un software basato sull'intelligenza artificiale (AI) che riconosce e segmenta i bordi delle celle RPE etichettati in modo fluorescente, quantifica 30 diverse metriche di forma e produce mappe di calore di ciascuna metrica per la visualizzazione 5,6.
Protocollo
I globi umani cadaveri sono stati ottenuti dall'Advanced Sight Network (Birmingham, AL). Il lavoro svolto sul tessuto cadavere è esentato dal NIH Institutional Review Board dal comitato etico di ricerca.
1. Spedizione del globo oculare
- Dopo l'enucleazione, spedire globi oculari freschi in un contenitore pieno di DPBS 1x ghiacciato con Ca 2+ e Mg2+.
NOTA: È meglio sezionare l'occhio entro 24 ore dopo l'enucleazione. La morfologia RPE non viene alterata durante questa finestra temporale.
2. Preparazione dello stampo in silicone
- Tagliare il fondo di 20 mm di un tubo a fondo tondo di 25 mm di diametro. Posizionarlo alla base di una pesatrice quadrata (81 mm x 81 mm x 25 mm).
- Miscelare i due componenti del Kit Elastomero di Silicio in rapporto 10:1, facendo attenzione a non intrappolare aria. Versare la miscela nel recipiente di pesatura contenente il pezzo sferico del tubo a fondo tondo.
- Polimerizzare il silicone a temperatura ambiente durante la notte. Rimuovere il battello di pesatura e il tubo a fondo tondo dallo stampo in silicone polimerizzato.
3. Dissezione RPE
- Pulire la sclera del globo dell'occhio fresco dei muscoli e del tessuto connettivo. Fissare l'occhio allo stampo in silicone utilizzando aghi da 27 G infilati attraverso la sclera. Riempire la cavità dello stampo con DPBS 1x con Ca 2+ e Mg2+.
NOTA: prestare attenzione a non violare la camera oculare. Gli aghi dovrebbero passare solo attraverso la sclera. - Utilizzare una siringa da 1 mL e un ago da 21 G per iniettare ~400 μL di soluzione di D-mannitolo 1.700 mOsm nel vitreo. Inserire l'ago attraverso la pars plana per evitare di perforare la camera anteriore dell'occhio. Lasciare l'occhio a temperatura ambiente per ~45 min.
- Tagliare la camera anteriore a livello della pars plana usando un paio di forbici e pinze sottili. Riempire la camera oculare posteriore con DPBS 1x con Ca 2+ e Mg2+. Sotto lo stereomicroscopio, localizzare la macula (la macchia gialla sulla retina).
- Se è disponibile una fresa vitrea chirurgica, rimuovere il vitreo e sostituirlo con DPBS 1x con Ca 2+ e Mg2+. In alternativa, prova a sollevare il vitreo con una pinza e taglialo con le forbici sottili.
- Prestando attenzione a preservare la regione maculare, tagliare l'occhio in quadranti: nasale, temporale, superiore e inferiore. Rimuovere gli aghi se sono di intralcio.
- Trasferire la camera posteriore dell'occhio farfallata in una capsula di Petri da 100 mm contenente DPBS 1x con Ca 2+ e Mg2+. Prima di rimuovere la retina, segnare il petalo che contiene la macula facendo un piccolo taglio (a forma di V) nel margine ciliare. Sollevare e tagliare tutto il vitreo che si trova sulla retina.
- Sollevare delicatamente la retina da più lati con una spatola curva o un paio di pinze per verificare se la retina si è staccata dall'RPE e per far circolare del liquido tra i due strati.
NOTA: La retina sarà ancora attaccata alla periferia (margini ciliari) e al nervo ottico. - Tagliare la retina dai margini ciliari in tutti i petali, assicurandosi di non graffiare l'RPE. Posizionare il tessuto in PFA al 4% e incubare per ~ 1 ora. Lavare 3x con DPBS 1x con Ca 2+ e Mg2+. Trasferire il tessuto in un contenitore riempito con DPBS 1x con Ca 2+ e Mg2+ e conservarlo a 4 °C.
NOTA: A questo punto, la retina è attaccata solo al nervo ottico. Questo è un punto di pausa in questo esperimento. - Trasferire il campione in una capsula di Petri da 100 mm contenente DPBS 1x con Ca 2+ e Mg2+. Perforare la testa del nervo ottico con un pugno bioptico da 1,5 mm e raccogliere la retina. Conservare la retina neurale in DPBS 1x con Ca 2+ e Mg2+ a 4 °C.
NOTA: Prima di perforare la testa del nervo ottico, assicurarsi di tagliare il nervo ottico il più possibile sul lato sclerale. Ciò aumenterà la precisione del pugno. La perforazione del nervo ottico dopo che il flatmount è stato fissato in PFA al 4% riduce il danno alle cellule RPE situate attorno al nervo ottico. - Rimuovere la sclera dall'RPE / coroide sollevando delicatamente lo strato RPE / coroide dalla periferia e tagliando i vasi coroideali e il tessuto connettivo che si trovano tra la sclera e l'RPE con un paio di forbici a molla Vannas. Una volta che l'RPE / coroide è completamente separato dalla sclera, raccogliere lo strato RPE / coroide. Trasferire il tessuto in un contenitore riempito con DPBS 1x con Ca 2+ e Mg2+ e conservarlo a 4°C.
Nota : in questo momento, l'esperimento può essere sospeso.
4. Colorazione
- Trasferire l'RPE/coroide in un pozzetto di una piastra a 6 pozzetti. Bloccare e permeabilizzare il campione in DPBS 1x con Ca 2+ e Mg 2+ con albumina sierica bovina (BSA) all'1%, 0,5% Tween20 e Triton X-100 allo 0,5% per 1 ora a temperatura ambiente.
- Incubare il campione con falloidina coniugata con un fluoroforo 647 ad una diluizione 1:250 in DPBS 1x con Ca 2+ e Mg 2+ con 1% BSA, 0,5% Tween20 e 0,5% Triton X-100 per 1 ora a temperatura ambiente. Lavare 3x in DPBS 1x con Ca 2+ e Mg2+.
- Trasferire il campione RPE/coroide su un vetrino da 50 mm x 75 mm e appiattirlo. Tagliare ogni "petalo" in due per rendere il campione più piatto. Presta attenzione alla macula. Disegnare un contorno del supporto piatto con una penna idrofobica.
- Per estinguere l'autofluorescenza della lipofuscina, aggiungere 500 μL della soluzione di quencher di autofluorescenza diluita a 1:20 in etanolo al 70% e incubare a temperatura ambiente per 2 minuti.
- Lavare accuratamente (almeno 3x) in DPBS 1x con Ca 2+ e Mg2+. Rimuovere il DPBS e aggiungere il supporto di montaggio. Posizionare un vetro di copertura sul supporto piatto e sigillare con lo smalto.
- Immagine del flatmount con un microscopio a fluorescenza (preferibilmente utilizzando un obiettivo 10x o 20x).
5. Analisi REShAPE
NOTA: Poiché l'algoritmo basato sull'intelligenza artificiale REShAPE è stato addestrato su immagini 10x e 20x, si consiglia vivamente di utilizzare un obiettivo 10x o 20x durante l'imaging. In caso contrario, le immagini dovranno essere ridimensionate di conseguenza.
- Se le immagini vengono acquisite con più di un canale fluorescente, isolare il canale utilizzato per acquisire i bordi delle celle. Esporta le immagini come file TIF in scala di grigi a 16 bit.
NOTA: le immagini acquisite con l'estensione .czi non devono essere esportate come file TIF, ma il canale contenente i bordi delle celle deve comunque essere isolato. - Installare il software su piattaforme Windows x64 o Linux (Centos 7).
NOTA: il software e le istruzioni per l'installazione sono disponibili all'indirizzo https://github.com/nih-nei/REShAPE. - Aprire il software (Figura 1).
- Nella scheda Directory selezionare le cartelle di input e output. Nella directory di input, selezionare la cartella che contiene le immagini. Nella directory di output, lasciare che il software cambi automaticamente il percorso in "directory di input / Elaborato". In alternativa, modificare manualmente la directory di output.
NOTA: il software scorrerà tutte le immagini contenute nella cartella di input. - Nella scheda Opzioni segmentazione NN, scegliete le seguenti impostazioni per la segmentazione delle immagini:
- Dimensione batch: digitare un valore pari a 20 nella casella di testo come buon punto di partenza, ma ridurlo se il sistema esaurisce le unità di elaborazione grafica (GPU).
NOTA: l'immagine originale è suddivisa in riquadri più piccoli per l'elaborazione. La dimensione del batch specifica quante tessere possono essere elaborate contemporaneamente. - Usa GPU?: se il sistema non dispone di risorse GPU sufficienti, seleziona la casella No.
- Scala interna: utilizzando il menu a discesa, modificare il ridimensionamento interno da Nessuno nei casi in cui le immagini non sono state acquisite con un obiettivo 10x o 20x. Ad esempio, ridimensiona le immagini 40x a metà delle dimensioni utilizzando il pulsante 1/2 . Le opzioni di ridimensionamento disponibili nel menu a discesa sono le seguenti: 5x, 2x, Nessuna, 1/2, 1/5.
NOTA: l'apprendimento automatico è stato addestrato con immagini 10x e 20x. Potrebbe non riconoscere i bordi delle celle da immagini scattate ad altri ingrandimenti. In questo caso, le immagini binarie appariranno completamente nere. - Filtro Arti: utilizzare la linguetta del filtro artefatto per rimuovere particelle molto luminose (detriti o piccole parti della retina o del tessuto connettivo attorno al nervo ottico) che possono essere presenti nell'immagine e possono interferire con la segmentazione dei bordi cellulari. Per impostazione predefinita non viene utilizzato alcun filtro. Le opzioni di filtro artificiale nel menu a discesa sono le seguenti: Regolare, Forte e Debole.
- Dimensione batch: digitare un valore pari a 20 nella casella di testo come buon punto di partenza, ma ridurlo se il sistema esaurisce le unità di elaborazione grafica (GPU).
- Nella scheda Opzioni immagine affiancata , inserisci un valore nella casella di testo per specificare la quantità di sovrapposizione tra i riquadri immagine regolando il parametro Pad Tiles By . Una sovrapposizione di 100 pixel di solito funziona bene.
- Nella scheda Opzioni grafica di output , ottimizza le impostazioni per la generazione della mappa di calore:
- Ricostruisci immagini piastrellate?: se il sistema non dispone di risorse sufficienti per l'analisi di immagini di grandi dimensioni, selezionare la casella No per consentire al software di completare l'analisi salvando singole tessere senza ricostruire l'intera immagine.
- Crea immagini a colori: per generare mappe termiche, seleziona Tutto dal menu a discesa.
- Per Colorare, scegli una delle diverse tavolozze di colori disponibili nel menu a discesa: Termico, Verde, Mpl-magma, Fase, Fuoco, Jet, Ciano Caldo.
- Per Formato immagine, salva le immagini come TIF o PNG selezionando una delle opzioni nella scheda.
- Per quanto riguarda la funzione Usa limiti manuali? , selezionare la casella No per consentire al software di utilizzare i valori minimo e massimo rilevati in ogni immagine. Seleziona la casella Sì per regolare manualmente l'intervallo di valori per ogni mappa termica metrica della forma e fai clic sul pulsante Imposta limiti per scegliere gli intervalli per i singoli parametri inserendo i valori nelle caselle di testo. Dopo aver modificato i valori di interesse, fare clic su Salva. Fare clic su Carica impostazioni predefinite per ripristinare tutti i limiti.
NOTA: utilizzare i limiti manuali se è necessario confrontare le mappe di calore di più immagini, ad esempio quando si confrontano gli effetti di diversi composti sulla forma delle cellule. In questo modo, viene utilizzato lo stesso intervallo di valori. L'insieme dei valori da immettere manualmente varia a seconda del tipo di campione. Si consiglia di eseguire alcune iterazioni per scegliere l'intervallo ottimale.
- Nella scheda Restrizioni dimensione cella per analisi, selezionare una soglia di dimensioni cella per l'analisi:
- In Dimensioni cella inferiori, inserire nella casella di testo la dimensione della cella più piccola da includere nell'analisi.
- In Dimensioni cella superiore, inserire nella casella di testo la dimensione della cella più grande da includere nell'analisi.
NOTA: l'unità di dimensione della cella cambia da pixel a micrometri quadrati a seconda dell'opzione scelta nella scheda Conversione automatica unità .
- Nella scheda Conversione automatica unità, scegliere l'unità preferita per l'analisi:
- In Converti pixel in unità reali?, seleziona la casella No per eseguire l'analisi in unità di pixel. Selezionare la casella Sì per eseguire l'analisi in micrometri.
- In Lunghezza della barra di scala (Pixel), immettete il valore in pixel nella casella di testo.
- In Lunghezza della barra di scala (Micron), immettere nella casella di testo la distanza corrispondente in micrometri.
- Per avviare l'analisi, premere Vai.
NOTA: Il software può anche misurare la vitalità cellulare quando le cellule sono colorate con 4',6-diamidino-2-fenilindolo (DAPI) e ioduro di propidio (scheda opzioni CZI ), ma questo non si applica ai flatmount RPE.
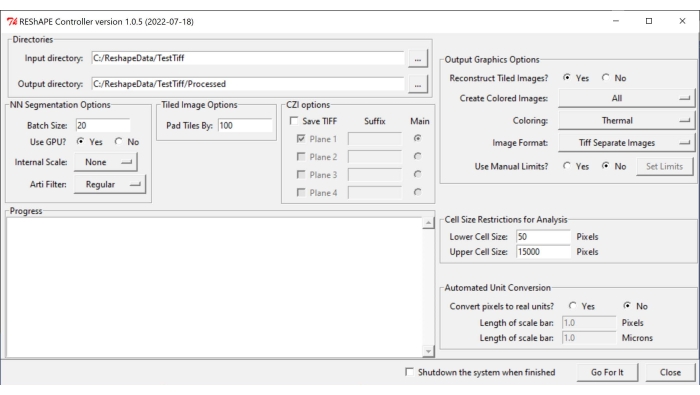
Figura 1: Interfaccia utente grafica REShAPE. La GUI ha diverse schede per selezionare le directory di lavoro (scheda Directory ), modificare le opzioni di segmentazione (schede Opzioni di segmentazione NN e Opzioni immagine affiancata ), specificare i parametri per l'analisi (Restrizioni dimensione cella per analisi e Conversione automatica unità ) e per la generazione di mappe di calore (scheda Opzioni grafica di output ). Abbreviazione: GUI = interfaccia utente grafica. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Risultati
Questo protocollo produce un'immagine single-plane di un flatmount, in cui vengono misurate la posizione della cella e 30 metriche di forma per ogni cella RPE correttamente identificata (Figura 2). Una cartella denominata "Elaborato" viene generata automaticamente all'interno della directory di input. Questa cartella contiene quattro sottodirectory, denominate "Analisi", "Codifica colore", "File combinati" e "Immagini segmentate", nonché alcuni file temporanei generati durante l'analisi. La cartella "File combinati" contiene un foglio di calcolo con tutte le misure della forma e un foglio di calcolo con le frequenze dei conteggi delle celle adiacenti di tutti i file combinati. La cartella "Analisi" contiene un foglio di calcolo con tutte le misure della forma e un foglio di calcolo con le frequenze dei conteggi delle celle vicine per ogni immagine separatamente. La directory "Segmented Images" contiene le maschere binarie finali dei bordi delle celle RPE; Può essere utilizzato per valutare la qualità della segmentazione. La directory "Color Coded" contiene mappe di calore per ogni misurazione della forma per visualizzare i modelli di forma in ogni immagine. Le definizioni metriche e le abbreviazioni delle forme sono riportate nella Tabella 1.
A volte i flatmount RPE possono contenere pezzi residui di retina che non sono stati rimossi in modo pulito, specialmente intorno al nervo ottico. La colorazione con falloidina del campione provoca un forte segnale proveniente dalla retina e questo può causare problemi per la segmentazione del bordo delle cellule RPE. Alcune tessere appariranno completamente nere, mentre le tessere circostanti mostreranno una normale segmentazione. Altri oggetti luminosi che potrebbero essere presenti nell'immagine causeranno anche la generazione di tessere nere (Figura 3). In questi casi, scegliendo una delle opzioni di filtraggio (Debole, Regolare, Forte) disponibili nel menu a tendina Filtro Arti si eviterà la formazione di tessere nere.
REShAPE accetta immagini in scala di grigi a 8 o 16 bit come input ma non immagini RGB. L'utilizzo di immagini RGB per l'analisi REShAPE produrrà immagini binarie interamente nere. In questo caso, la conversione delle immagini RGB in scala di grigi produrrà immagini binarie segmentate correttamente (Figura 4). In alcune occasioni in cui i bordi RPE non vengono riconosciuti correttamente, ad esempio, se la colorazione non è ottimale o se il campione è danneggiato da un graffio (Figura 5A), grandi grumi di cellule possono essere identificati come una singola cella molto grande (Figura 5B). In questo caso, gli oggetti di grandi dimensioni possono essere esclusi dall'analisi riducendo la soglia di dimensione della cella (Figura 5C). Questa operazione può essere ottenuta inserendo un valore inferiore nella casella di testo Dimensioni cella superiore . Tuttavia, ciò comporterà una modifica dell'intervallo della mappa di calore. Se un ricercatore sceglie di farlo, è anche possibile mantenere l'intervallo di mappe di calore originale (Figura 5D) selezionando la casella Sì nella funzione Usa limiti manuali? . Successivamente, il ricercatore deve fare clic con il pulsante sinistro del mouse sul pulsante Imposta limiti e inserire i valori desiderati nelle caselle di testo per specificare i limiti manuali.
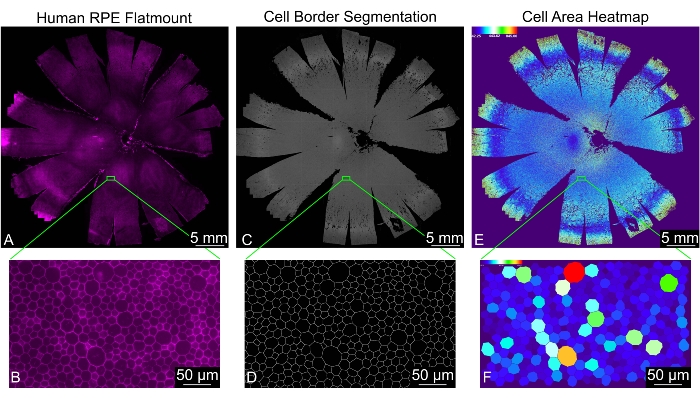
Figura 2: Analisi morfometrica completa di un intero monostrato di RPE umano. (A) Una vista a basso ingrandimento di un intero RPE/coroide umano flatmount (magenta: falloidina). (B) Una vista ingrandita delle cellule RPE colorate con falloidina. (C) segmentazione generata da REShAPE dei bordi delle celle RPE per un intero RPE/coroide umano flatmount e (D) la corrispondente vista ingrandita. (E) Una mappa di calore generata dal software che illustra l'area delle celle delle singole celle RPE nell'intero flatmount umano. La scala termica nell'angolo in alto a sinistra mostra l'intervallo di valori utilizzati. (F) La corrispondente visualizzazione ingrandita che mostra le singole celle RPE colorate per area. Barre della scala = (B,D,F) 50 μm, (A,C,E) 5 mm. Abbreviazione: RPE = epitelio pigmentato retinico. Fare clic qui per visualizzare una versione ingrandita di questa figura.
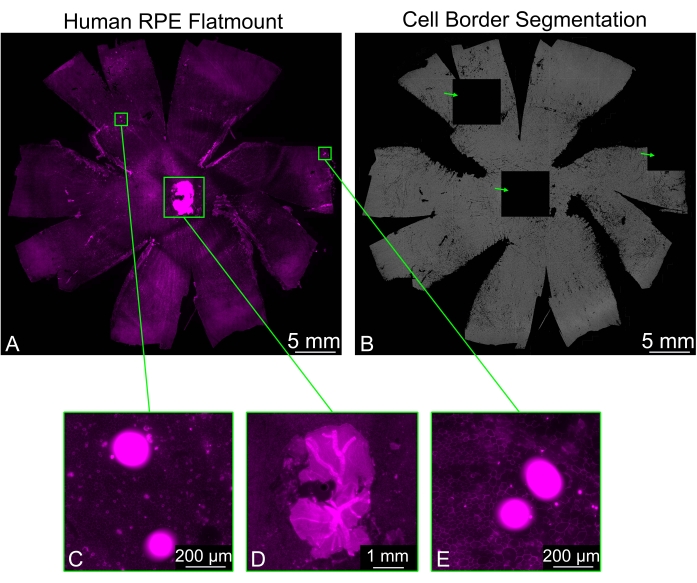
Figura 3: Filtraggio di artefatti luminosi. (A) Un RPE umano a montaggio piatto colorato per i bordi delle celle (magenta: falloidina) può presentare aree luminose (rettangoli verdi) che interferiscono con la segmentazione. (B) La segmentazione del bordo della cella RPE dell'intero flatmount contiene tre piastrelle completamente nere (frecce verdi) corrispondenti a regioni luminose di fluorescenza. (C,E) Due delle piastrelle nere corrispondono ad aree contenenti punti luminosi, che sono probabilmente alcuni detriti. (D) Una delle piastrelle nere è stata generata da un pezzo di retina neurale attorno al nervo ottico che non è stato rimosso correttamente. I pezzi di retina neurale sono considerevolmente più luminosi dello strato RPE e ostacolano la segmentazione cellulare. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 4: Requisito dell'immagine di input. Le celle RPE colorate per i bordi delle celle sono state salvate come (A) RGB o come (B) immagini in scala di grigi a 16 bit per l'analisi REShAPE. (C) L'output dell'analisi dell'immagine RBG è un'immagine binaria nera, (D) mentre l'analisi dell'immagine in scala di grigi produce un binario correttamente segmentato dei bordi delle celle. REShAPE può analizzare solo immagini in scala di grigi a 8 o 16 bit. Barre di scala = 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 5: Risultati non ottimali . (A) Un'immagine di una porzione del monostrato RPE in cui le cellule colorate con falloidina sono state accidentalmente graffiate. (B) Una mappa di calore delle celle RPE colorate dalla dimensione dell'area della cella. Una soglia di dimensioni delle celle superiori di grandi dimensioni include oggetti di grandi dimensioni nell'analisi. (C) Una mappa termica dell'area della cella in cui è stata scelta una soglia di dimensione superiore della cella più piccola per escludere oggetti di grandi dimensioni dall'analisi. (D) Una mappa di calore dell'area della cella in cui è stata scelta una soglia di dimensione della cella superiore più piccola e sono stati impostati limiti manuali per mantenere l'intervallo di mappe di calore utilizzato originariamente. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella 1: parametri REShAPE. La tabella riporta la definizione di ciascun parametro e le abbreviazioni utilizzate nei fogli di calcolo grezzi (file "_Data.csv") e per le mappe di calore. Clicca qui per scaricare questa tabella.
Discussione
La separazione coerente ed efficiente di RPE umano e retine può essere ottenuta utilizzando questo protocollo. Questo metodo consente lo studio delle differenze regionali nella forma RPE attraverso intere retine umane5. Un passo cruciale nel protocollo è la separazione fisica dell'RPE e della retina. Se i due tessuti non sono completamente staccati in alcune aree, si dovrebbe sollevare delicatamente la retina, assicurandosi di non rompere i tessuti. L'analisi REShAPE di grandi flatmount può richiedere l'uso di sistemi con notevoli risorse RAM. In questo caso, il riassemblaggio dell'intera immagine può essere disabilitato per consentire al software di completare correttamente l'analisi nonostante la mancanza di risorse di elaborazione.
La principale limitazione dell'utilizzo di REShAPE per segmentare i flatmount RPE umani è che l'algoritmo AI è stato per lo più addestrato su immagini di RPE derivato da cellule staminali pluripotenti indotte. Di conseguenza, la segmentazione dei flatmount RPE umani è meno accurata. Le cellule RPE di donatori anziani contengono una grande quantità di lipofuscina7 e l'ampio spettro della sua autofluorescenza interferisce con la segmentazione del bordo cellulare. In futuro, verranno utilizzate più immagini di supporti piatti RPE per migliorare la segmentazione dei bordi delle celle in questo tipo di campione. Nonostante questa limitazione, REShAPE è stato specificamente addestrato a riconoscere e segmentare i bordi delle celle RPE e offre prestazioni migliori rispetto ad altri metodi esistenti, come la segmentazione delle celle RPE di Voronoi8 e CellProfiler9 .
Inoltre, rispetto alla segmentazione manuale 10, REShAPE offre il vantaggio di analizzare rapidamente immagini di grandi dimensioni (sono stati testati ~130.000 pixel x130.000 pixel). In conclusione, questo metodo di dissezione è altamente riproducibile e può essere facilmente esteso ad altri modelli animali. Inoltre, il software può essere utilizzato per studiare la forma RPE in piani oculari o in modelli di coltura cellulare per esaminare l'effetto di determinati trattamenti. Infine, la versatilità di REShAPE lo rende ampiamente applicabile per l'analisi di altri tipi di cellule epiteliali.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo il nucleo istologico del National Eye Institute (NEI) per l'uso di Zeiss Axio Scan.Z1. Ringraziamo anche i donatori, le loro famiglie, l'Advancing Sight Network e il Lions Eye Institute per la loro generosità. Questo lavoro è stato sostenuto dai fondi IRP NEI (numero di sovvenzione ZIA EY000533-04).
Materiali
| Name | Company | Catalog Number | Comments |
| Biopsy punch 1.5 mm | Acuderm Inc. | P1525 | |
| Bovine albumin | MP Biomedicals | 160069 | |
| Coverglass 50 x 75 mm, #1.5 thickness | Brain Research Laboratories | 5075-1.5D | |
| Curved spatula | Katena | K3-6600 | |
| D-Mannitol | Sigma | M9546 | |
| DPBS 1x with Ca2+ and Mg2+ | Gibco | 14040-133 | |
| Fine Scissors | Fine Science Tools | 14558-11 | |
| Fluormount-G | Southern Biotech | 0100-01 | |
| Forceps - Dumont #5 | Fine Science Tools | 11252-23 | |
| Microscope slides 50 x 75 x 1.2 mm | Brain Research Laboratories | 5075 | |
| Needles 21 G x 1-1/2" hypodermic | Becton Dickinson (BD) | 305167 | |
| Needles 27 G x 1-1/4" hypodermic | Becton Dickinson (BD) | 305136 | |
| Paraformaldehyde 16% | Electron Microscopy Sciences | 15710 | |
| Petri dish 100 mm | Corning | 430167 | |
| Phalloidin-iFluor 647 | Abcam | ab176759 | |
| Razor blades | PAL (Personna) | 62-0177 | |
| Round bottom tubes 50 mL | Newegg | 9SIA4SR9M88854 | |
| Silicon Elastomer Kit | Dow Corning Corporation | 4019862 | |
| Square weighing boat (81 mm x 81 mm x 25 mm) | Sigma | W2876 | |
| Surgical Vitrectomy System | BD Visitrec | 585100 | optional |
| Syringe 1 mL | Becton Dickinson (BD) | 309659 | |
| Triton X-100 | Sigma | T9284 | |
| TrueBlack | Biotium | 23007 | autofluorescence quencher |
| Tween 20 | Affymetrix | 20605 | |
| Vannas Spring Scissors - 3 mm cutting edge | Fine Science Tools | 15000-10 |
Riferimenti
- Endo, E. G., Yao, X. Y., Marmor, M. F. Pigment adherence as a measure of retinal adhesion: Dependence on temperature. Investigative Ophthalmology and Visual Science. 29 (9), 1390-1396 (1988).
- Marmor, M. F. Retinal detachment from hyperosmotic intravitreal injection. Investigative Ophthalmology and Visual Science. 18 (12), 1237-1244 (1979).
- Marmor, M. F., Martin, L. J., Tharpe, S. Osmotically induced retinal detachment in the rabbit and primate. Electron microscopy of the pigment epithelium. Investigative Ophthalmology and Visual Science. 19 (9), 1016-1029 (1980).
- Ban, Y., Rizzolo, L. A culture model of development reveals multiple properties of RPE tight junctions. Molecular Vision. 3, 18(1997).
- Ortolan, D., et al. Single-cell-resolution map of human retinal pigment epithelium helps discover subpopulations with differential disease sensitivity. Proceedings of the National Academy of Sciences of the United States of America. 119 (19), 2117553119(2022).
- Schaub, N. J., et al. Deep learning predicts function of live retinal pigment epithelium from quantitative microscopy. Journal of Clinical Investigation. 130 (2), 1010-1023 (2020).
- Beatty, S., Koh, H. -H., Phil, M., Henson, D., Boulton, M. The role of oxidative stress in the pathogenesis of age-related macular degeneration. Survey of Ophthalmology. 45 (2), 115-134 (2000).
- Liu, Z., Kocaoglu, O. P., Miller, D. T. 3D imaging of retinal pigment epithelial cells in the living human retina. Investigative Ophthalmology and Visual Science. 57 (9), 533-543 (2016).
- Bhatia, S. K., et al. Analysis of RPE morphometry in human eyes. Molecular Vision. 22, 898-916 (2016).
- vonder Emde, L., et al. Histologic cell shape descriptors for the retinal pigment epithelium in age-related macular degeneration: A comparison to unaffected eyes. Translational Vision Science & Technology. 11 (8), 19(2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon