Method Article
Vollblutassay mit dualer Co-Stimulation zur antigenspezifischen Analyse der Immunität des Wirts gegen pilzliche und virale Krankheitserreger
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Vollblutbasierte Immunoassays bieten ein einfaches und ressourceneffizientes Werkzeug zur Analyse der antigenspezifischen Immunität für Diagnose- und Forschungszwecke. Dieser Artikel bietet ein optimiertes Vollblut-basiertes Protokoll mit dualer Co-Stimulation für eine umfassende Analyse der Immunität des Wirts gegen pilzliche und virale Krankheitserreger, einschließlich einer Version mit geringem Volumen für pädiatrische Patienten und Kleintiere.
Zusammenfassung
Eine schnelle und ressourceneffiziente Probenverarbeitung, ein hoher Durchsatz und eine hohe Robustheit sind entscheidend für eine effektive wissenschaftliche und klinische Anwendung fortschrittlicher antigenspezifischer Immunoassays. Traditionell beruhen solche Immunoassays, insbesondere die antigenspezifische T-Zell-Analyse mittels Durchflusszytometrie oder enzymgebundene Immunsorbent-Spot-Assays, häufig auf der Isolierung von mononukleären Zellen des peripheren Blutes. Dieser Prozess ist zeitaufwändig, unterliegt vielen präanalytischen Störfaktoren und erfordert große Blutmengen. Vollblutbasierte Assays bieten eine einfache Alternative mit erhöhter präanalytischer Robustheit und geringeren Anforderungen an das Blutvolumen. Darüber hinaus ermöglichen Vollblut-basierte Assays die Konservierung interzellulärer Interaktionen, die von Assays mit isolierten Zelluntergruppen nicht erfasst werden. Kürzlich wurde ein verfeinerter Vollblut-Immunoassay mit dualer anti-CD28- und anti-CD49d-Co-Stimulation für eine umfassende Analyse sowohl antigenspezifischer T-Zell-Funktionen als auch komplexer interzellulärer Wechselwirkungen als Reaktion auf verschiedene pilzliche und virale Antigene vorgeschlagen. Dieses Protokoll bietet eine Anleitung für die Vorbereitung von Stimulationsröhrchen, die Blutstimulation und die nachgelagerte Probenverarbeitung für die Durchflusszytometrie, Zytokinsekretionsassays und Transkriptionsanalysen. Dazu gehört ein validiertes und funktionell äquivalentes, bisher unveröffentlichtes Protokoll mit geringem Volumen (250 μl), um das durchflusszytometrische und zytokinbasierte T-Zell-Monitoring für Studien an pädiatrischen Patienten oder präklinische Studien an Kleintieren (z. B. Mäusen) zugänglicher zu machen. Insgesamt bieten diese Protokolle einen vielseitigen Werkzeugkasten für die komplexe antigenspezifische Immunanalyse sowohl in der klinischen als auch in der translationalen Forschung.
Einleitung
Die Quantifizierung und Charakterisierung der antigenspezifischen Immunität, insbesondere der spezifischen T-Zell-Antworten, ist für die Immunbiologie und Impfforschung sowie für einige diagnostische Tests von zentraler Bedeutung. Traditionell stützten sich antigenspezifische Immunoassays häufig auf isolierte mononukleäre Zellen des peripheren Blutes (PBMCs). Die Isolierung dieser Zellen ist jedoch zeit- und ressourcenintensiv und erfordert oft relativ große Blutmengen. Um die Aktivierung von Granulozyten und die anschließende Störung der T-Zellen während der präanalytischen Lagerung1 zu verhindern, ist außerdem eine schnelle Verarbeitung der Proben von größter Bedeutung, die in der klinischen Praxis oft nicht durchführbar ist. Diese Einschränkungen behindern die Praktikabilität antigenspezifischer Immunoassays in Hochdurchsatz-Forschungsszenarien und klinischen Routinen. Daher hat die Entwicklung von einfach zu handhabenden und potenziell automatisierbaren Vollblut-basierten Ansätzen in den letzten Jahren neue Anwendungsbereiche für Immunoassays eröffnet. Den derzeit kommerziell erhältlichen Systemen fehlen jedoch in der Regel optimale co-stimulatorische Umgebungen für T-Zellen und sie sind anfällig für präanalytische Verzögerungen. Zum Beispiel hat ein weit verbreiteter Vollblut-basierter IFN-γ-Freisetzungsassay eine positive zu negative Reversionsrate von 19 % nach 6 Stunden präanalytischer Blutlagerung2. Um diese Einschränkungen zu überwinden, wurden optimierte Protokolle mit dualer Anti-CD28- und Anti-CD49d-Co-Stimulation entwickelt 3,4,5,6.
Das hier vorgestellte Protokoll ermöglicht eine genaue und reproduzierbare Quantifizierung und Charakterisierung von antigenspezifischen T-Zellen, die Bewertung von Antigen-induzierten Zytokinantworten und andere (durchflusszytometrische oder transkriptionelle) funktionelle Immunmarker aus einem minimalen Blutvolumen, d.h. 500 μL Blut pro Stimulationsröhrchen. Zu den weiteren Vorteilen dieses Protokolls gehören die geringe Hands-on-Zeit, die hohe Resilienz gegenüber präanalytischen Störfaktoren und die Erhaltung funktioneller interzellulärer Interaktionen in einer relativ physiologischen ex vivo-Umgebung . Die Vergleichbarkeit der vollblutbasierten durchflusszytometrischen antigenspezifischen T-Zell-Charakterisierung mit Daten, die aus herkömmlichen PBMC-basierten Assays gewonnen wurden, wurde bereits im Rahmen der moldspezifischen T-Zell-Quantifizierung gezeigt6. Darüber hinaus entfällt durch die direkte Stimulation des Blutes der Probanden die Notwendigkeit einer Supplementierung mit autolom, allogenem oder sogar xenogenem Serum, das üblicherweise für eine optimale PBMC-Stimulation erforderlich ist. Der Wegfall der Zellisolierung reduziert auch die Scher- und Temperaturbelastung und verbessert dadurch die Lebensfähigkeit der Zelle. Am wichtigsten ist, dass Vollblut-basierte Assays Granulozytenpopulationen erhalten, die während der Gradientenzentrifugation für die Isolierung von PBMCs verloren gehen7. Dabei werden bei diesem Assay-Aufbau funktionelle Interaktionsschleifen zwischen Granulozyten und mononukleären Zellen erhalten und erfasst4.
Bemerkenswert ist, dass dieses Protokoll nur minimale Modifikationen erfordert, um unterschiedliche Auslesemodalitäten zu berücksichtigen, und sogar eine duale Analyse der Zytokinfreisetzung und der Transkriptionsreaktionen von derselben Stimulationsröhre ermöglicht. Während Zytokine nach der Stimulation aus dem Kulturüberstand analysiert werden, kann das Zellpellet für die RNA-Isolierung mit anschließender transkriptomischer Analyse verwendet werden. Der allgemeine Arbeitsablauf für die verschiedenen Auslesemodalitäten ist in Abbildung 1 zusammengefasst.
In den letzten Jahren wurde eine zunehmende Anzahl von Vollblut-basierten Assays für das pathogen-reaktive Immunmonitoring in Forschung und Klinik entwickelt, z. B. für Mycobacterium tuberculosis 8,9, Bordetella pertussis3, Orientia tsutsugamushi10 und SARS-CoV-2 5,11,12. Zum Beispiel wurde ein zuvor etabliertes System für mehrere Antigene verwendet, darunter M. tuberculosis, Influenza-A-Virus und SARS-CoV-2, verwendet jedoch keine co-stimulatorischen Faktoren, die für die Stimulation von T-Helfer (Th)-Zellen optimiert sind 13,14,15. Auch wenn das für diese Assays erforderliche Blutvolumen bereits deutlich geringer ist als das für herkömmliche PBMC-basierte Assays oder kommerziell erhältliche Vollblutstimulationskits, könnte ein noch kleineres Probenvolumen für Anwendungen in der Pädiatrie, Neonatologie, bei Patienten auf der Intensivstation (ICU) und in der präklinischen Forschung in Kleintiermodellen gerechtfertigt sein. Zum Beispiel ergibt selbst die terminale Blutentnahme von Mäusen (z. B. durch Herzpunktion) in der Regel ein Maximum von 0,7-1 ml Blut. Daher wurde im Rahmen dieses Protokolls die Möglichkeit evaluiert, zuvor etablierte Vollblut-basierte Immunoassay-Protokolle 4,6 zur präzisen Quantifizierung und Charakterisierung von antigen-reaktiven T-Zell-Antworten ab 250 μl Blutvolumen pro Stimulationsröhrchen weiter herunterzuskalieren.
Protokoll
Der Ethikrat der Ludwig-Maximilians-Universität München (Projektnummer 21-0689) hat die Entnahme von menschlichen Blutproben genehmigt. Zusätzlich wurden Teile der repräsentativen Datensätze unter der Corona-Register-Studiennummer 20-426 generiert, die ebenfalls von der Ethikkommission der Ludwig-Maximilians-Universität München genehmigt wurde. Die Einwilligung nach Aufklärung wurde eingeholt.
1. Vorbereitung von Vollblut-Stimulationsröhrchen
HINWEIS: Dieser Schritt wurde von Lauruschkat et al.4 und Weis et al.6 übernommen. Tabelle 1 fasst die detaillierten Reagenzkonzentrationen und -volumina zusammen.
- Bereiten Sie unter sterilen Bedingungen (sterile Werkbank) 2,7 mL Blutentnahmeröhrchen ohne Antikoagulans, mit Antigenen und co-stimulatorischen Antikörpern vor. Geben Sie 1 μg/ml α-CD28 und 1 μg/ml α-CD49d in jedes Röhrchen, einschließlich der Negativkontrolle. Berücksichtigen Sie bei der Bestimmung der Endkonzentrationen (250 μl oder 500 μl) das Blutvolumen, das für die Vollblutstimulation hinzugefügt werden soll. Um die Kammergeometrie und die Zugänglichkeit der Probe zu erhalten, ziehen Sie den Kolben nicht aus dem Blutentnahmeröhrchen zurück.
- Bestimmen Sie auf der Grundlage des Studiendesigns, ob die negativen Kontrollröhrchen entweder keinen zusätzlichen Stimulus enthalten sollten (unstimulierte Hintergrundkontrolle, wie in Tabelle 1 gezeigt) oder Antigene verwenden sollten, für die die Studienkohorte keine oder nur minimale antigenspezifische T-Zellen beherbergen sollte (z. B. ein HIV-Peptidpool bei HIV-seronegativen Personen 16).
- Für die antigenspezifische Stimulation optimieren Sie in Vorversuchen die idealen Konzentrationen von Antigenen durch Titration. Um die folgenden repräsentativen Daten zu generieren, verwenden Sie die folgenden optimierten Antigenkonzentrationen: 1,2 μg/ml HSV-1-Lysat, 0,6 nM/Peptid/ml CMV pp65, 0,6 nM/Peptid/ml SARS-CoV-2 Prot_S, 50 μg/ml Aspergillus fumigatus lysat und 1 ng/ml CRX-527.
- Schließen Sie eine Positivkontrolle ein, insbesondere für Studien an lymphopenischen Patienten oder Patienten, die eine immunsuppressive Pharmakotherapie erhalten. Verwenden Sie eine CPI-Positivkontrolllösung (0,6 nM/Peptid/ml), bestehend aus Cytomegalievirus-, Parainfluenzavirus- und Influenzaviruspeptiden.
HINWEIS: Alternativ können synthetische Stimuli wie PMA (10 μg/ml) ± Ionomycin (1 μg/ml) verwendet werden, aber sie induzieren weniger physiologische Reaktionen und können die Expression von Oberflächenantigenen beeinflussen, die üblicherweise als Populationsmarker für die Durchflusszytometrie verwendet werden (insbesondere CD417). Beachten Sie, dass co-stimulatorische Antikörper in Verbindung mit den meisten synthetischen Positivkontrollreizen nicht benötigt werden und sich sogar negativ auf die Lebensfähigkeit und Reaktionsfähigkeit der Zellen auswirken können. - Um das Reagenzvolumen über alle Stimulationsröhrchen hinweg zu harmonisieren, fügen Sie RPMI 1640 Medium bis zu den folgenden Gesamtvolumina hinzu: Full-Scale-Assay für die Durchflusszytometrie: 50 μl; Full-Scale-Assay für Zytokinanalyse und Transkriptomik: 500 μl; Kleiner Assay für die Durchflusszytometrie: 25 μl; Kleiner Assay für die Zytokinanalyse und Transkriptomik: 250 μL.
HINWEIS: Für die Herstellung mehrerer Sätze von Stimulationsröhrchen wird empfohlen, Mastermixe mit allen Zutaten herzustellen, da das Kontaminationsrisiko geringer ist und das Volumen an co-stimulatorischen Faktoren und Stimuli gering ist. - Verwenden Sie die vorbereiteten Röhrchen sofort oder kryokonservieren Sie sie bei -20 °C. Die meisten Reagenzien können bis zu 4 Wochen gelagert werden; Validieren Sie jedoch die maximalen Lagerzeiten für neue Reagenzien/Stimuli im Voraus.
2. Stimulation und Inkubation von Vollblutproben
- Etwa 30-60 min vor der Vollblutstimulation die gebrauchsfertigen Stimulationsröhrchen auftauen und bei Raumtemperatur aufbewahren.
- Entnehmen Sie venöses Blut vom Spender/Patienten mit Hilfe von Blutentnahmeröhrchen mit Lithium-Heparin-Antikoagulans. Stellen Sie sicher, dass das entnommene Blutvolumen den individuellen Versuchsanforderungen entspricht, d. h. für 3 Bedingungen des kleinen Assays, einschließlich Negativ- und Positivkontrolle, sind mindestens 750 μl erforderlich. Die kommerziell erworbenen Blutentnahmeröhrchen füllen Sie vollständig, um Heparin-Arbeitskonzentrationen von 16 - 25 IE/mL Blut zu erreichen.
HINWEIS: Die demografischen Daten der gesunden erwachsenen Probanden, die zur Erstellung der repräsentativen Datensätze rekrutiert wurden, sind in Tabelle 2 dargestellt. - Übertragen Sie die erforderliche Menge heparinisiertes Vollblut (250 μl bzw. 500 μl für kleine bzw. vollständige Assays) in die Stimulationsröhrchen, indem Sie sie unter einer sterilen Werkbank pipettieren.
HINWEIS: Wenn keine sterile Werkbank verfügbar ist oder wenn mit Proben gearbeitet wird, die ein minimales Infektionsrisiko aufweisen (z. B. Blut von vorgescreenten Probanden oder Tieren), können die Proben mit sterilen Spritzen in die Stimulationsröhrchen überführt werden. Diese Methode ermöglicht auch die Probenverarbeitung am Krankenbett unmittelbar nach der Blutentnahme, wodurch die präanalytische Lagerung und die Beeinträchtigung der T-Zellen minimiert werden. Desinfizieren Sie vor der Blutinjektion die Gummidichtungen sowohl des Blutentnahmeschlauchs als auch aller Stimulationsschläuche ausgiebig mit einem alkoholischen Desinfektionsmittel. Lassen Sie das Desinfektionsmittel mindestens 1 Minute trocknen. - Drehen Sie die Stimulationsröhren vorsichtig 5x-10x um. Legen Sie die Stimulationsschläuche in einen 37 °C heißen Inkubator. Beachten Sie, dass ein CO2 - Inkubator nicht erforderlich ist. Um eine unspezifische Aktivierung von Neutrophilen zu verhindern, kühlen Sie das Blutentnahmeröhrchen nicht.
HINWEIS: Dieses Protokoll wurde für die präanalytische Blutlagerung von bis zu 8 Stunden bei Raumtemperatur validiert, um den Probentransport zu ermöglichen. Wenn möglich, ist es vorzuziehen, Vollblut in die Stimulationsröhrchen an der Entnahmestelle zu injizieren und das Blut in den Stimulationsröhrchen zu einem Zentrallabor zu transportieren, da co-stimulatorische Faktoren die Lebensfähigkeit der Lymphozyten verbessern18,19. - Nur bei Proben, die für durchflusszytometrische Assays mit intrazellulärer Färbung verwendet werden, ist Brefeldin A (Endkonzentration von 10 μg/ml) nach 4 Stunden Inkubation in jedes Stimulationsröhrchen zu geben.
- Bereiten Sie eine 1 mg/ml-Brefeldin-A-Lösung in RPMI vor und fügen Sie 1 μl der Lösung pro 100 μl Probenvolumen hinzu (d. h. vorgemischter Stimulationscocktail + Vollblutvolumen). Fügen Sie Brefeldin A hinzu, indem Sie die Röhrchen öffnen und unter einer sterilen Werkbank pipettieren. Verschließen Sie die Röhrchen und geben Sie sie für weitere 16-18 Stunden bei 37 °C (insgesamt 20-22 Stunden) in den 37 °C-Inkubator zurück.
- Für Zytokinsekretionsassays oder Transkriptionsanalysen ist die Zugabe von Brefeldin A nicht zu verzehren und die Proben kontinuierlich bei 37 °C für 24-26 h zu inkubieren.
HINWEIS: Brefeldin A hemmt den Golgi-Apparat und unterdrückt dadurch die Zytokinsekretion und den Transport von Proteinen zur Zelloberfläche. Da die Sekretion oder Oberflächenexpression letztendlich zu einem Proteinverlust bzw. -abbau nach der Reinternalisierung führt, sind Exozytose-Inhibitoren für die intrazelluläre Akkumulation und Färbung von Zytokinen und einigen Aktivierungsmarkern (z. B. CD154) für Durchflusszytometrie-Studien unerlässlich. Brefeldin A stört jedoch auch physiologische zelluläre Prozesse, die im Transkriptom sichtbar sind, und macht Zytokinsekretionsassays aus Kulturüberständenungültig 20,21, was die Verwendung von zwei getrennten Reagenzgläsern (eines mit und eines ohne Brefeldin A) erforderlich macht, um diese Messungen parallel durchzuführen.
3. Vorbereitung der Proben für die Durchflusszytometrie
- Geben Sie 500 μl 0,5 M EDTA-Lösung in jedes Stimulationsröhrchen und inkubieren Sie die Probe 15 Minuten lang bei Raumtemperatur, um anhaftende Zellen von der Oberfläche der Röhrchen zu lösen.
- Übertragen Sie die Proben in neue 15-ml-Zentrifugenröhrchen. Spülen Sie die Stimulationsröhrchen mit 1 ml Erythrozyten-Lysepuffer, um die restlichen Blutzellen zu sammeln, und geben Sie dann den Puffer und die Zellen in dieselben 15-ml-Zentrifugenröhrchen.
- Zentrifugieren Sie die 15 mL Röhrchen für 7 min bei 600 x g. Entsorgen Sie den Überstand vorsichtig.
- Resuspendieren Sie das Blutzellenpellet in einem Erythrozyten-Lysepuffer (Abbildung 2A). Verwenden Sie 5 ml Erythrozyten-Lysepuffer für 500 μl Vollblut und 3 ml für 250 μl Vollblut.
HINWEIS: Optimale Erythrozyten-Lysebedingungen müssen für den verwendeten Puffer (intern hergestelltes oder kommerzielles Produkt) vorab bewertet werden. Die hier bereitgestellten Anweisungen wurden für den in der Materialtabelle aufgeführten Erythrozyten-Lysepuffer optimiert. - Inkubieren Sie die Proben bei Raumtemperatur, bis die Proben klar erscheinen (Abbildung 2B). Um eine Granulozytenlyse und -aggregation zu verhindern, darf die Inkubation 6 Minuten nicht überschreiten. Ein Indikator für eine erfolgreiche Erythrozytenlyse ist die Fähigkeit, die Zahlen und den Maßstab auf dem 15-ml-Röhrchen durch die Flüssigkeit hindurch zu sehen (Abbildung 2C).
- Zentrifugieren Sie die 15 mL Röhrchen für 7 min bei 600 x g. Entsorgen Sie den Überstand vorsichtig, wiederholen Sie die Schritte 3.4 - 3.5, wenn das Küvettenpellet noch auffällig rot ist.
- Resuspendieren Sie das Zellpellet in 1 mL HBSS und überführen Sie die Zellen in 2 mL Reaktionsröhrchen.
- Zentrifugieren Sie die 2 mL Röhrchen für 5 min bei 400 x g. Entsorgen Sie den Überstand vorsichtig.
- Führen Sie die durchflusszytometrische Färbung gemäß den Anweisungen des Herstellers für das intrazelluläre Färbekit und die verwendeten Antikörper durch.
HINWEIS: Das Antikörper-Panel, das zur Generierung des repräsentativen Datensatzes verwendet wurde, ist in Tabelle 3 zusammengefasst.
4. Vorbereitung der Proben für Zytokin-Assays
- Nach Schritt 2.6 wird das verdünnte Blut aus den Stimulationsröhrchen in 1,5-ml-Röhrchen überführt.
- Zentrifugieren Sie die 1,5 mL Röhrchen für 20 min bei 2000 x g. Pipettieren Sie den Überstand vorsichtig in ein frisches 1,5-ml-Röhrchen und verwenden Sie es entweder sofort für die Zytokinanalyse oder kryokonservieren Sie die Überstände bei -80 °C.
- Zentrifugieren Sie den Überstand erneut 5 Minuten lang bei ≥ 7000 x g (in 1,5-ml-Röhrchen), um vor der Analyse, insbesondere nach dem Auftauen, verbleibende Zelltrümmer zu entfernen. Führen Sie je nach Zytokin-Assay-Protokoll eine Vorverdünnung der Proben durch.
- Resuspendieren Sie das Zellpellet in 1 mL RNA-Schutzpuffer und kryokonservieren Sie es bei -80 °C für die anschließende RNA-Isolierung. Alternativ können Sie das Zellpellet in Lysepuffer resuspendieren, um eine sofortige RNA-Isolierung gemäß den Anweisungen für das RNA-Isolationskit (oder das hauseigene Protokoll) zu erhalten.
HINWEIS: Falls erforderlich, kann je nach nachfolgendem RNA-Verarbeitungsprotokoll ein Erythrozyten-Lyseschritt ähnlich 3.4-3.5 hinzugefügt werden, bevor der RNA-Schutzpuffer hinzugefügt wird.
Ergebnisse
Multimodale Analyse antigenspezifischer Immunantworten nach Vollblutstimulation mit Erreger-assoziierten Antigenen
Um einen repräsentativen Datensatz zu generieren, wurde ein gesunder erwachsener Spender ausgewählt, der seropositiv für HSV-1 und CMV war und eine SARS-CoV-2-Impfung erhalten hatte. Neben einer unstimulierten Kontrolle wurden folgende Stimuli wie oben beschrieben verwendet: Herpes-simplex-Virus 1 (HSV)-Lysat (Herstellerempfehlung, unveröffentlichte Daten), Cytomegalievirus (CMV) pp6522, schweres akutes respiratorisches Syndrom Coronavirus 2 (SARS-CoV-2) Prot_S 5,23, Aspergillus fumigatuslysat (ein ubiquitärer Umwelterreger)4,24, CRX-527 (ein Toll-like-Rezeptor-4-Stimulus auf der Basis von Lipopolysaccharid, der an sich keine T-Zellen aktivieren sollte)25 und CPI (Positivkontrolle für die Aktivierung von CD4+-T-Zellen, bestehend aus CMV-, Parainfluenzavirus- und Influenzaviruspeptiden)26. Die durchflusszytometrischen Daten und die Gating-Strategie sind in Abbildung 3 dargestellt. Im Allgemeinen wird empfohlen, so viele Lymphozyten wie möglich zu messen, wenn seltene Zellpopulationen (50.000 - 100.000 Lymphozyten) anvisiert werden, da die Präzision und Zuverlässigkeit der Messungen von der Gesamtzahl der Ereignisse abhängen27. Die unstimulierte Probe wird für das Gating einzelner Zelluntergruppen (z. B. CD3+CD4+-Zellen) verwendet. Die Gates für CD45RO und CCR7 für die Gedächtnis-T-Zell-Phänotypisierung sollten auf die gesamte CD3+CD4+-Population gesetzt und dann auf die Aktivierungsmarker-positiven Populationen übertragen werden, da die niedrigen Ereigniszahlen in letzteren oft eine eindeutige Identifizierung unterschiedlicher Populationen verhindern. Für einzelne Gates können geringfügige Anpassungen erforderlich sein, z. B. um Unterschiede in der Lebensfähigkeit der Lymphozyten zu berücksichtigen. Die Hochregulierung von CD154 (oder CD40-Liganden) wurde als globaler, konsistenter und schnell induzierter Aktivierungsmarker für T-Helferzellen beschrieben28,29. IFN-γ gilt als einer der prominentesten und Typ-1-spezifischen T-Zell-Aktivierungsmarker30,31. Wichtig ist, dass dieser Assay mit verschiedenen zusätzlichen Aktivierungs-, Erschöpfungs- und Zytokinmarkern getestet und veröffentlicht wurde (siehe Tabelle 4 und24).
Die Häufigkeiten der Aktivierungsmarker-positiven Populationen in der unstimulierten Stichprobe stellen einen unspezifischen Hintergrund dar und wurden von den Antigen-stimulierten Häufigkeiten abgezogen. Nach Subtraktion des unspezifischen Hintergrunds hatte der repräsentative Spender 0,75 % (HSV), 0,09 % (CMV), 0,06 % (SARS-CoV-2), 0,34 % (A. fumigatus), 0,00 % (CRX-527) bzw. 1,21 % (CPI) spezifische CD154+/CD3+CD4+ T-Helferzellen. Die IFN-γ-Expression kann auf ähnliche Weise analysiert werden, was zu 0,12 % (HSV), 0,07 % (CMV), 0,03 % (SARS-CoV-2), 0,04 % (A. fumigatus), 0 % (CRX-527) und 0,74 % (CPI) IFN-γ+/CD3+CD4+ -Zellen führt.
T-Zell-Populationen können weiter unterteilt werden in naive T-Zellen (T N, CD45RO-CCR7+), T-Zellen des zentralen Gedächtnisses (TCM, CD45RO+CCR7+), Effektor-Gedächtnis-T-Zellen (TEM, CD45RO+CCR7-) und Effektor-T-Zellen (und Effektor-Gedächtnis-T-Zellen, die CD45RA reexprimieren, TE/TEMRA, CD45RO-CCR7-). Unter den globalen CD3+CD4+ T-Zellen hatte der repräsentative Spender 38,34 %T N, 38,33 % TCM, 20,77 % TEM bzw. 2,56 % TE/TEMRA, wie anhand der unstimulierten Probe bestimmt wurde (Abbildung 3A). Unter den antigenspezifischen reaktiven T-Helferzellen (CD154+IFN-γ+) waren jedoch TCM und TEM mit Mittelwerten von 22,14 % bzw. 73,97 % die mit Abstand prominentesten Untergruppen.
Daten zu weiteren Leukozytenpopulationen wurden bereits mit dieser Methode veröffentlicht24. Die verwendeten Antikörperkombinationen sind in Tabelle 4 als weitere Referenz aufgeführt.
Um das volle Potenzial dieser Methode zu zeigen, wurde außerdem ein IFN-γ-ELISA an einem zweiten Satz stimulierter Proben (ohne Zusatz von Brefeldin A) durchgeführt. Um eine Überschreitung des Nachweisbereichs des IFN-γ-ELISA-Kits zu verhindern, wurde das Plasma aus CPI-stimulierten Proben 1:4 vorverdünnt. Die folgenden IFN-γ-Konzentrationen wurden gemessen und pro ml des Blutvolumens des Probanden normalisiert, d. h. sowohl um die Verdünnung in den Stimulationsröhrchen als auch um die Prä-ELISA-Verdünnungen korrigiert: 0 pg/ml (unstimuliert), 69,4 pg/ml (HSV), 471 pg/ml (CMV), 17,8 pg/ml (SARS-CoV-2), 61,9 pg/ml (A. fumigatus), 34,0 pg/ml (CRX-527) und 1958 pg/ml (CPI).
Schließlich wurde RNA aus denselben Proben mit konsistenten Ergebnissen isoliert. Die mittlere Ausbeute betrug 719 ng, wobei das mittlere Absorptionsverhältnis von 260 nm/280 nm 1,98 betrug.
Insgesamt zeigt dieser Datensatz, dass das vorgestellte Protokoll ein facettenreiches Auslesespektrum und eine gleichzeitige Analyse verschiedener infektionsassoziierter Antigene unter Verwendung eines minimalen Blutvolumens, d.h. insgesamt 8 ml für mehrere Stimuli und Auslesemodalitäten, ermöglicht.
Repräsentativer Datensatz für transkriptionelle Analysen zur Verfolgung der Impfantwort mit antigenstimuliertem Vollblut
Als Proof-of-Principle für transkriptionelle Studien, die an antigenstimuliertem Vollblut durchgeführt wurden, wurde 9 gesunden erwachsenen Probanden unmittelbar vor und 1 Monat nach der ersten Auffrischungsimpfung mit BNT162b2 (SARS-CoV-2-mRNA-Impfstoff)32,33 7-9 Monate nach der ersten Zwei-Dosen-Impfstoffserie Blut entnommen. Die durchschnittliche RNA-Ausbeute aus 500 μl unstimuliertem und Prot_S-stimuliertem Vollblut betrug 1,1 μg hochreiner RNA mit einem mittleren Absorptionsverhältnis von 260/280 von 1,99. Nach der nCounter-Analyse wurden die RNA-Zahlen auf die 12 Haushaltsgene des Panels (geometrisches Mittel) normalisiert. Danach wurde das Verhältnis von normalisierten mRNA-Zahlen in Prot_S-stimulierten Proben zu unstimulierten Hintergrundkontrollen für jeden Probanden und jedes Gen bestimmt. Es wurden mediane zu mediane Verhältnisse von Messungen nach und vor der Impfung bestimmt, und die Analyse der Signalanreicherung wurde mit dem in der Materialtabelle aufgeführten Softwarepaket durchgeführt. Die Anreicherung der kanonischen Signalwege wurde bei einem absoluten z-Score-Wert ≥ 1,25 und einem Benjamini-Hochberg-adjustierten p-Wert < 0,05 als signifikant angesehen. Signifikant unterschiedlich angereicherte Signalwege sind in Abbildung 4A zusammengefasst, und ein vereinfachtes Netzwerk von hintergrundadjustierten Veränderungen der Reaktion auf Prot_S nach der Impfung im Vergleich zu vor der Impfung ist in Abbildung 4B dargestellt. Darüber hinaus ist eine stärkere hintergrundadjustierte Induktion repräsentativer Gene im Zusammenhang mit der Antigen-präsentierenden Zellreifung und der Prot_S-induzierten T-Zell-Aktivierung nach der Auffrischungsimpfung in Abbildung 4C (p < 0,01-0,03) dargestellt. Schließlich wurde ein Anstieg der hintergrundadjustierten Prot_S-spezifischen Typ-1-T-Helferzellen (CD69+IFN-γ+) nach der Impfung bei den meisten Spendern durch Durchflusszytometrie mit einem zweiten Satz Stimulationsröhrchen bestätigt (p = 0,03, Abbildung 4D).
Vergleich von virusreaktiven T-Zell-Antworten in vollblutbasierten und kleinvolumigen Immunoassay-Protokollen
Als nächstes wurden die Häufigkeiten von virusreaktiven CD154+IFN-γ+ T-Helferzellen (CD3+CD4+ Zellen) bei gesunden Probanden unter Verwendung der Vollblut-Antigenstimulationsprotokolle (500 μL, WB) und des kleinen Volumens (250 μL, WBS) verglichen (Abbildung 5). Wie bereits berichtet34, wurden bei beiden Protokollen trotz dualer Co-Stimulation minimale unspezifische Hintergrundfrequenzen von CD154+IFN-γ+ -Zellen beobachtet (Mittelwerte 0,010% bzw. 0,011% für WB und WBS). Bemerkenswert ist, dass, obwohl unspezifische Hintergrundreaktionen von antigenspezifischen Reaktionen subtrahiert werden, sie immer noch zu einer erhöhten Ungenauigkeit des Assays beitragen, wie zuvor diskutiertwurde 27. Erhöhte Hintergrundsignale (d. h. >0,07-0,1 % CD154+ Th-Zellen oder >0,05 % CD154+IFN-γ+ Zellen) könnten auf eine Probenkontamination, eine akute Infektion des Probanden oder das Ergebnis einer unangemessenen präanalytischen Probenhandhabung hinweisen.
Die mittlere HSV-Lysat-reaktive T-Zell-Häufigkeit bei seropositiven Spendern (n = 5) betrug 0,151 % bzw. 0,107 % im WB- und WBS-System, verglichen mit 0,012 % bzw. 0,004 % bei seronegativen Spendern (n = 4). Mit dem CMV pp65-Peptidpool hatten seropositive Donatoren (n = 4) 0,041 % und 0,049 % reaktive T-Zellen im Vergleich zu 0,001 % und 0,004 % bei seronegativen Spendern (n = 5). Die Korrelationskoeffizienten von Lin betrugen 0,868 für HSV und 0,985 für CMV, was auf eine starke Korrelation hindeutet (Abbildung 5A). Insbesondere CMV-spezifische T-Zell-Tests können bei gesunden seropositiven Probanden, die keine kürzlichen Reaktivierungsereignisse hatten, negativ sein. Sowohl das Gesamt- als auch das Antigen-reaktive CD3+CD4+ T-Helferzell-Repertoire wurden anhand der CCR7- und CD45RO-Expression weiter differenziert (Abbildung 5B). Beruhigend ist, dass die Ergebnisse, die mit den WB- und WBS-Protokollen erzielt wurden, sowohl für die Gesamt- als auch für die Antigen-reaktive Population vergleichbar waren. Erwartungsgemäß war bei beiden Assays der Anteil differenzierter Gedächtnis-Th-Zellen (d.h. Effektor-Gedächtniszellen) bei den antigenreaktiven T-Zellen höher als bei der gesamten Th-Zell-Population. Erwartungsgemäß wurden nur wenige naive T-Zellen durch virale Stimulanzien aktiviert (Abbildung 5B).
Darüber hinaus wurden in einer weiteren Reihe von Experimenten stimulierte Kulturüberstände mittels IFN-γ ELISA analysiert (Abbildung 5C). Es wurde ein minimaler unspezifischer Hintergrund beobachtet (Mittelwert von 1,29 pg/mL bzw. 2,18 pg/mL in WB- bzw. WBS-Protokollen). Blutproben von HSV-seropositiven Spendern zeigten mittlere hintergrundadjustierte HSV-induzierte IFN-γ Konzentrationen von 111 pg/ml bzw. 125 pg/ml im WB- bzw. WBS-System. Im Gegensatz dazu lagen die IFN-γ-Konzentrationen in seronegativen Proben in beiden Systemen konstant unter 10 pg/ml (Lins Konkordanzkorrelationskoeffizient = 0,972, Abbildung 5C). Ebenso ergab die pp65-Stimulation von Blut von CMV-seropositiven Spendern mittlere IFN-γ-Konzentrationen von 258 pg/ml bzw. 272 pg/ml in WB- und WBS-Systemen, während in beiden Systemen mit seronegativen Proben eine minimale pp65-induzierte IFN-γ-Sekretion beobachtet wurde (Lin-Konkordanz-Korrelationskoeffizient = 0,953, Abbildung 5C).
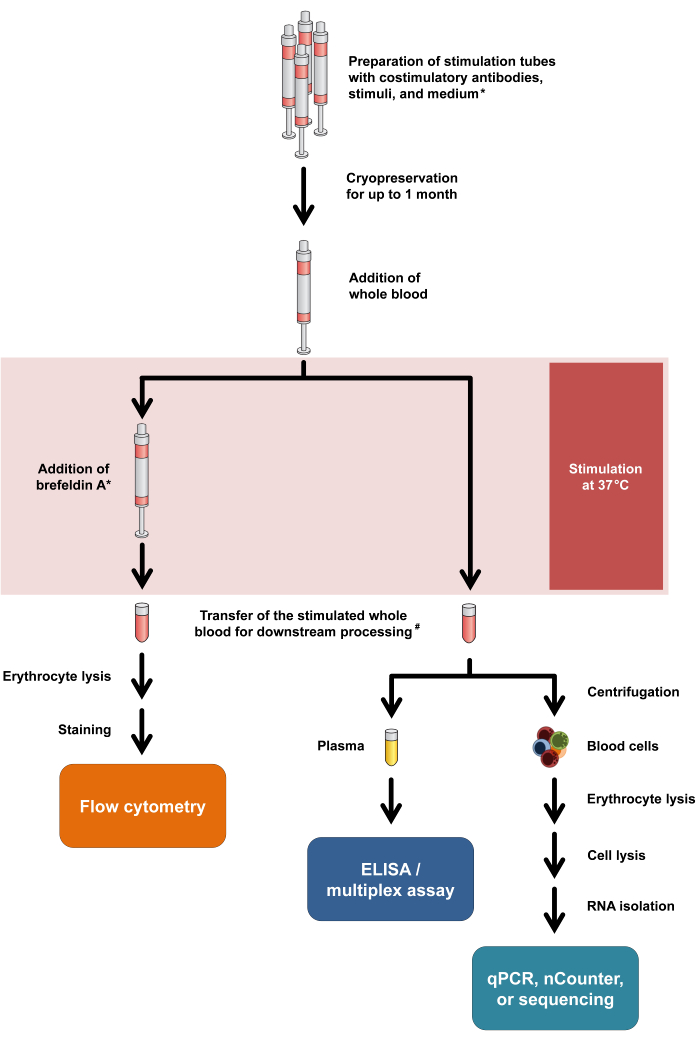
Abbildung 1: Flussdiagramm mit Zusammenfassung der experimentellen Verfahren und Messwerte. Sternchen bedeutet, dass Brefeldin A für einige T-Zell-Aktivierungsmarker (z. B. CD154) und die intrazelluläre Zytokinfärbung benötigt wird. Siehe Protokollschritte 2.5 und 2.6. #: Blut für die Durchflusszytometrie wird zunächst in 15-ml-Zentrifugenröhrchen für die Erythrozytenlyse überführt, während Blut für Zytokin-Assays und Transkriptomik in 1,5-ml-Mikrozentrifugenröhrchen überführt wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Erythrozytenlyse. Nachdem stimuliertes Blut in (A) Erythrozyten-Lysepuffer resuspendiert wurde, wird es inkubiert, bis (B) die Flüssigkeit klar erscheint, jedoch nicht länger als 6 Minuten. (C) Bei Verwendung eines abgestuften 15-ml-Röhrchens sollte dessen Zunder durch die zunehmend durchscheinende Probe sichtbar werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
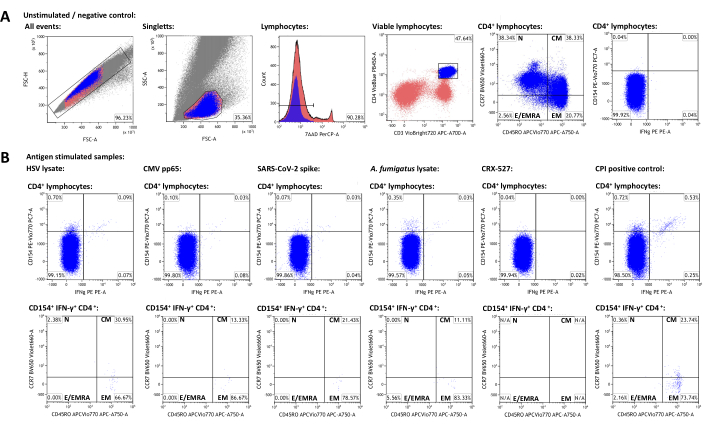
Abbildung 3: Repräsentativer Datensatz und durchflusszytometrische Gating-Schemata. (A) Singulett-Ereignisse werden durch FSC-A- und FSC-H-Eigenschaften identifiziert. Von diesen werden die Lymphozyten mit FSC-A und SSC-A gesteuert. Lymphozyten werden in CD3+CD4+ T-Helferzellen differenziert. CD45RO- und CCR7-Expressionsniveaus werden für die Phänotypisierung von Gedächtnis- und Effektorzellpopulationen verwendet. IFN-γ und CD154 wurden als Aktivierungsmarker verwendet. Die Gates wurden basierend auf der IFN-γ-CD154-Population in der unstimulierten Probe gesetzt. Anschließend wurden Gates auf die stimulierten Proben übertragen (B). Die Charakterisierung der Gedächtnispopulationen der aktivierten T-Zellen erfolgte durch Übertragung des CCR7/CD45RO-Quadrantengatters von der CD3+CD4+-Population auf die IFN-γ+CD154+CD3+CD4+-Populationen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
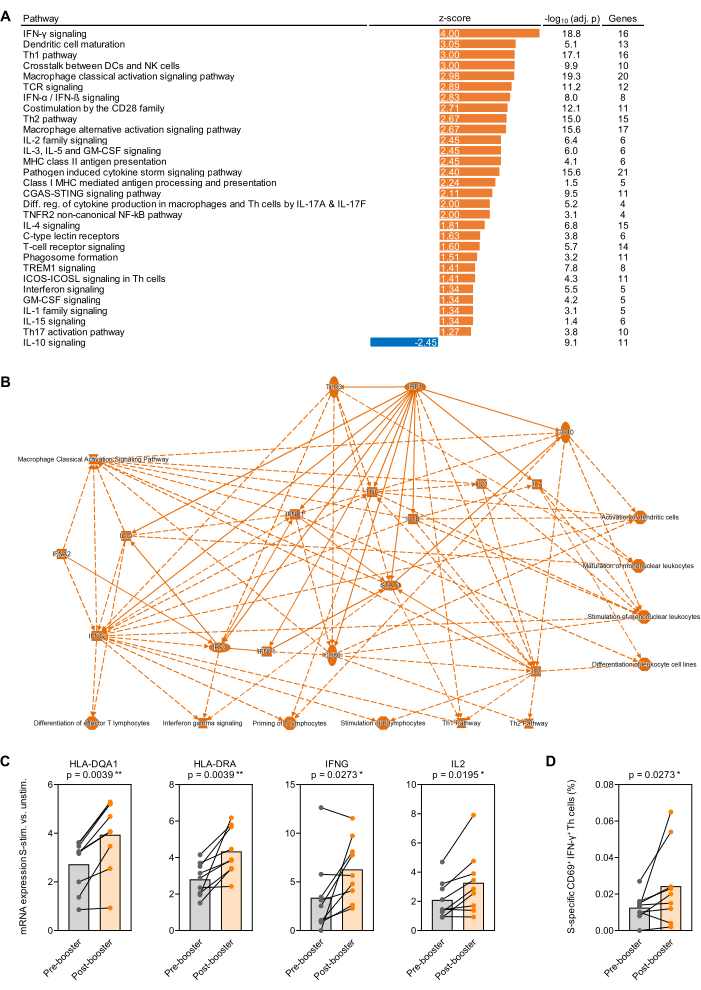
Abbildung 4: Antigen-induzierte transkriptionelle Veränderungen nach SARS-CoV-2-Auffrischungsimpfung. (A) Die hintergrundadjustierte Anreicherung der Transkriptionswege in SARS-CoV-2-Spike-Protein (Prot_S)-stimuliertem Vollblut nach und vor der (ersten) BNT162b2-mRNA-Auffrischungsimpfung wurde bei 9 gesunden erwachsenen Probanden analysiert. Immunbezogene kanonische Signalwege mit einem Benjamini-Hochberg-adjustierten (BH-adj.) p-Wert <0,05 (-log10[BH-adj. p] >1,3) und einem absoluten z-Score >1,25) sind dargestellt. (B) Vereinfachtes Netzwerk, das Gene und Signalwege zusammenfasst, die nach einer Auffrischungsimpfung stärker mit S-stimuliertem Vollblut angereichert sind als vor der Auffrischungsimpfung. (C) Hintergrundadjustierte Expressionsniveaus repräsentativer Gene, die mit der Antigenpräsentation und der T-Zell-Aktivierung in Prot_S-stimuliertem Vollblut vor und nach der Auffrischungsimpfung assoziiert sind. Gepaarter Wilcoxon-Test. (D) Hintergrundkorrigierte Häufigkeiten von Prot_S-spezifischen IFN-γ+CD69+ T-Zellen vor und nach der Auffrischungsimpfung. Gepaarter Wilcoxon-Test. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
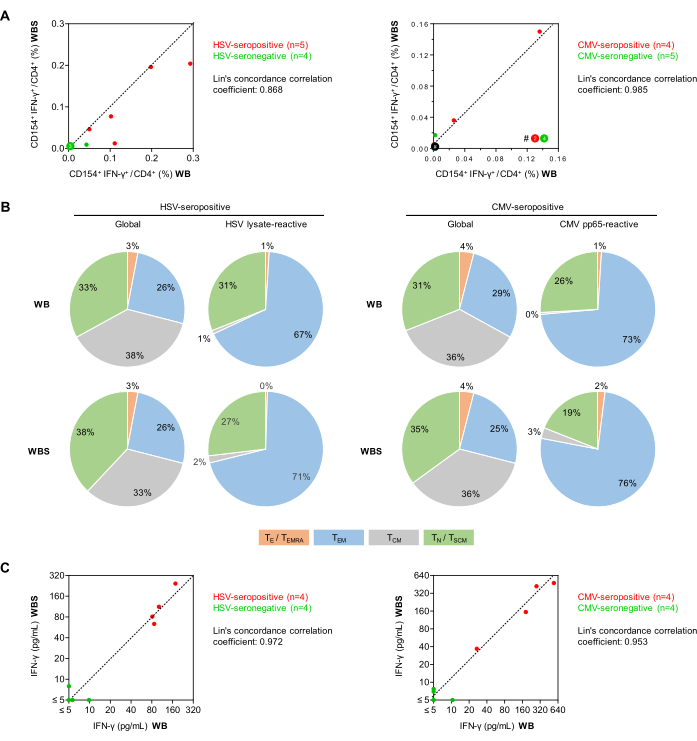
Abbildung 5: Antivirale T-Zell-Reaktivität in der Durchflusszytometrie und im ELISA. (A) Es werden Korrelationsdiagramme von hintergrundkorrigierten CD154+IFN-γ+/CD3+CD4+ T-Zellfrequenzen (Durchflusszytometrie) aus den etablierten (WB) und kleinvolumigen (WBS) Vollblutassays gezeigt. Grüne und rote Punkte stehen für Probanden, die seronegativ bzw. seropositiv für das getestete Virus sind. (B) Unter Verwendung der durchflusszytometrischen Beurteilung der CD45RO- und CCR7-Expression wurden Gedächtnis-/Effektor-T-Zell-Phänotypen zwischen globalen CD3+CD4+ T-Zellen und antigenreaktiven IFN-γ+CD154+CD3+CD4+ T-Zellen nach Stimulation mit HSV-Lysat oder CMV pp65 unter Verwendung von Blut von seropositiven Spendern (n = 5 bzw. 4) bestimmt. Mittelwertverteilungen werden angezeigt. Grün: naive T-Zellen (T N), CD45RO-CCR7+. Grau: T-Zellen mit Zentralspeicher (TCM), CD45RO+CCR7+. Blau: Effektor-Gedächtnis-T-Zellen (TEM), CD45RO+CCR7-. Orange: Effektor-T-Zellen und Effektor-Memory-T-Zellen, die CD45RA (TE/TEMRA) reexprimieren, CD45RO-CCR7-. (C) Korrelationsdiagramme der hintergrundkorrigierten IFN-γ-Freisetzung (ELISA), gemessen mit den WB- und WBS-Assays. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Inhalt der Stimulus-Sonde. Die Bestandskonzentrationen von Stimuli und Lösungsmitteln werden zusammengefasst. Der Vollbluttest (WB) wird mit 500 μl lithiumheparinisiertem Vollblut durchgeführt, während für die kleine Version (WBS) nur 250 μl Blut benötigt werden. Bei WSP sind alle Reagenzienvolumina halb so groß wie WB-Volumina. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Demografische Daten gesunder erwachsener Probanden, die zur Erstellung der repräsentativen Datensätze herangezogen wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Repräsentatives durchflusszytometrisches Panel für die T-Zell-Analyse. Dieses durchflusszytometrische Panel wurde verwendet, um den repräsentativen Datensatz zu generieren. Die Ergebnisse sind in Abbildung 3 detailliert dargestellt. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 4: Zuvor veröffentlichte durchflusszytometrische Panels. Daten, die diese Antikörperkombinationen verwenden, wurden zuvor von Tappe et al. veröffentlicht.24. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Antigenspezifische Immunoassays geben Einblicke in Wirt-Mikroben-Interaktionen, sind für die Impf- und Immuntherapieforschung von zentraler Bedeutung und werden zunehmend als diagnostische und prognostische Modalitäten bei Patienten mit opportunistischen Infektionen anerkannt35. Dieses Protokoll beschreibt ein einfaches Antigenstimulationssystem, das eine robuste und multimodale Analyse der antigenspezifischen Immunität mit minimalen Blutvolumina (250-500 μL pro Antigen) ermöglicht. Das verkleinerte 250-μl-Protokoll ergab eine hervorragende Korrelation von antigenspezifischen T-Zell-Frequenzen, Phänotypen und Zytokinproduktion im Vergleich zum zuvor etablierten 500-μl-Protokoll. Trotz der Verfügbarkeit von Lösungen mit kleinem Volumen für einige Schritte der Probenverarbeitung36 kann nach Kenntnis der Autoren kein derzeit verfügbares kommerzielles System die Antigenstimulation und die facettenreiche funktionelle Analyse von T-Zell-gesteuerten funktionellen Immunantworten durch Durchflusszytometrie, Zytokinfreisetzungsassays und Transkriptomik aus Blutvolumina von 250-500 μl zuverlässig unterstützen. Das am weitesten verbreitete kommerzielle System, das ein ähnliches Spektrum von Forschungsanwendungen ermöglicht, verwendet ein Blutvolumen von 1 ml in einer Stimulationsumgebung von 3 ml, was zu erheblich höheren Kosten und Mengen an benötigten Antigenen im Vergleich zu dem hier vorgestellten Protokoll führt 13,14,15.
Trotz kontinuierlicher Optimierung von Vollblut-basierten Protokollen für die durchflusszytometrische Quantifizierung von antigenspezifischen T-Zellen 6,37,38 haben durchflusszytometrische Messungen mehrere Nachteile. Insbesondere sind sie nach wie vor aufwendig und aufgrund der erheblichen Variabilität zwischen den Bedienern (z. B. des subjektiven Gating-Prozesses) und der unterschiedlichen Gerätekonfigurationen, Kompensationsprotokolle und Erfassungsparameter zwischen den Labors schwer zu standardisieren. Obwohl eine standardisierte Befundung39 und die Verwendung automatisierter Analyse- und Gating-Software die Standardisierung und Vergleichbarkeit von zunehmend komplexeren mehrfarbigen Datensätzen 40,41 verbessern könnten, wurde das hier beschriebene Stimulationsprotokoll so konzipiert, dass es verschiedene nicht-durchflusszytometrische Auslesemodalitäten aufnehmen kann.
Insbesondere Zytokinfreisetzungsassays können mit geringem Zeitaufwand und relativ kostengünstigen Geräten durchgeführt werden und sind für klinische Routineanwendungen oft leicht standardisiert. Darüber hinaus kann, wie in früheren Studien mit diesem Protokoll gezeigt wurde, mit modernen Multiplex-Assays eine Vielzahl von Zytokinantworten aus minimalen Probenvolumina gemessen werden, was eine Profilerstellung komplexer Zytokinsignaturen in Forschungsumgebungen ermöglicht24,42. Bemerkenswert ist, dass dieses robuste Protokoll mit dualer Co-Stimulation eine zuverlässige Quantifizierung antigenspezifischer Zytokinantworten bei nicht-lymphopenischen Patienten (>800 Lymphozyten/μl Blut) ermöglicht, selbst bei Patienten, die iatrogene Immunsuppression erhalten26,34. Ein Nachteil von Zytokin-Release-Assays, insbesondere bei Patienten mit Leukopenie, ist, dass sekretierte Zytokine nicht auf einzelne Zellpopulationen zurückgeführt werden können. In einigen Fällen kann dies durch den Einsatz von zellspezifischen Stimuli gemildert werden, sofern verfügbar. Eine Kombination der Zytokinkonzentrationen mit anderen Auslesemodalitäten und/oder eine Anpassung der Zytokinantworten auf der Grundlage der klinischen Hämatologie (d. h. vollständiges Blutbild mit Leukozytendifferenzierung) kann jedoch erforderlich sein. Insbesondere ermöglicht das hier vorgestellte Protokoll eine Kombination von Zytokinauslesungen und transkriptionellen Signaturen aus derselben Probe, wodurch eine konkordante Analyse von gut definierten transkriptionellen Aktivierungsmarkern ermöglicht wird, die den globalen Zytokinsignaturen einen zellulären Kontext und Spezifität hinzufügen könnten.
Ein zukünftiger Schritt hin zu einer vollständigen Standardisierung und noch besseren klinischen Praktikabilität wäre die vollständige Automatisierung dieser Assays von der Probenverarbeitung bis zur Analytauslesung. Auch wenn die präzise automatisierte Isolierung einzelner Zellpopulationen erfolgreich etabliert wurde43,44, erfordert die antigenspezifische T-Zell-Analyse immer noch intermittierende Handgriffe durch das Laborpersonal. Der Wegfall der Zellisolierung und des Umgangs mit anfälligen PBMC sowie die Verwendung kommerzieller, automatisierungsfähiger Stimulationsröhrchen könnten jedoch die Implementierung einfacher, vollautomatischer Vollblut-basierter Arbeitsabläufe für funktionelle Immunoassays erleichtern.
Insgesamt bergen vielseitige Vollblut-basierte Protokolle, wie das hierin vorgestellte, ein erhebliches Versprechen, die Anwendungen von antigenspezifischen funktionellen Immunoassays auf neue Patientenkohorten und Forschungsbereiche auszuweiten, einschließlich präklinischer Studien an Kleintieren. Antigenspezifische funktionelle Immunoassays sind in Mausmodellen derzeit weitgehend nicht durchführbar oder erfordern das Pooling von Blut mehrerer Tiere und/oder die Verwendung von nicht standardisierten Zellextrakten wie z.B. Splenozyten. Angesichts des wachsenden Interesses an immuntherapeutischen Interventionen zur Stärkung der Wirtsabwehr gegen opportunistische Infektionen (z. B. Immun-Checkpoint-Inhibitoren, hämatopoetische Wachstumsfaktoren, Zytokine usw.) und des Anstiegs innovativer Impftechnologien wird erwartet, dass antigenspezifische funktionelle Immunoassays eine zunehmende Rolle sowohl in der präklinischen Erforschung von Infektionskrankheiten als auch in der klinischen Anwendung in verschiedenen Patientenpopulationen spielen werden. Das hier vorgestellte robuste, kostengünstige und einfach zu bedienende Antigenstimulationssystem mit geringem Volumen kann umfassende antigenspezifische Immunanalysen in unerschlossenen Bereichen ermöglichen. Darüber hinaus könnte die präanalytische Robustheit dieses einfachen Protokolls Möglichkeiten für eine verbesserte Integration von Immunoassay-Anwendungen in die klinische Routine schaffen, die es uns ermöglicht, einem personalisierten, biomarkergesteuerten Management von Infektionskrankheiten einen Schritt näher zu kommen.
Offenlegungen
Es bestehen keine Interessenkonflikte, die von den Autoren offenzulegen sind.
Danksagungen
Wir danken den Abteilungen Klinische Chemie und Infektionsserologie am Institut für Laboratoriumsmedizin und Mikrobiologie des Universitätsklinikums Augsburg für die Durchführung der Serum-Antikörpermessungen. Wir danken Dr. Friederike Liesche-Starnecker und dem Institut für Pathologie und Molekulare Diagnostik des Universitätsklinikums Augsburg für die Bereitstellung von Transkriptomik-Einrichtungen. Wir danken Marie Freitag vom Universitätsklinikum Augsburg für die Unterstützung bei der Impflogistik und der Probenbeschaffung. Wir danken Dr. Olaf Kniemeyer und dem Hans-Knöll-Institut in Jena für die Bereitstellung von Aspergillus fumigatus lysat. Die Forschungsinitiative Bay-VOC (Förderkennzeichen GE2-2452-200-D37666/2022), das Bayerische Staatsministerium für Wissenschaft und Kunst sowie die Universität Augsburg unterstützten diese Arbeiten.
Materialien
| Name | Company | Catalog Number | Comments |
| 7-AAD Solution | Miltenyi Biotec | 130-111-568 | |
| Anti-IFN-g-PE, human, REA600, 100 tests | Miltenyi Biotec | 130-113-498 | |
| Aspergillus fumigatus lysate | N/A | N/A | Kindly provided by the Hans-Knoell Institute Jena, Germany |
| Brilliant Violet 650 anti-human CD197 (CCR7) Antibody | BioLegend | 353234 | |
| Buffer EL | Qiagen | 79217 | Erythrocyte lysis buffer |
| CD154-PE-Vio770, human, REA238, 100 tests | Miltenyi Biotec | 130-113-614 | |
| CD279 (PD1)-VioBright 515, human, 100 tests | Miltenyi Biotec | 130-120-386 | |
| CD28 pure, human - functional grade | Miltenyi Biotec | 130-093-375 | |
| CD3-VioBright R720, human, REA613, 100 tests | Miltenyi Biotec | 130-127-377 | |
| CD45RO-APC-Vio770, human, REA611, 100 tests | Miltenyi Biotec | 130-113-557 | |
| CD49d pure, human | Miltenyi Biotec | 130-093-279 | |
| CD4-VioBlue, human, REA623, 100 tests | Miltenyi Biotec | 130-114-534 | |
| CD69-PE-Vio615, human, REA824, 100 tests | Miltenyi Biotec | 130-112-617 | |
| CO2 Incubator | PHCbi | MCO-170AICD-PE | |
| CPI Positive Control Solution | ImmunoSpot | CTL-CPI-001 | |
| CRX-527 | Invivogen | tlrl-crx527 | |
| CytExpert Acquisition and Analysis Software | Beckman Coulter | Version 2.4 | Flow cytometer operating softwware |
| CytoFlex S B2-R3-V4-Y4 | Beckman Coulter | B75408 | Flow cytometer |
| GraphPad Prism | GraphPad | Version 10.1.0 | |
| Herpes simplex virus 1 lysate | AID Autoimmun Diagnostika | ELSP 5916K | |
| HU IFN G Uncoated ELISA 2X96T PLT | Thermo Fisher Scientific | 88-7316-22 | |
| HydroFLex Microplate Washer | Tecan | 30220085 | |
| Infinite M Plex | Tecan | 30213614 | Multimode Microplate Reader |
| Kaluza Analysis Software | Beckman Coulter | Version 2.1.00003.20057 | Flow cytometric data analysis software |
| MACS Inside Stain Kit | Miltenyi Biotec | 130-090-477 | |
| Mastercycler X50l | Eppendorf | 6303000010 | |
| Nanodrop One | Thermo Fisher Scientific | ND-ONE-W | |
| nCounter Sprint Cartridge | Nanostring | 100078 | |
| nCounter Sprint Profiler | Nanostring | 100170 | |
| nCounter Sprint Reagent Pack | Nanostring | 100077 | |
| nSolver | Nanostring | Version 4.0 | Nanostring nCounter data analysis software |
| Octeniderm farblos 250 ml FL | Schuelke | 118211 | |
| Omnifix F Solo, 1 ml, ohne Kanüle, 3-teilig | Braun | 9161406V | Syringes |
| PepTivator CMV pp65, human, 60nmol | Miltenyi Biotec | 130-093-435 | |
| PepTivator SARS-CoV-2 Prot_S, research grade, for stimulation of 1×108 cells | Miltenyi Biotec | 130-126-700 | |
| QIAshredder | Qiagen | 79654 | |
| RNA Protect | Qiagen | 76526 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| RPMI 1640 Medium, GlutaMAX Supplement, HEPES | Thermo Fisher Scientific | 72400047 | |
| Safe 2020 1.5 Microbiological Safety Cabinet | Thermo Fisher Scientific | 51026959 | |
| S-Monovette lithium-heparin, 4.9 ml | Sarstedt | 04.1939.001 | Blood collection tubes |
| S-Monovette neutral, 2.7 ml | Sarstedt | 05.1729 | Stimulation environment tubes |
| Sterican Safety G 19 x 1 1/2'' 1,1 x 40 mm | Braun | 4670052S-01 | Needles |
| Stop Solution, 100 ML | Thermo Fisher Scientific | BMS409.0100 | |
| Wash Buffer 20X, 500 ML | Thermo Fisher Scientific | BMS408.0500 | |
| Water, 1 l | Carl Roth | 3478.1 | |
| XT Hs Exhaustion CSO | Nanostring | 115000466 | Nanostring Immune Exhaustion Panel |
Referenzen
- Mckenna, K. C., Beatty, K. M., Vicetti Miguel, R., Bilonick, R. A. Delayed processing of blood increases the frequency of activated CD11b+ CD15+ granulocytes which inhibit t cell function. J Immunol Methods. 341 (1-2), 68-75 (2009).
- Doberne, D., Gaur, R. L., Banaei, N. Preanalytical delay reduces sensitivity of quantiferon-tb gold in-tube assay for detection of latent tuberculosis infection. J Clin Microbiol. 49 (8), 3061-3064 (2011).
- Corbiere, V., et al. A semi high-throughput whole blood-based flow cytometry assay to detect and monitor bordetella pertussis-specific th1, th2 and th17 responses. Front Immunol. 14, 1101366 (2023).
- Lauruschkat, C. D., et al. Development of a simple and robust whole blood assay with dual co-stimulation to quantify the release of t-cellular signature cytokines in response to aspergillus fumigatus antigens. J Fungi (Basel). 7 (6), 462 (2021).
- Riou, C., et al. simplified whole blood-based multiparameter assay to quantify and phenotype SARS-COV-2-specific T-cells. Eur Respir J. 59 (1), 2100285 (2022).
- Weis, P., et al. Development and evaluation of a whole blood-based approach for flow cytometric quantification of CD154+ mould-reactive T cells. Med Mycol. 58 (2), 187-196 (2020).
- Boyum, A. Isolation of lymphocytes, granulocytes and macrophages. Scand J Immunol. 5, 9-15 (1976).
- Chen, G., Wang, H., Wang, Y. Clinical application of quantiferon-tb gold in-tube in the diagnosis and treatment of tuberculosis. Eur J Clin Microbiol Infect Dis. 39 (4), 607-612 (2020).
- Manuel, O., Kumar, D. Quantiferon-tb gold assay for the diagnosis of latent tuberculosis infection. Expert Rev Mol Diagn. 8 (3), 247-256 (2008).
- Inthawong, M., et al. A whole blood intracellular cytokine assay optimised for field site studies demonstrates polyfunctionality of CD4+ t cells in acute scrub typhus. PLoS Negl Trop Dis. 17 (3), e0010905 (2023).
- Johnson, S. A., et al. Evaluation of quantiferon SARS-COV-2 interferon-gamma release assay following SARS-COV-2 infection and vaccination. Clin Exp Immunol. 212 (3), 249-261 (2023).
- Kruttgen, A., et al. Evaluation of the quantiferon SARS-COV-2 interferon-ɣ release assay in mrna-1273 vaccinated health care workers. J Virol Methods. 298, 114295 (2021).
- Duffy, D., et al. Standardized whole blood stimulation improves immunomonitoring of induced immune responses in multi-center study. Clin Immunol. 183, 325-335 (2017).
- Duffy, D., et al. Functional analysis via standardized whole-blood stimulation systems defines the boundaries of a healthy immune response to complex stimuli. Immunity. 40 (3), 436-450 (2014).
- Smith, N., et al. Defective activation and regulation of type i interferon immunity is associated with increasing COVID-19 severity. Nat Commun. 13 (1), 7254 (2022).
- Lauruschkat, C. D., et al. CD4+ T cells are the major predictor of hcmv control in allogeneic stem cell transplant recipients on letermovir prophylaxis. Front Immunol. 14, 1148841 (2023).
- Pelchen-Matthews, A., Parsons, I. J., Marsh, M. Phorbol ester-induced downregulation of cd4 is a multistep process involving dissociation from p56lck, increased association with clathrin-coated pits, and altered endosomal sorting. J Exp Med. 178 (4), 1209-1222 (1993).
- Boise, L. H., et al. CD28 costimulation can promote t cell survival by enhancing the expression of bcl-xl. Immunity. 3 (1), 87-98 (1995).
- Udagawa, T., Woodside, D. G., Mcintyre, B. W. Alpha 4 beta 1 (CD49d/CD29) integrin costimulation of human t cells enhances transcription factor and cytokine induction in the absence of altered sensitivity to anti-cd3 stimulation. J Immunol. 157 (5), 1965-1972 (1996).
- Baumann, J., et al. Golgi stress-induced transcriptional changes mediated by mapk signaling and three ets transcription factors regulate mcl1 splicing. Mol Biol Cell. 29 (1), 42-52 (2018).
- Miller, S. G., Carnell, L., Moore, H. H. Post-golgi membrane traffic: Brefeldin a inhibits export from distal golgi compartments to the cell surface but not recycling. J Cell Biol. 118 (2), 267-283 (1992).
- Zandvliet, M. L., et al. Co-ordinated isolation of CD8(+) and CD4(+) t cells recognizing a broad repertoire of cytomegalovirus pp65 and ie1 epitopes for highly specific adoptive immunotherapy. Cytotherapy. 12 (7), 933-944 (2010).
- Kramer, K. J., et al. Single-cell profiling of the antigen-specific response to bnt162b2 sars-cov-2 rna vaccine. Nat Commun. 13 (1), 3466 (2022).
- Tappe, B., et al. COVID-19 patients share common, corticosteroid-independent features of impaired host immunity to pathogenic molds. Front Immunol. 13, 954985 (2022).
- Stover, A. G., et al. Structure-activity relationship of synthetic toll-like receptor 4 agonists. J Biol Chem. 279 (6), 4440-4449 (2004).
- Page, L., et al. Impact of immunosuppressive and antifungal drugs on pbmc- and whole blood-based flow cytometric CD154(+) Aspergillus fumigatus specific t-cell quantification. Med Microbiol Immunol. 209 (5), 579-592 (2020).
- Hedley, B. D., Keeney, M. Technical issues: Flow cytometry and rare event analysis. Int J Lab Hematol. 35 (3), 344-350 (2013).
- Bacher, P., Scheffold, A. Flow-cytometric analysis of rare antigen-specific T cells. Cytometry A. 83 (8), 692-701 (2013).
- Frentsch, M., et al. Direct access to CD4+ t cells specific for defined antigens according to cd154 expression. Nat Med. 11 (10), 1118-1124 (2005).
- Osum, K. C., Jenkins, M. K. Toward a general model of CD4(+) t cell subset specification and memory cell formation. Immunity. 56 (3), 475-484 (2023).
- Lighvani, A. A., et al. T-bet is rapidly induced by interferon-gamma in lymphoid and myeloid cells. Proc Natl Acad Sci U S A. 98 (26), 15137-15142 (2001).
- Lamb, Y. N. Bnt162b2 mrna COVID-19 vaccine: First approval. Drugs. 81 (4), 495-501 (2021).
- Arbel, R., et al. Bnt162b2 vaccine booster and mortality due to COVID-19. N Engl J Med. 385 (26), 2413-2420 (2021).
- Lauruschkat, C. D., et al. T-cell immune surveillance in allogenic stem cell transplant recipients: Are whole blood-based assays ready to challenge elispot. Open Forum Infect Dis. 8 (1), 547 (2021).
- Albert-Vega, C., et al. Immune functional assays, from custom to standardized tests for precision medicine. Front Immunol. 9, 2367 (2018).
- . Biolegend Available from: https://www.biolegend.com/de-de/products/dry-human-aim-assay-cocktail-kit-23591?GroupID=GROUP28 (2024)
- Lauruschkat, C. D., et al. Susceptibility of a. Fumigatus-specific t-cell assays to pre-analytic blood storage and pbmc cryopreservation greatly depends on readout platform and analytes. Mycoses. 61 (8), 549-560 (2018).
- Wurster, S., et al. Quantification of a. Fumigatus-specific cd154+ t-cells-preanalytic considerations. Med Mycol. 55 (2), 223-227 (2017).
- Britten, C. M., et al. T cell assays and miata: The essential minimum for maximum impact. Immunity. 37 (1), 1-2 (2012).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
- Le Lann, L., et al. Standardization procedure for flow cytometry data harmonization in prospective multicenter studies. Sci Rep. 10 (1), 11567 (2020).
- Lauruschkat, C. D., et al. Chronic occupational mold exposure drives expansion of aspergillus-reactive type 1 and type 2 t-helper cell responses. J Fungi (Basel). 7 (9), 698 (2021).
- Pascual-Garcia, M., et al. Lif regulates cxcl9 in tumor-associated macrophages and prevents cd8(+) t cell tumor-infiltration impairing anti-pd1 therapy. Nat Commun. 10 (1), 2416 (2019).
- Paull, D., et al. Automated, high-throughput derivation, characterization and differentiation of induced pluripotent stem cells. Nat Meth. 12 (9), 885-892 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten