Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Rasterkraftmikroskopische Messungen von Knorpel in intakten und regenerierenden Axolotl-Gliedmaßen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
In diesem Protokoll zeigen wir, wie Axolotl-Gewebe für die Rasterkraftmikroskopie (AFM) vorbereitet und Indentationsmessungen in intaktem und regenerierendem Gliedmaßenknorpel durchgeführt werden können.
Zusammenfassung
Mechanische Kräfte liefern wichtige Signale für die normale Zellfunktion und Musterbildung in sich entwickelnden Geweben, und ihre Rolle wurde während der Embryogenese und Pathogenese umfassend untersucht. Vergleichsweise wenig ist über diese Signale während der Regeneration von Tieren bekannt.
Der Axolotl ist ein wichtiger Modellorganismus für die Erforschung der Regeneration, da er in der Lage ist, viele Organe und Gewebe nach Verletzungen, einschließlich fehlendem Knorpel und Knochen, vollständig wiederherzustellen. Aufgrund seiner entscheidenden Rolle als wichtigstes Stützgewebe im Wirbeltierkörper erfordert die Wiedererlangung der Skelettfunktion während der Regeneration sowohl die Wiederherstellung der fehlenden Strukturen als auch deren mechanische Eigenschaften. Dieses Protokoll beschreibt eine Methode zur Aufbereitung von Axolotl-Gliedmaßenproben für die Rasterkraftmikroskopie (Rasterkraftmikroskopie), die den Goldstandard für die Untersuchung mechanischer Eigenschaften von Zellen und Geweben mit hoher räumlicher Auflösung darstellt.
Unter Ausnutzung der regenerativen Fähigkeiten des Axolotls wurde in dieser Studie die Steifigkeit des Gliedmaßenknorpels während der Homöostase und in zwei Stadien der Gliedmaßenregeneration gemessen: der Gewebehistolyse und der Knorpelkondensation. Wir zeigen, dass AFM ein wertvolles Werkzeug ist, um Einblicke in die dynamische Gewebeumstrukturierung und die mechanischen Veränderungen, die während der Regeneration auftreten, zu gewinnen.
Einleitung
Das Skelett, insbesondere Knorpel und Knochen, stellt bei Wirbeltieren die wichtigste mechanische Unterstützung für die Weichteile des Körpers dar. Daher ist es wahrscheinlich, dass jede Beschädigung des Skelettsystems die Funktionalität und sogar das Überleben stark beeinträchtigt. Beim Menschen sind Knochenbrüche eine der häufigsten traumatischen Verletzungen1, von denen die meisten innerhalb weniger Wochen heilen, aber 5 % bis 10 % von ihnen verzögern die Heilung oder erholen sich nie vollständig 2,3. Darüber hinaus ist der Mensch nicht in der Lage, sich von einem ausgedehnten Knochen- oder Knorpelverlust zu erholen 4,5. Einige Salamander können jedoch eine Vielzahl von Körperstrukturen regenerieren, einschließlich ganzer Gliedmaßen6, was sie zu einem idealen Modell für die Untersuchung der Skelettregeneration macht.
Der Axolotl (Ambystoma mexicanum) ist eine Salamanderart, bei der die Regeneration der Gliedmaßen ausgiebig untersucht wurde. Dieser Prozess findet in vier aufeinanderfolgenden, aber sich überschneidenden Hauptphasen statt: 1) Wundheilung, 2) Entzündung/Histolyse, 3) Blastembildung und 4) Blastemwachstum/-differenzierung (überprüft in 7,8). Nach der Amputation wandern die Keratinozyten, die an die Verletzungsstelle grenzen, schnell, verschließen die Wunde und bilden das Wundepithel (WE). Bei der anschließenden Entzündung und Histolyse werden Krankheitserreger eliminiert, Ablagerungen und geschädigte Zellen beseitigt und die extrazelluläre Matrix (EZM) unter der Amputationsoberfläche wieder aufgebaut9. Die Gewebehistolyse ist essentiell für die Regeneration der Gliedmaßen10, wobei die Sekretion proteolytischer Enzyme nicht nur für den gesamten EZM-Umbau entscheidend ist, sondern auch für die Freisetzung der Zellen, aus denen das Blastem entsteht, und um bioaktive Moleküle freizusetzen, die in der EZM selbst sequestriert werden8. Tatsächlich haben Studien in vielen regenerativen Kontexten und Modellorganismen gezeigt, dass die einzigartigen Materialeigenschaften der EZM während der Histolyse in der Lage sind, Dedifferenzierungsprozesse zu induzieren oder die Wanderung von Zellen in Richtung der Verletzungsstelle zu lenken (überprüft in11). Darüber hinaus hat sich gezeigt, dass die Resorption von verkalktem Gewebe in den späten Stadien der Histolyse der Schlüssel für die korrekte Integration neu gebildeter Skelettelemente der Gliedmaßenist 12. Nach der Histolysephase wird das Blastem unter dem Wundepithel (WE) als Anhäufung von undifferenzierten, mehrlinienigen Vorläuferzellen gebildet, die aus dedifferenzierten reifen Gewebezellen oder residenten Stammzellen resultieren. Blastemzellen vermehren sich und differenzieren sich in alle fehlenden Zelltypen. Schließlich findet die Morphogenese der Gliedmaßen statt, bei der das Skelettgewebe durch die Kondensation von Chondroprogenitoren, die aus periskeletalen Zellen und transdifferenzierten dermalen Fibroblasten gewonnen werden, regeneriert wird 13,14,15.
Obwohl viele der biochemischen Signale, die Veränderungen in der Zellidentität und der EZM-Zusammensetzung regulieren, identifiziert wurden 10,13,14,16,17,18, blieben die mechanischen Eigenschaften des Gewebes während der verschiedenen Phasen der Regeneration der Gliedmaßen sowie ihr Einfluss auf die Regeneration weitgehend unerforscht. Viele Studien haben gezeigt, dass Zellen mechanische Signale wahrnehmen und integrieren, die ihr Schicksal und Verhalten in verschiedenen Kontexten regulieren (überprüft in 19,20). Daher wird die Ergänzung unseres zellulären und molekularen Wissens über die Regeneration von Gliedmaßen durch gewebemechanische Messungen unser Verständnis dieser Prozesse erheblich verbessern.
Es wurden verschiedene Techniken entwickelt, die eine mechanische Charakterisierung und Kraftmanipulation biologischer Proben ermöglichen21. Unter diesen Techniken hat sich die Rasterkraftmikroskopie (AFM) zum Goldstandard in der Mechanobiologie entwickelt, bei der die viskoelastischen Eigenschaften biologischer Proben mit hoher räumlicher Auflösung durch Eindrücken mit einem hochempfindlichen Kraftsensor, dem AFM-Cantilever, untersucht werden. Da diese Technik einen direkten Kontakt mit der Probe erfordert, werden in der Regel Gewebeschnitte erzeugt, was in einigen Fällen eine Herausforderung darstellen kann. Daher müssen die Präparationsbedingungen für jede einzelne Probe angepasst und optimiert werden, damit sie so nah wie möglich an den physiologischen Bedingungen bleiben kann und minimale Artefakte erzeugt werden23. Dieses Protokoll beschreibt, wie die Gewebesteifigkeit in Axolotl-Gliedmaßen mit Hilfe von AFM gemessen wird, wobei der Schwerpunkt auf Knorpelgewebe in intaktem Zustand, während der Histolyse und in Knorpelkondensationsstadien liegt (Abbildung 1 und Abbildung 2). Diese Methode kann auch für die Messung anderer Gewebetypen erweitert werden.
Protokoll
Axolotl (Ambystoma mexicanum) wurden in der Axolotl-Anlage des Zentrums für Regenerative Therapien Dresden (CRTD) der Technischen Universität Dresden (TUD) gezüchtet. Eine vollständige Beschreibung der Haltungsbedingungen finden Sie unter24. Kurz gesagt, die Zimmer wurden bei 20-22 °C mit einem 12/12 h Tag-Nacht-Zyklus gehalten. Alle Handhabungen und chirurgischen Eingriffe wurden in Übereinstimmung mit den Richtlinien der lokalen Ethikkommission durchgeführt und von der Landesdirektion Sachsen, Deutschland, genehmigt.
In dieser Studie wurden für alle Experimente weiße (d/d) Axolotl verwendet, ein natürlich vorkommender Mutantenstamm ohne Körperpigmentierung (wenige bis keine Melanophoren und Xanthophoren), mit Iridophoren nur in der Iris der Augen. In dieser Studie wurden Axolotl mit einer Größe von 8-15 cm von der Schnauze bis zum Schwanz (5-7 Monate alt) ohne geschlechtsspezifische Verzerrung verwendet.
1. Vorbereitung
- Bereiten Sie eine 10%ige (w/v) Benzocain-Stammlösung vor, die für die Anästhesie und Euthanasie der Axolotl verwendet wird (siehe unten). Mischen Sie dazu in einer volumetrischen Flasche 50 g Benzocain mit 100% Ethanol, bis der Grenzwert von 500 mL erreicht ist.
- Bereiten Sie eine 0,03%ige (w/v) Benzocain-Stammlösung vor.
- Mischen Sie für 1 l 50 ml 10x Tris-gepufferte Kochsalzlösung (TBS) mit 30 ml 10 % (w/v) Benzocain und 5 ml 4000 % (w/v) Holtfreter-Lösungen mit 915 mL destilliertem Wasser und mischen Sie es über Nacht mit einem magnetischen Rührstab.
- Für 1 l 10x TBS-Lösung mischen Sie 24,2 g Trizma-Base und 90 g NaCl mit 990 mL deionisiertem Wasser. Mit einem magnetischen Rührstäbchen gut mischen. Fügen Sie dann etwa 10 ml konzentriertes HCl (12 M oder 37%) hinzu und stellen Sie den pH-Wert auf 8 ein.
- Für 1 l 4000%ige (w/v) Holtfreter-Lösung werden 158,4 g NaCl, 11,13 g MgSO4,7H 2O, 5,36 g CaCl2,2H 2O und 2,88 g KCl mit deionisiertem Wasser bis zu 1 l Mischling gemischt.
- Bereiten Sie eine 0,01%ige (w/v) Benzocain-Verdünnung in Fäkalienwasser für die Anästhesie vor, indem Sie die 0,03%ige (w/v) Stammlösung 1:3 verdünnen.
- Für 1 l Lösung verwenden Sie 333 ml der Benzocain-Stammlösung plus 666 ml Wasser aus dem Fäkalientank.
- Bereiten Sie eine 5 mg/ml Butorphanoltartrat-Stammlösung zur Analgesie vor. Mischen Sie dazu 100 mg Butorphanol (+)-Tartratsalz mit 20 mL Reinstwasser. Aliquote bei 4 °C lagern.
- Am Tag der Amputationen 0,5 mg/l Butorphanoltartrat Arbeitslösung frisch zubereiten, um die Schmerzen der Tiere nach chirurgischen Eingriffen zu verringern, indem Sie die Stammlösung 1:10.000 in Fäkalienwasser verdünnen.
- Bei Tieren, die kleiner als 8 cm sind, mischen Sie 20 μl Stammlösung in 200 ml Wasser aus dem Fäkalienlager.
- Bei größeren Tieren mischen Sie 60 μl Stammlösung in 600 ml Wasser aus dem Fäkalienlager.
- Bereiten Sie sterile amphibische phosphatgepufferte Kochsalzlösung (APBS), eine 80%ige (v/v) PBS-Lösung, vor und halten Sie sie bei Raumtemperatur (RT), indem Sie 800 mL DPBS mit 200 mL deionisiertem Wasser mischen.
- Bereiten Sie 1 mg/ml Insulin-Stammlösung vor. Mischen Sie dazu 250 mg Insulinpulver mit 25 mL HCl 0,1 M und rühren Sie langsam, bis es sich aufgelöst hat. Fügen Sie während des Rührens 225 mL APBS hinzu, bis die Lösung klar ist. Sterilfiltrieren Sie und lagern Sie 4 mL Aliquots bei -20 °C.
- Steriles Kulturmedium (alle % (v/v): 62,5 % L15-Medium, 10 % hitzeinaktiviertes FBS, 1 % Penicillin/Streptomycin, 1 % Insulin, 1 % L-Glutamin) vorbereiten und bis zum Tag der Anwendung bei 4 °C aufbewahren. Äquilibrieren Sie am Versuchstag vor der Anwendung auf RT.
- Verwenden Sie für 400 ml Nährmedium 250 ml L15-Medium, 40 ml hitzeinaktiviertes FBS, 4 ml Penicillin/Streptomycin, 4 ml Insulinlösung (aus Schritt 1.7), 4 ml L-Glutamin und 98 ml deionisiertes steriles Wasser. Unter sterilen Bedingungen und einem Sterilfilter nach dem Mischen aller Komponenten zubereiten. Bereiten Sie 15 ml Aliquote vor.
- Bereiten Sie 2,5 % und 3 % (w/v) Agarose mit niedrigem Schmelzpunkt (lmp) in APBS-Lösung vor und erhitzen Sie sie bei 70 °C, um Agarose vollständig aufzulösen. Aliquots in 1,5-ml-Röhrchen zubereiten und bis zum Tag der Anwendung bei 4 °C lagern.
- Für 20 mL 2,5 % (w/v) lmp-Agarose mischen Sie 0,5 g lmp-Agarose mit 20 mL APBS in einem 50 mL Röhrchen und erhitzen Sie es in einem Wasserbad bei 70 °C, bis die Agarose vollständig geschmolzen ist. Die Lösung muss klar und transparent werden. Aliquots noch warm zubereiten.
- Für 20 mL 3 % (w/v) lmp-Agarose mischen Sie 0,6 g lmp-Agarose mit 20 mL APBS und gehen Sie wie oben beschrieben vor.
- (Optional): Für 20 mL 1 % (w/v) lmp-Agarose mischen Sie 0,2 g lmp-Agarose mit 20 mL APBS und gehen Sie wie oben beschrieben vor.
HINWEIS: Die Schmelzzeit hängt von der Konzentration der lmp-Agarose ab und kann zwischen 15 und 40 min liegen.
- Verwenden Sie für Amputationen und Gewebemontage ein Hellfeldstereoskop.
- Montieren Sie Petrischalen aus Kunststoff mit einem Durchmesser von 100 mm, Pinzetten, Skalpelle und chirurgische Scheren für Amputationen und Gewebeentnahmen.
- Bereiten Sie Petrischalen aus Kunststoff mit einem Durchmesser von 35 mm für die Eindruckmessungen vor.
- Bereiten Sie ~1 cm lange Zylinder mit ~1 cm Durchmesser vor. Erhitzen Sie dazu einen Kartonschneider unter einer Bunsenbrennerflamme und schneiden Sie mit der erhitzten Klinge ein 15 mL Röhrchen ab.
- 1 cm klein schneiden,2 Quadrate Parafilm und aufbewahren.
- Bereiten Sie einen Metallic- oder Kaltblock bei -20 °C vor, indem Sie ihn mindestens 1 h im Gefrierschrank ruhen lassen.
- Bereiten Sie Pasteur-Pipetten aus Kunststoff vor.
- (Optional): Fixiermittel MEMFa-Lösung (3-(N-Morpholin)propansulfonsäure [MOPS] 0,1 M pH 7,4, Ethylenglykol-bis (2-aminoethylether)-N,N,N′,N′-TETRAESSIGSÄURE [EGTA] 2 mM, MgSO4,7H 2O 1 mM, 3,7 % Formaldehyd) herstellen. Fügen Sie dazu 10,465 g MOPS, 0,123 g MgSO4,7H 2O, 2 mL 0,5 M EDTA pH 8,0 hinzu und fügen Sie Wasser bis 45 mL hinzu. Fügen Sie 5 mL Formaldehyd 37% (w/v) zu einem Gesamtvolumen von 50 mL hinzu.
2. Reagenzien
- In der Materialtabelle finden Sie die für diese Arbeit verwendeten Reagenzien, aber es können auch andere kommerzielle Anbieter verwendet werden.
3. Axolotl-Amputation und Regeneration der Gliedmaßen
- Vor allen experimentellen Verfahren betäuben Sie das Tier 20 Minuten lang mit 0,01 % (w/v) Benzocain, verdünnt in Wasser aus dem Haltungsbecken (Schritt 1,3), und stellen Sie sicher, dass die Tiere tief betäubt sind und nicht auf taktile Reize reagieren.
- Nehmen Sie den Axolotl aus der Anästhesie und legen Sie ihn auf eine 100 mm Petrischale, in der sich ein Papiertuch befindet, das mit anästhesiehaltigem Wasser angefeuchtet ist. Richten Sie die Gliedmaße senkrecht zur Körperachse aus und platzieren Sie die Platte zur besseren Visualisierung unter dem Stereoskop.
HINWEIS: Hier kam ein Stereoskop mit einer kompakten Schwanenhalslampe zum Einsatz. - Amputieren Sie die Extremität mit einem scharfen, sterilen Skalpell unmittelbar distal zum verkalkten Bereich der zeugopodialen Region (Radius/Ulna) (Abbildung 1A).
HINWEIS: Unter Einhaltung des 3R-Tierschutzprinzips wird empfohlen, die Gliedmaßen, die ursprünglich durch die Amputation entfernt wurden, als intakte Kontrollen zu verwenden. - Lassen Sie das Tier auf der Schüssel und decken Sie es 15 Minuten lang mit einem befeuchteten Seidenpapier ab, das mit benzocainhaltigem Wasser getränkt ist, damit die Blutgerinnung und der Wundverschluss stattfinden können.
- Die Tiere sind in einen Tank mit frischem Wasser und Analgetika (Butorphanoltartrat, 0,5 mg/l, aus Schritt 1.5) zu setzen.
- Bringen Sie die Tiere 24 Stunden nach der postoperativen analgetischen Behandlung in ihr ursprüngliches Becken mit frischem Warmhaltewasser.
- Lassen Sie die Tiere sich bis zum gewünschten Stadium des Interesses regenerieren.
HINWEIS: Die analysierten Gliedmaßen wurden am Tag der Amputation für die intakte Phase und 5 Tage nach der Amputation (dpa) für die Histolysephase bei 5 Monate alten Tieren entnommen. Das Knorpelkondensationsstadium wurde bei 7 Monate alten Tieren mit 21 dpa gemessen.
4. Gewebemontage und -verarbeitung für Messungen
- Die 1,5 mL Röhrchen mit 2,5 % bzw. 3 % (w/v) lmp-Agarose (aus Schritt 1.9) werden auf einem Thermoblock bei 70 °C erhitzt, bis die Agarose vollständig geschmolzen ist. Tauschen Sie die Rohre gegen einen anderen Thermoblock bei 37 °C aus und lassen Sie die Temperatur ausgleichen, bevor Sie sie verwenden.
HINWEIS: Hier wurden 2,5 % lmp-Agarose zur Messung von regenerierendem Gewebe und 3 % für intaktes Gewebe verwendet. - Decken Sie eine Seite der 1 cm langen Zylinder (aus Schritt 1.13) mit einem der Parafilm-Stücke (Schritt 1.14) ab, so dass sie an der Unterseite vollständig verschlossen sind.
- Äquilibrieren Sie 15 ml-Aliquots des Kulturmediums auf RT (aus Schritt 1.8)
- Betäuben Sie die Tiere, indem Sie sie mindestens 20 Minuten lang in anästhesiehaltiges Wasser (0,01 % (w/v) Benzocain, verdünnt im Fäkalienwasser) tauchen.
- Bilden Sie die Gliedmaßen unter einem Stereoskop mit einer Software ab, die quantitative Messungen ermöglicht. Messen Sie die Länge der interessierenden Struktur und berechnen Sie, wie viel Gewebe vom distalen Ende der Extremität entfernt werden muss, bis der interessierende Abschnitt erreicht ist.
- Um Gliedmaßen zu regenerieren, sammeln Sie sie, indem Sie mit einem Skalpell und/oder einer chirurgischen Schere auf Höhe des Ellbogens schneiden und überschüssiges Gewebe aus der Gliedmaße herauspräparieren. Lassen Sie das Gewebe in der APBS-Lösung, während Sie den nächsten Schritt vorbereiten. Stellen Sie sicher, dass der Schnitt quer (90°) zur unteren Armachse erfolgt, um eine ebene Oberfläche zu erzeugen. Bei den intakten Gliedmaßen entfernen Sie die Hand, indem Sie die Karpalregion durchschneiden.
- Euthanasieren Sie die Tiere, indem Sie sie mindestens 20 Minuten lang einer tödlichen Dosis Anästhesie (0,1 % Benzocain) aussetzen. Fügen Sie dazu das erforderliche Volumen von 10 % (w/v) Benzocainlösung hinzu, um eine Konzentration von 0,1 % (w/v) zu erreichen.
- Wenn Tiere in 100 ml 0,01 % (w/v) Benzocainlösung anästhesiert wurden, fügen Sie 900 μl der 10 % (w/v) Benzocainlösung hinzu.
- Spülen Sie die Gliedmaßen, indem Sie sie in APBS-Lösung tauchen.
- Stellen Sie sicher, dass sich die Pasteur-Pipetten und der Thermoblock (stabilisiert bei 37 °C mit Agarose-Aliquoten) in der Nähe der Arbeitsstation befinden. Nehmen Sie den kalten Block aus dem -20 °C heißen Gefrierschrank und legen Sie den Zylinder mit dem mit Parafilm überzogenen Ende nach unten darauf.
- Greifen Sie nach dem präparierten Glied und entfernen Sie die überschüssige Flüssigkeit vorsichtig mit Seidenpapier. Legen Sie das Glied auf einen sauberen Teller, geben Sie das geschmolzene lmp-Agarose darauf und bewegen Sie das Glied kurz in der Agarose, um das restliche APBS von der Hautoberfläche zu verdrängen.
- Arbeiten Sie schnell und platzieren Sie den Wurfarm im Zylinder und stellen Sie sicher, dass er vertikal ausgerichtet ist, wobei der Interessenbereich nach oben zeigt.
- Während Sie die Extremität vorsichtig mit einer Pinzette festhalten, geben Sie lmp-Agarose in den Zylinder, bis das Gewebe vollständig bedeckt ist. Entfernen Sie vorsichtig die Pinzette, bevor die Agarose erstarrt.
- Den Zylinder aus dem Kaltblock nehmen und die Agarose ca. 30 s bei RT vollständig erstarren lassen.
- Bringen Sie den gewebehaltigen Agaroseblock sofort in den Vibratomraum, zusammen mit den sterilen Kulturmedium-Aliquoten und APBS, bei RT.
- Entfernen Sie den Parafilm von der Unterseite des Zylinders und befestigen Sie die Agarose, die das Gewebe enthält, mit Cyanacrylatkleber auf der Vibratomstufe. Stellen Sie sicher, dass sowohl Agarose als auch Probe auf den Tisch geklebt sind.
- Tauchen Sie die Bühne zum Schneiden in APBS ein.
HINWEIS: Zu den intakten Geweben gehört der steifere Knochen/Knorpel, während regenerierendes Gewebe weicher ist. Daher müssen die Schwingungsparameter entsprechend angepasst werden. In dieser Studie wurden folgende Parameter verwendet: Intaktes Gewebe (Amplitude 1,2 mm und Geschwindigkeit 0,1 mm/s) und regenerierendes Gewebe (Amplitude 0,9 mm und Geschwindigkeit 0,4 mm/s). - Beginnen Sie mit dem Schnitt der Agarose in kurzen Schritten (z. B. in 100 μm-Schritten), bis die Gewebespitze erreicht ist. Anschließend wird der Gewebeblock durchtrennt, bis der distale Teil des Gewebes (berechnet in Schritt 4.5) entfernt ist. Auf diese Weise wird ein querschnittlicher Querschnitt des Interessengebiets leicht zugänglich.
HINWEIS: Der entnommene Gewebeschnitt enthält die unmittelbar an die mit dem AFM untersuchte Oberfläche angrenzende Oberfläche und kann als Referenz für die Gewebestruktur dienen. Daher kann es für die direkte komplementäre Analyse verwendet oder für die anschließende Färbung fixiert werden (siehe Abschnitt 6). - Entfernen Sie den gewebehaltigen Block vorsichtig mit einer Rasierklinge aus dem Vibratomime-Stadium und entfernen Sie alle Spuren von Kleber. Kleben Sie den Block sofort auf eine 35-mm-Petrischale aus Kunststoff mit chirurgischem Gewebekleber und fügen Sie ca. 2 ml Kulturmedium bei RT hinzu, um sicherzustellen, dass das Gewebe vollständig bedeckt ist.
HINWEIS: Die freiliegende Gewebeoberfläche im Agaroseblock ist die zu untersuchende Oberfläche.
5. Messungen mit AFM
- Bereiten Sie den Ausleger mindestens 1 Tag vor der Messung für die Messungen vor.
HINWEIS: Für diese Arbeit wurden spitzenlose Silikon-Cantilever mit Polystyrolkügelchen (Durchmesser 20 μm) funktionalisiert, und die Perlen-Cantilever-Bindung wurde mindestens 1 Tag lang verstärkt, bevor sie für Eindringmessungen verwendet wurde.- Befestigen Sie die Raupen mit Epoxidkleber mit einer Verarbeitungszeit von 5 min an den Auslegern.
- Senken Sie den Ausleger zügig und mit minimalem Kontakt auf einen mit Leim beschichteten Glasobjektträger ab, so dass sich an seinem Ende ein kleines Leimtröpfchen bildet, und bringen Sie ihn unmittelbar danach mit einer geeigneten Raupe in Kontakt.
- Halten Sie den Ausleger-Raupen-Kontakt ~10 Minuten lang, bevor Sie den Ausleger mit einer gebundenen Raupe von der Oberfläche abheben.
- Kalibrieren Sie den Ausleger vor den Messungen.
HINWEIS: Der modifizierte Ausleger wurde vor jeder Versuchsreihe nach dem thermischen Rauschverfahren unter Verwendung der eingebauten Verfahren der AFM-Software kalibriert. - Setzen Sie die Petrischale mit dem Gewebeblock (aus Schritt 4.18) und dem Kulturmedium in den Petrischalenhalter des AFM ein und nehmen Sie ein Übersichtsbild in der Hellfeldmikroskopie auf (Abbildung 1B).
HINWEIS: Für Eindringprüfungen wird ein Aufbau verwendet, der mit einem Motortisch auf einem aufrechten Lichtmikroskop ausgestattet ist. Montieren Sie den AFM-Kopf, positionieren Sie den Cantilever-Ball über dem Gewebe und bringen Sie den Cantilever-Ball in die Nähe der Oberfläche. - Wählen Sie den spezifischen Interessenbereich aus, und zeichnen Sie ein Array von Kraft-Weg-Kurven auf (Abbildung 2A).
HINWEIS: Es wurden Relativkraftsollwerte von 2-25 nN verwendet, um vergleichbare Eindringtiefen von ~1-4 μm für Gewebe unterschiedlicher Steifigkeit zu erreichen, mit einer Annäherungs- und Rückzugsgeschwindigkeit von 7,5 μm/s, einer Z-Länge von 50 μm, einer Gittergröße von 70 μm x 70 μm mit 3 x 3 Punkten. - Erfassen Sie für jede sondierte Region ein Hellfeldmikroskopiebild, um die erhaltenen Werte mit jeder einzelnen Region im Gliedmaßenbereich zu verknüpfen.
- Bei allen Proben sind mindestens 3 verschiedene Regionen pro Gewebetyp zu untersuchen.
HINWEIS: In allen Fällen wurden in dieser Studie 4 Regionen in der Mitte des Knorpels und 3 Regionen in der Peripherie der Knorpelregion gemessen und das Gewebe maximal 1 h unter dem Mikroskop gehalten. - Sobald die Eindruckmessungen abgeschlossen sind, entsorgen oder fixieren Sie das Gewebe für die weitere Analyse.
HINWEIS: Die Proben wurden nach Abschluss der Messungen fixiert, indem sie in 2-ml-Röhrchen mit MEMFa-Lösung gelegt und über Nacht bei 4 °C fixiert wurden. Es wurde MEMFa (Schritt 1.17) verwendet, aber auch Formaldehydlösung 4 % (w/v) in PBS kann verwendet werden.
6. (Optional) Verarbeitung benachbarter Gewebeschnitte
- Wenn die angrenzenden Gewebeschnitte für die anschließende Färbung verwendet werden, legen Sie sie sofort nach dem Schnitt in ein kleines 2-ml-Röhrchen mit Fixiermittel und fixieren Sie sie über Nacht bei 4 °C.
- Um die Gewebearchitektur durch die Markierung des Aktin-Zytoskeletts und der Zellkerne aufzudecken, waschen Sie 3 Mal mit PBS für jeweils 5 Minuten, um das Fixiermittel zu entfernen, und färben Sie mit einer Lösung, die Alexa Fluor 488 konjugiertes Phalloidin (1:250) und Hoechst 33258 (1:10000) in PBS enthält, für 1 h bei RT auf einer Wippplattform.
- Als nächstes werden die gefärbten Proben auf Schalen mit Glasboden mit 1 % (w/v) lmp-Agarose in APBS immobilisiert, die bei 37 °C stabilisiert ist (aus Schritt 1.9). Stellen Sie sicher, dass die gewünschte Oberfläche zum Glas zeigt. Sobald Agarose fest wird, bedecken Sie die Proben mit PBS, um eine Austrocknung zu verhindern.
HINWEIS: Die intakte Probe und die Histolyseprobe werden mit einem inversen Konfokalmikroskop (10-fache Vergrößerung und 8 μm optische Schnitte) abgebildet. Die Bilder in Abbildung 1C sind maximale Projektionen von 8 optischen Abschnitten.
- Wenn der benachbarte Gewebeschnitt für die unmittelbare nachgelagerte Analyse verwendet wird, die frisches Gewebe erfordert (z. B. RNA-Extraktion, Inkubation mit lebenden Farbstoffen usw.), stellen Sie sicher, dass Sie schnell arbeiten und ein Zellkulturmedium (aus Schritt 1.8) verwenden, um die höchstmögliche Gewebeintegrität zu gewährleisten.
7. Datenanalyse und -darstellung
- Um den scheinbaren Elastizitätsmodul zu berechnen, analysieren Sie die Kraft-Weg-Kurven unter Verwendung des Hertz/Sneddon-Modells (Gl. 1) für einen sphärischen Eindringkörper unter Verwendung der JPK/Bruker-Datenverarbeitungssoftware unter der Annahme eines Poisson-Verhältnisses von 0,5.
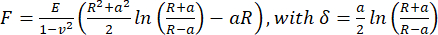 (Gl. 1)
(Gl. 1)
Dabei gilt: R: Eindringradius, E: Elastizitätsmodul, ν: Poissonzahl, a: Radius der kreisförmigen Kontaktfläche zwischen Eindringkörper und Probe, δ: Eindringtiefe. - Analysieren Sie für die viskoelastische Analyse die Annäherungsteile der Kraft-Eindringkurven in PyJibe 0.15.0 mit der Erweiterung "Hertz model corrected for viscoelasticity using Kelvin-Voigt-Maxwell (KVM)" (geschrieben von Paul Müller, https://github.com/AFM-analysis/PyJibe)25. Die Anpassungsfunktion basiert auf einem von Abuhattum et al.26 beschriebenen Modell, das Kelvin-Voigt-Maxwell-Modellelemente integriert.
- Verarbeiten Sie die Krafteindruckkurven vor, um den Kontaktpunkt zu schätzen, indem Sie eine stückweise Anpassung mit einer Linie (Basislinie) und einer polynomialen Anpassungsfunktion für das Anfahrteil verwenden.
- Passen Sie die Kraft-Eindring-Kurven an das "Hertz-Modell korrigiert für Viskoelastizität unter Verwendung des Kelvin-Voigt-Maxwell-Modells (KVM)) an". Aus der Passform erhalten Sie den unentspannten Elastizitätsmodul, den scheinbaren Elastizitätsmodul und die scheinbare Viskosität. Das Modell bietet Maxwell auch Entspannungs- und Eindringzeiten.
HINWEIS: In mehreren analysierten Krafteindringkurven (insbesondere im intakten Gewebe) waren die Maxwell-Relaxationszeiten signifikant größer oder kleiner als die Eindringzeit, was auf ein eher elastisches Verhalten bzw. mechanisches Kelvin-Voigt-Verhalten hinweist.
- Exportieren Sie alle Messungen in eine Tabelle und berechnen Sie die mittleren scheinbaren Elastizitätsmodule pro Gewebetyp und Probe.
- Plotten Sie die Daten und analysieren Sie sie statistisch mit geeigneter Software.
HINWEIS: Hier wird das GraphPad-Prisma verwendet, und bei der Beschreibung der Ergebnisse bezieht sich die Studie auf den Mittelwert ± SD der mittleren scheinbaren Elastizitätsmodule pro Probe (dargestellt in Abbildung 2D-F). - Zeigen Sie optische Schnitte aus konfokalen Bildern an, indem Sie sie mit der Fiji-Funktion für maximale Projektion projizieren. Passen Sie die Helligkeit und den Kontrast jedes einzelnen Kanals an, um eine optimale Visualisierung von Fluoreszenzfärbungen zu erzielen.
- Generieren Sie ein Abbildungspanel mit der entsprechenden Software.
HINWEIS: Zum Generieren der Panels wird Affinity Designer verwendet, und das Modell in Abbildung 1A wird mit Affinity Designer gezeichnet.
Ergebnisse
Unter Verwendung des oben beschriebenen Protokolls maßen wir den scheinbaren Elastizitätsmodul von knorpeligem Axolotl-Gliedmaßengewebe unter homöostatischen ("intakten") Bedingungen, während der frühen Knorpelhistolyse und späterer Knorpelkondensationsstadien (Abbildung 1A). Wir untersuchten auch die mechanischen Eigenschaften der Skelettelemente in verschiedenen Regionen, einschließlich ihres Zentrums und ihrer Peripherie, wie in den Bildern zu seh...
Diskussion
Hier demonstrieren wir eine Technik zur Messung der Knorpelsteifigkeit bei Axolotl-Gliedmaßen mit AFM. Diese Methode kann jedoch auch für die Untersuchung anderer Gewebetypen erweitert werden. Ein wichtiger Schritt für erfolgreiche AFM-Messungen ist die Probenvorbereitung, die sich bei Axolotl-Proben als besonders schwierig erwies. Wir fanden heraus, dass die Sondierung der Gewebeoberfläche, die noch in den Agaroseblock eingebettet war, der beste Weg war, um die Gewebeintegrität zu ...
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen
Danksagungen
Wir danken allen Mitgliedern des Sandoval-Guzmán-Labors für die kontinuierliche Unterstützung und Begleitung während der Entwicklung dieser Arbeit. Wir danken auch Anja Wagner, Beate Gruhl und Judith Konantz für ihr Engagement in der Axolotl-Pflege. Wir danken auch Paul Müller für die Bereitstellung von Codes für die AFM-Datenanalyse. Diese Arbeit wurde unterstützt von der Light Microscopy Facility der CMCB Technology Platform der TU Dresden. AT ist Fellow des Mildred Scheel Early Career Center Dresden P2, das von der Deutschen Krebshilfe gefördert wird. RA wird durch eine Eigene Stelle der Deutschen Forschungsgemeinschaft (DFG) – AI 214/1-1 – finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Affinity Designer | Affinity | version 1.10.4 | For figure assembling |
| Agarose Low Melt | Roth | 6351.1 | For sample preparation |
| Alexa Fluor 488 Phalloidin | Invitrogen | A12379 | To stain tissue |
| Axiozoom | Zeiss | To image samplea under the AFM | |
| Benzocaine | Sigma-Aldrich | E1501 | To anesthetize the animals |
| Butorphanol (+)-tartrate salt | Sigma-Aldrich | B9156 | As analgesic |
| Cantilever | NanoWorld | Arrow TL1 | For AFM indentation measurements |
| Cellhesion 200 setup equipped with a motorstage | JPK/Bruker | For AFM indentation measurements | |
| CellSense Entry | For imaging in Stereoscope Olympus UC90 | ||
| Dulbecco’s Phosphate Buffered Saline (DPBS, 1x) | Gibco | 14190-144 | To clean samples and section under vibratome |
| FIJI (ImageJ2) | https://imagej.net/software/fiji | version 2.9.0/1.53t | For image processing |
| GraphPad Prism | GraphPad Software | (version 8.4.3) | To graph and statistically analyze the data |
| Heat-inactivated FBS | Gibco | 10270-106 | For cell culture medium |
| Histoacryl glue (2-Butyl-Cyanoacrylate) | Braun | To glue sample to petri dishes | |
| Hoechst 33258 | Abcam | ab228550 | To stain tissue |
| Insulin | Sigma-Aldrich | I5500 | For cell culture medium |
| Inverted confocal microscope | Zeiss | 780 LSM | To image tissue sections |
| Inverted confocal microscope | Zeiss | 980 LSM | To image tissue sections |
| JPK/Bruker data processing software | JPK/Bruker | SPM 6.4 | To analyze force-distance curves |
| L15 medium (Leibovitz) | Sigma | L1518 | For cell culture medium |
| L-Glutamine | Gibco | 25030-024 | For cell culture medium |
| Penicillin/Streptomycin | Gibco | 15140-122 | For cell culture medium |
| polystyrene beads ( 20 µm diameter); ) | microParticles | For AFM indentation measurements | |
| Pyjibe | written by Paul Müller https://github.com/AFM-analysis/PyJibe | 0.15.0 | For viscoelastic analysis |
| Stereoscope Olympus SX10 | Olympus | SX10 | For limb amputations and tissue mounting |
| Stereoscope Olympus UC90 | Olympus | UC90 | For imaging |
| Vibratome Leica | Leica | VT 1200S | For tissue sectioning |
Referenzen
- Mills, L. A., Aitken, S. A., Simpson, A. H. R. W. The risk of non-union per fracture: current myths and revised figures from a population of over 4 million adults. Acta Orthop. 88 (4), 434-439 (2017).
- Calori, G. M., et al. Non-unions. Clin Cases Miner Bone Metab. 14 (2), 186-188 (2017).
- Nandra, R., Grover, L., Porter, K. Fracture non-union epidemiology and treatment. Trauma. 18 (1), 3-11 (2016).
- Nauth, A., Schemitsch, E., Norris, B., Nollin, Z., Watson, J. T. Critical-size bone defects: Is there a consensus for diagnosis and treatment. J Orthop Trauma. 32 (3 Suppl), S7-S11 (2018).
- Schemitsch, E. H. Size matters: Defining critical in bone defect size. J Orthop Trauma. 31 (10), S20-S22 (2017).
- Joven, A., Elewa, A., Simon, A. Model systems for regeneration: Salamanders. Development (Cambridge). 146 (14), 0 (2019).
- Aires, R., Keeley, S. D., Sandoval-Guzmán, T. Basics of Self-Regeneration. Cell Engineering and Regeneration. , (2020).
- Stocum, D. L. Mechanisms of urodele limb regeneration. Regeneration. 4 (4), 159-200 (2017).
- Bassat, E., Tanaka, E. M. The cellular and signaling dynamics of salamander limb regeneration. Curr Opin Cell Biol. 73, 117-123 (2021).
- Vinarsky, V., Atkinson, D. L., Stevenson, T. J., Keating, M. T., Odelberg, S. J. Normal newt limb regeneration requires matrix metalloproteinase function. Dev Biol. 279 (1), 86-98 (2005).
- Godwin, J., Kuraitis, D., Rosenthal, N. Extracellular matrix considerations for scar-free repair and regeneration: Insights from regenerative diversity among vertebrates. International J Biochem Cell Biol. 56, 47-55 (2014).
- Riquelme-Guzmán, C., et al. Osteoclast-mediated resorption primes the skeleton for successful integration during axolotl limb regeneration. ELife. 11, e79966 (2022).
- Gerber, T., et al. Single-cell analysis uncovers convergence of cell identities during axolotl limb regeneration. Science. 362 (6413), aaq0681 (2018).
- Kragl, M., et al. Cells keep a memory of their tissue origin during axolotl limb regeneration. Nature. 460 (7251), 60-65 (2009).
- Currie, J. D., et al. Live Imaging of Axolotl Digit Regeneration Reveals Spatiotemporal Choreography of Diverse Connective Tissue Progenitor Pools. Dev Cell. 39 (4), 411-423 (2016).
- Qin, T., et al. Single-cell RNA-seq reveals novel mitochondria-related musculoskeletal cell populations during adult axolotl limb regeneration process. Cell Death Differ. 28 (3), 1110-1125 (2021).
- Sandoval-Guzman, T., et al. Fundamental differences in dedifferentiation and stem cell recruitment during skeletal muscle regeneration in two salamander species. Cell Stem Cell. 14, 174-187 (2014).
- Currie, J. D., et al. The Prrx1limb enhancer marks an adult subpopulation of injury-responsive dermal fibroblasts. Biol Open. 8, bio043711 (2019).
- Wagh, K., Ishikawa, M., Garcia, D. A., Stavreva, D. A., Upadhyaya, A., Hager, G. L. Mechanical regulation of transcription: Recent advances. Trends Cell Biol. 31 (6), 457-472 (2021).
- d'Angelo, M., et al. The role of stiffness in cell reprogramming: A potential role for biomaterials in inducing tissue regeneration. Cells. 8 (9), 1036 (2019).
- Akhtar, R., Sherratt, M. J., Cruickshank, J. K., Derby, B. Characterizing the elastic properties of tissues. Mater Today (Kidlington). 14 (3), 96-105 (2011).
- Krieg, M., et al. Atomic force microscopy-based mechanobiology. Nat Rev Phys. 1 (1), 41-57 (2019).
- El Kirat, K., Burton, I., Dupres, V., Dufrene, Y. F. Sample preparation procedures for biological atomic force microscopy. J Microsc. 218 (3), 199-207 (2005).
- Riquelme-Guzmán, C., et al. Postembryonic development and aging of the appendicular skeleton in Ambystoma mexicanum. Dev Dyn. 251 (6), 1015-1034 (2021).
- López-Alonso, J., Eroles, M., Janel, S., et al. PyFMLab: Open-source software for atomic force microscopy microrheology data analysis [version 2; peer review: 2 approved]. Open Res Europe 2024. 3 (187), (2024).
- Abuhattum, S., Mokbel, D., Müller, P., Soteriou, D., Guck, J., Aland, S. An explicit model to extract viscoelastic properties of cells from AFM force-indentation curves. iScience. 25 (4), 104016 (2022).
- Glorieux, L., et al. In-depth analysis of the pancreatic extracellular matrix during development for next-generation tissue engineering. Int J Mol Sci. 24 (12), 10268 (2023).
- Hiratsuka, S., et al. Power-law stress and creep relaxations of single cells measured by colloidal probe atomic force microscopy. Jpn J Appl Phys. 48 (8 PART 3), 08JB17 (2009).
- Johnson, K. L., Kendall, K., Roberts, A. D. Surface energy and the contact of elastic solids. Proc R Soc Lond A. 324, 301-313 (1971).
- Kondiboyina, V., Duerr, T. J., Monaghan, J. R., Shefelbine, S. J. Material properties in regenerating axolotl limbs using inverse finite element analysis. J Mech Behav Biomed Mater. 150, 106341 (2024).
- Calve, S., Simon, H. Biochemical and mechanical environment cooperatively regulate skeletal muscle regeneration. FASEB J. 26 (6), 2538-2545 (2012).
- Calve, S., Simon, H. G. Extracellular control of limb regeneration. IUTAM Symposium on Cellular, Molecular and Tissue Mechanics. IUTAM Bookseries. 16, 257-266 (2010).
- Kaufmann, R. A., Kozin, S. H., Barnes, A., Kalluri, P. Changes in strain distribution along the radius and ulna with loading and interosseous membrane section. J Hand Surg Am. 27 (1), 93-97 (2002).
- Martin, A. R., et al. Measurement of distal forearm bone mineral density: Can different forearm segments be used interchangeably. J Clin Densitom. 2 (4), 381-387 (1999).
- Riquelme-Guzmán, C., et al. In vivo assessment of mechanical properties during axolotl development and regeneration using confocal Brillouin microscopy. Open Biol. 12 (6), 220078 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten