Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Proyección de imagen de dos fotones de atracción Microglial procesos hacia ATP o serotonina en rebanadas de cerebro agudo
* Estos autores han contribuido por igual
En este artículo
Resumen
Microglia, las células inmunes residentes del cerebro, responde rápidamente con cambios morfológicos a modificaciones de su entorno. Este protocolo describe cómo usar microscopía de dos fotones para estudiar la atracción de procesos de microglial hacia serotonina o ATP en rebanadas de cerebro agudo de ratones.
Resumen
Las células microgliales son residente células inmunes innatas del cerebro que exploración su entorno con sus largos procesos y constantemente, con disrupción de la homeostasis, experimentan rápidos cambios morfológicos. Por ejemplo, una lesión de láser induce en pocos minutos un crecimiento orientado de los procesos de microglial, también llamado "motilidad direccional", hacia el sitio de la lesión. Un efecto similar puede obtenerse mediante la entrega localmente ATP o serotonina (5-hidroxitriptamina [5-HT]). En este artículo se describe un protocolo para inducir un crecimiento direccional de microglial procesos hacia una aplicación local de ATP o 5-HT en rebanadas de cerebro agudo de ratones jóvenes y adultos y a esta atracción con el tiempo la imagen por microscopía multifotón. Se propone un método simple de cuantificación con software de análisis de imágenes gratis y de código abierto. El tiempo limitado, disminuyendo con la edad, durante el cual las células permanecen en un estado fisiológico es un reto que todavía caracteriza a rebanadas de cerebro agudo. Este protocolo, por lo tanto, destacan algunas mejoras técnicas (cámara de medio interfaz aire-líquido, cámara con una doble la perfusión de la proyección de imagen) para optimizar la viabilidad de las células microgliales durante varias horas, especialmente en sectores de ratones adultos.
Introducción
Las células microgliales son macrófagos residentes del cerebro y desempeñan un papel en ambas condiciones fisiológicas y patológicas1,2. Tienen una morfología muy ramificada y son constantemente ampliando y contrayendo sus procesos3,4. Este comportamiento de "análisis" se cree para ser relacionados y necesarios para el estudio de su entorno. La plasticidad morfológica de microglía se expresa de tres modos. En primer lugar, algunos compuestos rápidamente modulan la morfología microglial: la adición de ATP5,6 o NMDA5,7 en el medio que baña las rebanadas de cerebro agudo incrementa la complejidad de las ramificaciones de la microglía, mientras que la noradrenalina disminuye6. Estos efectos son directamente mediados por receptores de la microglía (para ATP y de la noradrenalina) o requieren una liberación de ATP de las neuronas (para NMDA). En segundo lugar, la velocidad de crecimiento y retracción de procesos microglial, llamado "vigilancia", o la motilidad puede ser afectada por factores extracelulares8homeostasis interrupciones9,10y mutaciones9, 10,11. En tercer lugar, además de estos cambios isotrópicos de la morfología y motilidad, microglia tienen la capacidad de extender sus procesos direccionalmente hacia una pipeta entrega ATP3,5,12, 13 , 14, en la cultura, en rebanadas de cerebro agudo o en vivo, o entrega 5-HT en rebanadas de cerebro agudo15. Tal crecimiento orientado de los procesos de microglial, también llamado motilidad direccional, primero fue descrito como una respuesta a una lesión local laser3,4. Por lo tanto, fisiológico, puede ser relacionado con la respuesta a la lesión o necesaria para dirigir procesos de microglial hacia las sinapsis o las regiones del cerebro que requieren poda durante desarrollo15,16, o enfisiológicas 17 ,18,19 o situaciones patológicas9,18,19,20 en la edad adulta. Los tres tipos de cambios morfológicos se basan en diferentes mecanismos intracelulares11,13,20y un compuesto determinado no necesariamente modular todas ellas (por ejemplo, NMDA, que actúa indirectamente sobre microglia, tiene un efecto en la morfología pero no induce motilidad direccional5,7). Por lo tanto, cuando con el objetivo de caracterizar el efecto de un compuesto, una mutación o una patología en la microglia, es importante caracterizar los tres componentes de su plasticidad morfológica. Aquí, describimos un método para estudiar el crecimiento direccional de microglial procesos hacia una fuente local del compuesto, que es, aquí, ATP o 5-HT.
Hay varios modelos para estudiar la atracción de los procesos microglia: cultivos primarios en entorno 3D6,18,19, cerebral aguda rebanadas6,13,15y en vivo proyección de imagen3,13. El enfoque en vivo es lo mejor para preservar el estado fisiológico de la microglia. Sin embargo, la proyección de imagen intravital de regiones profundas requiere procedimientos quirúrgicos complejos y, por lo tanto, a menudo se limita a las capas corticales superficiales. El uso de cultivo primario de la microglia es la técnica más sencilla para probar un gran número de condiciones con un número limitado de animales. Sin embargo, es imposible obtener la misma morfología de la célula como en vivo, y las células pierden sus interacciones fisiológicas con neuronas y astrocitos. Rebanadas de cerebro agudo representan un compromiso entre estos dos enfoques. Este modelo permite a los investigadores a estudiar las estructuras del cerebro que de lo contrario son difíciles de llegar y a la imagen con alta resolución en vivo e investigar rebanadas de etapas neonatales, mientras que la microscopía transcraneal se realiza sobre todo en la edad adulta. Por último, permite observar en tiempo real los efectos de la aplicación local de la droga y a repetir experimentos utilizando un número limitado de animales. Sin embargo, un problema con las rebanadas de cerebro agudo es el poco tiempo (unas horas) durante el cual las células siguen vivas, en particular para rebanadas de ratones más de dos semanas y el potencial cambio de morfología de la microglia por vez21,22 .
Aquí, describimos un protocolo para preparar rebanadas de cerebro agudo de jóvenes y adultos Cx3cr1GFP / + ratones hasta dos meses de edad, con la preservación de la morfología de la microglia y motilidad durante varias horas. A continuación, describimos cómo usar estas láminas para estudiar la atracción de los procesos de microglial hacia compuestos como ATP o 5-HT.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los experimentos fueron aprobados por el Comité de ética local (Comité de Darwin, acuerdos #1170 y #10921).
1. preparación de Micropipetas de vidrio para la aplicación Local de compuestos
- Preparar pipetas de borosilicato Capillares de vidrio de pared delgada con un tirador de electrodo. Ajustar los parámetros para obtener pipetas con un 4-5 μm de diámetro en su extremo. Figura 2D muestra una pipeta en trasmitida en la ampliación baja.
2. las soluciones
- Asegurar sólo material de vidrio que ha sido limpiada por un ciclo de autoclave, seguido por enjuague de 2 x - 3 x con agua ultrapura, se utilizará. Nunca uso cristalería que ha estado en contacto con paraformaldehido.
-
Preparar un 2 mol· L-1 CaCl2 solución madre disolviendo 14,7 mg de CaCl2·2H2O en 50 mL de agua de alta pureza (agua ultrapura, resistencia 18.2 MΩ; los rastros de metal en agua destilada o agua del grifo pueden conducir a la calidad subóptima de la rebanada debido a efectos pro-oxidativos).
- Almacenar esta solución a temperatura ambiente durante un máximo de un mes.
-
En el día del experimento, preparar 1 L de solución de colina-aCSF (líquido cefalorraquídeo artificial), cuya composición es 110 mmol· Colina de L-1 Cl, 25 mmol· Glucosa de L-1 , 25 mmol· L-1 NaHCO3, 7 mmol· L-1 MgCl2, 11.6 mmol· L-1 ácido ascórbico, 3.1 mmol· Piruvato de sodio L-1 , 2.5 mmol· L-1 KCl, 1.25 mmol· L-1 NaH2PO4y 0,5 mmol· L-1 CaCl2, 0,5.
- Para preparar esta solución, agregar, en el siguiente orden, a un matraz de 1 L graduado: 0,186 g de KCl, 0,195 g de NaH2PO4, 2,04 g de ácido ascórbico, 2,1 g de NaHCO3y 4,5 g de glucosa.
- Llenar aproximadamente la mitad del volumen final con agua ultrapura y agitar hasta disolución completa.
- Añadir 0,34 g de piruvato de sodio y g 15,36 de colina Cl.
Nota: Es conveniente disolver primero la colina Cl con 5 a 10 mL de la solución preparada en el paso 2.3.2 antes de agregar a la solución de todo. - Agregar 7 mL de 1 mol· L-1 MgCl2 y 250 μl de 2 mol· L-1 CaCl2 (preparado en el paso 2.2) a la solución.
- Llene el matraz aforado hasta 1 L con agua ultrapura.
- Con un osmómetro de presión de vapor, compruebe que la osmolaridad es de entre 300 y 310 mΩ. Si no, ajuste con glucosa.
- Compruebe el pH después de carbogenation (es decir, burbujas con "carbogen", una mezcla de 95% O25% CO2) y ajustar, si es necesario, a 7.3-7.4 con 10 M NaOH.
- Transferir la solución a una botella de vidrio para almacenamiento. Mantener la botella en la nevera hasta su uso (paso 3.1).
Nota: Se recomienda para hacer una solución fresca en el día del experimento. Sin embargo, si es necesario, colina aCSF puede almacenarse 2 días a 4 ° C.
-
En el día del experimento, preparar 1 L de una solución de la aCSF, cuya composición es 124 mmol· L-1 NaCl, 26.2 mmol· L-1 NaHCO3, 25 mmol· Glucosa de-1 L, 2,5 mmol· L-1 KCl, 2 mmol· L-1 CaCl2, 1 mmol· L-1 MgCl2y 1.25 mmol· L-1 NaH2PO4.
- Para preparar esta solución, agregar, en el siguiente orden, a un matraz aforado: 0,150 g de NaH2PO4, 0,186 g de KCl, 2,2 g de NaHCO3, 4,5 g de glucosa y 7,3 g de NaCl. La solución a un volumen de 1 L con agua ultrapura y agite vigorosamente en una placa de agitación.
- Añadir 1 mL de 1 mol· L-1 MgCl2 y 1 mL de 2 mol· L-1 CaCl2 a la solución y el traslado de la aCSF solución a una botella de vidrio para almacenamiento.
- Si la osmolaridad es de 300-310 mΩ· L-1 y, si no, ajustarlo con la glucosa.
- Compruebe el pH después de carbogenation (es decir, burbujas con "carbogen") y ajústelo si es necesario, a 7.3-7.4 con 10 M NaOH.
- Transferir la solución a una botella de vidrio para almacenamiento. Mantener la botella en la nevera hasta su uso (paso 3.1).
Nota: Se recomienda para hacer una solución fresca en el día del experimento. Sin embargo, una alternativa es preparar un 10 x solución madre que contiene NaCl NaHCO3, KCl y NaH2PO4 a 10 veces la concentración final, que puede ser almacenado por no más de una semana a 4 ° C. Hacer la aCSF final en el día del experimento al diluir la solución madre de 10 x con agua ultrapura y agregar la glucosa, CaCl2y MgCl2.
- Preparar las soluciones de drogas en el día del experimento. Utilizar la solución de la aCSF para llevarlos a las concentraciones finales que son, aquí, µmol· 500 L-1 de la ATP y µmol· 5 L-1 de 5-HT.
Nota: ATP, se puede preparar una solución madre (p. ej., 50 mM ATP en agua), almacenados en forma alícuotas a-20 ° C y diluir con aCSF a la concentración final en el día del experimento. En cambio, la solución de 5-HT (serotonina-HCl) debe ser preparada de polvo en el día del experimento, en 1 mg·mL-1 en agua a 4 ° C para evitar la oxidación de la 5-HT y diluida en aCSF en el momento del experimento.
3. preparación de rebanadas cerebrales agudas
-
Preparación de la zona de disección
- Preparar 70 mL de colina-aCSF helada en un vaso de precipitado de 80 mL colocado en hielo, para perfusión cardiaca, enfriamiento rápido del cerebro y de corte. Preparar 150 mL de colina-aCSF de 200 mL cristalizando el plato, colocada en un baño de agua caliente mantenido a 32 ° C. Colocar un colador de malla de nylon en la cristalización para retener las rebanadas. Esto se utilizará para dejar las rodajas recuperar durante 10 min después de rebanar.
- Por lo menos 30 minutos antes de comenzar la disección (sección 3.2), empezar a burbujear estas dos soluciones (70 mL de colina-aCSF sobre hielo) y 150 mL de colina-aCSF a 32 ° C con carbogen. Mantener carbogenation constante durante todo el procedimiento.
- Preparar el dispositivo de la cámara de la interfaz (figura 1), que se utilizará para mantener sectores hasta su uso.
- En una alimento sellado caja (10 x 10 cm o 10 cm de diámetro, 8 cm de altura), instalada en un agitador magnético, colocar un mL 200 cristalizando el plato con una barra de imán.
- Añadir 200 mL de aCSF en este plato la cristalización y coloque el soporte de rebanada de interfaz 3D impreso en la parte superior (titular de rebanada de la interfaz se compone de dos perfectamente partes de la instalación, con una malla de poliamida que se extendía entre ellos, figura 1A, B).
- Quitar exceso volumen del plato para mantener sólo una delgada película de solución cubriendo la malla interfaz rebanada del titular de la cristalización. Más adelante se creará un borde fino de la solución que rodea las rebanadas (pero sin que los cubre).
- Poner unos cuantos milímetros de la aCSF en la parte inferior de la caja de alimento y empezar a burbujear con carbogen (en el primer uso, hacer un pequeño agujero en la pared de la caja de sellado de alimentos para asegurarse de que la tubería puede introducirlo en la caja).
- Cierre la caja sellada manteniendo constante carbogenation. Esto creará un humidificado 95% O25% CO2 ambiente rico en el que las rebanadas serán transferidas después de su recuperación en colina-aCSF y mantenidas antes que imagen. Este dispositivo es en adelante la "cámara de interfaz" (figura 1).
-
Disección de cerebro y rebanar
- Anestesiar el ratón con una inyección intraperitoneal de 50 mg·mL-1 pentobarbital (0,15 mL/20 g de peso corporal de ratón), inmovilizarlo, exponer el corazón y realizar una perfusión cardiaca con 10 mL de helado, carbogenated, colina aCSF (vea el paso 3.1.1), con una bomba peristáltica. Observar la palidez del hígado como indicador de una buena perfusión. La perfusión dura menos de 5 minutos.
- El ratón de decapitar y cortar la piel para exponer el cráneo. Con las tijeras grandes, dos cortes transversales se aplican desde el gran agujero y un corte sagital durante mucho tiempo y con unas pinzas finas, retire las placas de cráneo.
- Rápidamente y con cuidado extraer el cerebro (en menos de 1 min) y lugar para 1 minuto en el vaso de precipitados de 80 mL que contiene el restante (~ 60 mL) helada colina-aCSF (aún bajo constante carbogenation), con el fin de enfriarlo.
- Transferir el cerebro sobre un papel de filtro previamente mojado con la aCSF.
- Cortar el cerebro según la región cerebral de interés y un mejor ángulo de corte. Por ejemplo, para la imagen del tálamo o del hipocampo en rebanadas coronales, corte con una hoja de bisturí el cerebelo y, luego, unos 2 mm de los extremos rostrales y caudales del cerebro.
Nota: Es importante eliminar partes del cerebro que son demasiado rostral o caudal demasiado porque cuanto menor sea la región a recortar antes de llegar a la zona de interés, más rápido el corte. Se recomienda un tiempo total de corte (paso 3.2.7) de menos de 20 minutos. - Para rebanadas coronales, colocar y pegar (con pegamento de cianocrilato) la cara caudal del cerebro en un 10 cm plato de Petri, pegado en el bloque de corte de una máquina de cortar del vibrante y posición en la cámara reservorio de la máquina de cortar vibrante que se coloca en una cámara más grande lleno de hielo. Luego, llene la caja Petri con todos lo restantes helada colina-aCSF.
- Manteniendo constante el 95% O25% CO2 burbujas de la colina helada-aCSF, cortar rebanadas coronales de 300 μm de espesor (velocidad: 0,08 mm·s-1, la vibración de la cuchilla: 60 Hz, amplitud de la vibración: 1 m m).
- Recoger los trozos de cerebro con una transferencia desechable de boca ancha (4 mm de diámetro) pipetear, uno por uno después de cada solo paso de la hoja, para evitar la acumulación de componentes tóxicos liberados por la periferia de las láminas. Tenga cuidado para evitar burbujas de aire durante la transferencia y coloque cada rebanada en la colina aCSF a 32 º C durante unos 10 minutos para la recuperación.
- Con la pipeta, coloque las rebanadas en pedazos de limpieza papel cubierto con una gota de colina-aCSF. Aspirar el exceso de colina-aCSF y, con la espátula, coloque las rodajas, en la limpieza de lentes, en el acoplamiento de la cámara de la interfaz que contiene aCSF carbogenated a temperatura ambiente (véase 3.1.3.5). Deje que la rodaja recupera en este ambiente durante al menos 30 minutos.
Nota: Después de esto, los trozos están listos y pueden utilizarse para los ratones de la microglia, proyección de imagen de hasta 6 h después de la extracción de cerebro de jóvenes (menos de un mes de edad) y hasta 4 h después de la extracción de cerebro de los adultos de dos meses de edad.
4. dos fotones microscopía
- Parámetros de ajuste
- Encienda el sistema multifotón (detectores híbridos, láser, escáner, modulador electro-óptico, microscopio).
- Ajustar el láser en 920 nm, compruebe que el láser está bloqueado por el modo y la energía en 5% - 15% y el aumento en 10%. Esto corresponde a una potencia de 3-5 mW en el objetivo. Asegurarse de que contratan a los detectores de nondescanned e instalación los filtros adecuados de emisión y excitación.
- Definir los parámetros del proyección de imagen de software a los siguientes valores: por el tamaño del marco, 1024 x 1024 píxeles correspondiente a un área de 295.07 x 295.07 μm; para el zoom, 2. Si la señal es muy ruidosa, se aplican un promedio de línea de 2. Para la dinámica de pixel, ajustar el software de imagen a 12 bits o más.
Nota: Imágenes con un valor más alto de bits permiten a los investigadores distinguir diferencias más pequeñas en la intensidad de fluorescencia que las imágenes con un valor de bit inferior: un cambio de un valor de gris en una imagen de 8 bits corresponde a un cambio de 16 valores de grises de 12 bits y de 256 grises valores i n una imagen de 16 bits. Por lo tanto, imágenes de bits más altas son más apropiados para el análisis cuantitativo, pero como su tamaño aumenta con la profundidad de bits, capacidad de almacenamiento y computación potencia puede llegar a ser limitantes. - Seleccione el modo de barrido XYZT con un rango Z-intervalo 2 μm y un intervalo de T de 2 minutos.
Nota: X, y y z resolución están determinados por el teorema de muestreo de Nyquist. Una magnitud Z alrededor de 0.8 sería óptimo para resolver procesos de microglia (con un diámetro de < 1 μm), pero es la limitación de la resolución óptica de microscopía multifotón (a 920 nm con un objetivo de NA 0.95, la resolución axial es alrededor de 1 μm). De barrera física, en un experimento de la proyección de imagen en vivo, la sensibilidad o signal to noise ratio, la resolución, la velocidad y la materia del tiempo de observación total. Teniendo en cuenta todos estos parámetros, un paso z de 2 μm (al igual que en numerosos estudios3,11,14), un tamaño de 1024 x 1024 píxeles y una adquisición de alta velocidad utilizando un escáner resonante acoplado a detectores de HyD (it toma alrededor de 15 s para adquirir planes de z 50) fueron seleccionados aquí. La frecuencia de adquisiciones es una serie XYZT cada 2 min y la duración total es de 30 minutos. Si la configuración no es rápido o bastante sensible, es posible reducir la resolución lateral (hasta 512 x 512) o el número de rodajas z (por proyección de imagen exclusivamente en la profundidad de z que exhibe la fluorescencia más fuerte [es decir, no el más profundas rodajas z donde la fluorescencia es débil), o para disminuir la velocidad del escáner. La resolución axial también puede ser disminuida al aumentar el z paso hasta 3 μm, pero como esto puede afectar la cuantificación, deben realizarse todos los experimentos que se comparará con el mismo paso de z.
Nota: Es posible realizar experimentos similares en rebanadas del CX3CR1creER-YFP ratones18, una línea de ratón utilizada para inducir la supresión genética en microglia solamente y en que microglia constitutivamente expresan proteína amarilla fluorescente (YFP). Sin embargo, el nivel de expresión de YFP es muy bajo en comparación con la proteína fluorescente verde (GFP) en CX3CR1GFP / + ratones; así, la proyección de imagen es posible pero difícil y requiere la optimización de los parámetros de adquisición. Se recomienda ajustarlos como sigue. - Ajustar el láser en 970 nm (que está mejor adaptada a excitación de YFP de 920 nm), la potencia al 50% y la ganancia al 50%, que corresponde a una energía del laser con el objetivo de 5-6 mW.
- Establecer un promedio de línea de 4 (o más) para mejorar la relación señal a ruido.
- Posicionamiento de la rebanada y de la micropipeta de vidrio y la aplicación local del compuesto
- Conecte la bomba peristáltica a la cámara de grabación, 30 min antes de iniciar la grabación. Después de limpiar el sistema de perfusión todo con 50 mL de agua ultrapura, iniciar la perfusión de la cámara de grabación con aCSF (50 mL) contenida en un vaso de vidrio en carbogenation constante. Durante todo el experimento, mantenga la aCSF circulante a 32 ° C con un microheater en línea o con un calentador Peltier.
Nota: Una cámara de perfusión específica con la perfusión de la parte superior e inferior está diseñada para optimizar la oxigenación en ambos lados de la rebanada. La cámara de perfusión se compone de dos perfectamente partes de la instalación, con una malla de poliamida que se extendía entre ellos (figura 2A, B). En comparación con otros tipos de cámaras, donde el segmento está colocando directamente sobre un cubreobjetos de vidrio, esta cámara reduce la muerte neuronal en la parte inferior de la rebanada, mejora la viabilidad y reduce los movimientos de corte inducidos por la inflamación. - Con una pipeta de transferencia desechable de boca ancha, transferir la rebanada del cerebro para ser reflejada en el vaso de la aCSF Retire el papel de la lente, que se hunden (como una prueba que no se ha fijado ninguna burbuja de aire) y transferir a la cámara de grabación (perfusión).
- Coloque un soporte de rebanada (una horquilla hecha de platino con las dos ramas Unidas por hilos de nylon paralelo) en el segmento para minimizar el movimiento de la rebanada debido al flujo de perfusión.
- Utilizar la iluminación de campo claro a la región cerebral de interés (tiempo de exposición: 50 a 80 ms) usando un objetivo de bajo aumento (5 X o 10 X). Cambiar al mayor objetivo de inmersión de agua aumento (25 x con un lente x 0,35) y ajustar la posición.
Nota: Evitar a los campos de la imagen cerca de hilos de rosca de nylon del titular de la corte ya que pueden bloquear la luz y deformarse localmente la rebanada. Asegúrese de que el área de interés es plana. Si es necesario, retire la rebanada con el fin de reposicionar el segmento o el titular de la corte. - Utilizar la iluminación de fluorescencia para localizar las células microgliales fluorescente para ser reflejada en el campo (tiempo de exposición: 250-500 ms).
Nota: Este paso permite a los investigadores para comprobar la presencia de células en la región de interés y de su intensidad de fluorescencia y para controlar la cantidad de detritos celulares. - Rellene la pipeta con 10 μl de la aCSF con ATP, 5-HT, o la droga de interés en su concentración final. Punto de la punta hacia abajo y sacuda suavemente la pipeta llena de drogas para eliminar las posibles burbujas de aire atrapados en la punta.
Nota: Si la solución que se inyecta tiende a formar burbujas, considere el uso de pipetas de borosilicato con un filamento interno. La salida de ATP fuera de la pipeta puede atraer procesos microglial incluso antes de la inyección (si esto ocurre, será visible en la etapa de análisis). Aunque esto debería ser moderado con la concentración de ATP utilizado (500 µmol· L-1), si es un problema, considere prefilling la micropipeta con 2 mL de la aCSF antes de añadir el ATP (u otro compuesto) solución paso 4.2.6. - Monte la pipeta llena en un soporte de pipeta, conectado con tubos transparentes a una jeringa de 5 mL con un émbolo colocado en la posición de 5 mL. El titular de la pipeta se está montado en un micromanipulador de tres ejes.
- Bajo iluminación de campo claro, utilizar el instrumental quirúrgico para colocar la pipeta en el centro del campo. Para un centrado óptimo y reproducible, mostrar y utilizar las reglas de la imagen.
- Inferior de la pipeta suavemente hacia el segmento, controlar y ajustar ligeramente el objetivo al mismo tiempo, hasta que la punta toque la superficie de la rodaja. Detener el descenso de la pipeta en cuanto es visible que se ha tocado el sector permite la punta de la pipeta penetrar 80-100 μm de la superficie de la rodaja (ver figura 3B).
- Ajustar el láser (vea los parámetros antedichos) y encienda el microscopio al modo de multifotón. Asegúrese de que la cámara es defendida de cualquier fuente de luz (por ejemplo, una pantalla de ordenador). Conecte los detectores de nondescanned y ajuste la ganancia. Utilice una tabla de búsqueda (LUT) con un límite superior codificados por colores para no saturar los píxeles de la imagen.
- Determinar el espesor de la rebanada para ser reflejada (es decir, el superiores e inferiores z-posiciones donde la fluorescencia es detectable [generalmente entre 220 y 290 μm en total]).
Nota: En la superficie de la rebanada, hay una mayor densidad de procesos y posiblemente de la microglía, a menudo con una morfología inusual, en comparación con el interior de la rebanada. Esta acumulación será más llamativa con el tiempo (es decir,., más visible en la última que en la primera rebanada del cerebro a ser reflejada). Por lo tanto, los planos de z en el primero ~ 30 μm no deben utilizarse para el análisis y pueden ser saltados incluso para la adquisición. - Iniciar la grabación de una duración total de 30 minutos (o más si así lo desea) y después de una base de 5 minutos, aplicar localmente el compuesto a probar (sin necesidad de interrumpir la proyección de imagen). Para hacer esto, Presione lentamente el émbolo de la jeringa conectada a la micropipeta de 5 mL a la posición de 1 mL (en cerca de 5 s). Resistencia al presionar el émbolo debe sentirse inmediatamente. Si no es así, la punta puede ser rota.
Nota: Un experimentador entrenado, las inyecciones con este método son reproducibles, pero como alternativa a la manipulación manual de una jeringa, la pipeta puede vincularse a un sistema de eyección automática de la presión para permitir un mejor control del volumen entregado. La inyección crea una distorsión física de la rebanada en el sitio de la inyección. Esta distorsión es visible a posteriori en los primeros dos o tres imágenes después de la inyección pero no debe ser visible en la cuarta imagen, (es decir, 8 min después de la inyección). Si persiste, considere cambiar los parámetros para la preparación de la pipeta. - Al final de la adquisición (30 min), deseche la micropipeta y retire el segmento. Si se desea, fijar el segmento para más inmunomarcación. Por ejemplo, el método de instantánea está optimizado para la fijación y tinción de gruesas rebanadas de23.
- Antes de comenzar un nuevo sector de la imagen, hacer la película 2D (sección 5.1) para comprobar eso microglia tienen una morfología normal y se están moviendo y, por tanto, que las rodajas estén sanas.
- Conecte la bomba peristáltica a la cámara de grabación, 30 min antes de iniciar la grabación. Después de limpiar el sistema de perfusión todo con 50 mL de agua ultrapura, iniciar la perfusión de la cámara de grabación con aCSF (50 mL) contenida en un vaso de vidrio en carbogenation constante. Durante todo el experimento, mantenga la aCSF circulante a 32 ° C con un microheater en línea o con un calentador Peltier.
5. Análisis de la atracción de los procesos de Microglial
-
Proyección 2D y corrección de la deriva
- Abra el archivo (. LIF) con Fiji24.
- Si es necesario, hacer un substack (Substack de imagen/pilas/herramientas/hacer) con sólo los z-planos de interés. Por ejemplo, excluir los planos z correspondiente a la superficie de la rebanada, si han sido adquiridos pero no deben utilizarse para el análisis (véase la nota después del paso 4.2.11) y los planos más profundos de z con ninguna fluorescencia. La pila final generalmente contiene 90-110 z-rebanadas (180-220 μm).
- Iniciar la función de proyecto Z (Proyecto imagen/pilas/Z") y seleccione el tipo de proyección de Máxima intensidad para hacer las proyecciones de la pila de z en cada punto del tiempo.
- Lanzar el MultiStackReg plugin (Plugin/registro/MultiStackReg), seleccionar acción 1: Alinee el y transformación: cuerpo rígido corregir derivas leve que puedan haber ocurrido durante la adquisición. Guardar esta película 2D como un archivo nuevo (.tiff).
-
Procesamiento de datos
- Abra este nuevo archivo con hielo25.
- Dibujar una región circular de R1 de interés (ROI) de 35 μm de diámetro, centrado en el sitio de inyección (identificado en particular por la sombra de la pipeta y la distorsión creada en el momento de la inyección).
- Usar el plugin evolución de intensidad de retorno de la inversión y medir la intensidad media en el tiempo en R1.
- Guardar los resultados a una. Archivo XLS.
-
Cuantificación y representación de los resultados
- Para cuantificar la respuesta microglial con el tiempo, determinar en cada momento
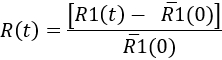
Here,'R1(0) es la media de los valores de R1(t) antes de la inyección. Entonces, los resultados pueden ser representados como una cinética de la respuesta microglial, o en un momento específico del punto (véase figura 7).
- Para cuantificar la respuesta microglial con el tiempo, determinar en cada momento
Access restricted. Please log in or start a trial to view this content.
Resultados
Este protocolo describe un método para inducir, observar y cuantificar el crecimiento orientado de microglial procesos hacia un compuesto localmente aplicado, por ejemplo, ATP o 5-HT, en cerebral aguda tajadas de joven o adulto (al menos hasta dos meses) ratones. Entre los factores que contribuyen al mantenimiento de rodajas de cerebro de animales adultos en un estado saludable durante varias horas es el uso de dos herramientas diseñadas para optimizar la supervivencia de la célula a d...
Access restricted. Please log in or start a trial to view this content.
Discusión
Al mantener, a diferencia de en disociado o organotypic de la rebanada cultura, una integridad estructural con los ajustes de red limitada, rebanadas de cerebro agudo permiten a los investigadores para el estudio de microglia en su entorno fisiológico. Sin embargo, una de las limitaciones más importantes es el hecho de que el procedimiento de corte crea lesiones que rápidamente pueden poner en peligro la viabilidad de las neuronas, especialmente en el cerebro adulto. Como microglia son particularmente reactivas a dañ...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a la célula y tejido de imágenes de la Institut du Fer à Moulin, donde se han realizado todos los análisis y adquisición de la imagen. Este trabajo ha sido apoyado en parte por el Centre National de la Recherche Scientifique, el Institut National de la Santé et de la Recherche Médicale, las Ciencias de la Universidad de Sorbonne y por donaciones de Sorbonne Universités-Pierre et Marie Curie Universidad ( Programa Emergence-UPMC 2011/2014), la Fondation pour la Recherche sur le Cerveau, Fondation de France, la Fondation pour la Recherche Médicale "Equipe FRM DEQ2014039529", el Ministerio francés de la investigación (Agence Nationale pour la Recherche ANR-17-CE16-0008 y el Avenir Investissements programa "Bio-Psy empresa Labex" ANR-11-IDEX-0004-02) y una investigación en colaboración en el programa de neurociencia computacional, Agencia Nacional de la ciencia francés Fundación Nacional para la investigación (número: 1515686). Todos los autores están afiliados a la investigación de los grupos que son miembros de la escuela de París de Neurociencia (PEV) y de la empresa Labex Bio-Psy. F.E. es estudiante de doctorado asociado a Sorbonne Université, Colegio Doctoral, F-75005 París, Francia y está financiado por la empresa Labex Bio-Psy. V.M. es becario posdoctoral financiado por la investigación colaborativa en el programa de neurociencia computacional, Agencia Nacional de la ciencia francés Fundación Nacional para la investigación (número: 1515686). Los autores agradecen a Marta Kolodziejczak que participaron en la iniciación del proyecto.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| for pipettes preparation | |||
| Clark Borosilicate Thin Wall Capillaries | Harvard Apparatus | 30-0065 | Borosilicate Thin Wall without Filament, 1.5 mm OD, 1.17 mm ID, 75 mm L , Pkg. of 225 |
| DMZ Universal Puller | Zeitz Instrumente | ||
| Name | Company | Catalog Number | Comments |
| for solutions | |||
| Calcium Chloride dihydrate (CaCl2) | Sigma | C5080 | |
| Choline Chloride | Sigma | C7527 | |
| D-(+)-Glucose | Sigma | G8270 | |
| L-Ascorbic acid | Sigma | A5960 | |
| Magnesium Chloride solution 1 M (MgCl2) | Sigma | 63020 | |
| Potassium chloride SigmaUltra >99.0% (KCl) | Sigma | P9333 | |
| Sodium bicarbonate (NaHCO3) | Sigma | S5761 | |
| Sodium Chloride (NaCl) | Sigma | S5886 | |
| Sodium phosphate monobasic | Sigma | S5011 | |
| Sodium pyruvate | Sigma | P2256 | |
| Ultrapure water | MilliQ | for all the solutions | |
| Name | Company | Catalog Number | Comments |
| for slice preparation | |||
| 2x 200 mL crystalizing dishes | |||
| 80 mL Pyrex beaker | |||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For mice perfusion and 2-photon chamber perfusion (aCSF) |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | ||
| Dolethal | Vetoquinol | Dolethal 50 mg/mL | |
| Filter papers (Whatman) | Sigma | WHA1001042 | Whatman qualitative filter paper, Grade 1 (Pore size: 11µM) |
| Fine Scissors - Sharp | Fine Science Tools | 14060-60 | |
| Food box 10 cm diameter, 8 cm Height | |||
| glue (ethyl cyanoacrylate) | Loctite | super glue 3 power flex | |
| Hippocampal Tool (spatula) | Fine Science Tools | 10099-15 | The largest extremity has to be angled at 90° |
| Ice | |||
| Iris Forceps (curved) | Moria | MC31 | |
| Lens cleaning tissue | THOR LABS | ||
| Nylon mesh strainer | diameter 7 cm | ||
| Razor blades | Electron Microscopy Sciences | 72000 | For the slicer |
| scalpel blade | |||
| Slice interface holder | home-made, the file for 3D printing is provided in Supplemental Material | ||
| Surgical Scissors - Sharp | Fine Science Tools | 14002-14 | |
| Vibrating slicer | Thermo Scientific | 720-2709 | Model: HM 650V (Vibrating blade microtome) |
| Water bath | Set at 32 °C (first recovery step) | ||
| Name | Company | Catalog Number | Comments |
| for slice imaging | |||
| × 25 0.95 NA water-immersion objective | Leica Microsystems (Germany) | HCX Irapo | |
| 2-photon MP5 upright microscope with resonant scanners (8 kHz) and two HyD Hybrid detectors | Leica Microsystems (Germany) | ||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For 2-photon chamber perfusion with aCSF |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | I1501L50R2A001 | |
| Chameleon Ultra2 Ti:sapphire laser | Coherent (Germany) | ||
| disposable transfer pipettes , wide mouth | ThermoFischer scientific | for example : 232-11 | 5.8 mL with fin tip, but we cut it (approx 7 cm) to have a 4 mm diameter mouth |
| emission filter SP680 | Leica Microsystems (Germany) | ||
| fluorescent cube containing a 525/50 emission filter and a 560 dichroic filter (for fluorescence collection) | Leica Microsystems (Germany) | ||
| glass beaker with 50 mL of ACSF to maintain constant perfusion of the slice | |||
| Heating system | Warner Instrument Corporation | Automatic Heater Controller TC-324B | to maintain perfusion solution at 32 °C |
| perfusion chamber | home-made, the file for 3D printing is provided in Supplemental Material | ||
| slice holder ("harp") | home made : hairpin made of platinum with the two branches joined by parallel nylon threads | ||
| Name | Company | Catalog Number | Comments |
| for slice stimulation | |||
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A-26209 | to be prepared ex-temporaneously : 1 mg/mL (3 mM) stock solution prepared the day of the experiment, kept at 4 °C (a few hours) and diluted just before use |
| Fluorescein (optional) | Sigma | F-6377 | use at 1 µM final |
| Micromanipulator | Luigs and Neumann | SM7 | connected to the micropipette holde |
| Micropipette holder | same as for eletrophysiology | ||
| Serotonin hydrochloride | Sigma | H-9523 | aliquots of 50mM stock solution in H20 kept at -20 °C. 500 µM solution prepared the day of the experiment. |
| Syringe 5 mL (without needle) | Terumo medical products | SS+05S1 | |
| Transparent tubing | Fischer Scientific | 11750105 | Saint Gobain Performance Plastics™ Tygon™ E-3603 Non-DEHP Tubing |
| Name | Company | Catalog Number | Comments |
| for image analysis | |||
| Fiji | https://fiji.sc | Schindelin, J. et al Nat. Methods (2012) doi 10.1038 | |
| Icy | Institut Pasteur | http://icy.bioimageanalysis.org | de Chaumont, F. et al. Nat. Methods (2012) |
| Name | Company | Catalog Number | Comments |
| mice | |||
| CX3CR1-GFP mice | Jung et al, 2000 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. | |
| CX3CR1creER-YFP mice | Parkhurst et al 2013 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. |
Referencias
- Salter, M. W., Stevens, B. Microglia emerge as central players in brain disease. Nature Publishing Group. 23 (9), 1018-1027 (2017).
- Tay, T. L., Savage, J., Hui, C. W., Bisht, K., Tremblay, M. -È Microglia across the lifespan: from origin to function in brain development, plasticity and cognition. The Journal of Physiology. , (2016).
- Davalos, D., et al. ATP mediates rapid microglial response to local brain injury in vivo. Nature Neuroscience. 8 (6), 752-758 (2005).
- Nimmerjahn, A. Resting Microglial Cells Are Highly Dynamic Surveillants of Brain Parenchyma in Vivo. Science. 308 (5726), 1314-1318 (2005).
- Dissing-Olesen, L., et al. Activation of neuronal NMDA receptors triggers transient ATP-mediated microglial process outgrowth. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10511-10527 (2014).
- Gyoneva, S., Traynelis, S. F. Norepinephrine modulates the motility of resting and activated microglia via different adrenergic receptors. Journal of Biological Chemistry. 288 (21), 15291-15302 (2013).
- Eyo, U. B., et al. Neuronal hyperactivity recruits microglial processes via neuronal NMDA receptors and microglial P2Y12 receptors after status epilepticus. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10528-10540 (2014).
- Hristovska, I., Pascual, O. Deciphering Resting Microglial Morphology and Process Motility from a Synaptic Prospect. Frontiers in Integrative Neuroscience. 9, 1231(2016).
- Avignone, E., Lepleux, M., Angibaud, J., Nägerl, U. V. Altered morphological dynamics of activated microglia after induction of status epilepticus. Journal of Neuroinflammation. 12, 202(2015).
- Abiega, O., et al. Neuronal Hyperactivity Disturbs ATP Microgradients, Impairs Microglial Motility, and Reduces Phagocytic Receptor Expression Triggering Apoptosis/Microglial Phagocytosis Uncoupling. PLoS Biology. 14 (5), e1002466(2016).
- Madry, C., et al. Microglial Ramification, Surveillance, and Interleukin-1β Release Are Regulated by the Two-Pore Domain K+Channel THIK-1. Neuron. 97 (2), 299-312 (2018).
- Honda, S., et al. Extracellular ATP or ADP induce chemotaxis of cultured microglia through Gi/o-coupled P2Y receptors. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 21 (6), 1975-1982 (2001).
- Haynes, S. E., et al. The P2Y12 receptor regulates microglial activation by extracellular nucleotides. Nature Neuroscience. 9 (12), 1512-1519 (2006).
- Wu, L. -J., Vadakkan, K. I., Zhuo, M. ATP-induced chemotaxis of microglial processes requires P2Y receptor-activated initiation of outward potassium currents. Glia. 55 (8), 810-821 (2007).
- Kolodziejczak, M., et al. Serotonin Modulates Developmental Microglia via 5-HT 2BReceptors: Potential Implication during Synaptic Refinement of Retinogeniculate Projections. ACS Chemical Neuroscience. 6 (7), 1219-1230 (2015).
- Schafer, D. P., et al. Microglia Sculpt Postnatal Neural Circuits in an Activity and Complement-Dependent Manner. Neuron. 74 (4), 691-705 (2012).
- Pfeiffer, T., Avignone, E., Nägerl, U. V. Induction of hippocampal long-term potentiation increases the morphological dynamics of microglial processes and prolongs their contacts with dendritic spines. Scientific Reports. 6, 32422(2016).
- Parkhurst, C. N., et al. Microglia Promote Learning-Dependent Synapse Formation through Brain-Derived Neurotrophic Factor. Cell. 155 (7), 1596-1609 (2013).
- Wu, Y., Dissing-Olesen, L., Macvicar, B. A., Stevens, B. Microglia: Dynamic Mediators of Synapse Development and Plasticity. Trends in Immunology. 36 (10), 605-613 (2015).
- Ohsawa, K., et al. P2Y12 receptor-mediated integrin-beta1 activation regulates microglial process extension induced by ATP. Glia. 58 (7), 790-801 (2010).
- Kurpius, D., Wilson, N., Fuller, L., Hoffman, A., Dailey, M. E. Early activation, motility, and homing of neonatal microglia to injured neurons does not require protein synthesis. Glia. 54 (1), 58-70 (2006).
- Stence, N., Waite, M., Dailey, M. E. Dynamics of microglial activation: a confocal time-lapse analysis in hippocampal slices. Glia. 33 (3), 256-266 (2001).
- Dissing-Olesen, L., Macvicar, B. A. Fixation and Immunolabeling of Brain Slices: SNAPSHOT Method. Current Protocols in Neuroscience. 71, 1.23.1-12 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Aitken, P. G., et al. Preparative methods for brain slices: a discussion. Journal of Neuroscience Methods. 59 (1), 139-149 (1995).
- Paris, I., et al. ProMoIJ: A new tool for automatic three-dimensional analysis of microglial process motility. Glia. 66 (4), 828-845 (2018).
- Pagani, F., et al. Defective microglial development in the hippocampus of Cx3cr1 deficient mice. Frontiers in Cellular Neuroscience. 9 (229), 111(2015).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods in Molecular Biology. , Clifton, NJ. 221-242 (2014).
- Mainen, Z. F., et al. Two-photon imaging in living brain slices. Methods. 18 (2), San Diego, CA. 231-239 (1999).
- Tanaka, Y., Tanaka, Y., Furuta, T., Yanagawa, Y., Kaneko, T. The effects of cutting solutions on the viability of GABAergic interneurons in cerebral cortical slices of adult mice. Journal of Neuroscience Methods. 171 (1), 118-125 (2008).
- Gyoneva, S., et al. Systemic inflammation regulates microglial responses to tissue damage in vivo. Glia. 62 (8), 1345-1360 (2014).
- Heindl, S., et al. Automated Morphological Analysis of Microglia After Stroke. Frontiers in Cellular Neuroscience. 12, 106(2018).
- Dailey, M. E., Eyo, U., Fuller, L., Hass, J., Kurpius, D. Imaging microglia in brain slices and slice cultures. Cold Spring Harbor Protocols. 12 (12), 1142-1148 (2013).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados