È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging del due-fotone di attrazione dei processi Microglial verso ATP o serotonina nelle fette del cervello acuto
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Microglia, le cellule immunitarie residente del cervello, di rispondere rapidamente con i cambiamenti morfologici a modificazioni del loro ambiente. Questo protocollo viene descritto come utilizzare microscopia del due-fotone per studiare l'attrazione dei processi microglial verso la serotonina o ATP nelle fette acuta del cervello dei topi.
Abstract
Cellule della microglia sono cellule immuni innate residente del cervello che costantemente la scansione loro ambiente con i loro processi lunghi e, al momento di rottura dell'omeostasi, subiscono rapidi cambiamenti morfologici. Ad esempio, una lesione laser induce in pochi minuti una crescita orientata dei processi microglial, chiamato anche "motilità direzionale", verso il luogo della ferita. Un effetto simile può essere ottenuto fornendo localmente ATP o serotonina (5-hydroxytryptamine [5-HT]). In questo articolo, descriviamo un protocollo per indurre una crescita direzionale dei processi microglial verso un'applicazione locale di ATP o 5-HT in fettine di cervello acuto di topi giovani e adulti e per questa attrazione nel tempo della battuta da microscopia multifotonica. Viene proposto un semplice metodo di quantificazione con software di analisi di immagini gratuito e open-source. Una sfida che ancora caratterizza fette del cervello acuto è il periodo di tempo limitato, diminuisce con l'età, durante il quale le cellule rimangono in uno stato fisiologico. Questo protocollo, quindi, evidenzia alcuni miglioramenti tecnici (sezione imaging camera con un'aspersione doppia interfaccia medio, aria-liquido) volti ad ottimizzare la vitalità delle cellule microgliali sopra parecchie ore, soprattutto in fette da topi adulti.
Introduzione
Cellule della microglia sono macrofagi residenti del cervello e svolgono un ruolo in entrambe le condizioni fisiologiche e patologiche1,2. Hanno una morfologia altamente ramificata e sono costantemente estendere e ritrarre i loro processi3,4. Questo comportamento "scansione" si crede di essere connessi e necessari per l'indagine del loro ambiente. La plasticità morfologica di microglia si esprime in tre modalità. In primo luogo, alcuni composti modulano rapidamente microglial morfologia: l'aggiunta di ATP5,6 o NMDA5,7 nel mezzo di balneazione fette del cervello acuto aumenta la complessità delle ramificazioni microglial, considerando che la noradrenalina diminuisce6. Questi effetti sono direttamente mediati dai recettori microglial (per ATP e noradrenalina) o richiedano un rilascio di ATP dai neuroni (per NMDA). In secondo luogo, la velocità di crescita e la ritrazione dei processi microglial, chiamato motilità o "sorveglianza", può essere influenzata da fattori extracellulari8, omeostasi interruzioni9,10o mutazioni9, 10,11. In terzo luogo, oltre a queste modifiche isotropiche di morfologia e la motilità, microglia hanno la capacità di estendere i loro processi direzionalmente verso una pipetta fornire ATP3,5,12, 13 , 14, nella cultura, in fettine di cervello acuto o in vivo, o consegna 5-HT nel cervello acuto fette15. Tale crescita orientata dei processi microglial, chiamato anche motilità direzionale, in primo luogo è stato descritto come una risposta a un locale laser lesione3,4. Pertanto, fisiologicamente, può essere relazionato con la risposta alla lesione o necessaria per il targeting microglial processi verso sinapsi o regioni del cervello che richiedono potatura durante sviluppo15,16, o nel fisiologico17 ,18,19 o situazioni patologiche9,18,19,20 in età adulta. I tre tipi di cambiamenti morfologici si basano su diversi meccanismi intracellulari11,13,20, e un composto dato non necessariamente modula tutti loro (ad es., NMDA, che agisce indirettamente sul microglia, ha un effetto sulla morfologia ma non induce la motilità direzionale5,7). Pertanto, al fine di caratterizzare l'effetto di un composto, una mutazione o una patologia sulla microglia, è importante caratterizzare i tre componenti della loro plasticità morfologica. Qui, descriviamo un metodo per studiare la crescita direzionale dei processi microglial verso una fonte locale di composto, che è, qui, ATP o 5-HT.
Ci sono diversi modelli per lo studio di attrazione dei processi microglia: colture primarie in ambiente 3D6,18,19, cerebrale acuto fette6,13,15e in vivo 3,13di imaging. Approccio in vivo è il migliore per mantenere lo stato fisiologico di microglia. Tuttavia, videomicroscopia imaging di regioni profonde richiede interventi chirurgici complessi e, pertanto, è spesso limitato a strati corticali superficiali. L'uso di colture primarie di microglia è la tecnica più semplice per testare un gran numero di condizioni con un numero limitato di animali. Tuttavia, è Impossibile ottenere la stessa morfologia delle cellule come in vivo, e le cellule perdono la loro interazioni fisiologiche con neuroni e astrociti. Fette del cervello acuto rappresentano un compromesso tra questi due approcci. Questo modello permette ai ricercatori di studiare le strutture cerebrali che altrimenti sono difficili di raggiungere e di immagine ad alta risoluzione in vivo, per indagare fette dalle fasi neonatale, mentre transcranial microscopia è effettuata principalmente nell'età adulta. Infine, rende possibile osservare in tempo reale gli effetti dell'applicazione della droga locale e di ripetere gli esperimenti durante l'utilizzo di un numero limitato di animali. Tuttavia, un problema con le fette di cervello acuto è il tempo limitato (poche ore) durante il quale le cellule rimangono vive, in particolare per le sezioni da topi più vecchi di due settimane e il potenziale di cambiamento della morfologia di microglia sopra tempo21,22 .
Qui, descriviamo un protocollo per preparare fette di cervello acuto di giovani e adulti Cx3cr1GFP / + topi fino a due mesi, con la conservazione della motilità e morfologia di microglia per diverse ore. Abbiamo, quindi, descrivere come utilizzare queste fette per studiare l'attrazione dei processi microglial verso composti come ATP o 5-HT.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Tutti gli esperimenti sono stati approvati dal comitato etico locale (Comitato di Darwin, accordi 1170 # e #10921).
1. preparazione del vetro Micropipette per l'applicazione locale di composti
- Preparare pipette da borosilicato tubi capillari in vetro a parete sottile con un estrattore di elettrodo. Regolare i parametri per ottenere pipette con un diametro di 4-5 µm alle loro estremità. Figura 2D Mostra una pipetta in campo chiaro a basso ingrandimento.
2. soluzioni
- Garantire tale cristalleria unica che è stata pulita da un ciclo di autoclave, seguito da risciacquo x 2 - 3 volte con acqua ultrapura, verrà utilizzato. Mai utilizzare vetreria che è stato a contatto con paraformaldeide.
-
Preparare un 2 mol · L-1 CaCl2 soluzione sciogliendo 14,7 mg di CaCl2·2H2O in 50 mL di acqua di elevata purezza (acqua ultrapura, resistenza 18,2 MΩ; le tracce di metallo in acqua distillata o acqua di rubinetto possono condurre alla qualità fetta non ottimale a causa effetti pro-ossidativi).
- Conservare questa soluzione madre a temperatura ambiente per un massimo di un mese.
-
Il giorno dell'esperimento, preparare 1 L di soluzione di colina-aCSF (liquido cerebrospinale artificiale), cui la composizione è 110 mmol· Colina di L-1 Cl, 25 mmol· Glucosio-1 L, 25 mmol· L-1 NaHCO3, mmol· 7 L-1 MgCl2, 11,6 mmol· Acido ascorbico L-1 , 3,1 mmol· Piruvato di sodio-1 L, 2,5 mmol· L-1 KCl, 1.25 mmol· L-1 NaH2PO4e 0,5 mmol· L-1 CaCl2, 0,5.
- Per preparare questa soluzione, aggiungere, nell'ordine seguente, in un pallone da 1 L è laureato: 0,186 g di KCl, 0,195 g di NaH2PO4, 2,04 g di acido ascorbico, 2,1 g di NaHCO3e 4,5 g di glucosio.
- Circa la metà del volume finale riempire con acqua ultrapura e mescolare fino a completa dissoluzione.
- Aggiungere 0,34 g di sodio piruvato e 15,36 di colina Cl.
Nota: Conviene prima sciogliere la colina Cl con 5-10 mL della soluzione preparata al punto 2.3.2 prima di aggiungere l'intera soluzione. - Aggiungere 7 mL di 1 mol · L-1 MgCl2 e 250 µ l di 2 mol · L-1 CaCl2 (preparata al punto 2.2) alla soluzione.
- Riempire il matraccio graduato fino a 1 L con acqua ultrapura.
- Con una pressione di vapore Osmometro, verifica che l'osmolarità è tra 300 e 310 mΩ. In caso contrario, regolare con glucosio.
- Controllare il pH dopo carbogenation (cioè, pullulante di "carbogen", un mix di 95% O25% CO2) e regolare, se necessario, a 7,3-7,4 con NaOH 10 M.
- Trasferire la soluzione in una bottiglia di vetro per lo stoccaggio. Tenere il flacone in frigorifero fino all'uso (punto 3.1).
Nota: Si raccomanda di fare una soluzione fresca il giorno dell'esperimento. Tuttavia, se necessario, Colina-aCSF possono essere conservati fino a due giorni a 4 ° C.
-
Il giorno dell'esperimento, preparare 1 L di una soluzione di aCSF, cui la composizione è 124 mmol· L-1 NaCl, 26,2 mmol· L-1 NaHCO3, 25 mmol· Glucosio-1 L, 2,5 mmol· L-1 KCl, 2 mmol· L-1 CaCl2, 1 mmol· L-1 MgCl2e 1.25 mmol· L-1 NaH2PO4.
- Per preparare questa soluzione, aggiungere, nell'ordine seguente, in un pallone tarato da: 0,150 g di NaH2PO4, 0,186 g di KCl, 2,2 g di NaHCO3, 4,5 g di glucosio e 7,3 g di NaCl. Portare la soluzione a un volume di 1 L con acqua ultrapura e mescolate energicamente su una piastra stir.
- Aggiungere 1 mL di 1 mol · L-1 MgCl2 e 1 mL di 2 mol · L-1 CaCl2 alla soluzione e al trasferimento della soluzione aCSF per una bottiglia di vetro per lo stoccaggio.
- Verificare se l'osmolarità è 300-310 mΩ· L-1 e, se non, regolarla con glucosio.
- Controllare il pH dopo carbogenation (cioè, pullulante di "carbogen") e regolarlo, se necessario, a 7,3-7,4 con NaOH 10 M.
- Trasferire la soluzione in una bottiglia di vetro per lo stoccaggio. Tenere il flacone in frigorifero fino all'uso (punto 3.1).
Nota: Si raccomanda di fare una soluzione fresca il giorno dell'esperimento. Tuttavia, in alternativa è possibile preparare un 10 x soluzione contenente NaCl, NaHCO3, KCl e NaH2PO4 a 10 volte la concentrazione finale, che possono essere conservati per non più di una settimana a 4 ° C. Rendere il aCSF finale il giorno dell'esperimento diluendo la soluzione stock 10x con acqua ultrapura e aggiungendo il glucosio e CaCl2MgCl2.
- Preparare le soluzioni di droga il giorno dell'esperimento. Utilizzare la soluzione aCSF per portarli alle concentrazioni finali che sono, qui, µmol· 500 L-1 per ATP e 5 µmol· L-1 per la 5-HT.
Nota: Per l'ATP, una soluzione di riserva può essere preparata (ad esempio 50 mM ATP in acqua), memorizzati in forma aliquotato a-20 ° C e diluita con aCSF alla concentrazione finale il giorno dell'esperimento. Al contrario, la soluzione di 5-HT (serotonina-HCl) deve essere preparata dalla polvere il giorno dell'esperimento, a 1 mg·mL-1 in acqua, conservata a 4 ° C per evitare l'ossidazione di 5-HT e diluita in aCSF al tempo dell'esperimento.
3. preparazione di fettine di cervello acuto
-
Preparazione della zona di dissezione
- Preparare 70 mL di colina-aCSF ghiacciata in un becher da mL 80 posizionato su ghiaccio, da utilizzarsi per aspersione cardiaca, rapido raffreddamento del cervello e affettare. Preparare 150 mL di colina-aCSF in 200 mL di cristallizzare il piatto, posizionato in un bagno di acqua riscaldata a 32 ° C. Posto un setaccio di maglia di nylon nel piatto di cristallizzazione per conservare le fette. Questo servirà per consentire le fette ripristinare per 10 minuti appena dopo il taglio.
- Almeno 30 min prima di iniziare la dissezione (sezione 3.2), avviare bubbling di queste due soluzioni (70 mL di colina-aCSF su ghiaccio) e 150 mL di colina-aCSF a 32 ° C con carbogen. Mantenere costante carbogenation durante l'intera procedura.
- Preparare il dispositivo di alloggiamento di interfaccia (Figura 1), che verrà utilizzato per tenere fette fino al loro utilizzo.
- In una scatola sigillata cibo (10 x 10 cm o 10 cm di diametro, altezza 8 cm), installata su un agitatore magnetico, posizionare un 200 mL cristallizzare il piatto con una barra magnete.
- Aggiungere 200 mL di aCSF in questo piatto di cristallizzazione e collocare il supporto per fetta interfaccia 3D-stampato su di esso (il titolare di fetta di interfaccia è composto da due parti perfettamente raccordo, con una maglia di poliammide tesa tra loro, Figura 1A, B).
- Rimuovere il volume in eccesso dal piatto cristallizzazione per mantenere solo una sottile pellicola di soluzione che copre la maglia di titolare di fetta del interfaccia. Più tardi questo creerà un cerchio bene della soluzione che circondano le fette (ma senza ricoprirle).
- Mettere un paio di millimetri di aCSF nella parte inferiore della finestra di cibo e avviare pullulante di carbogen (al primo utilizzo, fare un piccolo foro nella parete del cibo sigillato per assicurarsi che la tubazione possa entrare nella scatola).
- Chiudere la scatola sigillata mantenendo costante carbogenation. Questo creerà un umidificata 95% O25% CO2 ambiente ricco in cui le fette saranno trasferite dopo il loro recupero in colina-aCSF e gestite prima sono evidenziati chiaramente. Questo dispositivo è in appresso denominata "camera interfaccia" (Figura 1).
-
Dissezione del cervello e affettare
- Anestetizzare il mouse con un'iniezione intraperitoneale di 50 mg·mL-1 pentobarbital (0,15 mL/20 g di peso corporeo del mouse), immobilizzare e, esporre il cuore ed eseguire una perfusione cardiaca con 10 mL di ghiacciata, carbogenated, Colina-aCSF (vedere il passaggio 3.1.1), con una pompa peristaltica. Osservare il pallore del fegato come un indicatore di una buona perfusione. La perfusione dura meno di 5 min.
- Decapitare il mouse e tagliare la pelle per esporre il cranio. Con grandi forbici, applicare due tagli trasversali da grande orifizio e un taglio lungo sagittale e, utilizzando una pinzetta, togliere le piastre di cranio.
- Rapidamente e delicatamente estrarre il cervello (in meno di 1 min) e posizionarlo per 1 min nel becher 80 mL contenente il restante (~ 60 mL) ghiacciata Colina aCSF (ancora in fase di costante carbogenation), al fine di raffreddarlo.
- Trasferire il cervello su una carta da filtro precedentemente bagnato con aCSF.
- Tagliare il cervello secondo la regione del cervello di interesse e comodo angolo di taglio. Ad esempio, per realizzare l'immagine del talamo o ippocampo su fette coronali, tagliato con un bisturi il cervelletto e, poi, circa 2 mm dall'estremità rostrale e caudale del cervello.
Nota: È importante rimuovere parti del cervello che sono troppo rostrale o troppo caudale perché il più piccolo della regione di tagliare prima di raggiungere l'area di interesse, più velocemente l'affettamento. Si raccomanda un tempo totale per affettare (punto 3.2.7) di meno di 20 min. - Per le sezioni coronali, posizione e colla (con colla cianoacrilato) la faccia caudale del cervello su un 10cm di Petri, incollato sul blocco di taglio di un'affettatrice vibrante e posizionarlo nel vano serbatoio dell'affettatrice vibra, che è posizionata in una camera più grande riempito con ghiaccio. Poi, riempire la capsula di Petri con tutti i restanti ghiacciata Colina-aCSF.
- Mantenendo costante il 95% O25% CO2 ribolle di gelida Colina-aCSF, tagliare 300 µm di spessore fette coronali (velocità: 0,08 mm·s-1, lama vibrazione: 60 Hz, ampiezza di vibrazione: 1 mm).
- Raccogliere le fette di cervello con un trasferimento monouso a bocca larga (4 mm di diametro) pipettare, uno dopo ogni singolo passaggio della lama, per evitare l'accumulo di componenti tossici rilasciati dalla periferia delle fette. Fare attenzione a evitare bolle d'aria durante il trasferimento e posizionare ogni fetta in colina-aCSF a 32 ° C per circa 10 min per il recupero.
- Con la pipetta, sistemare le fettine su pezzi di pulizia delle lenti carta condita con una goccia di colina-aCSF. Aspirare l'eccesso di colina-aCSF e, con la spatola, mettere le fette, posate sopra il tessuto di pulizia delle lenti, sulla maglia della camera interfaccia contenente aCSF carbogenated a temperatura ambiente (Vedi 3.1.3.5). Lasciate che la fetta di recuperare in questo ambiente per almeno 30 min.
Nota: Dopo questo, le fette sono pronte e possono essere utilizzate per i topi di microglia imaging per fino a 6 h dopo l'estrazione del cervello da giovani (meno di un mese-vecchio) e fino a 4 h dopo l'estrazione del cervello da adulti di due mesi.
4. due-fotone microscopia
- Impostazione dei parametri
- Accendere il sistema multifotonica (rilevatori di ibrido, laser, scanner, modulatore elettro-ottico, microscopio).
- Sintonizzare il laser 920 nm, verificare che il laser è mode-locking e definire la potenza al 5% - 15% e il guadagno al 10%. Ciò corrisponde ad una potenza di 3-5 mW nell'ambito dell'obiettivo. Verificare che i rilevatori di nondescanned sono impegnati e i filtri di eccitazione e di emissione appropriati installati.
- Impostare i parametri del software di imaging per i seguenti valori: per le dimensioni della cornice, 1024 x 1024 pixel corrispondente ad un'area di 295.07 x 295.07 µm; per lo zoom, 2. Se il segnale è molto rumoroso, applicare una media di linea di 2. Per la dinamica di pixel, è possibile impostare il software di imaging a 12 bit o più.
Nota: Immagini con un più alto valore di bit permettono ai ricercatori di distinguere le più piccole differenze nell'intensità di fluorescenza rispetto alle immagini con un valore di bit inferiore: un cambiamento di un valore di grigio in un'immagine di 8 bit corrisponderebbe ad un cambiamento di 16 valori di grigio in un 12-bit e 256 grigi i valori i n un'immagine a 16 bit. Pertanto, immagini più bit sono più appropriati per l'analisi quantitativa, ma la loro dimensione aumenta con la profondità di bit, capacità di memorizzazione e calcolo potenza può diventare limitante. - Selezionare la modalità di scansione XYZT con una gamma di Z-intervallo 2 µm e un T-intervallo di 2 min.
Nota: La x, y e z ad alta risoluzione sono determinati dal teorema del campionamento di Nyquist. Una dimensione di Z-passo intorno a 0,8 sarebbe ottima per risolvere i processi microglia (con un diametro di < 1 µm), ma è limitante la risoluzione ottica di microscopia multifotonica (920 nm con un obiettivo di NA 0,95, la risoluzione assiale è di circa 1 µm). In cima a quello barriera fisica, in un esperimento di live-imaging, la sensibilità o signal-to-noise ratio, la risoluzione, la velocità e la questione del tempo di osservazione totale. Prendendo in considerazione tutti questi parametri, un z-passo di 2 µm (come in numerosi studi3,11,14), dimensioni di un'immagine di 1024 x 1024 pixel e una velocità d'acquisizione utilizzando uno scanner di risonanza accoppiato a rivelatori di HyD (it richiede circa 15 s per acquisire 50 z-piani) sono stati selezionati qui. La frequenza delle acquisizioni è uno XYZT serie ogni 2 minuti e la durata totale è di 30 min. Se il set-up non è abbastanza sensibile o veloce, è possibile ridurre la risoluzione laterale (fino a 512 x 512) o il numero di fette di z (da formazione immagine esclusivamente nella z-profondità che esibisce il più forte fluorescenza [cioè, non il più profonde z-fette dove fluorescenza è debole]), o per diminuire la velocità dello scanner. La risoluzione assiale può essere diminuita anche aumentando il z-passo fino a 3 µm, ma, questo potrebbe pregiudicare la quantificazione, tutti gli esperimenti da confrontare devono essere eseguiti con lo stesso z-passo.
Nota: È possibile effettuare esperimenti simili su fette da CX3CR1creER-YFP topi18, una linea di mouse utilizzata per indurre la delezione genetica nella microglia solo e nel quale microglia esprimono costitutivamente la proteina fluorescente gialla (YFP). Tuttavia, il livello di espressione di YFP è molto basso rispetto alla proteina fluorescente verde (GFP) in CX3CR1GFP / + topi; così, la formazione immagine è possibile ma impegnativo e richiede l'ottimizzazione dei parametri di acquisizione. Si consiglia di regolare come segue. - Sintonizzare il laser su 970 nm (che è meglio adattato all'eccitazione di YFP di 920 nm), la potenza al 50% e il guadagno al 50%, che corrisponde ad una potenza del laser nell'ambito dell'obiettivo di 5-6 mW.
- Impostare una media di linea di 4 (o più) per migliorare il rapporto segnale-rumore.
- Posizionamento della fetta e la micropipetta di vetro, e l'applicazione locale del composto
- Collegare la pompa peristaltica per la camera di registrazione, 30 min prima di iniziare la registrazione. Dopo la pulizia del sistema di perfusione intero con 50 mL di acqua ultrapura, iniziare la perfusione di camera di registrazione con aCSF (50 mL) contenute in un becher di vetro sotto costante carbogenation. In tutto l'esperimento, è necessario mantenere il aCSF circolanti a 32 ° C con un Jajuravan inline o un riscaldatore Peltier.
Nota: Un alloggiamento specifico aspersione con aspersione superiore e inferiore è progettato per ottimizzare l'ossigenazione su entrambi i lati della fetta. La camera di perfusione è composto di due parti perfettamente raccordo, con una maglia di poliammide tesa fra loro (Figura 2A, B). Rispetto ad altri tipi di camere, dove la fetta si posa direttamente sul vetrino coprioggetti, quest'Aula riduce la morte neuronale nella parte inferiore della fetta, migliora la vitalità e riduce i movimenti di fetta indotti dal relativo gonfiamento. - Con una pipetta di trasferimento monouso bocca larga, trasferire la fetta di cervello essere imaged aCSF Becher per rimuovere la carta per lenti, lasciarlo affondare (come una prova che nessuna bolla di aria è collegata) e trasferirlo alla camera di registrazione (perfusione).
- Posizionare un fetta titolare (un tornante in platino con due rami Uniti da fili di nylon paralleli) sulla fetta per ridurre al minimo movimento fetta dovuta al flusso di perfusione.
- Utilizzare l'illuminazione in campo chiaro per indirizzare la regione del cervello di interesse (tempo di esposizione: 50 a 80 ms) utilizzando un obiettivo di basso ingrandimento (5x o 10x). Passare al più alto obiettivo a immersione in acqua ingrandimento (25x con una lente x 0,35) e regolare la posizione.
Nota: Evitare di campi di immagine ai fili di nylon del titolare della fetta che possono bloccare la luce e localmente deformare la fetta. Assicurarsi che l'area di interesse è piatta. Se necessario, rimuovere il titolare fetta al fine di riposizionare la fetta e/o il titolare di fetta. - Utilizzare l'illuminazione a fluorescenza per individuare le cellule microglial fluorescente essere imaged nel campo (tempo di esposizione: 250-500 ms).
Nota: Questo passaggio permette ai ricercatori di controllare la presenza di cellule nella regione di interesse e la loro intensità di fluorescenza e di controllare per la quantità di detriti cellulari. - Recupero informazioni la pipetta con 10 µ l di aCSF con ATP, 5-HT, o il farmaco di interesse alla sua concentrazione finale. Puntare la punta verso il basso e agitare delicatamente la pipetta piena di droga per rimuovere eventuali bolle d'aria intrappolate nella punta.
Nota: Se la soluzione deve essere iniettato tende a formare delle bolle, considerare l'utilizzo di pipette di borosilicato con un filamento interno. Perdita di ATP fuori la pipetta in grado di attrarre microglial processi anche prima dell'iniezione (in questo caso, sarà visibile nella fase di analisi). Anche se questo dovrebbe essere moderato con la concentrazione di ATP utilizzato (500 µmol· L-1), se è un problema, considera prefilling la micropipetta con 2 mL di aCSF prima dell'aggiunta di ATP (o altro composto) soluzione al punto 4.2.6. - Montare la pipetta riempita in un supporto di pipetta, collegato con tubo trasparente per una siringa da 5 mL, con un pistone posizionato in corrispondenza della posizione 5 mL. Il pipetta titolare stesso è montato su un micromanipolatore di tre assi.
- Sotto illuminazione in campo chiaro, è possibile utilizzare il micromanipolatore per posizionare la pipetta al centro del campo. Per un centraggio ottimale e riproducibile, visualizzare e utilizzare i righelli sull'immagine.
- Abbassare la pipetta dolcemente verso la fetta, controllando e regolando l'obiettivo allo stesso tempo, fino a quando la punta della pipetta leggermente tocca la superficie della fetta. Permette di fermare la discesa della pipetta, non appena è visibile che la fetta è stata toccata la punta della pipetta di penetrare 80-100 µm della superficie della fetta (Vedi Figura 3B).
- Sintonizzare il laser (vedere i parametri di cui sopra) e passare il microscopio alla modalità multifotonica. Assicurarsi che la camera è schermata da qualsiasi fonte di luce (per esempio, uno schermo di computer). Accendere i rilevatori di nondescanned e impostare il guadagno. Utilizzare una tabella di ricerca (LUT) con un limite massimo con codifica a colori per evitare di saturare i pixel nell'immagine.
- Determinare lo spessore della fetta a essere imaged (cioè, la superiore e inferiore z-posizioni dove la fluorescenza è rilevabile [solitamente tra 220 e 290 µm in totale]).
Nota: Alla superficie della fetta, c'è un aumento della densità dei processi e possibilmente della microglia, spesso con una morfologia insolita, in confronto all'interno della fetta. Questo accumulo sarà più evidente con il tempo (cioè., più visibile negli ultimi rispetto alla prima fetta di cervello essere imaged). Di conseguenza, i z-Plane nel primo ~ 30 µm non devono essere utilizzati per l'analisi e possono anche essere saltati per l'acquisizione. - Avviare la registrazione per una durata complessiva di 30 minuti (o più se lo si desidera) e dopo una previsione di 5 min, localmente applicare il composto da testare (senza interrompere l'imaging). Per effettuare questa operazione, premere lentamente lo stantuffo della siringa collegata per la micropipetta, da 5 mL per la posizione di 1 mL (circa 5 s). Resistenza quando premendo lo stantuffo deve essere sentito immediatamente. In caso contrario, il suggerimento potrebbe essere rotto.
Nota: Per uno sperimentatore addestrato, le iniezioni con questo metodo sono riproducibili, ma in alternativa alla manipolazione manuale di una siringa, la pipetta potrebbe essere collegata ad un sistema di espulsione automatica pressione per consentire un migliore controllo del volume trasportato. L'iniezione crea una distorsione fisica della fetta presso il sito di iniezione. Questa distorsione è visibile un posteriori nei primi due o tre immagini dopo l'iniezione, ma non dovrebbe essere visibile la quarta immagine, (cioè, 8 min dopo l'iniezione). Se persiste, provare a modificare i parametri per la preparazione di pipetta. - Al termine dell'acquisizione (30 min), scartare la micropipetta e rimuovere la fetta. Se si desidera fissare la fetta per ulteriori immunolabeling. Ad esempio, il metodo SNAPSHOT è ottimizzato per la fissazione e colorazione di spesse fette di23.
- Prima di iniziare una nuova fetta di immagine, il film in 2D (vedere paragrafo 5.1) al fine di controllare tale microglia hanno una morfologia normale e si stanno muovendo e, quindi, che le fette sono sane.
- Collegare la pompa peristaltica per la camera di registrazione, 30 min prima di iniziare la registrazione. Dopo la pulizia del sistema di perfusione intero con 50 mL di acqua ultrapura, iniziare la perfusione di camera di registrazione con aCSF (50 mL) contenute in un becher di vetro sotto costante carbogenation. In tutto l'esperimento, è necessario mantenere il aCSF circolanti a 32 ° C con un Jajuravan inline o un riscaldatore Peltier.
5. analisi dell'attrazione di Microglial processi
-
Proiezione 2D e correzione di deriva
- Aprire il file (. LIF) con Fiji24.
- Se necessario, fare un substack (Immagine/pile/strumenti/Make Substack) con solo i z-Plane di interesse. Ad esempio, escludere i z-Plane corrispondente alla superficie della fetta, se essi sono stati acquisiti, ma non devono essere utilizzati per l'analisi (vedere la nota dopo il passaggio 4.2.11) e i più profondi z-Plane senza fluorescenza. Lo stack finale contiene generalmente 90-110 z-fette (180-220 µm).
- Lanciare la funzione Z project (Progetto di immagine/pile/Z") e selezionare il tipo di proiezione Max intensità per rendere le proiezioni dello z-stack acquisito in ogni momento.
- Lanciare il plugin MultiStackReg (Plugin/registrazione/MultiStackReg), selezionando azione 1: allineare e trasformazione: corpo rigido per correggere lievi derive che possono essersi verificati durante l'acquisizione. Salvare questo film 2D come un nuovo file (TIFF).
-
Trattamento dei dati
- Aprire il nuovo file con Icy25.
- Disegnare un'area circolare di R1 di interesse (ROI) di 35 µm di diametro, centrato sul sito di iniezione (identificato in particolare dall'ombra della pipetta e la distorsione creato al momento dell'iniezione).
- Utilizzare il plugin evoluzione intensità ROI e misurare l'intensità media nel tempo in R1.
- Salvare i risultati di un. File XLS.
-
Quantificazione e rappresentazione dei risultati
- Per quantificare la risposta di microglia nel corso del tempo, determinare in ogni momento
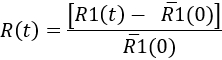
Here,'R1(0) è la media dei valori di R1(t) prima dell'iniezione. Quindi, i risultati possono essere rappresentati come una cinetica della risposta microglial, o in un momento specifico punto (Vedi Figura 7).
- Per quantificare la risposta di microglia nel corso del tempo, determinare in ogni momento
Access restricted. Please log in or start a trial to view this content.
Risultati
Questo protocollo descrive un metodo per indurre, osservare e quantificare la crescita orientata dei processi microglial verso un composto applicato localmente, ad esempio, ATP o 5-HT, nel cervello acuto fette da giovane o adulto (almeno fino a due mesi) topi. Tra i fattori che contribuiscono al mantenimento di fette del cervello da animali adulti in uno stato integro per diverse ore è l'uso di due strumenti progettati per ottimizzare la sopravvivenza delle cellule a due passi del protoc...
Access restricted. Please log in or start a trial to view this content.
Discussione
Mantenendo, a differenza di in dissociato o organotipiche affettare cultura, un'integrità strutturale con regolazioni di rete limitata, fettine di cervello acuto permettono ai ricercatori di studiare microglia nel loro ambiente fisiologico. Tuttavia, uno dei principali limiti è il fatto che la procedura per affettare crea le lesioni che possono rapidamente compromettere la vitalità dei neuroni, specialmente nel cervello adulto. Come la microglia è particolarmente reattivo al danno cellulare, è importante limitare il...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Ringraziamo la cellula e tessuto Imaging Facility del Institut du Fer à Moulin, dove sono state eseguite tutte le analisi e acquisizione delle immagini. Questo lavoro è stato supportato in parte dal Centre National de la Recherche Scientifique, Institut National de la Santé et de la Recherche Médicale, le scienze di Université Sorbonne e dalle sovvenzioni dalla Sorbonne Universités-Pierre et Marie Curie Università ( Programma Emergence-UPMC 2011/2014), la Fondation pour la Recherche sur le Cerveau, Fondation de France, la Fondation pour la Recherche Médicale "Equipe FRM DEQ2014039529", il Ministero francese della ricerca (Agence Nationale pour la Recherche Programma di ANR-17-CE16-0008 e l'Avenir Investissements "Bio-Psy Labex" ANR-11-IDEX-0004-02) e una ricerca collaborativa nel programma di neuroscienze computazionali, National Science Foundation/francese Agenzia nazionale per la ricerca (numero: 1515686). Tutti gli autori sono affiliati per ricercare gruppi che sono membri del Paris School of Neuroscience (PEV) e del Labex Bio-Psy. F.E. è uno studente di dottorato di ricerca affiliato Sorbonne Université, Collège Doctoral, F-75005 Parigi, Francia ed è finanziato dal Labex Bio-Psy. V.M. è un post-dottorato finanziato la ricerca collaborativa nel programma di neuroscienze computazionali, National Science Foundation/francese Agenzia nazionale per la ricerca (numero: 1515686). Gli autori ringraziano Marta Kolodziejczak che hanno partecipato all'inizio del progetto.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| for pipettes preparation | |||
| Clark Borosilicate Thin Wall Capillaries | Harvard Apparatus | 30-0065 | Borosilicate Thin Wall without Filament, 1.5 mm OD, 1.17 mm ID, 75 mm L , Pkg. of 225 |
| DMZ Universal Puller | Zeitz Instrumente | ||
| Name | Company | Catalog Number | Comments |
| for solutions | |||
| Calcium Chloride dihydrate (CaCl2) | Sigma | C5080 | |
| Choline Chloride | Sigma | C7527 | |
| D-(+)-Glucose | Sigma | G8270 | |
| L-Ascorbic acid | Sigma | A5960 | |
| Magnesium Chloride solution 1 M (MgCl2) | Sigma | 63020 | |
| Potassium chloride SigmaUltra >99.0% (KCl) | Sigma | P9333 | |
| Sodium bicarbonate (NaHCO3) | Sigma | S5761 | |
| Sodium Chloride (NaCl) | Sigma | S5886 | |
| Sodium phosphate monobasic | Sigma | S5011 | |
| Sodium pyruvate | Sigma | P2256 | |
| Ultrapure water | MilliQ | for all the solutions | |
| Name | Company | Catalog Number | Comments |
| for slice preparation | |||
| 2x 200 mL crystalizing dishes | |||
| 80 mL Pyrex beaker | |||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For mice perfusion and 2-photon chamber perfusion (aCSF) |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | ||
| Dolethal | Vetoquinol | Dolethal 50 mg/mL | |
| Filter papers (Whatman) | Sigma | WHA1001042 | Whatman qualitative filter paper, Grade 1 (Pore size: 11µM) |
| Fine Scissors - Sharp | Fine Science Tools | 14060-60 | |
| Food box 10 cm diameter, 8 cm Height | |||
| glue (ethyl cyanoacrylate) | Loctite | super glue 3 power flex | |
| Hippocampal Tool (spatula) | Fine Science Tools | 10099-15 | The largest extremity has to be angled at 90° |
| Ice | |||
| Iris Forceps (curved) | Moria | MC31 | |
| Lens cleaning tissue | THOR LABS | ||
| Nylon mesh strainer | diameter 7 cm | ||
| Razor blades | Electron Microscopy Sciences | 72000 | For the slicer |
| scalpel blade | |||
| Slice interface holder | home-made, the file for 3D printing is provided in Supplemental Material | ||
| Surgical Scissors - Sharp | Fine Science Tools | 14002-14 | |
| Vibrating slicer | Thermo Scientific | 720-2709 | Model: HM 650V (Vibrating blade microtome) |
| Water bath | Set at 32 °C (first recovery step) | ||
| Name | Company | Catalog Number | Comments |
| for slice imaging | |||
| × 25 0.95 NA water-immersion objective | Leica Microsystems (Germany) | HCX Irapo | |
| 2-photon MP5 upright microscope with resonant scanners (8 kHz) and two HyD Hybrid detectors | Leica Microsystems (Germany) | ||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For 2-photon chamber perfusion with aCSF |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | I1501L50R2A001 | |
| Chameleon Ultra2 Ti:sapphire laser | Coherent (Germany) | ||
| disposable transfer pipettes , wide mouth | ThermoFischer scientific | for example : 232-11 | 5.8 mL with fin tip, but we cut it (approx 7 cm) to have a 4 mm diameter mouth |
| emission filter SP680 | Leica Microsystems (Germany) | ||
| fluorescent cube containing a 525/50 emission filter and a 560 dichroic filter (for fluorescence collection) | Leica Microsystems (Germany) | ||
| glass beaker with 50 mL of ACSF to maintain constant perfusion of the slice | |||
| Heating system | Warner Instrument Corporation | Automatic Heater Controller TC-324B | to maintain perfusion solution at 32 °C |
| perfusion chamber | home-made, the file for 3D printing is provided in Supplemental Material | ||
| slice holder ("harp") | home made : hairpin made of platinum with the two branches joined by parallel nylon threads | ||
| Name | Company | Catalog Number | Comments |
| for slice stimulation | |||
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A-26209 | to be prepared ex-temporaneously : 1 mg/mL (3 mM) stock solution prepared the day of the experiment, kept at 4 °C (a few hours) and diluted just before use |
| Fluorescein (optional) | Sigma | F-6377 | use at 1 µM final |
| Micromanipulator | Luigs and Neumann | SM7 | connected to the micropipette holde |
| Micropipette holder | same as for eletrophysiology | ||
| Serotonin hydrochloride | Sigma | H-9523 | aliquots of 50mM stock solution in H20 kept at -20 °C. 500 µM solution prepared the day of the experiment. |
| Syringe 5 mL (without needle) | Terumo medical products | SS+05S1 | |
| Transparent tubing | Fischer Scientific | 11750105 | Saint Gobain Performance Plastics™ Tygon™ E-3603 Non-DEHP Tubing |
| Name | Company | Catalog Number | Comments |
| for image analysis | |||
| Fiji | https://fiji.sc | Schindelin, J. et al Nat. Methods (2012) doi 10.1038 | |
| Icy | Institut Pasteur | http://icy.bioimageanalysis.org | de Chaumont, F. et al. Nat. Methods (2012) |
| Name | Company | Catalog Number | Comments |
| mice | |||
| CX3CR1-GFP mice | Jung et al, 2000 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. | |
| CX3CR1creER-YFP mice | Parkhurst et al 2013 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. |
Riferimenti
- Salter, M. W., Stevens, B. Microglia emerge as central players in brain disease. Nature Publishing Group. 23 (9), 1018-1027 (2017).
- Tay, T. L., Savage, J., Hui, C. W., Bisht, K., Tremblay, M. -È Microglia across the lifespan: from origin to function in brain development, plasticity and cognition. The Journal of Physiology. , (2016).
- Davalos, D., et al. ATP mediates rapid microglial response to local brain injury in vivo. Nature Neuroscience. 8 (6), 752-758 (2005).
- Nimmerjahn, A. Resting Microglial Cells Are Highly Dynamic Surveillants of Brain Parenchyma in Vivo. Science. 308 (5726), 1314-1318 (2005).
- Dissing-Olesen, L., et al. Activation of neuronal NMDA receptors triggers transient ATP-mediated microglial process outgrowth. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10511-10527 (2014).
- Gyoneva, S., Traynelis, S. F. Norepinephrine modulates the motility of resting and activated microglia via different adrenergic receptors. Journal of Biological Chemistry. 288 (21), 15291-15302 (2013).
- Eyo, U. B., et al. Neuronal hyperactivity recruits microglial processes via neuronal NMDA receptors and microglial P2Y12 receptors after status epilepticus. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10528-10540 (2014).
- Hristovska, I., Pascual, O. Deciphering Resting Microglial Morphology and Process Motility from a Synaptic Prospect. Frontiers in Integrative Neuroscience. 9, 1231(2016).
- Avignone, E., Lepleux, M., Angibaud, J., Nägerl, U. V. Altered morphological dynamics of activated microglia after induction of status epilepticus. Journal of Neuroinflammation. 12, 202(2015).
- Abiega, O., et al. Neuronal Hyperactivity Disturbs ATP Microgradients, Impairs Microglial Motility, and Reduces Phagocytic Receptor Expression Triggering Apoptosis/Microglial Phagocytosis Uncoupling. PLoS Biology. 14 (5), e1002466(2016).
- Madry, C., et al. Microglial Ramification, Surveillance, and Interleukin-1β Release Are Regulated by the Two-Pore Domain K+Channel THIK-1. Neuron. 97 (2), 299-312 (2018).
- Honda, S., et al. Extracellular ATP or ADP induce chemotaxis of cultured microglia through Gi/o-coupled P2Y receptors. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 21 (6), 1975-1982 (2001).
- Haynes, S. E., et al. The P2Y12 receptor regulates microglial activation by extracellular nucleotides. Nature Neuroscience. 9 (12), 1512-1519 (2006).
- Wu, L. -J., Vadakkan, K. I., Zhuo, M. ATP-induced chemotaxis of microglial processes requires P2Y receptor-activated initiation of outward potassium currents. Glia. 55 (8), 810-821 (2007).
- Kolodziejczak, M., et al. Serotonin Modulates Developmental Microglia via 5-HT 2BReceptors: Potential Implication during Synaptic Refinement of Retinogeniculate Projections. ACS Chemical Neuroscience. 6 (7), 1219-1230 (2015).
- Schafer, D. P., et al. Microglia Sculpt Postnatal Neural Circuits in an Activity and Complement-Dependent Manner. Neuron. 74 (4), 691-705 (2012).
- Pfeiffer, T., Avignone, E., Nägerl, U. V. Induction of hippocampal long-term potentiation increases the morphological dynamics of microglial processes and prolongs their contacts with dendritic spines. Scientific Reports. 6, 32422(2016).
- Parkhurst, C. N., et al. Microglia Promote Learning-Dependent Synapse Formation through Brain-Derived Neurotrophic Factor. Cell. 155 (7), 1596-1609 (2013).
- Wu, Y., Dissing-Olesen, L., Macvicar, B. A., Stevens, B. Microglia: Dynamic Mediators of Synapse Development and Plasticity. Trends in Immunology. 36 (10), 605-613 (2015).
- Ohsawa, K., et al. P2Y12 receptor-mediated integrin-beta1 activation regulates microglial process extension induced by ATP. Glia. 58 (7), 790-801 (2010).
- Kurpius, D., Wilson, N., Fuller, L., Hoffman, A., Dailey, M. E. Early activation, motility, and homing of neonatal microglia to injured neurons does not require protein synthesis. Glia. 54 (1), 58-70 (2006).
- Stence, N., Waite, M., Dailey, M. E. Dynamics of microglial activation: a confocal time-lapse analysis in hippocampal slices. Glia. 33 (3), 256-266 (2001).
- Dissing-Olesen, L., Macvicar, B. A. Fixation and Immunolabeling of Brain Slices: SNAPSHOT Method. Current Protocols in Neuroscience. 71, 1.23.1-12 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Aitken, P. G., et al. Preparative methods for brain slices: a discussion. Journal of Neuroscience Methods. 59 (1), 139-149 (1995).
- Paris, I., et al. ProMoIJ: A new tool for automatic three-dimensional analysis of microglial process motility. Glia. 66 (4), 828-845 (2018).
- Pagani, F., et al. Defective microglial development in the hippocampus of Cx3cr1 deficient mice. Frontiers in Cellular Neuroscience. 9 (229), 111(2015).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods in Molecular Biology. , Clifton, NJ. 221-242 (2014).
- Mainen, Z. F., et al. Two-photon imaging in living brain slices. Methods. 18 (2), San Diego, CA. 231-239 (1999).
- Tanaka, Y., Tanaka, Y., Furuta, T., Yanagawa, Y., Kaneko, T. The effects of cutting solutions on the viability of GABAergic interneurons in cerebral cortical slices of adult mice. Journal of Neuroscience Methods. 171 (1), 118-125 (2008).
- Gyoneva, S., et al. Systemic inflammation regulates microglial responses to tissue damage in vivo. Glia. 62 (8), 1345-1360 (2014).
- Heindl, S., et al. Automated Morphological Analysis of Microglia After Stroke. Frontiers in Cellular Neuroscience. 12, 106(2018).
- Dailey, M. E., Eyo, U., Fuller, L., Hass, J., Kurpius, D. Imaging microglia in brain slices and slice cultures. Cold Spring Harbor Protocols. 12 (12), 1142-1148 (2013).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon