Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Zwei-Photon Imaging der Mikroglia Prozesse Anziehung in Richtung ATP oder Serotonin im akuten Gehirnscheiben
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Mikroglia, die ansässigen Immunzellen des Gehirns, reagieren schnell auf Änderungen ihrer Umgebung mit morphologischen Veränderungen. Dieses Protokoll beschreibt, wie zwei-Photonen-Mikroskopie zu verwenden, um die Anziehungskraft der Mikroglia Prozesse gegenüber Serotonin oder ATP in akuten Hirnschnitten von Mäusen zu studieren.
Zusammenfassung
Mikroglia-Zellen sind resident angeborenen immunen Zellen des Gehirns, die ständig ihre Umgebung mit ihren langen Prozessen absuchen und bei Störung der Homöostase, schnelle morphologische Veränderungen durchlaufen. Zum Beispiel induziert eine Laser-Läsion in wenigen Minuten eine ausgerichtete Wachstum von Mikroglia Prozesse, auch genannt "direktionale Motilität", in Richtung der Stelle der Verletzung. Eine ähnliche Wirkung kann erzielt werden, durch die Bereitstellung lokal ATP oder Serotonin (5-Hydroxytryptamine [5-HT]). In diesem Artikel beschreiben wir ein Protokoll eine gerichtete Wachstum der Mikroglia Prozesse in Richtung einer lokalen Anwendung von ATP oder 5-HT in akuten Hirnschnitten von jungen und Erwachsenen Mäusen zu induzieren, um diese Attraktion im Laufe der Zeit durch multiphoton Mikroskopie Bild. Eine einfache Methode zur Quantifizierung mit freien und Open-Source Bildanalyse-Software wird vorgeschlagen. Eine Herausforderung, die nach wie vor akuten Gehirnscheiben charakterisiert ist die begrenzte Zeit, verringern mit dem Alter, in dem die Zellen in einem physiologischen Zustand bleiben. Dieses Protokoll ist somit Höhepunkte einige technischen Verbesserungen (Mittel, Luft-Flüssigkeit Schnittstelle Kammer, Kammer mit einer doppelten Perfusion imaging) zur Optimierung der Lebensfähigkeit der Mikroglia-Zellen über mehrere Stunden, vor allem in Scheiben von Erwachsenen Mäusen.
Einleitung
Mikroglia-Zellen sind resident Makrophagen des Gehirns und eine Rolle in beiden physiologischen und pathologischen Bedingungen1,2. Sie sind haben eine stark verzweigte Morphologie und ständig erweitern und ihre Prozesse3,4einfahren. Dieses "Scan" Verhalten wird geglaubt, um Verwandte und notwendig, um den Überblick über ihre Umgebung werden. Die morphologische Plastizität der Mikroglia drückt sich in drei Modi. Zunächst einige Verbindungen schnell modulieren Mikroglia Morphologie: die Zugabe von ATP5,6 oder NMDA5,7 mittelfristig akute Gehirnscheiben Baden erhöht die Komplexität der Mikroglia Verzweigungen während Noradrenalin es6 sinkt. Diese Effekte sind direkt vermittelt durch Mikroglia-Rezeptoren (für ATP und Noradrenalin) oder erfordern eine ATP-Freisetzung von Neuronen (NMDA). Zweitens kann die Wachstum und Retraktion Geschwindigkeit der Mikroglia Prozesse, Motilität oder "Surveillance", genannt extrazellulären Faktoren8, Homöostase Störungen9,10oder Mutationen9betroffen sein, 10,11. Drittens: Neben diesen isotropen Änderungen der Morphologie und Motilität Mikroglia haben die Fähigkeit, ihre Prozesse gerichtet in Richtung einer Pipette liefert ATP3,5,12, zu verlängern 13 , 14, in der Kultur, in akuten Gehirnscheiben oder in vivo, oder liefern 5-HT in akuten Gehirn Scheiben15. Solches orientierte Wachstum Mikroglia Prozesse, auch genannt gerichtete Motilität, wurde als Reaktion auf eine lokale Laser Läsion3,4erstbeschrieben. Also physiologisch, es kann im Zusammenhang mit der Reaktion auf eine Verletzung oder erforderlich für targeting Mikroglia Prozesse in Richtung Synapsen oder Hirnregionen erfordern Rebschnitt während Entwicklung15,16, oder im physiologischen17 ,18,19 oder pathologischen Situationen9,18,19,20 im Erwachsenenalter. Die drei Arten von morphologischen Veränderungen setzen auf verschiedene intrazelluläre Mechanismen11,13,20, und einer bestimmte Verbindung nicht unbedingt modulieren alle von ihnen (z. B. NMDA, die indirekt auf wirkt Mikroglia, wirkt auf die Morphologie aber nicht dazu veranlassen, direktionale Motilität5,7). Mit dem Ziel, die Wirkung des eine Verbindung, eine Mutation oder eine Pathologie auf Mikroglia charakterisieren, ist es daher wichtig, die drei Komponenten ihre morphologische Plastizität zu charakterisieren. Hier beschreiben wir eine Methode zur Untersuchung der gerichteten Wachstums der Mikroglia Prozesse in Richtung einer lokalen Quelle Verbindung, was hier ist, ATP oder 5-HT.
Es gibt mehrere Modelle, Mikroglia Prozesse Attraktion zu studieren: Primärkulturen 3D-Umgebung6,18,19, akute Gehirn Scheiben6,13,15, und in Vivo Imaging-3,13. Der in-vivo-Ansatz ist die beste, die physiologische Zustand der Mikroglia zu bewahren. Allerdings intravitalen Bildgebung des tiefen Regionen erfordert komplexe chirurgische Eingriffe und daher ist es oft auf oberflächliche kortikalen Schichten beschränkt. Die Verwendung von Mikroglia Primärkultur ist die einfachste Technik, um eine große Anzahl von Bedingungen mit einer begrenzten Anzahl von Tieren zu testen. Dennoch ist es unmöglich, die gleiche Zelle Morphologie wie in Vivo zu erhalten, und Zellen verlieren ihre physiologischen Wechselwirkungen mit Neuronen und Astrozyten. Akute Gehirnscheiben sind ein Kompromiss zwischen diesen beiden Ansätzen. Dieses Modell erlaubt Forschern, Hirnstrukturen zu studieren, die sonst schwer zu erreichen und zu Bild mit hoher Auflösung in-vivo und Scheiben von Neugeborenen Stadien zu untersuchen sind, während die transkranielle Mikroskopie meistens im Erwachsenenalter durchgeführt wird. Schließlich macht es möglich, die Auswirkungen von lokalen Zulassungsantrag in Echtzeit zu beobachten, und versuche zu wiederholen, wobei eine begrenzte Anzahl von Tieren. Dennoch ist ein Problem mit akuten Gehirnscheiben der begrenzten Zeit (einige Stunden), während die Zellen am Leben, insbesondere für Scheiben von Mäusen, die älter als zwei Wochen, und die mögliche Änderung der Mikroglia Morphologie über Zeit21,22 bleiben .
Hier beschreiben wir ein Protokoll zur akuten Hirnschnitten von jungen und Erwachsenen Cx3cr1 bereitenGLP / + Mäuse bis zu zwei Monate alt, mit der Erhaltung der Mikroglia Morphologie und Motilität für mehrere Stunden. Wir beschreiben, wie diese Scheiben zu verwenden, um die Anziehungskraft der Mikroglia Prozesse in Richtung Verbindungen wie ATP oder 5-HT zu studieren.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Experimente wurden durch die lokale Ethikkommission genehmigt (Darwin Ausschuss, Vereinbarungen #1170 und #10921).
1. Vorbereitung von Glas Mikropipetten für die lokale Anwendung von Verbindungen
- Bereiten Sie Pipetten aus Borosilikat dünnwandige Glaskapillaren mit einer Elektrode Abzieher. Passen Sie die Parameter um Pipetten mit 4-5 µm Durchmesser an der Extremität zu erhalten. Abb. 2D zeigt eine Pipette im Hellfeld bei kleiner Vergrößerung.
(2) Lösungen
- Stellen Sie sicher, dass nur Gläser, die durch ein Autoklav Zyklus gereinigt wurde, gefolgt von Spülung 2 X - 3 X mit Reinstwasser, verwendet werden. Nie verwenden Glaswaren, die Kontakt mit Paraformaldehyd stattgefunden hat.
-
Bereiten Sie eine 2 mol· L-1 CaCl2 Stammlösung durch Auflösen von 14,7 mg CaCl2·2H2O in 50 mL Wasser von hoher Reinheit (Reinstwasser, Widerstand 18.2 MΩ; die Spuren des Metalls in destilliertem Wasser oder Leitungswasser können führen zu suboptimalen Slice Qualität aufgrund Pro-oxidative Wirkung).
- Speichern Sie diese Stammlösung bei Raumtemperatur für maximal einen Monat.
-
Am Tag des Experiments bereiten Sie 1 L Cholin-ACFS (künstliche Liquor cerebrospinalis) Lösung, deren Zusammensetzung 110 Mmol· ist L-1 Cholin Cl, 25 mmol· L-1 Glukose, 25 mmol· L-1 Nahco33, 7 mmol· L-1 MgCl2, 11,6 mmol· L-1 Ascorbinsäure, 3.1 mmol· L-1 Natrium Pyruvat, 2,5 mmol· L-1 KCl, 1,25 mmol· L-1 NaH2PO4und 0,5 mmol· L-1 CaCl2, 0,5.
- Zur Vorbereitung dieser Lösung hinzufügen, in der folgenden Reihenfolge in eine 1 L Schloss Kolben: 0,186 g KCl, 0,195 g NaH2PO42,04 g der Säure Ascorbinsäure, 2,1 g Nahco33und 4,5 g Glukose.
- Füllen Sie ca. zur Hälfte das Endvolumen mit Reinstwasser und rühren Sie bis zur vollständigen Auflösung.
- 0,34 g Natrium Pyruvat und 15,36 g Cholin Cl zugeben.
Hinweis: Es ist bequem, erste auflösen Cholin Cl mit 5 bis 10 mL der Lösung in 2.3.2 Schritt vorbereitet, bevor Sie die gesamte Lösung hinzufügen. - Fügen Sie 1 Mol· 7 mL L-1 MgCl2 und 250 µL 2 mol· L-1 CaCl2 (in Schritt 2.2 zubereitet) und die Lösung.
- Füllen Sie den Messkolben bis zu 1 L mit Reinstwasser.
- Überprüfen Sie mit einem Dampfdruck XR, dass die Osmolarität zwischen 300 und 310 mΩ. Wenn dies nicht der Fall ist, passen Sie es mit Glucose.
- Den pH-Wert nach Carbogenation (d. h. mit "Carbogen" sprudelt, eine Mischung aus 95 % O25 % CO2) zu überprüfen und gegebenenfalls zu 7,3-7,4 mit 10 M NaOH einstellen.
- Übertragen Sie die Lösung in eine Glasflasche zur Aufbewahrung. Halten Sie die Flasche in den Kühlschrank bis zur Verwendung (Schritt 3.1).
Hinweis: Es wird empfohlen, eine frische Lösung am Tag des Experiments zu machen. Allerdings kann bei Bedarf Cholin-ACFS zwei Tage bei 4 ° c gelagert werden
-
Am Tag des Experiments bereiten Sie 1 L einer ACFS Lösung, deren Zusammensetzung 124 Mmol· ist L-1 NaCl, 26,2 mmol· L-1 Nahco33, 25 mmol· L-1 Glukose, 2,5 mmol· L-1 KCl, 2 mmol· L-1 CaCl2, 1 mmol· L-1 MgCl2und 1,25 mmol· L-1 NaH2PO4.
- Zur Vorbereitung dieser Lösung hinzufügen, in der folgenden Reihenfolge zu einem Messkolben: 0,150 g NaH2PO4, 0,186 g KCl, 2,2 g Nahco33, 4,5 g Glukose und 7,3 g NaCl. Die Lösung auf ein Volumen von 1 L mit Reinstwasser zu bringen und es auf einem Teller rühren kräftig rühren.
- Fügen Sie 1 mL 1 mol· L-1 MgCl2 und 1 mL 2 mol· L-1 CaCl2 zur Lösung und zum Transfer der ACFS Lösung um eine Glasflasche zur Aufbewahrung.
- Überprüfen Sie, ob die Osmolarität 300-310 mΩ· L-1 und wenn nicht, stellen Sie es mit Glucose.
- Überprüfen Sie den pH-Wert nach Carbogenation (d. h. mit "Carbogen" blubbern) und gegebenenfalls auf 7,3-7,4 mit 10 M NaOH einstellen.
- Übertragen Sie die Lösung in eine Glasflasche zur Aufbewahrung. Halten Sie die Flasche in den Kühlschrank bis zur Verwendung (Schritt 3.1).
Hinweis: Es wird empfohlen, eine frische Lösung am Tag des Experiments zu machen. Jedoch ist eine Alternative zur Vorbereitung eine 10-fach-Stammlösung mit NaCl, Nahco33KCl und NaH2PO4 10 x die Endkonzentration, die nicht mehr als eine Woche bei 4 ° c aufbewahrt werden können Machen Sie die endgültige ACFS am Tag des Experiments durch Verdünnen der 10-fach-Stammlösung mit Reinstwasser und der Glukose, CaCl2und MgCl2hinzufügen.
- Bereiten Sie die Droge Lösungen am Tag des Experiments. Verwenden Sie die ACFS Lösung um zu bringen, die Endkonzentrationen sind hier 500 µmol· L-1 für ATP und 5 µmol· L-1 für 5-HT.
Hinweis: Für ATP, eine Stammlösung hergestellt werden kann (z. B. 50 mM ATP im Wasser), in regelmÄÑig Form bei-20 ° C gelagert und verdünnt mit ACFS auf die Endkonzentration am Tag des Experiments. Im Gegensatz dazu muss die 5-HT (Serotonin-HCl) Lösung werden aus Pulver am Tag des Experiments, bei 1 Mg·mL-1 im Wasser vorbereitet, gehalten bei 4 ° C, 5-HT-Oxidation zu vermeiden und verdünnt in ACFS zum Zeitpunkt des Experiments.
3. Vorbereitung des akuten Gehirnscheiben
-
Vorbereitung des Bereichs Dissektion
- Bereiten Sie 70 mL eiskaltes Cholin-ACFS in einer 80-mL-Becherglas auf Eis für kardiale Perfusion, schnelle Abkühlung des Gehirns und schneiden zu verwendende gelegt. Bereiten Sie 150 mL Cholin-ACFS in ein 200 mL kristallisieren Gericht, platziert in einem beheizten Wasserbad auf 32 ° c gehalten Legen Sie ein Nylon-Sieb in der kristallisierenden Teller, die Scheiben zu behalten. Hiermit werden die Scheiben für 10 min gerade schneiden erholen zu lassen.
- Mindestens 30 min vor Beginn der Dissektion (Abschnitt 3.2), beginnen diese zwei Lösungen (70 mL Cholin-ACFS auf Eis) und 150 mL von Cholin-ACFS bei 32 ° C mit Carbogen sprudeln. Konstante Carbogenation während des gesamten Verfahrens zu erhalten.
- Bereiten Sie die Kammer Schnittstellengerät (Abbildung 1), die verwendet wird, um die Scheiben zu halten, bis ihre Verwendung.
- In einem versiegelten Lebensmittel-Box (10 x 10 cm oder 10 cm im Durchmesser, Höhe 8 cm), installiert auf einem Magnetrührer, legen Sie eine 200 mL kristallisieren Gericht mit einer Bar Magnet.
- Fügen Sie 200 mL ACFS in diesem kristallisierenden Gericht und 3D-gedruckten Schnittstelle Slice Platzhalter oben drauf (die Schnittstelle Stück Halter besteht aus zwei perfekt passende Teile, mit Polyamid Gewebe erstreckte sich zwischen ihnen, Abb. 1A, B).
- Entfernen Sie überschüssige Menge aus der kristallisierenden Schale, nur eine dünne Schicht der Lösung, die das Netz von der Schnittstelle Stück Halter zu halten. Dadurch entsteht später eine feine Felge Lösung rund um die Scheiben (aber ohne abdecken).
- Legen Sie ein paar Millimeter der ACFS am unteren Rand der Lebensmittel-Box und starten Sie sprudelt es mit Carbogen (beim ersten Gebrauch machen ein kleines Loch in der verschlossenen Lebensmitteln Box Wand um sicherzustellen, dass die Schläuche kann Feld eingeben).
- Schließen Sie das versiegelte und gleichzeitig konstante Carbogenation. Dadurch wird eine befeuchtete 95 % O25 % CO2 reichhaltige Umgebung erstellt, in der die Scheiben nach ihrer Genesung in Cholin-ACFS übertragen und gepflegt, bevor sie abgebildet sind. Dieses Gerät ist nachfolgend als die "Schnittstelle Kammer" (Abbildung 1).
-
Gehirn sezieren und schneiden
- Die Maus mit einer intraperitonealen Injektion von 50 Mg·mL-1 Pentobarbital (0,15 mL/20 g Maus Körpergewicht) zu betäuben, es zu immobilisieren aussetzen das Herz und führen eine kardiale Perfusion mit 10 mL der eiskalte, Carbogenated, Cholin-ACFS (siehe Schritt 3.1.1), mit einer peristaltischen Pumpe. Beobachten Sie die Blässe der Leber als Indikator für eine gute Durchblutung. Die Perfusion dauert weniger als 5 Minuten.
- Enthaupten Sie die Maus zu und schneiden Sie die Haut um den Schädel zu entlarven. Gelten Sie mit großen Scheren zwei transversale Schnitten von großen Foramen und einem langen sagittaler Schnitt und mit feinen Pinzette, die Schädel-Platten zu entfernen.
- Schnell und schonend extrahieren Sie des Gehirns (in weniger als 1 min) und legen Sie sie für 1 min in den 80-mL-Becherglas mit den verbleibenden (~ 60 mL) eiskalte Cholin-ACFS (noch unter konstanten Carbogenation), um es abzukühlen.
- Übertragen Sie das Gehirn auf ein Filterpapier vorher nass mit ACFS.
- Schneiden Sie das Gehirn nach der Region des Gehirns von Interesse und bevorzugten Winkel des Schneidens. Zum Beispiel um den Thalamus oder der Hippocampus auf koronalen Scheiben image, schneiden Sie mit einer Skalpellklinge das Kleinhirn und dann, etwa 2 mm von der Höhlung und kaudalen Extremitäten des Gehirns.
Hinweis: Es ist wichtig, Gehirn Teile entfernen, die auch rostral oder auch kaudalen sind denn je kleiner der Region zu trimmen, bevor man den Bereich von Interesse, desto schneller schneiden. Gesamtzeit für slicing (Schritt 3.2.7) von weniger als 20 Minuten wird empfohlen. - Positionieren Sie für koronalen Scheiben und kleben Sie (mit Cyanacrylat-Klebstoff) der kaudalen Gesichts des Gehirns auf einer 10 cm Petrischale, des Schneidblockes eines vibrierenden Slicers aufgeklebt und positionieren Sie es in die Vorratskammer des vibrierenden Slicers, die in einer größeren Kammer angeordnet ist mit Eis gefüllt. Füllen Sie die Petrischale mit all den restlichen eiskalte Cholin-ACFS.
- Während 95 % O25 % CO2 konstant zu halten von den eiskalten Cholin-ACFS, sprudeln 300 µm dicken koronale Scheiben schneiden (Geschwindigkeit: 0,08 Mm·s-1, Klinge Vibration: 60 Hz, Schwingungsamplitude: 1 mm).
- Sammeln pipette die Gehirnscheiben mit einem breit-Mund (4 mm Durchmesser) Einweg-Transfer, eins nach dem anderen nach jedem Durchgang der Klinge, um die Ansammlung von toxischen Bestandteilen, veröffentlicht von der Peripherie der Scheiben zu vermeiden. Achten Sie darauf, um Luftblasen zu vermeiden, während der Übertragung und jede Scheibe in die Cholin-ACFS bei 32 ° C für ca. 10 min für die Wiederherstellung.
- Mit der Transferpipette, legen Sie die Scheiben auf Objektiv Reinigung Papier gekrönt mit einem Rückgang von Cholin-ACFS. Das Übermaß an Cholin-ACFS Aspirieren und mit dem Spatel, legen Sie die Scheiben, auf das Objektiv Reinigung Gewebe auf dem Netz der Schnittstelle Kammer mit Carbogenated ACFS bei Raumtemperatur gelegt (siehe 3.1.3.5). Lassen Sie die Scheibe in diesem Umfeld für mindestens 30 Minuten zu erholen.
Hinweis: Danach die Scheiben sind bereit und können für Mikroglia imaging für bis zu 6 h nach der Gehirn-Extraktion von jung (weniger als ein Monat alt) Mäuse und bis zu 4 h nach der Gehirn-Extraktion von zwei Monate alten Erwachsenen verwendet werden.
4. zwei-Photonen-Mikroskopie
- Parameter-Einstellung
- Das multiphoton System (Hybrid-Detektoren, Laser, Scanner, elektrooptischen Modulator, Mikroskop) einschalten.
- Optimieren den Laser bei 920 nm, überprüfen Sie, ob der Laser modengekoppelten ist, und legen Sie die Macht in 5-15 % und der Gewinn von 10 %. Dies entspricht einer Leistung von 3 bis 5 mW im Rahmen des Ziels. Sicherstellen Sie, dass die nondescanned Detektoren tätig sind und die entsprechenden Emissions- und Erregung Filter installiert.
- Parameter der imaging-Software auf die folgenden Werte festlegen: für die Frame-Größe 1024 x 1024 Pixel entspricht einer Fläche von 295.07 x 295.07 µm; für den Zoom, 2. Wenn das Signal sehr laut ist, gelten Sie einen Linie-Durchschnitt von 2. Setzen Sie für die Pixel-Dynamik die Software zur Abbilderstellung bei 12 Bit oder mehr.
Hinweis: Bilder mit einer höheren Bit-Wert erlauben Forschern, kleinere Unterschiede in der Fluoreszenzintensität als Bilder mit einer geringeren Bit-Wert zu unterscheiden: eine Änderung von einem Grauwert in einem 8-Bit-Bild einem Wechsel von 16 Grauwerte in einem 12-Bit und 256 grau entspräche Werte i n eine 16-Bit-Bild. Daher höhere Bit-Bilder sind besser geeignet für Quantitative Analyse, aber da ihre Größe mit Bit-Tiefe, Speicherkapazität und computing erhöht werden macht begrenzen. - Wählen Sie den Scanmodus XYZT mit einem Z-Intervall auf 2 µm und einem T-Intervall von 2 min.
Hinweis: Die x-, y- und Z-Auflösung richten sich nach dem Nyquist-Abtasttheorem. Eine Z-Schrittweite um 0,8 wäre optimal, Mikroglia Prozesse zu lösen (mit einem Durchmesser von < 1 µm), aber die optische Auflösung des multiphoton Mikroskopie begrenzt (bei 920 nm mit 0,95 NA Zielsetzung, die axiale Auflösung ist um 1 µm). Hinzu kommt, dass physikalische Barriere, in ein live-Imaging-Experiment, die Empfindlichkeit oder Signal-Rausch-Verhältnis, die Auflösung, die Geschwindigkeit und die gesamte Beobachtung Zeit Angelegenheit. Unter Berücksichtigung all diese Parameter, Z-Schritt 2 µm (wie in zahlreichen Studien3,11,14), einer Bildgröße von 1024 x 1024 Pixel und einer High-Speed-Übernahme mit einem Resonanz-Scanner gekoppelt an HyD Detektoren (es dauert ca. 15 s 50 Z-Pläne zu erwerben) wurden hier ausgewählt. Die Häufigkeit der Akquisitionen ist eine XYZT Serie alle 2 min und die Gesamtdauer beträgt 30 Minuten. Wenn das Setup nicht schnell oder empfindlich genug ist, ist es möglich, die laterale Auflösung (bis zu 512 x 512) oder die Anzahl der Z-Scheiben zu reduzieren (durch Bildgebung ausschließlich in die Z-Tiefe, die die stärkste Fluoreszenz aufweist [d. h. nicht die tiefste Z-Scheiben wo Fluoreszenz ist schwach]), oder um die Geschwindigkeit des Scanners zu verringern. Die axiale Auflösung kann auch durch eine Erhöhung des Z-Schritt bis zu 3 µm verringert werden, aber da dies die Quantifizierung auswirken kann, sollten alle Experimente verglichen werden mit der gleichen Z-Schritt durchgeführt werden.
Hinweis: Es ist möglich, ähnliche Experimente auf Scheiben aus CX3CR1creER-YFP Mäuse18, eine Mauslinie verwendet, um genetische Deletion im Mikroglia nur zu induzieren, und in welche Mikroglia konstitutiv express gelb fluoreszierenden Proteins (YFP) durchführen. Die Expression von YFP ist jedoch sehr gering im Vergleich zu grün fluoreszierendes Protein (GFP) in CX3CR1GLP / + Mäuse; so Bildgebung ist möglich, aber anspruchsvoll und erfordert die Optimierung der Aufnahmeparameter. Es wird empfohlen, diese wie folgt anzupassen. - Optimieren den Laser bei 970 nm (welche ist besser angepasst an YFP Erregung als 920 nm), die Macht bei 50 % und der Gewinn bei 50 %, das entspricht einer Laserleistung unter der Zielsetzung von 5 bis 6 mW.
- Legen Sie einen Durchschnitt von Linie 4 (oder mehr), das Signal-Rausch-Verhältnis zu verbessern.
- Positionierung des Segments und das Glas Mikropipette und die lokale Anwendung der Verbindung
- Anschließen Sie die peristaltische Pumpe zur Aufnahme Kammer, 30 min vor Beginn der Aufzeichnung. Beginnen Sie nach der Reinigung des ganze Perfusion Systems mit 50 mL Reinstwasser die Perfusion der Aufnahme Kammer mit ACFS (50 mL) in ein Becherglas unter ständiger Carbogenation enthalten. Während das Experiment halten Sie die zirkulierenden ACFS auf 32 ° C mit einer Inline-Microheater oder eine Peltier Heizung.
Hinweis: Eine spezifische Perfusion Kammer mit oberen und unteren Perfusion soll die Sauerstoffversorgung auf beiden Seiten der Scheibe zu optimieren. Die Perfusion Kammer besteht aus zwei perfekt passende Teile, mit Polyamid-Gewebe erstreckte sich zwischen ihnen (Abbildung 2A, B). Im Vergleich mit anderen Arten von Kammern, wo die Scheibe direkt auf einem Glas Deckgläschen Verlegung ist, dieser Kammer neuronalen Tod im Unterteil der Scheibe reduziert, verbessert die Rentabilität und die Slice-Bewegungen induziert durch die Schwellung reduziert. - Übertragen Sie mit einer Transferpipette breit-Mund Einweg-die Gehirn-Scheibe abgebildet werden, um das ACFS Becherglas, entfernen das Objektiv Papier, lassen es sinken (als Nachweis, dass keine Luftblase angeschlossen ist), und übertragen Sie es auf der Aufnahme (Perfusion) Kammer.
- Positionieren Sie einen Slice-Halter (eine Haarnadel von Platin mit den beiden Zweigen verbunden durch parallele Nylonfäden gemacht) auf die Scheibe um Scheibe Bewegung aufgrund der Perfusion zu minimieren.
- Verwenden Sie die Hellfeld Beleuchtung auf die Region des Gehirns von Interesse (Belichtungszeit: 50 bis 80 ms) mit einer niedrigen Vergrößerung Ziel (5 X oder 10 X). Wechseln Sie zu der höheren Vergrößerung (25 X mit einem 0,35 X Objektiv) Wasser eintauchen Objektiv und passen Sie die Position.
Hinweis: Vermeiden Sie zu Bildfelder in der Nähe der Scheibe Inhaber Nylonfäden lokal verformen die Scheibe und das Licht zu blockieren können. Stellen Sie sicher, dass das Interessengebiet flach ist. Entfernen Sie ggf. die Scheibe Inhaber um das Segment zu positionieren und/oder die Slice-Inhaber. - Die Fluoreszenz-Beleuchtung verwenden, um fluoreszierende Microglial Zellen im Bereich abgebildet finden (Belichtungszeit: 250-500 ms).
Hinweis: Dieser Schritt erlaubt Forschern, die Anwesenheit von Zellen in der Region von Interesse und deren Fluoreszenzintensität zu prüfen und für die Höhe der Zelltrümmer steuern. - Die Pipette mit 10 µL ACFS Hinterfüllung mit ATP, 5-HT oder das Medikament auf die Endkonzentration von Interesse. Zeigen Sie die Spitze nach unten und schütteln Sie die Droge-gefüllte Pipette entfernen Sie eventuell vorhandene Luftbläschen in der Spitze gefangen.
Hinweis: Wenn die Lösung injiziert werden tendenziell Bläschen bilden, Borosilikat Pipetten mit einem internen Filament in Betracht. Austritt von ATP aus der Pipette kann Mikroglia Prozesse schon vor der Injektion zu gewinnen (in diesem Fall wird es an den Analyseschritt sichtbar sein). Obwohl dies sollte moderat mit der ATP-Konzentration verwendet (500 µmol· L-1), wenn es ein Problem ist, betrachten die Mikropipette mit 2 mL ACFS vor dem Hinzufügen der ATP (oder andere Verbindung) prefilling Lösung bei Schritt 4.2.6. - Montieren Sie die gefüllte Pipette in eine Pipette Halter, verbunden mit transparenten Schlauch an einer 5 mL Spritze mit einem Kolben an der 5 mL-Position positioniert. Die Pipette Halterung selbst ist auf einem dreiachsigen Mikromanipulator montiert.
- Verwenden Sie unter Hellfeld Beleuchtung die Mikromanipulator, um die Pipette in der Mitte des Feldes zu positionieren. Zeigen Sie für eine reproduzierbare und optimale Zentrierung an und verwenden Sie der Herrscher auf dem Bild.
- Senken Sie die Pipette vorsichtig in Richtung der Scheibe, Steuerung und Anpassung das Ziel zur gleichen Zeit, bis die Pipettenspitze leicht berührt die Oberfläche der Scheibe. Den Abstieg der Pipette zu stoppen, sobald es sichtbar ist, dass die Scheibe berührt wurde die Pipettenspitze, 80-100 µm von der Oberfläche des Segments zu durchdringen kann (siehe Abb. 3 b).
- Einstellen des Lasers (siehe die oben genannten Parameter) und das Mikroskop in den multiphoton-Modus zu wechseln. Stellen Sie sicher, dass die Kammer von jeder Lichtquelle (z. B. einem Computer-Bildschirm) abgeschirmt ist. Einschalten der nondescanned Detektoren und des Gewinns. Verwenden Sie eine Lookup-Tabelle (LUT) mit einer farbcodierten Obergrenze um zu vermeiden, die Pixel im Bild zu sättigen.
- Bestimmen Sie die Dicke der Scheibe zu abgebildet werden (d. h. den oberen und unteren Z-Positionen, wo Fluoreszenz nachweisbar [in der Regel zwischen 220 und 290 µm insgesamt]).
Hinweis: An der Oberfläche der Scheibe ist eine erhöhte Dichte von Prozessen und möglicherweise der Mikroglia, oft mit einer ungewöhnlichen Morphologie, im Vergleich mit der Innenseite der Scheibe. Diese Ansammlung wird deutlicher, mit der Zeit (dh., sichtbarer in den letzten als in der ersten Gehirn Slice abgebildet werden). Daher Z-Flugzeuge in den ersten ~ 30 µm sollten nicht für die Analyse verwendet werden und können auch für den Erwerb übersprungen werden. - Starten Sie die Aufnahme einer Gesamtdauer von 30 min (oder mehr wenn gewünscht) und nach 5 min Basislinie vor Ort gelten Sie die Verbindung getestet werden (ohne Unterbrechung der Bildgebung). Um dies zu tun, drücken Sie langsam den Kolben der Spritze angeschlossen an die Mikropipette von 5 mL auf 1 mL (ca. 5 s). Widerstand, wenn den Kolben drücken muss sofort spürbar. Wenn dies nicht der Fall ist, die Spitze gebrochen werden könnte.
Hinweis: Für einen ausgebildeten Experimentator die Injektionen mit dieser Methode sind reproduzierbar, aber alternativ auf die manuelle Manipulation einer Spritze, die Pipette auf eine automatisierte Druck-Auswurf-System ermöglicht eine bessere Kontrolle über die Lautstärke geliefert verknüpft werden könnte. Die Injektion schafft eine körperliche Verzerrung des Segments an der Injektionsstelle. Diese Verzerrung ist sichtbar Nachhinein in den ersten zwei oder drei Bilder nach der Injektion sollte jedoch nicht auf dem vierten Bild (d. h. 8 min nach der Injektion) sichtbar. Wenn es anhält, in Erwägung die Parameter für die Vorbereitung der Pipette. - Am Ende des Erwerbs (30 min) verwerfen Sie die Mikropipette und entfernen Sie der Scheibe zu. Falls gewünscht, beheben Sie die Scheibe für weitere Immunolabeling. Zum Beispiel die SNAPSHOT-Methode ist optimiert für die Fixierung und Färbung der dicke Scheiben23.
- Vor dem Start zum Bild eines neuen Slices, 2D Film (Abschnitt 5.1) zu machen, um zu überprüfen, dass Mikroglia haben eine normale Morphologie und bewegen und damit, dass die Scheiben gesund sind.
- Anschließen Sie die peristaltische Pumpe zur Aufnahme Kammer, 30 min vor Beginn der Aufzeichnung. Beginnen Sie nach der Reinigung des ganze Perfusion Systems mit 50 mL Reinstwasser die Perfusion der Aufnahme Kammer mit ACFS (50 mL) in ein Becherglas unter ständiger Carbogenation enthalten. Während das Experiment halten Sie die zirkulierenden ACFS auf 32 ° C mit einer Inline-Microheater oder eine Peltier Heizung.
5. Analyse der Attraktion der Mikroglia Prozesse
-
2D Projektion und Drift-Korrektur
- Öffnen Sie die Datei (. LIF) mit Fidschi24.
- Wenn nötig, machen Sie eine Substack (Bild/Stapel/Tools/Make Substack) nur die Z-Flugzeuge von Interesse. Zum Beispiel ausschließen, die Z-Ebenen entsprechend an die Oberfläche der Scheibe, wenn sie erworben wurden, aber nicht für die Analyse verwendet werden sollen (siehe Hinweis nach Schritt 4.2.11) und den tiefsten Z-Ebenen mit keine Fluoreszenz. Der letzte Stapel enthält in der Regel 90-110 Z-Scheiben (180-220 µm).
- Starten Sie die Funktion Z Projekt (Bild/Stapel/Z Projekt") und wählen Sie den Projektionstyp Max Intensität zu den Projektionen des Z-Stack zu jedem Zeitpunkt erworben.
- Starten Sie das MultiStackReg -Plugin (Plugin/Registrierung/MultiStackReg), Auswahl Aktion 1: richten Sie und Transformation: Rigid Body leichte Abweichungen zu korrigieren, die beim Erwerb stattgefunden haben kann. Speichern Sie diese 2D Film als eine neue Datei (.TIF).
-
Datenverarbeitung
- Öffnen Sie diese neue Datei mit eisigen25.
- Zeichnen einer kreisförmigen R1 -Region of Interest (ROI) von 35 µm im Durchmesser, zentriert auf die Injektionsstelle (gekennzeichnet vor allem durch den Schatten von der Pipette und die Verzerrung erstellt zum Zeitpunkt der Injektion).
- Verwenden Sie das Plugin ROI Intensität Evolution und Messen Sie die mittlere Intensität im Laufe der Zeit in R1.
- Speichern Sie die Ergebnisse ein. XLS-Datei.
-
Quantifizierung und Darstellung der Ergebnisse
- Zur Quantifizierung der Mikroglia Antwort im Laufe der Zeit zu bestimmen, zu jedem Zeitpunkt
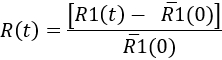
Here,'R1(0) ist der Mittelwert der R1(t) Werte vor der Injektion. Dann die Ergebnisse als eine kinetische der Mikroglia Antwort dargestellt werden können, oder zu einem bestimmten Zeitpunkt zeigen (siehe Abbildung 7).
- Zur Quantifizierung der Mikroglia Antwort im Laufe der Zeit zu bestimmen, zu jedem Zeitpunkt
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Dieses Protokoll beschreibt eine Methode zum auslösen, zu beobachten und zu quantifizieren das orientierte Wachstum der Mikroglia Prozesse in Richtung einer lokal applizierten Verbindung, z. B. ATP oder 5-HT, in akuten Gehirn Scheiben von Jugendlichen oder Erwachsenen (mindestens bis zu zwei Monate alten) Mäuse. Zu den Faktoren, die zur Aufrechterhaltung Hirnschnitten von erwachsenen Tieren in einem gesunden Zustand für mehrere Stunden ist die Verwendung von zwei Tools zur Optimierung ...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Durch die Beibehaltung, anders als in dissoziiert oder organotypischen schneiden Kultur, eine strukturelle Integrität mit begrenzten Anpassungen, akute Gehirnscheiben erlauben Forschern, Mikroglia in ihrer physiologischen Umgebung zu studieren. Eine wichtige Einschränkung ist jedoch die Tatsache, dass die Slice Prozedur Verletzungen erstellt, die schnell die Lebensfähigkeit von Neuronen, besonders im erwachsenen Gehirn beeinträchtigen können. Mikroglia sind besonders reaktiv zu Zellschäden, ist es wichtig, neuronal...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Wir danken der Zelle und Gewebe Imaging Facility des Institut du Fer À Moulin, wo alle Bildaufnahme und Analysen durchgeführt wurden. Diese Arbeit wurde teilweise unterstützt durch das Centre National De La Recherche Scientifique, Institut National De La Santé et De La Recherche Médicale, Université Sorbonne Wissenschaften, und durch Zuschüsse von Sorbonne Universités-Pierre et Marie Curie-Universität () Emergence-UPMC Programm 2011/2014), die Fondation pour la Recherche Sur le Cerveau, Fondation de France, die Fondation pour la Recherche Médicale "Equipe FRM DEQ2014039529", dem französischen Forschungsministerium (Agence Nationale Pour la Recherche ANR-17-CE16-0008 und des Investissements Avenir Programm "Bio-Psy Labex" ANR-11-IDEX-0004-02) und Kooperationsforschung in Computational Neuroscience Programm, National Science Foundation/Französisch National Agency for Research (Nummer: 1515686). Alles, was die Autoren angehören, um Forschung Gruppen, die Mitglieder der Paris School of Neuroscience (ENP) und der Bio-Psy Labex sind. Z.B. ist ein Ph.d. Student angegliedert Sorbonne Université, Collège Doktoranden, F-75005 Paris, Frankreich, und wird durch die Bio-Psy Labex finanziert. Caracterización ist ein Post-Doktorand geförderten Sonderforschungsbereichs Computational Neuroscience Programm, National Science Foundation/Französisch National Agency for Research (Nummer: 1515686). Die Autoren danken Marta Kolodziejczak, die bei der Initiierung des Projekts teilgenommen.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| for pipettes preparation | |||
| Clark Borosilicate Thin Wall Capillaries | Harvard Apparatus | 30-0065 | Borosilicate Thin Wall without Filament, 1.5 mm OD, 1.17 mm ID, 75 mm L , Pkg. of 225 |
| DMZ Universal Puller | Zeitz Instrumente | ||
| Name | Company | Catalog Number | Comments |
| for solutions | |||
| Calcium Chloride dihydrate (CaCl2) | Sigma | C5080 | |
| Choline Chloride | Sigma | C7527 | |
| D-(+)-Glucose | Sigma | G8270 | |
| L-Ascorbic acid | Sigma | A5960 | |
| Magnesium Chloride solution 1 M (MgCl2) | Sigma | 63020 | |
| Potassium chloride SigmaUltra >99.0% (KCl) | Sigma | P9333 | |
| Sodium bicarbonate (NaHCO3) | Sigma | S5761 | |
| Sodium Chloride (NaCl) | Sigma | S5886 | |
| Sodium phosphate monobasic | Sigma | S5011 | |
| Sodium pyruvate | Sigma | P2256 | |
| Ultrapure water | MilliQ | for all the solutions | |
| Name | Company | Catalog Number | Comments |
| for slice preparation | |||
| 2x 200 mL crystalizing dishes | |||
| 80 mL Pyrex beaker | |||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For mice perfusion and 2-photon chamber perfusion (aCSF) |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | ||
| Dolethal | Vetoquinol | Dolethal 50 mg/mL | |
| Filter papers (Whatman) | Sigma | WHA1001042 | Whatman qualitative filter paper, Grade 1 (Pore size: 11µM) |
| Fine Scissors - Sharp | Fine Science Tools | 14060-60 | |
| Food box 10 cm diameter, 8 cm Height | |||
| glue (ethyl cyanoacrylate) | Loctite | super glue 3 power flex | |
| Hippocampal Tool (spatula) | Fine Science Tools | 10099-15 | The largest extremity has to be angled at 90° |
| Ice | |||
| Iris Forceps (curved) | Moria | MC31 | |
| Lens cleaning tissue | THOR LABS | ||
| Nylon mesh strainer | diameter 7 cm | ||
| Razor blades | Electron Microscopy Sciences | 72000 | For the slicer |
| scalpel blade | |||
| Slice interface holder | home-made, the file for 3D printing is provided in Supplemental Material | ||
| Surgical Scissors - Sharp | Fine Science Tools | 14002-14 | |
| Vibrating slicer | Thermo Scientific | 720-2709 | Model: HM 650V (Vibrating blade microtome) |
| Water bath | Set at 32 °C (first recovery step) | ||
| Name | Company | Catalog Number | Comments |
| for slice imaging | |||
| × 25 0.95 NA water-immersion objective | Leica Microsystems (Germany) | HCX Irapo | |
| 2-photon MP5 upright microscope with resonant scanners (8 kHz) and two HyD Hybrid detectors | Leica Microsystems (Germany) | ||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For 2-photon chamber perfusion with aCSF |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | I1501L50R2A001 | |
| Chameleon Ultra2 Ti:sapphire laser | Coherent (Germany) | ||
| disposable transfer pipettes , wide mouth | ThermoFischer scientific | for example : 232-11 | 5.8 mL with fin tip, but we cut it (approx 7 cm) to have a 4 mm diameter mouth |
| emission filter SP680 | Leica Microsystems (Germany) | ||
| fluorescent cube containing a 525/50 emission filter and a 560 dichroic filter (for fluorescence collection) | Leica Microsystems (Germany) | ||
| glass beaker with 50 mL of ACSF to maintain constant perfusion of the slice | |||
| Heating system | Warner Instrument Corporation | Automatic Heater Controller TC-324B | to maintain perfusion solution at 32 °C |
| perfusion chamber | home-made, the file for 3D printing is provided in Supplemental Material | ||
| slice holder ("harp") | home made : hairpin made of platinum with the two branches joined by parallel nylon threads | ||
| Name | Company | Catalog Number | Comments |
| for slice stimulation | |||
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A-26209 | to be prepared ex-temporaneously : 1 mg/mL (3 mM) stock solution prepared the day of the experiment, kept at 4 °C (a few hours) and diluted just before use |
| Fluorescein (optional) | Sigma | F-6377 | use at 1 µM final |
| Micromanipulator | Luigs and Neumann | SM7 | connected to the micropipette holde |
| Micropipette holder | same as for eletrophysiology | ||
| Serotonin hydrochloride | Sigma | H-9523 | aliquots of 50mM stock solution in H20 kept at -20 °C. 500 µM solution prepared the day of the experiment. |
| Syringe 5 mL (without needle) | Terumo medical products | SS+05S1 | |
| Transparent tubing | Fischer Scientific | 11750105 | Saint Gobain Performance Plastics™ Tygon™ E-3603 Non-DEHP Tubing |
| Name | Company | Catalog Number | Comments |
| for image analysis | |||
| Fiji | https://fiji.sc | Schindelin, J. et al Nat. Methods (2012) doi 10.1038 | |
| Icy | Institut Pasteur | http://icy.bioimageanalysis.org | de Chaumont, F. et al. Nat. Methods (2012) |
| Name | Company | Catalog Number | Comments |
| mice | |||
| CX3CR1-GFP mice | Jung et al, 2000 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. | |
| CX3CR1creER-YFP mice | Parkhurst et al 2013 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. |
Referenzen
- Salter, M. W., Stevens, B. Microglia emerge as central players in brain disease. Nature Publishing Group. 23 (9), 1018-1027 (2017).
- Tay, T. L., Savage, J., Hui, C. W., Bisht, K., Tremblay, M. -È Microglia across the lifespan: from origin to function in brain development, plasticity and cognition. The Journal of Physiology. , (2016).
- Davalos, D., et al. ATP mediates rapid microglial response to local brain injury in vivo. Nature Neuroscience. 8 (6), 752-758 (2005).
- Nimmerjahn, A. Resting Microglial Cells Are Highly Dynamic Surveillants of Brain Parenchyma in Vivo. Science. 308 (5726), 1314-1318 (2005).
- Dissing-Olesen, L., et al. Activation of neuronal NMDA receptors triggers transient ATP-mediated microglial process outgrowth. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10511-10527 (2014).
- Gyoneva, S., Traynelis, S. F. Norepinephrine modulates the motility of resting and activated microglia via different adrenergic receptors. Journal of Biological Chemistry. 288 (21), 15291-15302 (2013).
- Eyo, U. B., et al. Neuronal hyperactivity recruits microglial processes via neuronal NMDA receptors and microglial P2Y12 receptors after status epilepticus. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10528-10540 (2014).
- Hristovska, I., Pascual, O. Deciphering Resting Microglial Morphology and Process Motility from a Synaptic Prospect. Frontiers in Integrative Neuroscience. 9, 1231(2016).
- Avignone, E., Lepleux, M., Angibaud, J., Nägerl, U. V. Altered morphological dynamics of activated microglia after induction of status epilepticus. Journal of Neuroinflammation. 12, 202(2015).
- Abiega, O., et al. Neuronal Hyperactivity Disturbs ATP Microgradients, Impairs Microglial Motility, and Reduces Phagocytic Receptor Expression Triggering Apoptosis/Microglial Phagocytosis Uncoupling. PLoS Biology. 14 (5), e1002466(2016).
- Madry, C., et al. Microglial Ramification, Surveillance, and Interleukin-1β Release Are Regulated by the Two-Pore Domain K+Channel THIK-1. Neuron. 97 (2), 299-312 (2018).
- Honda, S., et al. Extracellular ATP or ADP induce chemotaxis of cultured microglia through Gi/o-coupled P2Y receptors. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 21 (6), 1975-1982 (2001).
- Haynes, S. E., et al. The P2Y12 receptor regulates microglial activation by extracellular nucleotides. Nature Neuroscience. 9 (12), 1512-1519 (2006).
- Wu, L. -J., Vadakkan, K. I., Zhuo, M. ATP-induced chemotaxis of microglial processes requires P2Y receptor-activated initiation of outward potassium currents. Glia. 55 (8), 810-821 (2007).
- Kolodziejczak, M., et al. Serotonin Modulates Developmental Microglia via 5-HT 2BReceptors: Potential Implication during Synaptic Refinement of Retinogeniculate Projections. ACS Chemical Neuroscience. 6 (7), 1219-1230 (2015).
- Schafer, D. P., et al. Microglia Sculpt Postnatal Neural Circuits in an Activity and Complement-Dependent Manner. Neuron. 74 (4), 691-705 (2012).
- Pfeiffer, T., Avignone, E., Nägerl, U. V. Induction of hippocampal long-term potentiation increases the morphological dynamics of microglial processes and prolongs their contacts with dendritic spines. Scientific Reports. 6, 32422(2016).
- Parkhurst, C. N., et al. Microglia Promote Learning-Dependent Synapse Formation through Brain-Derived Neurotrophic Factor. Cell. 155 (7), 1596-1609 (2013).
- Wu, Y., Dissing-Olesen, L., Macvicar, B. A., Stevens, B. Microglia: Dynamic Mediators of Synapse Development and Plasticity. Trends in Immunology. 36 (10), 605-613 (2015).
- Ohsawa, K., et al. P2Y12 receptor-mediated integrin-beta1 activation regulates microglial process extension induced by ATP. Glia. 58 (7), 790-801 (2010).
- Kurpius, D., Wilson, N., Fuller, L., Hoffman, A., Dailey, M. E. Early activation, motility, and homing of neonatal microglia to injured neurons does not require protein synthesis. Glia. 54 (1), 58-70 (2006).
- Stence, N., Waite, M., Dailey, M. E. Dynamics of microglial activation: a confocal time-lapse analysis in hippocampal slices. Glia. 33 (3), 256-266 (2001).
- Dissing-Olesen, L., Macvicar, B. A. Fixation and Immunolabeling of Brain Slices: SNAPSHOT Method. Current Protocols in Neuroscience. 71, 1.23.1-12 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Aitken, P. G., et al. Preparative methods for brain slices: a discussion. Journal of Neuroscience Methods. 59 (1), 139-149 (1995).
- Paris, I., et al. ProMoIJ: A new tool for automatic three-dimensional analysis of microglial process motility. Glia. 66 (4), 828-845 (2018).
- Pagani, F., et al. Defective microglial development in the hippocampus of Cx3cr1 deficient mice. Frontiers in Cellular Neuroscience. 9 (229), 111(2015).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods in Molecular Biology. , Clifton, NJ. 221-242 (2014).
- Mainen, Z. F., et al. Two-photon imaging in living brain slices. Methods. 18 (2), San Diego, CA. 231-239 (1999).
- Tanaka, Y., Tanaka, Y., Furuta, T., Yanagawa, Y., Kaneko, T. The effects of cutting solutions on the viability of GABAergic interneurons in cerebral cortical slices of adult mice. Journal of Neuroscience Methods. 171 (1), 118-125 (2008).
- Gyoneva, S., et al. Systemic inflammation regulates microglial responses to tissue damage in vivo. Glia. 62 (8), 1345-1360 (2014).
- Heindl, S., et al. Automated Morphological Analysis of Microglia After Stroke. Frontiers in Cellular Neuroscience. 12, 106(2018).
- Dailey, M. E., Eyo, U., Fuller, L., Hass, J., Kurpius, D. Imaging microglia in brain slices and slice cultures. Cold Spring Harbor Protocols. 12 (12), 1142-1148 (2013).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten