Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Two-photon Imaging d’Attraction des microglies processus vers l’ATP ou la sérotonine cérébrale aiguë tranches
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
La microglie, les résidents cellules immunitaires du cerveau, répondre rapidement avec des changements morphologiques aux modifications de leur environnement. Ce protocole décrit comment utiliser la microscopie biphotonique à étudier l’attraction des microglies processus vers la sérotonine ou l’ATP en tranches cérébrale aiguë des souris.
Résumé
Les cellules microgliales sont des cellules immunitaires innées résidents du cerveau qui constamment analyser leur environnement avec leurs longs prolongements et, à la perturbation de l’homéostasie, subissent des changements morphologiques rapides. Par exemple, une lésion laser induit en quelques minutes une croissance axée sur des processus microgliales, également appelé « motilité directionnelle », vers l’endroit de la blessure. Un effet similaire peut être obtenu en fournissant localement ATP ou la sérotonine (5-hydroxytryptamine [5-HT]). Dans cet article, nous décrivons un protocole pour induire une croissance directionnelle des microglies processus vers une application locale d’ATP ou 5-HT dans des tranches de cerveau aiguë des souris jeunes et adultes et à l’image de cette attraction au fil du temps par la microscopie multiphoton. On propose une méthode simple de quantification avec analyse d’images gratuit et open source. Un défi qui caractérise encore les tranches cérébrale aiguë est le temps limité, diminuant avec l’âge, au cours de laquelle les cellules demeurent dans un état physiologique. Ce protocole, par conséquent, souligne certaines améliorations techniques (imagerie chambre avec une perfusion double chambre interface medium, air liquide) visant à optimiser la viabilité des cellules microgliales pendant plusieurs heures, surtout dans les tranches de souris adultes.
Introduction
Les cellules microgliales sont les macrophages résidents du cerveau et jouent un rôle dans les deux conditions physiologiques et pathologiques1,2. Ils ont une morphologie très ramifiée et sont constamment étendre et rétracter leur processus3,4. Ce comportement de « balayage » est censé être liées et nécessaires à l’enquête de leur environnement. La plasticité morphologique des microglies est exprimée en trois modes. Tout d’abord, certains composés modulent rapidement microgliales morphologie : l’addition d’ATP5,6 ou NMDA5,7 , dans le milieu de baignade tranches cérébrale aiguë accroît la complexité des microglies ramifications, Alors que la noradrénaline diminue il6. Ces effets sont directement médiées par les récepteurs microgliales (par l’ATP et de la noradrénaline) ou nécessitent une libération d’ATP des neurones (pour NMDA). En second lieu, la vitesse de croissance et rétraction des processus microgliales, appelé la motilité ou « surveillance », peut être affectée par des facteurs extracellulaires8, perturbations de l’homéostasie du9,10ou mutations9, 10,11. En troisième lieu, en plus de ces changements isotropes de morphologie et de la motilité, microglie ont la capacité d’étendre leurs processus déviés vers une pipette livrant ATP3,5,12, 13 , 14, la culture, en tranches cérébrale aiguë ou in vivo, ou livraison de 5-HT dans cérébrale aiguë tranches15. Une telle croissance axée sur des processus microgliales, également appelé motilité directionnelle, a été décrite comme une réponse à un laser local lésion3,4. Ainsi, physiologiquement, il peut être lié à la réponse à une lésion ou requis pour le ciblage des microglies processus vers les synapses ou régions cérébrales nécessitant l’élagage pendant développement15,16, ou enphysiologiques 17 ,18,19 ou situations pathologiques9,18,19,20 , à l’âge adulte. Les trois types de changements morphologiques s’appuient sur différents mécanismes intracellulaires de13,11,20, et une substance donnée ne pas nécessairement modulé chacun d’eux (p. ex., NMDA, qui agit indirectement sur la microglie, a un effet sur la morphologie, mais n’induit pas de motilité directionnelle5,,7). Par conséquent, lorsque le but de caractériser les effets d’un composé, une mutation ou une pathologie sur la microglie, il est important de caractériser les trois composantes de leur plasticité morphologique. Nous décrivons ici une méthode pour étudier la croissance directionnelle des microglies processus vers une source locale de composé, qui est, ici, ATP ou 5-HT.
Il existe plusieurs modèles pour étudier l’attraction des microglies processus : cultures primaires en environnement 3D6,18,19, cérébrale aiguë tranches6,13,15et in vivo imagerie des3,13. L’approche in vivo est le meilleur pour préserver l’état physiologique des cellules microgliales. Cependant, imagerie intravitale des régions profondes nécessite des interventions chirurgicales complexes et, par conséquent, il est souvent limité à des couches corticales superficielles. L’utilisation de culture primaire de cellules microgliales est la technique la plus simple pour tester un grand nombre de conditions avec un nombre limité d’animaux. Néanmoins, il est impossible d’obtenir la même morphologie cellulaire comme en vivo, et les cellules perdent leurs interactions physiologiques avec les neurones et les astrocytes. Tranches de cerveau aiguë représentent un compromis entre ces deux approches. Ce modèle permet aux chercheurs d’étudier les structures cérébrales qui sont par ailleurs difficiles d’atteindre d’images à haute résolution in vivo et d’enquêter sur des tranches de stades néonatales, attendu que transcrânienne microscopie est principalement effectuée à l’âge adulte. Enfin, il rend possible d’observer en temps réel les effets de la demande de drogue local et de répéter les expériences tout en utilisant un nombre limité d’animaux. Néanmoins, un problème avec des tranches de cerveau aigu est le peu de temps (quelques heures) au cours de laquelle les cellules demeurent vivantes, notamment pour les tranches de souris plus de deux semaines et la variation du potentiel de la morphologie des cellules microgliales au fil du temps21,22 .
Nous décrivons ici un protocole visant à préparer des tranches de cerveau aiguë de jeunes et d’adultes Cx3cr1GFP / + souris jusqu'à deux mois, avec la préservation de la morphologie de la microglie et motilité pendant plusieurs heures. Nous, ensuite, décrivent comment utiliser ces tranches pour étudier l’attraction des microglies processus vers composés comme ATP ou 5-HT.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les expériences ont été approuvées par le comité local d’éthique (Comité de Darwin, accords 1170 # et #10921).
1. préparation des Micropipettes de verre pour l’Application locale de composés
- Préparer les pipettes de borosilicate capillaires en verre à paroi mince avec un extracteur de l’électrode. Ajustez les paramètres pour obtenir des pipettes avec un 4-5 µm de diamètre à leur extrémité. Figure 2D montre une pipette en fond clair à faible grossissement.
2. les solutions
- S’assurer que seule verrerie qui a été nettoyé par un cycle d’autoclave, suivi par rinçage 2 x - 3 x avec l’eau ultrapure, seront utilisés. Jamais utiliser de verrerie qui a été au contact de paraformaldéhyde.
-
Préparer un 2 mol· L-1 CaCl2 solution mère en dissolvant 14,7 mg de CaCl2·2H2O dans 50 mL d’eau de grande pureté (eau ultrapure, résistance 18,2 MΩ ; les traces de métal dans l’eau distillée ou eau du robinet peuvent conduire à qualité suboptimales tranche due à effets pro-oxydatif).
- Conserver cette solution-mère à température ambiante pendant un maximum d’un mois.
-
Le jour de l’expérience, préparer 1 L de solution de choline-aCSF (artificiel liquide céphalo-rachidien), dont la composition est 110 mmol· Choline-1 de L Cl, 25 mmol· L-1 de glucose, 25 mmol· L-1 NaHCO3, mmol· 7 L-1 MgCl2, 11,6 mmol· L-1 d’acide ascorbique, mmol· 3.1 Pyruvate de sodium L-1 , mmol· 2,5 L-1 KCl, mmol· 1.25 L-1 NaH2PO4et mmol· 0,5 L-1 CaCl2, 0,5.
- Pour préparer cette solution, ajouter, dans l’ordre suivant, dans une fiole jaugée de 1 L est diplômé : 0,186 g de KCl, 0,195 g NaH2PO4, 2,04 g d’acide ascorbique, 2,1 g de NaHCO3et 4,5 g de glucose.
- Environ la moitié du volume final remplissez-le d’eau ultrapure et remuez jusqu'à dissolution complète.
- Ajouter 0,34 g de pyruvate de sodium et 15,36 g de choline Cl.
Remarque : Il est commode de première dissolution la choline Cl avec 5 à 10 mL de la solution préparée à l’étape 2.3.2 avant de l’ajouter à la solution entière. - Ajouter 7 mL de 1 mol· L-1 MgCl2 et 250 µL de 2 mol· L-1 CaCl2 (préparé à l’étape 2.2) à la solution.
- Remplir la fiole jaugée de 1 L avec de l’eau ultrapure.
- Avec une pression de vapeur osmomètre, vérifiez que l’osmolarité est entre 300 et 310 mΩ. Si ce n’est pas le cas, réglez-le au glucose.
- Vérifier le pH après carbogenation (bulles avec « carbogen », un mélange de 95 % O25 % CO2) et ajuster, si nécessaire, de 7,3 à 7,4 avec 10 M de NaOH.
- Transvaser la solution dans une bouteille en verre pour le stockage. Conserver la bouteille dans le réfrigérateur jusqu'à l’utilisation (étape 3.1).
Remarque : Il est recommandé de faire une solution fraîche le jour de l’expérience. Toutefois, si nécessaire, choline-FSCA peut être conservé jusqu'à deux jours à 4 ° C.
-
Le jour de l’expérience, préparer 1 L de solution aCSF, dont la composition est mmol· 124 L-1 NaCl, 26,2 mmol· L-1 NaHCO3, 25 mmol· L-1 de glucose, mmol· 2,5 L-1 KCl, 2 mmol· L-1 CaCl2, 1 mmol· L-1 MgCl2et 1,25 mmol· L-1 NaH2PO4.
- Pour préparer cette solution, ajouter, dans l’ordre suivant, dans une fiole jaugée : 0,150 g NaH2PO4, 0,186 g de KCl, 2,2 g de NaHCO3, 4,5 g de glucose et 7,3 g de NaCl. Porter la solution à un volume de 1 L avec de l’eau ultrapure et remuez vigoureusement sur une plaque de remuer.
- Ajouter 1 mL de 1 mol· L-1 MgCl2 et 1 mL de 2 mol· L-1 CaCl2 jusqu'à la solution et la solution de l’aCSF une bouteille en verre pour le stockage.
- Vérifier si l’osmolarité est 300-310 mΩ· L-1 et, si non, l’ajuster avec du glucose.
- Vérifier le pH après carbogenation (c.-à-d., bouillonnant avec « carbogen ») et ajuster, si nécessaire, de 7,3 à 7,4 avec 10 M de NaOH.
- Transvaser la solution dans une bouteille en verre pour le stockage. Conserver la bouteille dans le réfrigérateur jusqu'à l’utilisation (étape 3.1).
Remarque : Il est recommandé de faire une solution fraîche le jour de l’expérience. Toutefois, une alternative consiste à préparer un 10 x solution contenant NaCl, NaHCO3KCl et NaH2PO4 à 10 fois la concentration finale, qui peut être stocké pendant pas plus d’une semaine à 4 ° C. Faire le FSCA final le jour de l’expérience en diluant la solution 10 x avec de l’eau ultrapure et en ajoutant du glucose, CaCl2et MgCl2.
- Préparer les solutions de drogue le jour de l’expérience. Utiliser la solution de FSCA pour leur donner les concentrations finales qui sont, ici, µmol· 500 L-1 pour l’ATP et 5 µmol· L-1 pour la 5-HT.
Remarque : Pour l’ATP, une solution peut être préparée (par exemple, 50 mM ATP dans l’eau), stocké sous forme aliquoté à-20 ° C et dilué avec l’aCSF à la concentration finale le jour de l’expérience. En revanche, la solution de la 5-HT (sérotonine-HCl) doit être préparée à partir de poudre sur le jour de l’expérience, à 1 µg·ml-1 dans l’eau, maintenue à 4 ° C pour éviter l’oxydation de la 5-HT et diluée dans FSCA au moment de l’expérience.
3. préparation des tranches de cerveau aiguë
-
Préparation de la zone de dissection
- Préparation de 70 mL de choline-FSCA glacée dans un bécher de 80 mL mis dans la glace, à utiliser pour la perfusion cardiaque, refroidissement rapide du cerveau et de tranchage. Préparation de 150 mL de choline-FSCA dans un 200 mL cristallisant plat, placé dans un bain d’eau chauffée à 32 ° C. Place un tamis de maille en nylon dans le plat de cristallisation pour conserver les tranches. Cela servira à laisser les tranches de récupérer pendant 10 min juste après le tranchage.
- Au moins 30 min avant de commencer la dissection (section 3.2), bouillir ces deux solutions (70 mL de choline-aCSF sur glace) et 150 mL de choline-FSCA à 32 ° C avec carbogen. Maintenir la carbogenation constante pendant toute la procédure.
- Préparer le dispositif de chambre de l’interface (Figure 1), qui sera utilisé pour garder les tranches jusqu'à leur utilisation.
- Dans une aliment scellé boîte (10 x 10 cm ou 10 cm de diamètre, 8 cm de hauteur), installée sur un agitateur magnétique, placez un cristallisant plat avec une barre de 200 mL aimant.
- Ajouter 200 mL de FSCA dans ce plat de cristallisation et placez le support de tranche interface 3D-imprimé sur le dessus (le titulaire de la tranche d’interface est composé de deux parfaitement raccord parties, avec une maille de polyamide tendue entre eux, la Figure 1 a, B).
- Supprimer un volume excessif de la cristallisation plat pour garder seulement une fine pellicule de solution couvrant le maillage de la titulaire de la tranche pour l’interface. Plus tard, cela créera une jante fine de solution qui entoure les tranches (mais sans les couvrir).
- Mettre quelques millimètres du FSCA au bas de la boîte verte et bouillir avec carbogen (dès la première utilisation, faire un petit trou dans le mur de boîte d’aliment scellé pour s’assurer que le tuyau peut entrer dans la boîte).
- Fermez la boîte étanche tout en maintenant constante carbogenation. Cela créera un humidifié 95 % O25 % CO2 environnement riche dans lequel les tranches seront transférés après leur rétablissement en choline-aCSF et conservés avant ils sont imagés. Ce dispositif est ci-après dénommé la « chambre d’interface » (Figure 1).
-
Dissection du cerveau et découpage
- Anesthésier la souris avec une injection intrapéritonéale de 50 µg·ml-1 pentobarbital (0,15 mL/20 g de poids de corps de souris), immobiliser, exposer le cœur et effectuer une perfusion cardiaque avec 10 mL de glacé, carbogenated, choline-aCSF (voir l’étape 3.1.1), avec une pompe péristaltique. Observer la pâleur du foie comme indicateur d’une bonne perfusion. La perfusion dure moins de 5 min.
- Décapiter la souris et couper la peau afin d’exposer le crâne. Avec des grands ciseaux, appliquer deux coupes transversales du foramen grand et une coupe sagittale depuis longtemps et, à l’aide d’une pince fine, retirer les plaques du crâne.
- Doucement et rapidement extraire le cerveau (en moins de 1 min) et placez-le pendant 1 min dans le bécher de 80 mL contenant le restant (environ 60 mL) glacee choline-aCSF (toujours en constante carbogenation), afin de refroidir.
- Transférer le cerveau sur un papier-filtre préalablement mouillé avec FSCA.
- Découper le cerveau selon la région du cerveau d’intérêt et angle préféré de tranchage. Par exemple, pour l’image du thalamus ou l’hippocampe sur tranches coronales, découper avec une lame de bistouri le cervelet et, ensuite, environ 2 mm des extrémités rostrales et caudales du cerveau.
Remarque : Il est important d’enlever des parties de cerveau qui sont trop rostrale ou trop caudale parce que plus la région de couper avant d’atteindre la zone d’intérêt, plus vite le tranchage. Une durée totale de tranchage (étape 3.2.7) de moins de 20 min est recommandée. - Pour les tranches coronales, positionner et coller (avec de la colle cyanoacrylate) la face caudale du cerveau sur un 10 cm boîte de pétri, collé sur le bloc de coupe d’un trancheur vibrant et placez-la dans la chambre de réservoir du trancheur vibrant, qui est placé dans une chambre plus grande rempli de glaçons. Puis, remplissez la boîte de pétri avec tous le reste glacée choline-FSCA.
- Tout en gardant constant le 95 % O25 % CO2 bouillonnant de la choline glacee-aCSF, couper 300 µm d’épaisseur de tranches coronales (vitesse : 0,08 mm·s-1, de la vibration de la lame : 60 Hz, amplitude de vibration : 1 mm).
- Recueillir les tranches de cerveau avec un transfert jetables de large-bouche (4 mm de diamètre) pipetter, un par un après chaque passage unique de la lame, afin d’éviter l’accumulation de composés toxiques libérés par la périphérie des tranches. Prendre soin d’éviter les bulles d’air lors du transfert et de placer chaque tranche dans la choline-FSCA à 32 ° C pendant environ 10 min de récupération.
- Avec la pipette de transfert, déposer les tranches sur des morceaux de nettoyage de la lentille papier surmonté d’une goutte de choline-FSCA. Aspirer l’excès de choline-aCSF et, à l’aide de la spatule, déposer les tranches, posés sur le tissu de nettoyage de lentille, sur le maillage de la chambre d’interface contenant FSCA carbogenated à température ambiante (voir 3.1.3.5). Laisse la tranche récupérer dans cet environnement pendant au moins 30 min.
Remarque : Après cela, les tranches sont prêtes et peuvent être utilisés pour les souris de la microglie imagerie jusqu'à 6 h après l’extraction de cerveau de jeunes (moins d’un mois) et jusqu'à 4 h après l’extraction du cerveau des adultes âgés de 2 mois.
4. two-photon Microscopy
- Réglage des paramètres
- Allumez le système multiphotonique (détecteurs de l’hybride, laser, scanner, modulateur électro-optique, microscope).
- Régler le laser à 920 nm, vérifiez que le laser est verrouillée en mode et régler la puissance à 5 % - 15 % et le gain à 10 %. Cela correspond à une puissance de 3 à 5 mW au titre de l’objectif. Veiller à ce que les détecteurs nondescanned sont engagés et installé les filtres appropriés d’excitation et d’émission.
- Définir les paramètres du logiciel d’imagerie aux valeurs suivantes : pour la taille de trame, 1024 x 1024 pixels correspondant à une superficie de 295.07 x 295.07 µm ; pour le zoom, 2. Si le signal est très bruyant, appliquer une moyenne de la ligne 2. Pour la dynamique pixel, définissez le logiciel d’imagerie à 12 bits ou plus.
Remarque : Images avec une plus grande valeur de bit permettent aux chercheurs de distinguer les plus petites différences dans l’intensité de fluorescence que les images d’une valeur de bit inférieure : un changement d’une valeur de gris dans une image de 8 bits correspond à un changement des 16 valeurs de gris dans un 12 bits et de gris 256 valeurs i n une image 16 bits. Par conséquent, supérieur-bit images sont plus appropriées pour l’analyse quantitative, mais parce que leur taille augmente avec la profondeur de bit, capacité de stockage et de calcul puissance peut devenir limitant. - Sélectionnez le mode de balayage XYZT avec une gamme de Z-intervalle à 2 µm et un T-intervalle de 2 min.
Remarque : Le x, y et z de résolution sont déterminées par le théorème d’échantillonnage de Nyquist. Une taille de Z-étape autour de 0,8 serait optimale pour résoudre les microglies processus (d’un diamètre de < 1 µm), mais limite la résolution optique de la microscopie multiphoton (920 nm avec un objectif de NA 0,95, la résolution axiale est autour de 1 µm). En plus de cela barrière physique, dans une expérience de création d’images en direct, le ratio signal-bruit ou de la sensibilité, la résolution, la vitesse et la question du temps total d’observation. Compte tenu de tous ces paramètres, une z-étape de 2 µm (comme dans nombreuses études3,11,14), une taille d’image de 1024 x 1024 pixels et une acquisition à grande vitesse à l’aide d’un scanner résonant couplé à des détecteurs de HyD (il prend environ 15 s d’acquérir 50 z-plans) ont été sélectionnés ici. La fréquence des acquisitions est une série XYZT toutes les 2 min et la durée totale est de 30 min. Si l’installation n’est pas rapide ou assez sensible, il est possible de réduire la résolution latérale (jusqu'à 512 x 512) ou le nombre de z-tranches (par imagerie exclusivement dans la profondeur z qui présente la plus forte fluorescence [c'est-à-dire, pas les plus profonde de z-tranches où fluorescence est faible]), ou diminuer la vitesse du scanner. La résolution axiale peut également être diminuée en augmentant la z-étape jusqu'à 3 µm, mais comme cela peut impacter la quantification, toutes les expériences à comparer doivent être effectuées avec la même étape-z.
Remarque : Il est possible d’effectuer des expériences similaires sur des tranches de CX3CR1creER-YFP souris18, une lignée de souris utilisée pour induire la suppression génétique dans les cellules microgliales uniquement et dans laquelle la microglie constitutivement expriment la protéine fluorescente jaune (YFP). Cependant, le niveau d’expression de la YFP est très faible par rapport à la protéine fluorescente verte (GFP) dans CX3CR1GFP / + souris ; ainsi, l’imagerie est possible mais difficile et nécessite l’optimisation des paramètres d’acquisition. Il est recommandé de les ajuster comme suit. - Régler le laser à 970 nm (qui est mieux adapté à une excitation YFP que 920 nm), la puissance à 50 % et le gain à 50 %, ce qui correspond à une puissance laser sous l’objectif de 5 à 6 mW.
- Définir une moyenne de ligne de 4 (ou plus) afin d’améliorer le rapport signal-bruit.
- Positionnement de la tranche et de la micropipette de verre et l’application locale du composé
- Branchez la pompe péristaltique à la chambre d’enregistrement, 30 min avant de commencer l’enregistrement. Après le nettoyage du système de perfusion ensemble avec 50 mL d’eau ultrapure, commencer la perfusion de la chambre d’enregistrement avec le FSCA (50 mL) contenue dans un bécher de verre sous la constante carbogenation. Tout au long de l’expérience, garder le FSCA circulant à 32 ° C avec un microheater inline ou un radiateur Peltier.
Remarque : Une chambre de perfusion spécifiques avec perfusion haut et le bas est conçue pour optimiser l’oxygénation des deux côtés de la tranche. La chambre de perfusion se compose de deux parfaitement raccord parties, avec une maille de polyamide tendue entre eux (Figure 2 a, B). Par rapport aux autres types de chambres, où la tranche est directement posé sur une lamelle de verre, cette chambre réduit la mort neuronale dans la partie inférieure de la tranche, améliore la viabilité et réduit les mouvements de tranche induites par son gonflement. - Avec une pipette de transfert jetable de large-bouche, transférer la tranche de cerveau pour être photographié dans le bécher de FSCA pour enlever le papier de la lentille, laisser couler (comme une preuve qu’aucune bulle d’air n’est attaché) et transférer à la chambre d’enregistrement (perfusion).
- Positionner un support de tranche (une épingle à cheveux en platine avec les deux branches, rejoints par des threads parallèles en nylon) sur la tranche pour minimiser le mouvement de la tranche en raison du débit de perfusion.
- L’éclairement lumineux-zone permet de cibler la zone cérébrale concernée (temps d’exposition : 50 à 80 ms) à l’aide d’un objectif de faible grossissement (5 X ou 10 X). Basculez vers l’objectif à immersion d’eau grossissement (25 x avec un objectif x 0,35) plus élevé et ajuster la position.
Remarque : Éviter aux champs d’image proche de filets en nylon du détenteur de la tranche, car ils peuvent bloquer la lumière et déformer localement la tranche. Assurez-vous que la zone d’intérêt est plate. Le cas échéant, retirer le titulaire de la tranche afin de repositionner la tranche et/ou le titulaire de la tranche. - L’illumination par fluorescence permet de localiser des microglies fluorescent à être photographiée sur le champ (temps d’exposition : 250-500 ms).
Remarque : Cette étape permet aux chercheurs de vérifier la présence de cellules dans la région d’intérêt et leur intensité de fluorescence et de contrôler pour la quantité de débris cellulaires. - Remblayer la pipette avec 10 µL de FSCA avec ATP, 5-HT, ou le médicament d’intérêt à sa concentration finale. La pointe vers le bas et secouez légèrement la pipette remplie de drogue pour enlever les bulles d’air piégées dans la pointe.
Remarque : Si la solution à doser a tendance à former des bulles, envisagez d’utiliser des pipettes de borosilicate avec un filament interne. Fuite de l’ATP sur la pipette peut attirer microgliales processus même avant l’injection (dans ce cas, il sera visible à l’étape de l’analyse). Bien que cela devrait être modérée avec la concentration d’ATP utilisées (500 µmol· L-1), si c’est un problème, envisagez déclarants une micropipette avec 2 mL de FSCA avant l’ajout de l’ATP (ou tout autre composé) solution à étape 4.2.6. - Montez la pipette remplie dans un porte-pipette, connecté avec tube transparent à une seringue de 5 mL, avec un piston placé à la position de 5 mL. Le titulaire de la pipette est monté sur un micromanipulateur trois axes.
- Sous un éclairement lumineux-zone, utilisez le micromanipulateur pour positionner la pipette dans le centre du champ. Pour un centrage optimal et reproductible, afficher et utiliser les dirigeants sur l’image.
- Abaisser la pipette doucement vers la tranche, contrôle et réglage de l’objectif à la fois, jusqu'à ce que la pointe de pipette légèrement touche la surface de la tranche. Arrêtant la descente de la pipette, dès qu’il est visible que la tranche a été touchée permet l’embout de la pipette pénétrer de 80 à 100 µm de la surface de la tranche (voir la Figure 3 b).
- Régler le laser (voir les paramètres ci-dessus) et mettre le microscope multiphoton mode. Assurez-vous que la chambre est projetée de toute source de lumière (par exemple, un écran d’ordinateur). Mettre sur les détecteurs de nondescanned et régler le gain. Utiliser une table de correspondance (LUT) avec une limite supérieure codés par couleur pour éviter de saturer les pixels de l’image.
- Déterminer l’épaisseur de la tranche à être photographiée (c.-à-d., les majuscules et minuscules z-positions où la fluorescence est détectable [habituellement entre 220 et 290 µm au total]).
Remarque : À la surface de la tranche, il y a une densité accrue des processus et éventuellement des microglies, souvent avec une morphologie inhabituelle, par rapport à l’intérieur de la tranche. Cette accumulation sera plus frappante avec le temps (c.-à-d.., plus visible dans la dernière que dans la première tranche du cerveau à être photographiée). Par conséquent, les planes z dans le premier ~ 30 µm ne doivent pas être utilisés pour l’analyse et peuvent même être ignorées pour l’acquisition. - Démarrer l’enregistrement pour une durée totale de 30 min (ou plus si vous le souhaitez) et après une base de 5 min, localement appliquer le composé à tester (sans interrompre l’imagerie). Pour ce faire, appuyez lentement sur le piston de la seringue, connecté à la micropipette, depuis les 5 mL au poste 1 mL (environ 5 s). Résistance lorsque vous appuyez sur le piston doit se fait sentir immédiatement. Si ce n’est pas le cas, la pointe peut-être être rompue.
Remarque : Pour un expérimentateur formé, les injections avec cette méthode sont reproductibles, mais alternativement à la manipulation manuelle d’une seringue, la pipette pourrait être liée à un système d’éjection automatique de la pression pour permettre un meilleur contrôle du volume livré. L’injection crée une déformation physique de la tranche à l’endroit de l’injection. Cette distorsion est visible a posteriori dans les deux premières ou trois images après l’injection, mais ne doit pas être visible sur la quatrième image, (c'est-à-dire, 8 min après l’injection). Si elle persiste, pensez à modifier les paramètres pour la préparation de la pipette. - À la fin de l’acquisition (30 min), jeter une micropipette et enlever la tranche. Si vous le désirez, fixer la tranche pour plus amples immunomarquage. Par exemple, la méthode de capture instantanée est optimisée pour la fixation et coloration d’épaisses tranches de23.
- Avant de commencer une nouvelle tranche d’image, de faire le film 2D (section 5.1) afin de vérifier que les microglies ont une morphologie normale et se déplacent et, par conséquent, que les tranches sont en bonne santés.
- Branchez la pompe péristaltique à la chambre d’enregistrement, 30 min avant de commencer l’enregistrement. Après le nettoyage du système de perfusion ensemble avec 50 mL d’eau ultrapure, commencer la perfusion de la chambre d’enregistrement avec le FSCA (50 mL) contenue dans un bécher de verre sous la constante carbogenation. Tout au long de l’expérience, garder le FSCA circulant à 32 ° C avec un microheater inline ou un radiateur Peltier.
5. analyse de l’Attraction des microglies processus
-
Projection 2D et la correction de dérive
- Ouvrez le fichier (. FRV) avec les Fidji24.
- Si nécessaire, faire un substack (Image/Stacks/Tools/Make Substack) avec seulement les z-plans d’intérêt. Par exemple, exclure les planes z correspondant à la surface de la tranche s’ils ont été acquis, mais ne doivent ne pas être utilisés pour l’analyse (voir la NOTE après l’étape 4.2.11) et les z-plans plus profonds avec aucune fluorescence. La pile finale contient généralement 90-110 z-tranches (180-220 µm).
- Lancer la fonction projet Z (Image/Stacks/Z Project«) et sélectionnez le type de projection d’Intensité Max pour faire les projections de la z-pile acquis à chaque instant.
- Lancer le plugin MultiStackReg (Plugin/enregistrement/MultiStackReg), en sélectionnant Action 1 : aligner et Transformation : corps rigide pour corriger les dérives légères pouvant avoir eu lieu lors de l’acquisition. Enregistrer ce film 2D comme un nouveau fichier (.tiff).
-
Traitement des données
- Ouvrir ce nouveau fichier avec Icy25.
- Dessiner une zone circulaire de R1 d’intérêt (ROI) de 35 µm de diamètre, centrée sur le site d’injection (identifié notamment par l’ombre de la pipette et la distorsion créée au moment de l’injection).
- Utiliser le plugin evolution intensité ROI et mesurer l’intensité moyenne dans le temps en R1.
- Enregistrer les résultats à un. Fichier XLS.
-
La quantification et la représentation des résultats
- Afin de quantifier la réponse microgliales au fil du temps, de déterminer à chaque instant
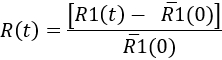
Here,'R1(0) est la moyenne des valeurs R1(t) avant l’injection. Ensuite, les résultats peuvent être représentés comme une cinétique de la réaction microgliale, ou à un moment précis point (voir Figure 7).
- Afin de quantifier la réponse microgliales au fil du temps, de déterminer à chaque instant
Access restricted. Please log in or start a trial to view this content.
Résultats
Ce protocole décrit une méthode pour induire, observer et quantifier la croissance orientée des microglies processus vers un composé appliqué localement, par exemple, l’ATP ou 5-HT, dans cérébrale aiguë tranches de jeune ou adulte (au moins jusqu'à deux mois) souris. Parmi les facteurs qui contribuent au maintien des tranches de cerveau d’animaux adultes en bon état pendant plusieurs heures est l’utilisation de deux outils conçus pour optimiser la survie des cellules à d...
Access restricted. Please log in or start a trial to view this content.
Discussion
Par le maintien, à la différence en dissocié ou organotypique trancher une intégrité structurelle avec les réglages de réseau limité, de la culture, des tranches de cerveau aiguë aux chercheurs d’étudier des microglies dans leur environnement physiologique. Cependant, une des principales limites est le fait que la procédure de tranchage crée des blessures qui peuvent rapidement compromettre la viabilité des neurones, particulièrement dans le cerveau adulte. Comme les microglies sont particulièrement réa...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions les cellules et tissus Imaging Facility de l’Institut du Fer à Moulin, où toutes les image acquisition et l’analyse ont été effectuées. Ce travail a été soutenu en partie par le Centre National de la Recherche Scientifique, l’Institut National de la Santé et de la Recherche Médicale, Sciences de l’Université de la Sorbonne et par des subventions de Sorbonne Universités-Pierre et Marie Curie université () Programme Emergence-UPMC 2011/2014), la Fondation pour la Recherche sur le Cerveau, la Fondation de France, la Fondation pour la Recherche Médicale « Equipe FRM DEQ2014039529 », le Ministère Français de la recherche (Agence Nationale pour la Recherche ANR-17-CE16-0008 et l’avenir des Investissements du programme « Bio-Psy Labex » ANR-11-IDEX-0004-02) et une recherche Collaborative dans le programme de neurosciences computationnelles, National Science Foundation/Français Agence nationale pour la recherche (numéro : 1515686). Tous les auteurs sont affiliés à la recherche des groupes qui sont membres de l’école des neurosciences de Paris (PEV) et de la Bio-Psy Labex. F.E. est étudiant au doctorat affilié à la Sorbonne Université, Collège Doctoral, F-75005 Paris, France et est financé par le Labex Bio-Psy. V.M. est stagiaire postdoctorale financé par la recherche Collaborative dans le programme de neurosciences computationnelles, National Science Foundation/Français Agence nationale pour la recherche (numéro : 1515686). Les auteurs remercient Marta Kolodziejczak qui ont participé à l’initiation du projet.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| for pipettes preparation | |||
| Clark Borosilicate Thin Wall Capillaries | Harvard Apparatus | 30-0065 | Borosilicate Thin Wall without Filament, 1.5 mm OD, 1.17 mm ID, 75 mm L , Pkg. of 225 |
| DMZ Universal Puller | Zeitz Instrumente | ||
| Name | Company | Catalog Number | Comments |
| for solutions | |||
| Calcium Chloride dihydrate (CaCl2) | Sigma | C5080 | |
| Choline Chloride | Sigma | C7527 | |
| D-(+)-Glucose | Sigma | G8270 | |
| L-Ascorbic acid | Sigma | A5960 | |
| Magnesium Chloride solution 1 M (MgCl2) | Sigma | 63020 | |
| Potassium chloride SigmaUltra >99.0% (KCl) | Sigma | P9333 | |
| Sodium bicarbonate (NaHCO3) | Sigma | S5761 | |
| Sodium Chloride (NaCl) | Sigma | S5886 | |
| Sodium phosphate monobasic | Sigma | S5011 | |
| Sodium pyruvate | Sigma | P2256 | |
| Ultrapure water | MilliQ | for all the solutions | |
| Name | Company | Catalog Number | Comments |
| for slice preparation | |||
| 2x 200 mL crystalizing dishes | |||
| 80 mL Pyrex beaker | |||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For mice perfusion and 2-photon chamber perfusion (aCSF) |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | ||
| Dolethal | Vetoquinol | Dolethal 50 mg/mL | |
| Filter papers (Whatman) | Sigma | WHA1001042 | Whatman qualitative filter paper, Grade 1 (Pore size: 11µM) |
| Fine Scissors - Sharp | Fine Science Tools | 14060-60 | |
| Food box 10 cm diameter, 8 cm Height | |||
| glue (ethyl cyanoacrylate) | Loctite | super glue 3 power flex | |
| Hippocampal Tool (spatula) | Fine Science Tools | 10099-15 | The largest extremity has to be angled at 90° |
| Ice | |||
| Iris Forceps (curved) | Moria | MC31 | |
| Lens cleaning tissue | THOR LABS | ||
| Nylon mesh strainer | diameter 7 cm | ||
| Razor blades | Electron Microscopy Sciences | 72000 | For the slicer |
| scalpel blade | |||
| Slice interface holder | home-made, the file for 3D printing is provided in Supplemental Material | ||
| Surgical Scissors - Sharp | Fine Science Tools | 14002-14 | |
| Vibrating slicer | Thermo Scientific | 720-2709 | Model: HM 650V (Vibrating blade microtome) |
| Water bath | Set at 32 °C (first recovery step) | ||
| Name | Company | Catalog Number | Comments |
| for slice imaging | |||
| × 25 0.95 NA water-immersion objective | Leica Microsystems (Germany) | HCX Irapo | |
| 2-photon MP5 upright microscope with resonant scanners (8 kHz) and two HyD Hybrid detectors | Leica Microsystems (Germany) | ||
| Antlia-3C Digital Peristaltic pump | DD Biolab | 178961 | For 2-photon chamber perfusion with aCSF |
| Carbogen 5% CO2/95% O2 | Air Liquide France Industrie | I1501L50R2A001 | |
| Chameleon Ultra2 Ti:sapphire laser | Coherent (Germany) | ||
| disposable transfer pipettes , wide mouth | ThermoFischer scientific | for example : 232-11 | 5.8 mL with fin tip, but we cut it (approx 7 cm) to have a 4 mm diameter mouth |
| emission filter SP680 | Leica Microsystems (Germany) | ||
| fluorescent cube containing a 525/50 emission filter and a 560 dichroic filter (for fluorescence collection) | Leica Microsystems (Germany) | ||
| glass beaker with 50 mL of ACSF to maintain constant perfusion of the slice | |||
| Heating system | Warner Instrument Corporation | Automatic Heater Controller TC-324B | to maintain perfusion solution at 32 °C |
| perfusion chamber | home-made, the file for 3D printing is provided in Supplemental Material | ||
| slice holder ("harp") | home made : hairpin made of platinum with the two branches joined by parallel nylon threads | ||
| Name | Company | Catalog Number | Comments |
| for slice stimulation | |||
| Adenosine 5′-triphosphate disodium salt hydrate (ATP) | Sigma | A-26209 | to be prepared ex-temporaneously : 1 mg/mL (3 mM) stock solution prepared the day of the experiment, kept at 4 °C (a few hours) and diluted just before use |
| Fluorescein (optional) | Sigma | F-6377 | use at 1 µM final |
| Micromanipulator | Luigs and Neumann | SM7 | connected to the micropipette holde |
| Micropipette holder | same as for eletrophysiology | ||
| Serotonin hydrochloride | Sigma | H-9523 | aliquots of 50mM stock solution in H20 kept at -20 °C. 500 µM solution prepared the day of the experiment. |
| Syringe 5 mL (without needle) | Terumo medical products | SS+05S1 | |
| Transparent tubing | Fischer Scientific | 11750105 | Saint Gobain Performance Plastics™ Tygon™ E-3603 Non-DEHP Tubing |
| Name | Company | Catalog Number | Comments |
| for image analysis | |||
| Fiji | https://fiji.sc | Schindelin, J. et al Nat. Methods (2012) doi 10.1038 | |
| Icy | Institut Pasteur | http://icy.bioimageanalysis.org | de Chaumont, F. et al. Nat. Methods (2012) |
| Name | Company | Catalog Number | Comments |
| mice | |||
| CX3CR1-GFP mice | Jung et al, 2000 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. | |
| CX3CR1creER-YFP mice | Parkhurst et al 2013 | male or females, P3 to 2 months-old ; we have backcrossed these mice on 129sv background. |
Références
- Salter, M. W., Stevens, B. Microglia emerge as central players in brain disease. Nature Publishing Group. 23 (9), 1018-1027 (2017).
- Tay, T. L., Savage, J., Hui, C. W., Bisht, K., Tremblay, M. -È Microglia across the lifespan: from origin to function in brain development, plasticity and cognition. The Journal of Physiology. , (2016).
- Davalos, D., et al. ATP mediates rapid microglial response to local brain injury in vivo. Nature Neuroscience. 8 (6), 752-758 (2005).
- Nimmerjahn, A. Resting Microglial Cells Are Highly Dynamic Surveillants of Brain Parenchyma in Vivo. Science. 308 (5726), 1314-1318 (2005).
- Dissing-Olesen, L., et al. Activation of neuronal NMDA receptors triggers transient ATP-mediated microglial process outgrowth. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10511-10527 (2014).
- Gyoneva, S., Traynelis, S. F. Norepinephrine modulates the motility of resting and activated microglia via different adrenergic receptors. Journal of Biological Chemistry. 288 (21), 15291-15302 (2013).
- Eyo, U. B., et al. Neuronal hyperactivity recruits microglial processes via neuronal NMDA receptors and microglial P2Y12 receptors after status epilepticus. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 34 (32), 10528-10540 (2014).
- Hristovska, I., Pascual, O. Deciphering Resting Microglial Morphology and Process Motility from a Synaptic Prospect. Frontiers in Integrative Neuroscience. 9, 1231(2016).
- Avignone, E., Lepleux, M., Angibaud, J., Nägerl, U. V. Altered morphological dynamics of activated microglia after induction of status epilepticus. Journal of Neuroinflammation. 12, 202(2015).
- Abiega, O., et al. Neuronal Hyperactivity Disturbs ATP Microgradients, Impairs Microglial Motility, and Reduces Phagocytic Receptor Expression Triggering Apoptosis/Microglial Phagocytosis Uncoupling. PLoS Biology. 14 (5), e1002466(2016).
- Madry, C., et al. Microglial Ramification, Surveillance, and Interleukin-1β Release Are Regulated by the Two-Pore Domain K+Channel THIK-1. Neuron. 97 (2), 299-312 (2018).
- Honda, S., et al. Extracellular ATP or ADP induce chemotaxis of cultured microglia through Gi/o-coupled P2Y receptors. The Journal of Neuroscience: The Official Journal of the Society for Neuroscience. 21 (6), 1975-1982 (2001).
- Haynes, S. E., et al. The P2Y12 receptor regulates microglial activation by extracellular nucleotides. Nature Neuroscience. 9 (12), 1512-1519 (2006).
- Wu, L. -J., Vadakkan, K. I., Zhuo, M. ATP-induced chemotaxis of microglial processes requires P2Y receptor-activated initiation of outward potassium currents. Glia. 55 (8), 810-821 (2007).
- Kolodziejczak, M., et al. Serotonin Modulates Developmental Microglia via 5-HT 2BReceptors: Potential Implication during Synaptic Refinement of Retinogeniculate Projections. ACS Chemical Neuroscience. 6 (7), 1219-1230 (2015).
- Schafer, D. P., et al. Microglia Sculpt Postnatal Neural Circuits in an Activity and Complement-Dependent Manner. Neuron. 74 (4), 691-705 (2012).
- Pfeiffer, T., Avignone, E., Nägerl, U. V. Induction of hippocampal long-term potentiation increases the morphological dynamics of microglial processes and prolongs their contacts with dendritic spines. Scientific Reports. 6, 32422(2016).
- Parkhurst, C. N., et al. Microglia Promote Learning-Dependent Synapse Formation through Brain-Derived Neurotrophic Factor. Cell. 155 (7), 1596-1609 (2013).
- Wu, Y., Dissing-Olesen, L., Macvicar, B. A., Stevens, B. Microglia: Dynamic Mediators of Synapse Development and Plasticity. Trends in Immunology. 36 (10), 605-613 (2015).
- Ohsawa, K., et al. P2Y12 receptor-mediated integrin-beta1 activation regulates microglial process extension induced by ATP. Glia. 58 (7), 790-801 (2010).
- Kurpius, D., Wilson, N., Fuller, L., Hoffman, A., Dailey, M. E. Early activation, motility, and homing of neonatal microglia to injured neurons does not require protein synthesis. Glia. 54 (1), 58-70 (2006).
- Stence, N., Waite, M., Dailey, M. E. Dynamics of microglial activation: a confocal time-lapse analysis in hippocampal slices. Glia. 33 (3), 256-266 (2001).
- Dissing-Olesen, L., Macvicar, B. A. Fixation and Immunolabeling of Brain Slices: SNAPSHOT Method. Current Protocols in Neuroscience. 71, 1.23.1-12 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- de Chaumont, F., et al. Icy: an open bioimage informatics platform for extended reproducible research. Nature Methods. 9 (7), 690-696 (2012).
- Aitken, P. G., et al. Preparative methods for brain slices: a discussion. Journal of Neuroscience Methods. 59 (1), 139-149 (1995).
- Paris, I., et al. ProMoIJ: A new tool for automatic three-dimensional analysis of microglial process motility. Glia. 66 (4), 828-845 (2018).
- Pagani, F., et al. Defective microglial development in the hippocampus of Cx3cr1 deficient mice. Frontiers in Cellular Neuroscience. 9 (229), 111(2015).
- Ting, J. T., Daigle, T. L., Chen, Q., Feng, G. Acute brain slice methods for adult and aging animals: application of targeted patch clamp analysis and optogenetics. Methods in Molecular Biology. , Clifton, NJ. 221-242 (2014).
- Mainen, Z. F., et al. Two-photon imaging in living brain slices. Methods. 18 (2), San Diego, CA. 231-239 (1999).
- Tanaka, Y., Tanaka, Y., Furuta, T., Yanagawa, Y., Kaneko, T. The effects of cutting solutions on the viability of GABAergic interneurons in cerebral cortical slices of adult mice. Journal of Neuroscience Methods. 171 (1), 118-125 (2008).
- Gyoneva, S., et al. Systemic inflammation regulates microglial responses to tissue damage in vivo. Glia. 62 (8), 1345-1360 (2014).
- Heindl, S., et al. Automated Morphological Analysis of Microglia After Stroke. Frontiers in Cellular Neuroscience. 12, 106(2018).
- Dailey, M. E., Eyo, U., Fuller, L., Hass, J., Kurpius, D. Imaging microglia in brain slices and slice cultures. Cold Spring Harbor Protocols. 12 (12), 1142-1148 (2013).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon