Method Article
Seguimiento de linaje y análisis clonal en el desarrollo de la corteza cerebral mediante el análisis de mosaico con marcadores dobles (MADM)
En este artículo
Resumen
Se presenta un protocolo para realizar el rastreo de linajes y el análisis genético funcional de genes candidatos a un solo nivel de células utilizando el análisis de mosaico con marcadores dobles (MADM). El análisis clonal de MADM proporciona un marco cuantitativo para medir el comportamiento proliferativo, la producción celular y la relación de linaje de los progenitores individuales y sus células hijas.
Resumen
A partir de un conjunto limitado de progenitores, la corteza cerebral de los mamíferos forma circuitos neuronales funcionales altamente organizados. Sin embargo, los mecanismos celulares y moleculares subyacentes que regulan las transiciones de linaje de las células madre neurales (NC) y la producción eventual de neuronas y glia en el neuroepitelio en desarrollo sigue sin estar claro. Los métodos para rastrear patrones de división NSC y mapear el linaje de las células relacionadas clonalmente han avanzado dramáticamente. Sin embargo, muchas técnicas contemporáneas de rastreo de linaje sufren de la falta de resolución celular del destino de las células progenie, que es esencial para descifrar los patrones de división de células progenitoras. Presentado es un protocolo que utiliza el análisis de mosaico con marcadores dobles (MADM) para realizar análisis clonales in vivo. MADM manipula de forma concomitante células progenitoras individuales y visualiza patrones de división precisos y progresión del linaje a una resolución de célula única sin precedentes. Los eventos de recombinación intercromosomal basados en MADM durante la fase G2-X de la mitosis, junto con CreERT2inducible temporalmente, proporcionan información exacta sobre las fechas de nacimiento de los clones y sus patrones de división. Por lo tanto, el rastreo de linaje MADM proporciona lecturas ópticas cualitativas y cuantitativas sin precedentes del modo de proliferación de progenitores de células madre a nivel de célula única. MADM también permite el examen de los mecanismos y requisitos funcionales de los genes candidatos en la progresión del linaje NSC. Este método es único en ese análisis comparativo de control y subclones mutantes se pueden realizar en el mismo entorno tisular in vivo. Aquí, el protocolo se describe en detalle, y se demuestran paradigmas experimentales para emplear MADM para el análisis clonal y el trazado de linaje en el desarrollo de la corteza cerebral. Es importante destacar que este protocolo se puede adaptar para realizar análisis clonales MADM en cualquier nicho de células madre murinas, siempre y cuando el controlador CreERT2 esté presente.
Introducción
La corteza cerebral es una estructura altamente organizada compuesta por seis capas distintas. La corteza contiene una amplia gama de tipos de células, incluyendo neuronas y glia, que interactúan para formar circuitos neuronales funcionales. La mayoría, si no todas, las neuronas de proyección excitatoria cortical y la glia se derivan de una piscina común de células madre neurales (NSC) conocidas como progenitores gliales radiales (BERÁN)1,2,,3. Los CCP se derivan de células madre neuroepiteliales (NEA) que componen el neuropitelio embrionario temprano. Por día embrionario 9 (E9) en ratones, los NESC comienzan a pasar a RGPs4. La progresión del linaje RGP requiere una regulación temporal y espacial precisa, y cuando este proceso se ve obstaculizado, trastornos neurológicos graves como la megalencefalia, la microcefalia, la lissencefalia o alteraciones como la esquizofrenia y el autismo pueden resultarde 5,,6. En E10, la mayoría de los RGPs se someten a divisiones proliferativas simétricas, lo que resulta en una expansión de la piscina de progenitores neuronales4,7. Los PMP eventualmente comienzan a dividirse asimétricamente, produciendo neuronas de proyección cortical de una manera definida temporalmente. A través de ondas consecutivas de neurogénesis, las neuronas recién nacidas migran a la placa cortical formando láminas corticales con neuronas de nacimiento temprano que ocupan capas profundas y neuronas de nacimiento tardío que residen en las capas superficiales8,,9,,10. Debido a que las neuronas piramidales relacionadas clonalmente migran radialmente a la corteza con muy poca dispersión tangencial, las células hijas tienden a formar una columna o estructura en forma de cono conocida como unidad radial neuronal4,11,12,13. Por E17, la expansión neurogénica embrionaria se completa en ratones14. Los SCP también pueden producir células ependimales y algunas clases de glia, incluyendo astrocitos y oligodendrocitos1,15,16,17,18,19. El potencial de los BAN para dar lugar tanto a las neuronas como a los astrocitos parece ser consistente en todas las regiones corticales18,con aproximadamente 1/6 de BAN neurogénicos también produciendo glia11.
Actualmente, los factores genéticos y epigenéticos que regulan la progresión temporal de una célula madre a lo largo de su linaje son en su mayoría desconocidos. Los patrones temporales de expresión génica pueden tener un impacto sustancial en las decisiones de linaje en los RGP20,21,22,23,24. Se desconoce cómo esta relación estrechamente tejida entre el patrón temporal y espacial conduce a la diversidad molecular de los tipos neuronales adultos en áreas corticales. Del mismo modo, cómo el potencial de células madre individuales y su salida celular se modula a nivel celular y molecular es una pregunta importante sin respuesta. Es de esperar que los estudios futuros aborden algunas de estas cuestiones, lo que en última instancia ampliará nuestra comprensión de la formación de circuitos corticales funcionales.
La neurobiología del desarrollo busca entender la relación de linaje que las células del cerebro comparten entre sí. Inicialmente, muy pocas herramientas de investigación estaban disponibles para esto, y muchos estudios tempranos se basaron en observaciones visuales de patrones de división en organismos transparentes como Caenorhabditis elegans25. En las últimas décadas se ha observado un aumento drástico en el número y la sofisticación de las técnicas disponibles13,,26,,27,,28,,29. La aparición del sistema de edición del genoma CRISPR-Cas9 permite la reconstrucción sintética de las relaciones de linaje celular mediante la introducción de códigos de barras de ADNen evolución 27,,30. Dos ejemplos recientes de estrategias de codificación de barras incluyen el uso de ARN guía de localización que dirige CRISPR-Cas9 a loci de código de barras de ADN específico o una deaminasa citidina fusionada con nickase Cas9 para apuntar a las regiones de repetición intercaladas endógenas31,,32. Estas tecnologías proporcionan enfoques altamente multiplexados a través de la introducción de códigos de barras que acumulan progresiva y establemente mutaciones únicas con el tiempo. Los enfoques de edición del genoma son muy valiosos porque permiten un análisis retroactivo de la relación entre dos celdas basadas en la herencia compartida de estos códigos de barras. Sin embargo, para leer los códigos de barras en células individuales, el tejido generalmente debe ser interrumpido, y por lo tanto se pierde información sobre la posición, morfología y números de celda absolutos de un progenitor individual.
Los paradigmas de etiquetado combinatorios preservan la información espacial y, en principio, también permiten la distinción entre clones estrechamente localizados o incluso superpuestos33,,34. Para que un método de trazado de linaje sea informativo, debe etiquetar los progenitores individuales y su progenie de una manera escasa e indeleble. En particular, los enfoques Brainbow35 y Confetti36,37 utilizan reporteros estocásticos multicolores a base de Cre que expresan una combinación de proteínas fluorescentes de un solo locus. El amplio número de combinaciones de colores simultáneas que se pueden lograr in vivo hacen de esta una poderosa herramienta al trazar clones y astrocitos de RGP cortical34. También se han desarrollado sistemas basados en transposon que proporcionan una integración genómica estable de los reporteros fluorescentes de codificación transgenes y permiten el trazado de linajes de progenitores corticales33,,38,,39,,40,,41. Los sistemas basados en Transposon tienen una ventaja adicional en el tiempo que el reportero construye una integración estable en el genoma, y por lo tanto etiqueta de manera confiable las células hijas relacionadas linealmente. Para trazar linajes de astrocitos específicamente, se han desarrollado una serie de métodos que implican electroporación de transposasas piggyBac incluyendo Star Track,que hace uso de una combinación de construcciones que codifican diferentes proteínas fluorescentes40,,42. Otro enfoque, los marcadores MAGIC,introduce los vectores Brainbow como transgenes transponibles. Esto se ha utilizado con éxito para rastrear los progenitores neuronales embrionarios y astrocitos34,43. Recientemente, se encontró que el análisis de mosaico por intercambio dual de casetes mediado por recombinase (MADR) etiquetó de forma estable las células mutantes que expresan elementos transgénicos a partir de loci cromosómico44definido con precisión. Estas potentes técnicas de etiquetado combinatorio in vivo han proporcionado numerosas perspectivas sobre la dinámica del linaje de las células progenitoras. Sin embargo, estos análisis se realizan en tejido fijo, proporcionando una instantánea de clones individuales en una etapa de desarrollo definida. Para observar los cambios en la dinámica de linaje de los clones individuales a lo largo del tiempo, es necesario aplicar45métodos crónicos de diagnóstico por imágenes in vivo similares a los realizados en el giro de dentranato adulto.
El análisis de mosaico con marcadores dobles (MADM) es un potente método de etiquetado de doble color que permite el trazado de linaje in vivo de células progenitoras individuales en ratones46,,47. Dos componentes son necesarios para que se produzcan eventos de etiquetado MADM: En primer lugar, los casetes MADM deben estar dirigidos a locis idénticos en cromosomas homólogos. Los casetes consisten en dos genes reporteros fluorescentes quiméricos, eGFP (verde, [G]) y dimer tándem Tomate (rojo, tdT[T]). El casete GT contiene el N-terminus de eGFP y el C-terminus de tdT, separados por un intrón que contiene un sitio loxP. El casete TG está construido inversamente, con el N-terminus de tdT y el C-terminus de eGFP. En segundo lugar, la expresión de Cre recombinase en la misma célula que contiene los casetes MADM dirigidos es esencial. En ausencia de Cre, los casetes quiméricos no expresan eGFP funcional o tdT porque sus secuencias de codificación se interrumpen. Los sitios loxP sirven como objetivo para la recombinación intercromosomal mediada por Cre, lo que resulta en la reconstitución de ambos casetes de expresión simultáneamente. Si la recombinación ocurre durante la fase G2 del ciclo celular seguida de la segregación X (G2-X), las dos células hijas expresarán cada una de las dos proteínas fluorescentes. La regulación temporal de la actividad de CreERT2 utilizando tamoxifeno (TM) proporciona información precisa sobre la fecha de nacimiento de los clones MADM y los patrones de división de su progenie (Figura 1A)29,46,47.
MADM puede potencialmente etiquetar sistemáticamente clones individuales con alta resolución de una sola célula en el cerebro del ratón similar a los métodos tradicionales pero inespecíficos y laboriosos como la tinción Golgi48 o el relleno de tinte49. Debido a que sólo el promotor que conduce CreERT2 determina la especificidad del tipo de célula del etiquetado MADM clonal, MADM puede en principio aplicarse para el trazado de linaje clonal a través de cualquier órgano y tejido murino47,,50,,51,,52. De hecho, los estudios ya han utilizado MADM para revelar las relaciones de linaje en clones derivados de diversos tejidos47,,50,,51,,52,,53,,54,,55,,56,,57,,58,,59. Se han aplicado paradigmas experimentales MADM para estudiar el linaje en neuronas de proyección cortical, glia y células madre postnatales en el neocórtex en desarrollo7,11,12,46,60,61,62,63,64,65. MADM también se ha utilizado para estudiar el linaje celular en el diurna de dentado adulto, tálamo, células de gránulos cerebelosos e interneurones a nivel clonal (ver Tabla 1 para una lista completa)47,53,54,56,57,66.
Una característica única de MADM es la capacidad de vincular genéticamente mutaciones distales a un casete MADM, creando así un mosaico genético (Figura 1B y Figura 2). Esto da como resultado células hijas de tipo salvaje etiquetadas con un marcador fluorescente (tdT en la Figura 1B)y hermanos mutantes homocigotos con el otro (eGFP en la Figura 1B)en un entorno heterocigoto sin etiquetar. MADM es único en ese análisis comparativo de control y subclones mutantes se pueden realizar en el mismo entorno tisular in vivo. Originalmente, los casetes MADM estaban dirigidos al locus47 rosa26, pero el análisis MADM de la función génica se limitó a genes distales al locus. Para superar (al menos parcialmente) esta limitación y ampliar las posibilidades de los análisis genéticos basados en MADM, los casetes MADM se acercaron a los centromeres de Chr.7 51, Chr. 1146y Chr. 1251. La orientación a los 19 autosomas de ratón con casetes MADM está en curso y permitirá estudiar prácticamente cualquier gen en el futuro, proporcionando una plataforma sin igual para el estudio de las relaciones de linaje de desarrollo en combinación con el análisis genético funcional.
Protocolo
Los protocolos de ratón fueron revisados por la instalación básica preclínica institucional (PCF) y el comité ético interno de IST Austria. Toda la cría y experimentación se realizaron bajo una licencia aprobada por el Ministerio Federal de Ciencia e Investigación de Austria de conformidad con las leyes austriacas y de la UE en animales.
1. Crianza de ratones experimentales para el análisis clonal de MADM
- Configurar apareamientos MADM experimentales cronometrando (>P56; CD-1) a última hora de la tarde (5:00 PM) y comprobar si hay enchufes vaginales por la mañana (8:00 AM). La mañana en que el enchufe está presente cuenta como día 0.5. Consulte la figura 2 para obtener información general sobre la configuración experimental de acoplamiento del ratón. Asegúrese de que los puntos de tiempo para la inducción TM de la actividad y el análisis de CreERT2 sean adecuados para abordar las preguntas experimentales.
NOTA: Para obtener más información, consulte la Figura 3 y los resultados representativos a continuación. - Para el muestreo postnatal, establecer crías para generar madres adoptivas en paralelo.
NOTA: Estos deben iniciarse hasta 1 o 2 días antes de la creación de crías experimentales.
2. Inducción TM en ratones MADM
- Preparar una solución de trabajo de 20 mg/ml TM disolviéndola en aceite de maíz en un tubo de centrífuga cónica de 15 ml o 50 ml y colocándola en una plataforma de balanceo durante 4 horas a temperatura ambiente (RT), asegurando que TM se disuelva por completo. Almacene la solución de trabajo a 4 oC cubierta con papel de aluminio y su uso en un plazo de 2 semanas.
- Para inducir eventos de recombinación MADM, entregue una sola inyección de TM por vía intraperitoneal (IP) utilizando una jeringa de tuberculina de 1 ml y una aguja de 25 G en una presa embarazada cronometrada. Dependiendo de la etapa de la neurogénesis cortical, inyectar TM entre E10-E15 a una dosis de 1 x 2 mg/ presa embarazada. Para los primeros puntos de tiempo (es decir, E10) utilice un máximo de 1 mg/presa embarazada (25 mg/kg) para prevenir complicaciones durante el embarazo11. Para puntos de tiempo entre E11-E15 utilice 2 mg/presa embarazada (50 mg/kg)7.
NOTA: Alternativamente, TM se puede administrar con un gavage oral para embarazos tardíos. - Para el análisis clonal de MADM a los puntos de tiempo postnatales, recuperar embriones vivos en E18-E19 a través de la cesárea, y luego criar cachorros con una madre adoptiva.
NOTA: Dependiendo del estado de salud de la mujer embarazada, puede que no sea necesario realizar una cesárea, pero la crianza de los cachorros con una madre adoptiva todavía es necesaria porque la madre tratada con TM original puede tener problemas para lactación. - Para recuperar embriones vivos por cesárea o para recuperar puntos de tiempo embrionarios para su análisis, sacrifique la presa embarazada por luxación cervical.
- Coloque el animal en posición supina y desinfecte el pelaje con un 70% de etanol. Haga una pequeña incisión en la piel en la parte inferior del abdomen por encima del útero utilizando fórceps quirúrgicos y tijeras. Haga una segunda incisión a través de los músculos y la pared muscular abdominal para revelar el peritoneo.
- Retire el útero separándose de los tejidos circundantes con tijeras. Transfiera el útero intacto a un guante lleno de agua tibia para aumentar la tasa de supervivencia del embrión hasta que cada uno se retire del amnion individualmente.
- Utilice tijeras y dedos con punta fina para abrir cuidadosamente las paredes uterinas para liberar embriones. No corte los cordones umbilicales demasiado cerca del cuerpo para evitar la pérdida extensa de sangre. Si los embriones se van a utilizar para el análisis, proceda al paso 3.9. Si se quiere que se aficionen a los cachorros, proceda al paso 2.8.
- Si se requiere el acogimiento, limpie a los cachorros antes de transferirlos a la madre adoptiva. Mientras limpia a los cachorros, presione suavemente el pecho de vez en cuando para iniciar la respiración. Coloque de nuevo en un segundo guante lleno de agua tibia para mejorar la tasa de supervivencia.
NOTA: Es importante eliminar suavemente cualquier amnion y/o placenta restante con una toalla de papel. - Antes de transferir cachorros a la madre adoptiva, retire a la madre adoptiva de su jaula, retire a los cachorros originales y sustitúyalos por los cachorros experimentales. Devuelva a la madre adoptiva a su jaula.
NOTA: Consulte la discusión para obtener sugerencias adicionales para mejorar el fomento de las tasas de aceptación. - Si se requiere genotipado, recoja las biopsias de dedo del dedo del dedo del dedo del de la parte del de la parte del otro o de la cola entre P6-P8.
NOTA: Realice este paso solo si las licencias experimentales de animales aprueban esta práctica.
3. Preparación de tejido para clones MADM en el cerebro
NOTA: Para experimentos que incluyan tejido posnatal (P4), continúe con el paso 3.1. Para los puntos de tiempo embrionarios y el postnatal temprano (P0-P3), continúe con el paso 3.9.
- Anestesiar el animal EXPERIMENTAL MADM con una inyección IP de una solución de ketamina/xilazina/acepromazina (65 mg, 13 mg y 2 mg/kg de peso corporal, respectivamente) y confirmar que el ratón no responde pellizcando la pata trasera.
NOTA: Tanto los ratones MADM masculinos como los hembras (fondo CD-1) se utilizan para el análisis. Si se requiere genotipado, recoja las biopsias de oído en este punto. - Coloque el animal anestesiado en la posición supina en la bandeja de cirugía de perfusión y desinfecte el pelaje con un 70% de etanol. Para comenzar la cirugía, haga cuidadosamente una incisión con tijeras y fórceps quirúrgicos a través de la capa externa de la piel y luego una segunda incisión a través de la capa muscular. Levante la punta del esternón y corte el tejido conectivo en los lados, teniendo especial cuidado para evitar cortar el hígado. La cavidad torácica será visible.
- Cortar el diafragma y levantar para revelar el corazón. Recorte cuidadosamente la caja torácica y colúncarla a la bandeja quirúrgica para exponer el corazón. Para los cachorros, retire la caja torácica por completo.
- Inserte una aguja con solución salina tamponada de fosfato (PBS) en el ventrículo inferior izquierdo (tejido más pálido). El uso de tijeras de iris pequeñas hace una incisión en el extremo posterior de la aurícula derecha (tejido rojo oscuro) para que la sangre drene.
- Realizar perfusión con PBS seguido inmediatamente de recién hecho, frío enfriado 4% paraformaldehído (PFA) preparado en PBS. Para los cachorros (P4-P10) utilice jeringas para realizar la perfusión. Llene una jeringa con 10 ml de PBS y otra con 10 ml de 4% de PFA. Asegúrese de que se hayan eliminado todas las burbujas de aire de las jeringas. Para animales mayores, utilice una bomba peristáltica.
- Comenzar a perfunder con PBS (10 ml a 2 x 4 ml/min en cachorros; 20 ml a 4 x 6 ml/min para adultos que utilizan una bomba peristáltica). El hígado se volverá de color amarillo claro y pálido si la aguja se coloca correctamente.
- Una vez completada, retire la aguja de los cachorros e inserte la aguja que contiene PFA en el mismo orificio. Para los adultos, detenga la bomba peristáltica antes de intercambiar la solución PBS con PFA helada, asegurándose de evitar burbujas en el tubo de absorción. Reanudar la perfusión con PFA (10 ml a 2 x 4 ml/min en cachorros; 30 ml a 4 x 6 ml/min para adultos que utilizan una bomba peristáltica).
- Cuando la perfusión está completa, decapitar el ratón y extraer el cerebro a través de una disección cuidadosa. Transfiera el cerebro al 4% de PFA. Utilice al menos 5 veces los volúmenes cerebrales (es decir, 5 x 10 ml de PFA en un tubo de centrífuga cónica de 15 ml) e incubar durante la noche a 4 oC para la fijación posterior a la perfusión para asegurar la fijación completa del tejido. Continúe con el paso 3.10.
- Para el tejido embrionario y el tejido postnatal temprano (es decir, P0-P3), después de realizar una sección cesárea, decapitar los embriones con tijeras. Si se requiere genotipado, recoja la cola del embrión en este punto. Disecciona inmediatamente el cerebro y transfiera a una placa de 12 pozos que contenga 2 x 3 ml de 4% PFA/pozo. Incubar durante la noche a 4oC para la postfijación.
- A la mañana siguiente, intercambie PFA con 10 ml (adulto) o 2 x 3 ml (embrión) de PBS y repita el lavado 3x durante 15 min a RT. Transfiera el tejido a una solución de sacarosa al 30% en tampón de fosfato (PB) y guárdelo a 4 oC en una plataforma de balanceo hasta que el tejido se hunda en la solución.
- Incrustar el cerebro en la temperatura de corte óptima (OCT) compuesto en un molde de incrustación, teniendo cuidado de orientar el cerebro para la sección coronal o sagital. Congele colocando el molde de incrustación sobre hielo seco hasta que el PTU se vuelva completamente opaco (10-15 min). Almacene el tejido a -80 oC hasta su uso posterior.
4. Preparación del tejido MADM para inmunohistoquímica
- Fije el bloque de tejido al disco de la muestra en el criostato aplicando un anillo de OCT al disco y colocando el bloque directamente en el OCT cuando comience a congelarse. Asegúrese de que el bloque esté correctamente orientado para el plano de corte deseado.
NOTA: Aquí se describe en detalle el apartado coronal para investigar clones CORticales MADM. - Ajuste la temperatura del bloque en el criostato a -20 oC y la temperatura de la hoja a -21 oC.
- Deje que el bloque de tejido se ajuste a la temperatura de la cámara montando el disco de la muestra en el soporte de la muestra y déjelo en criostato durante 5 minutos antes de comenzar la sección.
- Recortar el bloque en secciones gruesas (45-60 m) hasta que se alcance la región del tejido de interés.
- Una vez que el borde de la corteza es claramente visible, deje de secciones y bloquee la hoja. Asegúrese de que la hoja esté protegida antes de recortar el bloque.
- Recortar el exceso de OCT que rodea el tejido con una cuchilla, dejando 1 x 2 mm de OCT en todos los lados del cerebro.
- A continuación, oriente el bloque de modo que uno de los bordes laterales de la corteza esté orientado hacia abajo y el otro hacia arriba (es decir, el borde más rostral de la corteza es puntiagudo a la derecha).
- Comience a secciones con un espesor de 45 m para clones adultos y 30 m para clones embrionarios. Realice cada sección individualmente y utilice un pequeño cepillo para mantener el área debajo del cuchillo limpia de los restos que queden mientras recorta el bloque.
NOTA: Si esto no se hace y una sección cae, puede ser difícil determinar el orden correcto de los sectores. - Si las secciones comienzan a curvarse, recorte los bordes del bloque y/o ajuste cuidadosamente la placa antidesplaza de vidrio.
- Para el análisis de clones embrionarios, monte secciones directamente en una diapositiva esmerilada. Secar en una placa calefactora a 37oC antes de proceder directamente al paso 5.6.
NOTA: Se pueden agregar varias secciones a una diapositiva, pero asegúrese de que se mantiene el orden secuencial. - Para recoger clones adultos, prepare 24 placas de pozo que contengan 1 ml de PBS/pozo (normalmente, 5 x 6 placas por cerebro). A partir del primer pozo, con fórceps fríos recoge secciones seriales individuales en PBS en el orden de seccionamiento.
NOTA: El método de sección flotante se adopta para el tejido adulto para asegurarse de que no se pierda ninguna secciones y que las secciones montadas no contienen arrugas. - Deje de secciones una vez que se alcance el final del neocórtex.
- Para clones adultos, proceda al montaje de secciones flotantes.
NOTA: Las secciones se pueden mantener en PBS a 4 oC durante un máximo de 24 h.
5. Montaje de tejido adulto para imágenes
NOTA: Se requieren las siguientes herramientas: cepillo de pintura pequeño, plato Petri, PBS con 0,5% de interpolación (PBS-T), diapositivas de adhesión (Tabla de materiales),medio de montaje(Tabla de materiales),cubreobjetos y fórceps.
- Llene un plato de Petri con PBS-T.
NOTA: El detergente se utiliza para ayudar en el proceso de montaje. Si es necesario manchas para antígenos adicionales sensibles a detergentes (es decir, glicoproteínas), lo mejor es omitir la adición de Tween. - Coloque una diapositiva de adhesión en el PBS-T para que esté casi cubierta hasta la etiqueta.
- Transfiera la primera sección al PBS-T.
- Usando un pequeño pincel, maniobra la sección en el tobogán y organízala para preservar el orden de corte. Proceda de la misma manera con todas las secciones posteriores.
- Una vez que todas las secciones estén en posición, coloque la diapositiva (12 x 16 secciones /deslizamiento) en una cámara deslizante oscura. Levante ligeramente la tapa para permitir que las secciones se sequen por completo (10-20 min), asegurándose de que permanezcan adheridas en pasos posteriores.
- Si realiza inmunohistoquímica para antígenos adicionales, proceda directamente a la sección 6 o 7.
NOTA: Para los puntos de tiempo embrionarios, es necesario realizar pasos de inmunomanchas para al menos GFP y tdT (sección 6). Para clones adultos, esto sólo es necesario si la tinción para antígenos adicionales en paralelo (secciones 6 y 7). - Rehidratar y lavar secciones 1x con 1x PBS durante 5 min para eliminar el PBS-T residual. Aplicar 1 ml de 4o,6-diamidino-2-fenilindole (DAPI) diluido en 1x PBS (1 g/ml) en el portaobjetos, asegurando que todas las secciones estén cubiertas e incubar durante 15 min.
- Retire con cuidado el DAPI y lave 1x con 1 pbS durante 5 min. Retire el exceso de PBS y séquelo durante 1 x 2 minutos antes de incrustarlo en 110 oL del medio de montaje. Selle con un cubreobjetos de 24 x 60 mm y déjela secar durante al menos 3 horas antes de tomar imágenes.
6. Inmunostaining para GFP y tdT solamente
NOTA: Esta sección es necesaria para clones embrionarios.
- Coloque los portaobjetos horizontalmente en una cámara de incubación de diapositivas humidificadas. Marque los límites de la diapositiva con un marcador de cera para minimizar la cantidad de búfer necesario.
- Rehidratar secciones con 1x PBS. Para mejorar la calidad de la tinción, trabaje con tejido recién seccionado.
- Añadir 250 a 400 ml de tampón de bloqueo (0,5% Tritón X-100, 2 x 3% suero de burro normal en 1x PBS) por diapositiva, asegurando que todas las secciones estén cubiertas. Incubar durante 1 h.
NOTA: La concentración de detergente (Triton X-100 o Tween-20) variará en función de los anticuerpos primarios adicionales utilizados porque algunos antígenos son más sensibles a los detergentes que otros. - Elimine el búfer de bloqueo y añada anticuerpos primarios en el búfer de bloqueo a la diapositiva (300-400 l/deslizamiento).
NOTA: Un ejemplo de una reacción de anticuerpos primarios estándar para anti-GFP/anti-tdT (MADM) podría utilizar pollo anti-GFP (1:500) y anti-RFP de conejo (1:500). - Incubar con anticuerpos primarios durante la noche a 4oC.
NOTA: Los portaobjetos deben incubarse perfectamente horizontalmente con tampón que cubra todas las secciones. De lo contrario, pueden producirse manchas desiguales o deficientes. - Confirme a la mañana siguiente que el tampón de bloqueo con anticuerpos primarios todavía cubre todas las secciones de la diapositiva. Si no es así, repita el paso de incubación durante 3 x 4 h en RT.
- Retire los anticuerpos primarios y lave 4x con 1 PBS durante 10 minutos a RT.
- Añadir anticuerpos secundarios diluidos en el tampón de bloqueo para deslizar (300-400 l/deslizamiento): Alexa Fluor 488 anti-pollo IgG (1:500) y Cy3 anti-conejo IgG (1:500).
- Incubar en RT durante 2 h. Mantenga los portaobjetos cubiertos de luz para evitar el blanqueo de fluoróforos.
- Retire los anticuerpos secundarios y lave 2x con 1x PBS durante 10 min.
- Incubar con DAPI diluido en PBS (1:5.000) durante 15 min.
- Lavar las secciones 1x con 1x PBS durante 10 min.
- Retire el exceso de PBS y seque durante 1 x 2 min antes de incrustarlo en 110 ml del medio de montaje.
- Selle con un cubreobjetos de 24 x 60 mm y déjela secar durante al menos 3 horas antes de tomar imágenes. Diapositivas de imagen dentro de 1 a 2 semanas después de realizar inmunohistoquímica para garantizar una señal óptima.
7. Inmunostaining para GFP, tdT y antígenos adicionales
- Realice los pasos 6.1 a 6.3.
- Elimine el búfer de bloqueo y añada anticuerpos primarios en el búfer de bloqueo a la diapositiva (300-400 l/deslizamiento).
NOTA: Al tinción de tres o más antígenos (es decir, GFP, tdT y una proteína de interés) y el anticuerpo para la proteína de interés se levantó en conejo, se recomienda utilizar el anticuerpo primario anti-tdT (cabra) en una dilución de 1:500. Un ejemplo de reacción primaria de anticuerpos para tres antígenos con tinción alternativa de tdT podría utilizar pollo anti-GFP (1:500), anti-tdT de cabra (1:500) y anticuerpos contra la proteína de interés (es decir, conejo). - Realice los pasos 6.5 a 6.7.
- Añadir una mezcla de anticuerpos secundaria diluida en el tampón de bloqueo para deslizar (300-400 l/deslizamiento): Alexa Fluor 488 anti-pollo IgG (1:500), Cy3 anti-cabra IgG (1:500), y Alexa Fluor 647 anti-conejo IgG (1:500).
- Realice los pasos 6.9 a 6.14.
8. Adquisición y cuantificación de imágenes confocales de clones MADM
- Identificar y documentar secciones cerebrales que contienen clones y sus ubicaciones en la corteza.
NOTA: El número de secciones de un clon abarca variará dependiendo de cuándo se indujo el clon, el controlador CreERT2 y la hora del análisis. Este paso se puede realizar en un microscopio confocal o en un microscopio de epifluorescencia. - Con un microscopio confocal invertido, comience seleccionando y configurando las líneas y filtros láser correctos. Para cerebros MADM, seleccione DAPI, GFP y tdT (excitación: 358 nm, 488 nm y 554 nm, respectivamente; emisión máxima: 461 nm, 507 nm y 581 nm, respectivamente). Asegúrese de que el agujero esté ajustado en 1 unidad ventilar para una calidad de imagen óptima.
- Para ajustes específicos confocales, los clones de imagen con un objetivo de 20x y un zoom 1x. Para las imágenes que se utilizarán en las cuantificaciones, utilice un valor de permanencia de píxeles de velocidad de escaneo de 1,52 a 2,06 s (valores de 7 a 8 en el software de adquisición de imágenes) sin promediar. Ajuste la intensidad del láser y los ajustes de ganancia para cada canal según corresponda.
NOTA: Dependiendo de la calidad de imagen requerida, los ajustes para la velocidad de escaneo y el promedio pueden variar. - Una vez que el clon esté claramente identificado, organice las fichas de imagen para cubrir todas las secciones relevantes del clon. Ajuste la pila z de modo que todas las celdas etiquetadas por MADM en el clon se capturen con un intervalo de 1,5 m/z-pila de sectores. Ajuste la región en mosaico para que se capture todo el ancho de la corteza al crear imágenes del clon (es decir, desde la superficie pial hasta el cuerpo calloso).
- Imagen de clones individuales que abarcan varias secciones consecutivamente, asegurando que cualquier sección sin celdas dentro de un clon todavía se imagine con el propósito de reconstrucción 3D e interpretación correcta de la información espacial celular.
- Analice cada sección que contiene las células de un clon MADM secuencialmente desde el extremo rostral hasta el extremo caudal de la corteza. Distinguir las neuronas individuales y la glia en función de su morfología y/o tinción de marcadores. Registre la información posicional en paralelo en función de los límites de capa respectivos definidos por la tinción nuclear (DAPI).
NOTA: Consulte la Figura 4 para obtener resultados representativos para el análisis embrionario y la Figura 5 para obtener resultados representativos para el análisis de adultos.
9. Reconstrucción 3D serie de clones
NOTA: La reconstrucción 3D de clones individuales imágenes sobre secciones cerebrales seriales es útil para la visualización visual, así como para el análisis de arquitecturas clonales 3D y se puede realizar de acuerdo con los siguientes pasos.
- Coser y fusionar imágenes en mosaico confocales basadas en parámetros de adquisición utilizando software de adquisición de imágenes. Abra el archivo .czi y, a continuación, ejecute el método Stitching en la pestaña Processing del software ZEN (Zeiss).
- Exporte pilas de imágenes cosidas como planos z individuales en formato TIFF. Abra el archivo .czi cosido y, a continuación, ejecute el método de exportación de imágenes en la pestaña Procesamiento. Para imágenes multicanal, exporte como imágenes rojas/verdes/azules para el procesamiento posterior de imágenes.
- Repita los pasos 9.1 y 9.2 para cada sección de cerebro serie de un clon.
NOTA: Para una reconstrucción 3D precisa, también deben procesarse todas las secciones cerebrales dentro de un clon, incluidas las que no tienen células etiquetadas. - Concatenar imágenes individuales en una sola pila para empezar desde el plano z más rostral al más caudal utilizando software de procesamiento de imágenes de código abierto como ImageJ/Fiji67,68.
NOTA: Cualquier imagen en blanco en los bordes de cada sección del cerebro debe eliminarse en este punto. - Si es necesario, corrija la pila de imágenes obtenida del paso 9.4 para la desalineación utilizando un complemento ImageJ llamado "MultiStackReg" siguiendo los pasos 9.5.1 a 9.5.5. Si no se requiere alineación de imagen, continúe con el paso 9.6.
NOTA: Este plugin realiza la alineación de imágenes del canal con el mayor contraste (normalmente DAPI) y luego aplica la transformación grabada a los otros canales, lo que permite una alineación de imagen confiable de pilas multicanal. Un plugin auxiliar llamado "TurboReg" debe estar preinstalado.- En ImageJ, instale los plugins "MultiStackReg" y "TurboReg".
- Abra la pila de imágenes de imágenes clonadas obtenidas del paso 9.4 para alinearse. Divida los canales en DAPI (azul), GFP (verde) y tdT (rojo) en la opción Color en la pestaña Imagen.
- Ejecute el plugin "MultiStackReg" para alinear el canal DAPI mediante la transformación "Cuerpo rígido"y guarde el archivo de transformación.
- Aplique el archivo de transformación guardado a los otros dos canales utilizando "MultiStackReg".
- Combine los tres canales alineados y guarde la pila alineada.
- Para orientar el clon en ImageJ gire la pila de imágenes clonadas obtenidas del paso 9.4 (o paso 9.5.5 después de la alineación) en una orientación vertical con la superficie pial en la parte superior y el cuerpo calloso en la parte inferior. Recorte en el plano xy si es necesario.
- Tanto para la presentación cualitativa como para el análisis cuantitativo, genere una imagen de proyección z máxima (paso 9.8) o realice renderizado 3D (paso 9.9) del clon.
- En ImageJ, abra la pila de imágenes del paso 9.6 y seleccione la opción de proyección Z con el tipo de proyección Intensidad máxima. Esto generará una imagen de todo el clon proyectado en el mismo plano.
- En ImageJ, abra la pila de imágenes del paso 9.6 y seleccione la función z del proyecto 3D para generar una visualización 3D del clon que se puede girar.
NOTA: En este paso es importante introducir el intervalo de sectores correcto equivalente al grosor de las pilas z individuales durante la adquisición de imágenes. La herramienta de interpolación debe utilizarse para eliminar huecos entre sectores.
Resultados
MADM da como resultado la reconstitución de proteínas fluorescentes verdes y rojas funcionales con dos células hijas cada una expresando una de las dos proteínas fluorescentes sobre eventos de segregación cromosómica G2-X (Figura 1A). Debido a que los eventos MADM dan lugar a un etiquetado permanente y distinto de los dos linajes descendientes, se puede realizar una evaluación cuantificable de los linajes celulares hijas verdes y rojos (subclones). Se pueden determinar las variables que incluyen el patrón de división (por ejemplo, simétrico frente a asimétrico) y el potencial (por ejemplo, el número de progenie) del progenitor original. La cuantificación de cada subclón con etiqueta fluorescente es informativa cuando se determina retroactivamente si la célula progenitora original está siendo sometida a divisiones proliferativas simétricas, o divisiones neurogénicas asimétricas en el momento de la inducción de TM. Estudios previos agruparon clones de proyección excitatoria derivados de Emx1-CreERT2 o Nestin-CreERT2 en la corteza en dos clases amplias7,11,46. Los primeros, llamados "clones proliferativos simétricos", se componen en promedio de un número considerable de neuronas, con subclones verdes y rojos que contienen cuatro o más neuronas cada una. El segundo grupo, "clones asimétricos" define una clase de clones donde la subclone "minoría" contiene menos de tres neuronas y la subclone "mayoría", cuatro o más11. Estas definiciones son específicas de los CP corticales y pueden necesitar ser revisadas para otras regiones y tejidos cerebrales. Para ambas clases de clones corticales, la progenie se distribuirá a través de las capas superficiales y profundas.
Al diseñar estudios clonales MADM hay una serie de aspectos que deben tenerse en cuenta. El momento en que los eventos MADM son inducidos por la administración de TM es una consideración clave (Figura 3). Para clones MADM de neuronas de proyección excitatoria cortical (es decir, usando Emx1-CreERT2 o Nestin-CreERT2)en E10, casi todos los RG seguían siendo sometidos a divisiones simétricas11. Por lo tanto, la inducción en E10 con TM capturó múltiples rondas de amplificación de RGP proliferativa y resultó en clones con altos números de neuronas. Sin embargo, el número de RGPs en E10 era generalmente pequeño y por lo tanto la administración de TM generó muy pocos eventos MADM (a veces menos de uno por cerebro). La mayoría de los RGPs cambiaron de divisiones neurogénicas simétricas a asimétricas alrededor de E12. Para atacar clones neurogénicos estrictamente asimétricos, lo mejor era inducir en E12 o posterior (Figura 3). El tiempo entre la inducción de TM y la observación de eventos de recombinación MADM en la corteza tendió a ser inferior a 24 h. Las inyecciones de IP fueron el método preferido para administrar TM en etapas embrionarias para este método porque condujo a una mayor reproducibilidad en la inducción clonal. También es importante mantener la dosis TM al mínimo por dos razones. En primer lugar, si aumenta la tasa de recombinación MADM, la probabilidad de inducir múltiples, tal vez superpuestos, clones es mayor. En segundo lugar, si se administra demasiada TM, se puede observar una mayor tasa de aborto, reabsorción de embriones y tamaños de camada más pequeños. Se observaron abortos en aproximadamente la mitad de todas las presas embarazadas cuando se administraron inyecciones de TM en E10. Esta frecuencia disminuyó a partir de E11 y disminuyó a aproximadamente 1/3 de las presas embarazadas abortando. Para obtener un resumen de las dosis de TM, los tiempos de inducción y los controladores CreERT2 utilizados en estudios anteriores de MADM, consulte la Tabla 1. La actividad del reportero en ausencia de TM se observó con algunos conductores de CreERT2 inducibles por TM69. No se observó expresión ectópica o eventos de recombinación MADM en ausencia de TM con los controladores Emx1-CreERT2 de Nestin-CreERT2. Esto puede deberse en parte al hecho de que las transcombinaciones cromosómicas mediadas por TM se producen aproximadamente en 1:1,000 a 1:10,000 una frecuencia más baja que las recombinaciones cis, lo que reduce la probabilidad de etiquetado ECTmático de MADM.
Otro factor a tener en cuenta al planificar un experimento de análisis clonal de MADM es la duración del estudio. Al variar el tiempo entre la inducción TM y cuando se analizó el experimento (A) (ventana de tiempo) muestra la dinámica de células madre a lo largo del tiempo64. Ventanas de tiempo embrionarias cortas (es decir, TM/E11-A/E13; TM/E11-A/E16) capturó la dinámica de la neurogénesis embrionaria (Figura 4). La comparación de clones a partir de dos o más ventanas de tiempo proporciona información cuantitativa sobre el número de células producidas y cómo la distribución de las neuronas varía en diferentes etapas de la progresión del linaje64. Para capturar todo el potencial de los clones individuales, es necesario ampliar la ventana de tiempo analizada en los puntos de tiempo posnatales o adultos7,,11,,12. Ejemplos de clones neocorticales inducidos en el embrión y analizados en el adulto se muestran en la Figura 5. Cabe destacar que la neurogénesis cortical se completa principalmente y la gliogénesis aumenta por E17. Aproximadamente 1/6 RGP neurogénico también proceden a generar astrocitos y/u oligodendrocitos11.
Los clones simétricos se producen cuando los RGP se someten a una o más rondas de la división proliferativa11. Los clones de RGP inducidos entre E10-E12 eran en promedio de mayor tamaño y proporcionaban más características espaciales de la distribución final de la neurona(Figura 4A-C). Los clones con neuronas relativamente distribuidas por igual a través de capas profundas y superficiales tomaron una forma de "cilindro", mientras que los clones con neuronas más dispersas en capas superficiales que las capas más profundas desarrollaron una forma de "cono"11. Para capturar completamente la información espacial y morfológica de un clon, era necesario reconstruir computacionalmente cada clon utilizando imágenes secuenciales. Para medir la dispersión clonal, la dispersión lateral máxima (medida en todas las dimensiones) en capas superficiales (LII-VI) de un clon se comparó con la dispersión de neuronas en capas profundas (LV/LIV). Esta relación (distribución superior:distribución inferior) proporcionaba una lectura cuantificable de la forma general del clon.
Los clones asimétricos, donde el subclón minoritario era tres o menos, proporcionaron información sobre la salida neuronal de un solo RGP (Figura 4D-F y Figura 5A-F)7,11,12. La población mayoritaria (subclón grande) podría etiquetarse en rojo o verde, con un promedio de aproximadamente siete neuronas de proyección excitatoria por clon cuando se induce utilizando un Emx1-CreERT2 o Nestin-CreERT2(Figura 5G)7,11,12. El número total de células en un clon de MADM podría diseccionarse aún más mediante el análisis de la distribución de las neuronas en el subclón grande a través de capas superficiales y profundas. La población minoritaria (subclón pequeño) fue etiquetada por el color recíproco y era en promedio de 1 a 2 células por clon (Figura 5H). El "tamaño de la unidad" total, que era en promedio de 8 a 9 neuronas, se podía calcular añadiendo los subclones pequeños y grandes juntos (Figura 5I)7,11,12. Es importante tener en cuenta que mientras que la salida neuronal de los RGPs era altamente predecible, hubo un grado de heterogeneidad clonal12,,70.
La introducción de una mutación distal en el casete MADM permite la generación de mosaicos genéticos, proporcionando un método único para diseccionar los reguladores moleculares de la progresión del linaje de células madre. Como tal, MADM proporciona una plataforma experimental sin igual para estudiar la función autónoma celular de un gen (por ejemplo, su asociación a la microcefalia o macrocefalia). Al comparar clones inducidos en un mosaico genético MADM con clones inducidos en un control MADM, se puede generar una lectura altamente cuantitativa de los cambios en el número de neuronas y la distribución. Estudios anteriores basados en MADM cuantifican la función celular autónoma de Otx1 en la formación de microcefalia a nivel clonal (véase la Figura 6A-E para un ejemplo representativo)11. En otro estudio, el análisis clonal de MADM demostró que Ndel1 no regula de forma autónoma el número de neuronas de proyección, sino la capacidad de las neuronas recién nacidas para entrar o migrar dentro de la placa cortical, que más tarde forma la cortezaadulta 46. Estos estudios demostraron el carácter altamente cuantitativo del análisis clonal del MADM en el estudio de las funciones autónomas celulares de los genes que regulan el desarrollo cortical. Actualmente no hay ejemplos en la literatura utilizando MADM para estudiar genes implicados en la macrocefalia a nivel clonal. Sin embargo, en estudios futuros el análisis de genes relevantes para el control del tamaño cortical en general puede proporcionar información altamente deseable a nivel molecular y celular.

Figura 1: El principio MADM para el trazado de linajes y el análisis clonal a nivel de células madre únicas. (A) Para realizar el seguimiento de linaje y el análisis clonal con MADM, deben estar presentes dos componentes. En primer lugar, los casetes MADM deben estar dirigidos a locis idénticos en cromosomas homólogos. Los casetes consisten en dos genes reporteros fluorescentes quiméricos, eGFP (verde, [G]) y dimer tándem Tomate (rojo, tdT[T]). El casete GT contiene el N-terminus de eGFP y el C-terminus de tdT, separados por un intrón que contiene un sitio loxP. El casete TG está construido inversamente, con el N-terminus de tdT y el C-terminus de eGFP. En segundo lugar, la expresión de Cre recombinase debe producirse en la celda que contiene los casetes MADM de destino. Los sitios loxP sirven como objetivo para la recombinación intercromosomal mediada por Cre, lo que resulta en la reconstitución de ambos casetes de expresión simultáneamente. Si la recombinación ocurre durante la fase G2 del ciclo celular seguida de la segregación X (G2-X), las dos células hijas expresarán una de las dos proteínas fluorescentes. (B) Principio MADM para el análisis de mosaico genético a un solo nivel de clon. Los alelos mutantes (mutaciones puntuales, eliminaciones, inserciones, alelos condicionales con flanco loxP como se muestra en la Figura 1B,etc.) se pueden introducir distales en el casete TG-MADM a través de la recombinación meótica (ver Figura 2 y Hippenmeyer et al.46 para obtener más información sobre cómo introducir alelos mutantes en el sistema MADM). Si se produce una recombinación transcronómica mediada por G2-X Cre entre casetes MADM, da como resultado una célula mutante homocigótica GFP+ (GeneX-/-) para el gen de interés y una célula de tipo salvaje homocigótica tdT+ (GeneX+/+) en un entorno heterocigoto sin etiqueta46,47,71. Los resultados de etiquetado alternativos no utilizados en el análisis clonal (es decir, las células amarillas) se han descrito previamente en detalle11,46,47. Haga clic aquí para ver una versión más grande de esta figura.
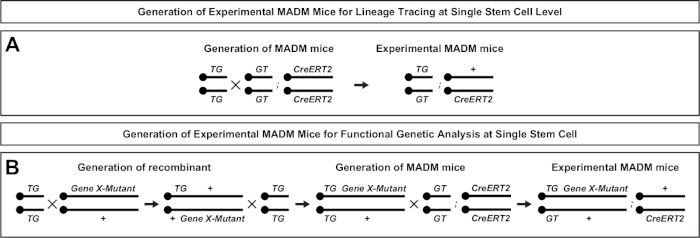
Figura 2: Esquemas de cría para la generación de ratones MADM experimentales para el trazado de linajes. Esquema de cría para la generación de ratones MADM(A)y Gene X MADM (B) experimentales MADM para análisis clonal. Para obtener más información sobre los paradigmas de cría MADM, véase Beattie et al.7 y Hippenmeyer et al.7,46. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Paradigmas del curso de tiempo para el análisis de linaje clonal basado en MADM. Esquema de las ventanas de tiempo de diseño experimental. Para los paradigmas de muestreo longitudinal, el punto de tiempo de la inducción del clon se mantuvo constante y el tiempo antes del análisis varió. En el muestreo de intervalos progresivos, el punto de tiempo del análisis se mantuvo constante, pero el tiempo de inducción varió. Se puede utilizar una combinación de uno o ambos enfoques en función de las preguntas abordadas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Análisis clonal del MADM en el neocórtex en desarrollo y adulto. Inducción de clones MADM mediados por TM en RGPs divisorias divisorias simétricamente proliferativas (TM en E10)(A-C)y neurogénicas asimétricamente neurogénicas (TM en E12) (D-F). Se representan clones MADM individuales in vivo en el desarrollo (TM/E10-A/E16 y TM/E12-A/E16) (B,E) y adultos (TM/E10-A/P21 y TM/E12-A/P21) (C,F) en MADM-11GT/TG; Nestin-CreERT2+/- (B,E) y MADM-11GT/TG; Emx1-CreERT2+/- (C,F). La producción de neuronas era independiente del color de la subclone y los subclones de mayoría/minoría verde podían compararse con los subclones de mayoría roja/minoría en condiciones de control7,11. Aproximadamente 1/6 de clones adultos también contenían astrocitos y/o oligodendrocitos, indicados por asteriscos blancos. Los paneles B y F se reproducen con permiso de Hippenmeyer et al.46 y Rulands y Simons72, respectivamente. CP - Placa cortical. Haga clic aquí para ver una versión más grande de esta figura.
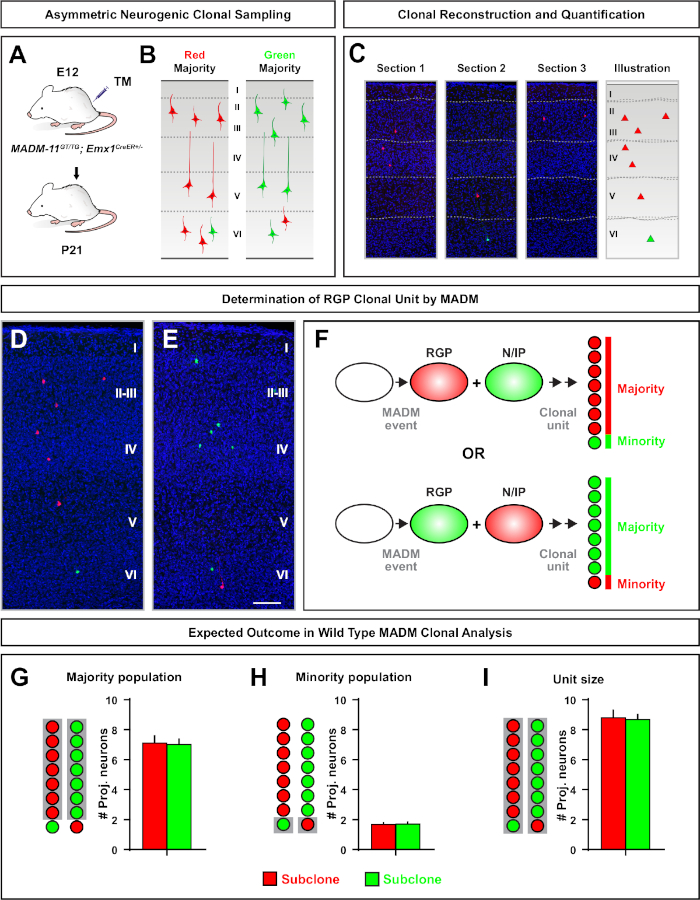
Figura 5: Análisis clonal de MADM para cuantificar la producción de neuronas mediadas por RGP. Análisis de la producción de neuronas excitatorias (unidad) por RGPs neurogénicos individuales a nivel clonal utilizando MADM7,11. (A) Paradigma experimental para inducir clones MADM en su mayoría asimétricos en la corteza en desarrollo. (B) Posibles resultados de clones asimétricos con la mayoría de subclone etiquetados en verde o rojo (C) Secciones consecutivas representativas que abarcan un único clon asimétrico neurogénico (D,E) imágenes de reconstrucción 3D de clones asimétricos representativos G2-X MADM con población mayoritaria en rojo (D) o verde (E) en MADM-11GT/TG; Emx1-CreERT2+/- con inducción TM en E12 y análisis en P21. Tenga en cuenta que las celdas etiquetadas en verde y rojo son de tipo salvaje. (F) Esquema que indica los dos posibles resultados experimentales del clon MADM. (G) Cuantificación del tamaño de la población mayoritaria derivada de la renovación de los RGPs en clones MADM-11. (H) Cuantificación del tamaño de la población minoritaria derivada de la renovación de los BDP en clones MADM-11. (I) Cuantificación del tamaño unitario de los clones asimétricos neurogénicos MADM-11. Los valores hipotéticos podrían representar la media de seM. Barra de escala de 100 m(D y E). TM - Tamoxifeno. Haga clic aquí para ver una versión más grande de esta figura.
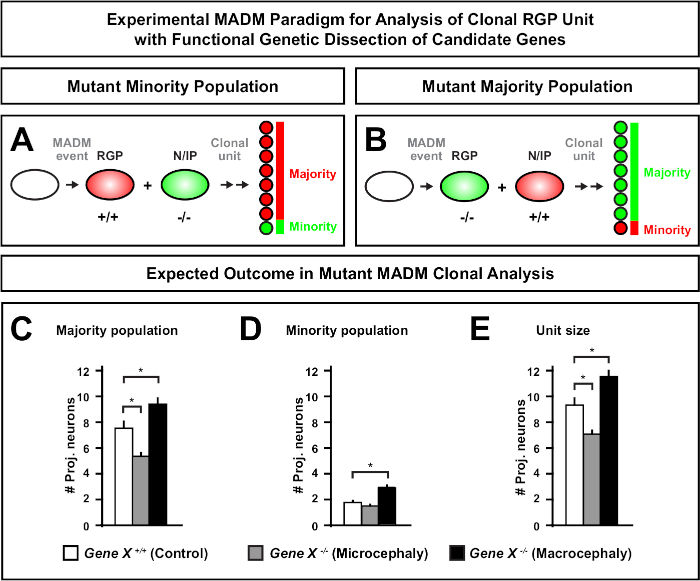
Figura 6: Análisis clonal de MADM para estudiar genes que conducen a la microcefalia y macrocefalia. El análisis clonal hipotético de MADM resulta al realizar la disección genética funcional de genes candidatos que conducen a microcefalia o macrocefalia. Para diseccionar las funciones celulares autónomas de un gen de interés (Gene X) sobre la producción de neuronas, MADM requiere que los alelos mutantes se introduzcan distales en los casetes MADM a través de la recombinación meótica (para obtener más información sobre cómo introducir alelos mutantes en el sistema MADM, véase también la Figura 2, Hippenmeyer et al.46, y Laukoter et al.46,73). (A,B) Esquema que indica paradigma MADM experimental para el análisis funcional de unidades CLONal RGP. El subclón mutante puede formar la población minoritaria (A) o mayoritaria (B). (C-E) Resultados hipotéticos del análisis clonal MADM al cuantificar los clones asimétricos de control MADM (barras blancas), microcefalia GENE-X MADM (barras grises) y barras negras macrocefalias Gene-X MADM). (C) Cuantificación del tamaño de la población mayoritaria. (D) Cuantificación del tamaño de la población minoritaria. (E) Cuantificación del tamaño unitario de los clones neurogénicos asimétricos. Los valores hipotéticos podrían representar la media de SEM. S - Escenario hipotético en el que la diferencia en el número de celda del subclón podría alcanzar significación, en relación con el control. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Estudios clonales MADM en la literatura. Resumen de los estudios en la literatura que contienen experimentos de linaje clon MADM, incluyendo el controlador CreERT2 utilizado, dosis TM, y el tiempo de inyección. Haga clic aquí para ver esta tabla (Haga clic con el botón derecho para descargar).
Discusión
Se describe un método para utilizar MADM para rastrear el linaje celular de los RGPs individuales in vivo en el neocórtex en desarrollo. Cuando se combinan con CreERT2inducible en TM, los eventos MADM se pueden cronomecer con precisión, proporcionando una lectura visual altamente cualitativa y cuantitativa de patrones de división de células madre a nivel de célula única. Al valorar la dosis de TM suministrada, en una situación ideal se puede obtener un promedio de menos de un clon por hemisferio cortical, proporcionando una separación espacial adecuada para distinguir inequívocamente los clones individuales. Al mantener la integridad del tejido, este método también captura información esencial sobre la posición, la morfología y los números de células absolutos. Los casetes MADM en Chr.11 7,11,12,46,56,57, en Chr. 751, y el MADM original en Rosa2647,53,59 se han utilizado en estudios de análisis clonal MADM. La alta resolución de células individuales proporciona una visión sin precedentes tanto de la morfología como de la relación clonal de las células hijas y permite la imagen en vivo de células madre proliferantes y clones emergentes46,,52.
La cesárea y el fomento de los cachorros para el análisis de clones en los puntos de tiempo postnatales es un paso necesario y crítico en el protocolo. Dependiendo del estado de salud de la presa embarazada tratada con TM, puede que no sea necesario realizar una cesárea. Sin embargo, criar a los cachorros con una madre adoptiva todavía es necesario, porque la madre tratada con TM puede tener problemas para lactación. No se han observado diferencias en la necesidad de fomentar con diferentes conductores de CreERT2. Tanto las líneas MADM como las madres adoptivas se mantienen sobre un fondo de CD-1. Si la cesárea no es necesaria, la presa de embarazo tratada con TM utilizada para generar cachorros experimentales puede ser reutilizada para crías experimentales adicionales de acuerdo con los principios del 3R (tenga en cuenta que esto sólo se puede hacer si las licencias experimentales de animales aprueban esta práctica). Las madres adoptivas se pueden utilizar para criar cachorros dentro de los 2 días después de dar a luz, pero se han observado tasas de éxito más altas cuando las madres adoptivas dan a luz el mismo día que los ratones experimentales que deben ser fomentados. Por lo tanto, es importante establecer apareamientos cronometrados para las madres adoptivas en paralelo a los apareamientos experimentales en el paso 1.1. Mantener un número de camada similar al de la camada original de la madre adoptiva puede mejorar la tasa de supervivencia de los cachorros acogidos, por lo que puede ser necesario eliminar de algunos a toda la camada original. Pasos adicionales que pueden mejorar el fomento incluye frotar los guantes del experimentador con basura y alimentos (para eliminar el aroma de los guantes); frotar a los cachorros suavemente después de la cesárea con fragmentos de la basura sucia de la madre adoptiva y el nido; y la colocación de los cachorros en estrecho contacto con los cachorros de la madre adoptiva antes de su colocación en la jaula de ratón adoptivo.
Al igual que en otros métodos de seguimiento de linaje basados en reporteros, se debe tener en cuenta cuidadosamente al elegir el controlador CreERT2 óptimo para experimentos clonales de MADM. En primer lugar, el promotor utilizado debe expresar la recombinase tanto temporal como espacialmente en la población progenitora de interés. Encontrar a este promotor puede ser un reto, porque algunos promotores pueden cambiar los patrones de expresión o quedar silenciados en diferentes etapas de desarrollo. Para mejorar la especificidad del tipo de celda se han utilizado varias recombinas específicas del sitio, cada una impulsada por promotores independientes. Cuando una o ambas recombinasas se expresan en la misma celda, esto etiqueta la celda y su progenie con un reportero fluorescente74,75,76,77. En resumen, es importante elegir un controlador CreERT2 que sea específico para la población de progenitores que se están analizando.
El paso más crítico de este método es la identificación de un clon, porque todas las celdas deben derivarse inequívocamente de un único evento de recombinación (paso 8.1). La valoración de la concentración de TM garantiza menos de un grupo de células rojas/verdes por hemisferio cerebral y maximiza la probabilidad de analizar un solo clon (paso 2.2)7,,11. Los clones deben descartarse si los clústeres de celdas vecinos se producen dentro de los 500 m del clon de interés. Por lo tanto, es importante examinar varias secciones antes y después de la aparición de un clon para asegurarse de que no hay eventos de recombinación adicionales cerca. Debido a la señal más débil de los fluoróforos, es necesario realizar inmunohistoquímica para eGFP y tdT en clones embrionarios (ver sección 6). Esto sólo se recomienda en clones adultos si se colan antígenos adicionales. Al capturar clones por imágenes, es importante capturar todo el ancho de la corteza donde se encuentra el clon (es decir, desde la superficie pial hasta el cuerpo calloso; consulte el paso 8.4) para no perder ninguna celda. Esto también facilita la alineación de la imagen durante el procesamiento de imágenes (sección 9). La sección 8 del protocolo requiere un microscopio confocal invertido, pero se puede adaptar dependiendo de la configuración del microscopio disponible. Se puede utilizar la microscopía de epifluorescencia, pero se recomienda la microscopía confocal porque esto conduce a una disminución de la contaminación por luz desde fuera del plano de enfoque. También es importante que la intensidad y ganancia del láser se ajusten para que las células verdes, rojas y amarillas puedan ser identificadas inequívocamente. Independientemente de la configuración, se recomienda utilizar un objetivo de al menos 20 veces para garantizar la separación espacial completa de las celdas estrechamente posicionadas. Además de registrar la profundidad cortical de todas las células (paso 8.6), las regiones corticales donde se encuentran los clones deben identificarse utilizando un atlas cerebral como el atlas de Allen Brain u otros mapas de coordenadas estereotaxicas. También se debe adoptar un paradigma de nomenclatura de archivos para asegurarse de que las imágenes clonadas sean fácilmente identificables. La siguiente información podría incluirse en el nombre del archivo: ID de imagen único, fecha de toma de la imagen, genotipo de animal, edad de inducción, edad de análisis, número de imagen en relación con el resto de las imágenes del mismo clon.
La introducción de una mutación distal en un casete MADM permite distintivamente la generación de mosaicos genéticos71 y permite la disección de reguladores moleculares de linaje y diversidad de tipo celular en el nivel clonal7,11,46,62. Para generar un mosaico genético con MADM, los casetes MADM deben estar vinculados meiotically al mismo cromosoma que el gen de interés (ver Figura 2 para el esquema de cría). Esto limita el análisis clonal actual con MADM a los genes ubicados en Chr.7 51, Chr. 1146, Chr. 1251y Chr. 6 distal al locus Rosa26 47. Estudios futuros utilizarán casetes MADM dirigidos a cualquier cromosoma, lo que permite el análisis de mosaico de prácticamente todos los genes del genoma del ratón a nivel clonal.
Por último, MADM no se limita al análisis de células progenitoras en el neocórtex en desarrollo. El estudio de muchos nichos de células madre podría beneficiarse de la capacidad de resolver arreglos espaciotemporales de células relacionadas clonalmente. Al aplicar MADM a otras regiones del cerebro, enfermedades (por ejemplo, cáncer), o en otros tejidos47,50,51,5252,53,54,55,56,57,58,59, estudios revelaron relaciones de linaje en clones derivados de diversas clases de progenitores y células madre (ver Tabla 1 para la lista actual de estudios clonales MADM). Otra aplicación futura interesante de MADM es combinarlo con reporteros funcionales o subcelulares adicionales, lo que aumentaría el grado de información que se puede adquirir de los clones.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a todos los miembros del laboratorio Hippenmeyer por su debate, el Centro de Bioimagen, el Centro de Ciencias de la Vida y el Centro Preclínico de IST Austria por su apoyo técnico. Esta labor fue apoyada por los fondos institucionales de IST Austria; R.B. recibió apoyo del programa Lise-Meitner del Fondo Científico Austriaco (FWF) (M 2416); N.A recibió apoyo del Fondo Austriaco de Ciencia (FWF) Firnberg-Programm (T 1031); GC recibió el apoyo del programa de investigación e innovación Horizonte 2020 de la Unión Europea en el marco del acuerdo de subvención Marie Sk-odowska-Curie No 754411 como becario postdoctoral de ISTplus; A.H. recibió el apoyo de una DOC (Beca Doctoral de la Academia Austriaca de Ciencias). Este estudio también fue apoyado por el Consejo Europeo de Investigación (ERC) en el marco del programa de investigación e innovación Horizonte 2020 de la Unión Europea (acuerdo de subvención no 725780 LinPro) a S.H.
Materiales
| Name | Company | Catalog Number | Comments |
| 1 mL tuberculin syringe (Omnifix Luer Lock) | Braun | 9204512N | |
| 1,4-diazabicyclooctane (DABCO) | Roth | 0718.2 | |
| 10 mL Syringe (Omnifix Luer Lock) | Braun | 8508429N | |
| 15 mL conical centrifuge | Sarstedt | 65.554.502 | |
| 24 multi-well dishes | Roth/Greiner Bio-one | CE56.1 | |
| 27- gauge x 3/4 needle (Sterican) | Braun | 16010256E | |
| Corn oil | Sigma | C8267-500ML | |
| Coverslips (24 x 60 mm #1) | Thermo Fisher Scientific (Menzel) | 15747592 | |
| Cryostat Cryostar NX70 | Thermo Fisher Scientific | 957000H | |
| Dako Pen (Wax marker) | Agilent | S200230-2 | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Invitrogen | D1306 | |
| Disposable microtome blade (MX35 Ultra) | Thermo Fisher Scientific | 705830 | |
| Fine Forceps (Dumont #5) | Fine Science Tools (FST) | 11254-20 | |
| Glass anti-roll plate | Histocom | M 449980 | |
| Glycerol | Sigma | G5516 | |
| LSM 800 Confocal | Zeiss | ||
| Mounting medium | 25 mg/mL DAPCO, 6 g Glycerol, 2.4 g Mowiol 4-88, 6 mL dH2O, 12 mL 0.2 M Tris-HCl (pH 8.5) | ||
| Mowiol 4-88 | Roth | 0713.2 | |
| Normal donkey serum | Innovative Research | IGDNSER100ML | |
| Paraformaldehyde | Sigma | 441244-1KG | |
| Peristaltic pump 323E/D 400RPM | Watson-Marlow | 036.3124.00A | |
| Sucrose | Sigma | S8501-5KG | |
| Superfrost plus glass slides | Thermo Fisher Scientific | J1800AMNT | |
| Tamoxifen | Sigma | T5648 | |
| Tissue Embedding mold T-12 (22mm square) | Polysciences Inc. | 18986-1 | |
| Tissue-Tek O.C.T | Sakura | 4583 | |
| Triton X-100 | Sigma | T8787-250ML | |
| Trizma hydrochloride | Sigma | 93363 | |
| Tween-20 | Sigma | P9416-100ML | |
| Software and Plugins: | |||
| Fiji | 1.52p | Fiji | |
| MultiStackReg | 1.45 | Download link | |
| TurboReg | EPFL Bioimaging | ||
| Zen Blue | 2.6 | Zeiss | |
| Experimental Models: Organisms/Strains: | |||
| Mouse: Emx1-CreER | The Jackson Laboratory | JAX:027784 | |
| Mouse: MADM-11-GT | The Jackson Laboratory | JAX:013749 | |
| Mouse: MADM-11-TG | The Jackson Laboratory | JAX:013751 | |
| Primary antibodies: | |||
| Chicken anti-GFP 1:500 | Aves Labs | GFP-1020 | |
| Goat anti-tdTomato 1:500 | Sicgen Antibodies | AB8181-200 | |
| Rabbit anti-RFP 1:500 | MBL | PM005 | |
| Secondary antibodies: | |||
| Donkey Anti-Chicken Alexa Fluor 488 1:500 | Jackson Immuno Research | 715-475-150 | |
| Donkey Anti-Goat Cy3 1:500 | Jackson Immuno Research | 705-165-147 | |
| Donkey Anti-Rabbit Cy3 1:500 | Jackson Immuno Research | 711-165-152 |
Referencias
- Malatesta, P., et al. Neuronal or glial progeny: regional differences in radial glia fate. Neuron. 37 (5), 751-764 (2003).
- Miyata, T., Kawaguchi, A., Okano, H., Ogawa, M. Asymmetric inheritance of radial glial fibers by cortical neurons. Neuron. 31 (5), 727-741 (2001).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30 (1), 465-502 (2014).
- Desikan, R. S., Barkovich, A. J. Malformations of cortical development. Annals of Neurology. 80 (6), 797-810 (2016).
- Gao, R., Penzes, P. Common mechanisms of excitatory and inhibitory imbalance in schizophrenia and autism spectrum disorders. Current Molecular Medicine. 15 (2), 146-167 (2015).
- Beattie, R., et al. Mosaic Analysis with Double Markers Reveals Distinct Sequential Functions of Lgl1 in Neural Stem Cells. Neuron. 94 (3), 517-533 (2017).
- Hippenmeyer, S. Molecular pathways controlling the sequential steps of cortical projection neuron migration. Advances in Experimental Medicine and Biology. 800, 1-24 (2014).
- Lodato, S., Arlotta, P. Generating neuronal diversity in the mammalian cerebral cortex. Annual Review of Cell and Developmental Biology. 31, 699-720 (2015).
- Hansen, A. H., Duellberg, C., Mieck, C., Loose, M., Hippenmeyer, S. Cell Polarity in Cerebral Cortex Development-Cellular Architecture Shaped by Biochemical Networks. Frontiers in Cellular Neuroscience. 11, 176 (2017).
- Gao, P., et al. Deterministic progenitor behavior and unitary production of neurons in the neocortex. Cell. 159 (4), 775-788 (2014).
- Llorca, A., et al. A stochastic framework of neurogenesis underlies the assembly of neocortical cytoarchitecture. eLife. 8, e51381 (2019).
- Ma, J., Shen, Z., Yu, Y. C., Shi, S. H. Neural lineage tracing in the mammalian brain. Current Opinion in Neurobiology. 50, 7-16 (2018).
- Caviness, V., Takahashi, T., Nowakowski, R. Numbers, time and neocortical neuronogenesis: a general developmental and evolutionary model. Trends in Neurosciences. 18 (9), 379-383 (1995).
- Schmechel, D. E., Rakic, P. A Golgi study of radial glial cells in developing monkey telencephalon: morphogenesis and transformation into astrocytes. Anatomy and Embryology. 156 (2), 115-152 (1979).
- Kessaris, N., et al. Competing waves of oligodendrocytes in the forebrain and postnatal elimination of an embryonic lineage. Nature Neuroscience. 9 (2), 173-179 (2006).
- Magavi, S., Friedmann, D., Banks, G., Stolfi, A., Lois, C. Coincident generation of pyramidal neurons and protoplasmic astrocytes in neocortical columns. The Journal of Neuroscience. 32 (14), 4762-4772 (2012).
- Anthony, T. E., Klein, C., Fishell, G., Heintz, N. Radial glia serve as neuronal progenitors in all regions of the central nervous system. Neuron. 41 (6), 881-890 (2004).
- Voigt, T. Development of glial cells in the cerebral wall of ferrets: direct tracing of their transformation from radial glia into astrocytes. The Journal of Comparative Neurology. 289 (1), 74-88 (1989).
- Amberg, N., Laukoter, S., Hippenmeyer, S. Epigenetic cues modulating the generation of cell-type diversity in the cerebral cortex. Journal of Neurochemistry. 149 (1), 12-26 (2019).
- Beattie, R., Hippenmeyer, S. Mechanisms of Radial Glia Progenitor Cell Lineage Progression. FEBS letters. 591 (24), 3993-4008 (2017).
- Telley, L., et al. Temporal patterning of apical progenitors and their daughter neurons in the developing neocortex. Science. 364 (6440), eaav2522 (2019).
- Oberst, P., et al. Temporal plasticity of apical progenitors in the developing mouse neocortex. Nature. 573 (7774), 370-374 (2019).
- Telley, L., et al. Sequential transcriptional waves direct the differentiation of newborn neurons in the mouse neocortex. Science. 351 (6280), 1443 (2016).
- Deppe, U., et al. Cell lineages of the embryo of the nematode Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 75 (1), 376-380 (1978).
- Woodworth, M. B., Girskis, K. M., Walsh, C. A. Building a lineage from single cells: genetic techniques for cell lineage tracking. Nature Reviews Genetics. 18 (4), 230-244 (2017).
- Masuyama, N., Mori, H., Yachie, N. DNA barcodes evolve for high-resolution cell lineage tracing. Current Opinion in Chemical Biology. 52, 63-71 (2019).
- Legue, E., Joyner, A. L. Chapter Ten-Genetic Fate Mapping Using Site-Specific Recombinases. Methods in Enzymology. 477, 153-181 (2010).
- Postiglione, M. P., Hippenmeyer, S. Monitoring neurogenesis in the cerebral cortex: an update. Future Neurology. 9 (3), 323-340 (2014).
- Espinosa-Medina, I., Garcia-Marques, J., Cepko, C., Lee, T. High-throughput dense reconstruction of cell lineages. Open Biology. 9 (12), 190229 (2019).
- Hwang, B., et al. Lineage tracing using a Cas9-deaminase barcoding system targeting endogenous L1 elements. Nature Communications. 10 (1), 1234 (2019).
- Kalhor, R., et al. Developmental barcoding of whole mouse via homing CRISPR. Science. 361 (6405), eaat9804 (2018).
- García-Moreno, F., Vasistha, N. A., Begbie, J., Molnár, Z. CLoNe is a new method to target single progenitors and study their progeny in mouse and chick. Development. 141 (7), 1589-1598 (2014).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Snippert, H. J., et al. Intestinal Crypt Homeostasis Results from Neutral Competition between Symmetrically Dividing Lgr5 Stem Cells. Cell. 143 (1), 134-144 (2010).
- Amitai-Lange, A., et al. A method for lineage tracing of corneal cells using multi-color fluorescent reporter mice. Journal of Visualized Experiments. (106), e53370 (2015).
- Vasistha, N. A., et al. Cortical and Clonal Contribution of Tbr2 Expressing Progenitors in the Developing Mouse Brain. Cerebral Cortex. 25 (10), 3290-3302 (2015).
- Chen, F., LoTurco, J. A method for stable transgenesis of radial glia lineage in rat neocortex by piggyBac mediated transposition. Journal of Neuroscience Methods. 207 (2), 172-180 (2012).
- Siddiqi, F., et al. Fate mapping by piggyBac transposase reveals that neocortical GLAST+ progenitors generate more astrocytes than Nestin+ progenitors in rat neocortex. Cerebral Cortex. 24 (2), 508-520 (2014).
- Yoshida, A., et al. Simultaneous expression of different transgenes in neurons and glia by combining in utero electroporation with the Tol2 transposon-mediated gene transfer system. Genes to Cells. 15 (5), 501-512 (2010).
- García-Marqués, J., López-Mascaraque, L. Clonal Identity Determines Astrocyte Cortical Heterogeneity. Cerebral Cortex. 23 (6), 1463-1472 (2013).
- Clavreul, S., et al. Cortical astrocytes develop in a plastic manner at both clonal and cellular levels. Nature Communications. 10 (1), 4884 (2019).
- Kim, G. B., et al. Rapid Generation of Somatic Mouse Mosaics with Locus-Specific, Stably Integrated Transgenic Elements. Cell. 179 (1), 251-267 (2019).
- Pilz, G. A., et al. Live imaging of neurogenesis in the adult mouse hippocampus. Science. 359 (6376), 658 (2018).
- Hippenmeyer, S., et al. Genetic Mosaic Dissection of Lis1 and Ndel1 in Neuronal Migration. Neuron. 68 (4), 695-709 (2010).
- Zong, H., Espinosa, J. S., Su, H. H., Muzumdar, M. D., Luo, L. Mosaic analysis with double markers in mice. Cell. 121 (3), 479-492 (2005).
- Ramón y Cajal, S. . Histologie du système nerveux de l'homme et des vertébrés. , (1911).
- Cowan, W. M. The emergence of modern neuroanatomy and developmental neurobiology. Neuron. 20 (3), 413-426 (1998).
- Ali, S. R., et al. Existing cardiomyocytes generate cardiomyocytes at a low rate after birth in mice. Proceedings of the National Academy of Sciences of the United States of America. 111 (24), 8850-8855 (2014).
- Hippenmeyer, S., Johnson, R. L., Luo, L. Mosaic analysis with double markers reveals cell-type-specific paternal growth dominance. Cell Reports. 3 (3), 960-967 (2013).
- Riccio, P., Cebrian, C., Zong, H., Hippenmeyer, S., Costantini, F. Ret and Etv4 promote directed movements of progenitor cells during renal branching morphogenesis. PLoS Biology. 14 (2), e1002382 (2016).
- Bonaguidi, M. A., et al. In vivo clonal analysis reveals self-renewing and multipotent adult neural stem cell characteristics. Cell. 145 (7), 1142-1155 (2011).
- Mayer, C., et al. Clonally Related Forebrain Interneurons Disperse Broadly across Both Functional Areas and Structural Boundaries. Neuron. 87 (5), 989-998 (2015).
- Muzumdar, M. D., et al. Clonal dynamics following p53 loss of heterozygosity in Kras-driven cancers. Nature Communications. 7, 12685 (2016).
- Shi, W., et al. Ontogenetic establishment of order-specific nuclear organization in the mammalian thalamus. Nature Neuroscience. 20, 516 (2017).
- Wong, S. Z. H., et al. In vivo clonal analysis reveals spatiotemporal regulation of thalamic nucleogenesis. PLoS Biology. 16 (4), e2005211 (2018).
- Xu, H. T., et al. Distinct Lineage-Dependent Structural and Functional Organization of the Hippocampus. Cell. 157 (7), 1552-1564 (2014).
- Brennand, K., Huangfu, D., Melton, D. All β Cells Contribute Equally to Islet Growth and Maintenance. PLoS Biology. 5 (7), e163 (2007).
- Ortiz-Alvarez, G., et al. Adult neural stem cells and multiciliated ependymal cells share a common lineage regulated by the geminin family members. Neuron. 102 (1), 159-172 (2019).
- Kaplan, E. S., Ramos-Laguna, K. A., Mihalas, A. B., Daza, R. A. M., Hevner, R. F. Neocortical Sox9+ radial glia generate glutamatergic neurons for all layers, but lack discernible evidence of early laminar fate restriction. Neural Development. 12 (1), 14 (2017).
- Lv, X., et al. TBR2 coordinates neurogenesis expansion and precise microcircuit organization via Protocadherin 19 in the mammalian cortex. Nature Communications. 10 (1), 3946 (2019).
- Mihalas, A. B., Hevner, R. F. Clonal analysis reveals laminar fate multipotency and daughter cell apoptosis of mouse cortical intermediate progenitors. Development. 145 (17), dev164335 (2018).
- Picco, N., et al. A mathematical insight into cell labelling experiments for clonal analysis. Journal of Anatomy. 235 (3), 687-696 (2019).
- Johnson, C. A., Ghashghaei, H. T. Sp2 regulates late neurogenic but not early expansive divisions of neural stem cells underlying population growth in the mouse cortex. Development. , (2020).
- Espinosa, J. S., Luo, L. Timing Neurogenesis and Differentiation: Insights from Quantitative Clonal Analyses of Cerebellar Granule Cells. The Journal of Neuroscience. 28 (10), 2301 (2008).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Liu, Y., et al. Tamoxifen-independent recombination in the RIP-CreER mouse. PLoS One. 5 (10), e13533 (2010).
- Klingler, E., Jabaudon, D. Do progenitors play dice?. eLife. 9, e54042 (2020).
- Hippenmeyer, S. Dissection of gene function at clonal level using mosaic analysis with double markers. Frontiers in Biology. 8 (6), 557-568 (2013).
- Rulands, S., Simons, B. D. Tracing cellular dynamics in tissue development, maintenance and disease. Current Opinion in Cell Biology. 43, 38-45 (2016).
- Laukoter, S., et al. Imprinted Cdkn1c genomic locus cell-autonomously promotes cell survival in cerebral cortex development. Nature Communications. 11 (1), 195 (2020).
- Daigle, T. L., et al. A Suite of Transgenic Driver and Reporter Mouse Lines with Enhanced Brain-Cell-Type Targeting and Functionality. Cell. 174 (2), 465-480 (2018).
- He, M., et al. Strategies and Tools for Combinatorial Targeting of GABAergic Neurons in Mouse Cerebral Cortex. Neuron. 91 (6), 1228-1243 (2016).
- Yamamoto, M., et al. A multifunctional reporter mouse line for Cre- and FLP-dependent lineage analysis. Genesis. 47 (2), 107-114 (2009).
- Plummer, N. W., et al. Expanding the power of recombinase-based labeling to uncover cellular diversity. Development. 142 (24), 4385 (2015).
- Imayoshi, I., Ohtsuka, T., Metzger, D., Chambon, P., Kageyama, R. Temporal regulation of Cre recombinase activity in neural stem cells. Genesis. 44 (5), 233-238 (2006).
- Sasaki, S., et al. Complete loss of Ndel1 results in neuronal migration defects and early embryonic lethality. Molecular and Cellular Biology. 25 (17), 7812-7827 (2005).
- Acampora, D., et al. Epilepsy and brain abnormalities in mice lacking the Otx1 gene. Nature Genetics. 14 (2), 218-222 (1996).
- Soeda, T., et al. Sox9-expressing precursors are the cellular origin of the cruciate ligament of the knee joint and the limb tendons. Genesis. 48 (11), 635-644 (2010).
- Klezovitch, O., Fernandez, T. E., Tapscott, S. J., Vasioukhin, V. Loss of cell polarity causes severe brain dysplasia in Lgl1 knockout mice. Genes & Development. 18 (5), 559-571 (2004).
- Pimeisl, I. M., et al. Generation and characterization of a tamoxifen-inducible EomesCreER mouse line. Genesis. 51 (10), 725-733 (2013).
- Nakagawa, N., et al. Memo1-Mediated Tiling of Radial Glial Cells Facilitates Cerebral Cortical Development. Neuron. 103 (5), 836-852 (2019).
- Nowotschin, S., et al. The T-box transcription factor Eomesodermin is essential for AVE induction in the mouse embryo. Genes & Development. 27 (9), 997-1002 (2013).
- Balordi, F., Fishell, G. Mosaic removal of hedgehog signaling in the adult SVZ reveals that the residual wild-type stem cells have a limited capacity for self-renewal. Journal of Neuroscience. 27 (52), 14248-14259 (2007).
- Liang, H., et al. Neural development is dependent on the function of specificity protein 2 in cell cycle progression. Development. 140 (3), 552-561 (2013).
- Guo, C., Yang, W., Lobe, C. G. A Cre recombinase transgene with mosaic, widespread tamoxifen-inducible action. Genesis. 32 (1), 8-18 (2002).
- Ahn, S., Joyner, A. L. In vivo analysis of quiescent adult neural stem cells responding to Sonic hedgehog. Nature. 437 (7060), 894-897 (2005).
- Storm, R., et al. The bHLH transcription factor Olig3 marks the dorsal neuroepithelium of the hindbrain and is essential for the development of brainstem nuclei. Development. 136 (2), 295-305 (2009).
- Koundakjian, E. J., Appler, J. L., Goodrich, L. V. Auditory neurons make stereotyped wiring decisions before maturation of their targets. Journal of Neuroscience. 27 (51), 14078-14088 (2007).
- Dor, Y., Brown, J., Martinez, O. I., Melton, D. A. Adult pancreatic β-cells are formed by self-duplication rather than stem-cell differentiation. Nature. 429 (6987), 41-46 (2004).
- Sohal, D. S., et al. Temporally regulated and tissue-specific gene manipulations in the adult and embryonic heart using a tamoxifen-inducible Cre protein. Circulation Research. 89 (1), 20-25 (2001).
- Ventura, A., et al. Restoration of p53 function leads to tumour regression in vivo. Nature. 445 (7128), 661-665 (2007).
- Johnson, L., et al. Somatic activation of the K-ras oncogene causes early onset lung cancer in mice. Nature. 410 (6832), 1111-1116 (2001).
- Tasic, B., et al. Extensions of MADM (mosaic analysis with double markers) in mice. PLoS One. 7 (3), e33332 (2012).
- Yu, J., Carroll, T. J., McMahon, A. P. Sonic hedgehog regulates proliferation and differentiation of mesenchymal cells in the mouse metanephric kidney. Development. 129 (22), 5301-5312 (2002).
- Zhao, H., et al. Role of fibroblast growth factor receptors 1 and 2 in the ureteric bud. Developmental Biology. 276 (2), 403-415 (2004).
- Schuchardt, A., D'Agati, V., Larsson-Blomberg, L., Costantini, F., Pachnis, V. Defects in the kidney and enteric nervous system of mice lacking the tyrosine kinase receptor Ret. Nature. 367 (6461), 380-383 (1994).
- Livet, J., et al. ETS gene Pea3 controls the central position and terminal arborization of specific motor neuron pools. Neuron. 35 (5), 877-892 (2002).
- Volz, K. S., et al. Pericytes are progenitors for coronary artery smooth muscle. eLife. 4, e10036 (2015).
- Cai, C. L., et al. A myocardial lineage derives from Tbx18 epicardial cells. Nature. 454 (7200), 104-108 (2008).
- Lasrado, R., et al. Lineage-dependent spatial and functional organization of the mammalian enteric nervous system. Science. 356 (6339), 722-726 (2017).
- Matsuoka, T., et al. Neural crest origins of the neck and shoulder. Nature. 436 (7049), 347-355 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados