Method Article
Traçage de lignée et analyse clonale dans le développement du cortex cérébral à l’aide de l’analyse mosaïque avec des marqueurs doubles (MADM)
Dans cet article
Résumé
Un protocole pour effectuer le traçage de lignée et l’analyse génétique fonctionnelle des gènes candidats au niveau d’une seule cellule à l’aide de l’analyse de mosaïque avec des marqueurs doubles (MADM) est présenté. L’analyse clonale de MADM fournit un cadre quantitatif pour mesurer le comportement prolifératif, la sortie cellulaire, et la relation de lignée des ancêtres individuels et de leurs cellules de fille.
Résumé
À partir d’un bassin limité d’ancêtres, le cortex cérébral des mammifères forme des circuits neuronaux fonctionnels très organisés. Cependant, les mécanismes cellulaires et moléculaires sous-jacents régulant les transitions de lignée des cellules souches neuronales (NSC) et la production éventuelle de neurones et de glie dans le neuroépithélium en développement restent flous. Les méthodes permettant de retracer les schémas de division du CSN et de cartographier la lignée des cellules liées au clonally ont progressé de façon spectaculaire. Cependant, de nombreuses techniques contemporaines de traçage de lignée souffrent de l’absence de résolution cellulaire du destin des cellules de descendance, ce qui est essentiel pour déchiffrer les modèles de division cellulaire progénitrice. Présenté est un protocole utilisant l’analyse de mosaïque avec des marqueurs doubles (MADM) pour effectuer l’analyse clonale in vivo. MADM manipule de façon concomitante les cellules progénitrices individuelles et visualise des schémas de division précis et la progression de la lignée à une résolution unique sans précédent. Les événements de recombinaison interchromosomiques basés à MADM pendant la phase G2-X de la mitose, ainsi que le CreERT2,inductible temporellement, fournissent des informations exactes sur les dates de naissance des clones et leurs modèles de division. Ainsi, le traçage de lignée MADM fournit des lectures optiques qualitatives et quantitatives sans précédent du mode de prolifération des progéniteurs de cellules souches au niveau des cellules uniques. MADM permet également d’examiner les mécanismes et les exigences fonctionnelles des gènes candidats dans la progression de la lignée NSC. Cette méthode est unique en ce que l’analyse comparative des sous-clones de contrôle et mutants peut être effectuée dans le même environnement tissulaire in vivo. Ici, le protocole est décrit en détail, et les paradigmes expérimentaux pour employer MADM pour l’analyse clonale et le traçage de lignée dans le cortex cérébral en développement sont démontrés. Fait important, ce protocole peut être adapté pour effectuer l’analyse clonale MADM dans n’importe quelle niche de cellules souches murines, tant que le pilote CreERT2 est présent.
Introduction
Le cortex cérébral est une structure très organisée composée de six couches distinctes. Le cortex contient un large éventail de types de cellules, y compris les neurones et la glie, qui interagissent pour former des circuits neuronaux fonctionnels. La plupart, sinon la totalité, des neurones de projection excitateurs corticaux et des glia sont dérivés d’un pool commun de cellules souches neuronales (NSC) connues sous le nom de progéniteurs gliaux radicaux (RGPs)1,2,3. Les RGP eux-mêmes sont dérivés de cellules souches neuroépithéliales (NESC) composant le neuroépithélium embryonnaire précoce. Par le jour embryonnaire 9 (E9) chez les souris, les NESC commencent à passer en RGPs4. La progression de la lignée RGP nécessite une régulation temporelle et spatiale précise, et lorsque ce processus est entravé, des troubles neurologiques graves tels que la mégalencéphalie, la microcéphalie, la lissencéphalie ou des déficiences telles que la schizophrénie et l’autisme peuvent entraîner5,6. À E10, la plupart des RPP subissent des divisions prolifératives symétriques, ce qui entraîne une expansion de la piscine progénitrice neuronale4,7. Les RMP finissent par commencer à se diviser de façon asymétrique, produisant des neurones de projection corticale d’une manière définie temporellement. Grâce à des ondes consécutives de neurogenèse, les neurones nouveau-nés migrent dans la plaque corticale formant laminee corticale avec des neurones nés tôt occupant des couches profondes et des neurones nés tardivement résidant dans les couches superficielles8,9,10. Étant donné que les neurones pyramidaux liés à la clonally migrent radialement dans le cortex avec très peu de dispersion tangentielle, les cellules filles ont tendance à former une structure en forme de colonne ou de cône appelée unité radiale neuronale4,11,12,13. Par E17, l’expansion neurogénique embryonnaire est complète chez les souris14. Les RGP peuvent également produire des cellules épendymales et certaines classes de glia, y compris les astrocytes et les oligodendrocytes1,15,16,17,18,19. Le potentiel des RMP pour donner naissance à la fois aux neurones et aux astrocytes semble être cohérent dans toutes les régions corticales18, avec environ 1/6 de RMP neurogéniques produisant également glia11.
Actuellement, les facteurs génétiques et épigénétiques régulant la progression temporelle d’une cellule souche le long de sa lignée sont pour la plupart inconnus. Les modèles temporels d’expression génique peuvent avoir un impact substantiel sur les décisions de lignée dans les RPP20,21,22,23,24. Comment cette relation étroitement liée entre les modèles temporels et spatiaux conduit à la diversité moléculaire des types neuronaux adultes à travers les zones corticales n’est pas connue. De même, la façon dont le potentiel individuel de cellules souches et sa production cellulaire est modulé au niveau cellulaire et moléculaire est une question importante sans réponse. Nous espérons que les études futures aborderont certaines de ces questions, ce qui permettra d’élargir notre compréhension de la formation fonctionnelle de circuits corticaux.
La neurobiologie du développement cherche à comprendre la relation de lignée que les cellules du cerveau partagent les unes avec les autres. Initialement, très peu d’outils de recherche étaient disponibles pour cela, et de nombreuses premières études se sont appuyées sur des observations visuelles des modèles de division dans des organismes transparents tels que Caenorhabditis elegans25. Au cours des dernières décennies ont vu une augmentation spectaculaire du nombre et de la sophistication des techniques disponibles13,26,27,28,29. L’émergence du système d’édition du génome CRISPR-Cas9 permet la reconstruction synthétique des relations de lignée cellulaire en introduisant l’évolution des codes à barres d’ADN27,30. Deux exemples récents de stratégies de codage à barres incluent l’utilisation de l’ARN guide homing qui dirige CRISPR-Cas9 à des loci de code-barres d’ADN spécifiques ou une cytidine deaminase fusionnée avec nickase Cas9 pour cibler endogène intercalée régions répétées31,32. Ces technologies fournissent des approches fortement multiplexées par l’introduction de codes à barres qui accumulent progressivement et de façon stable des mutations uniques au fil du temps. Les approches d’édition du génome sont très précieuses parce qu’elles permettent une analyse rétroactive de la relation entre deux cellules en fonction de l’héritage partagé de ces codes à barres. Cependant, afin de lire les codes à barres dans les cellules individuelles, le tissu doit généralement être perturbé, et donc l’information concernant la position, la morphologie, et les nombres absolus de cellules d’un ancêtre individuel est perdu.
Les paradigmes d’étiquetage combinatoire préservent l’information spatiale et permettent en principe également la distinction entre les clones étroitement localisés ou même qui se chevauchent33,34. Pour qu’une méthode de traçage de lignée soit instructive, elle doit étiqueter les ancêtres individuels et leur progéniture d’une manière clairsemée et indélébile. Notamment, les approches Brainbow35 et Confetti36,37 utilisent des journalistes stochastiques à base de recombinase multicolores Cre qui expriment une combinaison de protéines fluorescentes à partir d’un seul locus. Cre Le nombre étendu de combinaisons de couleurs simultanées qui peuvent être réalisées in vivo en font un outil puissant lors du traçage des clones corticals RGP et astrocytes34. Des systèmes basés sur transposon fournissant l’intégration génomique stable des transgenes encodant des reporters fluorescents et permettant le traçage de lignée des progéniteurs corticaux ont également été développés33,38,39,40,41. Les systèmes à base de transposon ont un avantage supplémentaire en ce que le journaliste construit stably intégrer dans le génome, et donc l’étiquette fiable cellules filles liées à la ligne. Pour retracer spécifiquement les lignées d’astrocytes, un certain nombre de méthodes ont été développées qui impliquent l’électroporation de transposases piggyBac, y compris Star Track, qui fait usage d’une combinaison de constructions encodage différentes protéines fluorescentes40,42. Une autre approche, magic markers, introduit les vecteurs Brainbow comme transgènes transposables. Ceci a été utilisé avec succès pour tracer les progéniteurs embryonnaires de neurones et d’astrocytes34,43. Récemment, l’analyse de mosaïque par double échange de cassettes recombinassées (MADR) a été trouvée pour étiqueter les cellules mutantes exprimant des éléments transgéniques à partir de loci chromosomiques précisément définis44. Ces puissantes techniques d’étiquetage combinatoire in vivo ont fourni de nombreux aperçus de la dynamique de lignée des cellules progénitrices. Cependant, ces analyses sont effectuées sur des tissus fixes, fournissant un instantané des clones individuels à un stade de développement défini. Afin d’observer les changements dans la dynamique de lignée des clones simples au fil du temps, des méthodes d’imagerie in vivo chroniques similaires à celles effectuées dans le gyrus denté adulte doivent être appliquées45.
L’analyse de mosaïque avec des marqueurs doubles (MADM) est une méthode puissante d’étiquetage à deux couleurs qui permet le traçage in vivo de lignées individuelles de cellules progénitrices individuelles chez les souris46,47. Deux composants sont nécessaires pour que les événements d’étiquetage MADM se produisent : premièrement, les cassettes MADM doivent être ciblées sur des loci identiques sur les chromosomes homologues. Les cassettes se composent de deux gènes de reporter fluorescent chimérique, eGFP (vert, [G]) et de la tomate de dimère tandem (rouge, tdT[T]). La cassette GT contient le n-terminus d’eGFP et le C-terminus de tdT, séparés par un intron contenant un site loxP. La cassette TG est construite inversement, avec le N-terminus de tdT et le C-terminus d’eGFP. Deuxièmement, l’expression de cre recombinase dans la même cellule contenant les cassettes MADM ciblées est essentielle. En l’absence de Cre, les cassettes chimériques n’expriment pas eGFP fonctionnelle ou tdT parce que leurs séquences de codage sont perturbées. Les sites loxP servent de cible à la recombinaison interchromosomique pédiée par Cre, ce qui entraîne la reconstitution simultanée des deux cassettes d’expression. Si la recombinaison se produit pendant la phase G2 du cycle cellulaire suivie de la ségrégation X (G2-X), les deux cellules filles exprimeront chacune l’une des deux protéines fluorescentes. La régulation temporelle de l’activité creert2 à l’aide du tamoxifène (TM) fournit des informations précises sur la date de naissance des clones MADM et les schémas de division de leur progéniture (Figure 1A)29,46,47.
MADM peut potentiellement systématiquement étiqueter les clones individuels avec une haute résolution unicellulaire dans le cerveau de la souris semblable à des méthodes traditionnelles mais non spécifiques et laborieuses comme Golgi tache48 ou colorant remplissage49. Étant donné que seul le promoteur qui conduit CreERT2 détermine la spécificité du type cellulaire de l’étiquetage clonal MADM, MADM peut en principe être appliqué pour le traçage de lignée clonale dans n’importe quel organe et tissu murin47,50,51,52. En effet, des études ont déjà utilisé MADM pour révéler des relations de lignée dans des clones dérivés de tissus divers47,50,51,52,53,54,55,56,57,58,59. Madm paradigmes expérimentaux ont été appliqués à la lignée d’étude dans les neurones de projection corticale, glia, et les cellules souches postnatales dans le développement néocortex7,11,12,46,60,61,62,63,64,65. MADM a également été utilisé pour étudier la lignée cellulaire dans le gyrus denté adulte, thalamus, cellules granuleuses cérébellaires, et les interneurones au niveau clonal (voir le tableau 1 pour une liste complète)47,53,54,56,57,66.
Une caractéristique unique de MADM est la capacité de lier génétiquement les mutations distales à une cassette MADM, créant ainsi une mosaïque génétique (figure 1B et figure 2). Il en résulte des cellules filles de type sauvage étiquetées avec un marqueur fluorescent (tdT dans la figure 1B)et des frères et sœurs mutants homozygotes avec l’autre (eGFP dans la figure 1B)dans un environnement hétérozygote non étiqueté. MADM est unique en ce que l’analyse comparative des sous-clones de contrôle et mutants peut être effectuée dans le même environnement tissulaire in vivo. À l’origine, les cassettes MADM ont été ciblées dans le locus47 rosa26 , mais l’analyse MADM de la fonction génique a été limitée aux gènes distal au locus. Pour surmonter (au moins en partie) cette limitation et élargir les possibilités d’analyses génétiques basées sur MADM, les cassettes MADM ont été frappées à proximité des centromères de Chr. 751, Chr. 1146, et Chr. 1251. Cibler les 19 autosomes de souris avec des cassettes MADM est en cours et permettra à pratiquement n’importe quel gène d’être étudié à l’avenir, fournissant une plate-forme inégalée pour l’étude des relations de lignée développementale en combinaison avec l’analyse génétique fonctionnelle.
Protocole
Les protocoles de souris ont été examinés par l’installation de base préclinique institutionnelle (PCF) et le comité d’éthique interne de l’IST Autriche. Toutes les reproductions et les expérimentations ont été effectuées en vertu d’une licence approuvée par le Ministère fédéral autrichien de la science et de la recherche conformément aux lois autrichiennes et européennes sur les animaux.
1. Élevage de souris expérimentales pour l’analyse clonale MADM
- Configurer les accouplements expérimentaux chronométrés MADM (>P56; CD-1) en fin d’après-midi (17h00) et vérifiez les bouchons vaginaux le matin de la procédure (8h00). Le matin, la prise est présente compte comme jour 0,5. Voir la figure 2 pour une vue d’ensemble de la configuration expérimentale de l’accouplement de souris. Assurez-vous que les délais pour l’induction et l’analyse de CreERT2 par TM sont appropriés pour répondre aux questions expérimentales.
REMARQUE : Pour de plus amples renseignements, reportez-vous à la figure 3 et aux résultats représentatifs ci-dessous. - Pour l’échantillonnage postnatal, mettre en place des élevages pour générer des mères adoptives en parallèle.
REMARQUE : Ceux-ci doivent être démarrés jusqu’à 1 à 2 jours avant la mise en place de reproductions expérimentales.
2. Induction TM chez les souris MADM
- Préparer une solution de travail de 20 mg/mL TM en la dissolvant dans de l’huile de maïs dans un tube de centrifugeuse conique de 15 mL ou 50 ml et en le plaçant sur une plate-forme à bascule pendant environ 4 h à température ambiante (RT), assurant que tm est complètement dissous. Conserver la solution de travail à 4 °C recouverte de papier d’aluminium et l’utiliser dans les 2 semaines.
- Pour induire des événements de recombinaison MADM, livrer une seule injection de TM intrapéritoneally (IP) à l’aide d’une seringue de tuberculine de 1 mL et une aiguille de 25 G dans un barrage enceinte chronométré. Selon le stade de la neurogenèse corticale, injectez la TM entre E10−E15 à une dose de 1−2 mg/barrage enceinte. Pour les retards précoces (c.-à-d. E10) utiliser un maximum de 1 mg/mère enceinte (25 mg/kg) pour prévenir les complications pendant la grossesse11. Pour les délais entre E11-E15 utiliser 2 mg/barrage enceinte (50 mg/kg)7.
REMARQUE : Alternativement, tm peut être administré avec un gavage oral pour des grossesses tardives. - Pour l’analyse clonale DE MADM aux délais postnatals, récupérez les embryons vivants à E18−E19 par césarienne, puis élevez des chiots avec une mère adoptive.
REMARQUE : Selon l’état de santé de la femelle enceinte, il peut ne pas être nécessaire d’effectuer une césarienne, mais l’éducation des chiots avec une mère adoptive est toujours nécessaire parce que la mère d’origine traitée par TM peut avoir de la difficulté à allaiter. - Pour récupérer des embryons vivants par césarienne ou pour récupérer des délais embryonnaires pour l’analyse, sacrifiez le barrage enceinte par dislocation cervicale.
- Placer l’animal en position de supination et désinfecter la fourrure avec 70 % d’éthanol. Faire une petite incision dans la peau dans le bas-ventre au-dessus de l’utérus en utilisant des forceps chirurgicaux et des ciseaux. Faites une deuxième incision à travers les muscles et la paroi musculaire abdominale pour révéler le péritoine.
- Retirer l’utérus en se séparant des tissus environnants avec des ciseaux. Transférer l’utérus intact sur un gant rempli d’eau chaude pour augmenter le taux de survie de l’embryon jusqu’à ce que chacun soit retiré de l’amnion individuellement.
- Utilisez des ciseaux et des doigts fins pour ouvrir soigneusement les parois utérines pour libérer des embryons. Ne coupez pas les cordons ombilicals trop près du corps pour éviter une perte de sang importante. Si les embryons doivent être utilisés pour l’analyse, passez à l’étape 3.9. Si les chiots doivent être encouragés, passez à l’étape 2.8.
- Si l’accueil est nécessaire, nettoyez les chiots avant de les transférer à la mère adoptive. Tout en nettoyant les chiots, appuyez doucement sur la poitrine de temps en temps pour commencer à respirer. Remettre sur un deuxième gant rempli d’eau chaude pour améliorer le taux de survie.
REMARQUE : Il est important d’enlever doucement tout amnion et/ou placenta restants avec une serviette en papier. - Avant de transférer les chiots à la mère adoptive, retirez la mère adoptive de sa cage, retirez les chiots d’origine et remplacez-les par les chiots expérimentaux. Rends la mère adoptive dans sa cage.
REMARQUE : Voir la discussion pour obtenir d’autres suggestions visant à améliorer les taux d’acceptation. - Si le génotypage est nécessaire, recueillir des biopsies d’orteil ou de queue entre P6−P8.
REMARQUE : Effectuez cette étape uniquement si les licences expérimentales sur les animaux approuvent cette pratique.
3. Préparation tissulaire pour les clones MADM dans le cerveau
REMARQUE : Pour les expériences qui incluent le tissu postnatal (≥P4), passez à l’étape 3.1. Pour les délais embryonnaires et les débuts postnatals (P0−P3), continuez à l’étape 3.9.
- Anesthésier l’animal expérimental MADM avec une injection IP d’une solution kétamine/xylazine/acepromazine (65 mg, 13 mg et 2 mg/kg de poids corporel, respectivement) et confirmer que la souris ne réagit pas en pinçant la patte postérieure.
REMARQUE : Les souris MADM mâles et femelles (fond CD-1) sont utilisées pour l’analyse. Si le génotypage est nécessaire, recueillir des biopsies de l’oreille à ce stade. - Placez l’animal anesthésié en position supinée sur le plateau de chirurgie de perfusion et désinfectez la fourrure avec 70% d’éthanol. Pour commencer la chirurgie, faites soigneusement une incision avec des ciseaux et des forceps chirurgicaux à travers la couche externe de la peau, puis une deuxième incision à travers la couche musculaire. Soulevez la pointe du sternum et coupez le tissu conjonctif sur les côtés, en prenant une prudence supplémentaire pour éviter de couper le foie. La cavité thoracique sera visible.
- Coupez le diaphragme et soulevez pour révéler le cœur. Coupez soigneusement la cage thoracique et épinglez-vous sur le plateau chirurgical pour exposer le cœur. Pour les chiots, retirer complètement la cage thoracique.
- Insérez une aiguille avec du phosphate tamponné saline (PBS) dans le ventricule inférieur gauche (tissu plus pâle). À l’aide de petits ciseaux d’iris, faire une incision à l’extrémité postérieure de l’atrium droit (tissu rouge foncé) pour que le sang s’écoule.
- Effectuer la perfusion avec PBS suivie immédiatement par fraîchement fait, glace-froid 4% paraformaldéhyde (PFA) préparé dans PBS. Pour les chiots (P4−P10), utilisez des seringues pour effectuer la perfusion. Remplissez une seringue de 10 mL de PBS et une autre de 10 mL de 4 % de PFA. Assurez-vous que toutes les bulles d’air dans les seringues ont été enlevées. Pour les animaux plus âgés, utilisez une pompe péristaltique.
- Commencez à perfuser avec pbs (10 mL à 2−4 mL/min chez les chiots; 20 mL à 4−6 mL/min pour les adultes utilisant une pompe péristaltique). Le foie deviendra clair et jaune pâle si l’aiguille est positionnée correctement.
- Une fois terminée, retirer l’aiguille des chiots et insérer l’aiguille contenant de la PFA dans le même trou. Pour les adultes, arrêtez la pompe péristaltique avant d’échanger la solution PBS avec de la PFA glacée, en veillant à éviter les bulles dans les tubes d’absorption. Reprendre la perfusage avec la PFA (10 mL à 2−4 mL/min chez les chiots; 30 mL à 4−6 mL/min pour les adultes utilisant une pompe péristaltique).
- Lorsque la perfusion est terminée, décapitez la souris et retirez le cerveau par dissection soigneuse. Transférer le cerveau à 4% PFA. Utiliser au moins 5 fois les volumes cérébraux (c.-à-d. ~5−10 ml de PFA dans un tube de centrifugeuse conique de 15 mL) et incuber pendant la nuit à 4 °C pour la fixation postperfusion afin d’assurer la fixation complète du tissu. Continuez à l’étape 3.10.
- Pour les tissus embryonnaires et les tissus postnatals précoces (p0−P3), après avoir effectué une césarienne, décapitez les embryons avec des ciseaux. Si le génotypage est nécessaire, recueillir la queue de l’embryon à ce stade. Disséquer immédiatement le cerveau et transférer à une plaque de 12 puits contenant 2−3 mL de 4% PFA/puits. Incuber toute la nuit à 4 °C pour la postfixation.
- Échangez le lendemain matin PFA avec 10 mL (adulte) ou 2−3 mL (embryon) de PBS et répétez le lavage 3x pendant 15 min à RT. Transférer le tissu à la solution de saccharose à 30% dans le tampon de phosphate (PB) et conserver à 4 °C sur une plate-forme à bascule jusqu’à ce que le tissu s’enfonce dans la solution.
- Incorporer le cerveau dans la température de coupe optimale (OCT) composé dans un moule d’intégration, en prenant soin d’orienter le cerveau pour la section coronale ou sagittale. Congeler en plaçant le moule d’incorporation sur la glace sèche jusqu’à ce que l’OCT devienne complètement opaque (~10−15 min). Conserver les tissus à -80 °C jusqu’à ce qu’ils soient utilisés davantage.
4. Préparation du tissu MADM pour l’immunohistochimie
- Attachez le bloc tissulaire au disque du spécimen dans le cryostat en appliquant un anneau d’OPO sur le disque et en plaçant le bloc directement dans le PTOM lorsqu’il commence à geler. Assurez-vous que le bloc est correctement orienté pour le plan de coupe souhaité.
REMARQUE : Ici, la section de coronale pour étudier les clones corticaux de MADM est décrite en détail. - Réglez la température du bloc dans le cryostat à -20 °C et la température de la lame à -21 °C.
- Laissez le bloc tissulaire s’ajuster à la température de la chambre en montant le disque du spécimen sur le support du spécimen et laissez-le en cryostat pendant environ 5 min avant de commencer à sectionr.
- Couper le bloc en sections épaisses (45−60 μm) jusqu’à ce que la région d’intérêt des tissus soit atteinte.
- Une fois que le bord du cortex est clairement visible, arrêtez de sectionr et verrouillez la lame. Assurez-vous que la lame est protégée avant de couper le bloc.
- Couper l’excès d’OCT entourant le tissu à l’aide d’une lame, en laissant ~1−2 mm d’OCT de tous les côtés du cerveau.
- Ensuite, orienter le bloc de sorte que l’un des bords latéraux du cortex est orienté vers le bas et l’autre vers le haut (c.-à-d. le bord le plus rostral du cortex est pointé à droite).
- Commencer la section avec une épaisseur de 45 μm pour les clones adultes et de 30 μm pour les clones embryonnaires. Effectuez chaque section individuellement et utilisez une petite brosse pour garder la zone sous le couteau propre des débris laissés tout en coupant le bloc.
REMARQUE : Si cela n’est pas fait et qu’une section tombe, il peut être difficile de déterminer l’ordre correct des tranches. - Si les sections commencent à se courber, couper les bords du bloc et/ou ajuster soigneusement la plaque antiroll en verre.
- Pour l’analyse des clones embryonnaires, monter les sections directement sur une lame givrée. Sécher sur une plaque chauffante à 37 °C avant de passer directement à l’étape 5.6.
REMARQUE : Plusieurs sections peuvent être ajoutées à une diapositive, mais assurez-vous que l’ordre séquentiel est maintenu. - Pour recueillir les clones adultes, préparez 24 plaques de puits contenant 1 mL de PBS/puits (généralement, 5 à 6 plaques par cerveau). À partir du premier puits, avec des forceps froids recueillir des sections de série individuelles dans PBS dans l’ordre de sectionnement.
REMARQUE : La méthode de section flottante est adoptée pour les tissus adultes afin de s’assurer qu’aucune section n’est oubliée et que les sections montées ne contiennent pas de rides. - Arrêtez de sectionr une fois la fin du néocortex atteinte.
- Pour les clones adultes, passez au montage de sections flottantes.
REMARQUE : Les sections peuvent être conservées en PBS à 4 °C jusqu’à 24 h.
5. Montage de tissu adulte pour l’imagerie
REMARQUE : Les outils suivants sont nécessaires : petit pinceau, plat Petri, PBS avec 0,5 % d’interpolation (PBS-T), diapositives d’adhérence (Table des matériaux),support de montage(Table des matériaux),couvercles et forceps.
- Remplissez une boîte de Pétri avec PBS-T.
REMARQUE : Le détergent est utilisé pour faciliter le processus de montage. Si la coloration d’antigènes supplémentaires sensibles aux détergents (c.-à-d. les glycoprotéines) est nécessaire, il est préférable de sauter l’ajout de Tween. - Placez une diapositive d’adhérence dans le PBS-T afin qu’il soit presque couvert jusqu’à l’étiquette.
- Transférer la première section dans le PBS-T.
- À l’aide d’un petit pinceau, manœuvrez la section sur la glissière et arrangez-la pour préserver l’ordre de coupe. Procédez de la même manière avec toutes les autres sections.
- Une fois que toutes les sections sont en position, placez la diapositive (~12−16 sections/diapositive) dans une chambre de diapositive sombre. Soulevez légèrement le couvercle pour permettre aux sections de sécher complètement (~10−20 min), en veillant à ce qu’elles restent respectées dans les étapes suivantes.
- Si vous effectuez une immunohistorique pour des antigènes supplémentaires, passez directement à l’article 6 ou 7.
REMARQUE : Pour les délais embryonnaires, il est nécessaire d’effectuer des étapes d’immunostaining pour au moins GFP et tdT (section 6). Pour les clones adultes, cela n’est nécessaire que si la coloration pour des antigènes supplémentaires en parallèle (sections 6 et 7). - Réhydrater et laver les sections 1x avec 1x PBS pendant 5 min pour enlever le PBS-T résiduel. Appliquer ~1 mL de 4′,6-diamidino-2-phénylindole (DAPI) dilué en 1x PBS (1 μg/mL) à la diapositive, en veillant à ce que toutes les sections soient couvertes et incuber pendant 15 min.
- Retirez soigneusement le DAPI et lavez 1x avec 1x PBS pendant 5 min. Retirez l’excès de PBS et séchez pendant ~1−2 min avant de l’intégrer dans 110 μL de support de montage. Sceller avec un couvercle de 24 x 60 mm et laisser sécher pendant au moins 3 h avant l’imagerie.
6. Immunostaining pour GFP et tdT seulement
REMARQUE : Cette section est nécessaire pour les clones embryonnaires.
- Placez les diapositives horizontalement dans une chambre d’incubation humidifiée. Marquez les limites de diapositives à l’aide d’un marqueur de cire pour minimiser la quantité de tampon requise.
- Réhydrater les sections avec 1x PBS. Pour améliorer la qualité de coloration, travaillez avec des tissus fraîchement sectionnés.
- Ajouter 250 à 400 μL de tampon de blocage (0,5 % de Triton X-100, 2−3 % de sérum d’âne normal en 1x PBS) par diapositive, en veillant à ce que toutes les sections soient couvertes. Incuber pendant 1 h.
REMARQUE : La concentration de détergent (Triton X-100 ou Tween-20) variera selon les anticorps primaires supplémentaires utilisés parce que certains antigènes sont plus sensibles aux détergents que d’autres. - Supprimez la mémoire tampon de blocage et ajoutez des anticorps primaires dans la mémoire tampon de blocage à la diapositive (300−400 μL/slide).
REMARQUE : Un exemple de réaction primaire standard d’anticorps pour l’anti-GFP/anti-tdT (MADM) pourrait utiliser le poulet anti-GFP (1:500) et le lapin anti-RFP (1:500). - Incuber avec des anticorps primaires pendant la nuit à 4 °C.
REMARQUE : Les lames doivent être incubées parfaitement horizontalement avec un tampon couvrant toutes les sections. Dans le cas contraire, des taches inégales ou médiocres peuvent en résulter. - Confirmez le lendemain matin que la mémoire tampon de blocage avec des anticorps primaires couvre toujours toutes les sections de la diapositive. Si ce n’est pas le cas, répétez l’étape d’incubation pendant 3−4 h à RT.
- Retirez les anticorps primaires et lavez 4x avec 1x PBS pendant 10 min à RT.
- Ajouter des anticorps secondaires dilués dans la mémoire tampon de blocage pour glisser (300−400 μL/slide) : Alexa Fluor 488 anti-poulet IgG (1:500) et Cy3 anti-rabbit IgG (1:500).
- Incuber à RT pendant 2 h. Gardez les lames couvertes de la lumière pour éviter le blanchiment des fluorophores.
- Retirer les anticorps secondaires et laver 2x avec 1x PBS pendant 10 min.
- Incuber avec DAPI dilué en PBS (1:5,000) pendant 15 min.
- Laver les sections 1x avec 1x PBS pendant 10 min.
- Retirer l’excès de PBS et sécher pendant environ 1−2 min avant d’être intégré dans 110 μL de milieu de montage.
- Sceller avec 24 x 60 mm de couvercle et laisser sécher pendant au moins 3 h avant l’imagerie. L’image glisse dans les 1 à 2 semaines suivant l’immunohistochimie pour assurer un signal optimal.
7. Immunostaining pour GFP, tdT, et antigènes supplémentaires
- Effectuez les étapes 6.1−6.3.
- Supprimez la mémoire tampon de blocage et ajoutez des anticorps primaires dans la mémoire tampon de blocage à la diapositive (300−400 μL/slide).
REMARQUE : Lorsque la coloration de trois antigènes ou plus (c.-à-d. GFP, tdT et protéine d’intérêt) et de l’anticorps pour la protéine d’intérêt a été soulevée dans le lapin, il est recommandé d’utiliser l’anticorps primaire anti-tdT (chèvre) à une dilution de 1:500. Un exemple d’une réaction primaire d’anticorps pour trois antigènes avec la coloration tdT alternative pourrait employer l’anti-GFP de poulet (1:500), l’anti-tdT de chèvre (1:500), et l’anticorps contre la protéine d’intérêt (c.-à-d. le lapin). - Effectuez les étapes 6.5−6.7.
- Ajouter un mélange d’anticorps secondaire dilué dans le tampon de blocage pour glisser (300−400 μL/slide) : Alexa Fluor 488 anti-poulet IgG (1:500), Cy3 anti-chèvre IgG (1:500) et Alexa Fluor 647 anti-rabbit IgG (1:500).
- Effectuez les étapes 6.9−6.14.
8. Acquisition et quantification d’images confocales de clones MADM
- Identifier et documenter les sections cérébrales contenant des clones et leurs emplacements dans le cortex.
REMARQUE : Le nombre de sections d’une portée de clone varie en fonction du moment où le clone a été induit, du pilote CreERT2 et du moment de l’analyse. Cette étape peut être effectuée soit sur un microscope confocal ou un microscope épifluorescence. - À l’aide d’un microscope confocal inversé, commencez par sélectionner et configurer les lignes et filtres laser corrects. Pour les cerveaux MADM, sélectionnez DAPI, GFP et tdT (excitation : 358 nm, 488 nm et 554 nm, respectivement; émission de pointe : 461 nm, 507 nm et 581 nm, respectivement). Assurez-vous que le trou d’épingle est réglé sur 1 unité aérée pour une qualité d’imagerie optimale.
- Pour les paramètres spécifiques confocal, clones d’image avec un objectif 20x et un zoom 1x. Pour les images qui seront utilisées dans les quantifications, utilisez une valeur de 1,52−2,06 μs (valeurs 7−8 dans le logiciel d’acquisition d’images) sans moyenne. Ajuster l’intensité laser et gagner les réglages pour chaque canal, le cas échéant.
REMARQUE : Selon la qualité d’image requise, les paramètres de vitesse de numérisation et de moyenne peuvent varier. - Une fois que le clone est clairement identifié, disposer les tuiles d’imagerie pour couvrir toutes les sections pertinentes dans le clone. Réglez la pile z de sorte que toutes les cellules étiquetées MADM du clone soient capturées avec un intervalle de 1,5 μm/z-stack. Ajuster la région carrelée de sorte que toute la largeur du cortex soit capturée lors de l’imagerie du clone (c.-à-d. de la surface piale au corpus callosum).
- Clones individuels d’image couvrant plusieurs sections consécutivement, s’assurant que toutes les sections sans cellules dans un clone sont encore photographiés aux fins de la reconstruction 3D et l’interprétation correcte de l’information spatiale cellulaire.
- Analyser chaque section contenant les cellules d’un clone MADM séquentiellement du rostral à l’extrémité caudale du cortex. Distinguer les neurones individuels et les glia en fonction de leur morphologie et/ou de leur coloration des marqueurs. Enregistrer l’information positionnelle en parallèle en fonction des limites respectives des couches définies par la coloration nucléaire (DAPI).
REMARQUE : Voir la figure 4 pour les résultats représentatifs de l’analyse embryonnaire et la figure 5 pour les résultats représentatifs pour l’analyse chez les adultes.
9. Reconstruction en série 3D des clones
REMARQUE : La reconstruction 3D de clones individuels photographiés sur des sections cérébrales en série est utile pour l’affichage visuel ainsi que pour l’analyse des architectures clonales 3D et peut être effectuée selon les étapes suivantes.
- Stitch et fusible des images carrelées confocales basées sur des paramètres d’acquisition à l’aide d’un logiciel d’acquisition d’images. Ouvrez le fichier .czi, puis exécutez la méthode Stitching sous l’onglet Traitement dans le logiciel ZEN (Zeiss).
- Exporter des piles d’images cousues sous forme de z-planes individuels au format TIFF. Ouvrez le fichier .czi cousu, puis exécutez la méthode Exportation d’images sous l’onglet Traitement. Pour les images multicanaux, exportez sous forme d’images rouge/verte/bleue pour le traitement ultérieur de l’image.
- Répétez les étapes 9.1 et 9.2 pour chaque section cérébrale en série d’un clone.
REMARQUE : Pour une reconstruction 3D précise, toutes les sections cérébrales d’un clone, y compris celles qui n’ont pas de cellules étiquetées, doivent également être traitées. - Concatenate images individuelles dans une seule pile dans l’ordre à partir de la plus rostral à la plus caudale z-plane en utilisant un logiciel de traitement d’image open source tels que ImageJ / Fidji67,68.
REMARQUE : Toutes les images vierges sur les bords de chaque section du cerveau doivent être enlevées à ce stade. - Si nécessaire, corrigez la pile d’images obtenue à partir de l’étape 9.4 pour le désalignement à l’aide d’un plugin ImageJ appelé «MultiStackReg» en suivant les étapes 9.5.1−9.5.5. Si l’alignement de l’image n’est pas nécessaire, passez à l’étape 9.6.
REMARQUE : Ce plugin effectue l’alignement d’image du canal avec le contraste le plus élevé (habituellement DAPI) puis applique la transformation enregistrée aux autres canaux, permettant ainsi un alignement d’image fiable des piles multicanaux. Un plugin auxiliaire appelé "TurboReg" doit être préinstallé.- Dans ImageJ, installez les plugins «MultiStackReg» et« TurboReg».
- Ouvrez la pile d’images de clones obtenues à partir de l’étape 9.4 à aligner. Divisez les canaux en DAPI (bleu), GFP (vert) et tdT (rouge) dans l’option Couleur sous l’onglet Image.
- Exécutez le plugin «MultiStackReg» pour aligner le canal DAPI par la transformation «Rigid Body» et enregistrer le fichier de transformation.
- Appliquez le fichier de transformation enregistré aux deux autres canaux à l’aide de «MultiStackReg».
- Fusionnez les trois canaux alignés et enregistrez la pile alignée.
- Pour orienter le clone dans ImageJ, la pile de clones obtenue à partir de l’étape 9.4 (ou étape 9.5.5 après alignement) dans une orientation verticale avec la surface pial sur le dessus et le corpus callosum en bas. Rogner dans le plan xy si nécessaire.
- Pour la présentation qualitative et l’analyse quantitative, générer une image de projection z maximale (étape 9.8) ou effectuer un rendu 3D (étape 9.9) du clone.
- Dans ImageJ, ouvrez la pile d’images à partir de l’étape 9.6 et sélectionnez Option de projection Z avec intensité maximalede type de projection . Cela générera une image de l’ensemble du clone projeté sur le même plan.
- Dans ImageJ, ouvrez la pile d’images à partir de l’étape 9.6 et sélectionnez fonction z 3D Project pour générer une visualisation 3D du clone qui peut être tournée.
REMARQUE : Dans cette étape, il est important d’entrer l’intervalle de tranches correct équivalent à l’épaisseur des piles z individuelles lors de l’acquisition de l’image. L’outil d’interpolation doit être utilisé pour éliminer les écarts entre les tranches.
Résultats
MADM entraîne la reconstitution de protéines fluorescentes vertes et rouges fonctionnelles avec deux cellules filles exprimant chacune l’une des deux protéines fluorescentes lors d’événements de ségrégation chromosomique G2-X (Figure 1A). Étant donné que les événements MADM entraînent une étiquetage permanent et distinct des deux lignées descendantes, l’évaluation quantifiable des lignées de cellules de fille verte et rouge (subclones) peut être effectuée. Les variables, y compris le modèle de division (p. ex., symétrique par rapport à l’asymétrique) et le potentiel (p. ex., le nombre de descendants) de l’ancêtre d’origine peuvent être déterminées. La quantification de chaque subclone fluorescente est instructive lorsqu’on détermine rétroactivement si la cellule progénitrice d’origine subit des divisions prolifératives symétriques ou des divisions neurogéniques asymétriques au moment de l’induction de la TM. Des études antérieures ont regroupé emx1-CreerT2 ou Nestin-CreERT2 ont dérivé des clones de projection excitatoire dans le cortex en deux grandes classes7,11,46. Les premiers, appelés « clones prolifératifs symétriques », sont composés en moyenne d’un nombre considérable de neurones, avec des sous-clones verts et rouges contenant quatre neurones ou plus chacun. Le deuxième groupe, les « clones asymétriques », définit une classe de clones où le sous-clone « minoritaire » contient moins de trois neurones et le sous-clone « majoritaire », quatre ou plus11. Ces définitions sont spécifiques aux RPP corticaux et peuvent devoir être revues pour d’autres régions et tissus du cerveau. Pour les deux classes de clones corticaux, la descendance sera distribuée dans les couches superficielles et profondes.
Lors de la conception des études clonales MADM, il ya un certain nombre d’aspects qui doivent être pris en considération. Le moment où les événements MADM sont induits par l’administration de TM est une considération clé (Figure 3). Pour les clones de neurones de projection excitatoire corticale MADM (c.-à-d. en utilisant Emx1-CreERT2 ou Nestin-CreERT2)à E10, presque tous les RMP subissaient encore des divisions symétriques11. Par conséquent, l’induction à E10 avec TM capturé plusieurs tours d’amplification proliférative RGP et a abouti à des clones avec des nombres de neurones élevés. Cependant, le nombre de RMP à E10 était généralement faible et donc l’administration TM généré très peu d’événements MADM (parfois moins d’un par cerveau). La majorité des RPP sont passés de divisions symétriques à des divisions neurogéniques asymétriques aux environ e12. Pour cibler les clones neurogéniques strictement asymétriques, il était préférable d’induire à E12 ou plus tard (figure 3). Le temps entre l’induction de TM et l’observation des événements de recombinaison de MADM dans le cortex tendait à être inférieur à 24 h. Les injections de PI étaient la méthode préférée pour administrer la TM aux stades embryonnaires de cette méthode parce qu’elle a mené à une plus grande reproductibilité dans l’induction clonale. Il est également important de réduire au minimum la dose de TM pour deux raisons. Tout d’abord, si le taux de recombinaison MADM augmente, la probabilité d’induire des clones multiples, peut-être qui se chevauchent, est plus élevée. Deuxièmement, si trop de TM est livré, un taux accru d’avortement, de réabsorption d’embryon, et de plus petites tailles de litière peuvent être observés. Des avortements dans environ la moitié de tous les barrages enceintes ont été observés lorsque des injections de TM ont été livrées à l’E10. Cette fréquence a diminué à partir de l’E11 et a diminué à environ 1/3 des barrages enceintes avorter. Pour un résumé des doses de TM, des temps d’induction et des pilotes CreERT2 utilisés dans les études précédentes madm, reportez-vous au tableau 1. L’activité des journalistes en l’absence de TM a été observée avec certains conducteurs de TM-inductibles CreERT2 69. L’expression ectopique ou les événements de recombinaison MADM en l’absence de TM n’a pas été observé avec le Emx1-CreERT2 des pilotes Nestin-CreERT2. Cela peut s’expliquer en partie par le fait que les transcombinaisons chromosomiques sous médiation TM se produisent vers 1:1 000 à 1:10 000 fréquence inférieure à la cis-recombinaison, ce qui réduit la probabilité d’étiquetage ectopique MADM.
Un autre facteur à considérer lors de la planification d’une expérience d’analyse clonale MADM est la durée de l’étude. La variation de la durée entre l’induction tm et le moment où l’expérience a été analysée (A) (fenêtre temporelle) affiche la dynamique des cellules souches au fil du temps64. Fenêtres de temps embryonnaires courtes (c.-à-d. TM/E11−A/E13; TM/E11−A/E16) a capturé la dynamique de la neurogenèse embryonnaire (figure 4). La comparaison des clones à partir de deux fenêtres de temps ou plus fournit un aperçu quantitatif du nombre de cellules produites et de la façon dont la distribution des neurones varie à différents stades de la progression de la lignée64. Pour capturer l’ensemble du potentiel des clones individuels, il est nécessaire de prolonger la fenêtre de temps analysée dans les délais postnatals ou adultes7,11,12. Des exemples de clones néocortiques induits dans l’embryon et analysés chez l’adulte sont présentés à la figure 5. Il est à noter que la neurogenèse corticale est principalement terminée et la gliogenèse augmente par E17. Environ 1/6 RGP neurogénique procèdent également à la génération d’astrocytes et/ou d’oligodendrocytes11.
Les clones symétriques se produisent lorsque les RPP subissent un ou plusieurs tours de divisionproliférative 11. Les clones RGP induits entre E10−E12 étaient en moyenne de plus grande taille et fournissaient plus de caractéristiques spatiales de la distribution finale des neurones (figure 4A-C). Les clones avec des neurones relativement également répartis à travers les couches profondes et superficielles ont pris une forme de « cylindre » tandis que les clones avec des neurones plus dispersés dans les couches superficielles que les couches plus profondes ont développé une forme « cône »11. Pour capturer entièrement les informations spatiales et morphologiques d’un clone, il a été nécessaire de reconstruire chaque clone de manière computationnelle à l’aide d’images séquentielles. Pour mesurer la dispersion clonale, la dispersion latérale maximale (mesurée dans toutes les dimensions) dans les couches superficielles (LII−VI) d’un clone a été comparée à la dispersion des neurones dans les couches profondes (LV/LIV). Ce ratio (distribution supérieure:distribution inférieure) a fourni une lecture quantifiable de la forme globale du clone.
Les clones asymétriques, dont le sous-clone minoritaire était de trois ou moins, ont fourni un aperçu de la sortie neuronale d’un seul RGP (figure 4D-F et figure 5A-F)7,11,12. La population majoritaire (grand sous-clone) pourrait être étiquetée en rouge ou en vert, avec une moyenne d’environ sept neurones de projection excitateurs par clone lorsqu’elle est induite à l’aide d’un Emx1-CreERT2 ou Nestin-CreERT2(Figure 5G)7,11,12. Le nombre total de cellules dans un clone MADM pourrait être disséqué en analysant la distribution des neurones dans le grand subclone à travers les couches superficielles et profondes. La population minoritaire (petit sous-clone) a été étiquetée par la couleur réciproque et était en moyenne de 1 à 2 cellules par clone (figure 5H). La « taille totale de l’unité », qui était en moyenne de 8 à 9 neurones, pouvait être calculée en ajoutant les petites et grandes sous-clones ensemble (Figure 5I)7,11,12. Il est important de noter que si la production neuronale des RGP était très prévisible, il y avait un certain degré d’hétérogénéité clonale12,70.
L’introduction d’une mutation distale à la cassette MADM permet la génération de mosaïques génétiques, fournissant une méthode unique pour disséquer les régulateurs moléculaires de la progression de la lignée des cellules souches. En tant que tel, MADM fournit une plate-forme expérimentale inégalée pour étudier la fonction cellulaire autonome d’un gène (par exemple, son association à la microcéphalie ou à la macrocéphalie). En comparant les clones induits dans une mosaïque génétique MADM à des clones induits dans un MADM de contrôle, une lecture très quantitative des changements dans le nombre et la distribution des neurones peut être générée. Des études antérieures basées sur le MADM ont quantifié la fonction cellulaire autonome d’Otx1 dans la formation de microcéphalie au niveau clonal (voir la figure 6A-E pour un exemple représentatif)11. Dans une autre étude, l’analyse clonale de MADM a démontré que Ndel1 ne régule pas de façon autonome les nombres de neurones de projection, mais plutôt la capacité des neurones nouveau-nés à entrer ou à migrer dans la plaque corticale, qui forme plus tard le cortex adulte46. Ces études ont démontré la nature hautement quantitative de l’analyse clonale MADM dans l’étude des fonctions cellulaires autonomes des gènes régulant le développement cortical. Il n’y a actuellement aucun exemple dans la littérature utilisant MADM pour étudier des gènes impliqués dans la macrocéphalie au niveau clonal. Cependant, dans les études futures l’analyse des gènes pertinents pour le contrôle de la taille corticale en général peut fournir des aperçus hautement souhaitables au niveau moléculaire et cellulaire.

Figure 1 : Principe MADM pour le traçage des lignées et l’analyse clonale au niveau des cellules souches uniques. (A) Pour effectuer le traçage des lignées et l’analyse clonale avec MADM, deux composantes doivent être présentes. Tout d’abord, les cassettes MADM doivent être ciblées sur des loci identiques sur les chromosomes homologues. Les cassettes se composent de deux gènes de reporter fluorescent chimérique, eGFP (vert, [G]) et de la tomate de dimère tandem (rouge, tdT[T]). La cassette GT contient le n-terminus d’eGFP et le C-terminus de tdT, séparés par un intron contenant un site loxP. La cassette TG est construite inversement, avec le N-terminus de tdT et le C-terminus d’eGFP. Deuxièmement, l’expression de la recombinase de Cre doit se produire dans la cellule contenant les cassettes MADM ciblées. Les sites loxP servent de cible à la recombinaison interchromosomique à médiation cré, ce qui entraîne la reconstitution simultanée des deux cassettes d’expression. Si la recombinaison se produit pendant la phase G2 du cycle cellulaire suivie de la ségrégation X (G2-X), les deux cellules filles exprimeront l’une des deux protéines fluorescentes. (B) Principe MADM pour l’analyse génétique de mosaïque à un seul niveau de clone. Les allèles mutants (mutations ponctuelles, suppressions, insertions, allèles conditionnels flanqués loxP tels qu’ils sont représentés à la figure 1B,etc.) peuvent être introduits distal à la cassette TG-MADM via la recombinaison méiotique (voir figure 2 et Hippenmeyer et al.46 pour plus de détails sur la façon d’introduire les allèles mutants dans le système MADM). Si une transcombinaison interchromosomique préanée G2-X Cre se produit entre les cassettes MADM, il en résulte une cellule mutante GFP+ homozygotes (GeneX-/-) pour le gène d’intérêt et une cellule de type sauvage homozygote tdT+ (GeneX+/+) dans un environnement hétérozygote non étiqueté46,47,71. Les autres résultats d’étiquetage non utilisés dans l’analyse clonale (c.-à-d. les cellules jaunes) ont déjà été décrits en détail11,46,47. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
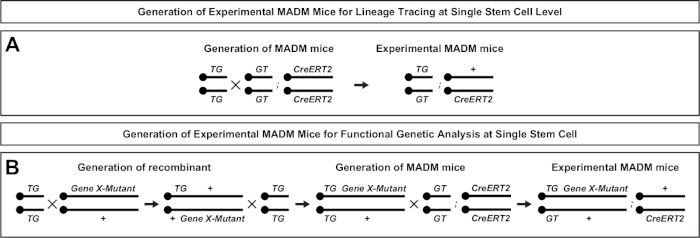
Figure 2 : Schémas de reproduction pour la génération de souris EXPÉRIMENTALEs MADM pour le traçage des lignées. Schéma de reproduction pour la génération de souris MADM (A) et Gene X MADM (B) expérimentales MADM pour l’analyse clonale. Pour plus d’informations sur les paradigmes d’élevage MADM, voir Beattie et coll.7 et Hippenmeyer et al.7,46. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Paradigmes de cours temporel pour l’analyse de lignée clonale basée sur MADM. Schéma des fenêtres de temps de conception expérimentale. Pour les paradigmes d’échantillonnage longitudinal, le temps d’induction du clone est demeuré constant et la durée avant l’analyse a varié. Dans l’échantillonnage progressif par intervalles, le temps d’analyse est demeuré constant, mais le temps d’induction variait. Une combinaison d’une ou des deux approches peut être utilisée en fonction des questions abordées. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Analyse clonale MADM dans le néocortex en développement et adulte. Induction de clone MADM à médiation TM en prolifération symétrique (TM à E10) (A-C) et asymétriquement neurogénique (TM à E12) (D-F) divisant les RGG. Sont représentés des clones MADM individuels in vivo dans les développements (TM/E10−A/E16 et TM/E12−A/E16) (B,E) et adultes (TM/E10−A/P21 et TM/E12−A/P21) (C,F) dans MADM-11GT/TG; Nestin-CreERT2+/- (B,E) et MADM-11GT/TG; Emx1-CreERT2+/- (C,F). La sortie des neurones était indépendante de la couleur subclone et les sous-clones de majorité/minorité verte pouvaient être comparés aux sous-clones de majorité/minorité rouge dans des conditions de contrôle7,11. Environ 1/6 des clones adultes contenaient également des astrocytes et/ou des oligodendrocytes, indiqués par des astérisques blancs. Les panneaux B et F sont reproduits avec la permission d’Hippenmeyer et coll.46 et Rulands et Simons72, respectivement. CP = Plaque corticale. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
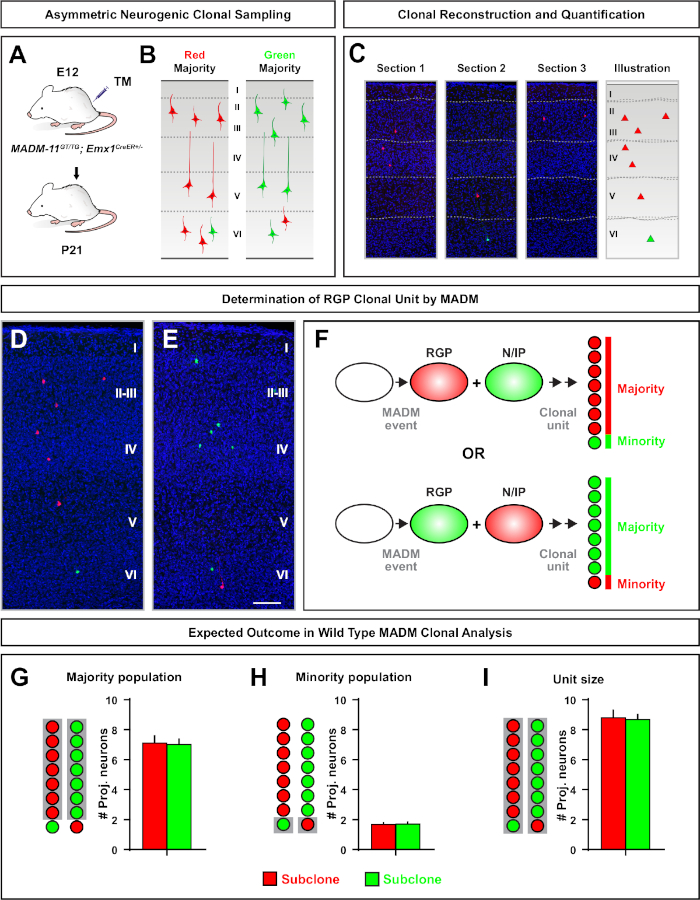
Figure 5 : Analyse clonale MADM pour quantifier la sortie des neurones médiés par le RGP. Analyse de la production de neurones excitateurs (unité) par des RMP neurogéniques individuels au niveau clonal à l’aide de MADM7,11. (A) Paradigme expérimental pour induire principalement des clones madm asymétriques dans le cortex en développement. (B) Résultats possibles de clone asymétrique avec la sous-clone majoritaire étiquetée en vert ou rouge (C) Sections consécutives représentatives couvrant un clone asymétrique neurogénique unique (D,E) images de reconstruction 3D de clones représentatifs asymétriques G2-X MADM avec population majoritaire en rouge (D) ou vert (E) dans MADM-11GT/TG; Emx1-CreERT2+/- avec induction TM à e12 et analyse à P21. Notez que les cellules étiquetées vertes et rouges sont de type sauvage. (F) Schéma indiquant les deux résultats expérimentaux possibles de clone de MADM. (G) Quantification de la taille de la population majoritaire résultant du renouvellement des RGP dans les clones MADM-11. (H) Quantification de la taille de la population minoritaire résultant du renouvellement des RGP dans les clones MADM-11. II) Quantification de la taille unitaire des clones nocurgies asymétriques de MADM-11. Les valeurs hypothétiques pourraient représenter la moyenne ± SEM. Barre d’échelle = 100 μm (D et E). TM = Tamoxifène. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
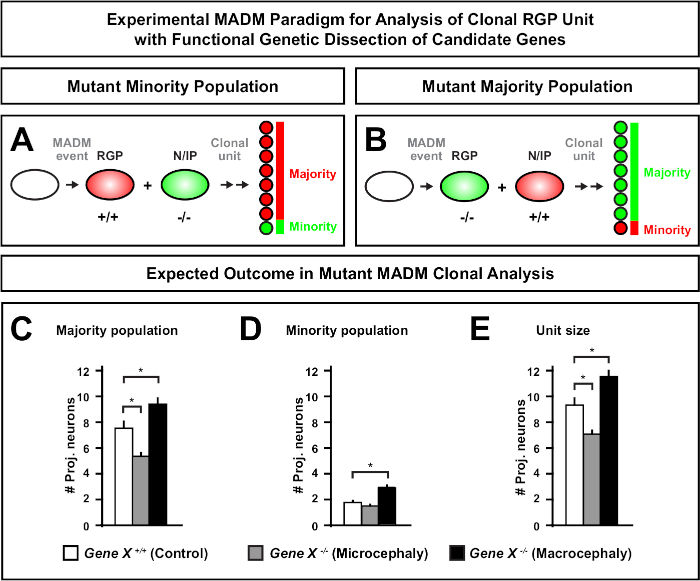
Figure 6 : Analyse clonale madm pour étudier les gènes qui mènent à la microcéphalie et à la macrocéphalie. Résultats hypothétiques d’analyse clonale de MADM lors de l’exécution de la dissection génétique fonctionnelle des gènes candidats qui mènent à la microcéphalie ou à la macrocéphalie. Pour disséquer les fonctions cellulaires autonomes d’un gène d’intérêt (Gene X) sur la sortie des neurones, MADM exige que les allèles mutants soient introduits distal dans les cassettes MADM via la recombinaison méiotique (pour plus de détails comment introduire les allèles mutants dans le système MADM voir aussi figure 2, Hippenmeyer et al.46, et Laukoter et al.46,73). (A,B) Schématique indiquant le paradigme expérimental de MADM pour l’analyse fonctionnelle des unités clonales de RGP. Le subclone mutant peut former la population minoritaire (A) ou majoritaire (B). (C-E) Résultats hypothétiques de l’analyse clonale MADM lors de la quantification du contrôle MADM (barres blanches), de la microcéphalie gene-X MADM (barres grises) et des barres noires de macrocéphalie de Gene-X MADM). (C) Quantification de la taille de la population majoritaire. (D) Quantification de la taille de la population minoritaire. EE) Quantification de la taille unitaire des clones neurogéniques asymétriques. Les valeurs hypothétiques pourraient représenter la moyenne ± SEM. S = Scénario hypothétique où la différence dans le nombre de cellules subclones pourrait atteindre l’importance, par rapport au contrôle. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Tableau 1 : Études clonales MADM dans la littérature. Résumé des études dans la littérature contenant des expériences de lignée clonale MADM, y compris le conducteur de CreERT2 utilisé, la dose de TM, et le temps d’injection. Veuillez cliquer ici pour voir ce tableau (cliquez avec le bouton droit pour télécharger).
Discussion
Une méthode d’utilisation de MADM pour suivre la lignée cellulaire des RGPs individuels in vivo dans le néocortex en développement est décrite. Lorsqu’ils sont combinés avec TM-inductible CreERT2, les événements MADM peuvent être chronométrés avec précision, fournissant une lecture visuelle très qualitative et quantitative des modèles de division de cellules souches au niveau des cellules uniques. En titrant la dose de TM livrée, dans une situation idéale, on peut obtenir en moyenne moins d’un clone par hémisphère cortical, ce qui permet d’obtenir une séparation spatiale adéquate pour distinguer sans ambiguïté les clones individuels. En maintenant l’intégrité des tissus, cette méthode capture également des informations essentielles concernant la position, la morphologie et les nombres absolus de cellules. Cassettes MADM sur Chr. 117,11,12,46,56,57, sur Chr. 751, et le MADM original à Rosa2647,53,,59 ont été utilisés dans les études d’analyse clonale MADM. La haute résolution des cellules individuelles fournit un aperçu sans précédent de la morphologie et de la relation clonale des cellules filles et permet l’imagerie vivante des cellules souches proliférantes et des clones émergents46,52.
La césarienne et la promotion des chiots pour l’analyse des clones aux délais postnatals est une étape nécessaire et critique dans le protocole. Selon l’état de santé du barrage enceinte traité par TM, il peut ne pas être nécessaire d’effectuer une césarienne. Cependant, il est encore nécessaire d’élever les chiots avec une mère adoptive, car la mère traitée au TM peut avoir de la difficulté à allaiter. Aucune différence n’a été observée dans la nécessité de favoriser avec différents conducteurs CreERT2. Les lignes MADM et les mères adoptives sont maintenues sur un fond de CD-1 hors-de-race. Si la césarienne n’est pas nécessaire, le barrage enceinte traité par TM utilisé pour produire des chiots expérimentaux peut être réutilisé pour d’autres reproductions expérimentales conformément aux principes de la 3R (notez que cela ne peut être fait que si les licences expérimentales animales approuvent cette pratique). Les mères adoptives peuvent être utilisées pour favoriser les chiots dans les 2 jours suivant leur accouchement, mais des taux de réussite plus élevés ont été observés lorsque les mères adoptives accouchent le même jour que les souris expérimentales qui doivent être encouragées. Par conséquent, il est important de mettre en place des accouplements chronométrés pour les mères adoptives en parallèle aux accouplements expérimentaux à l’étape 1.1. Le maintien d’un numéro de litière semblable à celui de la litière de la mère adoptive peut améliorer le taux de survie des chiots placés en famille d’accueil, et il peut donc être nécessaire d’enlever certains de ceux-ci à toutes les portées d’origine. D’autres mesures qui peuvent améliorer l’accueil comprennent frotter les gants de l’expérimentateur avec de la litière et de la nourriture (pour enlever l’odeur des gants); frotter les chiots doucement après la césarienne avec des fragments de la litière et du nid souillés de la mère adoptive; et le placement des chiots en contact étroit avec les chiots de la mère adoptive avant leur placement dans la cage de la souris d’accueil.
Comme dans d’autres méthodes de traçage de lignée basées sur le journaliste, il faut examiner attentivement lors du choix du pilote CreERT2 optimal pour les expériences clonales MADM. Tout d’abord, le promoteur utilisé doit exprimer la recombinase à la fois temporellement et spatialement dans la population progénitrice d’intérêt. Trouver ce promoteur peut être difficile, parce que certains promoteurs peuvent changer les modèles d’expression ou se réduire au silence à différents stades de développement. Pour améliorer la spécificité du type de cellule, plusieurs recombinas spécifiques au site, chacune pilotée par des promoteurs distincts, ont été utilisées. Lorsque l’une ou les deux recombinases sont exprimées dans la même cellule, cela étiquette la cellule et sa progéniture avec un journaliste fluorescent74,75,76,77. En résumé, il est important de choisir un pilote CreERT2 spécifique à la population d’ancêtres analysés.
L’étape la plus critique de cette méthode est l’identification d’un clone, car toutes les cellules doivent être dérivées sans ambiguïté d’un seul événement de recombinaison (étape 8.1). La titration de la concentration de TM assure moins d’un groupe de cellules rouges/vertes par hémisphère cérébral et maximise la probabilité d’analyser un seul clone (étape 2.2)7,11. Les clones doivent être jetés si les amas voisins de cellules se produisent à moins de 500 μm du clone d’intérêt. Par conséquent, il est important d’examiner plusieurs sections avant et après l’apparition d’un clone pour s’assurer qu’il n’y a pas d’événements de recombinaison supplémentaires à proximité. En raison du signal plus faible des fluorophores, il est nécessaire d’effectuer l’immunohistorique pour eGFP et tdT dans les clones embryonnaires (voir la section 6). Ceci n’est recommandé que chez les clones adultes que si des antigènes supplémentaires seront colabés. Lors de l’imagerie des clones, il est important de capturer toute la largeur du cortex où se trouve le clone (c.-à-d. de la surface piale au corpus callosum; voir l’étape 8.4) pour ne manquer aucune cellule. Cela facilite également l’alignement de l’image pendant le traitement de l’image (section 9). La section 8 du protocole nécessite un microscope confocal inversé, mais peut être adaptée en fonction de la configuration du microscope disponible. La microscopie d’épifluorescence peut être employée, mais la microscopie confocale est recommandée parce que ceci mène à une diminution de la contamination de lumière de l’extérieur du plan de mise au point. Il est également important que l’intensité et le gain du laser soient ajustés de façon à ce que les cellules vertes, rouges et jaunes puissent être identifiées sans ambiguïté. Indépendamment de la configuration, il est recommandé d’utiliser un objectif d’au moins 20x pour assurer une séparation spatiale complète des cellules étroitement positionnées. En plus d’enregistrer la profondeur corticale de toutes les cellules (étape 8.6), les régions corticales où se trouvent les clones doivent être identifiées à l’aide d’un atlas cérébral tel que l’atlas du cerveau Allen ou d’autres cartes de coordonnées stéréotaxiques. Un paradigme de nommage de fichier devrait également être adopté pour s’assurer que les images de clones sont facilement identifiables. Les informations suivantes pourraient être incluses dans le nom du fichier : ID d’image unique, image de date a été prise, génotype de l’animal, âge d’induction, âge d’analyse, numéro d’image par rapport au reste des images du même clone.
L’introduction d’une mutation distale à une cassette MADM permet distinctement la génération de mosaïques génétiques71 et permet la dissection des régulateurs moléculaires de la diversité de lignée et de type cellulaire au niveau clonal7,11,46,62. Pour générer une mosaïque génétique avec MADM, les cassettes MADM doivent être liées de façon meiotiquement au même chromosome que le gène d’intérêt (voir la figure 2 pour le schéma de reproduction). Cela limite l’analyse clonale actuelle avec MADM aux gènes situés sur Chr. 751, Chr. 1146, Chr. 1251, et Chr. 6 distal au rosa26 locus47. Les études futures utiliseront des cassettes MADM ciblées sur n’importe quel chromosome, permettant l’analyse en mosaïque de pratiquement tous les gènes du génome de la souris au niveau clonal.
Enfin, MADM ne se limite pas à l’analyse des cellules progénitrices du néocortex en développement. L’étude de nombreuses niches de cellules souches pourrait bénéficier de la capacité de résoudre les arrangements spatiotemporal des cellules clonales. En appliquant MADM à d’autres régions du cerveau, les maladies (par exemple, le cancer), ou dans d’autres tissus47,50,51,52,53,54,55,56,57,58,59,études ont révélé des relations de lignée dans des clones dérivés de diverses classes de progénitrice et de cellules souches (voir le tableau 1 pour la liste actuelle des études clonalales MADM). Une autre application intéressante future de MADM est de le combiner avec des journalistes fonctionnels ou subcellulaires supplémentaires, ce qui augmenterait le degré d’information qui peut être acquise à partir de clones.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions tous les membres du laboratoire Hippenmeyer pour leur discussion, le Centre de bioimagerie, le Centre des sciences de la vie et l’Installation préclinique de l’IST Autriche pour leur soutien technique. Ce travail a été soutenu par les fonds institutionnels de l’IST Autriche; R.B. a reçu le soutien du programme Lise-Meitner du Fonds autrichien pour la science (FWF) (M 2416); N.A a reçu le soutien du Fonds autrichien pour la science (FWF) Firnberg-Programm (T 1031); GC a reçu le soutien du programme de recherche et d’innovation Horizon 2020 de l’Union européenne dans le cadre de l’accord de subvention Marie Skłodowska-Curie no 754411 en tant que boursier postdoctoral ISTplus; A.H. a reçu le soutien d’un ÖAW DOC (Bourse doctorale de l’Académie autrichienne des sciences). Cette étude a également été soutenue par le Conseil européen de la recherche (CER) dans le cadre du programme de recherche et d’innovation Horizon 2020 de l’Union européenne (accord de subvention n° 725780 LinPro) à S.H.
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL tuberculin syringe (Omnifix Luer Lock) | Braun | 9204512N | |
| 1,4-diazabicyclooctane (DABCO) | Roth | 0718.2 | |
| 10 mL Syringe (Omnifix Luer Lock) | Braun | 8508429N | |
| 15 mL conical centrifuge | Sarstedt | 65.554.502 | |
| 24 multi-well dishes | Roth/Greiner Bio-one | CE56.1 | |
| 27- gauge x 3/4 needle (Sterican) | Braun | 16010256E | |
| Corn oil | Sigma | C8267-500ML | |
| Coverslips (24 x 60 mm #1) | Thermo Fisher Scientific (Menzel) | 15747592 | |
| Cryostat Cryostar NX70 | Thermo Fisher Scientific | 957000H | |
| Dako Pen (Wax marker) | Agilent | S200230-2 | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Invitrogen | D1306 | |
| Disposable microtome blade (MX35 Ultra) | Thermo Fisher Scientific | 705830 | |
| Fine Forceps (Dumont #5) | Fine Science Tools (FST) | 11254-20 | |
| Glass anti-roll plate | Histocom | M 449980 | |
| Glycerol | Sigma | G5516 | |
| LSM 800 Confocal | Zeiss | ||
| Mounting medium | 25 mg/mL DAPCO, 6 g Glycerol, 2.4 g Mowiol 4-88, 6 mL dH2O, 12 mL 0.2 M Tris-HCl (pH 8.5) | ||
| Mowiol 4-88 | Roth | 0713.2 | |
| Normal donkey serum | Innovative Research | IGDNSER100ML | |
| Paraformaldehyde | Sigma | 441244-1KG | |
| Peristaltic pump 323E/D 400RPM | Watson-Marlow | 036.3124.00A | |
| Sucrose | Sigma | S8501-5KG | |
| Superfrost plus glass slides | Thermo Fisher Scientific | J1800AMNT | |
| Tamoxifen | Sigma | T5648 | |
| Tissue Embedding mold T-12 (22mm square) | Polysciences Inc. | 18986-1 | |
| Tissue-Tek O.C.T | Sakura | 4583 | |
| Triton X-100 | Sigma | T8787-250ML | |
| Trizma hydrochloride | Sigma | 93363 | |
| Tween-20 | Sigma | P9416-100ML | |
| Software and Plugins: | |||
| Fiji | 1.52p | Fiji | |
| MultiStackReg | 1.45 | Download link | |
| TurboReg | EPFL Bioimaging | ||
| Zen Blue | 2.6 | Zeiss | |
| Experimental Models: Organisms/Strains: | |||
| Mouse: Emx1-CreER | The Jackson Laboratory | JAX:027784 | |
| Mouse: MADM-11-GT | The Jackson Laboratory | JAX:013749 | |
| Mouse: MADM-11-TG | The Jackson Laboratory | JAX:013751 | |
| Primary antibodies: | |||
| Chicken anti-GFP 1:500 | Aves Labs | GFP-1020 | |
| Goat anti-tdTomato 1:500 | Sicgen Antibodies | AB8181-200 | |
| Rabbit anti-RFP 1:500 | MBL | PM005 | |
| Secondary antibodies: | |||
| Donkey Anti-Chicken Alexa Fluor 488 1:500 | Jackson Immuno Research | 715-475-150 | |
| Donkey Anti-Goat Cy3 1:500 | Jackson Immuno Research | 705-165-147 | |
| Donkey Anti-Rabbit Cy3 1:500 | Jackson Immuno Research | 711-165-152 |
Références
- Malatesta, P., et al. Neuronal or glial progeny: regional differences in radial glia fate. Neuron. 37 (5), 751-764 (2003).
- Miyata, T., Kawaguchi, A., Okano, H., Ogawa, M. Asymmetric inheritance of radial glial fibers by cortical neurons. Neuron. 31 (5), 727-741 (2001).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30 (1), 465-502 (2014).
- Desikan, R. S., Barkovich, A. J. Malformations of cortical development. Annals of Neurology. 80 (6), 797-810 (2016).
- Gao, R., Penzes, P. Common mechanisms of excitatory and inhibitory imbalance in schizophrenia and autism spectrum disorders. Current Molecular Medicine. 15 (2), 146-167 (2015).
- Beattie, R., et al. Mosaic Analysis with Double Markers Reveals Distinct Sequential Functions of Lgl1 in Neural Stem Cells. Neuron. 94 (3), 517-533 (2017).
- Hippenmeyer, S. Molecular pathways controlling the sequential steps of cortical projection neuron migration. Advances in Experimental Medicine and Biology. 800, 1-24 (2014).
- Lodato, S., Arlotta, P. Generating neuronal diversity in the mammalian cerebral cortex. Annual Review of Cell and Developmental Biology. 31, 699-720 (2015).
- Hansen, A. H., Duellberg, C., Mieck, C., Loose, M., Hippenmeyer, S. Cell Polarity in Cerebral Cortex Development-Cellular Architecture Shaped by Biochemical Networks. Frontiers in Cellular Neuroscience. 11, 176 (2017).
- Gao, P., et al. Deterministic progenitor behavior and unitary production of neurons in the neocortex. Cell. 159 (4), 775-788 (2014).
- Llorca, A., et al. A stochastic framework of neurogenesis underlies the assembly of neocortical cytoarchitecture. eLife. 8, e51381 (2019).
- Ma, J., Shen, Z., Yu, Y. C., Shi, S. H. Neural lineage tracing in the mammalian brain. Current Opinion in Neurobiology. 50, 7-16 (2018).
- Caviness, V., Takahashi, T., Nowakowski, R. Numbers, time and neocortical neuronogenesis: a general developmental and evolutionary model. Trends in Neurosciences. 18 (9), 379-383 (1995).
- Schmechel, D. E., Rakic, P. A Golgi study of radial glial cells in developing monkey telencephalon: morphogenesis and transformation into astrocytes. Anatomy and Embryology. 156 (2), 115-152 (1979).
- Kessaris, N., et al. Competing waves of oligodendrocytes in the forebrain and postnatal elimination of an embryonic lineage. Nature Neuroscience. 9 (2), 173-179 (2006).
- Magavi, S., Friedmann, D., Banks, G., Stolfi, A., Lois, C. Coincident generation of pyramidal neurons and protoplasmic astrocytes in neocortical columns. The Journal of Neuroscience. 32 (14), 4762-4772 (2012).
- Anthony, T. E., Klein, C., Fishell, G., Heintz, N. Radial glia serve as neuronal progenitors in all regions of the central nervous system. Neuron. 41 (6), 881-890 (2004).
- Voigt, T. Development of glial cells in the cerebral wall of ferrets: direct tracing of their transformation from radial glia into astrocytes. The Journal of Comparative Neurology. 289 (1), 74-88 (1989).
- Amberg, N., Laukoter, S., Hippenmeyer, S. Epigenetic cues modulating the generation of cell-type diversity in the cerebral cortex. Journal of Neurochemistry. 149 (1), 12-26 (2019).
- Beattie, R., Hippenmeyer, S. Mechanisms of Radial Glia Progenitor Cell Lineage Progression. FEBS letters. 591 (24), 3993-4008 (2017).
- Telley, L., et al. Temporal patterning of apical progenitors and their daughter neurons in the developing neocortex. Science. 364 (6440), eaav2522 (2019).
- Oberst, P., et al. Temporal plasticity of apical progenitors in the developing mouse neocortex. Nature. 573 (7774), 370-374 (2019).
- Telley, L., et al. Sequential transcriptional waves direct the differentiation of newborn neurons in the mouse neocortex. Science. 351 (6280), 1443 (2016).
- Deppe, U., et al. Cell lineages of the embryo of the nematode Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 75 (1), 376-380 (1978).
- Woodworth, M. B., Girskis, K. M., Walsh, C. A. Building a lineage from single cells: genetic techniques for cell lineage tracking. Nature Reviews Genetics. 18 (4), 230-244 (2017).
- Masuyama, N., Mori, H., Yachie, N. DNA barcodes evolve for high-resolution cell lineage tracing. Current Opinion in Chemical Biology. 52, 63-71 (2019).
- Legue, E., Joyner, A. L. Chapter Ten-Genetic Fate Mapping Using Site-Specific Recombinases. Methods in Enzymology. 477, 153-181 (2010).
- Postiglione, M. P., Hippenmeyer, S. Monitoring neurogenesis in the cerebral cortex: an update. Future Neurology. 9 (3), 323-340 (2014).
- Espinosa-Medina, I., Garcia-Marques, J., Cepko, C., Lee, T. High-throughput dense reconstruction of cell lineages. Open Biology. 9 (12), 190229 (2019).
- Hwang, B., et al. Lineage tracing using a Cas9-deaminase barcoding system targeting endogenous L1 elements. Nature Communications. 10 (1), 1234 (2019).
- Kalhor, R., et al. Developmental barcoding of whole mouse via homing CRISPR. Science. 361 (6405), eaat9804 (2018).
- García-Moreno, F., Vasistha, N. A., Begbie, J., Molnár, Z. CLoNe is a new method to target single progenitors and study their progeny in mouse and chick. Development. 141 (7), 1589-1598 (2014).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Snippert, H. J., et al. Intestinal Crypt Homeostasis Results from Neutral Competition between Symmetrically Dividing Lgr5 Stem Cells. Cell. 143 (1), 134-144 (2010).
- Amitai-Lange, A., et al. A method for lineage tracing of corneal cells using multi-color fluorescent reporter mice. Journal of Visualized Experiments. (106), e53370 (2015).
- Vasistha, N. A., et al. Cortical and Clonal Contribution of Tbr2 Expressing Progenitors in the Developing Mouse Brain. Cerebral Cortex. 25 (10), 3290-3302 (2015).
- Chen, F., LoTurco, J. A method for stable transgenesis of radial glia lineage in rat neocortex by piggyBac mediated transposition. Journal of Neuroscience Methods. 207 (2), 172-180 (2012).
- Siddiqi, F., et al. Fate mapping by piggyBac transposase reveals that neocortical GLAST+ progenitors generate more astrocytes than Nestin+ progenitors in rat neocortex. Cerebral Cortex. 24 (2), 508-520 (2014).
- Yoshida, A., et al. Simultaneous expression of different transgenes in neurons and glia by combining in utero electroporation with the Tol2 transposon-mediated gene transfer system. Genes to Cells. 15 (5), 501-512 (2010).
- García-Marqués, J., López-Mascaraque, L. Clonal Identity Determines Astrocyte Cortical Heterogeneity. Cerebral Cortex. 23 (6), 1463-1472 (2013).
- Clavreul, S., et al. Cortical astrocytes develop in a plastic manner at both clonal and cellular levels. Nature Communications. 10 (1), 4884 (2019).
- Kim, G. B., et al. Rapid Generation of Somatic Mouse Mosaics with Locus-Specific, Stably Integrated Transgenic Elements. Cell. 179 (1), 251-267 (2019).
- Pilz, G. A., et al. Live imaging of neurogenesis in the adult mouse hippocampus. Science. 359 (6376), 658 (2018).
- Hippenmeyer, S., et al. Genetic Mosaic Dissection of Lis1 and Ndel1 in Neuronal Migration. Neuron. 68 (4), 695-709 (2010).
- Zong, H., Espinosa, J. S., Su, H. H., Muzumdar, M. D., Luo, L. Mosaic analysis with double markers in mice. Cell. 121 (3), 479-492 (2005).
- Ramón y Cajal, S. . Histologie du système nerveux de l'homme et des vertébrés. , (1911).
- Cowan, W. M. The emergence of modern neuroanatomy and developmental neurobiology. Neuron. 20 (3), 413-426 (1998).
- Ali, S. R., et al. Existing cardiomyocytes generate cardiomyocytes at a low rate after birth in mice. Proceedings of the National Academy of Sciences of the United States of America. 111 (24), 8850-8855 (2014).
- Hippenmeyer, S., Johnson, R. L., Luo, L. Mosaic analysis with double markers reveals cell-type-specific paternal growth dominance. Cell Reports. 3 (3), 960-967 (2013).
- Riccio, P., Cebrian, C., Zong, H., Hippenmeyer, S., Costantini, F. Ret and Etv4 promote directed movements of progenitor cells during renal branching morphogenesis. PLoS Biology. 14 (2), e1002382 (2016).
- Bonaguidi, M. A., et al. In vivo clonal analysis reveals self-renewing and multipotent adult neural stem cell characteristics. Cell. 145 (7), 1142-1155 (2011).
- Mayer, C., et al. Clonally Related Forebrain Interneurons Disperse Broadly across Both Functional Areas and Structural Boundaries. Neuron. 87 (5), 989-998 (2015).
- Muzumdar, M. D., et al. Clonal dynamics following p53 loss of heterozygosity in Kras-driven cancers. Nature Communications. 7, 12685 (2016).
- Shi, W., et al. Ontogenetic establishment of order-specific nuclear organization in the mammalian thalamus. Nature Neuroscience. 20, 516 (2017).
- Wong, S. Z. H., et al. In vivo clonal analysis reveals spatiotemporal regulation of thalamic nucleogenesis. PLoS Biology. 16 (4), e2005211 (2018).
- Xu, H. T., et al. Distinct Lineage-Dependent Structural and Functional Organization of the Hippocampus. Cell. 157 (7), 1552-1564 (2014).
- Brennand, K., Huangfu, D., Melton, D. All β Cells Contribute Equally to Islet Growth and Maintenance. PLoS Biology. 5 (7), e163 (2007).
- Ortiz-Alvarez, G., et al. Adult neural stem cells and multiciliated ependymal cells share a common lineage regulated by the geminin family members. Neuron. 102 (1), 159-172 (2019).
- Kaplan, E. S., Ramos-Laguna, K. A., Mihalas, A. B., Daza, R. A. M., Hevner, R. F. Neocortical Sox9+ radial glia generate glutamatergic neurons for all layers, but lack discernible evidence of early laminar fate restriction. Neural Development. 12 (1), 14 (2017).
- Lv, X., et al. TBR2 coordinates neurogenesis expansion and precise microcircuit organization via Protocadherin 19 in the mammalian cortex. Nature Communications. 10 (1), 3946 (2019).
- Mihalas, A. B., Hevner, R. F. Clonal analysis reveals laminar fate multipotency and daughter cell apoptosis of mouse cortical intermediate progenitors. Development. 145 (17), dev164335 (2018).
- Picco, N., et al. A mathematical insight into cell labelling experiments for clonal analysis. Journal of Anatomy. 235 (3), 687-696 (2019).
- Johnson, C. A., Ghashghaei, H. T. Sp2 regulates late neurogenic but not early expansive divisions of neural stem cells underlying population growth in the mouse cortex. Development. , (2020).
- Espinosa, J. S., Luo, L. Timing Neurogenesis and Differentiation: Insights from Quantitative Clonal Analyses of Cerebellar Granule Cells. The Journal of Neuroscience. 28 (10), 2301 (2008).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Liu, Y., et al. Tamoxifen-independent recombination in the RIP-CreER mouse. PLoS One. 5 (10), e13533 (2010).
- Klingler, E., Jabaudon, D. Do progenitors play dice?. eLife. 9, e54042 (2020).
- Hippenmeyer, S. Dissection of gene function at clonal level using mosaic analysis with double markers. Frontiers in Biology. 8 (6), 557-568 (2013).
- Rulands, S., Simons, B. D. Tracing cellular dynamics in tissue development, maintenance and disease. Current Opinion in Cell Biology. 43, 38-45 (2016).
- Laukoter, S., et al. Imprinted Cdkn1c genomic locus cell-autonomously promotes cell survival in cerebral cortex development. Nature Communications. 11 (1), 195 (2020).
- Daigle, T. L., et al. A Suite of Transgenic Driver and Reporter Mouse Lines with Enhanced Brain-Cell-Type Targeting and Functionality. Cell. 174 (2), 465-480 (2018).
- He, M., et al. Strategies and Tools for Combinatorial Targeting of GABAergic Neurons in Mouse Cerebral Cortex. Neuron. 91 (6), 1228-1243 (2016).
- Yamamoto, M., et al. A multifunctional reporter mouse line for Cre- and FLP-dependent lineage analysis. Genesis. 47 (2), 107-114 (2009).
- Plummer, N. W., et al. Expanding the power of recombinase-based labeling to uncover cellular diversity. Development. 142 (24), 4385 (2015).
- Imayoshi, I., Ohtsuka, T., Metzger, D., Chambon, P., Kageyama, R. Temporal regulation of Cre recombinase activity in neural stem cells. Genesis. 44 (5), 233-238 (2006).
- Sasaki, S., et al. Complete loss of Ndel1 results in neuronal migration defects and early embryonic lethality. Molecular and Cellular Biology. 25 (17), 7812-7827 (2005).
- Acampora, D., et al. Epilepsy and brain abnormalities in mice lacking the Otx1 gene. Nature Genetics. 14 (2), 218-222 (1996).
- Soeda, T., et al. Sox9-expressing precursors are the cellular origin of the cruciate ligament of the knee joint and the limb tendons. Genesis. 48 (11), 635-644 (2010).
- Klezovitch, O., Fernandez, T. E., Tapscott, S. J., Vasioukhin, V. Loss of cell polarity causes severe brain dysplasia in Lgl1 knockout mice. Genes & Development. 18 (5), 559-571 (2004).
- Pimeisl, I. M., et al. Generation and characterization of a tamoxifen-inducible EomesCreER mouse line. Genesis. 51 (10), 725-733 (2013).
- Nakagawa, N., et al. Memo1-Mediated Tiling of Radial Glial Cells Facilitates Cerebral Cortical Development. Neuron. 103 (5), 836-852 (2019).
- Nowotschin, S., et al. The T-box transcription factor Eomesodermin is essential for AVE induction in the mouse embryo. Genes & Development. 27 (9), 997-1002 (2013).
- Balordi, F., Fishell, G. Mosaic removal of hedgehog signaling in the adult SVZ reveals that the residual wild-type stem cells have a limited capacity for self-renewal. Journal of Neuroscience. 27 (52), 14248-14259 (2007).
- Liang, H., et al. Neural development is dependent on the function of specificity protein 2 in cell cycle progression. Development. 140 (3), 552-561 (2013).
- Guo, C., Yang, W., Lobe, C. G. A Cre recombinase transgene with mosaic, widespread tamoxifen-inducible action. Genesis. 32 (1), 8-18 (2002).
- Ahn, S., Joyner, A. L. In vivo analysis of quiescent adult neural stem cells responding to Sonic hedgehog. Nature. 437 (7060), 894-897 (2005).
- Storm, R., et al. The bHLH transcription factor Olig3 marks the dorsal neuroepithelium of the hindbrain and is essential for the development of brainstem nuclei. Development. 136 (2), 295-305 (2009).
- Koundakjian, E. J., Appler, J. L., Goodrich, L. V. Auditory neurons make stereotyped wiring decisions before maturation of their targets. Journal of Neuroscience. 27 (51), 14078-14088 (2007).
- Dor, Y., Brown, J., Martinez, O. I., Melton, D. A. Adult pancreatic β-cells are formed by self-duplication rather than stem-cell differentiation. Nature. 429 (6987), 41-46 (2004).
- Sohal, D. S., et al. Temporally regulated and tissue-specific gene manipulations in the adult and embryonic heart using a tamoxifen-inducible Cre protein. Circulation Research. 89 (1), 20-25 (2001).
- Ventura, A., et al. Restoration of p53 function leads to tumour regression in vivo. Nature. 445 (7128), 661-665 (2007).
- Johnson, L., et al. Somatic activation of the K-ras oncogene causes early onset lung cancer in mice. Nature. 410 (6832), 1111-1116 (2001).
- Tasic, B., et al. Extensions of MADM (mosaic analysis with double markers) in mice. PLoS One. 7 (3), e33332 (2012).
- Yu, J., Carroll, T. J., McMahon, A. P. Sonic hedgehog regulates proliferation and differentiation of mesenchymal cells in the mouse metanephric kidney. Development. 129 (22), 5301-5312 (2002).
- Zhao, H., et al. Role of fibroblast growth factor receptors 1 and 2 in the ureteric bud. Developmental Biology. 276 (2), 403-415 (2004).
- Schuchardt, A., D'Agati, V., Larsson-Blomberg, L., Costantini, F., Pachnis, V. Defects in the kidney and enteric nervous system of mice lacking the tyrosine kinase receptor Ret. Nature. 367 (6461), 380-383 (1994).
- Livet, J., et al. ETS gene Pea3 controls the central position and terminal arborization of specific motor neuron pools. Neuron. 35 (5), 877-892 (2002).
- Volz, K. S., et al. Pericytes are progenitors for coronary artery smooth muscle. eLife. 4, e10036 (2015).
- Cai, C. L., et al. A myocardial lineage derives from Tbx18 epicardial cells. Nature. 454 (7200), 104-108 (2008).
- Lasrado, R., et al. Lineage-dependent spatial and functional organization of the mammalian enteric nervous system. Science. 356 (6339), 722-726 (2017).
- Matsuoka, T., et al. Neural crest origins of the neck and shoulder. Nature. 436 (7049), 347-355 (2005).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon