Method Article
Lineage Tracing und Clonal Analysis bei der Entwicklung von Zerebralparese mit Mosaikanalyse mit Doppelmarkern (MADM)
In diesem Artikel
Zusammenfassung
Es wird ein Protokoll zur Verfolgung und funktionellen genetischen Analyse von Kandidatengenen auf Einzelzellebene mithilfe von Mosaikanalysen mit Doppelmarkern (MADM) vorgestellt. Die CLonale MADM-Analyse bietet einen quantitativen Rahmen, um das proliferative Verhalten, die zelluläre Leistung und die Abstammungsbeziehung einzelner Vorfahren und ihrer Tochterzellen zu messen.
Zusammenfassung
Ausgehend von einem begrenzten Pool von Vorläufern bildet die Säugetiergroßrinde hochorganisierte funktionelle neuronale Schaltkreise. Die zugrunde liegenden zellulären und molekularen Mechanismen, die Linienübergänge neuronaler Stammzellen (NSCs) und die letztendliche Produktion von Neuronen und Glia im sich entwickelnden Neuroepithel regulieren, bleiben jedoch unklar. Methoden zum Nachverfolgen von NSC-Divisionsmustern und zur Kartierung der Abstammung klonal verwandter Zellen haben sich dramatisch weiterentwickelt. Jedoch, viele zeitgenössische Linie Tracing-Techniken leiden unter dem Mangel an zellulärer Auflösung der Nachkommenzell Schicksal, die für die Entschlüsselung VorläuferzellTeilung Muster ist wesentlich. Präsentiert wird ein Protokoll, das Mosaikanalysen mit Doppelmarkern (MADM) verwendet, um in vivo clonale Analysen durchzuführen. MADM manipuliert gleichzeitig einzelne Vorläuferzellen und visualisiert präzise Teilungsmuster und Linienverlauf bei beispielloser Einzelzellauflösung. MADM-basierte interchromosomale Rekombinationsereignisse während der G2-X-Phase der Mitose, zusammen mit zeitlich induzierbarem CreERT2,liefern genaue Informationen über die Geburtsdaten von Klonen und deren Teilungsmuster. Somit bietet die MADM-Linienverfolgung beispiellose qualitative und quantitative optische Auslesungen des Proliferationsmodus von Stammzellvorläufern auf Einzelzellebene. MADM ermöglicht auch die Untersuchung der Mechanismen und funktionellen Anforderungen von Kandidatengenen in der NSC-Linienprogression. Diese Methode ist einzigartig, da eine vergleichende Analyse von Kontroll- und mutierten Subklonen in der gleichen Gewebeumgebung in vivo durchgeführt werden kann. Hier wird das Protokoll ausführlich beschrieben, und experimentelle Paradigmen zur Verwendung von MADM für die klonale Analyse und Linienverfolgung in der sich entwickelnden Großhirnrinde werden demonstriert. Wichtig ist, dass dieses Protokoll angepasst werden kann, um MADM Klonanalyse in jeder murinen Stammzellnische durchzuführen, solange der CreERT2 Treiber vorhanden ist.
Einleitung
Die Großhirnrinde ist eine hochorganisierte Struktur, die aus sechs unterschiedlichen Schichten besteht. Der Kortex enthält eine Vielzahl von Zelltypen, einschließlich Neuronen und Glia, die interagieren, um funktionelle neuronale Schaltkreise zu bilden. Die meisten, wenn nicht alle, kortikalen exzitatorischen Projektionsneuronen und Glia werden aus einem gemeinsamen Pool von neuronalen Stammzellen (NSCs) abgeleitet, die als radiale Gliavorläufer (RGPs)1,2,3bekannt sind. RGPs selbst werden aus neuroepithelialen Stammzellen (NESCs) abgeleitet, die das frühe embryonale Neuroepithel bilden. Am embryonalen Tag 9 (E9) bei Mäusen beginnen NESCs mit dem Übergang zu RGPs4. Die Progression der RGP-Linien erfordert eine genaue zeitliche und räumliche Regulation, und wenn dieser Prozess behindert wird, können schwere neurologische Störungen wie Megalenzephalie, Mikrozephalie, Lissenzephalie oder Beeinträchtigungen wie Schizophrenie und Autismus5,6führen. Bei E10 durchlaufen die meisten RGPs symmetrische proliferative Divisionen, was zu einer Erweiterung des neuronalen Vorläuferpools4,7führt. RGPs beginnen schließlich, sich asymmetrisch zu teilen, wodurch kortikale Projektionsneuronen in zeitlich definierter Weise produziert werden. Durch aufeinander folgende Wellen der Neurogenese wandern neugeborene Neuronen in die kortikale Platte bildende kortikale Laminae mit frühgeborenen Neuronen, die tiefe Schichten besetzen, und spätgeborenen Neuronen, die in den oberflächlichen Schichten8,9,10leben. Da klonal verwandte pyramidale Neuronen radial in den Kortex mit sehr geringer tangentialer Dispersion wandern, neigen Tochterzellen dazu, eine säulenförmige oder kegelförmige Struktur zu bilden, die als neuronale radiale Einheit4,11,12,13bezeichnet wird. Durch E17 ist die embryonale neurogene Expansion bei Mäusen abgeschlossen14. RGPs können auch ependymale Zellen und einige Klassen von Glia produzieren, einschließlich Astrozyten und Oligodendrozyten1,15,16,17,18,19. Das Potenzial von RGPs, sowohl Neuronen als auch Astrozyten hervorrufen zu können, scheint in allen kortikalen Regionen18konsistent zu sein, wobei etwa 1/6 der neurogenen RGPs ebenfalls Glia11produzieren.
Derzeit sind die genetischen und epigenetischen Faktoren, die das zeitliche Fortschreiten einer Stammzelle entlang ihrer Abstammung regulieren, weitgehend unbekannt. Zeitliche Muster der Genexpression können erhebliche Auswirkungen auf Abstammungsentscheidungen in RGPs20,21,22,23,24haben. Wie diese eng maschinale Beziehung zwischen zeitlicher und räumlicher Musterung zur molekularen Vielfalt adulter neuronaler Typen über kortikale Bereiche hinweg führt, ist nicht bekannt. Ebenso ist die Frage, wie das individuelle Stammzellpotential und seine zelluläre Leistung auf zellulärer und molekularer Ebene moduliert wird, eine wichtige unbeantwortete Frage. Zukünftige Studien werden hoffentlich einige dieser Fragen angehen und letztendlich unser Verständnis der funktionellen kortikalen Schaltungsbildung erweitern.
Die Entwicklungsneurobiologie versucht, die Abstammungsbeziehung zu verstehen, die Zellen im Gehirn miteinander teilen. Anfangs standen nur sehr wenige Forschungsinstrumente dafür zur Verfügung, und viele frühe Studien stützten sich auf visuelle Beobachtungen von Teilungsmustern in transparenten Organismen wie Caenorhabditis elegans25. In den letzten Jahrzehnten hat eine dramatische Zunahme der Anzahl und Raffinesse der verfügbaren Techniken13,26,27,28,29gesehen. Die Entstehung des CRISPR-Cas9 Genom-Editing-Systems ermöglicht eine synthetische Rekonstruktion von Zelllinienbeziehungen durch die Einführung sich entwickelnder DNA-Barcodes27,30. Zwei aktuelle Beispiele für Barcoding-Strategien umfassen die Verwendung von Homing-Guide-RNA, die CRISPR-Cas9 auf bestimmte DNA-Barcode-Loci oder eine Cytidin-Deaminase lenkt, die mit Nickase Cas9 verschmolzen ist, um endogene interspersierte Wiederholungsbereiche31,32zu zielen. Diese Technologien bieten hochmultiplexierte Ansätze durch die Einführung von Barcodes, die im Laufe der Zeit schrittweise und stabil einzigartige Mutationen ansammeln. Genom-Editing-Ansätze sind sehr wertvoll, da sie eine rückwirkende Analyse der Beziehung zwischen zwei beliebigen Zellen basierend auf der gemeinsamen Vererbung dieser Barcodes ermöglichen. Um die Barcodes in einzelnen Zellen lesen zu können, muss jedoch in der Regel das Gewebe gestört werden, so dass Informationen über Position, Morphologie und absolute Zellzahlen eines einzelnen Vorläufers verloren gehen.
Kombinatorische Etikettierungsparadigmen bewahren räumliche Informationen und ermöglichen grundsätzlich auch die Unterscheidung zwischen eng lokalisierten oder sogar überlappenden Klonen33,34. Damit eine Linienverfolgungsmethode informativ ist, muss sie einzelne Vorläufer und ihre Nachkommen spärlich und unauslöschlich kennzeichnen. Bemerkenswert ist, dass die Brainbow35 und Konfetti36,37 Ansätze verwenden stochastische multicolor Cre Recombinase-basierte Reporter, die eine Kombination von fluoreszierenden Proteinen aus einem einzigen Ort auszudrücken. Die große Anzahl gleichzeitiger Farbkombinationen, die in vivo erreicht werden können, machen dies zu einem leistungsstarken Werkzeug bei der Verfolgung kortikaler RGP-Klone und Astrozyten34. Transposonbasierte Systeme, die eine stabile genomische Integration von Transgenen ermöglichen, die fluoreszierende Reporter kodieren und die Linienverfolgung von kortikalen Vorläufern ermöglichen, wurden ebenfalls33,38,39,40,41entwickelt. Transposonbasierte Systeme haben einen zusätzlichen Vorteil, da der Reporter stabil in das Genom integriert und damit linienbezogene Tochterzellen zuverlässig kennzeichnen kann. Um Astrozytenlinien speziell zu verfolgen, wurden eine Reihe von Methoden entwickelt, die die Elektroporation von PiggyBac-Transposasen einschließlich Star Trackbeinhalten, die eine Kombination von Konstrukten verwendet, die verschiedene fluoreszierende Proteine40,42kodieren. Ein anderer Ansatz, MAGIC Marker, führt Brainbow-Vektoren als transponierbare Transgene ein. Dies wurde erfolgreich verwendet, um embryonale neuronale und Astrozyten-Vorläufer34,43zu verfolgen. Kürzlich wurde eine Mosaikanalyse durch dualen rekombinatoren Kassettenaustausch (MADR) gefunden, um mutierte Zellen stabil zu kennzeichnen, die transgene Elemente aus genau definierten chromosomalen Loci44exlastebar machen. Diese leistungsstarken in vivo kombinatorischen Etikettierungstechniken haben zahlreiche Einblicke in die Abstammungsdynamik von Vorläuferzellen gegeben. Diese Analysen werden jedoch auf festem Gewebe durchgeführt und liefern eine Momentaufnahme einzelner Klone in einem definierten Entwicklungsstadium. Um Veränderungen in der Abstammungsdynamik einzelner Klone im Laufe der Zeit zu beobachten, müssen chronische in vivo-Bildgebungsmethoden angewendet werden, die denen im adulten Dentate-Gyrus ähneln45.
Die Mosaikanalyse mit Doppelmarkern (MADM) ist eine leistungsstarke Dual-Color-Labeling-Methode, die die In-vivo-Linienverfolgung einzelner Vorläuferzellen in Mäusen46,47ermöglicht. Zwei Komponenten sind für MADM-Etikettierungsereignisse notwendig: Erstens müssen MADM-Kassetten auf identische Loci auf homologen Chromosomen ausgerichtet sein. Kassetten bestehen aus zwei chimären fluoreszierenden Reportergen, eGFP (grün, [G]) und Tandem-Dimer Tomato (rot, tdT[T]). Die GT-Kassette enthält den N-Terminus von eGFP und den C-Terminus von tdT, getrennt durch ein Intron, das eine loxP-Site enthält. Die TG-Kassette ist umgekehrt mit dem N-Terminus von tdT und dem C-Terminus von eGFP konstruiert. Zweitens ist die Cre Expression der Cre-Recombinase in derselben Zelle, die die zielgerichteten MADM-Kassetten enthält, von wesentlicher Bedeutung. In Ermangelung von Credrücken die chimerischen Kassetten keine funktionelle eGFP oder tdT aus, da ihre Codierungssequenzen gestört sind. Die loxP-Standorte dienen als Ziel für die cre-mediale interchromosomale Rekombination, was zur gleichzeitigen Rekonstitution beider Expressionskassetten führt. Wenn eine Rekombination während der G2-Phase des Zellzyklus gefolgt von X-Segregation (G2-X) auftritt, exprimieren die beiden Tochterzellen jeweils eines der beiden fluoreszierenden Proteine. Die zeitliche Regulierung der CreERT2-Aktivität mit Tamoxifen (TM) liefert genaue Informationen über das Geburtsdatum von MADM-Klonen und die Teilungsmuster ihrer Nachkommen (Abbildung 1A)29,46,47.
MADM kann potenziell systematisch einzelne Klone mit hoher Einzelzellauflösung im Maushirn ähnlich wie herkömmliche, aber unspezifische und mühsame Methoden wie Golgi Färbung48 oder Farbstofffüllung49kennzeichnen. Da nur der Promotor, der CreERT2 antreibt, die Zelltypspezifität der klonalen MADM-Etikettierung bestimmt, kann MADM grundsätzlich für die klonale Linienverfolgung in allen murinen Organen undGeweben 47,50,51,52angewendet werden. Tatsächlich haben Studien bereits MADM verwendet, um Abstammungsbeziehungen in Klonen zu offenbaren, die aus verschiedenen Geweben abgeleitet sind47,50,51,52,53,54,55,56,57,58,59. MADM experimentelle Paradigmen wurden angewendet, um Linie in kortikalen Projektion neuronen, glia, und postnatalen Stammzellen in der sich entwickelnden Neocortex7,,11,12,46,60,61,62,63,64,65. MADM wurde auch verwendet, um Zelllinien in den erwachsenen Dentate Gyrus, Thalamus, Kleinhirn-Granulat-Zellen und Interneuronen auf klonaler Ebene zu untersuchen (siehe Tabelle 1 für eine vollständige Liste)47,53,54,56,57,66.
Ein einzigartiges Merkmal von MADM ist die Fähigkeit, Mutationen genetisch mit einer MADM-Kassette zu verknüpfen und so ein genetisches Mosaik zu erzeugen (Abbildung 1B und Abbildung 2). Dies führt zu wildlebenden Tochterzellen, die mit einem fluoreszierenden Marker (tdT in Abbildung 1B)und homozygoten mutierten Geschwistern mit den anderen (eGFP in Abbildung 1B) in einer unbeschrifteten heterozygoten Umgebung beschriftet sind. MADM ist einzigartig, da eine vergleichende Analyse von Kontroll- und mutierten Subklonen in der gleichen Gewebeumgebung in vivo durchgeführt werden kann. Ursprünglich wurden MADM-Kassetten in den Rosa26-Lokus 47gezielt, aber die MADM-Analyse der Genfunktion beschränkte sich auf Gene, die auf den Ort distal distal waren. Um diese Einschränkung (zumindest teilweise) zu überwinden und die Möglichkeiten für MADM-basierte Genanalysen zu erweitern, wurden MADM-Kassetten in der Nähe der Zentromere von Chr. 751, Chr. 1146und Chr. 1251eingeschlagen. Die Ausrichtung auf alle 19 Maus-Autosomen mit MADM-Kassetten ist in Arbeit und ermöglicht es, praktisch jedes Gen in der Zukunft zu untersuchen, was eine unvergleichliche Plattform für die Untersuchung von Entwicklungslinienbeziehungen in Kombination mit funktioneller genetischer Analyse bietet.
Protokoll
Mausprotokolle wurden von der institutionellen präklinischen Kerneinrichtung (PCF) und der internen Ethikkommission des IST Austria überprüft. Alle Zucht- und Experimente wurden unter einer vom Bundesministerium für Wissenschaft und Forschung genehmigten Lizenz nach österreichischem und EU-Tierrecht durchgeführt.
1. Züchtung von Versuchsmäusen für die CLonalanalyse MADM
- Einrichten von zeitzeitlichen experimentellen MADM-Paarungen (>P56; CD-1) am späten Nachmittag (17:00 Uhr) und überprüfen Sie am morgen (8:00 Uhr) vaginale Stecker. Der Morgen, an dem der Stecker vorhanden ist, zählt als Tag 0.5. Siehe Abbildung 2 für eine Übersicht über das experimentelle Mauspaarungs-Setup. Stellen Sie sicher, dass Zeitpunkte für die TM-Induktion von CreERT2-Aktivität und -Analyse geeignet sind, um experimentelle Fragen zu beantworten.
HINWEIS: Weitere Informationen finden Sie in Abbildung 3 und den nachstehenden repräsentativen Ergebnissen. - Für die postnatale Probenahme, richten Sie Zuchten ein, um Pflegemütter parallel zu generieren.
HINWEIS: Diese sollten bis zu 1 bis 2 Tage vor der Einrichtung experimenteller Zuchten gestartet werden.
2. TM Induktion bei MADM-Mäusen
- Bereiten Sie eine 20 mg/ml TM Arbeitslösung vor, indem Sie sie in Maisöl in einem 15 ml oder 50 ml konischen Zentrifugenrohr auflösen und auf einer Schaukelplattform für 4 h bei Raumtemperatur (RT) platzieren, um sicherzustellen, dass TM vollständig aufgelöst wird. Arbeitslösung bei 4 °C mit Aluminiumfolie überzogen und innerhalb von 2 Wochen verwenden.
- Um MADM-Rekombinationsereignisse zu induzieren, liefern Sie eine einmalige Injektion von TM intraperitoneal (IP) mit einer 1 ml Tuberkulinspritze und einer 25 G Nadel in eine zeitgefällige schwangere Mutter. Je nach Stadium der kortikalen Neurogenese, injizieren TM zwischen E10-E15 in einer Dosis von 1,2 mg/schwangere Mutter. Für frühe Zeitpunkte (d. h. E10) verwenden Sie maximal 1 mg/schwangere Mutter (25 mg/kg), um Komplikationen während der Schwangerschaft zu verhindern11. Für Zeitpunkte zwischen E11-E15 verwenden Sie 2 mg/schwangere Mutter (50 mg/kg)7.
HINWEIS: Alternativ kann TM bei späten Schwangerschaften mit einer oralen Gavage verabreicht werden. - Für madM klonale Analyse postnatale Zeitpunkte, erholen lebende Embryonen bei E18-E19 durch Kaiserschnitt, und dann welpen mit einer Pflegemutter aufziehen.
HINWEIS: Je nach Gesundheitszustand der schwangeren Frau kann es nicht notwendig sein, einen Kaiserschnitt durchzuführen, aber die Aufzucht von Welpen bei einer Pflegemutter ist immer noch erforderlich, da die ursprüngliche TM-behandelte Mutter Probleme beim Laktieren haben kann. - Um lebende Embryonen per Kaiserschnitt zu bergen oder embryonale Zeitpunkte für die Analyse abzurufen, opfern Sie die schwangere Mutter durch Zervixverrenkung.
- Das Tier in eine Supine-Position geben und Fell mit 70% Ethanol desinfizieren. Machen Sie einen kleinen Schnitt in der Haut im Unterbauch über der Gebärmutter mit chirurgischen Zangen und Schere. Machen Sie einen zweiten Schnitt durch die Muskeln und die Bauchmuskelwand, um das Peritoneum zu offenbaren.
- Entfernen Sie die Gebärmutter, indem Sie sich mit einer Schere vom umgebenden Gewebe trennen. Übertragen Sie intakte Gebärmutter auf einen mit warmem Wasser gefüllten Handschuh, um die Überlebensrate des Embryos zu erhöhen, bis jeder einzeln aus dem Amnion entfernt wird.
- Verwenden Sie eine fein gekippte Schere und Finger, um die Gebärmutterwände vorsichtig zu öffnen, um Embryonen freizusetzen. Schneiden Sie die Nabelschnüre nicht zu nah am Körper, um einen ausgedehnten Blutverlust zu verhindern. Wenn Embryonen für die Analyse verwendet werden sollen, fahren Sie mit Schritt 3.9 fort. Wenn Welpen gefördert werden sollen, fahren Sie mit Schritt 2.8 fort.
- Wenn Pflege erforderlich ist, reinigen Sie die Welpen, bevor Sie sie an die Pflegemutter überführen. Während der Reinigung der Welpen, drücken Sie vorsichtig die Brust von Zeit zu Zeit, um die Atmung zu initiieren. Legen Sie wieder auf einen zweiten Handschuh mit warmem Wasser gefüllt, um die Überlebensrate zu verbessern.
HINWEIS: Es ist wichtig, alle verbleibenden Amnion und/oder Plazenta mit einem Papiertuch vorsichtig zu entfernen. - Bevor Sie Welpen an die Pflegemutter überführen, entfernen Sie die Pflegemutter aus ihrem Käfig, entfernen Sie die ursprünglichen Welpen und ersetzen Sie sie durch die experimentellen Welpen. Bringen Sie die Pflegemutter in ihren Käfig zurück.
HINWEIS: Weitere Vorschläge zur Verbesserung der Akzeptanzraten finden Sie in der Diskussion. - Wenn Genotypisierung erforderlich ist, sammeln Zehen- oder Schwanzbiopsien zwischen P6-P8.
HINWEIS: Führen Sie diesen Schritt nur aus, wenn Tierversuchslizenzen diese Praxis genehmigen.
3. Gewebevorbereitung für MADM-Klone im Gehirn
HINWEIS: Bei Experimenten, die postnatales Gewebe enthalten, fahren Sie mit Schritt 3.1 fort. Für embryonale Zeitpunkte und frühe postnatale (P0-P3), setzen Sie Schritt 3.9 fort.
- Anästhetisieren Sie das experimentelle MADM-Tier mit einer IP-Injektion einer Ketamin/Xylazin/Acepromazin-Lösung (65 mg, 13 mg bzw. 2 mg/kg Körpergewicht) und bestätigen Sie, dass die Maus nicht reagiert, indem sie die Hinterpfote kneift.
HINWEIS: Sowohl männliche als auch weibliche MADM-Mäuse (CD-1-Hintergrund) werden zur Analyse verwendet. Wenn Genotypisierung erforderlich ist, sammeln Sie an dieser Stelle Ohrbiopsien. - Legen Sie das anästhesierte Tier in die Rückenlage auf die Perfusionschirurgie und desinfizieren Sie Fell mit 70% Ethanol. Um mit der Operation zu beginnen, machen Sie vorsichtig einen Schnitt mit schere und chirurgischen Zangen durch die äußere Schicht der Haut und dann einen zweiten Schnitt durch die Muskelschicht. Heben Sie die Spitze des Brustbeins und schneiden Sie Bindegewebe an den Seiten, mit besonderer Vorsicht, um zu vermeiden, die Leber zu schneiden. Die Brusthöhle wird sichtbar sein.
- Schnippt das Zwerchfell und hebt, um das Herz zu offenbaren. Schneiden Sie den Rippenkäfig und den Stift vorsichtig auf die chirurgische Schale, um das Herz freizulegen. Für Welpen, entfernen Sie den Rippenkäfig vollständig.
- Setzen Sie eine Nadel mit Phosphat gepufferter Saline (PBS) in den unteren linken Ventrikel (Palergewebe) ein. Mit einer kleinen Irisschere wird am hinteren Ende des rechten Vorhofs (dunkelrotes Gewebe) ein Schnitt einstrichen, damit das Blut abfließen kann.
- Perfusion mit PBS durchführen, gefolgt von frisch zubereitetem, eiskaltem 4% Paraformaldehyd (PFA), das in PBS hergestellt wird. Für Welpen (P4-P10) verwenden Sie Spritzen, um Perfusion durchzuführen. Füllen Sie eine Spritze mit 10 ml PBS und eine andere mit 10 ml 4% PFA. Stellen Sie sicher, dass alle Luftblasen in den Spritzen entfernt wurden. Für ältere Tiere verwenden Sie eine peristaltische Pumpe.
- Beginnen Sie mit PBS (10 ml bei 2 x 4 ml/min in Welpen; 20 ml bei 4 x 6 ml/min für Erwachsene mit einer peristaltischen Pumpe). Die Leber wird klar und hellgelb, wenn die Nadel richtig positioniert ist.
- Nach Abschluss der Nadel von den Welpen entfernen und die Nadel mit PFA in dasselbe Loch einsetzen. Für Erwachsene, stoppen Sie die peristaltische Pumpe vor dem Austausch der PBS-Lösung mit eiskaltem PFA, um sicherzustellen, Blasen in den Aufnahmeschläuchen zu vermeiden. Fortsetzen der Durchflage mit PFA (10 ml bei 2 x 4 ml/min in Welpen; 30 ml bei 4 x 6 ml/min für Erwachsene mit einer peristaltischen Pumpe).
- Wenn die Durchblutung abgeschlossen ist, enthaupten Sie die Maus und entfernen Sie das Gehirn durch sorgfältige Zerlegung. Übertragen Sie gehirn auf 4% PFA. Verwenden Sie mindestens das 5-fache der Hirnvolumina (d. h. 5 x 10 ml PFA in einem 15 ml konischen Zentrifugenrohr) und inkubieren Sie über Nacht bei 4 °C für die Postperfusionsfixierung, um eine vollständige Fixierung des Gewebes zu gewährleisten. Fahren Sie mit Schritt 3.10 fort.
- Für embryonales Gewebe und frühes postnatales Gewebe (d.h. P0-P3) enthaupten Sie die Embryonen nach durchführung eines Kaiserschnitts mit einer Schere. Wenn Genotypisierung erforderlich ist, sammeln Sie den Schwanz des Embryos an dieser Stelle. Sezieren Sie das Gehirn sofort aus und übertragen Sie es auf eine 12-Well-Platte, die 2 x 3 ml 4% PFA/Well enthält. Über Nacht bei 4 °C für die Nachfixierung inkubieren.
- Am nächsten Morgen PFA mit 10 ml (Erwachsene) oder 2 x 3 ml (Embryo) PBS austauschen und 3x für 15 min bei RT wiederholen. Gewebe in 30% Saccharoselösung im Phosphatpuffer (PB) übertragen und bei 4 °C auf einer Schaukelplattform lagern, bis das Gewebe in der Lösung sinkt.
- Betten Sie das Gehirn in eine optimale Schnitttemperatur (OCT) Verbindung in eine Einbettform, wobei darauf geachtet wird, das Gehirn entweder für koronare oder sagittale Schnitte zu orientieren. Einfrieren, indem Sie die Einbettform auf Trockeneis legen, bis OCT vollständig undurchsichtig wird (ca. 10 bis 15 min). Gewebe bis zur weiteren Verwendung bei -80 °C lagern.
4. Herstellung von MADM-Gewebe für die Immunhistochemie
- Befestigen Sie den Gewebeblock an der Probenscheibe im Kryostat, indem Sie einen RING von OCT auf die Scheibe auftragen und den Block direkt in das OCT legen, wenn er zu frieren beginnt. Stellen Sie sicher, dass der Block korrekt auf die gewünschte Schnittebene ausgerichtet ist.
HINWEIS: Hier wird die koronale Schnitte zur Untersuchung kortikaler MADM-Klone ausführlich beschrieben. - Stellen Sie die Blocktemperatur im Kryostat auf -20 °C und die Klingentemperatur auf -21 °C ein.
- Lassen Sie den Gewebeblock an die Kammertemperatur anpassen, indem Sie die Probenscheibe am Probenhalter montieren und in Kryostat für 5 min lassen, bevor Sie mit dem Schnitt beginnen.
- Schneidblock in dicken Abschnitten (45 x 60 m), bis der Gewebebereich von Interesse erreicht ist.
- Sobald der Rand des Kortex deutlich sichtbar ist, stoppen Sie das Schnitten und verriegeln Sie die Klinge. Stellen Sie sicher, dass die Klinge abgeschirmt ist, bevor Sie den Block zuschneiden.
- Trim überschüssiges OCT, das das Gewebe umgibt, mit einer Klinge, so dass auf allen Seiten des Gehirns 1 bis 2 mm OCT übrig bleiben.
- Richten Sie den Block dann so aus, dass einer der seitlichen Ränder des Kortex nach unten und der andere nach oben ausgerichtet ist (d. h. die rostralste Kante des Kortex ist nach rechts gerichtet).
- Beginnen Sie mit dem Schneiden mit einer Dicke von 45 m für erwachsene Klone und 30 m für embryonale Klone. Führen Sie jeden Abschnitt einzeln aus und verwenden Sie eine kleine Bürste, um den Bereich unter dem Messer sauber von schmutzen den Block zu halten.
HINWEIS: Wenn dies nicht geschieht und ein Abschnitt fällt, kann es schwierig sein, die richtige Reihenfolge der Slices zu bestimmen. - Wenn die Abschnitte zu krümmen beginnen, trimmen Sie die Ränder des Blocks und/oder passen Sie die Glasantirollplatte sorgfältig an.
- Für die embryonale Klonanalyse, montieren Sie Abschnitte direkt an einem mattierten Schlitten. Trocknen Sie auf einer Heizplatte bei 37 °C, bevor Sie direkt zu Schritt 5.6 übergehen.
HINWEIS: Eine Folie können mehrere Abschnitte hinzugefügt werden, stellen jedoch sicher, dass die sequenzielle Reihenfolge beibehalten wird. - Um erwachsene Klone zu sammeln, bereiten Sie 24 Brunnenplatten vor, die 1 ml PBS/Well enthalten (typischerweise 5 x 6 Platten pro Gehirn). Ab dem ersten Brunnen, mit kalter Zange sammeln einzelne serielle Abschnitte in PBS in der Reihenfolge der Schnitte.
HINWEIS: Die Methode des schwimmenden Abschnitts wird für erwachsenes Gewebe verwendet, um sicherzustellen, dass keine Abschnitte übersehen werden und dass montierte Abschnitte keine Falten enthalten. - Beenden Sie das Schnitten, sobald das Ende des Neocortex erreicht ist.
- Für erwachsene Klone, fahren Sie mit der Montage schwimmender Abschnitte fort.
HINWEIS: Abschnitte können in PBS bei 4 °C für bis zu 24 h aufbewahrt werden.
5. Montage von erwachsenem Gewebe für die Bildgebung
HINWEIS: Folgende Werkzeuge sind erforderlich: kleiner Pinsel, Petrischale, PBS mit 0,5% Tween (PBS-T), Haftschlitten (Materialtabelle), Montagemedium (Materialtabelle), Abdeckungen und Zangen.
- Füllen Sie eine Petrischale mit PBS-T.
HINWEIS: Reinigungsmittel wird verwendet, um den Montageprozess zu unterstützen. Wenn eine Färbung für zusätzliche Antigene erforderlich ist, die empfindlich auf Reinigungsmittel reagieren (d. h. Glykoproteine), ist es am besten, die Zugabe von Tween zu überspringen. - Legen Sie eine Haftrutsche in das PBS-T, so dass es fast bis zum Etikett verdeckt ist.
- Übertragen Sie den ersten Abschnitt in das PBS-T.
- Mit einem kleinen Pinsel, manövrieren Sie den Abschnitt auf die Folie und ordnen Sie es, um die Reihenfolge des Schneidens zu erhalten. Gehen Sie mit allen weiteren Abschnitten auf die gleiche Weise vor.
- Sobald alle Abschnitte in Position sind, legen Sie die Folie (12-16 Abschnitte/Folie) in eine dunkle Schiebekammer. Heben Sie den Deckel leicht an, damit die Abschnitte vollständig trocknen können (ca. 10 bis 20 min), um sicherzustellen, dass sie in den nachfolgenden Schritten verbleiben.
- Wenn Sie eine Immunhistochemie für zusätzliche Antigene durchführen, fahren Sie direkt mit Abschnitt 6 oder 7 fort.
HINWEIS: Für embryonale Zeitpunkte ist es notwendig, Immunfärbungsschritte für mindestens GFP und tdT durchzuführen (Abschnitt 6). Bei erwachsenen Klonen ist dies nur erforderlich, wenn zusätzliche Antigene parallel gefärbt werden (Abschnitte 6 und 7). - Rehydratisieren und waschen Sie Abschnitte 1x mit 1x PBS für 5 min, um Rest-PBS-T zu entfernen. Tragen Sie 1 ml 4,6-Diamidino-2-Phenylindole (DAPI) in 1x PBS (1 g/ml) verdünnt auf die Rutsche auf, um sicherzustellen, dass alle Abschnitte abgedeckt sind und 15 min inkubieren.
- Entfernen Sie die DAPI vorsichtig und waschen Sie 1x mit 1x PBS für 5 min. Entfernen Sie überschüssige PBS und trocknen Sie für 1 x 2 min vor der Einbettung in 110 l Montagemedium. Mit einem 24 x 60 mm Abdeckschlupf abdichten und vor der Bildgebung mindestens 3 h trocknen lassen.
6. Immunostainierung nur für GFP und tdT
HINWEIS: Dieser Abschnitt ist für embryonale Klone erforderlich.
- Legen Sie die Dias horizontal in eine befeuchtete Gleitinkubationskammer. Markieren Sie Foliengrenzen mit einem Wachsmarker, um die erforderliche Puffermenge zu minimieren.
- Rehydrieren Sie Abschnitte mit 1x PBS. Um die Färbequalität zu verbessern, arbeiten Sie mit frisch geschnittenem Gewebe.
- Fügen Sie 250 bis 400 L Blockierpuffer (0,5 % Triton X-100, 2,3 % normales Eselsserum in 1x PBS) pro Folie hinzu, um sicherzustellen, dass alle Abschnitte abgedeckt sind. Inkubieren für 1 h.
HINWEIS: Die Konzentration von Reinigungsmitteln (Triton X-100 oder Tween-20) variiert je nach den zusätzlichen primären Antikörpern, die verwendet werden, da einige Antigene empfindlicher auf Reinigungsmittel reagieren als andere. - Entfernen Sie den Blockierungspuffer, und fügen Sie dem Dia primäre Antikörper im Blockierungspuffer hinzu (300 x 400 L/Folie).
HINWEIS: Ein Beispiel für eine standardprimäre Antikörperreaktion für Anti-GFP/Anti-TdT (MADM) könnte Hühneranti-GFP (1:500) und Kaninchen-Anti-RFP (1:500) verwenden. - Mit Primärantikörpern über Nacht bei 4 °C inkubieren.
HINWEIS: Die Folien müssen perfekt horizontal mit Puffer, der alle Abschnitte abdeckt, inkubiert werden. Andernfalls kann es zu ungleichmäßigen oder schlechten Färbungen kommen. - Bestätigen Sie am nächsten Morgen, dass der Sperrpuffer mit primären Antikörpern weiterhin alle Abschnitte auf der Folie abdeckt. Wenn dies nicht der Fall ist, wiederholen Sie den Inkubationsschritt für 3 x 4 h bei RT.
- Primäre Antikörper entfernen und 4x mit 1x PBS 10 min bei RT waschen.
- Fügen Sie sekundäre Antikörper, die im Sperrpuffer verdünnt werden, zum Dia (300 x 400 l/Rutsche) hinzu: Alexa Fluor 488 Anti-Huhn IgG (1:500) und Cy3 Anti-Kaninchen-IgG (1:500).
- Inkubieren Sie bei RT für 2 h. Halten Sie Dias von Licht bedeckt, um das Bleichen von Fluorophoren zu verhindern.
- Sekundäre Antikörper entfernen und 2x mit 1x PBS 10 min waschen.
- Inkubieren Sie mit DAPI in PBS (1:5.000) für 15 min verdünnt.
- Waschabschnitte 1x mit 1x PBS für 10 min.
- Entfernen Sie überschüssige PBS und trocknen Sie für 1 x 2 min, bevor Sie in 110 l Montagemedium einbetten.
- Mit 24 x 60 mm Abdeckschlupf abdichten und vor der Bildgebung mindestens 3 h trocknen lassen. Das Bild gleitet innerhalb von 1 bis 2 Wochen nach der Durchführung der Immunhistochemie, um ein optimales Signal zu gewährleisten.
7. Immunostainierung für GFP, tdT und zusätzliche Antigene
- Führen Sie die Schritte 6.1-6.3 aus.
- Entfernen Sie den Blockierungspuffer, und fügen Sie dem Dia primäre Antikörper im Blockierungspuffer hinzu (300 x 400 L/Folie).
HINWEIS: Bei der Färbung von drei oder mehr Antigenen (d. h. GFP, tdT und einem Protein von Interesse) und dem Antikörper für das Protein von Interesse wurde bei Kaninchen erhöht, wird empfohlen, den Anti-TdT (Ziege) primärer Antikörper bei einer Verdünnung von 1:500 zu verwenden. Ein Beispiel für eine primäre Antikörperreaktion für drei Antigene mit alternativer tdT-Färbung könnte Hühneranti-GFP (1:500), Ziegenanti-TdT (1:500) und Antikörper gegen das Protein von Interesse (d.h. Kaninchen) verwenden. - Führen Sie die Schritte 6.5-6.7 aus.
- Fügen Sie einen sekundären Antikörper-Mix in Blockierpuffer verdünnt zu gleiten (300 x 400 l / Rutsche): Alexa Fluor 488 Anti-Huhn IgG (1:500), Cy3 Anti-Ziegen IgG (1:500), und Alexa Fluor 647 Anti-Kaninchen-IgG (1:500).
- Führen Sie die Schritte 6.9-6.14 aus.
8. Konfokale Bildaufnahme und Quantifizierung von MADM-Klonen
- Identifizieren und dokumentieren Sie Gehirnabschnitte, die Klone und deren Positionen im Kortex enthalten.
HINWEIS: Die Anzahl der Abschnitte, die ein Klon umfasst, hängt davon ab, wann der Klon induziert wurde, der CreERT2-Treiber und der Zeitpunkt der Analyse. Dieser Schritt kann entweder mit einem konfokalen Mikroskop oder einem Epifluoreszenzmikroskop durchgeführt werden. - Verwenden Sie ein invertiertes konfokales Mikroskop, indem Sie zunächst die richtigen Laserlinien und Filter auswählen und konfigurieren. Wählen Sie für MADM-Gehirne DAPI, GFP und tdT (Erregung: 358 nm, 488 nm bzw. 554 nm; Spitzenemission: 461 nm, 507 nm bzw. 581 nm). Stellen Sie sicher, dass das Loch auf 1 luftige Einheit eingestellt ist, um eine optimale Bildqualität zu erzielen.
- Für konfokale spezifische Einstellungen, Bild-Klone mit einem 20-fachen Objektiv und 1x Zoom. Verwenden Sie für Bilder, die in Quantifizierungen verwendet werden, einen Pixelwert für die Scangeschwindigkeit von 1,52 bis 2,06 s (Werte 7 bis 8 in der Bildaufnahmesoftware) ohne Mittelung. Passen Sie die Laserintensitäts- und Verstärkungseinstellungen für jeden Kanal entsprechend an.
HINWEIS: Je nach gewünschter Bildqualität können die Einstellungen für Scangeschwindigkeit und Mittelung variieren. - Sobald der Klon eindeutig identifiziert ist, ordnen Sie bildgebende Kacheln an, um alle relevanten Abschnitte im Klon abzudecken. Passen Sie den Z-Stack so an, dass alle MADM-markierten Zellen im Klon mit einem Intervall von 1,5 m/z-Stack-Slice erfasst werden. Passen Sie den gekachelten Bereich so an, dass die gesamte Breite des Kortex beim Bilden des Klons erfasst wird (d. h. von der Pialoberfläche bis zum Corpus callosum).
- Bild einzelne Klone, die mehrere Abschnitte nacheinander erstrecken, um sicherzustellen, dass alle Abschnitte ohne Zellen innerhalb eines Klons weiterhin zum Zweck der 3D-Rekonstruktion und korrekten Interpretation von Zell-Rauminformationen abgebildet werden.
- Analysieren Sie jeden Abschnitt, der Zellen eines MADM-Klons enthält, sequenziell vom Rostral bis zum kaudalen Ende des Kortex. Unterscheiden Sie einzelne Neuronen und Glia basierend auf ihrer Morphologie und/oder Markerfärbung. Erfassen Sie Positionsinformationen parallel basierend auf den jeweiligen Schichtgrenzen, die durch kerntechnische Färbung (DAPI) definiert werden.
ANMERKUNG: Repräsentative Ergebnisse für die Embryonalanalyse finden Sie in Abbildung 4 und in Abbildung 5 für repräsentative Ergebnisse für die Erwachsenenanalyse.
9. Serielle 3D-Rekonstruktion von Klonen
HINWEIS: Die 3D-Rekonstruktion einzelner Klone, die über serielle Gehirnabschnitte abgebildet sind, ist sowohl für die visuelle Darstellung als auch für die Analyse von 3D-Klonarchitekturen nützlich und kann gemäß den folgenden Schritten durchgeführt werden.
- Heften und verschmelzen konfokale gekachelte Bilder basierend auf Aufnahmeparametern mit Bildaufnahmesoftware. Öffnen Sie .czi-Datei, und führen Sie dann die Stitching-Methode unter der Registerkarte Verarbeitung in der ZEN-Software (Zeiss) aus.
- Exportieren Sie genähte Bildstapel als einzelne Z-Ebenen im TIFF-Format. Öffnen Sie die datei "Stitched .czi", und führen Sie dann die Image Export-Methode unter der Registerkarte Verarbeitung aus. Für Mehrkanalbilder als Rot/Grün/Blau-Bilder für die nachfolgende Bildverarbeitung exportieren.
- Wiederholen Sie die Schritte 9.1 und 9.2 für jeden seriellen Gehirnabschnitt eines Klons.
HINWEIS: Für eine genaue 3D-Rekonstruktion müssen auch alle Hirnabschnitte innerhalb eines Klons verarbeitet werden, einschließlich derer ohne beschriftete Zellen. - Verkörpern Sie einzelne Bilder in einem einzigen Stapel, um von der rostralsten bis zur kaudalsten Z-Ebene mit Open-Source-Bildverarbeitungssoftware wie ImageJ/Fiji67,68zu beginnen.
HINWEIS: Alle leeren Bilder an den Rändern jedes Gehirnabschnitts sollten an dieser Stelle entfernt werden. - Korrigieren Sie bei Bedarf den Abbild-Stack aus Schritt 9.4 für Fehlausrichtung mit einem ImageJ-Plugin namens "MultiStackReg", indem Sie die Schritte 9.5.1-9.5.5 verwenden. Wenn keine Bildausrichtung erforderlich ist, fahren Sie mit Schritt 9.6 fort.
HINWEIS: Dieses Plugin führt die Bildausrichtung des Kanals mit dem höchsten Kontrast (in der Regel DAPI) durch und wendet dann die aufgezeichnete Transformation auf die anderen Kanäle an, wodurch eine zuverlässige Bildausrichtung von Mehrkanal-Stacks ermöglicht wird. Ein Hilfs-Plugin namens "TurboReg" muss vorinstalliert werden.- Installieren Sie in ImageJ die Plugins "MultiStackReg" und "TurboReg".
- Öffnen Sie den Bildstapel der Klonbilder aus Schritt 9.4, die ausgerichtet werden sollen. Teilen Sie Kanäle in DAPI (blau), GFP (grün) und tdT (rot) unter Farbe unter Bild-Registerkarte auf.
- Führen Sie "MultiStackReg" Plugin aus, um den DAPI-Kanal durch die "Rigid Body"-Transformation auszurichten und die Transformationsdatei zu speichern.
- Wenden Sie die gespeicherte Transformationsdatei mit "MultiStackReg" auf die beiden anderen Kanäle an.
- Führen Sie alle drei ausgerichteten Kanäle zusammen, und speichern Sie den ausgerichteten Stapel.
- Um den Klon in ImageJ zu drehen Stapel von Klonbildern aus Schritt 9.4 (oder Schritt 9.5.5 nach Ausrichtung) in einer vertikalen Ausrichtung mit der pialen Oberfläche auf der Oberseite und dem Corpus callosum an der Unterseite erhalten. Bei Bedarf in der xy-Ebene zuschneiden.
- Generieren Sie für die qualitative Darstellung und quantitative Analyse ein maximales Z-Projektionsbild (Schritt 9.8) oder führen Sie 3D-Rendering (Schritt 9.9) des Klons durch.
- Öffnen Sie in ImageJ den Bildstapel aus Schritt 9.6, und wählen Sie die Option Z-Projektion mit Demlichtungstyp Max Intensitätaus. Dadurch wird ein Bild des gesamten Klons generiert, der auf derselben Ebene projiziert wird.
- Öffnen Sie in ImageJ den Bildstapel aus Schritt 9.6, und wählen Sie die 3D-Projekt-Z-Funktion aus, um eine 3D-Visualisierung des Klons zu generieren, die gedreht werden kann.
HINWEIS: In diesem Schritt ist es wichtig, das richtige Slice-Intervall einzugeben, das der Dicke einzelner Z-Stacks während der Bildaufnahme entspricht. Das Interpolationswerkzeug sollte verwendet werden, um Lücken zwischen Slices zu entfernen.
Ergebnisse
MADM führt zur Rekonstitution funktioneller grüner und roter fluoreszierender Proteine mit zwei Tochterzellen, die jeweils eines der beiden fluoreszierenden Proteine bei G2-X-Chromosomensegregationsereignissen exezieren (Abbildung 1A). Da MADM-Ereignisse zu einer dauerhaften und eindeutigen Beschriftung der beiden nachfolgenden Linien führen, kann eine quantifizierbare Bewertung grüner und roter Tochterzelllinien (Subklonen) durchgeführt werden. Variablen wie Divisionsmuster (z. B. symmetrisch versus asymmetrisch) und Potential (z. B. die Anzahl der Nachkommen) des ursprünglichen Vorläufers können bestimmt werden. Die Quantifizierung jedes fluoreszierend markierten Subklons ist informativ, wenn man rückwirkend bestimmt, ob die ursprüngliche Vorläuferzelle symmetrische proliferative Divisionen oder asymmetrische, neurogene Divisionen zum Zeitpunkt der TM-Induktion durchläuft. Frühere Studien gruppierten Emx1-CreERT2 oder Nestin-CreERT2 abgeleitete exzitatorische Projektionklone im Kortex in zwei breite Klassen7,11,46. Die ersten, als "symmetrische proliferative Klone" bezeichnet, bestehen im Durchschnitt aus einer beträchtlichen Anzahl von Neuronen, wobei sowohl grüne als auch rote Subklonen jeweils vier oder mehr Neuronen enthalten. Die zweite Gruppe, "asymmetrische Klone", definiert eine Klasse von Klonen, bei der der Unterklon "Minderheit" weniger als drei Neuronen und der "Mehrheit"-Subklon vier oder mehr11enthält. Diese Definitionen sind spezifisch für kortikale RGPs und müssen möglicherweise für andere Hirnregionen und Gewebe überprüft werden. Für beide Klassen von kortikalen Klonen wird Dienachkommen schaft in den oberflächlichen und tiefen Schichten verteilt.
Bei der Gestaltung von MADM-Klonstudien gibt es eine Reihe von Aspekten, die berücksichtigt werden müssen. Die Zeit, in der MADM-Ereignisse durch die Verabreichung von TM induziert werden, ist eine wichtige Überlegung (Abbildung 3). Bei kortikalen exzitatorischen Projektions-Neuron-MADM-Klonen (d.h. mit Emx1-CreERT2 oder Nestin-CreERT2) bei E10 waren fast alle RGPs noch in symmetrischen Teilungen11. Daher erfasste die Induktion bei E10 mit TM mehrere Runden proliferativer RGP-Verstärkung und führte zu Klonen mit hohen Neuronzahlen. Allerdings war die Anzahl der RGPs bei E10 im Allgemeinen gering und somit erzeugte die TM-Administration nur sehr wenige MADM-Ereignisse (manchmal weniger als eins pro Gehirn). Die Mehrheit der RGPs wechselte von symmetrischen zu asymmetrischen neurogenen Divisionen bei etwa E12. Um streng asymmetrische neurogene Klone ins Visier zu nehmen, war es am besten, bei E12 oder später zu induzieren (Abbildung 3). Die Zeit zwischen DER TM-Induktion und der Beobachtung von MADM-Rekombinationsereignissen im Kortex war tendenziell weniger als 24 h. IP-Injektionen waren die bevorzugte Methode zur Verabreichung von TM in embryonalen Stadien für diese Methode, da sie zu einer größeren Reproduzierbarkeit bei der klonalen Induktion führte. Es ist auch wichtig, die TM-Dosis aus zwei Gründen auf ein Minimum zu beschränken. Erstens, wenn die MADM-Rekombinationsrate steigt, ist die Wahrscheinlichkeit, mehrere, vielleicht überlappende Klone zu induzieren, höher. Zweitens: Wenn zu viel TM geliefert wird, kann eine erhöhte Abtreibungsrate, Embryo-Reabsorption und kleinere Wurfgrößen beobachtet werden. Abtreibungen in etwa der Hälfte aller schwangeren Muttermütter wurden beobachtet, als TM-Injektionen bei E10 abgegeben wurden. Diese Häufigkeit verringerte sich ab E11 und verringerte sich auf etwa 1/3 der abbrechenden trächtigen Muttermütter. Eine Zusammenfassung der TM-Dosen, Induktionszeiten und CreERT2-Treiber, die in früheren MADM-Studien verwendet wurden, finden Sie in Tabelle 1. Reporteraktivität in Abwesenheit von TM wurde bei einigen TM-induzierbaren CreERT2 Treibern69beobachtet. Bei den Emx1-CreERT2 von Nestin-CreERT2-Treibern wurden keine ektopischen Expressions- oder MADM-Rekombinationsereignisse in Abwesenheit von TM beobachtet. Dies kann teilweise darauf zurückzuführen sein, dass TM-vermittelte chromosomale Transrekombinationen bei etwa 1:1.000 bis 1:10.000 niedrigeren Frequenzen auftreten als cis-rekombinationen, was die Wahrscheinlichkeit einer ektopischen MADM-Kennzeichnung verringert.
Ein weiterer Faktor, der bei der Planung eines MADM-Klonanalyseexperiments zu berücksichtigen ist, ist die Dauer der Studie. Die Änderung der Zeitspanne zwischen der TM-Induktion und dem Zeitpunkt der Analyse des Experiments (A) (Zeitfenster) zeigt die Stammzelldynamik im Laufe der Zeit64an. Kurze embryonale Zeitfenster (d. h. TM/E11-A/E13; TM/E11-A/E16) erfasste die Dynamik der embryonalen Neurogenese (Abbildung 4). Der Vergleich von Klonen aus zwei oder mehr Zeitfenstern bietet quantitative Einblicke in die Anzahl der produzierten Zellen und wie die Neuronenverteilung in verschiedenen Stadien der Abstammungsprogressionvariiert 64. Um das gesamte Potenzial einzelner Klone zu erfassen, ist es notwendig, das analysierte Zeitfenster auf postnatale oder erwachsene Zeitpunkte7,11,12zu erweitern. Beispiele für neokortikale Klone, die im Embryo induziert und beim Erwachsenen analysiert wurden, sind in Abbildung 5dargestellt. Bemerkenswert ist, kortikale Neurogenese ist meist abgeschlossen und Gliogenese erhöht sich um E17. Ungefähr 1/6 neurogene RGP gehen auch um Astrozyten und/oder Oligodendrozyten zu erzeugen11.
Symmetrische Klone treten auf, wenn RGPs eine oder mehrere Runden der proliferativen Division11durchlaufen. Die zwischen E10-E12 induzierten RGP-Klone waren im Durchschnitt größer und lieferten mehr räumliche Merkmale der endgültigen Neuronverteilung (Abbildung 4A-C). Klone mit Neuronen, die relativ gleichmäßig über tiefe und oberflächliche Schichten verteilt waren, nahmen eine "Zylinder"-Form an, während Klone mit Neuronen, die in oberflächlichen Schichten verteilter waren als tiefere Schichten, eine "Kegel"-Form11entwickelten. Um die räumlichen und morphologischen Informationen eines Klons vollständig zu erfassen, war es notwendig, jeden Klon mit sequenziellen Bildern rechnerisch zu rekonstruieren. Zur Messung der klonalen Dispersion wurde die maximale laterale Dispersion (gemessen in allen Dimensionen) in oberflächlichen Schichten (LII-VI) eines Klons mit der Neuronendispersion in tiefen Schichten (LV/LIV) verglichen. Dieses Verhältnis (Verteilung oben: Verteilung niedriger) lieferte eine quantifizierbare Auslesung der gesamten Klonform.
Asymmetrische Klone, bei denen der Minderheitsunterklon drei oder weniger betrug, gaben Einblick in die neuronale Ausgabe eines einzelnen RGP (Abbildung 4D-F und Abbildung 5A-F)7,11,12. Die Mehrheitspopulation (großer Subklon) könnte entweder rot oder grün markiert werden, mit einem Durchschnitt von etwa sieben erregenden Projektionsneuronen pro Klon, wenn sie mit einem Emx1-CreERT2 oder Nestin-CreERT2induziert werden (Abbildung 5G)7,11,12. Die Gesamtzahl der Zellen in einem MADM-Klon könnte weiter seziert werden, indem die Verteilung von Neuronen im großen Subklon über oberflächliche und tiefe Schichten analysiert wird. Die Minderheitspopulation (kleiner Subklon) wurde durch die wechselseitige Farbe beschriftet und betrug durchschnittlich 1 x 2 Zellen pro Klon(Abbildung 5H). Die Gesamtgröße der "Einheit", die im Durchschnitt 8 bis 9 Neuronen betrug, konnte berechnet werden, indem die kleinen und großen Subklonen zusammengerechnet wurden (Abbildung 5I)7,11,12. Es ist wichtig zu beachten, dass, während die neuronale Leistung von RGPs war sehr vorhersehbar, gab es einen Grad der klonalen Heterogenität12,70.
Die Einführung einer Distalmutation in die MADM-Kassette ermöglicht die Erzeugung genetischer Mosaike und bietet eine einzigartige Methode, um die molekularen Regulatoren der Stammzelllinienprogression zu sezieren. Als solches bietet MADM eine unvergleichliche experimentelle Plattform, um die zellautonome Funktion eines Gens zu untersuchen (z. B. seine Assoziation zu Mikrozephalie oder Makrozephalie). Durch den Vergleich von In einem MADM-Genmosaik induzierten Klonen mit Klonen, die in einem Kontroll-MADM induziert werden, kann eine hochquantitative Auslesung von Veränderungen der Neuronenzahlen und -verteilung erzeugt werden. Frühere MADM-basierte Studien quantifizierten die zellautonome Funktion von Otx1 bei der Mikrozephaliebildung auf klonaler Ebene (siehe Abbildung 6A-E für ein repräsentatives Beispiel)11. In einer anderen Studie zeigte die CLonale MADM-Analyse, dass Ndel1 nicht zellautonom die Projektions-Neuronenzahlen reguliert, sondern die Fähigkeit neugeborener Neuronen, in die kortikale Platte ein- oder zu wandern, die später den erwachsenen Kortex46bildet. Diese Studien zeigten die hochquantitative Natur der CLonalanalyse von MADM bei der Untersuchung der zellautonomen Funktionen von Genen, die die kortikale Entwicklung regulieren. Es gibt derzeit keine Beispiele in der Literatur, die MADM verwenden, um Gene zu untersuchen, die in Makrozephalie auf klonaler Ebene involviert sind. In zukünftigen Studien kann die Analyse von Genen, die für die Kontrolle der kortikalen Größe im Allgemeinen relevant sind, jedoch sehr wünschenswerte Erkenntnisse auf molekularer und zellulärer Ebene liefern.

Abbildung 1: Das MADM-Prinzip für die Linienverfolgung und klonale Analyse auf Einzelstammzellebene. (A) Um die Linienverfolgung und die Klonanalyse mit MADM durchführen zu können, müssen zwei Komponenten vorhanden sein. Erstens müssen MADM-Kassetten auf identische Loci auf homologen Chromosomen ausgerichtet sein. Kassetten bestehen aus zwei chimären fluoreszierenden Reportergen, eGFP (grün, [G]) und Tandem-Dimer Tomato (rot, tdT[T]). Die GT-Kassette enthält den N-Terminus von eGFP und den C-Terminus von tdT, getrennt durch ein Intron, das eine loxP-Site enthält. Die TG-Kassette ist umgekehrt mit dem N-Terminus von tdT und dem C-Terminus von eGFP konstruiert. Zweitens muss die Cre Expression der Cre-Recombinase in der Zelle auftreten, die die zielgerichteten MADM-Kassetten enthält. Die loxP-Standorte dienen als Ziel für eine cre-mediale interchromosomale Rekombination, was zur gleichzeitigen Rekonstitution beider Expressionskassetten führt. Wenn während der G2-Phase des Zellzyklus, gefolgt von X-Segregation (G2-X), eine Rekombination auftritt, exprimieren die beiden Tochterzellen eines der beiden fluoreszierenden Proteine. (B) MADM-Prinzip für die genetische Mosaikanalyse auf einer einzigen Klonebene. Mutante Allele (Punktmutationen, Deletionen, Einfügungen, loxP-flankierte bedingte Allele, wie in Abbildung 1Busw.dargestellt) können distal in die TG-MADM-Kassette durch meiotische Rekombination eingeführt werden (siehe Abbildung 2 und Hippenmeyer et al.46 für Details zur Einführung von Mutantenallelen in das MADM-System). Wenn eine G2-X Cre recombinase-vermittelte interchromosomale Transrekombination zwischen MADM-Kassetten auftritt, ergibt sich eine GFP+ homozygote Mutantzelle (GeneX-/-) für das Gen von Interesse und eine tdT+ homozygote Wildtypzelle (GeneX+/+) in einer unbeschrifteten heterozygoten Umgebung46,47,71. Alternative Etikettierungsergebnisse, die nicht in der Klonanalyse verwendet werden (d. h. gelbe Zellen), wurden zuvor im Detail beschrieben11,46,47. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
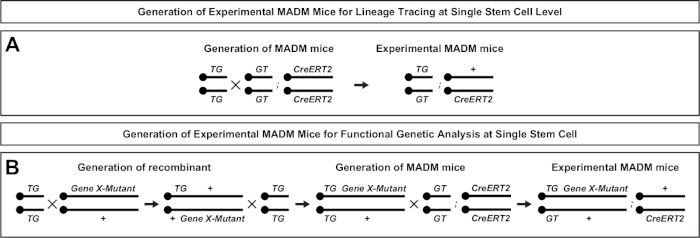
Abbildung 2: Zuchtschemata zur Erzeugung von experimentellen MADM-Mäusen zur Linienverfolgung. Zuchtschema für die Erzeugung von Kontroll-MADM (A) und Gene X MADM (B) experimentelle MADM-Mäuse für die klonale Analyse. Weitere Informationen zu MADM Zuchtparadigmen finden Sie unter Beattie et al.7 und Hippenmeyer et al.7,46. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Zeitkursparadigmen für madM-basierte klonale Linienanalyse. Schemat der experimentellen Entwurfszeitfenster. Bei Längsstichprobenparadigmen blieb der Zeitpunkt der Kloninduktion konstant, und die Dauer vor der Analyse variierte. Bei der progressiven Intervallstichprobe blieb der Zeitpunkt der Analyse konstant, aber der Zeitpunkt der Induktion variierte. Je nach den angesprochenen Fragen kann eine Kombination aus einem oder beiden Ansätzen verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: MADM-Clonalanalyse im entwicklungs- und erwachsenen Neocortex. TM-vermittelte MADM-Kloninduktion in symmetrisch proliferativer (TM bei E10) (A-C) und asymmetrisch neurogener (TM bei E12) (D-F) Teilung von RGPs. Abgebildet sind einzelne MADM-Klone in vivo in den sich entwickelnden (TM/E10-A/E16 und TM/E12-A/E16) (B,E) und Erwachsenen (TM/E10-A/P21 und TM/E12-A/P21) (C,F) in MADM-11GT/TG; Nestin-CreERT2+/- (B,E) und MADM-11GT/TG; Emx1-CreERT2+/- (C,F). Die Neuronenproduktion war unabhängig von Subklonfarbe und grüne Mehrheits-/Minderheitsunterklonen konnten mit roten Mehrheits-/Minderheitsunterklonen unter Kontrollbedingungen7,11verglichen werden. Ungefähr 1/6 der erwachsenen Klone enthielten auch Astrozyten und/oder Oligodendrozyten, die durch weiße Sternchen angezeigt wurden. Die Panels B und F werden mit Genehmigung von Hippenmeyer et al.46 bzw. Rulands und Simons72reproduziert. CP = Kortikale Platte. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
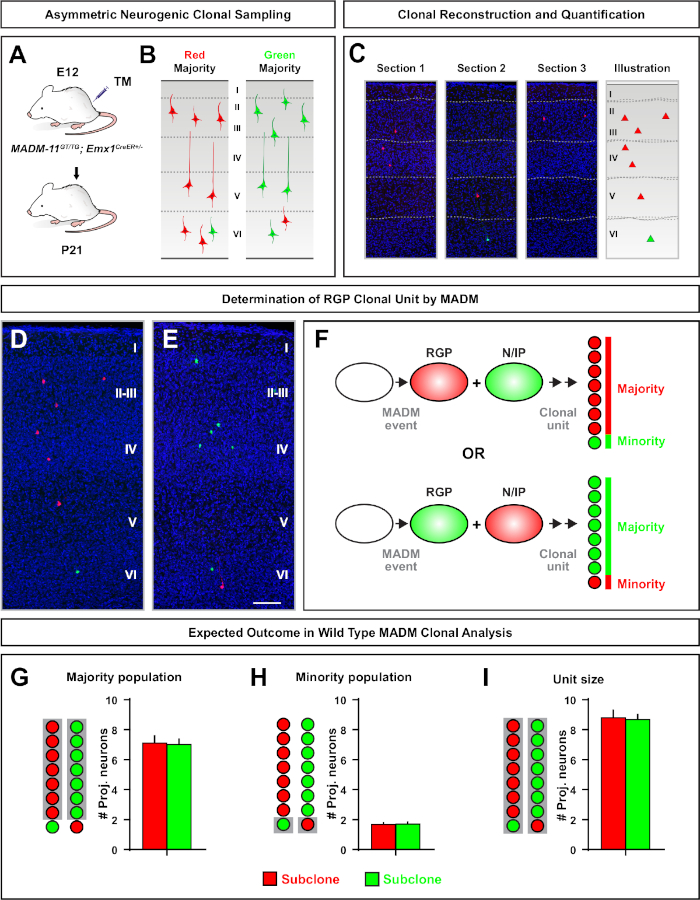
Abbildung 5: MADM-Klonanalyse zur Quantifizierung des RGP-vermittelten Neuronenausgangs. Analyse der exzitatorischen Neuronenproduktion (Einheit) durch einzelne neurogene RGPs auf klonaler Ebene mit MADM7,11. (A) Experimentelles Paradigma zur Induktion meist asymmetrischer MADM-Klone im sich entwickelnden Kortex. (B) Mögliche asymmetrische Klonergebnisse mit der Mehrheit der Subklone, die entweder in grün oder rot (C) repräsentative aufeinander folgende Abschnitte beschriftet sind, die einen einzelnen neurogenen asymmetrischen Klon (D,E) 3D-Rekonstruktionsbilder repräsentativer asymmetrischer G2-X MADM-Klone mit Mehrheitspopulation in rot (D) oder grün (E) in MADM-11GT/TGumfassen; Emx1-CreERT2+/- mit TM-Induktion bei E12 und Analyse bei P21. Beachten Sie, dass sowohl grüne als auch rot beschriftete Zellen wild sind. (F) Schematisch, der die beiden möglichen experimentellen MADM-Klonergebnisse anzeigt. (G) Quantifizierung der Größe der Mehrheitsbevölkerung, die sich aus der Erneuerung von RGPs in MADM-11-Klonen ergibt. (H) Quantifizierung der Größe der Minderheitsbevölkerung, die sich aus der Erneuerung von RGPs in MADM-11-Klonen ergibt. (I) Quantifizierung der einheitlichen Größe asymmetrischer neurogener MADM-11-Klone. Hypothetische Werte könnten für den Mittelwert von SEM stehen. Skalenbalken = 100 m (D und E). TM = Tamoxifen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
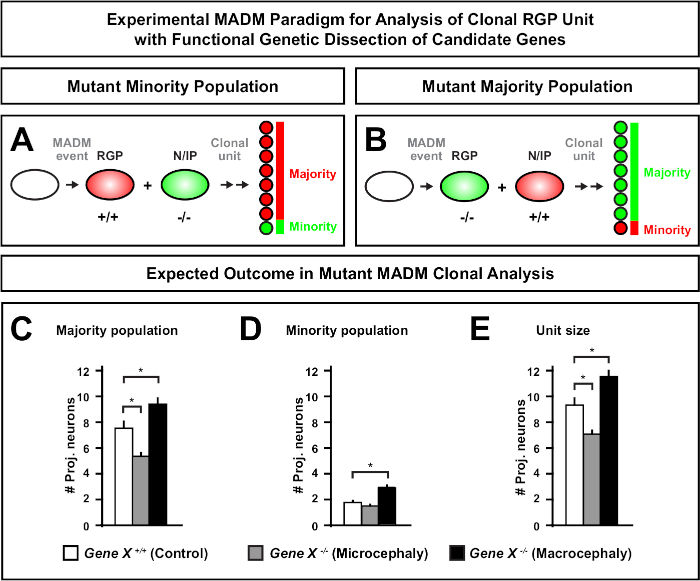
Abbildung 6: MADM-Klonanalyse zur Untersuchung von Genen, die zu Mikrozephalie und Makrozephalie führen. Hypothetische MADM-Clonalanalyseergebnisse bei der funktionellen genetischen Zerlegung von Kandidatengenen, die zu Mikrozephalie oder Makrozephalie führen. Um die zellautonomen Funktionen eines Gens von Interesse (Gene X) auf neuronenAusgang zu sezieren, erfordert MADM, dass mutierte Allele distal in die MADM-Kassetten durch meiotische Rekombination eingeführt werden (detailsweise, wie man mutierte Allele in das MADM-System einführen kann, siehe auch Abbildung 2, Hippenmeyer et al.46und Laukoter et al.46,73). (A,B) Schematische Darstellung des experimentellen MADM-Paradigmas für die funktionelle Analyse klonaler RGP-Einheiten. Der mutierte Unterklon kann entweder die Minderheitspopulation (A) oder die Mehrheitspopulation (B) bilden. (C-E) Hypothetische MADM-Clonalanalyseergebnisse bei der Quantifizierung von Kontroll-MADM (weiße Balken), Gene-X MADM-Mikrozephalie (graue Balken) und Gene-X MADM-Makrozephalie-Schwarzbalken) asymmetrische Klone. (C) Quantifizierung der Größe der Mehrheitsbevölkerung. (D) Quantifizierung der Größe der Minderheitsbevölkerung. (E) Quantifizierung der einheitlichen Größe asymmetrischer neurogener Klone. Hypothetische Werte könnten einen Mittelwert darstellen. S = Hypothetisches Szenario, in dem die Differenz in der Unterklonzellenzahl im Verhältnis zur Kontrolle signifziert werden könnte. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: MADM-Klonstudien in der Literatur. Zusammenfassung der Studien in der Literatur, die MADM klonale Abstammungsexperimente enthalten, einschließlich Desontolischer CreERT2-Treiber, TM-Dosis und Zeitpunkt der Injektion. Bitte klicken Sie hier, um diese Tabelle anzuzeigen (Rechtsklick zum Herunterladen).
Diskussion
Eine Methode zur Verwendung von MADM zum Verfolgen der Zellabstammung einzelner RGPs in vivo im sich entwickelnden Neocortex wird beschrieben. In Kombination mit TM-induzierbarem CreERT2können MADM-Ereignisse präzise getaktet werden, was eine hochqualitative und quantitative visuelle Auslesung von Stammzellteilungsmustern auf Einzelzellebene ermöglicht. Durch die Titrate der gelieferten TM-Dosis kann in idealer Situation durchschnittlich weniger als ein Klon pro kortikaler Hemisphäre erreicht werden, was eine angemessene räumliche Trennung zur eindeutigen Unterscheidung einzelner Klone bietet. Durch die Aufrechterhaltung der Gewebeintegrität erfasst diese Methode auch wichtige Informationen über Position, Morphologie und absolute Zellzahlen. MADM Kassetten auf Chr. 117,11,12,46,56,57, auf Chr. 751, und das original MADM bei Rosa2647,53,59 wurden in MADM klonalen Analysestudien verwendet. Die hohe Auflösung einzelner Zellen bietet einen beispiellosen Einblick in die Morphologie und die klonale Beziehung von Tochterzellen und ermöglicht die livee Bildgebung von sich ausbreitenden Stammzellen und entstehenden Klonen46,52.
Kaiserschnitt und Förderung von Welpen für die Analyse von Klonen an postnatalen Zeitpunkten ist ein notwendiger und kritischer Schritt im Protokoll. Abhängig vom Gesundheitszustand der mit TM behandelten schwangeren Mutter kann es nicht notwendig sein, einen Kaiserschnitt durchzuführen. Die Aufzucht der Welpen bei einer Pflegemutter ist jedoch weiterhin erforderlich, da die mit tm behandelten Mütter Probleme beim Laktieren haben kann. Es wurden keine Unterschiede in der Notwendigkeit der Förderung mit verschiedenen CreERT2-Fahrern beobachtet. Sowohl MADM-Linien als auch Pflegemütter werden auf einem ausgezüchteten CD-1-Hintergrund gepflegt. Wenn ein Kaiserschnitt nicht erforderlich ist, kann der mit TM behandelten trächtigen Muttertier, der zur Erzeugung experimenteller Welpen verwendet wird, gemäß den Grundsätzen von 3R für zusätzliche Versuchszuchten wiederverwendet werden (beachten Sie, dass dies nur möglich ist, wenn Tierversuchslizenzen diese Praxis genehmigen). Pflegemütter können für die Pflege von Welpen innerhalb von 2 Tagen nach der Geburt verwendet werden, aber höhere Erfolgsraten wurden beobachtet, wenn Pflegemütter am selben Tag wie die experimentellen Mäuse gebären, die gefördert werden müssen. Daher ist es wichtig, in Schritt 1.1 zeitzeitliche Paarungen für Pflegemütter parallel zu experimentellen Paarungen einzurichten. Die Aufrechterhaltung einer ähnlichen Wurfzahl wie der Wurf der ursprünglichen Pflegemutter kann die Überlebensrate von pflegefördernden Welpen verbessern, und daher kann die Entfernung einiger der ursprünglichen Abfälle erforderlich sein. Weitere Schritte, die die Pflege verbessern können, umfassen das Reiben der Handschuhe des Experimentators mit Wurf und Nahrung (um den Duft der Handschuhe zu entfernen); Reiben der Welpen sanft nach dem Kaiserschnitt mit Fragmenten der Pflegemutter verschmutzten Wurf und Nest; und Platzierung der Welpen in engem Kontakt mit den Welpen der Pflegemutter vor ihrer Platzierung im Pflegemauskäfig.
Wie bei anderen reporterbasierten Linienverfolgungsmethoden muss bei der Auswahl des optimalen CreERT2-Treibers für MADM-Klonexperimente sorgfältig berücksichtigt werden. Erstens muss der verwendete Promotor die Rekombinantase sowohl zeitlich als auch räumlich in der Vorläuferpopulation von Interesse ausdrücken. Diesen Promoter zu finden, kann eine Herausforderung sein, da einige Promoter Ausdrucksmuster ändern oder in verschiedenen Entwicklungsstadien zum Schweigen gebracht werden können. Zur Verbesserung der Zelltypspezifität wurden mehrere standortspezifische Rekombinatoasen verwendet, die jeweils von separaten Promotoren angetrieben werden. Wenn eine oder beide Rekombinanten in derselben Zelle exprimiert werden, wird die Zelle und ihre Nachkommen mit einem fluoreszierenden Reporter74,75,76,77. Zusammenfassend ist es wichtig, einen CreERT2-Treiber zu wählen, der spezifisch für die Population der untersuchten Vorfahren ist.
Der wichtigste Schritt bei dieser Methode ist die Identifizierung eines Klons, da alle Zellen eindeutig von einem einzelnen Rekombinationsereignis abgeleitet werden müssen (Schritt 8.1). Die Titration der TM-Konzentration gewährleistet weniger als einen Cluster rot/grüner Zellen pro Gehirnhälfte und maximiert die Wahrscheinlichkeit, einen einzelnen Klon zu analysieren (Schritt 2.2)7,11. Klone sollten verworfen werden, wenn benachbarte Zellcluster innerhalb von 500 m des Klons von Interesse auftreten. Daher ist es wichtig, mehrere Abschnitte vor und nach dem Auftreten eines Klons zu untersuchen, um sicherzustellen, dass sich keine zusätzlichen Rekombinationsereignisse in der Nähe befinden. Aufgrund des schwächeren Signals der Fluorophore ist es notwendig, die Immunhistochemie für eGFP und tdT in embryonalen Klonen durchzuführen (siehe Abschnitt 6). Dies wird nur bei erwachsenen Klonen empfohlen, wenn zusätzliche Antigene mitetikettiert werden. Bei der Abbildung von Klonen ist es wichtig, die gesamte Breite des Kortex zu erfassen, in dem sich der Klon befindet (d. h. von der Pialoberfläche bis zum Corpus callosum; siehe Schritt 8.4), um keine Zellen zu verpassen. Dies erleichtert auch die Bildausrichtung während der Bildverarbeitung (Abschnitt 9). Abschnitt 8 des Protokolls erfordert ein invertiertes konfokales Mikroskop, kann aber je nach verfügbarem Mikroskop angepasst werden. Epifluoreszenzmikroskopie kann verwendet werden, aber konfokale Mikroskopie wird empfohlen, da dies zu einer Abnahme der Lichtkontamination von außerhalb der Fokusebene führt. Es ist auch wichtig, dass die Laserintensität und -verstärkung so eingestellt wird, dass grüne, rote und gelbe Zellen eindeutig identifiziert werden können. Unabhängig vom Setup wird empfohlen, ein Ziel von mindestens 20x zu verwenden, um eine vollständige räumliche Trennung von eng positionierten Zellen zu gewährleisten. Zusätzlich zur Aufzeichnung der kortikalen Tiefe aller Zellen (Schritt 8.6) müssen kortikale Bereiche, in denen sich die Klone befinden, mit einem Hirnatlas wie dem Allen Brain Atlas oder anderen stereotaxic-Koordinatenkarten identifiziert werden. Außerdem sollte ein Dateibenennungsparadigma gewählt werden, um sicherzustellen, dass Klonbilder leicht identifizierbar sind. Die folgenden Informationen könnten in die Dateibenennung aufgenommen werden: eindeutige Bild-ID, Datumsbild wurde aufgenommen, Genotyp des Tieres, Alter der Induktion, Alter der Analyse, Bildnummer in Bezug auf den Rest der Bilder aus demselben Klon.
Die Einführung einer Mutation distal in einer MADM-Kassette ermöglicht unverwechselbar die Erzeugung von genetischen Mosaiken71 und ermöglicht die Zerlegung molekularer Regulatoren von Abstammung und Zelltypvielfalt auf klonaler Ebene7,11,46,62. Um ein genetisches Mosaik mit MADM zu erzeugen, müssen die MADM-Kassetten meiotisch mit demselben Chromosom wie das Gen von Interesse verknüpft sein (siehe Abbildung 2 für Zuchtschema). Dies beschränkt die aktuelle klonale Analyse mit MADM auf Gene, die sich auf Chr. 751, Chr. 1146, Chr. 1251und Chr. 6 distal auf den Rosa26 Lokus47befinden. Zukünftige Studien werden MADM-Kassetten verwenden, die auf jedes Chromosom ausgerichtet sind, was die Mosaikanalyse praktisch aller Gene des Mausgenoms auf klonaler Ebene ermöglicht.
Schließlich beschränkt sich MADM nicht auf die Analyse von Vorläuferzellen im sich entwickelnden Neocortex. Die Untersuchung vieler Stammzellnischen könnte von der Fähigkeit profitieren, räumlich zeitliche Anordnungen klonal verwandter Zellen zu lösen. Durch die Anwendung von MADM auf andere Regionen des Gehirns, Krankheitszustände (z. B. Krebs) oder in anderen Geweben47,50,51,52,53,54,55,56,57,58,59, Studien ergaben Abstammungsbeziehungen in Klonen, die aus verschiedenen Klassen von Vorläufer- und Stammzellen stammen (siehe Tabelle 1 für aktuelle Liste der CLonalstudien madM). Eine weitere interessante zukünftige Anwendung von MADM ist es, es mit zusätzlichen funktionalen oder subzellulären Reportern zu kombinieren, die den Grad der Informationen erhöhen würden, die von Klonen erworben werden können.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken allen Mitgliedern des Hippenmeyer-Labors für die Diskussion, der Bioimaging Facility, Life Science Facility und der Pre-Clinical Facility am IST Austria für die technische Unterstützung. Diese Arbeit wurde von den institutionellen Fonds des IST Austria unterstützt; R.B. erhielt Unterstützung vom Programm DesÖsterreichischen Wissenschaftsfonds (FWF) Lise-Meitner (M 2416); N.A erhielt Unterstützung vom Wissenschaftsfonds (FWF) Firnberg-Programm (T 1031); GC erhielt Unterstützung aus dem Forschungs- und Innovationsprogramm Horizont 2020 der Europäischen Union im Rahmen der Marie-Skodowska-Curie-Zuschussvereinbarung Nr. 754411 als POSTdoktorandin von ISTplus; Unterstützung erhielt A.H. von einem ÖAW DOC (Doktorandenstipendium der Österreichischen Akademie der Wissenschaften). Diese Studie wurde auch vom Europäischen Forschungsrat (ERC) im Rahmen des Forschungs- und Innovationsprogramms Horizont 2020 der Europäischen Union (Grant Agreement No 725780 LinPro) an S.H. unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1 mL tuberculin syringe (Omnifix Luer Lock) | Braun | 9204512N | |
| 1,4-diazabicyclooctane (DABCO) | Roth | 0718.2 | |
| 10 mL Syringe (Omnifix Luer Lock) | Braun | 8508429N | |
| 15 mL conical centrifuge | Sarstedt | 65.554.502 | |
| 24 multi-well dishes | Roth/Greiner Bio-one | CE56.1 | |
| 27- gauge x 3/4 needle (Sterican) | Braun | 16010256E | |
| Corn oil | Sigma | C8267-500ML | |
| Coverslips (24 x 60 mm #1) | Thermo Fisher Scientific (Menzel) | 15747592 | |
| Cryostat Cryostar NX70 | Thermo Fisher Scientific | 957000H | |
| Dako Pen (Wax marker) | Agilent | S200230-2 | |
| DAPI (4',6-Diamidino-2-Phenylindole, Dihydrochloride) | Invitrogen | D1306 | |
| Disposable microtome blade (MX35 Ultra) | Thermo Fisher Scientific | 705830 | |
| Fine Forceps (Dumont #5) | Fine Science Tools (FST) | 11254-20 | |
| Glass anti-roll plate | Histocom | M 449980 | |
| Glycerol | Sigma | G5516 | |
| LSM 800 Confocal | Zeiss | ||
| Mounting medium | 25 mg/mL DAPCO, 6 g Glycerol, 2.4 g Mowiol 4-88, 6 mL dH2O, 12 mL 0.2 M Tris-HCl (pH 8.5) | ||
| Mowiol 4-88 | Roth | 0713.2 | |
| Normal donkey serum | Innovative Research | IGDNSER100ML | |
| Paraformaldehyde | Sigma | 441244-1KG | |
| Peristaltic pump 323E/D 400RPM | Watson-Marlow | 036.3124.00A | |
| Sucrose | Sigma | S8501-5KG | |
| Superfrost plus glass slides | Thermo Fisher Scientific | J1800AMNT | |
| Tamoxifen | Sigma | T5648 | |
| Tissue Embedding mold T-12 (22mm square) | Polysciences Inc. | 18986-1 | |
| Tissue-Tek O.C.T | Sakura | 4583 | |
| Triton X-100 | Sigma | T8787-250ML | |
| Trizma hydrochloride | Sigma | 93363 | |
| Tween-20 | Sigma | P9416-100ML | |
| Software and Plugins: | |||
| Fiji | 1.52p | Fiji | |
| MultiStackReg | 1.45 | Download link | |
| TurboReg | EPFL Bioimaging | ||
| Zen Blue | 2.6 | Zeiss | |
| Experimental Models: Organisms/Strains: | |||
| Mouse: Emx1-CreER | The Jackson Laboratory | JAX:027784 | |
| Mouse: MADM-11-GT | The Jackson Laboratory | JAX:013749 | |
| Mouse: MADM-11-TG | The Jackson Laboratory | JAX:013751 | |
| Primary antibodies: | |||
| Chicken anti-GFP 1:500 | Aves Labs | GFP-1020 | |
| Goat anti-tdTomato 1:500 | Sicgen Antibodies | AB8181-200 | |
| Rabbit anti-RFP 1:500 | MBL | PM005 | |
| Secondary antibodies: | |||
| Donkey Anti-Chicken Alexa Fluor 488 1:500 | Jackson Immuno Research | 715-475-150 | |
| Donkey Anti-Goat Cy3 1:500 | Jackson Immuno Research | 705-165-147 | |
| Donkey Anti-Rabbit Cy3 1:500 | Jackson Immuno Research | 711-165-152 |
Referenzen
- Malatesta, P., et al. Neuronal or glial progeny: regional differences in radial glia fate. Neuron. 37 (5), 751-764 (2003).
- Miyata, T., Kawaguchi, A., Okano, H., Ogawa, M. Asymmetric inheritance of radial glial fibers by cortical neurons. Neuron. 31 (5), 727-741 (2001).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Taverna, E., Götz, M., Huttner, W. B. The Cell Biology of Neurogenesis: Toward an Understanding of the Development and Evolution of the Neocortex. Annual Review of Cell and Developmental Biology. 30 (1), 465-502 (2014).
- Desikan, R. S., Barkovich, A. J. Malformations of cortical development. Annals of Neurology. 80 (6), 797-810 (2016).
- Gao, R., Penzes, P. Common mechanisms of excitatory and inhibitory imbalance in schizophrenia and autism spectrum disorders. Current Molecular Medicine. 15 (2), 146-167 (2015).
- Beattie, R., et al. Mosaic Analysis with Double Markers Reveals Distinct Sequential Functions of Lgl1 in Neural Stem Cells. Neuron. 94 (3), 517-533 (2017).
- Hippenmeyer, S. Molecular pathways controlling the sequential steps of cortical projection neuron migration. Advances in Experimental Medicine and Biology. 800, 1-24 (2014).
- Lodato, S., Arlotta, P. Generating neuronal diversity in the mammalian cerebral cortex. Annual Review of Cell and Developmental Biology. 31, 699-720 (2015).
- Hansen, A. H., Duellberg, C., Mieck, C., Loose, M., Hippenmeyer, S. Cell Polarity in Cerebral Cortex Development-Cellular Architecture Shaped by Biochemical Networks. Frontiers in Cellular Neuroscience. 11, 176 (2017).
- Gao, P., et al. Deterministic progenitor behavior and unitary production of neurons in the neocortex. Cell. 159 (4), 775-788 (2014).
- Llorca, A., et al. A stochastic framework of neurogenesis underlies the assembly of neocortical cytoarchitecture. eLife. 8, e51381 (2019).
- Ma, J., Shen, Z., Yu, Y. C., Shi, S. H. Neural lineage tracing in the mammalian brain. Current Opinion in Neurobiology. 50, 7-16 (2018).
- Caviness, V., Takahashi, T., Nowakowski, R. Numbers, time and neocortical neuronogenesis: a general developmental and evolutionary model. Trends in Neurosciences. 18 (9), 379-383 (1995).
- Schmechel, D. E., Rakic, P. A Golgi study of radial glial cells in developing monkey telencephalon: morphogenesis and transformation into astrocytes. Anatomy and Embryology. 156 (2), 115-152 (1979).
- Kessaris, N., et al. Competing waves of oligodendrocytes in the forebrain and postnatal elimination of an embryonic lineage. Nature Neuroscience. 9 (2), 173-179 (2006).
- Magavi, S., Friedmann, D., Banks, G., Stolfi, A., Lois, C. Coincident generation of pyramidal neurons and protoplasmic astrocytes in neocortical columns. The Journal of Neuroscience. 32 (14), 4762-4772 (2012).
- Anthony, T. E., Klein, C., Fishell, G., Heintz, N. Radial glia serve as neuronal progenitors in all regions of the central nervous system. Neuron. 41 (6), 881-890 (2004).
- Voigt, T. Development of glial cells in the cerebral wall of ferrets: direct tracing of their transformation from radial glia into astrocytes. The Journal of Comparative Neurology. 289 (1), 74-88 (1989).
- Amberg, N., Laukoter, S., Hippenmeyer, S. Epigenetic cues modulating the generation of cell-type diversity in the cerebral cortex. Journal of Neurochemistry. 149 (1), 12-26 (2019).
- Beattie, R., Hippenmeyer, S. Mechanisms of Radial Glia Progenitor Cell Lineage Progression. FEBS letters. 591 (24), 3993-4008 (2017).
- Telley, L., et al. Temporal patterning of apical progenitors and their daughter neurons in the developing neocortex. Science. 364 (6440), eaav2522 (2019).
- Oberst, P., et al. Temporal plasticity of apical progenitors in the developing mouse neocortex. Nature. 573 (7774), 370-374 (2019).
- Telley, L., et al. Sequential transcriptional waves direct the differentiation of newborn neurons in the mouse neocortex. Science. 351 (6280), 1443 (2016).
- Deppe, U., et al. Cell lineages of the embryo of the nematode Caenorhabditis elegans. Proceedings of the National Academy of Sciences of the United States of America. 75 (1), 376-380 (1978).
- Woodworth, M. B., Girskis, K. M., Walsh, C. A. Building a lineage from single cells: genetic techniques for cell lineage tracking. Nature Reviews Genetics. 18 (4), 230-244 (2017).
- Masuyama, N., Mori, H., Yachie, N. DNA barcodes evolve for high-resolution cell lineage tracing. Current Opinion in Chemical Biology. 52, 63-71 (2019).
- Legue, E., Joyner, A. L. Chapter Ten-Genetic Fate Mapping Using Site-Specific Recombinases. Methods in Enzymology. 477, 153-181 (2010).
- Postiglione, M. P., Hippenmeyer, S. Monitoring neurogenesis in the cerebral cortex: an update. Future Neurology. 9 (3), 323-340 (2014).
- Espinosa-Medina, I., Garcia-Marques, J., Cepko, C., Lee, T. High-throughput dense reconstruction of cell lineages. Open Biology. 9 (12), 190229 (2019).
- Hwang, B., et al. Lineage tracing using a Cas9-deaminase barcoding system targeting endogenous L1 elements. Nature Communications. 10 (1), 1234 (2019).
- Kalhor, R., et al. Developmental barcoding of whole mouse via homing CRISPR. Science. 361 (6405), eaat9804 (2018).
- García-Moreno, F., Vasistha, N. A., Begbie, J., Molnár, Z. CLoNe is a new method to target single progenitors and study their progeny in mouse and chick. Development. 141 (7), 1589-1598 (2014).
- Loulier, K., et al. Multiplex Cell and Lineage Tracking with Combinatorial Labels. Neuron. 81 (3), 505-520 (2014).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Snippert, H. J., et al. Intestinal Crypt Homeostasis Results from Neutral Competition between Symmetrically Dividing Lgr5 Stem Cells. Cell. 143 (1), 134-144 (2010).
- Amitai-Lange, A., et al. A method for lineage tracing of corneal cells using multi-color fluorescent reporter mice. Journal of Visualized Experiments. (106), e53370 (2015).
- Vasistha, N. A., et al. Cortical and Clonal Contribution of Tbr2 Expressing Progenitors in the Developing Mouse Brain. Cerebral Cortex. 25 (10), 3290-3302 (2015).
- Chen, F., LoTurco, J. A method for stable transgenesis of radial glia lineage in rat neocortex by piggyBac mediated transposition. Journal of Neuroscience Methods. 207 (2), 172-180 (2012).
- Siddiqi, F., et al. Fate mapping by piggyBac transposase reveals that neocortical GLAST+ progenitors generate more astrocytes than Nestin+ progenitors in rat neocortex. Cerebral Cortex. 24 (2), 508-520 (2014).
- Yoshida, A., et al. Simultaneous expression of different transgenes in neurons and glia by combining in utero electroporation with the Tol2 transposon-mediated gene transfer system. Genes to Cells. 15 (5), 501-512 (2010).
- García-Marqués, J., López-Mascaraque, L. Clonal Identity Determines Astrocyte Cortical Heterogeneity. Cerebral Cortex. 23 (6), 1463-1472 (2013).
- Clavreul, S., et al. Cortical astrocytes develop in a plastic manner at both clonal and cellular levels. Nature Communications. 10 (1), 4884 (2019).
- Kim, G. B., et al. Rapid Generation of Somatic Mouse Mosaics with Locus-Specific, Stably Integrated Transgenic Elements. Cell. 179 (1), 251-267 (2019).
- Pilz, G. A., et al. Live imaging of neurogenesis in the adult mouse hippocampus. Science. 359 (6376), 658 (2018).
- Hippenmeyer, S., et al. Genetic Mosaic Dissection of Lis1 and Ndel1 in Neuronal Migration. Neuron. 68 (4), 695-709 (2010).
- Zong, H., Espinosa, J. S., Su, H. H., Muzumdar, M. D., Luo, L. Mosaic analysis with double markers in mice. Cell. 121 (3), 479-492 (2005).
- Ramón y Cajal, S. . Histologie du système nerveux de l'homme et des vertébrés. , (1911).
- Cowan, W. M. The emergence of modern neuroanatomy and developmental neurobiology. Neuron. 20 (3), 413-426 (1998).
- Ali, S. R., et al. Existing cardiomyocytes generate cardiomyocytes at a low rate after birth in mice. Proceedings of the National Academy of Sciences of the United States of America. 111 (24), 8850-8855 (2014).
- Hippenmeyer, S., Johnson, R. L., Luo, L. Mosaic analysis with double markers reveals cell-type-specific paternal growth dominance. Cell Reports. 3 (3), 960-967 (2013).
- Riccio, P., Cebrian, C., Zong, H., Hippenmeyer, S., Costantini, F. Ret and Etv4 promote directed movements of progenitor cells during renal branching morphogenesis. PLoS Biology. 14 (2), e1002382 (2016).
- Bonaguidi, M. A., et al. In vivo clonal analysis reveals self-renewing and multipotent adult neural stem cell characteristics. Cell. 145 (7), 1142-1155 (2011).
- Mayer, C., et al. Clonally Related Forebrain Interneurons Disperse Broadly across Both Functional Areas and Structural Boundaries. Neuron. 87 (5), 989-998 (2015).
- Muzumdar, M. D., et al. Clonal dynamics following p53 loss of heterozygosity in Kras-driven cancers. Nature Communications. 7, 12685 (2016).
- Shi, W., et al. Ontogenetic establishment of order-specific nuclear organization in the mammalian thalamus. Nature Neuroscience. 20, 516 (2017).
- Wong, S. Z. H., et al. In vivo clonal analysis reveals spatiotemporal regulation of thalamic nucleogenesis. PLoS Biology. 16 (4), e2005211 (2018).
- Xu, H. T., et al. Distinct Lineage-Dependent Structural and Functional Organization of the Hippocampus. Cell. 157 (7), 1552-1564 (2014).
- Brennand, K., Huangfu, D., Melton, D. All β Cells Contribute Equally to Islet Growth and Maintenance. PLoS Biology. 5 (7), e163 (2007).
- Ortiz-Alvarez, G., et al. Adult neural stem cells and multiciliated ependymal cells share a common lineage regulated by the geminin family members. Neuron. 102 (1), 159-172 (2019).
- Kaplan, E. S., Ramos-Laguna, K. A., Mihalas, A. B., Daza, R. A. M., Hevner, R. F. Neocortical Sox9+ radial glia generate glutamatergic neurons for all layers, but lack discernible evidence of early laminar fate restriction. Neural Development. 12 (1), 14 (2017).
- Lv, X., et al. TBR2 coordinates neurogenesis expansion and precise microcircuit organization via Protocadherin 19 in the mammalian cortex. Nature Communications. 10 (1), 3946 (2019).
- Mihalas, A. B., Hevner, R. F. Clonal analysis reveals laminar fate multipotency and daughter cell apoptosis of mouse cortical intermediate progenitors. Development. 145 (17), dev164335 (2018).
- Picco, N., et al. A mathematical insight into cell labelling experiments for clonal analysis. Journal of Anatomy. 235 (3), 687-696 (2019).
- Johnson, C. A., Ghashghaei, H. T. Sp2 regulates late neurogenic but not early expansive divisions of neural stem cells underlying population growth in the mouse cortex. Development. , (2020).
- Espinosa, J. S., Luo, L. Timing Neurogenesis and Differentiation: Insights from Quantitative Clonal Analyses of Cerebellar Granule Cells. The Journal of Neuroscience. 28 (10), 2301 (2008).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature Methods. 9 (7), 671-675 (2012).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Liu, Y., et al. Tamoxifen-independent recombination in the RIP-CreER mouse. PLoS One. 5 (10), e13533 (2010).
- Klingler, E., Jabaudon, D. Do progenitors play dice?. eLife. 9, e54042 (2020).
- Hippenmeyer, S. Dissection of gene function at clonal level using mosaic analysis with double markers. Frontiers in Biology. 8 (6), 557-568 (2013).
- Rulands, S., Simons, B. D. Tracing cellular dynamics in tissue development, maintenance and disease. Current Opinion in Cell Biology. 43, 38-45 (2016).
- Laukoter, S., et al. Imprinted Cdkn1c genomic locus cell-autonomously promotes cell survival in cerebral cortex development. Nature Communications. 11 (1), 195 (2020).
- Daigle, T. L., et al. A Suite of Transgenic Driver and Reporter Mouse Lines with Enhanced Brain-Cell-Type Targeting and Functionality. Cell. 174 (2), 465-480 (2018).
- He, M., et al. Strategies and Tools for Combinatorial Targeting of GABAergic Neurons in Mouse Cerebral Cortex. Neuron. 91 (6), 1228-1243 (2016).
- Yamamoto, M., et al. A multifunctional reporter mouse line for Cre- and FLP-dependent lineage analysis. Genesis. 47 (2), 107-114 (2009).
- Plummer, N. W., et al. Expanding the power of recombinase-based labeling to uncover cellular diversity. Development. 142 (24), 4385 (2015).
- Imayoshi, I., Ohtsuka, T., Metzger, D., Chambon, P., Kageyama, R. Temporal regulation of Cre recombinase activity in neural stem cells. Genesis. 44 (5), 233-238 (2006).
- Sasaki, S., et al. Complete loss of Ndel1 results in neuronal migration defects and early embryonic lethality. Molecular and Cellular Biology. 25 (17), 7812-7827 (2005).
- Acampora, D., et al. Epilepsy and brain abnormalities in mice lacking the Otx1 gene. Nature Genetics. 14 (2), 218-222 (1996).
- Soeda, T., et al. Sox9-expressing precursors are the cellular origin of the cruciate ligament of the knee joint and the limb tendons. Genesis. 48 (11), 635-644 (2010).
- Klezovitch, O., Fernandez, T. E., Tapscott, S. J., Vasioukhin, V. Loss of cell polarity causes severe brain dysplasia in Lgl1 knockout mice. Genes & Development. 18 (5), 559-571 (2004).
- Pimeisl, I. M., et al. Generation and characterization of a tamoxifen-inducible EomesCreER mouse line. Genesis. 51 (10), 725-733 (2013).
- Nakagawa, N., et al. Memo1-Mediated Tiling of Radial Glial Cells Facilitates Cerebral Cortical Development. Neuron. 103 (5), 836-852 (2019).
- Nowotschin, S., et al. The T-box transcription factor Eomesodermin is essential for AVE induction in the mouse embryo. Genes & Development. 27 (9), 997-1002 (2013).
- Balordi, F., Fishell, G. Mosaic removal of hedgehog signaling in the adult SVZ reveals that the residual wild-type stem cells have a limited capacity for self-renewal. Journal of Neuroscience. 27 (52), 14248-14259 (2007).
- Liang, H., et al. Neural development is dependent on the function of specificity protein 2 in cell cycle progression. Development. 140 (3), 552-561 (2013).
- Guo, C., Yang, W., Lobe, C. G. A Cre recombinase transgene with mosaic, widespread tamoxifen-inducible action. Genesis. 32 (1), 8-18 (2002).
- Ahn, S., Joyner, A. L. In vivo analysis of quiescent adult neural stem cells responding to Sonic hedgehog. Nature. 437 (7060), 894-897 (2005).
- Storm, R., et al. The bHLH transcription factor Olig3 marks the dorsal neuroepithelium of the hindbrain and is essential for the development of brainstem nuclei. Development. 136 (2), 295-305 (2009).
- Koundakjian, E. J., Appler, J. L., Goodrich, L. V. Auditory neurons make stereotyped wiring decisions before maturation of their targets. Journal of Neuroscience. 27 (51), 14078-14088 (2007).
- Dor, Y., Brown, J., Martinez, O. I., Melton, D. A. Adult pancreatic β-cells are formed by self-duplication rather than stem-cell differentiation. Nature. 429 (6987), 41-46 (2004).
- Sohal, D. S., et al. Temporally regulated and tissue-specific gene manipulations in the adult and embryonic heart using a tamoxifen-inducible Cre protein. Circulation Research. 89 (1), 20-25 (2001).
- Ventura, A., et al. Restoration of p53 function leads to tumour regression in vivo. Nature. 445 (7128), 661-665 (2007).
- Johnson, L., et al. Somatic activation of the K-ras oncogene causes early onset lung cancer in mice. Nature. 410 (6832), 1111-1116 (2001).
- Tasic, B., et al. Extensions of MADM (mosaic analysis with double markers) in mice. PLoS One. 7 (3), e33332 (2012).
- Yu, J., Carroll, T. J., McMahon, A. P. Sonic hedgehog regulates proliferation and differentiation of mesenchymal cells in the mouse metanephric kidney. Development. 129 (22), 5301-5312 (2002).
- Zhao, H., et al. Role of fibroblast growth factor receptors 1 and 2 in the ureteric bud. Developmental Biology. 276 (2), 403-415 (2004).
- Schuchardt, A., D'Agati, V., Larsson-Blomberg, L., Costantini, F., Pachnis, V. Defects in the kidney and enteric nervous system of mice lacking the tyrosine kinase receptor Ret. Nature. 367 (6461), 380-383 (1994).
- Livet, J., et al. ETS gene Pea3 controls the central position and terminal arborization of specific motor neuron pools. Neuron. 35 (5), 877-892 (2002).
- Volz, K. S., et al. Pericytes are progenitors for coronary artery smooth muscle. eLife. 4, e10036 (2015).
- Cai, C. L., et al. A myocardial lineage derives from Tbx18 epicardial cells. Nature. 454 (7200), 104-108 (2008).
- Lasrado, R., et al. Lineage-dependent spatial and functional organization of the mammalian enteric nervous system. Science. 356 (6339), 722-726 (2017).
- Matsuoka, T., et al. Neural crest origins of the neck and shoulder. Nature. 436 (7049), 347-355 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten