Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Registro de la actividad de la red en circuitos nociceptivos espinales utilizando matrices de microelectrodos
En este artículo
Resumen
Se describe el uso combinado de la tecnología de matriz de microelectrodos y la estimulación química inducida por 4-aminopiridina para investigar la actividad nociceptiva a nivel de red en el asta dorsal de la médula espinal.
Resumen
Los roles y la conectividad de tipos específicos de neuronas dentro del asta dorsal de la médula espinal (DH) se están delineando a un ritmo rápido para proporcionar una visión cada vez más detallada de los circuitos que sustentan el procesamiento del dolor espinal. Sin embargo, los efectos de estas conexiones para una actividad de red más amplia en la DH siguen siendo menos conocidos porque la mayoría de los estudios se centran en la actividad de neuronas individuales y microcircuitos pequeños. Alternativamente, el uso de matrices de microelectrodos (AMUMA), que pueden monitorear la actividad eléctrica en muchas células, proporciona una alta resolución espacial y temporal de la actividad neuronal. Aquí, se describe el uso de AMUMA con cortes de médula espinal de ratón para estudiar la actividad de DH inducida por circuitos de DH químicamente estimulantes con 4-aminopiridina (4-AP). La actividad rítmica resultante está restringida a la DH superficial, estable en el tiempo, bloqueada por tetrodotoxina, y puede ser investigada en diferentes orientaciones de corte. Juntos, esta preparación proporciona una plataforma para investigar la actividad del circuito DH en tejidos de animales ingenuos, modelos animales de dolor crónico y ratones con función nociceptiva genéticamente alterada. Además, los registros de MEA en cortes de médula espinal estimulados con 4-AP se pueden utilizar como una herramienta de detección rápida para evaluar la capacidad de los nuevos compuestos antinociceptivos para interrumpir la actividad en la DH de la médula espinal.
Introducción
Las funciones de tipos específicos de interneuronas inhibitorias y excitatorias dentro de la DH de la médula espinal se están descubriendo a un ritmo rápido 1,2,3,4. Juntas, las interneuronas constituyen más del 95% de las neuronas en la DH y están involucradas en el procesamiento sensorial, incluida la nocicepción. Además, estos circuitos interneuronales son importantes para determinar si las señales periféricas ascienden al neuroeje para llegar al cerebro y contribuyen a la percepción del dolor 5,6,7. Hasta la fecha, la mayoría de los estudios han investigado el papel de las neuronas DH a nivel de análisis de una sola célula o de todo el organismo utilizando combinaciones de electrofisiología intracelular in vitro, etiquetado neuroanatómico y análisis conductual in vivo 1,3,8,9,10,11,12,13,14 . Estos enfoques han avanzado significativamente en la comprensión del papel de poblaciones específicas de neuronas en el procesamiento del dolor. Sin embargo, sigue habiendo una brecha en la comprensión de cómo los tipos específicos de células y los pequeños macrocircuitos influyen en grandes poblaciones de neuronas a nivel de microcircuito para posteriormente dar forma a la salida de la DH, las respuestas conductuales y la experiencia del dolor.
Una tecnología que puede investigar la función de macrocircuito o multicelular es la matriz de microelectrodos (MEA)15,16. Los AMUMA se han utilizado para investigar la función del sistema nervioso durante varias décadas17,18. En el cerebro, han facilitado el estudio del desarrollo neuronal, la plasticidad sináptica, el cribado farmacológico y las pruebas de toxicidad17,18. Se pueden utilizar tanto para aplicaciones in vitro como in vivo, dependiendo del tipo de MEA. Además, el desarrollo de los AMUMA ha evolucionado rápidamente, con diferentes números de electrodos y configuraciones ahora disponibles19. Una ventaja clave de los AMUMA es su capacidad para evaluar simultáneamente la actividad eléctrica en muchas neuronas con alta precisión espacial y temporal a través de múltiples electrodos15,16. Esto proporciona una lectura más amplia de cómo las neuronas interactúan en circuitos y redes, en condiciones de control y en presencia de compuestos aplicados localmente.
Un desafío de las preparaciones de DH in vitro es que los niveles de actividad en curso suelen ser bajos. Aquí, este desafío se aborda en los circuitos dh de la médula espinal utilizando el bloqueador de canales K + dependiente de voltaje, 4-aminopryidine (4-AP), para estimular químicamente los circuitos DH. Este fármaco se ha utilizado previamente para establecer la actividad eléctrica síncrona rítmica en la DH de cortes agudos de la médula espinal y en condiciones agudas in vivo 20,21,22,23,24. Estos experimentos han utilizado parches unicelulares y registro extracelular o imágenes de calcio para caracterizar la actividad inducida por 4-AP 20,21,22,23,24,25. En conjunto, este trabajo ha demostrado el requisito de transmisión sináptica excitatoria e inhibitoria y sinapsis eléctricas para la actividad rítmica inducida por 4-AP. Por lo tanto, la respuesta 4-AP se ha visto como un enfoque que desenmascara los circuitos polisinápticos nativos de DH con relevancia biológica en lugar de como un epifenómeno inducido por fármacos. Además, la actividad inducida por 4-AP exhibe un perfil de respuesta similar a los fármacos analgésicos y antiepilépticos como las condiciones de dolor neuropático y se ha utilizado para proponer nuevas dianas farmacológicas analgésicas basadas en la columna vertebral como las conexinas 20,21,22.
Aquí, se describe una preparación que combina MEA y activación química de la DH espinal con 4-AP para estudiar este circuito nociceptivo en el macrocircuito, o nivel de análisis de red. Este enfoque proporciona una plataforma estable y reproducible para investigar circuitos nociceptivos en condiciones ingenuas y neuropáticas "similares al dolor". Esta preparación también es fácilmente aplicable para probar la acción a nivel de circuito de analgésicos conocidos y para detectar analgésicos novedosos en la médula espinal hiperactiva.
Protocolo
Se realizaron estudios en ratones machos y hembras c57Bl/6 de 3 a 12 meses de edad. Todos los procedimientos experimentales se realizaron de acuerdo con el Comité de Ética y Cuidado Animal de la Universidad de Newcastle (protocolos A-2013-312 y A-2020-002).
1. Electrofisiología in vitro
- Preparación de soluciones para la preparación y grabación de cortes de médula espinal
- Líquido cefalorraquídeo artificial
NOTA: El líquido cefalorraquídeo artificial (aCSF) se utiliza en una cámara de incubación de interfaz, donde las rebanadas se almacenan hasta que comienza el registro y durante los experimentos como perfusato y diluyente para medicamentos. Véase la Tabla 1 para la composición detallada.
- Líquido cefalorraquídeo artificial
| Químico | aCSF (mM) | aCSF (g/100 ml) | ACSF sustituido por sacarosa (mM) | ACSF sustituido por sacarosa (g/100 ml) | ACSF con alto contenido de potasio (mM) | ACSF con alto contenido de potasio (g/100 ml) |
| Cloruro de sodio (NaCl) | 118 | 0.690 | - | - | 118 | 0.690 |
| Carbonato de hidrógeno sódico (NaHCO3) | 25 | 0.210 | 25 | 0.210 | 25 | 0.210 |
| Glucosa | 10 | 0.180 | 10 | 0.180 | 10 | 0.180 |
| Cloruro de potasio (KCl) | 2.5 | 0.019 | 2.5 | 0.019 | 4.5 | 0.034 |
| Fosfato de dihidrógeno sódico (NaH2PO4) | 1 | 0.012 | 1 | 0.012 | 1 | 0.012 |
| Cloruro de magnesio (MgCl2) | 1 | 0.01 | 1 | 0.01 | 1 | 0.01 |
| Cloruro de calcio (CaCl2) | 2.5 | 0.028 | 2.5 | 0.028 | 2.5 | 0.028 |
| Sacarosa | - | - | 250 | 8.558 | - | - |
Tabla 1: Composiciones artificiales del líquido cefalorraquídeo. Abreviatura: aCSF = líquido cefalorraquídeo artificial.
- Preparar aCSF que contenga (en mM) 118 NaCl, 25 NaHCO3, 10 glucosa, 2,5 KCl, 1 NaH2PO4, 1 MgCl2 y 2,5 CaCl2 añadiendo las cantidades requeridas de lo anterior, excluyendo CaCl2, a 2 L de agua destilada.
- Burbujear la solución anterior con carbógeno (95% O2, 5% CO2) durante 5 min y agregar CaCl2.
NOTA: Este paso evita la precipitación de CaCl2 , es decir, la solución no debe nublarse. Para la aplicación de fármacos durante los experimentos, diluya las soluciones de stock de fármacos en aCSF a las concentraciones finales deseadas.
- Líquido cefalorraquídeo artificial sustituido por sacarosa
NOTA: El aCSF sustituido por sacarosa se usa durante la disección y el corte de la médula espinal. Como indica el nombre, la sacarosa se sustituye por NaCl para reducir la excitación neuronal durante estos procedimientos mientras se mantiene la osmolaridad. Véase la Tabla 1 para la composición detallada.- Preparar aCSF sustituido por sacarosa que contenga (en mM) 250 sacarosa, 25 NaHCO3, 10 glucosa, 2,5 KCl, 1 NaH2PO4, 1 MgCl2 y 2,5 CaCl2 añadiendo las cantidades requeridas de todo lo anterior, excluido CaCl2, a 300 ml de agua destilada.
- Burbujear la solución con carbógeno durante 5 min y luego agregar CaCl2.
- Guarde la solución en un congelador de -80 °C durante aproximadamente 40 minutos o hasta que la solución forme una suspensión. Evite congelar sólidos y úselos mientras esté en consistencia de lodo.
- Preparación de matrices de microelectrodos
NOTA: La superficie de contacto del MEA requiere un pretratamiento para que sea hidrófilo.- Antes del experimento, llene bien el MEA con suero fetal bovino (FBS) o suero de caballo (HS) durante 30 minutos.
- Retire el FBS o HS y enjuague bien el MEA con aproximadamente cinco lavados de agua destilada hasta que el agua destilada ya no sea espumosa. Llene el pozo con aCSF, listo para su uso.
- Preparación aguda de la rebanada de la médula espinal
NOTA: La preparación de la rebanada de la médula espinal del ratón es como se describió anteriormente por Smith et al.2. Idealmente, la eliminación del agrandamiento lumbosacro no debe tomar más de 8-10 minutos (pasos 1.3.2-1.3.11 a continuación).- Anestesiar profundamente al ratón con 100 mg / kg de ketamina (i.p.) y luego decapitarlo con tijeras quirúrgicas grandes.
- Retire la piel sobre la región abdominal haciendo un pequeño corte en la piel a nivel de las caderas. Tire de la piel a ambos lados del corte rostralmente hasta que se elimine toda la piel, es decir, desde la parte superior de la caja torácica hasta la parte superior de la pelvis (tanto ventral como dorsalmente).
- Coloque el cuerpo sobre hielo y use un enfoque ventral para exponer la columna vertebral eliminando todas las vísceras y cortando las costillas laterales al esternón.
- Retire la caja torácica ventral, ambas escápulas (cortadas aproximadamente en T2) y las extremidades inferiores y la pelvis (cortadas aproximadamente en la parte superior del sacro).
- Transfiera la columna vertebral y la preparación de las costillas a un baño de disección que contenga sacarosa helada aCSF. Fije las cuatro esquinas de la preparación (superficie ventral hacia arriba) colocando alfileres a través de los músculos de la espalda baja y las costillas superiores unidas.
- Elimine todo el músculo y el tejido conectivo que recubre la superficie ventral de las vértebras con rongeurs e identifique la región vertebral sobre el agrandamiento lumbosacro, que se encuentra aproximadamente debajo de los cuerpos vertebrales T12 a L2.
- Retire un cuerpo vertebral que es caudal a la región de agrandamiento lumbosacro para proporcionar acceso a la médula espinal a medida que se encuentra en el canal vertebral.
- Usando tijeras curvas de resorte, corte a través de los pedículos vertebrales bilateralmente mientras levanta y tira del cuerpo vertebral rostralmente para separar los aspectos ventral y dorsal de las vértebras y exponer la médula espinal.
- Una vez que se extraen los cuerpos vertebrales para revelar el agrandamiento lumbosacro, limpie cuidadosamente las raíces restantes que anclan la médula espinal con tijeras de resorte hasta que la médula flote libremente.
- Aísle la médula espinal con cortes rostrales y caudales muy por encima y por debajo del agrandamiento lumbosacro, permitiendo que la región objetivo de la médula "flote libremente".
NOTA: La orientación de corte preferida determinará cómo se monta posteriormente el cable para la seccionamiento (Figura 1). - Para rodajas transversales, levante el segmento lumbosacro por una raíz unida y colóquelo sobre un bloque de poliestireno (espuma de poliestireno) precortado (1 cm x 1 cm x 1 cm) con un canal poco profundo cortado en el centro. Use adhesivo de cianoacrilato (consulte la Tabla de materiales) para unir el bloque y el cordón a la plataforma de seccionamiento y colóquelo en el baño de corte que contenga sacarosa helada aCSF (purín).
NOTA: El canal poco profundo ayuda a asegurar y orientar la médula espinal, con el lado dorsal expuesto y el extremo torácico de la médula en la parte inferior del bloque. - Para las rodajas sagitales, coloque una línea delgada de adhesivo de cianoacrilato en la plataforma de seccionamiento, levante el agrandamiento lumbosacro por una raíz adjunta y coloque el cordón a lo largo de la línea de pegamento, asegurando que una superficie lateral esté en el adhesivo y la otra mire hacia arriba. Colóquelo en el baño de corte que contenga sacarosa helada aCSF (purín).
- Para rodajas horizontales, coloque una línea delgada de adhesivo de cianoacrilato en la plataforma de seccionamiento. Levante el agrandamiento lumbosacro por una raíz unida y coloque el agrandamiento lumbosacro a lo largo de la línea de adhesivo, asegurando que la superficie ventral esté en el adhesivo y la superficie dorsal mire hacia arriba. Use raíces adheridas para colocar el cordón. Colóquelo en el baño de corte que contenga sacarosa helada aCSF (purín).
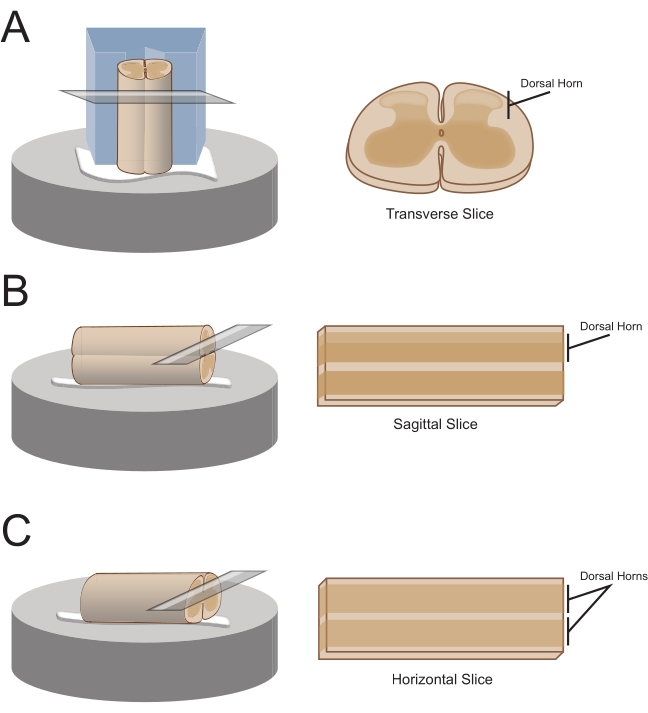
Figura 1: Orientaciones de corte de médula espinal, métodos de montaje y corte. (A) Las rodajas transversales requieren un bloque de corte de espuma de poliestireno con una ranura de soporte cortada en él. La médula espinal se apoya contra el bloque en el surco de soporte, el lado dorsal de la médula se aleja del bloque. El bloque y el cordón se pegan en una etapa de corte con adhesivo de cianoacrilato. (B) Las rodajas sagitales se preparan colocando una línea delgada de adhesivo de cianoacrilato en la etapa de corte y luego colocando la médula espinal de lado en el pegamento. (C) Las rodajas horizontales se preparan colocando una línea delgada de adhesivo de cianoacrilato en la etapa de corte y luego colocando el lado ventral de la médula espinal hacia abajo en el pegamento. Haga clic aquí para ver una versión más grande de esta figura.
- Obtenga rodajas de 300 μm de espesor (L1-L5, mismo espesor independientemente de la orientación) utilizando un microtomo vibratorio con los siguientes ajustes: velocidad 0,06 mm/s, amplitud 2,50 mm y calibrado dentro de ±0,02 desviación de amplitud de altura.
- Transfiera las rodajas a una cámara de incubación de interfaz de aire que contenga aCSF oxigenado.
- Antes de grabar, deje que las rodajas se equilibren durante 1 h a temperatura ambiente (20-24 °C).
- Grabaciones de matrices de microelectrodos
NOTA: Los siguientes pasos detallan cómo usar los datos de registro de experimentos basados en MEA en cortes de médula espinal. Se pueden utilizar varios diseños de MEA dependiendo del experimento. Los detalles de diseño de los AMUMA utilizados en estos experimentos se muestran en Cuadro 2 y Figura 2. La información detallada sobre el diseño ha sido publicada por Egert et al.26 y Thiebaud et al.27 para los AMUMA planos y tridimensionales (3D), respectivamente. Ambos tipos MEA están compuestos por 60 electrodos de nitruro de titanio, con una capa aislante de nitruro de silicio y pistas de nitruro de titanio y almohadillas de contacto.- Configuración experimental
- Encienda la computadora y la placa de interfaz, e inicie el software de grabación.
- Cargue la plantilla de grabación premontada (Figura 3A). Asigne un nombre a los archivos del día en la pestaña grabadora.
- Burbuja continua aCSF con carbógeno (5% CO2, 95% O2) durante la duración del experimento.
- Encienda el sistema de perfusión, que es controlado por una bomba peristáltica. Coloque la línea de entrada en aCSF y el extremo de entrada en un vaso de precipitados de desecho. Imprima las líneas de perfusión con aCSF.
- Preparar 4-AP y cualquier otra solución farmacológica diluyendo las existencias en 50 ml de aCSF a la concentración final requerida (por ejemplo, 200 μM para 4-AP).
- Coloque las soluciones de medicamentos en ollas de drogas y burbujee con carbógeno.
- Actividad 4-AP
- Después de la incubación, transfiera una sola rebanada de la incubadora utilizando una pipeta Pasteur de punta grande llena de aCSF.
- Coloque la rebanada en el pozo MEA y agregue aCSF adicional.
- Coloque la rebanada sobre la matriz de grabación de 60 electrodos con un pincel fino para el cabello corto. Evite poner en contacto los electrodos con el pincel o arrastrar el tejido a través de los electrodos, especialmente si usa matrices 3D.
NOTA: Dependiendo del diseño de MEA, esto se puede hacer con o sin la ayuda de un microscopio para un posicionamiento preciso. - Después de colocar la rebanada, coloque una red pesada sobre el tejido para mantenerlo en su lugar y promover un buen contacto con los electrodos MEA.
NOTA: Es posible que el segmento necesite un reposicionamiento después de la colocación de la red. - Coloque el MEA en el escenario de grabación (Figura 2A, B).
- Verifique la posición del tejido sobre los electrodos usando un microscopio invertido (aumento 2x) para confirmar que tantos electrodos como sea posible estén bajo el DH superficial (SDH). Asegúrese de que al menos 2-6 electrodos no entren en contacto con la rebanada, ya que estos electrodos son importantes para restar ruido y registrar artefactos durante el análisis (Figura 2E).
- Encienda la cámara, conéctela al dispositivo y tome una imagen de referencia de la rebanada en relación con el MEA para usarla durante el análisis.
- Presione Start DAQ en el software de grabación y confirme que todos los electrodos están recibiendo una señal clara.
NOTA: Si la señal es ruidosa, desenganche el cabezal y limpie tanto las almohadillas de contacto MEA como los contactos de resorte dorado con etanol al 70% (use una toallita de laboratorio para asegurarse de que las almohadillas y los contactos estén secos después de la limpieza). Si la señal sigue siendo ruidosa, apague los electrodos que funcionan mal en el software de grabación o anote para su exclusión más adelante durante el análisis. - Conecte las líneas de entrada y salida de perfusión al pozo MEA (previamente lleno de aCSF) y encienda el sistema de perfusión. Compruebe el caudal, idealmente 4-6 volúmenes de baño por minuto, y asegúrese de que el flujo de salida sea suficiente para evitar el desbordamiento del superfusado.
- Permita que el tejido se equilibre durante 5 minutos y luego registre 5 minutos de datos basales crudos y sin filtrar.
- Mueva la línea de entrada de perfusión de aCSF a una solución de 4-AP y espere 12 minutos para que la actividad rítmica inducida por 4-AP alcance el estado estacionario (2 minutos para que los medicamentos lleguen al baño y 10 minutos para que la actividad alcance su punto máximo y luego se estabilice).
- Registre 5 min de actividad inducida por 4-AP. Prepárese para grabaciones posteriores para probar los medicamentos o para verificar la estabilidad de 4-AP.
- Configuración experimental
| Diseños de matrices de microelectrodos | ||||
| Modelo de matriz de microelectrodos | 60MEA 200/30iR-Ti | 60-3DMEA 100/12/40iR-Ti | 60-3DMEA 200/12/50iR-Ti | 60MEA 500/30iR-Ti |
| Plana o tridimensional (3D) | Planar | 3D | 3D | Planar |
| Rejilla de electrodos | 8 x 8 | 8 x 8 | 8 x 8 | 6 x 10 |
| Espaciado de electrodos | 200 μm | 100 μm | 200 μm | 500 μm |
| Diámetro del electrodo | 30 μm | 12 μm | 12 μm | 30 μm |
| Altura del electrodo (3D) | N/A | 40 μm | 50 μm | N/A |
| Experimentos | Rodaja transversal | Rodaja transversal | Sagital + Horizontal | Sagital + Horizontal |
Tabla 2: Diseños de matrices de microelectrodos.
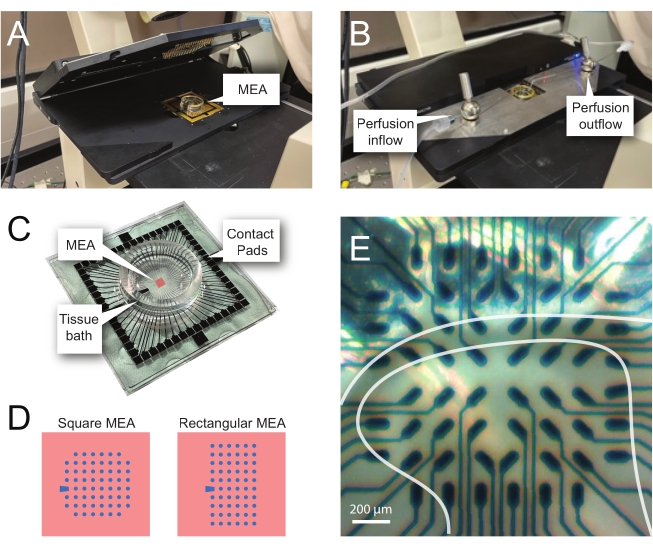
Figura 2: Posicionamiento del tejido en la matriz de microelectrodos. (A) La imagen muestra un cabezal MEA abierto con un MEA colocado en posición. (B) Igual que A con el cabezal MEA cerrado para grabaciones y el sistema de perfusión tisular en su lugar. (C) La imagen muestra un MEA suministrado por el fabricante. Se muestran las almohadillas de contacto, que interactúan con los resortes dorados del estadio principal, y el baño de tejido MEA que contiene la solución de baño de tejido y la rebanada de tejido. El área resaltada por el cuadrado rojo en el centro es la ubicación de la matriz de electrodos. (D) Los esquemas muestran las dos configuraciones de electrodos MEA utilizadas en este estudio, con más detalles presentados en la Tabla 2. El electrodo de referencia se denota por el trapecio azul. El diseño del electrodo MEA izquierdo muestra una configuración cuadrada de 60 electrodos, más utilizada en los modelos de trabajo presentados 60MEA200/30iR-Ti con electrodos de 30 μm de diámetro espaciados a 200 μm de distancia, o 200 μm espaciados y 100 μm espaciados 3-dimensionales MEA (60MEA200/12/50iR-Ti y 60MEA100/12/40iR-Ti) con electrodos de 12 μm de diámetro y 50 μm o 40 μm de altura, respectivamente. El diseño del electrodo MEA izquierdo muestra un diseño rectangular de 6 x 10 electrodos:60MEA500/30iR-Ti. (E) Imagen de alto aumento de un MEA cuadrado de 60MEA100/12/40iR-Ti con corte transversal de la médula espinal colocado para su grabación. La rebanada se encuentra en las filas de electrodos 3-8. La fila superior de electrodos, que no entran en contacto con ningún tejido, sirven como electrodos de referencia. El área SDH aparece como una banda semitransparente. En este caso, el SDH superpone los electrodos en las filas 4, 5 y 6 y las columnas 2, 3, 4, 5 y 7 del MEA. Barra de escala = 200 μm. Abreviaturas: MEA = matriz de microelectrodos; SDH = cuerno dorsal superficial. Haga clic aquí para ver una versión más grande de esta figura.
- Cambio de sectores
- Después de cada sesión de grabación, enjuague las líneas con aCSF.
- Retire el MEA del escenario.
- Retire la red y el tejido del pozo MEA, enjuáguelos bien con aCSF y repita los pasos anteriores con una nueva rebanada.
2. Tratamiento y análisis de datos
NOTA: Los siguientes pasos detallan cómo usar el software de análisis para experimentos MEA en cortes de médula espinal. Uno de los 60 electrodos sirve como referencia interna (marcado por un trapecio en la Figura 2 C, D), mientras que entre cuatro y veinticinco de los 59 restantes se colocan debajo de la SDH en una rebanada de médula espinal de ratón adulto. El análisis posterior detecta formas de onda de potencial de acción extracelular (EAP) y potencial de campo local (LFP) (ver Figura 3B para ejemplos) de la señal bruta en esta región.
- Procesamiento de datos sin procesar
- Abra el software de análisis y cargue el diseño de análisis prefabricado (Figura 3B).
- Abra el archivo de interés y anule la selección del electrodo de referencia (electrodo 15 en configuración de 8 x 8 MEA o electrodo E1 en configuración de 6 x 10 MEA) y cualquier electrodo que se considere excesivamente ruidoso.
- Establezca la ventana de tiempo para el análisis (0:00 → 5:00 min).
- Vaya a la pestaña Filtro multicanal . Seleccione Referencia compleja y seleccione los electrodos de referencia en función de la imagen tomada y las notas tomadas durante el experimento (es decir, los electrodos que no están debajo del tejido). Para aplicar y comprobar esto, pulse Explorar antes de continuar.
- Vaya a la pestaña filtro EAP y aplique un filtro Butterworth de paso altode 2º orden (corte de 200 Hz) para eliminar la actividad de LFP.
- Vaya a la pestaña filtro LFP y aplique un filtro Butterworth de paso de bandade 2º orden (frecuencias delta de 0.5-4 Hz) para eliminar la actividad de EAP.
- Vaya a la pestaña Detector EAP y seleccione Umbral automático. Marque las casillas de borde ascendente y descendente y establezca el tiempo muerto en 0,5 ms.
- Establezca umbrales positivos y negativos en función de los datos. Inspeccione los datos volviendo a la pantalla del analizador de datos sin procesar , moviendo el marcador de tiempo y, a continuación, volviendo a la pestaña del detector EAP y presionando Explorar. Repita hasta que esté convencido de que el umbral de detección establecido está capturando EAP sin capturar ruido / actividad no fisiológica. Utilice los electrodos de referencia para identificar el ruido/actividad no fisiológica.
NOTA: Es necesario asegurarse de que se detecte un número mínimo de EAP en los electrodos de referencia donde no se producirá actividad fisiológica. Sin embargo, las desviaciones bastante leves en la línea de base podrían detectarse falsamente como EAP. Esto es sin dejar de apuntar a maximizar el número de eventos reales detectados en los electrodos activos. - Vaya a la pestaña Detector LFP, seleccione el umbral manual, marque las casillas De borde ascendente y descendente y establezca el Tiempo muerto en 3 ms.
- Repita el paso 2.1.8 para un electrodo seleccionando un electrodo con actividad LFP. Una vez satisfecho, seleccione Aplicar a todos, ya que los umbrales solo se aplicarán a un solo electrodo al realizar el umbral manual.
- Al examinar los datos de LFP en la pestaña Detector , observe el número máximo de cruces de umbral para una forma de onda LFP y la separación de tiempo máximo de cruces de umbral para una forma de onda LFP para su uso en análisis posteriores.
- Presione Iniciar análisis.
- Cuando se complete el análisis, vaya a la pestaña Analizador de EAP y exporte los datos. Haga lo mismo en la pestaña analizador LFP .
- Repita este proceso para todos los demás archivos del mismo segmento.
- Después de la exportación de datos, convierta los archivos al formato xlsx para que puedan ser leídos por el script de programación utilizado. Asigne un nombre a los archivos de acuerdo con la siguiente convención para que el script proporcionado los lea: nombre del experimento (por ejemplo, datos de muestra) - número de segmento (por ejemplo, S1) - número de grabación (por ejemplo, R1) - tipo de actividad (por ejemplo, picos o SP, correspondientes a EAP o LFP, respectivamente).
NOTA: El análisis EAP descrito aquí trata el pico de canales individuales como una sola población, a pesar de que esta actividad comúnmente surgiría de múltiples neuronas en las proximidades del electrodo de grabación. Si se desea el número de neuronas que contribuyen a los EAP en un canal, se pueden aplicar técnicas de clasificación multipico descritas en otra parte para distinguir distintas poblaciones de picos en función de las características de la forma de onda28.
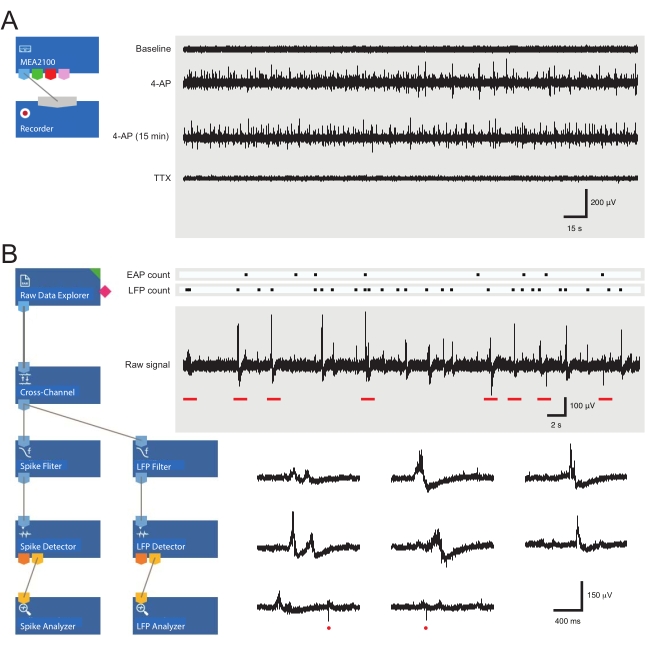
Figura 3: Diseños de herramientas de registro y análisis de datos y ejemplos de registros de matrices de microelectrodos que muestran el potencial de acción extracelular y las formas de onda del potencial de campo local. (A) Schematic muestra plantillas de grabación preconfiguradas utilizadas para la adquisición de datos MEA. La vinculación del MEA2100 y la herramienta de grabación (headstage/amplificador) permite nombrar y guardar los datos. Cuatro rastros de ejemplo de datos sin procesar (épocas derecha, de 5 minutos) fueron recolectados por un canal MEA que muestra actividad al inicio, 12 minutos después de la aplicación de 4-AP, otros 15 minutos después de la actividad establecida de 4-AP y después de la aplicación en baño de TTX (1 μM). Tenga en cuenta que la adición de 4-AP (segundo rastro) produce un claro aumento en el ruido de fondo y la actividad EAP / LFP. Es importante destacar que la actividad permanece relativamente estable durante al menos 15 minutos después de que se establece la actividad inducida por 4-AP (tercer rastro). La adición de TTX (1 μM) suprime toda actividad (traza inferior). (B) Schematic (izquierda) muestra la configuración del software del analizador para el análisis de datos. La herramienta de explorador de datos sin procesar se utiliza para importar grabaciones recopiladas por el software de grabación. Estos datos se ejecutan a través de una herramienta de filtro de canal cruzado que resta las señales de electrodos de referencia seleccionados de otros electrodos para eliminar el ruido de fondo. Los datos pasan a través del filtro EAP y las herramientas de filtro LFP para optimizar las relaciones señal-ruido para cada forma de onda. Después de este paso, los datos de ruta de EAP ingresan a la herramienta detector de EAP, donde se establecen los umbrales. Los EAP se detectan y luego se envían a la herramienta de análisis EAP donde las latencias de cada evento se registran y exportan como un txt. archivo. Se produce un flujo de trabajo idéntico para los datos de LFP utilizando un kit de herramientas de LFP correspondiente. Los rastros correctos muestran datos de un solo canal MEA que contiene varias formas de onda extracelulares. La ubicación de las señales EAP y LFP se resalta en los 'rásteres de conteo' anteriores. Las trazas inferiores son épocas de grabación superior (denotadas por barras rojas) que muestran formas de onda en una escala de tiempo expandida, incluidas varias señales LFP (tenga en cuenta la variedad de apariencias) y EAP extracelulares individuales (círculos rojos). Tenga en cuenta que la forma de onda y la polaridad de LFP / EAP varían en relación con el número de neuronas que producen estas señales, su proximidad al electrodo de grabación y su ubicación en relación con los electrodos cercanos. Abreviaturas: MEA = matriz de microelectrodos; EAP = potencial de acción extracelular; LFP = potencial de campo local; 4-AP = 4-aminopiridina; TTX = tetrodotoxina. Haga clic aquí para ver una versión más grande de esta figura.
- Análisis de sincronicidad
NOTA: La sincronicidad, o el número de eventos "coincidentes" entre dos electrodos, se determinó utilizando el criterio de coincidencia dentro del método de sincronización A-SPIKE descrito por Satuvuori et al. 29. El guión utilizado aquí solo compara los electrodos adyacentes entre sí para la eficiencia (es decir, vecinos horizontales, verticales y diagonales); sin embargo, el script podría reescribirse para comparar todos los electrodos si es necesario.- Realice análisis de datos utilizando un script de programación personalizado, que extrae las marcas de tiempo de latencia para cada electrodo de los archivos de .xlsx.
NOTA: Esto se puede hacer manualmente. - En el paso 2.1.11, registre el número máximo de cruces de umbral y la separación de tiempo máximo de los cruces de umbral para la forma de onda de un LFP. Modifique el script para introducir estos parámetros de definición de LFP para cada segmento antes de ejecutar el script.
NOTA: El umbral realizado anteriormente en el software de análisis captura claramente los EAP como un solo evento. Sin embargo, los LFP se componen de un número variable de picos dependiendo de la forma de la forma de onda y el número posterior de cruces de umbral por un evento. - Modifique el script para introducir los electrodos de interés antes del análisis.
- Para determinar la sincronicidad (definida en el script por marcos de tiempo modificables para que ocurra la actividad síncrona dentro), separe y analice las latencias extraídas para detectar eventos coincidentes.
NOTA: El script permite establecer el tiempo máximo entre eventos coincidentes. Estos se establecen en 20 ms para los EAP y 200 ms para los LFP. - Ejecute el script para extraer las marcas de tiempo de latencia.
NOTA: El archivo de salida .xlsx contiene las interpretaciones de los datos de latencia, que son recuentos de EAP y LFP, frecuencias y recuentos de eventos coincidentes para electrodos individuales y segmentos completos. Estos datos se utilizan para evaluar la frecuencia, los recuentos de EAP/LFP, el número de electrodos activos, el número de eventos coincidentes, el número de electrodos unidos y la fuerza media de estos enlaces.
- Realice análisis de datos utilizando un script de programación personalizado, que extrae las marcas de tiempo de latencia para cada electrodo de los archivos de .xlsx.
Resultados
Modelo de actividad en red en el asta dorsal de la médula espinal
La aplicación de 4-AP induce de manera confiable la actividad rítmica síncrona en la DH de la médula espinal. Dicha actividad se presenta como un aumento de los EAP y LFP. La señal posterior es una forma de onda de baja frecuencia, que se ha descrito previamente en las grabacionesMEA 30. Los cambios en la actividad de EAP y / o LFP después de la aplicación del medicamento reflejan una actividad neuronal a...
Discusión
A pesar de la importancia de la DH espinal en la señalización nociceptiva, el procesamiento y las respuestas conductuales y emocionales resultantes que caracterizan el dolor, los circuitos dentro de esta región siguen siendo poco conocidos. Un desafío clave en la investigación de este tema ha sido la diversidad de poblaciones de neuronas que comprenden estos circuitos 6,31,32. Los recientes avances en tecnologí...
Divulgaciones
Los autores no tienen conflictos de intereses que declarar.
Agradecimientos
Este trabajo fue financiado por el Consejo Nacional de Salud e Investigación Médica (NHMRC) de Australia (subvenciones 631000, 1043933, 1144638 y 1184974 a B.A.G. y R.J.C.) y el Instituto de Investigación Médica Hunter (subvención a B.A.G. y R.J.C.).
Materiales
| Name | Company | Catalog Number | Comments |
| 4-aminopyridine | Sigma-Aldrich | 275875-5G | |
| 100% ethanol | Thermo Fisher | AJA214-2.5LPL | |
| CaCl2 1M | Banksia Scientific | 0430/1L | |
| Carbonox (Carbogen - 95% O2, 5% CO2) | Coregas | 219122 | |
| Curved long handle spring scissors | Fine Science Tools | 15015-11 | |
| Custom made air interface incubation chamber | |||
| Foetal bovine serum | Thermo Fisher | 10091130 | |
| Forceps Dumont #5 | Fine Science Tools | 11251-30 | |
| Glucose | Thermo Fisher | AJA783-500G | |
| Horse serum | Thermo Fisher | 16050130 | |
| Inverted microscope | Zeiss | Axiovert10 | |
| KCl | Thermo Fisher | AJA383-500G | |
| Ketamine | Ceva | KETALAB04 | |
| Large surgical scissors | Fine Science Tools | 14007-14 | |
| Loctite 454 Instant Adhesive | Bolts and Industrial Supplies | L4543G | |
| MATLAB | MathWorks | R2018b | |
| MEAs, 3-Dimensional | Multichannel Systems | 60-3DMEA100/12/40iR-Ti, 60-3DMEA200/12/50iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in an 8x8 square grid. Electrodes are 12 µm in diameter, 40 µm (100/12/40) or 50 µm (200/12/50) high and equidistantly spaced 100 µm (100/12/40) or 200 µm (200/12/50) apart. |
| MEA headstage | Multichannel Systems | MEA2100-HS60 | |
| MEA interface board | Multichannel Systems | MCS-IFB 3.0 Multiboot | |
| MEA net | Multichannel Systems | ALA HSG-MEA-5BD | |
| MEA perfusion system | Multichannel Systems | PPS2 | |
| MEAs, Planar | Multichannel Systems | 60MEA200/30iR-Ti, 60MEA500/30iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in either a 8x8 square grid (200/30) or a 6x10 rectangular grid (500/30). Electrodes are 30 µm in diameter and equidistantly spaced 200 µm (200/30) or 500 µm (500/30) apart. |
| MgCl2 | Thermo Fisher | AJA296-500G | |
| Microscope camera | Motic | Moticam X Wi-Fi | |
| Multi Channel Analyser software | Multichannel Systems | V 2.17.4 | |
| Multi Channel Experimenter software | Multichannel Systems | V 2.17.4 | |
| NaCl | Thermo Fisher | AJA465-500G | |
| NaHCO3 | Thermo Fisher | AJA475-500G | |
| NaH2PO4 | Thermo Fisher | ACR207805000 | |
| Rongeurs | Fine Science Tools | 16021-14 | |
| Small spring scissors | Fine Science Tools | 91500-09 | |
| Small surgical scissors | Fine Science Tools | 14060-09 | |
| Sucrose | Thermo Fisher | AJA530-500G | |
| Superglue | cyanoacrylate adhesive | ||
| Tetrodotoxin | Abcam | AB120055 | |
| Vibration isolation table | Newport | VH3048W-OPT | |
| Vibrating microtome | Leica | VT1200 S |
Referencias
- Smith, K. M., et al. Calretinin positive neurons form an excitatory amplifier network in the spinal cord dorsal horn. eLife. 8, 49190 (2019).
- Smith, K. M., et al. Functional heterogeneity of calretinin-expressing neurons in the mouse superficial dorsal horn: implications for spinal pain processing. The Journal of physiology. 593 (19), 4319-4339 (2015).
- Boyle, K. A., et al. Defining a spinal microcircuit that gates myelinated afferent input: Implications for tactile allodynia. Cell Reports. 28 (2), 526-540 (2019).
- Browne, T. J., et al. Transgenic cross-referencing of inhibitory and excitatory interneuron populations to dissect neuronal heterogeneity in the dorsal horn. Frontiers in Molecular Neuroscience. 13, 32 (2020).
- Graham, B. A., Hughes, D. I. Rewards, perils and pitfalls of untangling spinal pain circuits. Current Opinion in Physiology. 11, 35-41 (2019).
- Todd, A. J. Neuronal circuitry for pain processing in the dorsal horn. Nature Reviews Neuroscience. 11 (12), 823-836 (2010).
- Hughes, D. I., Todd, A. J. Central nervous system targets: inhibitory interneurons in the spinal cord. Neurotherapeutics. 17 (3), 874-885 (2020).
- Duan, B., et al. Identification of spinal circuits transmitting and gating mechanical pain. Cell. 159 (6), 1417-1432 (2014).
- Hachisuka, J., Chiang, M. C., Ross, S. E. Itch and neuropathis itch. Pain. 159 (3), 603 (2018).
- Foster, E., et al. Targeted ablation, silencing, and activation establish glycinergic dorsal horn neurons as key components of a spinal gate for pain and itch. Neuron. 85 (6), 1289-1304 (2015).
- Bourane, S., et al. Identification of a spinal circuit for light touch and fine motor control. Cell. 160 (3), 503-515 (2015).
- Cheng, L., et al. Identification of spinal circuits involved in touch-evoked dynamic mechanical pain. Nature neuroscience. 20 (6), 804-814 (2017).
- Peirs, C., et al. Mechanical allodynia circuitry in the dorsal horn is defined by the nature of the injury. Neuron. 109 (1), 73-90 (2021).
- Huang, J., et al. Circuit dissection of the role of somatostatin in itch and pain. Nature Neuroscience. 21 (5), 707-716 (2018).
- Obien, M. E. J., Deligkaris, K., Bullmann, T., Bakkum, D. J., Frey, U. Revealing neuronal function through microelectrode array recordings. Frontiers in Neuroscience. 8, 423 (2015).
- Nam, Y., Wheeler, B. C. In vitro microelectrode array technology and neural recordings. Critical Reviews in Biomedical Engineering. 39 (1), 45-61 (2011).
- Johnstone, A. F., et al. Microelectrode arrays: a physiologically based neurotoxicity testing platform for the 21st century. Neurotoxicology. 31 (4), 331-350 (2010).
- Stett, A., et al. Biological application of microelectrode arrays in drug discovery and basic research. Analytical and Bioanalytical Chemistry. 377 (3), 486-495 (2003).
- Xu, L., et al. Trends and recent development of the microelectrode arrays (MEAs). Biosensors and Bioelectronics. 175 (1), 112854 (2020).
- Chapman, R. J., Cilia La Corte, P. F., Asghar, A. U. R., King, A. E. Network-based activity induced by 4-aminopyridine in rat dorsal horn in vitro is mediated by both chemical and electrical synapses. The Journal of Physiology. 587, 2499-2510 (2009).
- Ruscheweyh, R., Sandkühler, J. Epileptiform activity in rat spinal dorsal horn in vitro has common features with neuropathic pain. Pain. 105 (1-2), 327-338 (2003).
- Kay, C. W., Ursu, D., Sher, E., King, A. E. The role of Cx36 and Cx43 in 4-aminopyridine-induced rhythmic activity in the spinal nociceptive dorsal horn: an electrophysiological study in vitro. Physiological Reports. 4 (14), 12852 (2016).
- Jankowska, E., Lundberg, A., Rudomin, P., Sykova, E. Effects of 4-aminopyridine on synaptic transmission in the cat spinal cord. Brain Research. 240 (1), 117-129 (1982).
- Semba, K., Geller, H. M., Egger, M. D. 4-Aminopyridine induces expansion of cutaneous receptive fields of dorsal horn cells. Brain Research. 343 (2), 398-402 (1985).
- Ruscheweyh, R., Sandkühler, J. Long-range oscillatory Ca2+ waves in rat spinal dorsal horn. European Journal of Neuroscience. 22 (8), 1967-1976 (2005).
- Egert, U., et al. A novel organotypic long-term culture of the rat hippocampus on substrate-integrated multielectrode arrays. Brain Research Protocols. 2 (4), 229-242 (1998).
- Thiebaud, P., De Rooij, N., Koudelka-Hep, M., Stoppini, L. Microelectrode arrays for electrophysiological monitoring of hippocampal organotypic slice cultures. IEEE Transactions on Biomedical Engineering. 44 (11), 1159-1163 (1997).
- Rey, H. G., Pedreira, C., Quiroga, R. Q. Past, present and future of spike sorting techniques. Brain Research Bulletin. 119, 106-117 (2015).
- Satuvuori, E., et al. Measures of spike train synchrony for data with multiple time scales. Journal of Neuroscience Methods. 287, 25-38 (2017).
- Mendis, G. D. C., Morrisroe, E., Reid, C. A., Halgamuge, S. K., Petrou, S. Use of local field potentials of dissociated cultures grown on multi-electrode arrays for pharmacological assays. 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. , 952-956 (2016).
- Hughes, D. I., et al. Morphological, neurochemical and electrophysiological features of parvalbumin-expressing cells: a likely source of axo-axonic inputs in the mouse spinal dorsal horn. The Journal of Physiology. 590 (16), 3927-3951 (2012).
- Peirs, C., Seal, R. P. Neural circuits for pain: recent advances and current views. Science. 354 (6312), 578-584 (2016).
- Li, J., Baccei, M. L. Developmental regulation of membrane excitability in rat spinal lamina I projection neurons. Journal of Neurophysiology. 107 (10), 2604-2614 (2012).
- Li, J., Baccei, M. L. Pacemaker neurons within newborn spinal pain circuits. Journal of Neuroscience. 31 (24), 9010-9022 (2011).
- Sandkühler, J., Eblen-Zajjur, A. Identification and characterization of rhythmic nociceptive and non-nociceptive spinal dorsal horn neurons in the rat. Neuroscience. 61 (4), 991-1006 (1994).
- Lucas-Romero, J., Rivera-Arconada, I., Roza, C., Lopez-Garcia, J. A. Origin and classification of spontaneous discharges in mouse superficial dorsal horn neurons. Scientific Reports. 8 (1), 9735-9735 (2018).
- Antonio, L., et al. L. al. In vitro seizure like events and changes in ionic concentration. Journal of Neuroscience Methods. 260, 33-44 (2016).
- Avoli, M., Jefferys, J. G. Models of drug-induced epileptiform synchronization in vitro. Journal of Neuroscience Methods. 260, 26-32 (2016).
- Taccola, G., Nistri, A. Low micromolar concentrations of 4-aminopyridine facilitate fictive locomotion expressed by the rat spinal cord in vitro. Neuroscience. 126 (2), 511-520 (2004).
- Mitra, P., Brownstone, R. M. An in vitro spinal cord slice preparation for recording from lumbar motoneurons of the adult mouse. Journal of Neurophysiology. 107 (2), 728-741 (2012).
- Egert, U., Heck, D., Aertsen, A. Two-dimensional monitoring of spiking networks in acute brain slices. Experimental Brain Research. 142 (2), 268-274 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados