È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Registrazione dell'attività di rete nei circuiti nocicettivi spinali utilizzando array di microelettrodi
In questo articolo
Riepilogo
Viene delineato l'uso combinato della tecnologia microelectrode array e della stimolazione chimica indotta da 4-aminopiridina per studiare l'attività nocicettiva a livello di rete nel corno dorsale del midollo spinale.
Abstract
I ruoli e la connettività di specifici tipi di neuroni all'interno del corno dorsale del midollo spinale (DH) vengono delineati a un ritmo rapido per fornire una visione sempre più dettagliata dei circuiti alla base dell'elaborazione del dolore spinale. Tuttavia, gli effetti di queste connessioni per una più ampia attività di rete nel DH rimangono meno ben compresi perché la maggior parte degli studi si concentra sull'attività di singoli neuroni e piccoli microcircuiti. In alternativa, l'uso di array di microelettrodi (MEA), in grado di monitorare l'attività elettrica su molte cellule, fornisce un'elevata risoluzione spaziale e temporale dell'attività neurale. Qui, viene descritto l'uso di MEA con fette di midollo spinale di topo per studiare l'attività dh indotta da circuiti DH chimicamente stimolanti con 4-aminopiridina (4-AP). L'attività ritmica risultante è limitata al DH superficiale, stabile nel tempo, bloccata dalla tetrodotossina, e può essere studiata in diversi orientamenti delle fette. Insieme, questa preparazione fornisce una piattaforma per studiare l'attività del circuito DH nei tessuti di animali naïve, modelli animali di dolore cronico e topi con funzione nocicettiva geneticamente alterata. Inoltre, le registrazioni MEA in fette di midollo spinale stimolate da 4-AP possono essere utilizzate come strumento di screening rapido per valutare la capacità di nuovi composti antinocicettivi di interrompere l'attività nel DH del midollo spinale.
Introduzione
I ruoli di specifici tipi di interneuroni inibitori ed eccitatori all'interno del DH del midollo spinale vengono scoperti ad un ritmo rapido 1,2,3,4. Insieme, gli interneuroni costituiscono oltre il 95% dei neuroni nel DH e sono coinvolti nell'elaborazione sensoriale, compresa la nocicezione. Inoltre, questi circuiti interneuronici sono importanti per determinare se i segnali periferici salgono sul neuroassi per raggiungere il cervello e contribuire alla percezione del dolore 5,6,7. Ad oggi, la maggior parte degli studi ha studiato il ruolo dei neuroni DH a livello di analisi a singola cellula o intero organismo utilizzando combinazioni di elettrofisiologia intracellulare in vitro, etichettatura neuroanatomica e analisi comportamentale in vivo 1,3,8,9,10,11,12,13,14 . Questi approcci hanno notevolmente migliorato la comprensione del ruolo di specifiche popolazioni di neuroni nell'elaborazione del dolore. Tuttavia, rimane una lacuna nella comprensione di come specifici tipi di cellule e piccoli macro-circuiti influenzino grandi popolazioni di neuroni a livello di microcircuito per modellare successivamente l'output del DH, le risposte comportamentali e l'esperienza del dolore.
Una tecnologia in grado di studiare la funzione macro-circuito o multicellulare è il microelectrode array (MEA)15,16. I MEA sono stati utilizzati per studiare la funzione del sistema nervoso per diversi decenni17,18. Nel cervello, hanno facilitato lo studio dello sviluppo neuronale, della plasticità sinaptica, dello screening farmacologico e dei test di tossicità17,18. Possono essere utilizzati sia per applicazioni in vitro che in vivo, a seconda del tipo di MEA. Inoltre, lo sviluppo dei MEA si è evoluto rapidamente, con diversi numeri di elettrodi e configurazioni ora disponibili19. Un vantaggio chiave dei MEA è la loro capacità di valutare simultaneamente l'attività elettrica in molti neuroni con elevata precisione spaziale e temporale tramite più elettrodi15,16. Ciò fornisce una lettura più ampia di come i neuroni interagiscono in circuiti e reti, in condizioni di controllo e in presenza di composti applicati localmente.
Una sfida dei preparati DH in vitro è che i livelli di attività in corso sono in genere bassi. Qui, questa sfida viene affrontata nei circuiti DH del midollo spinale utilizzando il bloccante del canale K + voltaggio-gated, 4-aminopryidine (4-AP), per stimolare chimicamente i circuiti DH. Questo farmaco è stato precedentemente utilizzato per stabilire l'attività elettrica sincrona ritmica nel DH di fette acute del midollo spinale e in condizioni acute in vivo 20,21,22,23,24. Questi esperimenti hanno utilizzato patch monocellulare e registrazione extracellulare o imaging del calcio per caratterizzare l'attività indotta da 4-AP 20,21,22,23,24,25. Insieme, questo lavoro ha dimostrato il requisito della trasmissione sinaptica eccitatoria e inibitoria e delle sinapsi elettriche per l'attività ritmica indotta da 4-AP. Pertanto, la risposta 4-AP è stata vista come un approccio che smaschera i circuiti DH polisinaptici nativi con rilevanza biologica piuttosto che come un epifenomeno indotto da farmaci. Inoltre, l'attività indotta da 4-AP mostra un profilo di risposta simile ai farmaci analgesici e antiepilettici come le condizioni di dolore neuropatico ed è stata utilizzata per proporre nuovi bersagli farmacologici analgesici a base spinale come le connessioni 20,21,22.
Qui, viene descritta una preparazione che combina MEA e attivazione chimica del DH spinale con 4-AP per studiare questo circuito nocicettivo al macro-circuito, o a livello di rete di analisi. Questo approccio fornisce una piattaforma stabile e riproducibile per studiare i circuiti nocicettivi in condizioni ingenue e neuropatiche "simili al dolore". Questa preparazione è anche facilmente applicabile per testare l'azione a livello circuitale di analgesici noti e per lo screening di nuovi analgesici nel midollo spinale iperattivo.
Protocollo
Gli studi sono stati condotti su topi c57Bl/6 maschi e femmine di età compresa tra 3 e 12 mesi. Tutte le procedure sperimentali sono state eseguite in conformità con il Comitato per la cura degli animali e l'etica dell'Università di Newcastle (protocolli A-2013-312 e A-2020-002).
1. Elettrofisiologia in vitro
- Preparazione di soluzioni per la preparazione e la registrazione di fette di midollo spinale
- Liquido cerebrospinale artificiale
NOTA: Il liquido cerebrospinale artificiale (aCSF) viene utilizzato in una camera di incubazione di interfaccia, dove le fette vengono conservate fino all'inizio della registrazione e durante gli esperimenti sia come perfusato che come diluente per i farmaci. Vedere la Tabella 1 per la composizione dettagliata.
- Liquido cerebrospinale artificiale
| Chimico | aCSF (mM) | aCSF (g/100 mL) | ACSF (mM) sostituito dal saccarosio | ACSF sostituito con saccarosio (g/100 mL) | ACSF ad alto contenuto di potassio (mM) | ACSF ad alto contenuto di potassio (g/100 mL) |
| Cloruro di sodio (NaCl) | 118 | 0.690 | - | - | 118 | 0.690 |
| Carbonato acido di sodio (NaHCO3) | 25 | 0.210 | 25 | 0.210 | 25 | 0.210 |
| Glucosio | 10 | 0.180 | 10 | 0.180 | 10 | 0.180 |
| Cloruro di potassio (KCl) | 2.5 | 0.019 | 2.5 | 0.019 | 4.5 | 0.034 |
| Sodio diidrogeno fosfato (NaH2PO4) | 1 | 0.012 | 1 | 0.012 | 1 | 0.012 |
| Cloride di magnesio (MgCl2) | 1 | 0.01 | 1 | 0.01 | 1 | 0.01 |
| Cloruro di calcio (CaCl2) | 2.5 | 0.028 | 2.5 | 0.028 | 2.5 | 0.028 |
| Saccarosio | - | - | 250 | 8.558 | - | - |
Tabella 1: Composizioni del liquido cerebrospinale artificiale. Abbreviazione: aCSF = liquido cerebrospinale artificiale.
- Preparare un FCS contenente (in mM) 118 NaCl, 25 NaHCO3, 10 glucosio, 2,5 KCl, 1 NaH2PO4, 1 MgCl2 e 2,5 CaCl2 aggiungendo le quantità richieste di quanto sopra, escluso CaCl2, a 2 L di acqua distillata.
- Bolle la soluzione di cui sopra con carbogeno (95% O2, 5% CO2) per 5 minuti e aggiungere CaCl2.
NOTA: questo passaggio impedisce la precipitazione di CaCl2 , ovvero la soluzione non deve diventare torbida. Per l'applicazione di farmaci durante gli esperimenti, diluire le soluzioni stock di farmaci in aCSF alle concentrazioni finali desiderate.
- Liquido cerebrospinale artificiale sostituito con saccarosio
NOTA: L'aCSF sostituito con saccarosio viene utilizzato durante la dissezione e l'affettamento del midollo spinale. Come indicato dal nome, il saccarosio viene sostituito da NaCl per ridurre l'eccitazione neuronale durante queste procedure mantenendo l'osmolarità. Vedere la Tabella 1 per la composizione dettagliata.- Preparare l'aCSF sostitutivo con saccarosio contenente (in mM) 250 saccarosio, 25 NaHCO3, 10 glucosio, 2,5 KCl, 1 NaH2PO4, 1 MgCl2 e2,5 CaCl 2 aggiungendo le quantità richieste di tutto quanto sopra, escluso CaCl2, a 300 ml di acqua distillata.
- Bolle la soluzione con carbogen per 5 minuti e poi aggiungere CaCl2.
- Conservare la soluzione in un congelatore a -80 °C per circa 40 minuti o fino a quando la soluzione non forma un impasto. Evitare il congelamento del solido e utilizzare mentre si è in consistenza del liquame.
- Preparazione di array di microelettrodi
NOTA: La superficie di contatto del MEA richiede un pretrattamento per renderlo idrofilo.- Prima dell'esperimento, riempire bene il MEA con siero bovino fetale (FBS) o siero equino (HS) per 30 minuti.
- Rimuovere l'FBS o l'HS e risciacquare accuratamente MEA con circa cinque lavaggi di acqua distillata fino a quando l'acqua distillata non è più schiumosa. Riempire il pozzo con aCSF, pronto per l'uso.
- Preparazione acuta della fetta di midollo spinale
NOTA: La preparazione della fetta di midollo spinale del topo è come precedentemente descritta da Smith et al.2. Idealmente, la rimozione dell'allargamento lombosacrale non dovrebbe richiedere più di 8-10 minuti (passaggi 1.3.2-1.3.11 di seguito).- Anestetizzare profondamente il topo con 100 mg/kg di ketamina (i.p.) e poi decapitarlo con grandi forbici chirurgiche.
- Rimuovere la pelle sopra la regione addominale facendo un piccolo taglio nella pelle a livello dei fianchi. Tirare la pelle su entrambi i lati del taglio rostralmente fino a quando tutta la pelle viene rimossa, cioè dalla parte superiore della gabbia toracica alla parte superiore del bacino (sia ventralmente che dorsalmente).
- Posizionare il corpo sul ghiaccio e utilizzare un approccio ventrale per esporre la colonna vertebrale rimuovendo tutti i visceri e tagliando le costole laterali allo sterno.
- Rimuovere la gabbia toracica ventrale, entrambe le scapole (tagliate a circa T2), e gli arti inferiori e il bacino (tagliati approssimativamente nella parte superiore del sacro).
- Trasferire la colonna vertebrale e la preparazione delle costole in un bagno di dissezione contenente saccarosio ghiacciato aCSF. Fissare tutti e quattro gli angoli della preparazione (superficie ventrale verso l'alto) posizionando i perni attraverso i muscoli della parte bassa della schiena e le costole superiori attaccate.
- Rimuovere tutti i muscoli e il tessuto connettivo sovrastanti la superficie ventrale delle vertebre con i rongeurs e identificare la regione vertebrale sopra l'allargamento lombosacrale, che si trova approssimativamente sotto i corpi vertebrali da T12 a L2.
- Rimuovere un corpo vertebrale che è caudale alla regione di allargamento lombosacrale per fornire l'accesso al midollo spinale mentre si trova nel canale vertebrale.
- Usando forbici a molla curve, tagliare bilateralmente i frontoni vertebrali mentre si solleva e si tira il corpo vertebrale rostralmente per separare gli aspetti ventrali e dorsali delle vertebre ed esporre il midollo spinale.
- Una volta rimossi i corpi vertebrali per rivelare l'allargamento lombosacrale, cancellare con cura le radici rimanenti che ancorano il midollo spinale con forbici a molla fino a quando il cordone galleggia libero.
- Isolare il midollo spinale con tagli rostrali e caudali ben al di sopra e al di sotto dell'allargamento lombosacrale, consentendo alla regione target del cordone di "galleggiare liberamente".
NOTA: l'orientamento della sezione preferito determinerà la modalità di montaggio del cavo per il sezionamento (Figura 1). - Per le fette trasversali, sollevare il segmento lombosacrale con una radice attaccata e posizionarlo su un blocco di polistirene pretagliato (polistirolo) (1 cm x 1 cm x 1 cm) con un canale poco profondo tagliato al centro. Utilizzare l'adesivo cianoacrilato (vedere la tabella dei materiali) per fissare il blocco e il cavo alla piattaforma di sezionamento e posizionarlo nel bagno di taglio contenente saccarosio ghiacciato aCSF (liquame).
NOTA: Il canale poco profondo aiuta a fissare e orientare il midollo spinale, con il lato dorsale esposto e l'estremità toracica del cordone nella parte inferiore del blocco. - Per le fette sagittali, posare una sottile linea di adesivo cianoacrilato sulla piattaforma di sezionamento, sollevare l'allargamento lombosacrale con una radice attaccata e posizionare il cavo lungo la linea di colla, assicurandosi che una superficie laterale sia nell'adesivo e l'altra faccia verso l'alto. Metterlo nel bagno di taglio contenente saccarosio ghiacciato aCSF (liquame).
- Per le fette orizzontali, mettere una sottile linea di adesivo cianoacrilato sulla piattaforma di sezionamento. Sollevare l'allargamento lombosacrale con una radice attaccata e posizionare l'allargamento lombosacrale lungo la linea di adesivo, assicurandosi che la superficie ventrale sia nell'adesivo e la superficie dorsale sia rivolta verso l'alto. Utilizzare le radici attaccate per posizionare il cavo. Metterlo nel bagno di taglio contenente saccarosio ghiacciato aCSF (liquame).
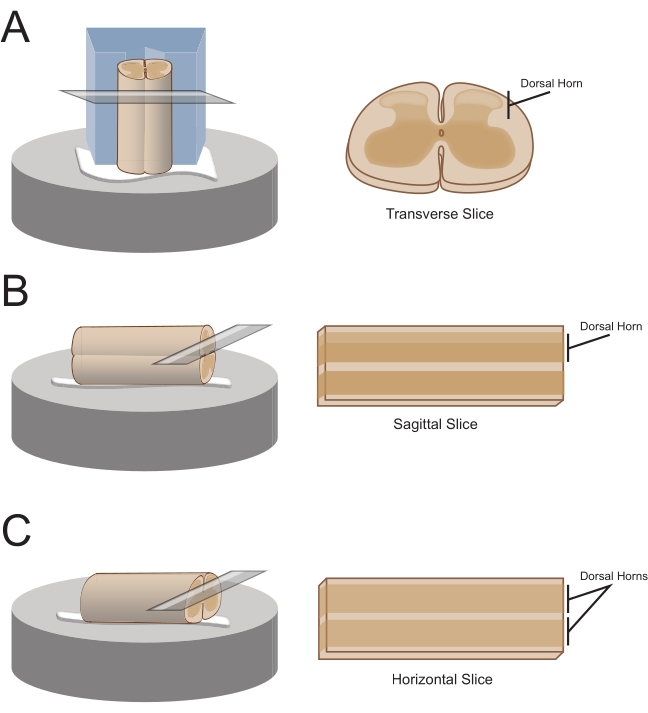
Figura 1: Orientamenti delle sezioni del midollo spinale, metodi di montaggio e taglio. (A) Le fette trasversali richiedono un blocco di taglio in polistirolo con una scanalatura di supporto tagliata al suo interno. Il midollo spinale è appoggiato contro il blocco nella scanalatura di supporto, il lato dorsale del cordone rivolto lontano dal blocco. Il blocco e la corda sono incollati su un palcoscenico di taglio con adesivo cianoacrilato. (B) Le fette sagittali vengono preparate posizionando una sottile linea di adesivo cianoacrilato sul gradino di taglio e quindi posizionando il midollo spinale su un lato sulla colla. (C) Le fette orizzontali vengono preparate posizionando una sottile linea di adesivo cianoacrilato sul palco di taglio e quindi posizionando il lato ventrale del midollo spinale verso il basso sulla colla. Fare clic qui per visualizzare una versione più grande di questa figura.
- Ottenere fette spesse 300 μm (L1-L5, stesso spessore indipendentemente dall'orientamento) utilizzando un microtomo vibrante con le seguenti impostazioni: velocità 0,06 mm/s, ampiezza 2,50 mm e calibrato entro ±0,02 di altezza deviazione dell'ampiezza.
- Trasferire le fette in una camera di incubazione dell'interfaccia dell'aria contenente aCSF ossigenato.
- Prima della registrazione, lasciare equilibrare le fette per 1 ora a temperatura ambiente (20-24 °C).
- Registrazioni con array di microelettrodi
NOTA: i passaggi seguenti descrivono in dettaglio come utilizzare i dati di registrazione da esperimenti basati su MEA su fette di midollo spinale. Diversi progetti MEA possono essere utilizzati a seconda dell'esperimento. I dettagli di progettazione per i MEA utilizzati in questi esperimenti sono mostrati in Tabella 2 e Figura 2. Informazioni dettagliate sulla progettazione sono state pubblicate da Egert et al.26 e Thiebaud et al.27 per MEA planari e tridimensionali (3D), rispettivamente. Entrambi i tipi MEA sono composti da 60 elettrodi di nitruro di titanio, con uno strato isolante di nitruro di silicio e cingoli e cuscinetti di contatto in nitruro di titanio.- Configurazione sperimentale
- Accendere il computer e la scheda di interfaccia e avviare il software di registrazione.
- Caricare il modello di registrazione preassemblato (Figura 3A). Assegna un nome ai file per il giorno nella scheda del registratore.
- Bolle continuamente aCSF con carbogeno (5% CO2, 95% O2) per tutta la durata dell'esperimento.
- Accendere il sistema di perfusione, che è controllato da una pompa peristaltica. Posizionare la linea di ingresso in aCSF e l'estremità di ingresso in un becher di scarto. Innescare le linee di perfusione con aCSF.
- Preparare 4-AP e qualsiasi altra soluzione farmacologica diluendo le scorte in 50 ml di aCSF alla concentrazione finale richiesta (ad esempio, 200 μM per 4-AP).
- Posizionare le soluzioni farmacologiche in vasi di droga e bollerle con carbogeno.
- Attività 4-AP
- Dopo l'incubazione, trasferire una singola fetta dall'incubatore utilizzando una pipetta Pasteur a punta larga riempita con aCSF.
- Posizionare bene la fetta nel MEA e aggiungere ulteriore aCSF.
- Posiziona la fetta sopra l'array di registrazione a 60 elettrodi usando un pennello a pelo corto. Evitare di contattare gli elettrodi con il pennello o trascinare il tessuto attraverso gli elettrodi, soprattutto se si utilizzano array 3D.
NOTA: A seconda del layout MEA, questo può essere fatto con o senza l'assistenza di un microscopio per un posizionamento accurato. - Dopo aver posizionato la fetta, posizionare una rete pesata sul tessuto per tenerlo in posizione e promuovere un buon contatto con gli elettrodi MEA.
NOTA: potrebbe essere necessario riposizionare la sezione dopo il posizionamento netto. - Posizionare il MEA nell'headstage di registrazione (Figura 2A,B).
- Controllare la posizione del tessuto sopra gli elettrodi utilizzando un microscopio invertito (ingrandimento 2x) per confermare che il maggior numero possibile di elettrodi si trova sotto il DH superficiale (SDH). Assicurarsi che almeno 2-6 elettrodi non entrino in contatto con la fetta poiché questi elettrodi sono importanti per sottrarre rumore e registrare artefatti durante l'analisi (Figura 2E).
- Accendere la fotocamera, collegarla al dispositivo e acquisire un'immagine di riferimento della sezione relativa al MEA da utilizzare durante l'analisi.
- Premere Avvia DAQ nel software di registrazione e verificare che tutti gli elettrodi ricevano un segnale chiaro.
NOTA: se il segnale è rumoroso, sganciare l'headstage e pulire sia i cuscinetti di contatto MEA che i contatti a molla dorata con etanolo al 70% (utilizzare una salvietta da laboratorio per assicurarsi che i pad e i contatti siano asciutti dopo la pulizia). Se il segnale è ancora rumoroso, spegnere gli elettrodi malfunzionanti nel software di registrazione o annotare per l'esclusione in un secondo momento durante l'analisi. - Collegare le linee di ingresso e di uscita della perfusione al pozzetto MEA (precedentemente riempito con aCSF) e accendere il sistema di perfusione. Controllare la portata, idealmente 4-6 volumi di bagno al minuto, e assicurarsi che il deflusso sia sufficiente per evitare il trabocco del superfusato.
- Lasciare che il tessuto si equilibri per 5 minuti e quindi registrare 5 minuti di dati di base grezzi e non filtrati.
- Spostare la linea di ingresso della perfusione da aCSF a una soluzione 4-AP e attendere 12 minuti affinché l'attività ritmica indotta da 4 AP raggiunga lo stato stazionario (2 minuti per i farmaci per raggiungere il bagno e 10 minuti per l'attività al picco e poi al plateau).
- Registra 5 minuti di attività indotta da 4 AP. Preparati per le registrazioni successive per testare i farmaci o per verificare la stabilità di 4-AP.
- Configurazione sperimentale
| Layout di array di microelettrodi | ||||
| Modello di array di microelettrodi | 60MEA 200/30iR-Ti | 60-3DMEA 100/12/40iR-Ti | 60-3DMEA 200/12/50iR-Ti | 60MEA 500/30iR-Ti |
| Planare o 3-Dimensionale (3D) | Planare | 3D | 3D | Planare |
| Griglia elettrodica | 8 x 8 | 8 x 8 | 8 x 8 | 6 x 10 |
| Spaziatura elettrodi | 200 μm | 100 μm | 200 μm | 500 μm |
| Diametro elettrodo | 30 μm | 12 μm | 12 μm | 30 μm |
| Altezza elettrodo (3D) | N/D | 40 μm | 50 μm | N/D |
| Esperimenti | Fetta trasversale | Fetta trasversale | Sagittale + Orizzontale | Sagittale + Orizzontale |
Tabella 2: Layout di array di microelettrodi.
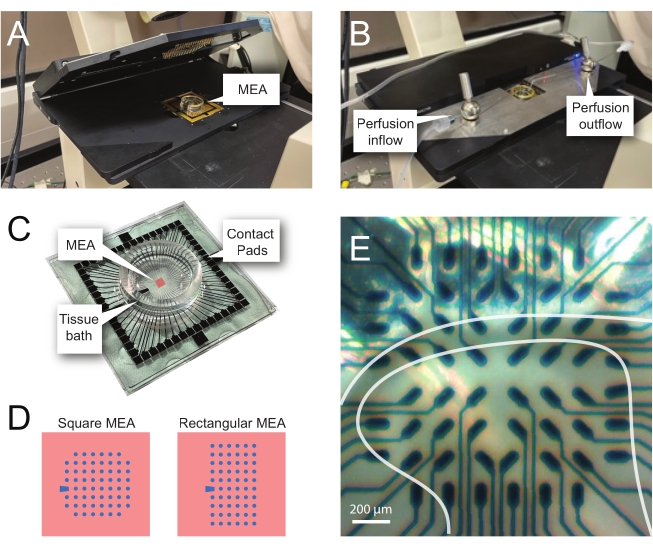
Figura 2: Posizionamento dei tessuti sull'array di microelettrodi. (A) L'immagine mostra un headstage MEA aperto con un MEA posto in posizione. (B) Come A con il palco di testa MEA chiuso per le registrazioni e il sistema di perfusione tissutale in atto. (C) L'immagine mostra un MEA fornito dal produttore. Vengono mostrati i cuscinetti di contatto, che si interfacciano con le molle dorate del palco di testa, e il bagno di tessuto MEA che contiene la soluzione di balneazione del tessuto e la fetta di tessuto. L'area evidenziata dal quadrato rosso al centro è la posizione dell'array di elettrodi. (D) Gli schemi mostrano le due configurazioni di elettrodi MEA utilizzate in questo studio, con ulteriori dettagli presentati nella Tabella 2. L'elettrodo di riferimento è indicato dal trapezio blu. Il layout dell'elettrodo MEA sinistro mostra una configurazione quadrata a 60 elettrodi, utilizzata maggiormente nei modelli di lavoro presentati 60MEA200/30iR-Ti con elettrodi di diametro 30 μm distanziati di 200 μm, o 200 μm distanziati e 100 μm distanziati MEA tridimensionali (60MEA200/12/50iR-Ti e 60MEA100/12/40iR-Ti) con elettrodi di 12 μm di diametro e 50 μm o 40 μm di altezza, rispettivamente. Il layout dell'elettrodo MEA sinistro mostra un layout rettangolare di elettrodi 6 x 10-60MEA500/30iR-Ti. (E) Immagine ad alto ingrandimento di un MEA quadrato 60MEA100/12/40iR-Ti con fetta di midollo spinale trasversale posizionata per la registrazione. La fetta si trova sulle file di elettrodi 3-8. La fila superiore di elettrodi, che non entrano in contatto con alcun tessuto, fungono da elettrodi di riferimento. L'area SDH viene visualizzata come una banda semitrasparente. In questo caso, l'SDH sovrasta gli elettrodi nelle righe 4, 5 e 6 e nelle colonne 2, 3, 4, 5 e 7 del MEA. Barra della scala = 200 μm. Abbreviazioni: MEA = matrice di microelettrodi; SDH = corno dorsale superficiale. Fare clic qui per visualizzare una versione più grande di questa figura.
- Modifica delle sezioni
- Dopo ogni sessione di registrazione, risciacquare le linee con aCSF.
- Rimuovere il MEA dal palco principale.
- Rimuovere bene la rete e il tessuto dal MEA, sciacquarli bene con aCSF e ripetere i passaggi precedenti con una nuova fetta.
2. Trattamento e analisi dei dati
NOTA: i seguenti passaggi descrivono in dettaglio come utilizzare il software di analisi per gli esperimenti MEA sulle fette del midollo spinale. Uno dei 60 elettrodi funge da riferimento interno (contrassegnato da un trapezio nella Figura 2 C, D), mentre tra quattro e venticinque dei restanti 59 sono posizionati sotto l'SDH in una fetta di midollo spinale di topo adulto. Analisi successive rilevano forme d'onda del potenziale d'azione extracellulare (EAP) e del potenziale di campo locale (LFP) (vedere la Figura 3B per esempi) dal segnale grezzo in questa regione.
- Elaborazione di dati grezzi
- Aprire il software di analisi e caricare il layout di analisi predefinito (Figura 3B).
- Aprire il file di interesse e deselezionare l'elettrodo di riferimento (elettrodo 15 in 8 x 8 MEA- o elettrodo E1 in configurazione 6 x 10 MEA) e gli eventuali elettrodi ritenuti eccessivamente rumorosi.
- Impostare la finestra temporale per l'analisi (0:00 → 5:00 min).
- Passare alla scheda Filtro tra canali . Selezionare Riferimento complesso e selezionare gli elettrodi di riferimento in base all'immagine scattata e alle note fatte durante l'esperimento (cioè, quegli elettrodi non sotto il tessuto). Per applicare e verificare questa operazione, premere Esplora prima di continuare.
- Passare alla scheda Filtro EAP e applicare un filtro Butterworth passa altodi 2° ordine (taglio a 200 Hz) per rimuovere l'attività LFP.
- Passare alla scheda filtro LFP e applicare un filtro Butterworth passa banda di 2° ordine (frequenze delta di 0,5-4 Hz) per rimuovere l'attività EAP.
- Passare alla scheda Rilevatore EAP e selezionare Soglia automatica. Spuntare le caselle di bordo In aumento e Caduta e impostare il tempo morto su 0,5 ms.
- Impostare soglie positive e negative in base ai dati. Esaminare i dati tornando alla schermata analizzatore di dati grezzi, spostando l'indicatore di tempo, quindi tornando alla scheda rilevatore EAP e premendo Esplora. Ripetere l'operazione fino a quando non si è certi che la soglia di rilevamento impostata stia catturando gli EAP senza catturare il rumore/l'attività non fisiologica. Utilizzare gli elettrodi di riferimento per identificare il rumore/attività non fisiologica.
NOTA: è necessario assicurarsi che un numero minimo di EAP sia rilevato negli elettrodi di riferimento in cui non si verificherà attività fisiologica. Tuttavia, deviazioni piuttosto lievi nel basale potrebbero essere erroneamente rilevate come EAP. Questo pur mirando a massimizzare il numero di eventi reali rilevati negli elettrodi attivi. - Passare alla scheda del rilevatore LFP , selezionare la soglia manuale, selezionare le caselle bordo in aumento e in caduta e impostare il tempo morto su 3 ms.
- Ripetere il passaggio 2.1.8 per un elettrodo selezionando un elettrodo con attività LFP. Una volta soddisfatto, selezionare Applica a tutti poiché le soglie verranno applicate solo a un singolo elettrodo quando si esegue la soglia manuale.
- Durante l'esame dei dati LFP nella scheda Rilevatore , prendere nota del numero massimo di passaggi di soglia per l'unica forma d'onda LFP e della separazione temporale massima degli incroci di soglia per l'unica forma d'onda LFP da utilizzare in analisi successive.
- Premere Avvia analisi.
- Al termine dell'analisi, passare alla scheda Analizzatore EAP ed esportare i dati. Fai lo stesso nella scheda Analizzatore LFP .
- Ripetere questa procedura per tutti gli altri file della stessa sezione.
- Dopo l'esportazione dei dati, convertire i file in formato xlsx in modo che possano essere letti dallo script di programmazione utilizzato. Assegna un nome ai file in base alla seguente convenzione affinché lo script fornito li legga: nome dell'esperimento (ad esempio, dati di esempio) - numero di sezione (ad esempio, S1) - numero di registrazione (ad esempio, R1) - tipo di attività (ad esempio, picchi o SP, corrispondenti rispettivamente a EAP o LFP).
NOTA: L'analisi EAP qui descritta tratta il picchio da singoli canali come una singola popolazione, anche se questa attività deriverebbe comunemente da più neuroni in prossimità dell'elettrodo di registrazione. Se si desidera il numero di neuroni che contribuiscono agli EAP in un canale, le tecniche di smistamento multispike descritte altrove possono essere applicate per distinguere popolazioni distinte di picchi in base alle caratteristiche della forma d'onda28.
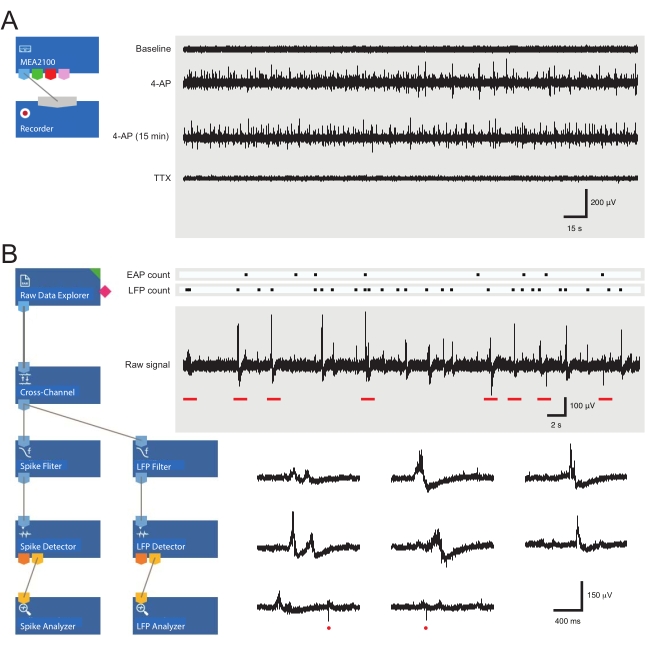
Figura 3: Layout degli strumenti di registrazione e analisi dei dati e registrazioni di array di microelettrodi di esempio che mostrano il potenziale d'azione extracellulare e le forme d'onda del potenziale di campo locale. (A) Schematico mostra modelli di registrazione preconfigurati utilizzati per l'acquisizione di dati MEA. Il collegamento dello strumento MEA2100 e di registrazione (headstage/amplificatore) consente di denominare e salvare i dati. Quattro tracce di esempio di dati grezzi (a destra, epoche di 5 minuti) sono state raccolte da un canale MEA che mostra l'attività al basale, 12 minuti dopo l'applicazione 4-AP, altri 15 minuti dopo l'attività 4-AP stabilita e dopo l'applicazione del bagno di TTX (1 μM). Nota, l'aggiunta di 4-AP (seconda traccia) produce un chiaro aumento del rumore di fondo e dell'attività EAP/LFP. È importante sottolineare che l'attività rimane relativamente stabile per almeno 15 minuti dopo che l'attività indotta da 4-AP è stata stabilita (terza traccia). L'aggiunta di TTX (1 μM) abolisce ogni attività (traccia inferiore). (B) Schematico (a sinistra) mostra la configurazione del software dell'analizzatore per l'analisi dei dati. Lo strumento di esplorazione dei dati grezzi viene utilizzato per importare le registrazioni raccolte dal software di registrazione. Questi dati vengono quindi eseguiti attraverso uno strumento di filtro a più canali che sottrae i segnali degli elettrodi di riferimento selezionati da altri elettrodi per rimuovere il rumore di fondo. I dati passano attraverso il filtro EAP e gli strumenti di filtro LFP per ottimizzare le relazioni segnale-rumore per ogni forma d'onda. Seguendo questo passaggio, i dati del percorso EAP entrano nello strumento di rilevamento EAP, dove vengono impostate le soglie. Gli EAP vengono rilevati e quindi inviati allo strumento di analisi EAP in cui le latenze di ciascun evento vengono registrate ed esportate come txt. file. Un flusso di lavoro identico si verifica per i dati LFP utilizzando un toolkit LFP corrispondente. Le tracce a destra mostrano i dati di un singolo canale MEA contenente varie forme d'onda extracellulari. La posizione dei segnali EAP e LFP è evidenziata nel "conteggio dei raster" di cui sopra. Le tracce inferiori sono epoche della registrazione superiore (indicate da barre rosse) che mostrano forme d'onda su una scala temporale espansa, inclusi vari segnali LFP (si noti la varietà di apparenze) e singoli EAP extracellulari (cerchi rossi). Nota, la forma d'onda E LA POLARITÀ LFP / EAP variano in relazione al numero di neuroni che producono questi segnali, alla loro vicinanza all'elettrodo di registrazione e alla loro posizione in relazione agli elettrodi vicini. Abbreviazioni: MEA = matrice di microelettrodi; EAP = potenziale d'azione extracellulare; LFP = potenziale di campo locale; 4-AP = 4-aminopiridina; TTX = tetrodotossina. Fare clic qui per visualizzare una versione più grande di questa figura.
- Analisi di sincronicità
NOTA: La sincronicità, o il numero di eventi "coincidenti" tra due elettrodi, è stata determinata utilizzando il criterio di coincidenza all'interno del metodo di sincronizzazione A-SPIKE delineato da Satuvuori et al. 29. Lo script utilizzato qui confronta solo gli elettrodi adiacenti l'uno all'altro per l'efficienza (cioè vicini orizzontali, verticali e diagonali); tuttavia, lo script potrebbe essere riscritto per confrontare tutti gli elettrodi, se necessario.- Eseguire l'analisi dei dati utilizzando uno script di programmazione personalizzato, che estrae i timestamp di latenza per ciascun elettrodo dai file .xlsx.
NOTA: questa operazione può essere eseguita manualmente. - Nel passaggio 2.1.11, registrare il numero massimo di passaggi di soglia e la separazione temporale massima degli attraversamenti di soglia per l'unica forma d'onda LFP. Modificare lo script per l'immissione di questi parametri di definizione LFP per ogni sezione prima di eseguire lo script.
NOTA: la soglia eseguita in precedenza nel software di analisi acquisisce chiaramente gli EAP come un singolo evento. Tuttavia, gli LFP sono composti da un numero variabile di picchi a seconda della forma della forma d'onda e del successivo numero di attraversamenti di soglia da parte di un evento. - Modificare lo script per inserire gli elettrodi di interesse prima dell'analisi.
- Per determinare la sincronicità (definita nello script da intervalli di tempo modificabili per l'attività sincrona all'interno), separare e analizzare le latenze estratte per rilevare eventi coincidenti.
NOTA: lo script consente di impostare il tempo massimo tra eventi coincidenti. Questi sono impostati a 20 ms per gli EAP e 200 ms per gli LFP. - Eseguire lo script per estrarre i timestamp di latenza.
NOTA: il file di output .xlsx contiene le interpretazioni dei dati di latenza, ovvero conteggi EAP e LFP, frequenze e conteggi di eventi coincidenti per singoli elettrodi e intere sezioni. Questi dati vengono utilizzati per valutare la frequenza, i conteggi EAP/LFP, il numero di elettrodi attivi, il numero di eventi coincidenti, il numero di elettrodi collegati e la forza media di questi collegamenti.
- Eseguire l'analisi dei dati utilizzando uno script di programmazione personalizzato, che estrae i timestamp di latenza per ciascun elettrodo dai file .xlsx.
Risultati
Modello di attività di rete nel corno dorsale del midollo spinale
L'applicazione di 4-AP induce in modo affidabile l'attività ritmica sincrona nel DH del midollo spinale. Tale attività si presenta come un aumento degli EAP e dei LFP. Il segnale successivo è una forma d'onda a bassa frequenza, che è stata precedentemente descritta nelle registrazioni MEA30. I cambiamenti nell'attività EAP e/ o LFP dopo l'applicazione del farmaco riflettono un'attività neurale alterata. Es...
Discussione
Nonostante l'importanza del DH spinale nella segnalazione nocicettiva, nell'elaborazione e nelle conseguenti risposte comportamentali ed emotive che caratterizzano il dolore, i circuiti all'interno di questa regione rimangono poco compresi. Una sfida chiave nello studio di questo problema è stata la diversità delle popolazioni di neuroni che comprendono questi circuiti 6,31,32. I recenti progressi nelle tecnologie transgeniche...
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Questo lavoro è stato finanziato dal National Health and Medical Research Council (NHMRC) dell'Australia (sovvenzioni 631000, 1043933, 1144638 e 1184974 a B.A.G. e R.J.C.) e dall'Hunter Medical Research Institute (sovvenzione a B.A.G. e R.J.C.).
Materiali
| Name | Company | Catalog Number | Comments |
| 4-aminopyridine | Sigma-Aldrich | 275875-5G | |
| 100% ethanol | Thermo Fisher | AJA214-2.5LPL | |
| CaCl2 1M | Banksia Scientific | 0430/1L | |
| Carbonox (Carbogen - 95% O2, 5% CO2) | Coregas | 219122 | |
| Curved long handle spring scissors | Fine Science Tools | 15015-11 | |
| Custom made air interface incubation chamber | |||
| Foetal bovine serum | Thermo Fisher | 10091130 | |
| Forceps Dumont #5 | Fine Science Tools | 11251-30 | |
| Glucose | Thermo Fisher | AJA783-500G | |
| Horse serum | Thermo Fisher | 16050130 | |
| Inverted microscope | Zeiss | Axiovert10 | |
| KCl | Thermo Fisher | AJA383-500G | |
| Ketamine | Ceva | KETALAB04 | |
| Large surgical scissors | Fine Science Tools | 14007-14 | |
| Loctite 454 Instant Adhesive | Bolts and Industrial Supplies | L4543G | |
| MATLAB | MathWorks | R2018b | |
| MEAs, 3-Dimensional | Multichannel Systems | 60-3DMEA100/12/40iR-Ti, 60-3DMEA200/12/50iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in an 8x8 square grid. Electrodes are 12 µm in diameter, 40 µm (100/12/40) or 50 µm (200/12/50) high and equidistantly spaced 100 µm (100/12/40) or 200 µm (200/12/50) apart. |
| MEA headstage | Multichannel Systems | MEA2100-HS60 | |
| MEA interface board | Multichannel Systems | MCS-IFB 3.0 Multiboot | |
| MEA net | Multichannel Systems | ALA HSG-MEA-5BD | |
| MEA perfusion system | Multichannel Systems | PPS2 | |
| MEAs, Planar | Multichannel Systems | 60MEA200/30iR-Ti, 60MEA500/30iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in either a 8x8 square grid (200/30) or a 6x10 rectangular grid (500/30). Electrodes are 30 µm in diameter and equidistantly spaced 200 µm (200/30) or 500 µm (500/30) apart. |
| MgCl2 | Thermo Fisher | AJA296-500G | |
| Microscope camera | Motic | Moticam X Wi-Fi | |
| Multi Channel Analyser software | Multichannel Systems | V 2.17.4 | |
| Multi Channel Experimenter software | Multichannel Systems | V 2.17.4 | |
| NaCl | Thermo Fisher | AJA465-500G | |
| NaHCO3 | Thermo Fisher | AJA475-500G | |
| NaH2PO4 | Thermo Fisher | ACR207805000 | |
| Rongeurs | Fine Science Tools | 16021-14 | |
| Small spring scissors | Fine Science Tools | 91500-09 | |
| Small surgical scissors | Fine Science Tools | 14060-09 | |
| Sucrose | Thermo Fisher | AJA530-500G | |
| Superglue | cyanoacrylate adhesive | ||
| Tetrodotoxin | Abcam | AB120055 | |
| Vibration isolation table | Newport | VH3048W-OPT | |
| Vibrating microtome | Leica | VT1200 S |
Riferimenti
- Smith, K. M., et al. Calretinin positive neurons form an excitatory amplifier network in the spinal cord dorsal horn. eLife. 8, 49190 (2019).
- Smith, K. M., et al. Functional heterogeneity of calretinin-expressing neurons in the mouse superficial dorsal horn: implications for spinal pain processing. The Journal of physiology. 593 (19), 4319-4339 (2015).
- Boyle, K. A., et al. Defining a spinal microcircuit that gates myelinated afferent input: Implications for tactile allodynia. Cell Reports. 28 (2), 526-540 (2019).
- Browne, T. J., et al. Transgenic cross-referencing of inhibitory and excitatory interneuron populations to dissect neuronal heterogeneity in the dorsal horn. Frontiers in Molecular Neuroscience. 13, 32 (2020).
- Graham, B. A., Hughes, D. I. Rewards, perils and pitfalls of untangling spinal pain circuits. Current Opinion in Physiology. 11, 35-41 (2019).
- Todd, A. J. Neuronal circuitry for pain processing in the dorsal horn. Nature Reviews Neuroscience. 11 (12), 823-836 (2010).
- Hughes, D. I., Todd, A. J. Central nervous system targets: inhibitory interneurons in the spinal cord. Neurotherapeutics. 17 (3), 874-885 (2020).
- Duan, B., et al. Identification of spinal circuits transmitting and gating mechanical pain. Cell. 159 (6), 1417-1432 (2014).
- Hachisuka, J., Chiang, M. C., Ross, S. E. Itch and neuropathis itch. Pain. 159 (3), 603 (2018).
- Foster, E., et al. Targeted ablation, silencing, and activation establish glycinergic dorsal horn neurons as key components of a spinal gate for pain and itch. Neuron. 85 (6), 1289-1304 (2015).
- Bourane, S., et al. Identification of a spinal circuit for light touch and fine motor control. Cell. 160 (3), 503-515 (2015).
- Cheng, L., et al. Identification of spinal circuits involved in touch-evoked dynamic mechanical pain. Nature neuroscience. 20 (6), 804-814 (2017).
- Peirs, C., et al. Mechanical allodynia circuitry in the dorsal horn is defined by the nature of the injury. Neuron. 109 (1), 73-90 (2021).
- Huang, J., et al. Circuit dissection of the role of somatostatin in itch and pain. Nature Neuroscience. 21 (5), 707-716 (2018).
- Obien, M. E. J., Deligkaris, K., Bullmann, T., Bakkum, D. J., Frey, U. Revealing neuronal function through microelectrode array recordings. Frontiers in Neuroscience. 8, 423 (2015).
- Nam, Y., Wheeler, B. C. In vitro microelectrode array technology and neural recordings. Critical Reviews in Biomedical Engineering. 39 (1), 45-61 (2011).
- Johnstone, A. F., et al. Microelectrode arrays: a physiologically based neurotoxicity testing platform for the 21st century. Neurotoxicology. 31 (4), 331-350 (2010).
- Stett, A., et al. Biological application of microelectrode arrays in drug discovery and basic research. Analytical and Bioanalytical Chemistry. 377 (3), 486-495 (2003).
- Xu, L., et al. Trends and recent development of the microelectrode arrays (MEAs). Biosensors and Bioelectronics. 175 (1), 112854 (2020).
- Chapman, R. J., Cilia La Corte, P. F., Asghar, A. U. R., King, A. E. Network-based activity induced by 4-aminopyridine in rat dorsal horn in vitro is mediated by both chemical and electrical synapses. The Journal of Physiology. 587, 2499-2510 (2009).
- Ruscheweyh, R., Sandkühler, J. Epileptiform activity in rat spinal dorsal horn in vitro has common features with neuropathic pain. Pain. 105 (1-2), 327-338 (2003).
- Kay, C. W., Ursu, D., Sher, E., King, A. E. The role of Cx36 and Cx43 in 4-aminopyridine-induced rhythmic activity in the spinal nociceptive dorsal horn: an electrophysiological study in vitro. Physiological Reports. 4 (14), 12852 (2016).
- Jankowska, E., Lundberg, A., Rudomin, P., Sykova, E. Effects of 4-aminopyridine on synaptic transmission in the cat spinal cord. Brain Research. 240 (1), 117-129 (1982).
- Semba, K., Geller, H. M., Egger, M. D. 4-Aminopyridine induces expansion of cutaneous receptive fields of dorsal horn cells. Brain Research. 343 (2), 398-402 (1985).
- Ruscheweyh, R., Sandkühler, J. Long-range oscillatory Ca2+ waves in rat spinal dorsal horn. European Journal of Neuroscience. 22 (8), 1967-1976 (2005).
- Egert, U., et al. A novel organotypic long-term culture of the rat hippocampus on substrate-integrated multielectrode arrays. Brain Research Protocols. 2 (4), 229-242 (1998).
- Thiebaud, P., De Rooij, N., Koudelka-Hep, M., Stoppini, L. Microelectrode arrays for electrophysiological monitoring of hippocampal organotypic slice cultures. IEEE Transactions on Biomedical Engineering. 44 (11), 1159-1163 (1997).
- Rey, H. G., Pedreira, C., Quiroga, R. Q. Past, present and future of spike sorting techniques. Brain Research Bulletin. 119, 106-117 (2015).
- Satuvuori, E., et al. Measures of spike train synchrony for data with multiple time scales. Journal of Neuroscience Methods. 287, 25-38 (2017).
- Mendis, G. D. C., Morrisroe, E., Reid, C. A., Halgamuge, S. K., Petrou, S. Use of local field potentials of dissociated cultures grown on multi-electrode arrays for pharmacological assays. 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. , 952-956 (2016).
- Hughes, D. I., et al. Morphological, neurochemical and electrophysiological features of parvalbumin-expressing cells: a likely source of axo-axonic inputs in the mouse spinal dorsal horn. The Journal of Physiology. 590 (16), 3927-3951 (2012).
- Peirs, C., Seal, R. P. Neural circuits for pain: recent advances and current views. Science. 354 (6312), 578-584 (2016).
- Li, J., Baccei, M. L. Developmental regulation of membrane excitability in rat spinal lamina I projection neurons. Journal of Neurophysiology. 107 (10), 2604-2614 (2012).
- Li, J., Baccei, M. L. Pacemaker neurons within newborn spinal pain circuits. Journal of Neuroscience. 31 (24), 9010-9022 (2011).
- Sandkühler, J., Eblen-Zajjur, A. Identification and characterization of rhythmic nociceptive and non-nociceptive spinal dorsal horn neurons in the rat. Neuroscience. 61 (4), 991-1006 (1994).
- Lucas-Romero, J., Rivera-Arconada, I., Roza, C., Lopez-Garcia, J. A. Origin and classification of spontaneous discharges in mouse superficial dorsal horn neurons. Scientific Reports. 8 (1), 9735-9735 (2018).
- Antonio, L., et al. L. al. In vitro seizure like events and changes in ionic concentration. Journal of Neuroscience Methods. 260, 33-44 (2016).
- Avoli, M., Jefferys, J. G. Models of drug-induced epileptiform synchronization in vitro. Journal of Neuroscience Methods. 260, 26-32 (2016).
- Taccola, G., Nistri, A. Low micromolar concentrations of 4-aminopyridine facilitate fictive locomotion expressed by the rat spinal cord in vitro. Neuroscience. 126 (2), 511-520 (2004).
- Mitra, P., Brownstone, R. M. An in vitro spinal cord slice preparation for recording from lumbar motoneurons of the adult mouse. Journal of Neurophysiology. 107 (2), 728-741 (2012).
- Egert, U., Heck, D., Aertsen, A. Two-dimensional monitoring of spiking networks in acute brain slices. Experimental Brain Research. 142 (2), 268-274 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon