Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Enregistrement de l’activité du réseau dans les circuits nociceptifs rachidiens à l’aide de réseaux de microélectrodes
Dans cet article
Résumé
L’utilisation combinée de la technologie des réseaux de microélectrodes et de la stimulation chimique induite par la 4-aminopyridine pour étudier l’activité nociceptive au niveau du réseau dans la corne dorsale de la moelle épinière est décrite.
Résumé
Les rôles et la connectivité de types spécifiques de neurones dans la corne dorsale de la moelle épinière (DH) sont délimités à un rythme rapide pour fournir une vue de plus en plus détaillée des circuits qui sous-tendent le traitement de la douleur vertébrale. Cependant, les effets de ces connexions sur l’activité plus large du réseau dans la DH restent moins bien compris car la plupart des études se concentrent sur l’activité de neurones uniques et de petits microcircuits. Alternativement, l’utilisation de réseaux de microélectrodes (MEA), qui peuvent surveiller l’activité électrique à travers de nombreuses cellules, fournit une résolution spatiale et temporelle élevée de l’activité neuronale. Ici, l’utilisation d’AME avec des tranches de moelle épinière de souris pour étudier l’activité de la DH induite par la stimulation chimique des circuits de DH avec la 4-aminopyridine (4-AP) est décrite. L’activité rythmique résultante est limitée à la DH superficielle, stable dans le temps, bloquée par la tétrodotoxine, et peut être étudiée dans différentes orientations de tranches. Ensemble, cette préparation fournit une plate-forme pour étudier l’activité du circuit DH dans les tissus d’animaux naïfs, de modèles animaux de douleur chronique et de souris ayant une fonction nociceptive génétiquement modifiée. En outre, les enregistrements MEA dans les tranches de moelle épinière stimulées par 4-AP peuvent être utilisés comme outil de dépistage rapide pour évaluer la capacité de nouveaux composés antinociceptifs à perturber l’activité dans la moelle épinière DH.
Introduction
Les rôles de types spécifiques d’interneurones inhibiteurs et excitateurs dans la moelle épinière DH sont découverts à un rythme rapide 1,2,3,4. Ensemble, les interneurones représentent plus de 95% des neurones de la DH et sont impliqués dans le traitement sensoriel, y compris la nociception. De plus, ces circuits interneurones sont importants pour déterminer si les signaux périphériques montent dans l’axe du neuroaxe pour atteindre le cerveau et contribuer à la perception de la douleur 5,6,7. À ce jour, la plupart des études ont étudié le rôle des neurones DH au niveau de l’analyse unicellulaire ou de l’organisme entier en utilisant des combinaisons d’électrophysiologie intracellulaire in vitro, de marquage neuroanatomique et d’analyse comportementale in vivo 1,3,8,9,10,11,12,13,14 . Ces approches ont considérablement fait progresser la compréhension du rôle de populations de neurones spécifiques dans le traitement de la douleur. Cependant, une lacune subsiste dans la compréhension de la façon dont des types cellulaires spécifiques et de petits macro-circuits influencent de grandes populations de neurones au niveau des microcircuits pour façonner ensuite la sortie de la DH, les réponses comportementales et l’expérience de la douleur.
Une technologie qui peut étudier la fonction de macro-circuit ou multicellulaire est le réseau de microélectrodes (MEA)15,16. Les MEA sont utilisés pour étudier le fonctionnement du système nerveux depuis plusieurs décennies17,18. Dans le cerveau, ils ont facilité l’étude du développement neuronal, de la plasticité synaptique, du dépistage pharmacologique et des tests de toxicité17,18. Ils peuvent être utilisés pour des applications in vitro et in vivo, selon le type de MEA. En outre, le développement des MEA a évolué rapidement, avec différents nombres et configurations d’électrodes désormais disponibles19. L’un des principaux avantages des MEA est leur capacité à évaluer simultanément l’activité électrique dans de nombreux neurones avec une grande précision spatiale et temporelle via plusieurs électrodes15,16. Cela fournit une lecture plus large de la façon dont les neurones interagissent dans les circuits et les réseaux, dans des conditions de contrôle et en présence de composés appliqués localement.
L’un des défis des préparations in vitro de DH est que les niveaux d’activité continue sont généralement faibles. Ici, ce défi est abordé dans les circuits DH de la moelle épinière en utilisant le bloqueur de canal K + voltage-dépendant, la 4-aminopryidine (4-AP), pour stimuler chimiquement les circuits DH. Ce médicament a déjà été utilisé pour établir une activité électrique synchrone rythmique dans la DH des tranches aiguës de la moelle épinière et dans des conditions aiguës in vivo 20,21,22,23,24. Ces expériences ont utilisé un patch unicellulaire et un enregistrement extracellulaire ou l’imagerie calcique pour caractériser l’activitéinduite par le 4-AP 20,21,22,23,24,25. Ensemble, ces travaux ont démontré l’exigence d’une transmission synaptique excitatrice et inhibitrice et de synapses électriques pour une activité rythmique induite par le 4-AP. Ainsi, la réponse 4-AP a été considérée comme une approche qui démasque les circuits DH polysynaptiques natifs ayant une pertinence biologique plutôt que comme un épiphénomène induit par un médicament. En outre, l’activité induite par le 4-AP présente un profil de réponse similaire aux analgésiques et aux antiépileptiques que les affections douloureuses neuropathiques et a été utilisée pour proposer de nouvelles cibles médicamenteuses analgésiques à base rachidienne telles que les connexines 20,21,22.
Ici, une préparation qui combine les MEA et l’activation chimique de la DH spinale avec le 4-AP pour étudier ce circuit nociceptif au niveau du macro-circuit, ou réseau d’analyse, est décrite. Cette approche fournit une plate-forme stable et reproductible pour étudier les circuits nociceptifs dans des conditions naïves et neuropathiques « semblables à la douleur ». Cette préparation est également facilement applicable pour tester l’action au niveau du circuit des analgésiques connus et pour dépister de nouveaux analgésiques dans la moelle épinière hyperactive.
Protocole
Des études ont été réalisées sur des souris mâles et femelles c57Bl/6 âgées de 3 à 12 mois. Toutes les procédures expérimentales ont été effectuées conformément au comité de soins et d’éthique des animaux de l’Université de Newcastle (protocoles A-2013-312 et A-2020-002).
1. Électrophysiologie in vitro
- Préparation de solutions pour la préparation et l’enregistrement de tranches de moelle épinière
- Liquide céphalo-rachidien artificiel
REMARQUE: Le liquide céphalo-rachidien artificiel (CSPa) est utilisé dans une chambre d’incubation d’interface, où les tranches sont stockées jusqu’au début de l’enregistrement et pendant les expériences sous forme de perfusat et de diluant pour les médicaments. Voir le tableau 1 pour la composition détaillée.
- Liquide céphalo-rachidien artificiel
| Chimique | aCSF (mM) | aCSF (g/100 mL) | CSF substitué par le saccharose (mM) | CSAf substitué par le saccharose (g/100 mL) | ACSF à haute teneur en potassium (mM) | ACSF à haute teneur en potassium (g/100 mL) |
| Chlorure de sodium (NaCl) | 118 | 0.690 | - | - | 118 | 0.690 |
| Hydrogénocarbonate de sodium (NaHCO3) | 25 | 0.210 | 25 | 0.210 | 25 | 0.210 |
| Glucose | 10 | 0.180 | 10 | 0.180 | 10 | 0.180 |
| Chlorure de potasium (KCl) | 2.5 | 0.019 | 2.5 | 0.019 | 4.5 | 0.034 |
| Dihydrogénophosphate de sodium (NaH2PO4) | 1 | 0.012 | 1 | 0.012 | 1 | 0.012 |
| Cloride de magnésium (MgCl2) | 1 | 0.01 | 1 | 0.01 | 1 | 0.01 |
| Chlorure de calcium (CaCl2) | 2.5 | 0.028 | 2.5 | 0.028 | 2.5 | 0.028 |
| Saccharose | - | - | 250 | 8.558 | - | - |
Tableau 1 : Compositions du liquide céphalo-rachidien artificiel. Abréviation : aCSF = liquide céphalo-rachidien artificiel.
- Préparer un CSF contenant (en mM) 118 NaCl, 25 NaHCO3, 10 glucose, 2,5 KCl, 1 NaH2PO4, 1 MgCl2 et 2,5 CaCl2 en ajoutant les quantités requises de ce qui précède, à l’exclusion du CaCl2, à 2 L d’eau distillée.
- Faire bouillonner la solution ci-dessus avec du carbogène (95% O2, 5% CO2) pendant 5 min et ajouter CaCl2.
REMARQUE: Cette étape empêche les précipitations de CaCl2 , c’est-à-dire que la solution ne doit pas devenir trouble. Pour l’application de médicaments pendant les expériences, diluer les solutions mères de médicaments dans l’aCSF aux concentrations finales souhaitées.
- Liquide céphalo-rachidien artificiel substitué au saccharose
REMARQUE: Le LSCa substitué par le saccharose est utilisé pendant la dissection et le tranchage de la moelle épinière. Comme son nom l’indique, le saccharose est substitué au NaCl pour réduire l’excitation neuronale au cours de ces procédures tout en maintenant l’osmolarité. Voir le tableau 1 pour la composition détaillée.- Préparer le CSAf substitué au saccharose contenant (en mM) 250 saccharose, 25 NaHCO3, 10 glucose, 2,5 KCl, 1 NaH2PO4, 1 MgCl2 et 2,5 CaCl2 en ajoutant les quantités requises de tout ce qui précède, à l’exclusion du CaCl2, à 300 mL d’eau distillée.
- Faire bouillonner la solution avec du carbogène pendant 5 min, puis ajouter caCl2.
- Conserver la solution dans un congélateur à -80 °C pendant environ 40 min ou jusqu’à ce que la solution forme une boue. Évitez de congeler solide et utilisez-le en consistance de boue.
- Préparation du réseau de microélectrodes
REMARQUE: La surface de contact de l’AEM nécessite un prétraitement pour la rendre hydrophile.- Avant l’expérience, remplissez bien le MEA avec du sérum fœtal bovin (FBS) ou du sérum de cheval (HS) pendant 30 min.
- Retirez le FBS ou le SH et rincez soigneusement le MEA avec environ cinq lavages d’eau distillée jusqu’à ce que l’eau distillée ne soit plus mousseuse. Remplissez le puits avec aCSF, prêt à l’emploi.
- Préparation aiguë de tranches de moelle épinière
REMARQUE: La préparation de tranches de moelle épinière de souris est comme décrit précédemment par Smith et al.2. Idéalement, l’élimination de l’élargissement lombo-sacré ne devrait pas prendre plus de 8 à 10 minutes (étapes 1.3.2 à 1.3.11 ci-dessous).- Anesthésiez profondément la souris avec 100 mg / kg de kétamine (i.p.), puis décapitez-la à l’aide de gros ciseaux chirurgicaux.
- Enlevez la peau sur la région abdominale en faisant une petite coupure dans la peau au niveau des hanches. Tirez la peau de chaque côté de la coupe de manière rostrale jusqu’à ce que toute la peau soit enlevée, c’est-à-dire du haut de la cage thoracique au sommet du bassin (ventrale et dorsale).
- Placez le corps sur la glace et utilisez une approche ventrale pour exposer la colonne vertébrale en enlevant tous les viscères et en coupant à travers les côtes latérales au sternum.
- Retirez la cage thoracique ventrale, les deux omoplates (coupées à environ T2) et les membres inférieurs et le bassin (coupés approximativement au sommet du sacrum).
- Transférer la colonne vertébrale et la préparation des côtes dans un bain de dissection contenant du saccharose glacé aCSF. Épinglez les quatre coins de la préparation (surface ventrale vers le haut) en plaçant des épingles à travers les muscles du bas du dos et les côtes supérieures attachées.
- Enlevez tous les muscles et le tissu conjonctif recouvrant la surface ventrale des vertèbres de rongeurs et identifiez la région vertébrale au-dessus de l’élargissement lombo-sacré, qui se trouve approximativement sous les corps vertébraux T12 à L2.
- Enlevez un corps vertébral qui est caudal à la région d’élargissement lombo-sacré pour donner accès à la moelle épinière car elle se trouve dans le canal vertébral.
- À l’aide de ciseaux à ressort incurvés, coupez les pédicules vertébraux bilatéralement tout en soulevant et en tirant le corps vertébral de manière rostrale pour séparer les aspects ventral et dorsal des vertèbres et exposer la moelle épinière.
- Une fois que les corps vertébraux sont retirés pour révéler l’élargissement lombo-sacré, dégagez soigneusement les racines restantes qui ancrent la moelle épinière avec des ciseaux à ressort jusqu’à ce que la moelle flotte librement.
- Isolez la moelle épinière avec des coupes rostrales et caudales bien au-dessus et au-dessous de l’élargissement lombo-sacré, permettant à la région cible de la moelle osseuse de « flotter librement ».
REMARQUE : L’orientation de tranche préférée déterminera la façon dont le cordon est ensuite monté pour le sectionnement (Figure 1). - Pour les tranches transversales, soulevez le segment lombo-sacré par une racine attachée et placez-le sur un bloc de polystyrène prédécoupé (polystyrène) (1 cm x 1 cm x 1 cm) avec un canal peu profond coupé au centre. Utilisez un adhésif cyanoacrylate (voir la Table des matériaux) pour fixer le bloc et le cordon à la plate-forme de sectionnement et placez-les dans le bain de coupe contenant du saccharose glacé aCSF (boue).
REMARQUE: Le canal peu profond aide à sécuriser et à orienter la moelle épinière, avec la face dorsale exposée et l’extrémité thoracique de la moelle au bas du bloc. - Pour les tranches sagittales, posez une fine ligne d’adhésif cyanoacrylate sur la plate-forme de sectionnement, soulevez l’élargissement lombo-sacré par une racine attachée et placez le cordon le long de la ligne de colle, en veillant à ce qu’une surface latérale soit dans l’adhésif et que l’autre soit orientée vers le haut. Placez-le dans le bain de coupe contenant du saccharose glacé aCSF (boue).
- Pour les tranches horizontales, placez une fine ligne d’adhésif cyanoacrylate sur la plate-forme de sectionnement. Soulevez l’élargissement lombo-sacré par une racine attachée et placez l’élargissement lombo-sacré le long de la ligne d’adhésif, en vous assurant que la surface ventrale est dans l’adhésif et que la surface dorsale est orientée vers le haut. Utilisez des racines attachées pour positionner le cordon. Placez-le dans le bain de coupe contenant du saccharose glacé aCSF (boue).
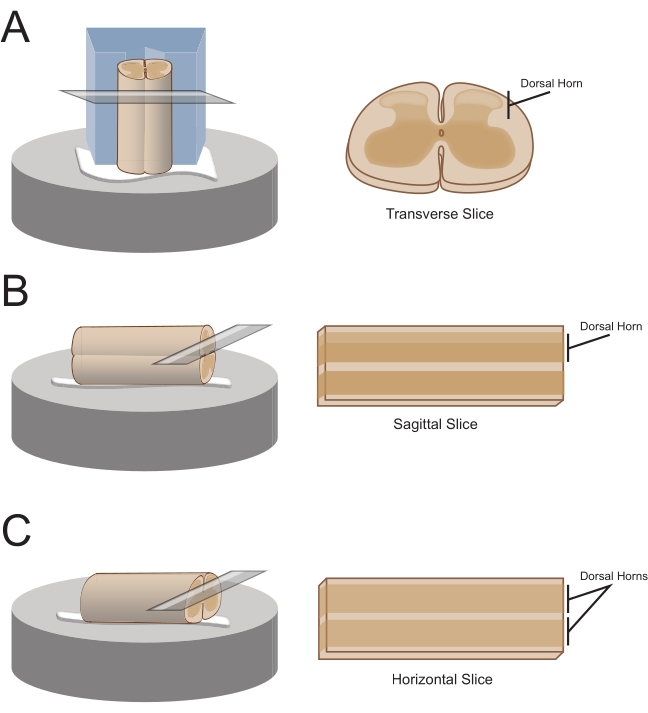
Figure 1 : Orientations des tranches de moelle épinière, méthodes de montage et de coupe. (A) Les tranches transversales nécessitent un bloc de coupe en polystyrène avec une rainure de support coupée dedans. La moelle épinière est posée contre le bloc dans la rainure de support, la face dorsale de la moelle faisant face au bloc. Le bloc et le cordon sont collés sur une étape de coupe avec de l’adhésif cyanoacrylate. (B) Les tranches sagittales sont préparées en plaçant une fine ligne d’adhésif cyanoacrylate sur l’étape de coupe, puis en positionnant la moelle épinière sur son côté sur la colle. (C) Les tranches horizontales sont préparées en plaçant une fine ligne d’adhésif cyanoacrylate sur la scène de coupe, puis en positionnant la face ventrale de la moelle épinière vers le bas sur la colle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Obtenez des tranches de 300 μm d’épaisseur (L1-L5, même épaisseur quelle que soit l’orientation) à l’aide d’un microtome vibrant avec les réglages suivants : vitesse 0,06 mm/s, amplitude 2,50 mm et calibré à ±0,02 degré de déviation d’amplitude de hauteur.
- Transférer les tranches dans une chambre d’incubation d’interface d’air contenant de l’aCSF oxygéné.
- Avant d’enregistrer, laissez les tranches s’équilibrer pendant 1 h à température ambiante (20-24 °C).
- Enregistrements en réseau de microélectrodes
REMARQUE: Les étapes suivantes détaillent comment utiliser les données d’enregistrement d’expériences MEA sur des tranches de moelle épinière. Plusieurs conceptions MEA peuvent être utilisées en fonction de l’expérience. Les détails de conception des MEA utilisés dans ces expériences sont présentés dans Tableau 2 et Graphique 2. Des informations détaillées sur la conception ont été publiées par Egert et al.26 et Thiebaud et coll.27 pour les MEA planaires et 3 dimensions (3D), respectivement. Les deux types MEA sont composés de 60 électrodes en nitrure de titane, avec une couche isolante en nitrure de silicium et des pistes et des coussinets de contact en nitrure de titane.- Configuration expérimentale
- Allumez l’ordinateur et la carte d’interface, puis démarrez le logiciel d’enregistrement.
- Chargez le modèle d’enregistrement pré-assemblé (Figure 3A). Nommez les fichiers de la journée dans l’onglet enregistreur.
- Bulle continue d’aCSF avec du carbogène (5% CO2, 95% O2) pendant toute la durée de l’expérience.
- Allumez le système de perfusion, qui est contrôlé par une pompe péristaltique. Placez la ligne d’entrée dans aCSF et l’extrémité d’entrée dans un bécher à déchets. Amorcer les lignes de perfusion avec aCSF.
- Préparer le 4-AP et toute autre solution médicamenteuse en diluant les bouillons dans 50 mL d’aCSF à la concentration finale requise (p. ex., 200 μM pour le 4-AP).
- Placez les solutions médicamenteuses dans des pots de médicaments et faites-les bouillonner de carbogène.
- Activité 4-AP
- Après l’incubation, transférer une seule tranche de l’incubateur à l’aide d’une pipette Pasteur à grande pointe remplie d’aCSF.
- Placez la tranche dans le puits MEA et ajoutez un aCSF supplémentaire.
- Placez la tranche sur le réseau d’enregistrement à 60 électrodes à l’aide d’un pinceau fin à cheveux courts. Évitez de contacter les électrodes avec le pinceau ou de faire glisser le tissu à travers les électrodes, surtout si vous utilisez des réseaux 3D.
REMARQUE: Selon la disposition MEA, cela peut être fait avec ou sans l’aide d’un microscope pour un positionnement précis. - Après avoir positionné la tranche, placez un filet lesté sur le tissu pour le maintenir en place et favoriser un bon contact avec les électrodes MEA.
REMARQUE : La tranche peut nécessiter un repositionnement après le placement net. - Placez le MEA dans la salle de tête d’enregistrement (Figure 2A,B).
- Vérifiez la position du tissu sur les électrodes à l’aide d’un microscope inversé (grossissement 2x) pour confirmer que le plus grand nombre possible d’électrodes sont sous le DH superficiel (SDH). Assurez-vous qu’au moins 2 à 6 électrodes n’entrent pas en contact avec la tranche, car ces électrodes sont importantes pour soustraire le bruit et enregistrer les artefacts pendant l’analyse (Figure 2E).
- Allumez l’appareil photo, connectez-le à l’appareil et prenez une image de référence de la tranche par rapport à l’AEM pour l’utiliser pendant l’analyse.
- Appuyez sur Démarrer le DAQ dans le logiciel d’enregistrement et vérifiez que toutes les électrodes reçoivent un signal clair.
REMARQUE: Si le signal est bruyant, déclipsez la scène de tête et nettoyez les coussinets de contact MEA et les contacts à ressort en or avec de l’éthanol à 70% (utilisez une lingette de laboratoire pour vous assurer que les tampons et les contacts sont secs après le nettoyage). Si le signal est toujours bruyant, éteignez les électrodes défectueuses dans le logiciel d’enregistrement ou notez l’exclusion plus tard pendant l’analyse. - Fixez les conduites d’entrée et de sortie de perfusion au puits MEA (précédemment rempli d’aCSF) et allumez le système de perfusion. Vérifiez le débit, idéalement 4 à 6 volumes de bain par minute, et assurez-vous que l’écoulement est suffisant pour éviter le débordement du superfusat.
- Laissez le tissu s’équilibrer pendant 5 minutes, puis enregistrez 5 minutes de données de base brutes et non filtrées.
- Déplacez la ligne d’entrée de perfusion de l’aCSF vers une solution 4-AP et attendez 12 min pour que l’activité rythmique induite par le 4-AP atteigne l’état d’équilibre (2 min pour que les médicaments atteignent le bain et 10 min pour que l’activité atteigne son pic puis plafonne).
- Enregistrez 5 min d’activité induite par 4-AP. Préparez-vous à des enregistrements ultérieurs pour tester les médicaments ou pour vérifier la stabilité du 4-AP.
- Configuration expérimentale
| Dispositions des réseaux de microélectrodes | ||||
| Modèle de réseau de microélectrodes | 60MEA 200/30iR-Ti | 60-3DMEA 100/12/40iR-Ti | 60-3DMEA 200/12/50iR-Ti | 60MEA 500/30iR-Ti |
| Planaire ou 3 dimensions (3D) | Planaire | 3D | 3D | Planaire |
| Grille d’électrodes | 8 x 8 | 8 x 8 | 8 x 8 | 6 x 10 |
| Espacement des électrodes | 200 μm | 100 μm | 200 μm | 500 μm |
| Diamètre de l’électrode | 30 μm | 12 μm | 12 μm | 30 μm |
| Hauteur de l’électrode (3D) | N/a | 40 μm | 50 μm | N/a |
| Expériences | Tranche transversale | Tranche transversale | Sagittal + Horizontal | Sagittal + Horizontal |
Tableau 2 : Dispositions des réseaux de microélectrodes.
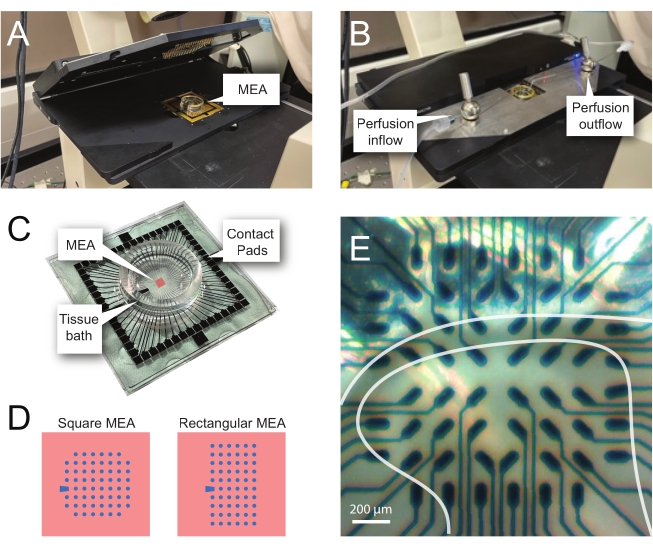
Figure 2 : Positionnement des tissus sur le réseau de microélectrodes. (A) L’image montre une salle de tête MEA ouverte avec un MEA placé en position. (B) Identique à A avec la scène de tête MEA fermée pour les enregistrements et le système de perfusion tissulaire en place. (C) L’image montre un MEA tel que fourni par le fabricant. Les coussinets de contact, qui interagissent avec les ressorts dorés de la scène de tête, et le bain de tissu MEA qui contient la solution de bain de tissu et la tranche de tissu sont montrés. La zone mise en évidence par le carré rouge au centre est l’emplacement du réseau d’électrodes. (D) Les schémas montrent les deux configurations d’électrodes MEA utilisées dans cette étude, avec plus de détails présentés dans le tableau 2. L’électrode de référence est désignée par le trapèze bleu. La disposition gauche des électrodes MEA montre une configuration carrée à 60 électrodes, utilisée le plus souvent dans les modèles de travail présentés 60MEA200/30iR-Ti avec des électrodes de 30 μm de diamètre espacées de 200 μm, ou des MEAs en 3 dimensions espacées de 200 μm et espacées de 100 μm (60MEA200/12/50iR-Ti et 60MEA100/12/40iR-Ti) avec des électrodes de 12 μm de diamètre et de 50 μm ou 40 μm de haut, respectivement. La disposition gauche de l’électrode MEA montre une disposition rectangulaire de 6 x 10 électrodes -60MEA500/30iR-Ti. (E) Image à fort grossissement d’un MEA carré 60MEA100/12/40iR-Ti avec tranche transversale de la moelle épinière positionnée pour l’enregistrement. La tranche se trouve sur les rangées d’électrodes 3-8. La rangée supérieure d’électrodes, qui n’entre en contact avec aucun tissu, sert d’électrodes de référence. La zone SDH apparaît comme une bande semi-transparente. Dans ce cas, le SDH recouvre les électrodes des lignes 4, 5 et 6 et des colonnes 2, 3, 4, 5 et 7 de l’AEM. Barre d’échelle = 200 μm. Abréviations : MEA = réseau de microélectrodes; SDH = corne dorsale superficielle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Modification des tranches
- Après chaque session d’enregistrement, rincez les lignes avec aCSF.
- Retirez le MEA de la scène de tête.
- Retirez bien le filet et le tissu du MEA, rincez-les bien avec de l’aCSF et répétez les étapes ci-dessus avec une nouvelle tranche.
2. Traitement et analyse des données
REMARQUE: Les étapes suivantes détaillent comment utiliser le logiciel d’analyse pour les expériences MEA sur les tranches de moelle épinière. L’une des 60 électrodes sert de référence interne (marquée par un trapèze sur la figure 2 C,D), tandis qu’entre quatre et vingt-cinq des 59 électrodes restantes sont positionnées sous la SDH dans une tranche de moelle épinière de souris adulte. Une analyse ultérieure détecte les formes d’onde du potentiel d’action extracellulaire (EAP) et du potentiel de champ local (LFP) (voir la figure 3B pour des exemples) à partir du signal brut dans cette région.
- Traitement des données brutes
- Ouvrez le logiciel d’analyse et chargez la mise en page d’analyse prédéfinie (Figure 3B).
- Ouvrez le fichier d’intérêt et désélectionnez l’électrode de référence (électrode 15 en 8 x 8 MEA ou électrode E1 en configuration 6 x 10 MEA) et toutes les électrodes jugées excessivement bruyantes.
- Définissez la fenêtre de temps pour l’analyse (0:00 → 5:00 min).
- Accédez à l’onglet Filtre multicouche . Sélectionnez Référence complexe et sélectionnez les électrodes de référence en fonction de l’image prise et des notes prises pendant l’expérience (c.-à-d. les électrodes qui ne se trouvent pas sous les tissus). Pour appliquer et vérifier cela, appuyez sur Explorer avant de continuer.
- Accédez à l’onglet Filtre EAP et appliquez un filtre Butterworth passe-hautde 2 e ordre (coupure de 200 Hz) pour supprimer l’activité LFP.
- Accédez à l’onglet Filtre LFP et appliquez un filtre Butterworth passe-banded’ordre 2 e (fréquences delta de 0,5 à 4 Hz) pour supprimer l’activité EAP.
- Accédez à l’onglet Détecteur EAP et sélectionnez Seuil automatique. Cochez les cases de bord Montant et Descendant et réglez le temps mort sur 0,5 ms.
- Définissez des seuils positifs et négatifs en fonction des données. Inspectez les données en revenant à l’écran de l’analyseur de données brutes, en déplaçant le marqueur temporel, puis en revenant à l’onglet Détecteur EAP et en appuyant sur Explorer. Répétez l’opération jusqu’à ce que vous soyez convaincu que le seuil de détection défini capture les PAE sans capturer le bruit ou l’activité non physiologique. Utilisez les électrodes de référence pour identifier le bruit ou l’activité non physiologique.
REMARQUE: Il est nécessaire de s’assurer qu’un nombre minimal de PAE sont détectés dans les électrodes de référence où l’activité physiologique ne se produira pas. Cependant, de légers écarts dans le niveau de référence pourraient être faussement détectés comme des PAE. Ceci tout en visant à maximiser le nombre d’événements réels détectés dans les électrodes actives. - Accédez à l’onglet Détecteur LFP , sélectionnez le seuil manuel, cochez les cases Bord montant et Descendant et réglez le temps mort sur 3 ms.
- Répétez l’étape 2.1.8 pour une électrode en sélectionnant une électrode avec une activité LFP. Une fois satisfait, sélectionnez Appliquer à tous car les seuils ne seront appliqués qu’à une seule électrode lors de la réalisation d’un seuillage manuel.
- Lors de l’examen des données LFP dans l’onglet Détecteur , notez le nombre maximal de franchissements de seuil pour la forme d’onde LFP et la séparation temporelle maximale des franchissements de seuil pour la forme d’onde LFP unique à utiliser dans une analyse ultérieure.
- Appuyez sur Démarrer l’analyse.
- Une fois l’analyse terminée, accédez à l’onglet Analyseur EAP et exportez les données. Faites de même sous l’onglet Analyseur LFP .
- Répétez ce processus pour tous les autres fichiers de la même tranche.
- Après l’exportation des données, convertissez les fichiers au format xlsx afin qu’ils puissent être lus par le script de programmation utilisé. Nommez les fichiers selon la convention suivante pour que le script fourni puisse les lire : nom de l’expérience (par exemple, données d’échantillon) - numéro de tranche (par exemple, S1) - numéro d’enregistrement (par exemple, R1) - type d’activité (par exemple, pics ou SP, correspondant respectivement aux EAP ou aux LFP).
REMARQUE: L’analyse EAP décrite ici traite le pic de canaux individuels comme une seule population, même si cette activité proviendrait généralement de plusieurs neurones à proximité de l’électrode d’enregistrement. Si le nombre de neurones contribuant aux PAE dans un canal est souhaité, les techniques de tri multipike décrites ailleurs peuvent être appliquées pour distinguer des populations distinctes de pics en fonction des caractéristiques de forme d’onde28.
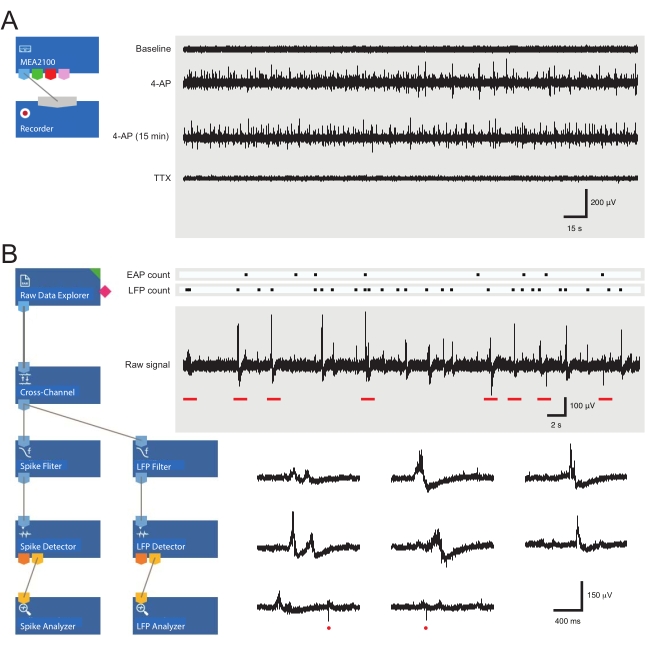
Figure 3: Dispositions d’outils d’enregistrement et d’analyse de données et exemples d’enregistrements de réseaux de microélectrodes montrant le potentiel d’action extracellulaire et les formes d’onde potentielles du champ local. (A) Schéma montre un modèle d’enregistrement préconfiguré utilisé pour l’acquisition de données MEA. La liaison du MEA2100 et de l’outil d’enregistrement (headstage/amplificateur) permet de nommer et d’enregistrer les données. Quatre exemples de traces de données brutes (à droite, époques de 5 minutes) ont été collectés par un canal MEA montrant l’activité au départ, 12 min après l’application 4-AP, 15 minutes supplémentaires après l’activité 4-AP établie et après l’application de TTX (1 μM). Notez que l’ajout de 4-AP (deuxième trace) produit une nette augmentation du bruit de fond et de l’activité EAP/LFP. Il est important de noter que l’activité reste relativement stable pendant au moins 15 minutes après l’établissement de l’activité induite par le 4-AP (troisième trace). L’ajout de TTX (1 μM) supprime toute activité (trace inférieure). (B) Schéma (à gauche) montre la configuration du logiciel de l’analyseur pour l’analyse des données. L’outil d’exploration de données brutes est utilisé pour importer des enregistrements collectés par un logiciel d’enregistrement. Ces données sont ensuite transmises à un outil de filtrage cross-canal qui soustrait le ou les signaux d’électrodes de référence sélectionnés d’autres électrodes pour éliminer le bruit de fond. Les données passent par le filtre EAP et les outils de filtrage LFP pour optimiser les relations signal-bruit pour chaque forme d’onde. Après cette étape, les données de chemin EAP entrent dans l’outil de détection EAP, où les seuils sont définis. Les PAE sont détectés, puis envoyés à l’outil d’analyse EAP où les latences de chaque événement sont enregistrées et exportées sous forme de txt. lime. Un flux de travail identique se produit pour les données LFP à l’aide d’une boîte à outils LFP correspondante. Les traces droites montrent les données d’un seul canal MEA contenant diverses formes d’onde extracellulaires. L’emplacement des signaux EAP et LFP est mis en évidence dans les « rasters de comptage » ci-dessus. Les traces inférieures sont des époques de l’enregistrement supérieur (désignées par des barres rouges) montrant des formes d’onde sur une échelle de temps étendue, y compris divers signaux LFP (notez la variété des apparences) et des EAP extracellulaires individuels (cercles rouges). Notez que la forme d’onde et la polarité LFP/EAP varient en fonction du nombre de neurones produisant ces signaux, de leur proximité avec l’électrode d’enregistrement et de leur emplacement par rapport à la ou aux électrodes voisines. Abréviations : MEA = réseau de microélectrodes; EAP = potentiel d’action extracellulaire; LFP = potentiel de champ local; 4-AP = 4-aminopyridine; TTX = tétrodotoxine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Analyse de synchronicité
NOTE: La synchronicité, ou le nombre d’événements « coïncidents » entre deux électrodes, a été déterminée en utilisant le critère de coïncidence dans la méthode de synchronisation A-SPIKE décrite par Satuvuori et al. 29. Le script utilisé ici ne compare que les électrodes adjacentes les unes aux autres pour l’efficacité (c.-à-d. les voisins horizontaux, verticaux et diagonals); cependant, le script pourrait être réécrit pour comparer toutes les électrodes si nécessaire.- Effectuez une analyse des données à l’aide d’un script de programmation personnalisé, qui extrait les horodatages de latence pour chaque électrode des fichiers .xlsx.
REMARQUE: Cela peut être fait manuellement. - À l’étape 2.1.11, consignez le nombre maximal de franchissements de seuil et la séparation temporelle maximale des franchissements de seuil pour la forme d’onde LFP unique. Modifiez le script d’entrée de ces paramètres définissant LFP pour chaque tranche avant d’exécuter le script.
REMARQUE : Le seuil précédemment effectué dans un logiciel d’analyse capture clairement les PAE comme un événement unique. Cependant, les LFP sont composés d’un nombre variable de pics en fonction de la forme d’onde et du nombre ultérieur de franchissements de seuil par un événement. - Modifiez le script pour saisir les électrodes d’intérêt avant l’analyse.
- Pour déterminer la synchronicité (définie dans le script par des délais modifiables pour que l’activité synchrone se produise à l’intérieur), séparez et analysez les latences extraites pour détecter les événements coïncidents.
REMARQUE : Le script permet de définir la durée maximale entre les événements coïncidents. Ceux-ci sont fixés à 20 ms pour les PAE et à 200 ms pour les PFL. - Exécutez le script pour extraire les horodatages de latence.
REMARQUE : Le fichier de sortie .xlsx contient les interprétations des données de latence, qui sont les comptes EAP et LFP, les fréquences et les comptes d’événements coïncidents pour des électrodes individuelles et des tranches entières. Ces données sont utilisées pour évaluer la fréquence, le nombre d’EAP/LFP, le nombre d’électrodes actives, le nombre d’événements coïncidents, le nombre d’électrodes liées et la force moyenne de ces liaisons.
- Effectuez une analyse des données à l’aide d’un script de programmation personnalisé, qui extrait les horodatages de latence pour chaque électrode des fichiers .xlsx.
Résultats
Modèle d’activité du réseau dans la corne dorsale de la moelle épinière
L’application de 4-AP induit de manière fiable une activité rythmique synchrone dans la moelle épinière DH. Cette activité se traduit par une augmentation des PAE et des PFL. Le dernier signal est une forme d’onde à basse fréquence, qui a déjà été décrite dans les enregistrements MEA30. Les changements dans l’activité du PAE et/ou de la LFP après l’application du médicament ref...
Discussion
Malgré l’importance de la DH spinale dans la signalisation nociceptive, le traitement et les réponses comportementales et émotionnelles qui en résultent qui caractérisent la douleur, les circuits de cette région restent mal compris. L’un des principaux défis dans l’étude de cette question a été la diversité des populations de neurones qui composent ces circuits 6,31,32. Les progrès récents des tech...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Ce travail a été financé par le National Health and Medical Research Council (NHMRC) d’Australie (subventions 631000, 1043933, 1144638 et 1184974 à B.A.G. et R.J.C.) et le Hunter Medical Research Institute (subvention à B.A.G. et R.J.C.).
matériels
| Name | Company | Catalog Number | Comments |
| 4-aminopyridine | Sigma-Aldrich | 275875-5G | |
| 100% ethanol | Thermo Fisher | AJA214-2.5LPL | |
| CaCl2 1M | Banksia Scientific | 0430/1L | |
| Carbonox (Carbogen - 95% O2, 5% CO2) | Coregas | 219122 | |
| Curved long handle spring scissors | Fine Science Tools | 15015-11 | |
| Custom made air interface incubation chamber | |||
| Foetal bovine serum | Thermo Fisher | 10091130 | |
| Forceps Dumont #5 | Fine Science Tools | 11251-30 | |
| Glucose | Thermo Fisher | AJA783-500G | |
| Horse serum | Thermo Fisher | 16050130 | |
| Inverted microscope | Zeiss | Axiovert10 | |
| KCl | Thermo Fisher | AJA383-500G | |
| Ketamine | Ceva | KETALAB04 | |
| Large surgical scissors | Fine Science Tools | 14007-14 | |
| Loctite 454 Instant Adhesive | Bolts and Industrial Supplies | L4543G | |
| MATLAB | MathWorks | R2018b | |
| MEAs, 3-Dimensional | Multichannel Systems | 60-3DMEA100/12/40iR-Ti, 60-3DMEA200/12/50iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in an 8x8 square grid. Electrodes are 12 µm in diameter, 40 µm (100/12/40) or 50 µm (200/12/50) high and equidistantly spaced 100 µm (100/12/40) or 200 µm (200/12/50) apart. |
| MEA headstage | Multichannel Systems | MEA2100-HS60 | |
| MEA interface board | Multichannel Systems | MCS-IFB 3.0 Multiboot | |
| MEA net | Multichannel Systems | ALA HSG-MEA-5BD | |
| MEA perfusion system | Multichannel Systems | PPS2 | |
| MEAs, Planar | Multichannel Systems | 60MEA200/30iR-Ti, 60MEA500/30iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in either a 8x8 square grid (200/30) or a 6x10 rectangular grid (500/30). Electrodes are 30 µm in diameter and equidistantly spaced 200 µm (200/30) or 500 µm (500/30) apart. |
| MgCl2 | Thermo Fisher | AJA296-500G | |
| Microscope camera | Motic | Moticam X Wi-Fi | |
| Multi Channel Analyser software | Multichannel Systems | V 2.17.4 | |
| Multi Channel Experimenter software | Multichannel Systems | V 2.17.4 | |
| NaCl | Thermo Fisher | AJA465-500G | |
| NaHCO3 | Thermo Fisher | AJA475-500G | |
| NaH2PO4 | Thermo Fisher | ACR207805000 | |
| Rongeurs | Fine Science Tools | 16021-14 | |
| Small spring scissors | Fine Science Tools | 91500-09 | |
| Small surgical scissors | Fine Science Tools | 14060-09 | |
| Sucrose | Thermo Fisher | AJA530-500G | |
| Superglue | cyanoacrylate adhesive | ||
| Tetrodotoxin | Abcam | AB120055 | |
| Vibration isolation table | Newport | VH3048W-OPT | |
| Vibrating microtome | Leica | VT1200 S |
Références
- Smith, K. M., et al. Calretinin positive neurons form an excitatory amplifier network in the spinal cord dorsal horn. eLife. 8, 49190 (2019).
- Smith, K. M., et al. Functional heterogeneity of calretinin-expressing neurons in the mouse superficial dorsal horn: implications for spinal pain processing. The Journal of physiology. 593 (19), 4319-4339 (2015).
- Boyle, K. A., et al. Defining a spinal microcircuit that gates myelinated afferent input: Implications for tactile allodynia. Cell Reports. 28 (2), 526-540 (2019).
- Browne, T. J., et al. Transgenic cross-referencing of inhibitory and excitatory interneuron populations to dissect neuronal heterogeneity in the dorsal horn. Frontiers in Molecular Neuroscience. 13, 32 (2020).
- Graham, B. A., Hughes, D. I. Rewards, perils and pitfalls of untangling spinal pain circuits. Current Opinion in Physiology. 11, 35-41 (2019).
- Todd, A. J. Neuronal circuitry for pain processing in the dorsal horn. Nature Reviews Neuroscience. 11 (12), 823-836 (2010).
- Hughes, D. I., Todd, A. J. Central nervous system targets: inhibitory interneurons in the spinal cord. Neurotherapeutics. 17 (3), 874-885 (2020).
- Duan, B., et al. Identification of spinal circuits transmitting and gating mechanical pain. Cell. 159 (6), 1417-1432 (2014).
- Hachisuka, J., Chiang, M. C., Ross, S. E. Itch and neuropathis itch. Pain. 159 (3), 603 (2018).
- Foster, E., et al. Targeted ablation, silencing, and activation establish glycinergic dorsal horn neurons as key components of a spinal gate for pain and itch. Neuron. 85 (6), 1289-1304 (2015).
- Bourane, S., et al. Identification of a spinal circuit for light touch and fine motor control. Cell. 160 (3), 503-515 (2015).
- Cheng, L., et al. Identification of spinal circuits involved in touch-evoked dynamic mechanical pain. Nature neuroscience. 20 (6), 804-814 (2017).
- Peirs, C., et al. Mechanical allodynia circuitry in the dorsal horn is defined by the nature of the injury. Neuron. 109 (1), 73-90 (2021).
- Huang, J., et al. Circuit dissection of the role of somatostatin in itch and pain. Nature Neuroscience. 21 (5), 707-716 (2018).
- Obien, M. E. J., Deligkaris, K., Bullmann, T., Bakkum, D. J., Frey, U. Revealing neuronal function through microelectrode array recordings. Frontiers in Neuroscience. 8, 423 (2015).
- Nam, Y., Wheeler, B. C. In vitro microelectrode array technology and neural recordings. Critical Reviews in Biomedical Engineering. 39 (1), 45-61 (2011).
- Johnstone, A. F., et al. Microelectrode arrays: a physiologically based neurotoxicity testing platform for the 21st century. Neurotoxicology. 31 (4), 331-350 (2010).
- Stett, A., et al. Biological application of microelectrode arrays in drug discovery and basic research. Analytical and Bioanalytical Chemistry. 377 (3), 486-495 (2003).
- Xu, L., et al. Trends and recent development of the microelectrode arrays (MEAs). Biosensors and Bioelectronics. 175 (1), 112854 (2020).
- Chapman, R. J., Cilia La Corte, P. F., Asghar, A. U. R., King, A. E. Network-based activity induced by 4-aminopyridine in rat dorsal horn in vitro is mediated by both chemical and electrical synapses. The Journal of Physiology. 587, 2499-2510 (2009).
- Ruscheweyh, R., Sandkühler, J. Epileptiform activity in rat spinal dorsal horn in vitro has common features with neuropathic pain. Pain. 105 (1-2), 327-338 (2003).
- Kay, C. W., Ursu, D., Sher, E., King, A. E. The role of Cx36 and Cx43 in 4-aminopyridine-induced rhythmic activity in the spinal nociceptive dorsal horn: an electrophysiological study in vitro. Physiological Reports. 4 (14), 12852 (2016).
- Jankowska, E., Lundberg, A., Rudomin, P., Sykova, E. Effects of 4-aminopyridine on synaptic transmission in the cat spinal cord. Brain Research. 240 (1), 117-129 (1982).
- Semba, K., Geller, H. M., Egger, M. D. 4-Aminopyridine induces expansion of cutaneous receptive fields of dorsal horn cells. Brain Research. 343 (2), 398-402 (1985).
- Ruscheweyh, R., Sandkühler, J. Long-range oscillatory Ca2+ waves in rat spinal dorsal horn. European Journal of Neuroscience. 22 (8), 1967-1976 (2005).
- Egert, U., et al. A novel organotypic long-term culture of the rat hippocampus on substrate-integrated multielectrode arrays. Brain Research Protocols. 2 (4), 229-242 (1998).
- Thiebaud, P., De Rooij, N., Koudelka-Hep, M., Stoppini, L. Microelectrode arrays for electrophysiological monitoring of hippocampal organotypic slice cultures. IEEE Transactions on Biomedical Engineering. 44 (11), 1159-1163 (1997).
- Rey, H. G., Pedreira, C., Quiroga, R. Q. Past, present and future of spike sorting techniques. Brain Research Bulletin. 119, 106-117 (2015).
- Satuvuori, E., et al. Measures of spike train synchrony for data with multiple time scales. Journal of Neuroscience Methods. 287, 25-38 (2017).
- Mendis, G. D. C., Morrisroe, E., Reid, C. A., Halgamuge, S. K., Petrou, S. Use of local field potentials of dissociated cultures grown on multi-electrode arrays for pharmacological assays. 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. , 952-956 (2016).
- Hughes, D. I., et al. Morphological, neurochemical and electrophysiological features of parvalbumin-expressing cells: a likely source of axo-axonic inputs in the mouse spinal dorsal horn. The Journal of Physiology. 590 (16), 3927-3951 (2012).
- Peirs, C., Seal, R. P. Neural circuits for pain: recent advances and current views. Science. 354 (6312), 578-584 (2016).
- Li, J., Baccei, M. L. Developmental regulation of membrane excitability in rat spinal lamina I projection neurons. Journal of Neurophysiology. 107 (10), 2604-2614 (2012).
- Li, J., Baccei, M. L. Pacemaker neurons within newborn spinal pain circuits. Journal of Neuroscience. 31 (24), 9010-9022 (2011).
- Sandkühler, J., Eblen-Zajjur, A. Identification and characterization of rhythmic nociceptive and non-nociceptive spinal dorsal horn neurons in the rat. Neuroscience. 61 (4), 991-1006 (1994).
- Lucas-Romero, J., Rivera-Arconada, I., Roza, C., Lopez-Garcia, J. A. Origin and classification of spontaneous discharges in mouse superficial dorsal horn neurons. Scientific Reports. 8 (1), 9735-9735 (2018).
- Antonio, L., et al. L. al. In vitro seizure like events and changes in ionic concentration. Journal of Neuroscience Methods. 260, 33-44 (2016).
- Avoli, M., Jefferys, J. G. Models of drug-induced epileptiform synchronization in vitro. Journal of Neuroscience Methods. 260, 26-32 (2016).
- Taccola, G., Nistri, A. Low micromolar concentrations of 4-aminopyridine facilitate fictive locomotion expressed by the rat spinal cord in vitro. Neuroscience. 126 (2), 511-520 (2004).
- Mitra, P., Brownstone, R. M. An in vitro spinal cord slice preparation for recording from lumbar motoneurons of the adult mouse. Journal of Neurophysiology. 107 (2), 728-741 (2012).
- Egert, U., Heck, D., Aertsen, A. Two-dimensional monitoring of spiking networks in acute brain slices. Experimental Brain Research. 142 (2), 268-274 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon