Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Aufzeichnung der Netzwerkaktivität in spinalen nozizeptiven Schaltkreisen unter Verwendung von Mikroelektrodenarrays
In diesem Artikel
Zusammenfassung
Die kombinierte Verwendung von Mikroelektroden-Array-Technologie und 4-Aminopyridin-induzierter chemischer Stimulation zur Untersuchung der nozizeptiven Aktivität auf Netzwerkebene im Rückenmarksdorsalhorn wird skizziert.
Zusammenfassung
Die Rollen und die Konnektivität bestimmter Arten von Neuronen innerhalb des Rückenmarkshorns (DH) werden mit hoher Geschwindigkeit abgegrenzt, um einen immer detaillierteren Überblick über die Schaltkreise zu erhalten, die der Verarbeitung von Wirbelsäulenschmerzen zugrunde liegen. Die Auswirkungen dieser Verbindungen auf eine breitere Netzwerkaktivität in der DH bleiben jedoch weniger gut verstanden, da sich die meisten Studien auf die Aktivität einzelner Neuronen und kleiner Mikroschaltkreise konzentrieren. Alternativ bietet die Verwendung von Mikroelektrodenarrays (MEAs), die die elektrische Aktivität in vielen Zellen überwachen können, eine hohe räumliche und zeitliche Auflösung der neuronalen Aktivität. Hier wird die Verwendung von MEAs mit Rückenmarksschnitten der Maus beschrieben, um die DH-Aktivität zu untersuchen, die durch chemisch stimulierende DH-Schaltkreise mit 4-Aminopyridin (4-AP) induziert wird. Die resultierende rhythmische Aktivität ist auf das oberflächliche DH beschränkt, über die Zeit stabil, durch Tetrodotoxin blockiert und kann in verschiedenen Scheibenorientierungen untersucht werden. Zusammen bietet dieses Präparat eine Plattform, um die DH-Schaltkreisaktivität im Gewebe von naiven Tieren, Tiermodellen chronischer Schmerzen und Mäusen mit genetisch veränderter nozizeptiver Funktion zu untersuchen. Darüber hinaus können MEA-Aufnahmen in 4-AP-stimulierten Rückenmarksschnitten als Schnellscreening-Werkzeug verwendet werden, um die Fähigkeit neuartiger antinozizeptiver Verbindungen zur Störung der Aktivität im Rückenmark DH zu beurteilen.
Einleitung
Die Rolle bestimmter Arten von inhibitorischen und exzitatorischen Interneuronen innerhalb des Rückenmarks DH wird mit einer schnellen Rate aufgedeckt 1,2,3,4. Zusammen machen Interneuronen über 95% der Neuronen im DH aus und sind an der sensorischen Verarbeitung, einschließlich der Nozizeption, beteiligt. Darüber hinaus sind diese Interneuronenschaltkreise wichtig, um festzustellen, ob periphere Signale die Neuroachse hinaufsteigen, um das Gehirn zu erreichen und zur Schmerzwahrnehmung beizutragen 5,6,7. Bisher haben die meisten Studien die Rolle von DH-Neuronen auf Einzelzell- oder Ganzorganismenebene untersucht, indem sie Kombinationen von intrazellulärer In-vitro-Elektrophysiologie, neuroanatomischer Markierung und In-vivo-Verhaltensanalyse verwendet haben 1,3,8,9,10,11,12,13,14 . Diese Ansätze haben das Verständnis der Rolle bestimmter Neuronenpopulationen bei der Schmerzverarbeitung erheblich verbessert. Es bleibt jedoch eine Lücke im Verständnis, wie bestimmte Zelltypen und kleine Makroschaltkreise große Populationen von Neuronen auf Mikroschaltkreisebene beeinflussen, um anschließend die Ausgabe der DH, Verhaltensreaktionen und die Schmerzerfahrung zu beeinflussen.
Eine Technologie, die die Funktion von Makroschaltungen oder Mehrzellen untersuchen kann, ist das Mikroelektrodenarray (MEA) 15,16. MEAs werden seit mehreren Jahrzehnten verwendet, um die Funktion des Nervensystems zu untersuchen17,18. Im Gehirn haben sie das Studium der neuronalen Entwicklung, der synaptischen Plastizität, des pharmakologischen Screenings und der Toxizitätstests erleichtert17,18. Sie können je nach Art der MEA sowohl für In-vitro- als auch für In-vivo-Anwendungen eingesetzt werden. Darüber hinaus hat sich die Entwicklung von MEAs rasant weiterentwickelt, wobei verschiedene Elektrodennummern und -konfigurationen jetzt verfügbarsind 19. Ein wesentlicher Vorteil von MEAs ist ihre Fähigkeit, die elektrische Aktivität in vielen Neuronen gleichzeitig mit hoher räumlicher und zeitlicher Genauigkeit über mehrere Elektroden zu bewerten15,16. Dies bietet eine breitere Ablesung dessen, wie Neuronen in Schaltkreisen und Netzwerken, unter Kontrollbedingungen und in Gegenwart von lokal applizierten Verbindungen interagieren.
Eine Herausforderung bei In-vitro-DH-Präparaten besteht darin, dass die laufende Aktivität in der Regel niedrig ist. Hier wird diese Herausforderung in Rückenmark-DH-Schaltungen mit dem spannungsgesteuerten K+-Kanalblocker 4-Aminopryidin (4-AP) angegangen, um DH-Schaltkreise chemisch zu stimulieren. Dieses Medikament wurde zuvor verwendet, um rhythmische synchrone elektrische Aktivität im DH von akuten Rückenmarksschnitten und unter akuten In-vivo-Bedingungen 20,21,22,23,24 zu etablieren. Diese Experimente haben Einzelzellpflaster und extrazelluläre Aufzeichnung oder Kalziumbildgebung verwendet, um die 4-AP-induzierte Aktivität20,21,22,23,24,25 zu charakterisieren. Zusammen hat diese Arbeit die Notwendigkeit einer exzitatorischen und inhibitorischen synaptischen Übertragung und elektrischer Synapsen für rhythmische 4-AP-induzierte Aktivität gezeigt. Daher wurde die 4-AP-Antwort als ein Ansatz angesehen, der native polysynaptische DH-Schaltkreise mit biologischer Relevanz und nicht als medikamenteninduziertes Epiphänomen entlarvt. Darüber hinaus zeigt die 4-AP-induzierte Aktivität ein ähnliches Ansprechprofil auf Analgetika und Antiepileptika wie neuropathische Schmerzzustände und wurde verwendet, um neuartige spinalbasierte Analgetika-Ziele wie die Connexine20,21,22 vorzuschlagen.
Hier wird ein Präparat beschrieben, das MEAs und chemische Aktivierung des spinalen DH mit 4-AP kombiniert, um diese nozizeptive Schaltung auf der Makroschaltungs- oder Netzwerkebene der Analyse zu untersuchen. Dieser Ansatz bietet eine stabile und reproduzierbare Plattform für die Untersuchung nozizeptiver Schaltkreise unter naiven und neuropathischen "schmerzähnlichen" Bedingungen. Dieses Präparat ist auch leicht anwendbar, um die Wirkung bekannter Analgetika auf Schaltkreisebene zu testen und neuartige Analgetika im hyperaktiven Rückenmark zu screenen.
Protokoll
Es wurden Studien an männlichen und weiblichen c57Bl/6-Mäusen im Alter von 3-12 Monaten durchgeführt. Alle experimentellen Verfahren wurden in Übereinstimmung mit der Tierpflege- und Ethikkommission der University of Newcastle durchgeführt (Protokolle A-2013-312 und A-2020-002).
1. In-vitro-Elektrophysiologie
- Herstellung von Lösungen für die Präparation und Aufzeichnung von Rückenmarksschnitten
- Künstliche Zerebrospinalflüssigkeit
HINWEIS: Künstliche Zerebrospinalflüssigkeit (aCSF) wird in einer Grenzflächeninkubationskammer verwendet, in der die Scheiben bis zum Beginn der Aufzeichnung und während der Experimente als Perfusat und Verdünnungsmittel für Medikamente gelagert werden. Die detaillierte Zusammensetzung ist Tabelle 1 zu entnehmen.
- Künstliche Zerebrospinalflüssigkeit
| Chemisch | aCSF (mM) | aCSF (g/100 ml) | Saccharose-substituierter aCSF (mM) | Saccharose-substituierter aCSF (g/100 ml) | Kaliumhohes aCSF (mM) | aCSF mit hohem Kaliumgehalt (g/100 ml) |
| Natriumchlorid (NaCl) | 118 | 0.690 | - | - | 118 | 0.690 |
| Natriumhydrogencarbonat (NaHCO 3) | 25 | 0.210 | 25 | 0.210 | 25 | 0.210 |
| Traubenzucker | 10 | 0.180 | 10 | 0.180 | 10 | 0.180 |
| Potasiumchlorid (KCl) | 2.5 | 0.019 | 2.5 | 0.019 | 4.5 | 0.034 |
| Natriumdihydrogenphosphat (NaH2PO4) | 1 | 0.012 | 1 | 0.012 | 1 | 0.012 |
| Magnesiumclolid (MgCl2) | 1 | 0.01 | 1 | 0.01 | 1 | 0.01 |
| Calciumchlorid (CaCl2) | 2.5 | 0.028 | 2.5 | 0.028 | 2.5 | 0.028 |
| Saccharose | - | - | 250 | 8.558 | - | - |
Tabelle 1: Zusammensetzung der künstlichen Zerebrospinalflüssigkeit Abkürzung: aCSF = künstliche Zerebrospinalflüssigkeit.
- Es wird aCSF mit (in mM) 118 NaCl, 25NaHCO3, 10 Glukose, 2,5 KCl, 1NaH2PO4, 1 MgCl2 und 2,5 CaCl2 hergestellt, indem die erforderlichen Mengen der vorstehenden, ausgenommen CaCl2, zu 2 L destilliertem Wasser hinzugefügt werden.
- Die obige Lösung mit Carbogen (95%O2, 5% CO 2) für 5 min blasen und CaCl2 hinzufügen.
HINWEIS: Dieser Schritt verhindert CaCl2-Niederschlag , d.h. die Lösung sollte nicht trüb werden. Für die Arzneimittelanwendung während der Experimente verdünnen Sie die Arzneimittelbestandslösungen in aCSF auf die gewünschten Endkonzentrationen.
- Saccharose-substituierte künstliche Zerebrospinalflüssigkeit
HINWEIS: Saccharose-substituiertes aCSF wird während der Dissektion und Rückenmarksschnitt verwendet. Wie der Name schon sagt, wird Saccharose durch NaCl ersetzt, um die neuronale Erregung während dieser Verfahren zu reduzieren und gleichzeitig die Osmolarität aufrechtzuerhalten. Die detaillierte Zusammensetzung ist Tabelle 1 zu entnehmen.- Saccharose-substituierte aCSF mit (in mM) 250 Saccharose, 25NaHCO3, 10 Glukose, 2,5 KCl, 1NaH2PO4, 1 MgCl2 und 2,5 CaCl2 werden hergestellt, indem die erforderlichen Mengen aller oben genannten, ausgenommen CaCl2, zu 300 ml destilliertem Wasser hinzugefügt werden.
- Blasen Sie die Lösung mit Carbogen für 5 min und fügen Sie dann CaCl2 hinzu.
- Lagern Sie die Lösung in einem -80 °C Gefrierschrank für ca. 40 min oder bis die Lösung eine Aufschlämmung bildet. Vermeiden Sie das Einfrieren von Feststoffen und verwenden Sie sie in der Güllekonsistenz.
- Vorbereitung von Mikroelektroden-Arrays
HINWEIS: Die Kontaktfläche des MEA erfordert eine Vorbehandlung, um es hydrophil zu machen.- Vor dem Experiment füllen Sie die MEA 30 min lang entweder mit fetalem Rinderserum (FBS) oder Pferdeserum (HS).
- Entfernen Sie das FBS oder HS und spülen Sie MEA gründlich mit etwa fünf Waschungen destilliertem Wasser ab, bis das destillierte Wasser nicht mehr schaumig ist. Füllen Sie den Brunnen mit aCSF, bereit für den Einsatz.
- Akute Rückenmarksschnittvorbereitung
HINWEIS: Die Vorbereitung der Rückenmarksschnitte der Maus ist wie zuvor von Smith et al.2 beschrieben. Idealerweise sollte die Entfernung der lumbosakralen Vergrößerung nicht länger als 8-10 Minuten dauern (Schritte 1.3.2-1.3.11 unten).- Betäuben Sie die Maus tief mit 100 mg/kg Ketamin (i.p.) und enthaupten Sie sie dann mit einer großen chirurgischen Schere.
- Entfernen Sie die Haut über der Bauchregion, indem Sie einen kleinen Schnitt in der Haut auf Höhe der Hüften machen. Ziehen Sie die Haut auf beiden Seiten des Schnitts rostral, bis die gesamte Haut entfernt ist, d. H. Von der Oberseite des Brustkorbs bis zur Oberseite des Beckens (sowohl ventral als auch dorsal).
- Legen Sie den Körper auf Eis und verwenden Sie einen ventralen Ansatz, um die Wirbelsäule freizulegen, indem Sie alle Eingeweide entfernen und die Rippen seitlich zum Brustbein schneiden.
- Entfernen Sie den ventralen Brustkorb, beide Schulterblätter (bei etwa T2 abgeschnitten) und die unteren Gliedmaßen und das Becken (etwa an der Oberseite des Kreuzbeins abgeschnitten).
- Übertragen Sie die Wirbelsäule und die Rippenvorbereitung in ein Sezierbad, das eiskalte Saccharose aCSF enthält. Stecken Sie alle vier Ecken des Präparats (ventrale Oberfläche nach oben), indem Sie Stifte durch die unteren Rückenmuskeln und die befestigten oberen Rippen legen.
- Entfernen Sie alle Muskeln und Bindegewebe, die die ventrale Oberfläche der Wirbel mit Rongeurs überlagern, und identifizieren Sie die Wirbelregion über der lumbosakralen Vergrößerung, die ungefähr unter den T12- bis L2-Wirbelkörpern liegt.
- Entfernen Sie einen Wirbelkörper, der kaudal zur lumbosakralen Vergrößerungsregion ist, um den Zugang zum Rückenmark zu ermöglichen, während es im Wirbelkanal sitzt.
- Schneiden Sie mit einer gekrümmten Federschere die Wirbelstiele bilateral durch, während Sie den Wirbelkörper rostral anheben und ziehen, um die ventralen und dorsalen Aspekte der Wirbel zu trennen und das Rückenmark freizulegen.
- Sobald die Wirbelkörper entfernt wurden, um die lumbosakrale Vergrößerung zu enthüllen, reinigen Sie vorsichtig die verbleibenden Wurzeln, die das Rückenmark mit einer Federschere verankern, bis das Kord frei schwebt.
- Isolieren Sie das Rückenmark mit rostralen und kaudalen Schnitten weit über und unter der lumbosakralen Vergrößerung, so dass die Zielregion des Rückenmarks "frei schweben" kann.
HINWEIS: Die bevorzugte Schnittausrichtung bestimmt, wie das Kabel anschließend zum Schneiden montiert wird (Abbildung 1). - Für Querschnitte heben Sie das lumbosakrale Segment durch eine befestigte Wurzel an und legen Sie es auf einen vorgeschnittenen Polystyrolblock (Styropor) (1 cm x 1 cm x 1 cm) mit einem flachen Kanalschnitt in der Mitte. Verwenden Sie Cyanacrylat-Klebstoff (siehe Materialtabelle), um den Block und die Schnur an der Trennplattform zu befestigen und in das Schneidbad zu legen, das eiskalte Saccharose aCSF (Aufschlämmung) enthält.
HINWEIS: Der flache Kanal hilft, das Rückenmark zu sichern und zu orientieren, wobei die Rückenseite freigelegt ist und das thorakale Ende des Schnabels am unteren Rand des Blocks liegt. - Für sagittale Scheiben legen Sie eine dünne Linie von Cyanacrylat-Klebstoff auf die Trennplattform, heben Sie die lumbosakrale Vergrößerung durch eine befestigte Wurzel an und legen Sie die Schnur entlang der Leimlinie, um sicherzustellen, dass sich eine seitliche Oberfläche im Klebstoff befindet und die andere nach oben zeigt. Legen Sie es in das Schneidbad, das eiskalte Saccharose aCSF (Slurry) enthält.
- Für horizontale Scheiben eine dünne Linie aus Cyanacrylat-Klebstoff auf die Trennplattform legen. Heben Sie die lumbosakrale Vergrößerung durch eine befestigte Wurzel an und platzieren Sie die lumbosakrale Vergrößerung entlang der Klebstofflinie, um sicherzustellen, dass sich die ventrale Oberfläche im Klebstoff befindet und die dorsale Oberfläche nach oben zeigt. Verwenden Sie angebrachte Wurzeln, um das Kabel zu positionieren. Legen Sie es in das Schneidbad, das eiskalte Saccharose aCSF (Slurry) enthält.
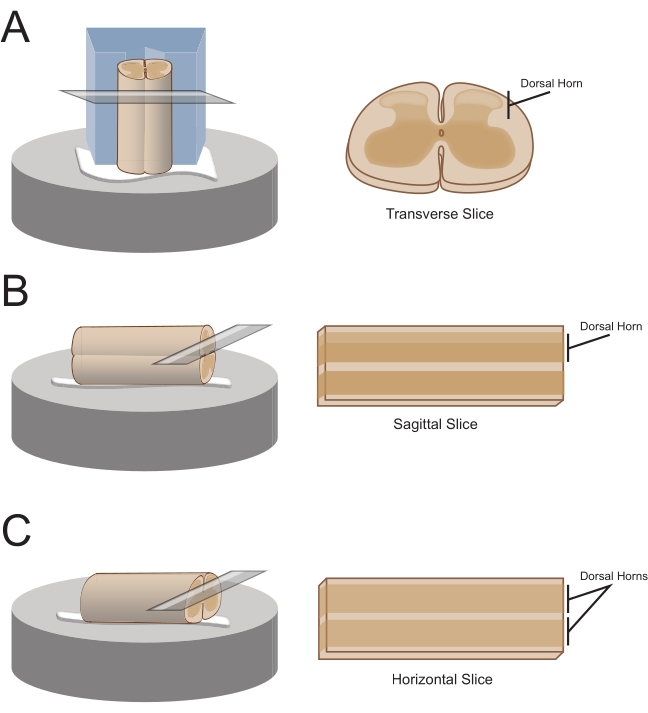
Abbildung 1: Orientierungen der Rückenmarksschnitte, Montage- und Schneidmethoden. (A) Querschnitte erfordern einen Styropor-Schneidblock, in den eine Stütznut geschnitten ist. Das Rückenmark ruht gegen den Block in der Stütznut, die dorsale Seite des Rückenmarks zeigt vom Block weg. Block und Kordel werden mit Cyanacrylat-Klebstoff auf eine Schneidstufe geklebt. (B) Sagittal-Scheiben werden hergestellt, indem eine dünne Linie Cyanacrylat-Klebstoff auf die Schneidstufe aufgetragen und dann das Rückenmark auf der Seite auf dem Kleber positioniert wird. (C) Horizontale Scheiben werden hergestellt, indem eine dünne Linie Cyanacrylatkleber auf die Schneidstufe gelegt und dann die ventrale Seite des Rückenmarks nach unten auf dem Kleber positioniert wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Erhalten Sie 300 μm dicke Scheiben (L1-L5, gleiche Dicke unabhängig von der Ausrichtung) mit einem vibrierenden Mikrotom mit den folgenden Einstellungen: Geschwindigkeit 0,06 mm/s, Amplitude 2,50 mm und kalibriert auf eine Amplitudenabweichung von ±0,02 Höhe.
- Übertragen Sie die Scheiben in eine Inkubationskammer mit Luftschnittstelle, die sauerstoffreiches aCSF enthält.
- Lassen Sie die Scheiben vor der Aufnahme 1 h bei Raumtemperatur (20-24 °C) ausgleichen.
- Mikroelektroden-Array-Aufnahmen
HINWEIS: In den folgenden Schritten wird detailliert beschrieben, wie Sie Aufzeichnungsdaten aus MEA-basierten Experimenten auf Rückenmarksschnitten verwenden. Je nach Experiment können mehrere MEA-Designs verwendet werden. Designdetails für MEAs, die in diesen Experimenten verwendet wurden, sind in Tabelle 2 und Abbildung 2. Detaillierte Designinformationen wurden von Egert et al. veröffentlicht.26 und Thiebaud et al.27 für planare bzw. 3-dimensionale (3D) MEAs. Beide MEA-Typen bestehen aus 60 Titannitrid-Elektroden mit einer Siliziumnitrid-Isolierschicht sowie Titannitrid-Spuren und Kontaktpads.- Versuchsaufbau
- Schalten Sie den Computer und die Schnittstellenkarte ein und starten Sie die Aufnahmesoftware.
- Laden Sie die vormontierte Aufzeichnungsvorlage (Abbildung 3A). Benennen Sie die Dateien für den Tag auf der Registerkarte Aufzeichnung.
- ACSF mit Carbogen (5% CO2, 95%O2) für die Dauer des Experiments kontinuierlich blasen.
- Schalten Sie das Perfusionssystem ein, das von einer Peristaltikpumpe gesteuert wird. Legen Sie die Einlassleitung in aCSF und das Einlassende in ein Abfallbecherglas. Grundieren Sie die Perfusionslinien mit aCSF.
- Bereiten Sie 4-AP und alle anderen Arzneimittellösungen vor, indem Sie die Bestände in 50 ml aCSF auf die erforderliche Endkonzentration (z. B. 200 μM für 4-AP) verdünnen.
- Legen Sie die Arzneimittellösungen in Medikamententöpfe und blasen Sie sie mit Carbogen auf.
- 4-AP-Aktivität
- Nach der Inkubation eine einzelne Scheibe aus dem Inkubator mit einer großspitzen Pasteur-Pipette übertragen, die mit aCSF gefüllt ist.
- Legen Sie das Segment in das MEA-Bohrloch und fügen Sie zusätzliches aCSF hinzu.
- Positionieren Sie die Scheibe mit einem feinen Kurzhaarpinsel über dem 60-Elektroden-Aufnahmefeld. Vermeiden Sie es, die Elektroden mit dem Pinsel zu berühren oder das Gewebe über die Elektroden zu ziehen, insbesondere wenn Sie 3D-Arrays verwenden.
HINWEIS: Je nach MEA-Layout kann dies mit oder ohne Hilfe eines Mikroskops für eine genaue Positionierung erfolgen. - Nachdem Sie die Scheibe positioniert haben, legen Sie ein gewichtetes Netz über das Gewebe, um es an Ort und Stelle zu halten und einen guten Kontakt mit MEA-Elektroden zu fördern.
HINWEIS: Das Slice muss nach der Nettoplatzierung möglicherweise neu positioniert werden. - Platzieren Sie die MEA in der Aufzeichnungskopfbühne (Abbildung 2A,B).
- Überprüfen Sie die Position des Gewebes über den Elektroden mit einem inversen Mikroskop (2-fache Vergrößerung), um zu bestätigen, dass sich so viele Elektroden wie möglich unter dem oberflächlichen DH (SDH) befinden. Stellen Sie sicher, dass mindestens 2-6 Elektroden den Schnitt nicht berühren, da diese Elektroden für die Subtraktion von Rauschen und die Aufzeichnung von Artefakten während der Analyse wichtig sind (Abbildung 2E).
- Schalten Sie die Kamera ein, schließen Sie sie an das Gerät an und nehmen Sie ein Referenzbild des Slices relativ zum MEA auf, das während der Analyse verwendet werden kann.
- Drücken Sie in der Aufzeichnungssoftware auf Datenerfassung starten , und vergewissern Sie sich, dass alle Elektroden ein klares Signal empfangen.
HINWEIS: Wenn das Signal laut ist, lösen Sie die Kopfstufe und reinigen Sie sowohl die MEA-Kontaktpads als auch die Goldfederkontakte mit 70% Ethanol (verwenden Sie ein Labortuch, um sicherzustellen, dass die Pads und Kontakte nach der Reinigung trocken sind). Wenn das Signal immer noch verrauscht ist, schalten Sie die fehlerhaften Elektroden in der Aufnahmesoftware aus oder notieren Sie sich den Ausschluss später während der Analyse. - Befestigen Sie die Perfusionseinlass- und -auslassleitungen an der MEA-Vertiefung (zuvor mit aCSF gefüllt) und schalten Sie das Perfusionssystem ein. Überprüfen Sie die Durchflussrate, idealerweise 4-6 Badvolumen pro Minute, und stellen Sie sicher, dass der Abfluss ausreicht, um einen Überlauf des Superfusats zu verhindern.
- Lassen Sie das Gewebe 5 Minuten lang ausgleichen und zeichnen Sie dann 5 Minuten rohe, ungefilterte Basisdaten auf.
- Verschieben Sie die Perfusionseinlassleitung von aCSF auf eine 4-AP-Lösung und warten Sie 12 Minuten, bis die 4-AP-induzierte rhythmische Aktivität einen stabilen Zustand erreicht hat (2 Minuten, bis Medikamente das Bad erreichen, und 10 Minuten, bis die Aktivität ihren Höhepunkt erreicht und dann ein Plateau erreicht).
- Zeichnen Sie 5 Minuten 4-AP-induzierte Aktivität auf. Seien Sie auf nachfolgende Aufnahmen vorbereitet, um die Medikamente zu testen oder die Stabilität von 4-AP zu überprüfen.
- Versuchsaufbau
| Mikroelektroden-Array-Layouts | ||||
| Mikroelektroden-Array-Modell | 60MEA 200/30iR-Ti | 60-3DMEA 100/12/40iR-Ti | 60-3DMEA 200/12/50iR-Ti | 60MEA 500/30iR-Ti |
| Planar oder 3-dimensional (3D) | Flach | 3D | 3D | Flach |
| Elektrodengitter | 8 x 8 | 8 x 8 | 8 x 8 | 6 x 10 Zoll |
| Elektrodenabstand | 200 μm | 100 μm | 200 μm | 500 μm |
| Elektrodendurchmesser | 30 μm | 12 μm | 12 μm | 30 μm |
| Elektrodenhöhe (3D) | N/A | 40 μm | 50 μm | N/A |
| Experimente | Querschnitt | Querschnitt | Sagittal + Horizontal | Sagittal + Horizontal |
Tabelle 2: Layouts von Mikroelektrodenarrays
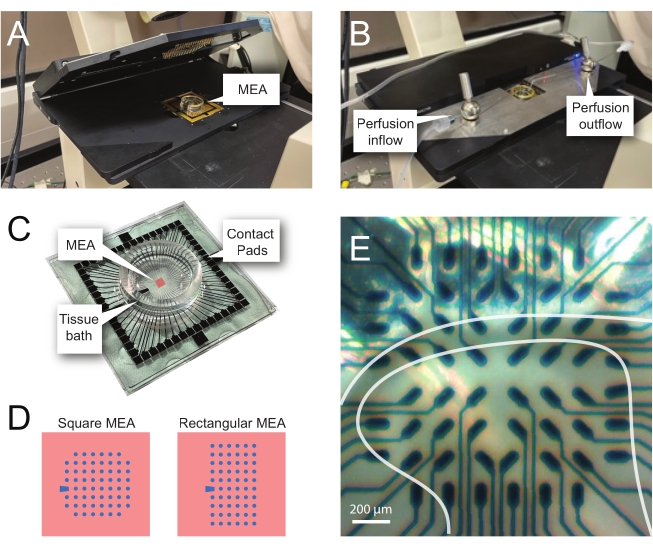
Abbildung 2: Gewebepositionierung auf dem Mikroelektrodenarray . (A) Das Bild zeigt eine offene MEA-Kopfbühne mit einem MEA in Position. (B) Wie A mit geschlossener MEA-Kopfstufe für Aufzeichnungen und vorhandenem Gewebeperfusionssystem. (C) Das Bild zeigt eine MEA, wie sie vom Hersteller geliefert wird. Gezeigt werden Kontaktpads, die mit den Goldfedern der Kopfbühne verbunden sind, und das MEA-Gewebebad, in dem sich die Gewebebadelösung und die Gewebescheibe befinden. Der Bereich, der durch das rote Quadrat in der Mitte hervorgehoben wird, ist die Position des Elektrodenarrays. (D) Die Schaltpläne zeigen die beiden in dieser Studie verwendeten MEA-Elektrodenkonfigurationen, wobei weitere Einzelheiten in Tabelle 2 dargestellt sind. Die Referenzelektrode ist durch das blaue Trapez gekennzeichnet. Das linke MEA-Elektrodenlayout zeigt eine quadratische Konfiguration mit 60 Elektroden, die am häufigsten in den vorgestellten Arbeitsmodellen 60MEA200/30iR-Ti mit Elektroden mit einem Durchmesser von 30 μm im Abstand von 200 μm oder 200 μm im Abstand und 100 μm im Abstand von 3-dimensionalen MEAs (60MEA200/12/50iR-Ti und 60MEA100/12/40iR-Ti) mit Elektroden mit einem Durchmesser von 12 μm und einer Höhe von entweder 50 μm oder 40 μm verwendet wird. beziehungsweise. Das linke MEA-Elektrodenlayout zeigt ein rechteckiges 6 x 10-Elektroden-Layout-60MEA500/30iR-Ti. (E) Hochvergrößerungsbild eines quadratischen MEA 60MEA100/12/40iR-Ti mit quer angeordnetem Rückenmarksschnitt für die Aufnahme. Die Scheibe sitzt auf den Elektrodenreihen 3-8. Die obere Reihe von Elektroden, die kein Gewebe berühren, dienen als Referenzelektroden. Der SDH-Bereich erscheint als halbtransparentes Band. In diesem Fall überlagert das SDH Elektroden in den Zeilen 4, 5 und 6 und den Spalten 2, 3, 4, 5 und 7 der MEA. Maßstabsleiste = 200 μm. Abkürzungen: MEA = Mikroelektrodenarray; SDH = oberflächliches Rückenhorn. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Ändern von Segmenten
- Nach jeder Aufnahmesitzung spülen Sie die Zeilen mit aCSF.
- Entfernen Sie die MEA von der Kopfbühne.
- Entfernen Sie das Netz und das Gewebe aus dem MEA-Brunnen, spülen Sie sie gut mit aCSF ab und wiederholen Sie die obigen Schritte mit einer neuen Scheibe.
2. Datenverarbeitung und -analyse
HINWEIS: In den folgenden Schritten wird beschrieben, wie die Analysesoftware für MEA-Experimente an Rückenmarksschnitten verwendet wird. Eine der 60 Elektroden dient als interne Referenz (markiert durch ein Trapez in Abbildung 2 C,D), während zwischen vier und fünfundzwanzig der verbleibenden 59 unter dem SDH in einer erwachsenen Maus-Rückenmarksscheibe positioniert sind. Die anschließende Analyse erkennt extrazelluläre Aktionspotential- (EAP) und lokale Feldpotentialwellenformen (LFP) (siehe Abbildung 3B für Beispiele) aus dem Rohsignal in diesem Bereich.
- Rohdatenverarbeitung
- Öffnen Sie die Analysesoftware und laden Sie das vorgefertigte Analyselayout (Abbildung 3B).
- Öffnen Sie die Datei von Interesse und deaktivieren Sie die Referenzelektrode (Elektrode 15 in 8 x 8 MEA- oder Elektrode E1 in 6 x 10 MEA-Konfiguration) und alle Elektroden, die als übermäßig laut erachtet werden.
- Legen Sie das Zeitfenster für die Analyse fest (0:00 → 5:00 min).
- Wechseln zur Registerkarte Cross-channel filter Wählen Sie Komplexe Referenz und wählen Sie die Referenzelektroden basierend auf dem aufgenommenen Bild und den während des Experiments gemachten Notizen (d. h. den Elektroden, die nicht unter Gewebe liegen) aus. Um dies anzuwenden und zu überprüfen, drücken Sie Durchsuchen , bevor Sie fortfahren.
- Wechseln Sie zur Registerkarte EAP-Filter und wenden Sie einen Hochpass-Butterworth-Filter 2. Ordnung an (200 Hz abgeschnitten), um LFP-Aktivitäten zuentfernen.
- Wechseln Sie zur Registerkarte LFP-Filter und wenden Sie einen Butterworth-Filter 2. Ordnung (Deltafrequenzen von 0,5-4 Hz) an, um EAP-Aktivitäten zuentfernen.
- Wechseln Sie zur Registerkarte EAP-Detektor, und wählen Sie Automatischer Schwellenwert aus. Aktivieren Sie die Kontrollkästchen Steigende und fallende Kante und stellen Sie die Totzeit auf 0,5 ms ein.
- Legen Sie basierend auf den Daten positive und negative Schwellenwerte fest. Überprüfen Sie die Daten, indem Sie zum Bildschirm des Rohdatenanalysators zurückkehren, die Zeitmarkierung verschieben und dann zur Registerkarte EAP-Detektor zurückkehren und auf Durchsuchen klicken. Wiederholen Sie den Vorgang, bis Sie sich vergewissert haben, dass der eingestellte Erkennungsschwellenwert EAPs erfasst, ohne Rauschen/nicht-physiologische Aktivitäten zu erfassen. Verwenden Sie die Referenzelektroden, um Rauschen/nicht-physiologische Aktivität zu identifizieren.
HINWEIS: Es muss sichergestellt werden, dass eine minimale Anzahl von EAPs in Referenzelektroden erkannt wird, in denen keine physiologische Aktivität auftritt. Eher geringfügige Abweichungen in der Baseline können jedoch fälschlicherweise als EAPs erkannt werden. Dies zielt gleichzeitig darauf ab, die Anzahl der in den aktiven Elektroden erkannten realen Ereignisse zu maximieren. - Wechseln Sie zur Registerkarte LFP-Detektor, wählen Sie den manuellen Schwellenwert aus, aktivieren Sie die Kontrollkästchen Steigende und fallende Kante und stellen Sie die Totzeit auf 3 ms ein.
- Wiederholen Sie Schritt 2.1.8 für eine Elektrode, indem Sie eine Elektrode mit LFP-Aktivität auswählen. Wenn Sie zufrieden sind, wählen Sie Auf alle anwenden aus, da die Schwellenwerte nur auf eine einzelne Elektrode angewendet werden, wenn eine manuelle Schwellenwertierung durchgeführt wird.
- Beachten Sie bei der Untersuchung der LFP-Daten auf der Registerkarte Detektor die maximale Anzahl von Schwellenwertübergängen für die eine LFP-Wellenform und die maximale Zeittrennung der Schwellenübergänge für die eine LFP-Wellenform zur Verwendung in der späteren Analyse.
- Drücken Sie Analyse starten.
- Wenn die Analyse abgeschlossen ist, wechseln Sie zur Registerkarte EAP Analyzer und exportieren Sie die Daten. Machen Sie dasselbe auf der Registerkarte LFP-Analysator .
- Wiederholen Sie diesen Vorgang für alle anderen Dateien aus demselben Segment.
- Konvertieren Sie die Dateien nach dem Datenexport in das xlsx-Format, damit sie vom verwendeten Programmierskript gelesen werden können. Benennen Sie die Dateien gemäß der folgenden Konvention, damit das bereitgestellte Skript sie lesen kann: Experimentname (z. B. Beispieldaten) - Slice-Nummer (z. B. S1) - Aufzeichnungsnummer (z. B. R1) - Aktivitätstyp (z. B. Spitzen oder SPs, entsprechend EAPs bzw. LFPs).
HINWEIS: Die hier beschriebene EAP-Analyse behandelt Spiking aus einzelnen Kanälen als eine einzige Population, obwohl diese Aktivität üblicherweise von mehreren Neuronen in unmittelbarer Nähe der Aufzeichnungselektrode ausgehen würde. Wenn die Anzahl der Neuronen, die zu EAPs in einem Kanal beitragen, gewünscht wird, können an anderer Stelle beschriebene Multispike-Sortiertechniken angewendet werden, um verschiedene Populationen von Spikes basierend auf Wellenformmerkmalenzu unterscheiden 28.
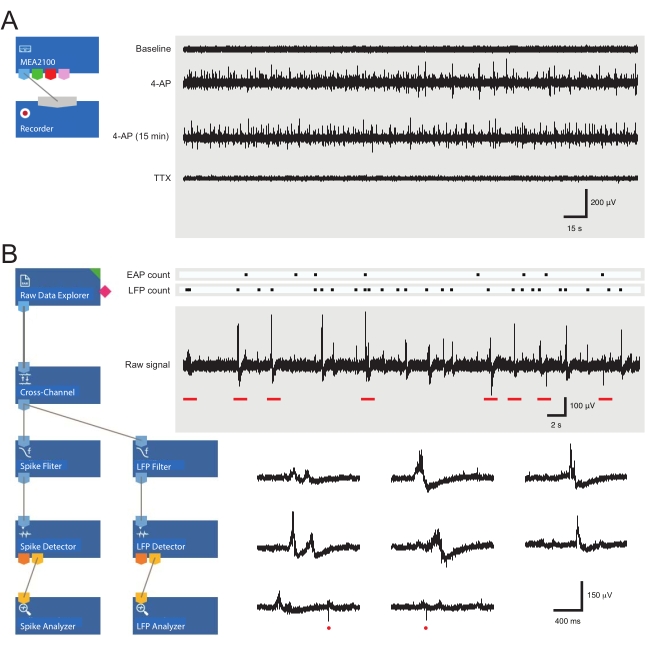
Abbildung 3: Datenaufzeichnungs- und Analysewerkzeuglayouts und Beispielaufzeichnungen von Mikroelektrodenarrays, die extrazelluläres Aktionspotential und lokale Feldpotentialwellenformen zeigen . (A) Der Schaltplan zeigt eine vorkonfigurierte Aufzeichnungsvorlage, die für die Erfassung von MEA-Daten verwendet wird. Durch die Verknüpfung des MEA2100 mit dem Aufzeichnungstool (Headstage/Verstärker) können die Daten benannt und gespeichert werden. Vier Beispielspuren von Rohdaten (rechts, 5-minütige Epochen) wurden von einem MEA-Kanal gesammelt, der die Aktivität zu Studienbeginn, 12 Minuten nach der 4-AP-Anwendung, weitere 15 Minuten nach der etablierten 4-AP-Aktivität und nach der Badanwendung von TTX (1 μM) zeigte. Beachten Sie, dass das Hinzufügen von 4-AP (zweite Spur) zu einem deutlichen Anstieg der Hintergrundgeräusche und der EAP / LFP-Aktivität führt. Wichtig ist, dass die Aktivität für mindestens 15 Minuten relativ stabil bleibt, nachdem eine 4-AP-induzierte Aktivität festgestellt wurde (dritte Spur). Durch die Zugabe von TTX (1 μM) wird die gesamte Aktivität aufgehoben (untere Spur). (B) Der Schaltplan (links) zeigt die Konfiguration der Analysesoftware für die Datenanalyse. Das Rohdaten-Explorer-Tool wird verwendet, um Aufzeichnungen zu importieren, die von der Aufzeichnungssoftware gesammelt wurden. Diese Daten werden dann durch ein Cross-Channel-Filterwerkzeug geleitet, das die ausgewählten Referenzelektrodensignale von anderen Elektroden subtrahiert, um Hintergrundgeräusche zu entfernen. Die Daten durchlaufen den EAP-Filter und die LFP-Filterwerkzeuge, um die Signal-Rausch-Beziehungen für jede Wellenform zu optimieren. Nach diesem Schritt gelangen die EAP-Pfaddaten in das EAP-Detektorwerkzeug, in dem Schwellenwerte festgelegt werden. EAPs werden erkannt und dann an das EAP-Analysetool gesendet, wo die Latenzen jedes Ereignisses aufgezeichnet und als txt exportiert werden. Datei. Ein identischer Workflow für LFP-Daten erfolgt unter Verwendung eines entsprechenden LFP-Toolkits. Rechte Spuren zeigen Daten von einem einzelnen MEA-Kanal, der verschiedene extrazelluläre Wellenformen enthält. Die Position von EAP- und LFP-Signalen wird in den obigen "Zählrastern" hervorgehoben. Untere Spuren sind Epochen aus der oberen Aufzeichnung (gekennzeichnet durch rote Balken), die Wellenformen auf einer erweiterten Zeitskala zeigen, einschließlich verschiedener LFP-Signale (beachten Sie die Vielfalt der Erscheinungen) und einzelner extrazellulärer EAPs (rote Kreise). Beachten Sie, dass LFP/EAP-Wellenform und Polarität relativ zur Anzahl der Neuronen, die diese Signale erzeugen, ihrer Nähe zur Aufnahmeelektrode und ihrer Position in Bezug auf die nahe gelegenen Elektroden, variieren. Abkürzungen: MEA = Mikroelektrodenarray; EAP = extrazelluläres Aktionspotential; LFP = lokales Feldpotential; 4-AP = 4-Aminopyridin; TTX = Tetrodotoxin. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Synchronitätsanalyse
HINWEIS: Die Synchronizität oder die Anzahl der "zufälligen" Ereignisse zwischen zwei Elektroden wurde unter Verwendung des Koinzidenzkriteriums innerhalb der von Satuvuori et al. beschriebenen A-SPIKE-Synchronisationsmethode bestimmt. 29. Das hier verwendete Skript vergleicht nur nebeneinander liegende Elektroden hinsichtlich ihrer Effizienz (d. h. horizontale, vertikale und diagonale Nachbarn); Das Skript könnte jedoch umgeschrieben werden, um bei Bedarf alle Elektroden zu vergleichen.- Führen Sie die Datenanalyse mit einem benutzerdefinierten Programmierskript durch, das Latenzzeitstempel für jede Elektrode aus den .xlsx Dateien extrahiert.
HINWEIS: Dies kann manuell erfolgen. - Zeichnen Sie in Schritt 2.1.11 die maximale Anzahl von Schwellenübergängen und die maximale Zeittrennung der Schwellenübergänge für die eine LFP-Wellenform auf. Ändern Sie das Skript für die Eingabe dieser LFP-definierenden Parameter für jedes Segment, bevor Sie das Skript ausführen.
HINWEIS: Schwellenwerte, die zuvor in der Analysesoftware durchgeführt wurden, erfassen EAPs eindeutig als ein einzelnes Ereignis. LFPs bestehen jedoch aus einer variablen Anzahl von Peaks, abhängig von der Form der Wellenform und der anschließenden Anzahl der Schwellenübergänge durch das eine Ereignis. - Ändern Sie das Skript, um die gewünschten Elektroden vor der Analyse einzugeben.
- Um die Synchronizität zu bestimmen (definiert im Skript durch veränderbare Zeitrahmen für synchrone Aktivitäten), trennen und analysieren Sie die extrahierten Latenzen, um zufällige Ereignisse zu erkennen.
HINWEIS: Das Skript lässt die maximale Zeit zwischen zufälligen Ereignissen zu. Diese sind auf 20 ms für EAPs und 200 ms für LFPs festgelegt. - Führen Sie das Skript aus, um Latenzzeitstempel zu extrahieren.
HINWEIS: Die .xlsx Ausgabedatei enthält die Interpretationen von Latenzdaten, bei denen es sich um EAP- und LFP-Anzahl, Frequenzen und gleichzeitige Ereigniszählungen für einzelne Elektroden und ganze Slices handelt. Diese Daten werden verwendet, um die Frequenz, die EAP/LFP-Zählung, die Anzahl der aktiven Elektroden, die Anzahl der gleichzeitig auftretenden Ereignisse, die Anzahl der verknüpften Elektroden und die durchschnittliche Stärke dieser Verbindungen zu bewerten.
- Führen Sie die Datenanalyse mit einem benutzerdefinierten Programmierskript durch, das Latenzzeitstempel für jede Elektrode aus den .xlsx Dateien extrahiert.
Ergebnisse
Modell der Netzwerkaktivität im Rückenmark Rückenhorn
Die Anwendung von 4-AP induziert zuverlässig eine synchrone rhythmische Aktivität im Rückenmark DH. Solche Aktivitäten stellen sich als verstärkte EAPs und LFPs dar. Das spätere Signal ist eine niederfrequente Wellenform, die zuvor in MEA-Aufnahmen30 beschrieben wurde. Veränderungen der EAP- und/oder LFP-Aktivität nach der Arzneimittelanwendung spiegeln eine veränderte neuronale Aktivität wider. Beispiele für U...
Diskussion
Trotz der Bedeutung der spinalen DH für die nozizeptive Signalgebung, Verarbeitung und die daraus resultierenden Verhaltens- und emotionalen Reaktionen, die den Schmerz charakterisieren, bleiben die Schaltkreise in dieser Region schlecht verstanden. Eine zentrale Herausforderung bei der Untersuchung dieses Problems war die Vielfalt der Neuronenpopulationen, aus denen diese Schaltkreisebestehen 6,31,32. Jüngste Fortschritte in ...
Offenlegungen
Die Autoren haben keine Interessenkonflikte zu erklären.
Danksagungen
Diese Arbeit wurde vom National Health and Medical Research Council (NHMRC) of Australia (Zuschüsse 631000, 1043933, 1144638 und 1184974 an B.A.G. und R.J.C.) und dem Hunter Medical Research Institute (Zuschuss an B.A.G. und R.J.C.) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 4-aminopyridine | Sigma-Aldrich | 275875-5G | |
| 100% ethanol | Thermo Fisher | AJA214-2.5LPL | |
| CaCl2 1M | Banksia Scientific | 0430/1L | |
| Carbonox (Carbogen - 95% O2, 5% CO2) | Coregas | 219122 | |
| Curved long handle spring scissors | Fine Science Tools | 15015-11 | |
| Custom made air interface incubation chamber | |||
| Foetal bovine serum | Thermo Fisher | 10091130 | |
| Forceps Dumont #5 | Fine Science Tools | 11251-30 | |
| Glucose | Thermo Fisher | AJA783-500G | |
| Horse serum | Thermo Fisher | 16050130 | |
| Inverted microscope | Zeiss | Axiovert10 | |
| KCl | Thermo Fisher | AJA383-500G | |
| Ketamine | Ceva | KETALAB04 | |
| Large surgical scissors | Fine Science Tools | 14007-14 | |
| Loctite 454 Instant Adhesive | Bolts and Industrial Supplies | L4543G | |
| MATLAB | MathWorks | R2018b | |
| MEAs, 3-Dimensional | Multichannel Systems | 60-3DMEA100/12/40iR-Ti, 60-3DMEA200/12/50iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in an 8x8 square grid. Electrodes are 12 µm in diameter, 40 µm (100/12/40) or 50 µm (200/12/50) high and equidistantly spaced 100 µm (100/12/40) or 200 µm (200/12/50) apart. |
| MEA headstage | Multichannel Systems | MEA2100-HS60 | |
| MEA interface board | Multichannel Systems | MCS-IFB 3.0 Multiboot | |
| MEA net | Multichannel Systems | ALA HSG-MEA-5BD | |
| MEA perfusion system | Multichannel Systems | PPS2 | |
| MEAs, Planar | Multichannel Systems | 60MEA200/30iR-Ti, 60MEA500/30iR-Ti | 60 titanium nitride (TiN) electrodes with 1 internal reference electrode, organised in either a 8x8 square grid (200/30) or a 6x10 rectangular grid (500/30). Electrodes are 30 µm in diameter and equidistantly spaced 200 µm (200/30) or 500 µm (500/30) apart. |
| MgCl2 | Thermo Fisher | AJA296-500G | |
| Microscope camera | Motic | Moticam X Wi-Fi | |
| Multi Channel Analyser software | Multichannel Systems | V 2.17.4 | |
| Multi Channel Experimenter software | Multichannel Systems | V 2.17.4 | |
| NaCl | Thermo Fisher | AJA465-500G | |
| NaHCO3 | Thermo Fisher | AJA475-500G | |
| NaH2PO4 | Thermo Fisher | ACR207805000 | |
| Rongeurs | Fine Science Tools | 16021-14 | |
| Small spring scissors | Fine Science Tools | 91500-09 | |
| Small surgical scissors | Fine Science Tools | 14060-09 | |
| Sucrose | Thermo Fisher | AJA530-500G | |
| Superglue | cyanoacrylate adhesive | ||
| Tetrodotoxin | Abcam | AB120055 | |
| Vibration isolation table | Newport | VH3048W-OPT | |
| Vibrating microtome | Leica | VT1200 S |
Referenzen
- Smith, K. M., et al. Calretinin positive neurons form an excitatory amplifier network in the spinal cord dorsal horn. eLife. 8, 49190 (2019).
- Smith, K. M., et al. Functional heterogeneity of calretinin-expressing neurons in the mouse superficial dorsal horn: implications for spinal pain processing. The Journal of physiology. 593 (19), 4319-4339 (2015).
- Boyle, K. A., et al. Defining a spinal microcircuit that gates myelinated afferent input: Implications for tactile allodynia. Cell Reports. 28 (2), 526-540 (2019).
- Browne, T. J., et al. Transgenic cross-referencing of inhibitory and excitatory interneuron populations to dissect neuronal heterogeneity in the dorsal horn. Frontiers in Molecular Neuroscience. 13, 32 (2020).
- Graham, B. A., Hughes, D. I. Rewards, perils and pitfalls of untangling spinal pain circuits. Current Opinion in Physiology. 11, 35-41 (2019).
- Todd, A. J. Neuronal circuitry for pain processing in the dorsal horn. Nature Reviews Neuroscience. 11 (12), 823-836 (2010).
- Hughes, D. I., Todd, A. J. Central nervous system targets: inhibitory interneurons in the spinal cord. Neurotherapeutics. 17 (3), 874-885 (2020).
- Duan, B., et al. Identification of spinal circuits transmitting and gating mechanical pain. Cell. 159 (6), 1417-1432 (2014).
- Hachisuka, J., Chiang, M. C., Ross, S. E. Itch and neuropathis itch. Pain. 159 (3), 603 (2018).
- Foster, E., et al. Targeted ablation, silencing, and activation establish glycinergic dorsal horn neurons as key components of a spinal gate for pain and itch. Neuron. 85 (6), 1289-1304 (2015).
- Bourane, S., et al. Identification of a spinal circuit for light touch and fine motor control. Cell. 160 (3), 503-515 (2015).
- Cheng, L., et al. Identification of spinal circuits involved in touch-evoked dynamic mechanical pain. Nature neuroscience. 20 (6), 804-814 (2017).
- Peirs, C., et al. Mechanical allodynia circuitry in the dorsal horn is defined by the nature of the injury. Neuron. 109 (1), 73-90 (2021).
- Huang, J., et al. Circuit dissection of the role of somatostatin in itch and pain. Nature Neuroscience. 21 (5), 707-716 (2018).
- Obien, M. E. J., Deligkaris, K., Bullmann, T., Bakkum, D. J., Frey, U. Revealing neuronal function through microelectrode array recordings. Frontiers in Neuroscience. 8, 423 (2015).
- Nam, Y., Wheeler, B. C. In vitro microelectrode array technology and neural recordings. Critical Reviews in Biomedical Engineering. 39 (1), 45-61 (2011).
- Johnstone, A. F., et al. Microelectrode arrays: a physiologically based neurotoxicity testing platform for the 21st century. Neurotoxicology. 31 (4), 331-350 (2010).
- Stett, A., et al. Biological application of microelectrode arrays in drug discovery and basic research. Analytical and Bioanalytical Chemistry. 377 (3), 486-495 (2003).
- Xu, L., et al. Trends and recent development of the microelectrode arrays (MEAs). Biosensors and Bioelectronics. 175 (1), 112854 (2020).
- Chapman, R. J., Cilia La Corte, P. F., Asghar, A. U. R., King, A. E. Network-based activity induced by 4-aminopyridine in rat dorsal horn in vitro is mediated by both chemical and electrical synapses. The Journal of Physiology. 587, 2499-2510 (2009).
- Ruscheweyh, R., Sandkühler, J. Epileptiform activity in rat spinal dorsal horn in vitro has common features with neuropathic pain. Pain. 105 (1-2), 327-338 (2003).
- Kay, C. W., Ursu, D., Sher, E., King, A. E. The role of Cx36 and Cx43 in 4-aminopyridine-induced rhythmic activity in the spinal nociceptive dorsal horn: an electrophysiological study in vitro. Physiological Reports. 4 (14), 12852 (2016).
- Jankowska, E., Lundberg, A., Rudomin, P., Sykova, E. Effects of 4-aminopyridine on synaptic transmission in the cat spinal cord. Brain Research. 240 (1), 117-129 (1982).
- Semba, K., Geller, H. M., Egger, M. D. 4-Aminopyridine induces expansion of cutaneous receptive fields of dorsal horn cells. Brain Research. 343 (2), 398-402 (1985).
- Ruscheweyh, R., Sandkühler, J. Long-range oscillatory Ca2+ waves in rat spinal dorsal horn. European Journal of Neuroscience. 22 (8), 1967-1976 (2005).
- Egert, U., et al. A novel organotypic long-term culture of the rat hippocampus on substrate-integrated multielectrode arrays. Brain Research Protocols. 2 (4), 229-242 (1998).
- Thiebaud, P., De Rooij, N., Koudelka-Hep, M., Stoppini, L. Microelectrode arrays for electrophysiological monitoring of hippocampal organotypic slice cultures. IEEE Transactions on Biomedical Engineering. 44 (11), 1159-1163 (1997).
- Rey, H. G., Pedreira, C., Quiroga, R. Q. Past, present and future of spike sorting techniques. Brain Research Bulletin. 119, 106-117 (2015).
- Satuvuori, E., et al. Measures of spike train synchrony for data with multiple time scales. Journal of Neuroscience Methods. 287, 25-38 (2017).
- Mendis, G. D. C., Morrisroe, E., Reid, C. A., Halgamuge, S. K., Petrou, S. Use of local field potentials of dissociated cultures grown on multi-electrode arrays for pharmacological assays. 38th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. , 952-956 (2016).
- Hughes, D. I., et al. Morphological, neurochemical and electrophysiological features of parvalbumin-expressing cells: a likely source of axo-axonic inputs in the mouse spinal dorsal horn. The Journal of Physiology. 590 (16), 3927-3951 (2012).
- Peirs, C., Seal, R. P. Neural circuits for pain: recent advances and current views. Science. 354 (6312), 578-584 (2016).
- Li, J., Baccei, M. L. Developmental regulation of membrane excitability in rat spinal lamina I projection neurons. Journal of Neurophysiology. 107 (10), 2604-2614 (2012).
- Li, J., Baccei, M. L. Pacemaker neurons within newborn spinal pain circuits. Journal of Neuroscience. 31 (24), 9010-9022 (2011).
- Sandkühler, J., Eblen-Zajjur, A. Identification and characterization of rhythmic nociceptive and non-nociceptive spinal dorsal horn neurons in the rat. Neuroscience. 61 (4), 991-1006 (1994).
- Lucas-Romero, J., Rivera-Arconada, I., Roza, C., Lopez-Garcia, J. A. Origin and classification of spontaneous discharges in mouse superficial dorsal horn neurons. Scientific Reports. 8 (1), 9735-9735 (2018).
- Antonio, L., et al. L. al. In vitro seizure like events and changes in ionic concentration. Journal of Neuroscience Methods. 260, 33-44 (2016).
- Avoli, M., Jefferys, J. G. Models of drug-induced epileptiform synchronization in vitro. Journal of Neuroscience Methods. 260, 26-32 (2016).
- Taccola, G., Nistri, A. Low micromolar concentrations of 4-aminopyridine facilitate fictive locomotion expressed by the rat spinal cord in vitro. Neuroscience. 126 (2), 511-520 (2004).
- Mitra, P., Brownstone, R. M. An in vitro spinal cord slice preparation for recording from lumbar motoneurons of the adult mouse. Journal of Neurophysiology. 107 (2), 728-741 (2012).
- Egert, U., Heck, D., Aertsen, A. Two-dimensional monitoring of spiking networks in acute brain slices. Experimental Brain Research. 142 (2), 268-274 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten