Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Observación del fotoconducto en Chlamydomonas reinhardtii
* Estos autores han contribuido por igual
En este artículo
Resumen
La mayoría de los organismos fotoautótrofos que nadan muestran cambios de comportamiento fotoinducidos (fotocomportamiento). El presente protocolo observa dicho fotocomportamiento en el organismo modelo Chlamydomonas reinhardtii.
Resumen
Para la supervivencia de los microorganismos fotótrofos móviles, es crucial estar en condiciones de luz adecuadas. En consecuencia, muestran comportamientos fotoinducidos (o fotoconducto) y alteran su dirección de movimiento en respuesta a la luz. Los fotoconductos típicos incluyen respuesta de fotochoque (o fotofóbico) y fototaxis. El fotoshock es una respuesta a un cambio repentino en la intensidad de la luz (por ejemplo, iluminación del flash), en el que los organismos dejan de moverse transitoriamente o retroceden. Durante la fototaxis, los organismos se mueven hacia la fuente de luz o en la dirección opuesta (llamada fototaxis positiva o negativa, respectivamente). El alga verde unicelular Chlamydomonas reinhardtii es un organismo excelente para estudiar el fotoconducto porque cambia rápidamente su patrón de natación modulando el latido de los cilios (también conocidos como flagelos) después de la fotorrecepción. Aquí, se muestran varios métodos simples para observar fotocomportadores en C. reinhardtii. La investigación sobre los fotoconductos de C. reinhardtii ha llevado al descubrimiento de mecanismos reguladores comunes entre los cilios eucariotas y las canalrodopsinas, que pueden contribuir a una mejor comprensión de las ciliopatías y al desarrollo de nuevos métodos optogenéticos.
Introducción
La luz es una fuente de energía indispensable para los organismos fotosintéticos, pero demasiada luz puede causar daño foto-oxidativo. Por lo tanto, los organismos fototróficos necesitan sobrevivir bajo luz de intensidad moderada, donde pueden fotosintetizar pero no sufrir daño foto-oxidativo1. En las plantas terrestres, los cloroplastos no pueden salir de la hoja y mostrar movimientos fotográficos en la célula; Los cloroplastos se desplazan a la periferia de la célula bajo alta luz y a la superficie celular bajo poca luz2, mientras que muchas algas móviles muestran fotoconductos que les permiten encontrar condiciones de luz adecuadas para la fotosíntesis y, por lo tanto, facilitan su supervivencia3.
Chlamydomonas reinhardtii es un alga verde unicelular considerada como un organismo modelo en campos de investigación como cilios (también conocidos como flagelos), fotosíntesis y fotoconducto. C. reinhardtii se presenta con una ojera y dos cilios por célula, utilizados para la fotorrecepción y la natación, respectivamente. La mancha ocular tiene dos componentes: canalrodopsinas (ChR), canales iónicos dependientes de la luz en la membrana plasmática y las capas de gránulos ricos en carotenoides ubicadas justo detrás de los ChR. La mancha ocular actúa como un receptor de luz direccional ya que las capas de gránulos ricas en carotenoides funcionan como un reflector de luz 4,5.
Los ChR se identificaron inicialmente como fotorreceptores causantes de fotocomportamiento en C. reinhardtii 6,7,8,9. Aunque dos isoformas, ChR1 y ChR2, se encuentran en la mancha ocular, los experimentos de derribo mostraron que ChR1 es el fotorreceptor primario para los fotoconductos10. A pesar de esto, ChR2 ha recibido más atención y ha desempeñado un papel central en el desarrollo de la optogenética, una técnica para controlar la excitación celular por la luz11. Por lo tanto, el estudio de los mecanismos reguladores que gobiernan los fotoconductos en C. reinhardtii promoverá la comprensión de la función ChR y mejorará la optogenética.
Después de la fotorrecepción, las células de C. reinhardtii muestran dos tipos de fotocomportadores: fototaxis y respuesta de fotochoque12. La fototaxis es el comportamiento de las células que nadan en la dirección de la fuente de luz o en la dirección opuesta, llamada fototaxis positiva o negativa, respectivamente. La respuesta de fotochoque es un comportamiento que las células muestran después de detectar un cambio repentino en la intensidad de la luz, como cuando se ilumina con un flash. Las células dejan de nadar o nadan hacia atrás (es decir, nadando con el cuerpo celular hacia adelante) durante un corto período, generalmente <1 s.
Los movimientos ciliares en C. reinhardtii están involucrados en sus fotocomportadores. Dos cilios generalmente laten como la natación de braza de un humano, y esto se modula para los fotocomportadores. Para los fototaxis, las fuerzas generadas por los dos cilios están desequilibradas por la modulación de la frecuencia de latido y la amplitud de la forma de onda de cada cilio13. El cilio más cercano a la mancha ocular se llama cilio cis, y el otro se llama cilio trans. Estos dos cilios difieren en varios puntos. Por ejemplo, la frecuencia de latido ciliar del transcilio in vitro es 30%-40% mayor14. Además, su sensibilidad a Ca2+ es diferente. La reactivación de los modelos de células desmembranadas15 mostró que el cilio cis late con más fuerza que el transcilio para Ca2+ <1 x 10−8 M, mientras que lo contrario es cierto para Ca2+ >1 x 10−7 M. Esta asimetría en la sensibilidad de Ca2+ es posiblemente importante para los giros fototácticos, ya que los mutantes que carecen de esta asimetría no exhiben fototaxisnormales 16,17. Por el contrario, la conversión de la forma de onda es necesaria para el fotoshock. La forma de onda ciliar se transforma de la forma de onda asimétrica en la natación hacia adelante a la forma de onda simétrica en la natación hacia atrás. Esta conversión de forma de onda también está regulada por Ca2+, en un umbral de 1 x 10−4 M18,19. Dado que los defectos en la regulación de los movimientos ciliares causan discinesia ciliar primaria en humanos, el estudio de los fotoconductos en C. reinhardtii podría ayudar a comprender mejor estas enfermedades y los desarrollos terapéuticos20.
Aquí, se demuestran cuatro métodos simples para observar fotocomportadores en C. reinhardtii . En primer lugar, se muestra un ensayo de fototaxis utilizando placas de Petri, y en segundo lugar, un ensayo de fototaxis contra gotas de suspensión celular. El fenómeno observado en ambos casos no es estrictamente fototaxis sino fotoacumulación, donde las células tienden a acumularse cerca del lado de la fuente de luz o del lado opuesto. En C. reinhardtii, la acumulación de fotos es causada principalmente por fototaxis de una manera que puede usarse como una aproximación a la fototaxis. En tercer lugar, se muestra un ensayo más riguroso para la fototaxis bajo un microscopio, y por último es un ensayo de fotoshock bajo un microscopio.
Access restricted. Please log in or start a trial to view this content.
Protocolo
En el presente estudio, se utilizó una cepa de tipo salvaje de Chlamydomonas reinhardtii, una progenie del cruce CC-124 x CC-125 con agg1+mt-,21. CC-124 y CC-125 se obtuvieron del Centro de Recursos de Chlamydomonas (ver Tabla de Materiales) y se mantuvieron en un medio de tris-acetato-fosfato (TAP)22, 1,5% de agarosa a 20-25 °C. Cualquier cepa móvil se puede utilizar para este protocolo.
1. Cultivo celular
- Cultive una cepa de interés de Chlamydomonas reinhardtii en medio líquido TAP con aireación burbujeando aire estéril en un período claro-oscuro de 12 h/12 h (período claro, ~50 μmol fotones·m−2·s−1 luz blanca) a 20-25 °C durante 2 días.
NOTA: Es necesario utilizar células en una fase de crecimiento logarítmico medio. El cultivo largo (>3 días, en la fase de crecimiento logarítmico tardío) hace que las células sean menos sensibles al estímulo de luz y aumenta el número de células muertas en el cultivo, lo que dificulta la lectura de los resultados.

Figura 1: Cultivo líquido después de 2 días de cultivo. A partir de una placa de agar TAP-1.5%, un trozo de células de tipo salvaje que llenan el bucle de platino se inoculó en ~ 150 ml de medio líquido TAP en un matraz. La densidad celular después del cultivo de 2 días fue de ~5.0 x 106 células/ml. Haga clic aquí para ver una versión más grande de esta figura.
2. Pretratamiento de las células
- Mezcle ~ 10 μL del cultivo con un volumen igual de solución de deciliación para evitar que las células naden y mida la densidad celular utilizando un contador celular o un hemocitómetro.
NOTA: Hay aproximadamente 1-5 x 106 células/ml después de un cultivo de 2 días (Figura 1). La composición de la solución de deciliación es la siguiente23: 40 mM de acetato de potasio, 1 mM de CaCl2, pH 4.5 ajustado con HCl. - Centrifugar la cantidad requerida de cultivo líquido a 1000 x g durante 3 min a temperatura ambiente.
NOTA: Un experimento requiere 3 ml de 2 x 107 células / ml para condiciones estándar para el ensayo de fototaxis en una placa de Petri. Si se van a realizar dos experimentos con 1 x 106 células/ml de cultivo, es necesario centrifugar 120 ml de cultivo. - Suspenda el pellet celular con la cantidad requerida de solución experimental de fotoconducto a ~2 x 107 células/ml (para el ensayo de fototaxis en plato) o 1 x 106 células/ml (para el ensayo de fototaxis a nivel celular o el ensayo de respuesta al fotochoque) y coloque la suspensión celular en un tubo cónico.
NOTA: La densidad celular no necesita ser controlada muy estrictamente para una estimación aproximada de los fotoconductos o puede cambiar dependiendo del propósito del ensayo. Solución experimental fotoconductual14: 5 mM HEPES (pH 7.4), 0.2 mM EGTA, 1 mM KCl, 0.3 mM CaCl2. Este paso de intercambio de búfer se puede omitir para un ensayo simple sobre la capacidad de exhibir fotocomportadores, y el experimento se puede realizar con el medio de cultivo. Sin embargo, debido a que la composición iónica de la solución afecta a los signos fototácticos24, y la composición iónica del medio después del cultivo puede no ser constante, se recomienda usar esta solución para un ensayo más riguroso. La sustitución con medio TAP fresco puede ser una opción. - Coloque el tubo bajo una luz roja tenue (fotones de 10-30 μmol·m−2·s−1) durante ~1 h (Figura 2).
NOTA: Este paso aumenta la sensibilidad de las células al estímulo de luz.

Figura 2: Suspensión celular bajo luz roja. Una luz blanca fluorescente regular cubierta con una lámina de celofán rojo. Un tubo que contiene la suspensión celular se coloca bajo ~10 μmol de fotones·m−2·s−1 luz roja. Haga clic aquí para ver una versión más grande de esta figura.
3. Ensayo de fototaxis utilizando la placa de Petri (el llamado "ensayo de plato")
- Coloque 2-3 ml de suspensión celular en una placa de Petri (3,5 cm), colóquela en una caja de luz (alternativamente, una hoja de papel blanca o una placa de plástico blanco), agite suavemente para distribuir uniformemente las células y adquiera una imagen antes de la iluminación.
NOTA: El tamaño del plato puede cambiar dependiendo del propósito. Dependiendo de la cepa y el estado del cultivo, las células pueden adherirse al fondo de la placa de Petri. En tal caso, las células atascadas deben eliminarse mediante pipeteo antes del ensayo. - Ilumine el plato desde un lado con un diodo emisor de luz verde (LED, consulte tabla de materiales) en un cuarto oscuro de escritorio (Figura 3).
NOTA: Las condiciones de luz típicas son λ = 525 nm y 50-100 μmol fotones·m−2·s−1. Consulte la discusión sobre la selección de la longitud de onda de la fuente de luz en la sección de discusión. Cubra tanto los platos como el LED con una caja o tela negra cuando se use un LED pequeño. - Déjalos durante ≥5 min y luego adquiere imágenes.
NOTA: El tiempo para la iluminación de la luz se puede cambiar dependiendo del tamaño o propósito de la placa de Petri (Figura 4). - Importe el archivo de imagen a Fiji (consulte la Tabla de materiales) para su cuantificación.
NOTA: Fiji es una distribución de ImageJ2, que agrupa muchos complementos. - Cambie la imagen en color a escala de grises a través de image > Tipo > 8 bits (Figura 5).
- Invertir blanco y negro a través de Editar > Invertir.
- Rodea todo el plato como una región de interés (ROI) y mide la densidad a través de Analizar > Medir.
NOTA: La densidad del plato total se puede calcular como (Área) x (Media). - Rodea la mitad del plato más cercano a la fuente de luz como ROI y mide la densidad.
- Calcular el índice fototáctico como la (densidad de células fototácticas) por (densidad de células totales).

Figura 3: Iluminación lateral para el ensayo de platos de fototaxis. (A) Una placa de Petri que contiene una suspensión celular colocada en una caja de luz en un cuarto oscuro de escritorio. Luz verde (placa LED de 525 nm, fotones de ~100 μmol·m−2·s−1) iluminada desde un lado. (B) Método alternativo de iluminación. Un LED tipo bala de cañón de 5 mm. (C) Para bloquear la luz del exterior, se puede usar una caja con un paño negro en el interior en lugar de un cuarto oscuro de escritorio. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Ejemplo de fototaxis negativa después de 5 min de iluminación lateral. (A) Suspensión celular de tipo salvaje en una placa de Petri iluminada durante 5 min. La mayoría de las células se acumularon en el lado opuesto de la fuente de luz. Estos datos pueden interpretarse como fototaxis negativos. (B) Imagen del plato desde la parte superior. Haga clic aquí para ver una versión más grande de esta figura.
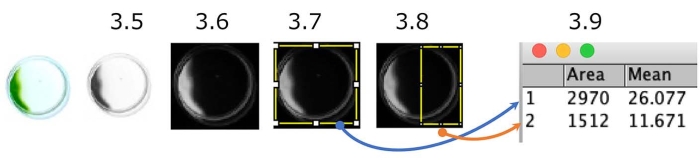
Figura 5: Cuantificación del ensayo de fototaxis del plato. Un ejemplo de células que muestran fototaxis negativas (la fuente de luz está en el lado derecho). La imagen en color se convierte en una escala de grises (paso 3.5.) y, a continuación, se invierte (paso 3.6.). Se delimitaron las regiones de interés (ROI), todo el plato (Paso 3.7.) y la mitad del plato de la fuente de luz (Paso 3.8.). Se midió la densidad de cada ROI (Paso 3.9.). En este caso, el índice fototáctico (IP) es de aproximadamente 0,18 ([1.512 x 11,671] / [2.970 x 26,077]). La IP es 1 o 0 cuando todas las células muestran fototaxis positiva o negativa, respectivamente. Haga clic aquí para ver una versión más grande de esta figura.
4. Ensayo de fototaxis utilizando gotitas de cultivo celular
- Coloque 25 μL de gotas de suspensión celular (Paso 2.4.) directamente sobre una placa de plástico blanco con una micropipeta.
- Ilumine las gotas de un lado con un LED verde en un cuarto oscuro de escritorio (Figura 6).
NOTA: Las condiciones de luz típicas son λ = 525 nm y 50-100 μmol fotones m−2 s−1. Cubra ambas placas y LED con una caja o un paño negro cuando se use un LED pequeño. - Déjalos durante 3 min y luego adquiere imágenes.
NOTA: Este ensayo es adecuado para una verificación rápida de la fototaxis de muchas muestras a la vez, como en el cribado de mutantes o el análisis de tétradas. Un cultivo celular en la fase logarítmica media cultivada en una placa de 96 pocillos se puede iluminar directamente para facilitar el rendimiento. En ambos casos, se puede omitir el Paso 2 (pretratamiento celular).

Figura 6: Ensayo de fototaxis de gotas. (A) Nueve gotas de una suspensión celular de 25 μL colocadas sobre una lámina de plástico blanco e iluminadas desde un lado por un LED verde. (B) Después de 3 minutos de iluminación. En cada gota, las células se acumularon en el lado de la fuente de luz (fototaxis positivas), se acumularon en el lado opuesto (fototaxis negativas) o se difundieron en la gota (sin fototaxis). Barra de escala = 1 cm. Haga clic aquí para ver una versión más grande de esta figura.
5. Ensayo de fototaxis bajo un microscopio
- Tome ~ 30 μL de suspensión celular (1 x 106 celdas / ml en solución experimental de fotoconducto) en un portaobjetos de vidrio y coloque un cobertor (18 mm x 18 mm) con espaciadores en la parte superior (Figura 7).
NOTA: Los espaciadores se pueden hacer con petróleo blanco o cintas adhesivas de doble cara en dos lados opuestos de un cobertor. La luz debe provenir de una dirección donde no haya espaciadores. - Ilumine la muestra desde un lado de la cubierta sin un espaciador con un LED verde y observe las células bajo un microscopio de campo oscuro con una lente de objetivo 10x bajo luz roja tenue (λ > 630 nm, ~5 μmol fotones·m−2·s−1, Figura 8).
NOTA: Evite exponer las celdas en el escenario a una luz que no sea LED o luz de observación, como la iluminación de la habitación o la luz de un monitor de PC. La ampliación adecuada de un objetivo depende del ángulo de visión de la cámara. Seleccione una lente con un aumento que permita que la celda nade durante ~ 2 s sin salir del campo de visión. Para rastrear celdas, es deseable una imagen de alto contraste; por lo tanto, se recomienda el uso de un condensador de campo oscuro para la observación microscópica. Sin embargo, otros condensadores, como el campo brillante, se pueden usar para observar fototaxis. Asegúrese de que la luz del LED brille en las celdas. - Grabe el movimiento de la célula durante ~ 20 s después de la iluminación de la luz utilizando un microscopio equipado con cámara.
NOTA: Unos segundos después del inicio de la iluminación LED verde, puede ocurrir una respuesta de fotoamortiguamiento y / o la orientación fototáctica (o signo) no es estable; por lo tanto, se recomienda una grabación de 20 s para la estabilización de la respuesta (Película 1).

Figura 7: Hacer espaciadores en los bordes de las cubiertas. (A) Se aplicó una capa delgada de vaselina a la palma de una mano. Una pequeña cantidad de petróleo blanco fue raspada con el borde de una funda. (B) Un espaciador en el borde de un coverslip. (C) Otro espaciador en el borde opuesto. Haga clic aquí para ver una versión más grande de esta figura.

Figura 8: Iluminación lateral bajo un microscopio. (A) Configuración de un LED verde. Un LED verde tipo bala de cañón se fija a la manguito y se fija al soporte junto al microscopio. Las células se observaron bajo un microscopio de campo oscuro con un filtro de corte afilado (λ > 630 nm). (B) Iluminación lateral por el LED verde. Haga clic aquí para ver una versión más grande de esta figura.
Película 1: Ensayo de fototaxis bajo un microscopio. Luz verde iluminada a ~0 s desde la derecha. En ese momento, las células tendían a nadar en una dirección aleatoria. Después de 0 s en el contador de tiempo, las células nadaron hacia la derecha o hacia la izquierda, mostrando fototaxis positivas o negativas. La luz se apagó a ~ 15 s cuando las células comenzaron a nadar en una dirección aleatoria nuevamente. Barra de escala = 100 μm. Haga clic aquí para descargar esta película.
6. Seguimiento de células fototácticas y dibujo de histograma polar
- Extraiga 1.5 s del video grabado, comenzando 15 s después de la iluminación.
NOTA: La duración de la trayectoria puede cambiar dependiendo de la velocidad de natación de la célula. Un video de 1.5 s contiene 46 cuadros a 30 fps de grabación. - Importe el archivo a Fiji como una secuencia de imágenes de formato de archivo de imagen de etiqueta a través de la secuencia de > de imagen de > Importar >.
- Ejecute el complemento "Seguimiento manual" para el seguimiento celular utilizando Complementos > Seguimiento > Seguimiento manual.
- Haga clic en Agregar pista y aparecerá el primer fotograma.
- Haga clic en una celda de interés y aparecerá el segundo fotograma.
- Mueva el control deslizante al último fotograma y haga clic en la misma celda que en el paso 6.5.
NOTA: No es necesario realizar un seguimiento de las celdas en cada fotograma. Solo el ángulo entre el eje de luz y el segmento de línea formado por el inicio y los puntos finales es esencial. - Repita los pasos 6.4.-6.6. para ~30 celdas.
NOTA: Seleccione las celdas que permanecen dentro del ángulo de visión durante todo el 1,5 s. Si la densidad celular es lo suficientemente baja y ninguna trayectoria de natación celular se cruza durante 1,5 s, se puede realizar un seguimiento automático. En ese caso, elija el complemento "MTrack2" (configuración típica: Tamaño mínimo del objeto = 1; Tamaño máximo del objeto = 10.000; Velocidad máxima = 100; Longitud mínima de la tachuela [Fotogramas] = 46). Se recomienda verificar la correspondencia entre las celdas del vídeo y los resultados del seguimiento mediante la obtención de los datos marcando ¿Mostrar texto? > Superponer puntos y líneas. - Copie y pegue los resultados en el software de hoja de cálculo (por ejemplo, Excel).
NOTA: Aquí, se muestra cómo dibujar un histograma polar con Excel. - Mida el ángulo entre el eje de luz y la dirección de natación de una celda por "=grados(atan2(x2-x1),-(y2-y1))", donde la posición inicial de la celda (corte #1) es (x1, y1), y la última posición (corte #46) es (x2, y1).
NOTA: En una imagen de Fiji, la esquina superior izquierda es el origen (0, 0). - Repita el paso 6.9. para todas las celdas medidas (típicamente ~30 celdas).
- Prepare una tabla de distribución de frecuencias para todos los datos obtenidos con los contenedores de -180° a +180°, divididos en 15° en ambos extremos y 30° para el resto del rango utilizando la función "FRECUENCIA".
NOTA: Después de seleccionar las columnas inmediatamente en las columnas de la papelera, ingrese lo siguiente en la columna superior = FRECUENCIA (data_array, bins_array) y presione mayús + ctrl + entrar (Figura suplementaria 1). - Vuelva a crear una tabla de distribución de frecuencias con los valores calculados en el paso 6.8. giró −90° para establecer el lado derecho a 0° (es decir, convertir el número en el rango de −15° a 15°, al rango de −105° a −75°), ya que, con el método anterior, se considera que las células que nadan hacia arriba nadan en un ángulo de 0° con respecto a la luz (es decir, muestran fototaxis positivos), aunque la luz proviene de la derecha (Figura suplementaria 2).
- Introduzca el valor de ángulo en el centro del rango de un contenedor de 30° (por ejemplo, 30° para 15°-45°), omitiendo una columna, e introduzca el recuento de muestras correspondiente a su derecha (Figura suplementaria 3).
- Convierta cada valor de recuento de muestras en un porcentaje e introduzca 0 en la columna correspondiente a la columna en blanco entre contenedores.
- Dibuje un gráfico de radar utilizando los valores de ángulo como etiquetas de eje horizontal y los valores percentuales (%) como leyenda (Figura suplementaria 4).
- Calcule el índice fototáctico (PI) para una mayor cuantificación promediando el cosθ (θ = el ángulo entre el eje de luz y la dirección de natación)16.
NOTA: PI es 0 cuando las células nadan en una dirección aleatoria y 1 o −1 cuando el 100% de las células muestran fototaxis positivas o negativas, respectivamente.
7. Ensayo de respuesta de fotochoque bajo un microscopio
- Coloque ~ 30 μL de suspensión celular (1 x 106 celdas / ml en la solución experimental de fotoconducto) en un portaobjetos de vidrio y coloque un capaplete (18 mm x 18 mm) con espaciadores en la parte superior, como en el Paso 4.1.
- Observe las células bajo un microscopio con luz roja tenue (λ > 630 nm, ~5 μmol fotones·m−2·s−1).
- Aplique la iluminación del flash con el flash de una cámara (película 2,3).
NOTA: Otra forma de inducir una respuesta de fotoamortiguamiento es eliminar rápidamente un filtro rojo a mano de la trayectoria de la luz de observación. Sin embargo, este método es más variable ya que la velocidad de eliminación del filtro varía de persona a persona (Película 4,5). - Registre el movimiento celular con un microscopio equipado con cámara.
- Para la cuantificación, realice uno o ambos de los siguientes: (1) calcule la proporción de células que muestran la respuesta de fotochoque por células totales16; (2) para las células que muestran la respuesta al fotoshock, mida el tiempo desde el estímulo del fotoshock hasta la recuperación de la natación hacia adelante25.
Película 2: Iluminación de fotoshock por el flash de una cámara. El flash de la cámara se mantuvo hasta el escenario del microscopio y se encendió. Haga clic aquí para descargar esta película.
Película 3: Respuesta de fotoamortiguamiento causada por un destello bajo un microscopio. Las células se observaron bajo una luz roja tenue. Se emitió un destello a ~0 s. Casi todas las células dejaron de nadar hacia adelante, nadaron hacia atrás durante un corto período y se recuperaron nadando hacia adelante. Barra de escala = 100 μm. Haga clic aquí para descargar esta película.
Película 4: Respuesta de fotoamortiguamiento causada por la eliminación de un filtro rojo bajo un microscopio. Las células se observaron bajo una luz roja tenue. El filtro rojo se eliminó a ~ 5 s. Casi todas las células dejaron de nadar hacia adelante, nadaron hacia atrás durante un corto período y se recuperaron nadando hacia adelante. Barra de escala = 100 μm. Haga clic aquí para descargar esta película.
Película 5: Eliminación de un filtro rojo. Eliminación rápida de un filtro rojo establecido en la trayectoria de la luz para entregar fotoamortiguamiento. Haga clic aquí para descargar esta película.
Access restricted. Please log in or start a trial to view this content.
Resultados
Aquí se muestran los ensayos típicos de respuesta de fototaxis y fotochoque de C. reinhardtii . Después de la estimación de la densidad celular, el cultivo celular de tipo salvaje (una progenie de la cruz CC-124 × CC-125 con agg1+ mt -)23 se lavó con una solución experimental de fotoconducto para el ensayo de plato de fototaxis. La suspensión celular se colocó bajo una luz roja tenue durante ~ 1 h. Se colocó una suspensión celular de 2 ml en una placa de Petri de 3,5 cm. La pla...
Access restricted. Please log in or start a trial to view this content.
Discusión
El protocolo actual es fácil y no requiere mucho tiempo. Si se sospecha que un mutante de C. reinhardtii presenta defectos en la fotorrecepción o el movimiento ciliar, este método podría servir como análisis fenotípico primario.
Sin embargo, existen algunos pasos críticos. Una es usar células en el experimento en la fase de crecimiento temprano a medio log. Después del cultivo durante largos períodos, las células se vuelven menos móviles, menos sensibles a la luz e incluso...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este estudio fue apoyado por subvenciones de la Sociedad Japonesa para la Promoción de la Ciencia KAKENHI (https://www.jsps.go.jp/english/index.html) a NU (19K23758, 21K06295), TH (16H06556) y KW (19H03242, 20K21420, 21H00420), de la Ohsumi Frontier Science Foundation (https://www.ofsf.or.jp/en/) a KW, y de la Alianza Dinámica para la Innovación Abierta Uniendo Humano, Medio Ambiente y Materiales (http://alliance.tagen.tohoku.ac.jp/english/) a NU, TH y KW.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SARSTEDT | 62.554.502 | |
| 5 mm Cannonball green LED | Optosupply | OSPG5161P | |
| 50 mL conical tube | SARSTEDT | 62.547.254 | |
| AC adaptor for the light box | ATTO | 2196161 | |
| Auto cell counter | DeNovix | CellDrop BF | |
| CaCl2 | Nakalai tesque | 06731-05 | |
| Camera flash | NEWWER | TT560 | |
| Centrifuge | KUBOTA | 2800 | |
| Chlamydomonas strains CC-124 and CC-125 | Chlamydomonas Resource Center | https://www.chlamycollection.org/ | |
| C-mout CCD camera | Wraymer | 1129HMN1/3 | |
| Desktop darkroom | Scientex | B-S8 | |
| Digital still camera | SONY | RX100II | |
| EGTA | Dojindo | G002 | |
| Fiji | https://fiji.sc/ | ||
| Green LED plate | CCS | ISLM-150X150-GG | |
| HCl | Fujifilm WAKO | 080-01066 | |
| HEPES | Dojindo | GB70 | |
| KCl | Nakalai tesque | 238514-75 | |
| Lightbox (Flat viewer) | ATTO | 2196160 | |
| Microscope | Olympus | BX-53 | |
| Petri dish (φ3.5 cm) | IWAKI | 1000-035 | |
| Pottasium acetate | Nakalai tesque | 28434-25 | |
| Power supply for the green LED plate | CCS | ISC-201-2 | |
| Red filter | Shibuya Optical | S-RG630 |
Referencias
- Demmig-Adams, B., Adams, W. W. Photoprotection and other responses of plants to high light stress. Annual Reviews Plant Physiology and Plant Molecular Biology. 43, 599-626 (1992).
- Wada, M. Chloroplast movement. Plant Science. 210, 177-182 (2013).
- Sgarbossa, A., Checcucci, G., Lenci, F. Photoreception and photomovements of microorganisms. Photochemical & Photobiological Sciences. 1 (7), 459-467 (2002).
- Ueki, N., et al. Eyespot-dependent determination of the phototactic sign in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 113 (19), 5299-5304 (2016).
- Foster, K. W., Smyth, R. D. Light antennas in phototactic algae. Microbiological Reviews. 44 (4), 572-630 (1980).
- Nagel, G., et al. Channelrhodopsin-1: a light-gated proton channel in green algae. Science. 296 (5577), 2395-2398 (2002).
- Nagel, G., et al. Channelrhodopsin-2, a directly light-gated cation-selective membrane channel. Proceedings of the National Academy of Sciences of the United States of America. 100 (24), 13940-13945 (2003).
- Sineshchekov, O. A., Jung, K. -H., Spudich, J. L. Two rhodopsins mediate phototaxis to low- and high-intensity light in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 99 (13), 8689-8694 (2002).
- Suzuki, T., et al. Archaeal-type rhodopsins in Chlamydomonas: model structure and intracellular localization. Biochemical and Biophysical Research Communications. 301 (3), 711-717 (2003).
- Berthold, P., et al. Channelrhodopsin-1 initiates phototaxis and photophobic responses in Chlamydomonas by immediate light-induced depolarization. Plant Cell. 20 (6), 1665-1677 (2008).
- Deisseroth, K., et al. Next-generation optical technologies for illuminating genetically targeted brain circuits. Journal of Neuroscience. 26 (41), 10380-10386 (2006).
- Wakabayashi, K., Isu, A., Ueki, N. Channelrhodopsin-dependent photo-behavioral responses in the unicellular green alga Chlamydomonas reinhardtii. Optogenetics (Advances in Experimental Medicine and Biology), 2nd ed. , Springer. 21-33 (2021).
- Rüffer, U., Nultsch, W. Flagellar photoresponses of Chlamydomonas cells held on micropipettes: II. Change in flagellar beat pattern. Cell Motility and the Cytoskeleton. 18 (4), 269-278 (1991).
- Kamiya, R., Hasegawa, E. Intrinsic difference in beat frequency between the two flagella of Chlamydomonas reinhardtii. Experimental Cell Research. 173, 299-304 (1987).
- Kamiya, R., Witman, G. B. Submicromolar levels of calcium control the balance of beating between the two flagella in demembranated models of Chlamydomonas. Journal of Cell Biology. 98 (1), 97-107 (1984).
- Okita, N., Isogai, N., Hirono, M., Kamiya, R., Yoshimura, K. Phototactic activity in Chlamydomonas 'non-phototactic' mutants deficient in Ca2+-dependent control of flagellar dominance or in inner-arm dynein. Journal of Cell Science. 118, 529-537 (2005).
- Horst, C. J., Witman, G. B. ptx1, a nonphototactic mutant of Chlamydomonas, lacks control of flagellar dominance. Journal of Cell Biology. 120 (3), 733-741 (1993).
- Hyams, J. S., Borisy, G. G. Isolated flagellar apparatus of Chlamydomonas: characterization of forward swimming and alteration of waveform and reversal of motion by calcium ions in vitro. Journal of Cell Science. 33, 235-253 (1978).
- Bessen, M., Fay, R. B., Witman, G. B. Calcium control of waveform in isolated flagellar axonemes of Chlamydomonas. Journal of Cell Biology. 86 (2), 446-455 (1980).
- Reiter, J. F., Leroux, M. R. Genes and molecular pathways underpinning ciliopathies. Nature Reviews Molecular Cell Biology. 18 (9), 533-547 (2017).
- Ide, T., et al. Identification of the agg1 mutation responsible for negative phototaxis in a "wild-type" strain of Chlamydomonas reinhardtii. Biochemistry and Biophysics Reports. 7, 379-385 (2016).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Finst, R. J., Kim, P. J., Quarmby, L. M. Genetics of the deflagellation pathway in Chlamydomonas. Genetics. 149 (2), 927-936 (1998).
- Morel-Laurens, N. Calcium control of phototactic orientation in Chlamydomonas reinhardtii: sign and strength of response. Photochemistry and Photobiology. 45 (1), 119-128 (1987).
- Wakabayashi, K., King, S. M. Modulation of Chlamydomonas reinhardtii flagellar motility by redox poise. Journal of Cell Biology. 173 (5), 743-754 (2006).
- Wakabayashi, K., Misawa, Y., Mochiji, S., Kamiya, R. Reduction-oxidation poise regulates the sign of phototaxis in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11280-11284 (2011).
- Harris, E. H. in The Chlamydomonas Sourcebook Second Edition. 1, Academic Press. Ch. 2 25-64 (2009).
- Mergenhagen, D. Circadian clock: genetic characterization of a short period mutant of Chlamydomonas reinhardii. European Journal of Cell Biology. 33 (1), 13-18 (1984).
- Ozasa, K., Lee, J., Song, S., Hara, M., Maeda, M. Two-dimensional optical feedback control of Euglena confined in closed-type microfluidic channels. Lab on a Chip. 11 (11), 1933-1940 (2011).
- Tanno, A., et al. The four-celled Volvocales green alga Tetrabaena socialis exhibits weak photobehavior and high-photoprotection ability. PLoS One. 16 (10), 0259138(2021).
- Ueno, Y., Aikawa, S., Kondo, A., Akimoto, S. Adaptation of light-harvesting functions of unicellular green algae to different light qualities. Photosynthesis Research. 139 (1-3), 145-154 (2019).
- Takahashi, T., Watanabe, M. Photosynthesis modulates the sign of phototaxis of wild-type Chlamydomonas reinhardtii. Effects of red background illumination and 3-(3',4'-dichlorophenyl)-1,1-dimethylurea. FEBS Letters. 336 (3), 516-520 (1993).
- Morishita, J., Tokutsu, R., Minagawa, J., Hisabori, T., Wakabayashi, K. I. Characterization of Chlamydomonas reinhardtii mutants that exhibit strong positive phototaxis. Plants (Basel). 10 (7), (2021).
- Fujiu, K., Nakayama, Y., Yanagisawa, A., Sokabe, M., Yoshimura, K. Chlamydomonas CAV2 encodes a voltage- dependent calcium channel required for the flagellar waveform conversion. Current Biology. 19 (2), 133-139 (2009).
- Inaba, K. Calcium sensors of ciliary outer arm dynein: functions and phylogenetic considerations for eukaryotic evolution. Cilia. 4 (1), 6(2015).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados