Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Beobachtung des Photoverhaltens bei Chlamydomonas reinhardtii
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Die meisten schwimmenden photoautotrophen Organismen zeigen photoinduzierte Verhaltensänderungen (Photobehavior). Das vorliegende Protokoll beobachtet das besagte Photoverhalten im Modellorganismus Chlamydomonas reinhardtii.
Zusammenfassung
Für das Überleben der beweglichen phototrophen Mikroorganismen ist es entscheidend, unter den richtigen Lichtbedingungen zu stehen. Folglich zeigen sie photoinduziertes Verhalten (oder Fotoverhalten) und ändern ihre Bewegungsrichtung als Reaktion auf Licht. Typische Photoverhaltensweisen sind Photoshock (oder photophobe) Reaktion und Phototaxis. Photoschock ist eine Reaktion auf eine plötzliche Änderung der Lichtintensität (z. B. Blitzbeleuchtung), bei der Organismen vorübergehend aufhören, sich zu bewegen oder sich rückwärts zu bewegen. Während der Phototaxis bewegen sich Organismen in Richtung der Lichtquelle oder in die entgegengesetzte Richtung (positive bzw. negative Phototaxis genannt). Die einzellige Grünalge Chlamydomonas reinhardtii ist ein ausgezeichneter Organismus, um das Photoverhalten zu untersuchen, da sie ihr Schwimmmuster schnell ändert, indem sie das Schlagen von Flimmerchen (auch bekannt als Flagellen) nach der Photorezeption moduliert. Hier werden verschiedene einfache Methoden zur Beobachtung des Photoverhaltens bei C. reinhardtii gezeigt. Die Erforschung des Photoverhaltens von C. reinhardtii hat zur Entdeckung gemeinsamer Regulationsmechanismen zwischen eukaryotischen Zilien und Kanalrhodopsinen geführt, die zu einem besseren Verständnis von Ziliopathien und zur Entwicklung neuer optogenetischer Methoden beitragen können.
Einleitung
Licht ist eine unverzichtbare Energiequelle für photosynthetische Organismen, aber zu viel Licht kann photooxidative Schäden verursachen. Daher müssen phototrophe Organismen unter Licht mittlerer Intensität überleben, wo sie Photosynthese betreiben können, aber keinen photooxidativen Schaden erleiden1. Bei Landpflanzen können sich Chloroplasten nicht aus dem Blatt bewegen und zeigen Fotobewegungen in der Zelle; Chloroplasten bewegen sich unter hohem Licht an die Peripherie der Zelle und bei schwachem Licht an die Zelloberfläche2, während viele bewegliche Algen Photoverhalten zeigen, das es ihnen ermöglicht, die richtigen Lichtbedingungen für die Photosynthese zu finden und so ihr Überleben zu erleichtern3.
Chlamydomonas reinhardtii ist eine einzellige Grünalge, die als Modellorganismus in Forschungsbereichen wie Zilien (auch bekannt als Flagellen), Photosynthese und Photoverhalten gilt. C. reinhardtii präsentiert sich mit einem Augenfleck und zwei Zilien pro Zelle, die für den Fotoempfang bzw. das Schwimmen verwendet werden. Der Augenfleck besteht aus zwei Komponenten: Kanalrhodopsinen (ChRs), lichtgesteuerten Ionenkanälen in der Plasmamembran und den Carotinoid-reichen Granulatschichten, die sich direkt hinter den ChRs befinden. Der Augenfleck fungiert als gerichteter Lichtrezeptor, da die Carotinoid-reichen Granulatschichten als Lichtreflektorfungieren 4,5.
ChRs wurden ursprünglich als Photorezeptoren identifiziert, die Photoverhalten in C. reinhardtii 6,7,8,9 verursachen. Obwohl zwei Isoformen, ChR1 und ChR2, im Augenfleck gefunden werden, zeigten Knock-Down-Experimente, dass ChR1 der primäre Photorezeptor für Photobehaviors10 ist. Trotzdem hat ChR2 mehr Aufmerksamkeit erhalten und spielte eine zentrale Rolle bei der Entwicklung der Optogenetik, einer Technik zur Kontrolle der Zellanregung durch Licht11. Daher wird die Untersuchung der regulatorischen Mechanismen, die das Photoverhalten bei C. reinhardtii steuern, das Verständnis der ChR-Funktion fördern und die Optogenetik verbessern.
Nach der Photorezeption zeigen C. reinhardtii-Zellen zwei Arten von Photoverhalten: Phototaxis und Photoschockantwort12. Phototaxis ist das Verhalten von Zellen, die in Richtung der Lichtquelle oder in die entgegengesetzte Richtung schwimmen, was als positive bzw. negative Phototaxis bezeichnet wird. Die Photoschockreaktion ist ein Verhalten, das Zellen zeigen, nachdem sie eine plötzliche Änderung der Lichtintensität wahrgenommen haben, z. B. wenn sie von einem Blitz beleuchtet werden. Zellen hören für kurze Zeit auf zu schwimmen oder schwimmen rückwärts (d.h. mit dem Zellkörper nach vorne schwimmen), typischerweise <1 s.
Ziliarbewegungen bei C. reinhardtii sind an seinem Photoverhalten beteiligt. Zwei Zilien schlagen normalerweise wie das Brustschwimmen eines Menschen, und dies wird für Photobehaviors moduliert. Bei Phototaxis sind die von den beiden Zilien erzeugten Kräfte durch die Modulation der Schlagfrequenz und der Wellenformamplitude jedesZiliums 13 ausgeglichen. Das Zilium, das dem Augenfleck am nächsten ist, heißt cis-cilium und das andere wird trans cilium genannt. Diese beiden Zilien unterscheiden sich in verschiedenen Punkten. Zum Beispiel ist die Ziliarschlagfrequenz von Transcilium in vitro 30% -40% höher14. Darüber hinaus ist ihre Ca2 + -Empfindlichkeit unterschiedlich. Die Reaktivierung demembranierter Zellmodelle15 zeigte, dass das cis-Zilium für Ca 2+ <1 x 10−8 M stärker schlägt als das trans cilium, während das Gegenteil für Ca2+ >1 x 10−7 M gilt. Diese Asymmetrie in Ca2+ Empfindlichkeit ist möglicherweise wichtig für phototaktische Drehungen, da Mutanten, denen diese Asymmetrie fehlt, keine normalen Phototaxis16,17 aufweisen. Umgekehrt ist die Wellenformkonvertierung für den Fotoschock notwendig. Die Ziliarwellenform wandelt sich von der asymmetrischen Wellenform im Vorwärtsschwimmen in die symmetrische Wellenform im Rückwärtsschwimmen um. Diese Wellenformkonvertierung wird auch durch Ca2+ reguliert, bei einem Schwellenwert von 1 x 10−4 M18,19. Da Defekte bei der Regulierung der Ziliarbewegungen beim Menschen eine primäre ziliäre Dyskinesie verursachen, könnte die Untersuchung des Photoverhaltens bei C. reinhardtii zu einem besseren Verständnis dieser Krankheiten und therapeutischen Entwicklungenbeitragen 20.
Hierin werden vier einfache Methoden zur Beobachtung des Photoverhaltens bei C. reinhardtii demonstriert. Erstens wird ein Phototaxis-Assay mit Petrischalen und zweitens ein Phototaxis-Assay gegen Zellsuspensionströpfchen gezeigt. Das in beiden Fällen beobachtete Phänomen ist nicht ausschließlich Phototaxis, sondern Photoakkumulation, bei der sich die Zellen in der Nähe der Lichtquellenseite oder der gegenüberliegenden Seite ansammeln. Bei C. reinhardtii wird die Photoakkumulation hauptsächlich durch Phototaxis in einer Weise verursacht, die als Annäherung an Phototaxis verwendet werden kann. Drittens wird ein strengerer Assay für Phototaxis unter einem Mikroskop gezeigt, und zuletzt ist ein Photoschock-Assay unter einem Mikroskop.
Access restricted. Please log in or start a trial to view this content.
Protokoll
In der vorliegenden Studie wurde ein Wildtypstamm von Chlamydomonas reinhardtii, einem Nachkommen des Kreuzes CC-124 x CC-125 mit agg1+mt-,21 verwendet. CC-124 und CC-125 wurden aus dem Chlamydomonas Resource Center gewonnen (siehe Materialtabelle) und auf einem Tris-Acetat-Phosphat (TAP)22, 1,5% Agarosemedium bei 20-25 °C gehalten. Jede bewegliche Dehnung kann für dieses Protokoll verwendet werden.
1. Zellkultur
- Kultur einer interessierenden Sorte von Chlamydomonas reinhardtii in TAP-flüssigem Medium mit Belüftung durch sprudelnde sterile Luft in einer 12 h/12 h Hell-Dunkel-Periode (Lichtperiode, ~50 μmol Photonen·m−2·s−1 weißes Licht) bei 20-25 °C für 2 Tage.
HINWEIS: Zellen in einer mittleren logarithmischen Wachstumsphase müssen verwendet werden. Lange Kultur (>3 Tage, in der spätlogarithmischen Wachstumsphase) macht die Zellen weniger empfindlich gegenüber dem Lichtreiz und erhöht die Anzahl der toten Zellen in der Kultur, was das Auslesen der Ergebnisse behindert.

Abbildung 1: Flüssigkultur nach 2-tägiger Kultivierung. Aus einer TAP-1,5% Agarplatte wurde ein Stück Wildtypzellen, die die Platinschleife füllten, in einem Kolben in ~ 150 ml flüssiges TAP-Medium eingeimpft. Die Zelldichte nach 2-tägiger Kultur betrug ~5,0 x 106 Zellen/ml. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
2. Vorbehandlung von Zellen
- Mischen Sie ~ 10 μL der Kultur mit einem gleichen Volumen an Dezitilisierungslösung, um das Schwimmen der Zellen zu stoppen, und messen Sie die Zelldichte mit einem Zellzähler oder Hämozytometer.
HINWEIS: Es gibt ungefähr 1-5 x 106 Zellen / ml nach einer 2-tägigen Kultur (Abbildung 1). Die Zusammensetzung der Dezisiliationslösung ist wie folgt23: 40 mM Kaliumacetat, 1 mM CaCl2, pH 4,5 mit HCl eingestellt. - Zentrifen Sie die erforderliche Menge an Flüssigkultur bei 1000 x g für 3 min bei Raumtemperatur.
HINWEIS: Ein Experiment erfordert 3 ml von 2 x 107 Zellen / ml für Standardbedingungen für den Phototaxis-Assay in einer Petrischale. Wenn zwei Experimente mit 1 x 106 Zellen / ml Kultur durchgeführt werden sollen, müssen 120 ml Kultur zentrifugiert werden. - Suspendieren Sie das Zellpellet mit der erforderlichen Menge an experimenteller Photobehavior-Lösung auf ~ 2 x 107 Zellen/ml (für den Dish-Phototaxis-Assay) oder 1 x 106 Zellen/ml (für den Phototaxis-Assay auf Zellebene oder den Photoshock-Response-Assay) und legen Sie die Zellsuspension in einen konischen Schlauch.
HINWEIS: Die Zelldichte muss für eine grobe Abschätzung des Photoverhaltens nicht sehr streng kontrolliert werden oder kann sich je nach Zweck des Assays ändern. Photobehavior-Versuchslösung14: 5 mM HEPES (pH 7,4), 0,2 mM EGTA, 1 mM KCl, 0,3 mM CaCl2. Dieser Pufferaustauschschritt kann für einen einfachen Assay über die Fähigkeit, Photoverhalten zu zeigen, weggelassen werden, und das Experiment kann mit dem Kulturmedium durchgeführt werden. Da jedoch die ionische Zusammensetzung der Lösung die phototaktischen Zeichen24 beeinflusst und die ionische Zusammensetzung des Mediums nach der Kultivierung möglicherweise nicht konstant ist, wird die Verwendung dieser Lösung für einen strengeren Assay empfohlen. Die Substitution durch frisches TAP-Medium kann eine Option sein. - Stellen Sie die Röhre für ~1 h unter schwaches rotes Licht (10-30 μmol Photonen·m−2·s−1) (Abbildung 2).
HINWEIS: Dieser Schritt erhöht die Empfindlichkeit der Zellen gegenüber dem Lichtreiz.

Abbildung 2: Zellsuspension bei rotem Licht. Ein normales fluoreszierendes weißes Licht, das mit einer roten Zellophanschicht bedeckt ist. Ein Röhrchen, das die Zellsuspension enthält, wird unter ~10 μmol Photonen·m−2·s−1 rotem Licht platziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
3. Phototaxis Assay mit der Petrischale (sog. "Dish Assay")
- Legen Sie 2-3 ml Zellsuspension in eine Petrischale (3,5 cm), legen Sie sie auf einen Lampenkasten (alternativ ein weißes Blatt Papier oder eine weiße Plastikplatte), schütteln Sie sie vorsichtig, um die Zellen gleichmäßig zu verteilen, und erhalten Sie vor der Beleuchtung ein Bild.
HINWEIS: Die Schalengröße kann sich je nach Verwendungszweck ändern. Abhängig von der Belastung und dem Zustand der Kultur können die Zellen am Boden der Petrischale haften. In einem solchen Fall müssen die festsitzenden Zellen vor dem Assay durch Pipettieren entfernt werden. - Beleuchten Sie die Schüssel von einer Seite mit einer grünen Leuchtdiode (LED, siehe Materialtabelle) in einer Desktop-Dunkelkammer (Abbildung 3).
HINWEIS: Typische Lichtverhältnisse sind λ = 525 nm und 50-100 μmol Photonen·m−2·s−1. Siehe die Diskussion zur Auswahl der Wellenlänge der Lichtquelle im Diskussionsabschnitt. Decken Sie sowohl das Geschirr als auch die LED mit einer Box oder einem schwarzen Tuch ab, wenn eine kleine LED verwendet wird. - Lassen Sie sie für ≥5 Minuten und nehmen Sie dann Bilder auf.
HINWEIS: Die Zeit für die Lichtbeleuchtung kann je nach Größe oder Zweck der Petrischale geändert werden (Abbildung 4). - Importieren Sie die Bilddatei zur Quantifizierung nach Fidschi (siehe Materialtabelle).
HINWEIS: Fidschi ist eine Distribution von ImageJ2, die viele Plugins bündelt. - Ändern Sie das Farbbild über Bild > Geben Sie > 8-Bit in Graustufen ein (Abbildung 5).
- Kehren Sie Schwarz-Weiß durch Bearbeiten > Umkehren um.
- Umgeben Sie das gesamte Gericht als Region of Interest (ROI) und messen Sie die Dichte durch Analyze > Measure.
HINWEIS: Die Dichte der Gesamtschale kann als (Fläche) x (Mittelwert) berechnet werden. - Umgeben Sie die Hälfte der Schale, die der Lichtquelle am nächsten ist, als ROI und messen Sie die Dichte.
- Berechnen Sie den phototaktischen Index als (Dichte der phototaktischen Zellen) pro (Dichte der Gesamtzellen).

Abbildung 3: Seitenbeleuchtung für den Phototaxis-Schalen-Assay . (A) Eine Petrischale mit einer Zellaufhängung, die auf einem Leuchtkasten in einer Desktop-Dunkelkammer platziert ist. Grünes Licht (525 nm LED-Platte, ~100 μmol Photonen·m−2·s−1) von der Seite beleuchtet. (B) Alternative Beleuchtungsmethode. Eine 5-mm-Cannonball-LED. (C) Um Licht von außen zu blockieren, kann anstelle einer Desktop-Dunkelkammer eine Box mit einem schwarzen Tuch auf der Innenseite verwendet werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 4: Beispiel für negative Phototaxis nach 5-minütiger Seitenbeleuchtung. (A) Wildtyp-Zellsuspension in einer 5 min beleuchteten Petrischale Die meisten Zellen sammelten sich auf der gegenüberliegenden Seite der Lichtquelle an. Diese Daten können als negative Phototaxis interpretiert werden. (B) Bild des Gerichts von oben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
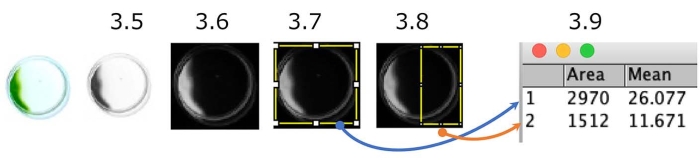
Abbildung 5: Quantifizierung des Schüssel-Phototaxis-Assays. Ein Beispiel für Zellen, die negative Phototaxis zeigen (die Lichtquelle befindet sich auf der rechten Seite). Das Farbbild wird in eine Graustufe konvertiert (Schritt 3.5.) und dann invertiert (Schritt 3.6.). Die interessierenden Regionen (ROI), die gesamte Schale (Schritt 3.7.) und die lichtquellenseitige Hälfte der Schale (Schritt 3.8.) wurden abgegrenzt. Die Dichte jedes ROI wurde gemessen (Schritt 3.9.). In diesem Fall beträgt der phototaktische Index (PI) etwa 0,18 ([1.512 x 11,671] / [2.970 x 26,077]). PI ist 1 oder 0, wenn alle Zellen positive bzw. negative Phototaxis zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
4. Phototaxis-Assay mit Zellkulturtröpfchen
- 25 μL Zellsuspensionströpfchen (Schritt 2.4.) werden mit einer Mikropipette direkt auf eine weiße Kunststoffplatte gelegt.
- Beleuchten Sie die Tröpfchen von einer Seite mit einer grünen LED in einer Desktop-Dunkelkammer (Abbildung 6).
HINWEIS: Typische Lichtverhältnisse sind λ = 525 nm und 50-100 μmol Photonen m−2 s−1. Decken Sie beide Platten und LED mit einer Box oder einem schwarzen Tuch ab, wenn eine kleine LED verwendet wird. - Lassen Sie sie für 3 Minuten stehen und nehmen Sie dann Bilder auf.
HINWEIS: Dieser Assay eignet sich für eine schnelle Überprüfung von Phototaxis vieler Proben auf einmal, z. B. im Mutantenscreening oder in der Tetradenanalyse. Eine Zellkultur in der Mid-Log-Phase, die in einer 96-Well-Platte gezüchtet wird, kann zur leichteren Leistung direkt beleuchtet werden. In beiden Fällen kann Schritt 2 (Zellvorbehandlung) entfallen.

Abbildung 6: Tropfen-Phototaxis-Assay. (A) Neun Tröpfchen einer 25-μL-Zellsuspension auf einer weißen Kunststofffolie platziert und von einer grünen LED von der Seite beleuchtet. (B) Nach 3 Minuten Beleuchtung. In jedem Tröpfchen sammelten sich Zellen entweder auf der Lichtquellenseite an (positive Phototaxis), sammelten sich auf der gegenüberliegenden Seite an (negative Phototaxis) oder diffundierten in das Tröpfchen (keine Phototaxis). Maßstabsleiste = 1 cm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
5. Phototaxis Assay unter dem Mikroskop
- Nehmen Sie ~ 30 μL Zellsuspension (1 x 106 Zellen/ml in experimenteller Photobehavior-Lösung) auf einen Glasobjektträger und legen Sie ein Deckglas (18 mm x 18 mm) mit Abstandshaltern oben auf (Abbildung 7).
HINWEIS: Die Abstandshalter können mit weißem Erdöl oder doppelseitigen Klebebändern auf zwei gegenüberliegenden Seiten eines Deckglases hergestellt werden. Das Licht muss aus einer Richtung kommen, in der es keine Abstandshalter gibt. - Beleuchten Sie die Probe von einer Seite des Deckglases ohne Abstandshalter mit einer grünen LED und beobachten Sie die Zellen unter einem Dunkelfeldmikroskop mit einer 10-fachen Objektivlinse unter schwachem rotem Licht (λ > 630 nm, ~5 μmol Photonen·m−2·s−1, Abbildung 8).
HINWEIS: Vermeiden Sie es, die Zellen auf der Bühne anderem Licht als LED oder Beobachtungslicht auszusetzen, z. B. Raumbeleuchtung oder Licht von einem PC-Monitor. Die geeignete Vergrößerung eines Objektivs hängt vom Blickwinkel der Kamera ab. Wählen Sie eine Linse mit einer Vergrößerung, die es der Zelle ermöglicht, ~ 2 s zu schwimmen, ohne das Sichtfeld zu verlassen. Um Zellen zu verfolgen, ist ein kontrastreiches Bild wünschenswert; Daher wird die Verwendung eines Dunkelfeldkondensators für die mikroskopische Beobachtung empfohlen. Andere Kondensatoren, wie z.B. Hellfelder, können jedoch zur Beobachtung von Phototaxis verwendet werden. Stellen Sie sicher, dass das Licht von LED auf die Zellen scheint. - Zeichnen Sie die Zellbewegung für ~ 20 s nach Lichtbeleuchtung mit einem mit einer Kamera ausgestatteten Mikroskop auf.
HINWEIS: Einige Sekunden nach Beginn der grünen LED-Beleuchtung kann eine Photoschockreaktion auftreten und / oder die phototaktische Ausrichtung (oder das Vorzeichen) ist nicht stabil; Daher wird eine 20-Sekunden-Aufnahme zur Ansprechstabilisierung empfohlen (Film 1).

Abbildung 7: Herstellen von Abstandshaltern an Deckschutzkanten . (A) Eine dünne Schicht Vaseline wurde auf die Handfläche einer Hand aufgetragen. Eine kleine Menge Weißöl wurde mit dem Rand eines Deckglases abgekratzt. (B) Ein Abstandshalter am Rand eines Deckglases. (C) Ein weiterer Abstandshalter an der gegenüberliegenden Kante. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 8: Seitliche Beleuchtung unter dem Mikroskop . (A) Einrichtung einer grünen LED. Eine grüne LED vom Typ Kanonenkugel wird am Muff befestigt und am Ständer neben dem Mikroskop befestigt. Die Zellen wurden unter einem Dunkelfeldmikroskop mit einem scharfen Schnittfilter (λ > 630 nm) beobachtet. (B) Seitliche Beleuchtung durch die grüne LED. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Film 1: Phototaxis-Assay unter dem Mikroskop. Grünes Licht leuchtet bei ~0 s von rechts. Zu diesem Zeitpunkt neigten die Zellen dazu, in eine zufällige Richtung zu schwimmen. Nach 0 s im Zeitzähler schwammen die Zellen entweder nach rechts oder links und zeigten positive oder negative Phototaxis. Das Licht wurde bei ~ 15 s ausgeschaltet, als die Zellen wieder in eine zufällige Richtung zu schwimmen begannen. Maßstabsleiste = 100 μm. Bitte klicken Sie hier, um diesen Film herunterzuladen.
6. Verfolgung phototaktischer Zellen und Polarhistogrammzeichnung
- Extrahieren Sie 1,5 s aus dem aufgenommenen Video, beginnend 15 s nach der Beleuchtung.
HINWEIS: Die Trajektoriendauer kann sich je nach Schwimmgeschwindigkeit der Zelle ändern. Ein 1,5-Sekunden-Video enthält 46 Bilder bei einer Aufnahme mit 30 fps. - Importieren Sie die Datei nach Fidschi als Tag-Bilddateiformat-Bildsequenz über Datei > > Bildsequenz importieren.
- Führen Sie das Plugin "Manual Tracking" für das Zell-Tracking mit Plugins > Tracking > Manual Tracking aus.
- Klicken Sie auf Spur hinzufügen, und der erste Frame wird angezeigt.
- Klicken Sie auf eine Zelle von Interesse, und der zweite Frame wird angezeigt.
- Bewegen Sie den Schieberegler zum letzten Frame, und klicken Sie auf dieselbe Zelle wie in Schritt 6.5.
HINWEIS: Es ist nicht notwendig, Zellen in jedem Frame zu verfolgen. Nur der Winkel zwischen der Lichtachse und dem Liniensegment, das durch den Start und die Endpunkte gebildet wird, ist wesentlich. - Wiederholen Sie die Schritte 6.4.-6.6. für ~30 Zellen.
HINWEIS: Markieren Sie Zellen, die während der gesamten 1,5 s innerhalb des Bildwinkels bleiben. Wenn die Zelldichte ausreichend niedrig ist und sich keine Zellschwimmbahnen für 1,5 s schneiden, kann ein automatisches Tracking durchgeführt werden. Wählen Sie in diesem Fall das Plugin "MTrack2" (typische Einstellungen: Minimale Objektgröße = 1; Maximale Objektgröße = 10.000; Maximale Geschwindigkeit = 100; Minimale Tack Länge [Frames] = 46). Es wird empfohlen, die Korrespondenz zwischen den Zellen im Video und den Tracking-Ergebnissen zu überprüfen, indem Sie die Daten abrufen, indem Sie Text anzeigen? > Überlagern Sie Punkte und Linien. - Kopieren Sie die Ergebnisse und fügen Sie sie in eine Tabellenkalkulationssoftware (z. B. Excel) ein.
HINWEIS: Hierin wird gezeigt, wie man ein Polarhistogramm mit Excel zeichnet. - Messen Sie den Winkel zwischen der Lichtachse und der Schwimmrichtung einer Zelle um "=degrees(atan2(x2-x1),-(y2-y1))", wobei die Startposition der Zelle (Slice #1) (x1, y1) und die letzte Position (Slice #46) (x2, y1) ist.
HINWEIS: In einem Fidschi-Bild ist die obere linke Ecke der Ursprung (0, 0). - Wiederholen Sie Schritt 6.9. für alle gemessenen Zellen (typischerweise ~30 Zellen).
- Bereiten Sie eine Häufigkeitsverteilungstabelle für alle mit den Behältern erhaltenen Daten von −180° bis +180° vor, unterteilt in 15° an beiden Enden und 30° für den Rest des Bereichs, indem Sie die Funktion "FREQUENCY" verwenden.
HINWEIS: Nachdem Sie die Spalten sofort zu den bin-Spalten ausgewählt haben, geben Sie in der oberen Spalte Folgendes ein =FREQUENCY (data_array, bins_array) und drücken Sie Umschalt + Strg + Eingabetaste (Ergänzende Abbildung 1). - Erstellen Sie eine Häufigkeitsverteilungstabelle mit den in Schritt 6.8 berechneten Werten neu. um −90° gedreht, um die rechte Seite auf 0° einzustellen (d. h. die Zahl im Bereich von −15° bis 15° in den Bereich von −105° bis −75° umzuwandeln), da bei der obigen Methode davon ausgegangen wird, dass Zellen, die nach oben schwimmen, in einem Winkel von 0° zum Licht schwimmen (d. h. positive Phototaxis zeigen), obwohl das Licht von rechts kommt (Ergänzende Abbildung 2).
- Geben Sie den Winkelwert in der Mitte des Bereichs eines 30°-Behälters ein (z. B. 30° für 15°-45°), überspringen Sie eine Spalte und geben Sie rechts die entsprechende Stichprobenanzahl ein (Ergänzende Abbildung 3).
- Konvertieren Sie jeden Stichprobenzählwert in einen Prozentsatz, und geben Sie 0 in die Spalte ein, die der leeren Spalte zwischen den Ablagen entspricht.
- Zeichnen Sie ein Radardiagramm mit den Winkelwerten als horizontalen Achsenbeschriftungen und den Prozentwerten (%) als Legende (Ergänzende Abbildung 4).
- Berechnen Sie den phototaktischen Index (PI) zur weiteren Quantifizierung durch Mittelung des cosθ (θ = der Winkel zwischen der Lichtachse und der Schwimmrichtung)16.
HINWEIS: PI ist 0, wenn Zellen in eine zufällige Richtung schwimmen, und 1 oder −1, wenn 100% der Zellen positive bzw. negative Phototaxis zeigen.
7. Photoshock response Assay unter dem Mikroskop
- Geben Sie ~ 30 μL Zellsuspension (1 x 106 Zellen/ml in der experimentellen Photobehavior-Lösung) auf einen Glasobjektträger und legen Sie ein Deckglas (18 mm x 18 mm) mit Abstandshaltern darauf, wie in Schritt 4.1.
- Beobachten Sie die Zellen unter einem Mikroskop mit schwachem rotem Licht (λ > 630 nm, ~5 μmol Photonen·m−2·s−1).
- Wenden Sie die Blitzbeleuchtung mit einem Kamerablitz an (Film 2,3).
HINWEIS: Eine andere Möglichkeit, eine Photoschockreaktion zu induzieren, besteht darin, einen Rotfilter schnell von Hand aus dem Beobachtungslichtpfad zu entfernen. Diese Methode ist jedoch variabler, da die Geschwindigkeit der Filterentfernung von Person zu Person variiert (Film 4,5). - Zeichnen Sie die Zellbewegung mit einem mit einer Kamera ausgestatteten Mikroskop auf.
- Führen Sie zur Quantifizierung eine oder beide der folgenden Aktionen aus: (1) Berechnen Sie das Verhältnis der Zellen, die die Photoschockreaktion zeigen, pro Gesamtzelle16; (2) Für Zellen, die die Photoschockreaktion zeigen, messen Sie die Zeit vom Photoschockreiz bis zur Erholung des Vorwärtsschwimmens25.
Film 2: Photoschockbeleuchtung durch einen Kamerablitz. Der Kamerablitz wurde bis zur Mikroskopstufe gehalten und eingeschaltet. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 3: Photoschockreaktion durch einen Blitz unter dem Mikroskop. Die Zellen wurden unter schwachem rotem Licht beobachtet. Ein Blitz wurde bei ~0 s emittiert. Fast alle Zellen hörten beim Vorwärtsschwimmen auf, schwammen für kurze Zeit rückwärts und erholten sich vorwärts schwimmend. Maßstabsleiste = 100 μm. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 4: Photoschockreaktion durch Entfernen eines Rotfilters unter dem Mikroskop. Die Zellen wurden unter schwachem rotem Licht beobachtet. Der Rotfilter wurde bei ~5 s entfernt. Fast alle Zellen hörten beim Vorwärtsschwimmen auf, schwammen für kurze Zeit rückwärts und erholten sich vorwärts schwimmend. Maßstabsleiste = 100 μm. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 5: Entfernen eines Rotfilters Schnelle Entfernung eines Rotfilters, der im Lichtweg eingestellt ist, um einen Fotoschock zu liefern. Bitte klicken Sie hier, um diesen Film herunterzuladen.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Typische C. reinhardtii Phototaxis und Photoshock Response Assays sind hier dargestellt. Nach der Schätzung der Zelldichte wurde die Wildtyp-Zellkultur (ein Nachkomme des Kreuzes CC-124 × CC-125 mit agg1+ mt -)23 mit Photobehavior-Experimentierlösung für den Phototaxis-Schalenassay gewaschen. Die Zellsuspension wurde für ~1 h unter schwaches rotes Licht gestellt. Eine 2 ml Zellsuspension wurde in eine 3,5 cm große Petrischale gegeben. Die Petrischale wurde sanft geschüttelt, auf ei...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Das vorliegende Protokoll ist einfach und nicht zeitaufwendig. Wenn bei einer C. reinhardtii-Mutante der Verdacht besteht, dass sie Defekte in der Photorezeption oder Ziliarbewegung aufweist, könnte diese Methode als primäre phänotypische Analyse dienen.
Es gibt jedoch einige kritische Schritte. Eine besteht darin, Zellen im Experiment in der frühen bis mittleren Log-Wachstumsphase zu verwenden. Nach längerer Kultivierung werden die Zellen weniger beweglich, lichtempfindlich und ...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Diese Studie wurde durch Zuschüsse der Japan Society for the Promotion of Science KAKENHI (https://www.jsps.go.jp/english/index.html) an NU (19K23758, 21K06295), TH (16H06556) und KW (19H03242, 20K21420, 21H00420), von der Ohsumi Frontier Science Foundation (https://www.ofsf.or.jp/en/) an KW und von der Dynamic Alliance for Open Innovation Bridging Human, Environment and Materials (http://alliance.tagen.tohoku.ac.jp/english/) an NU, TH und KW unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SARSTEDT | 62.554.502 | |
| 5 mm Cannonball green LED | Optosupply | OSPG5161P | |
| 50 mL conical tube | SARSTEDT | 62.547.254 | |
| AC adaptor for the light box | ATTO | 2196161 | |
| Auto cell counter | DeNovix | CellDrop BF | |
| CaCl2 | Nakalai tesque | 06731-05 | |
| Camera flash | NEWWER | TT560 | |
| Centrifuge | KUBOTA | 2800 | |
| Chlamydomonas strains CC-124 and CC-125 | Chlamydomonas Resource Center | https://www.chlamycollection.org/ | |
| C-mout CCD camera | Wraymer | 1129HMN1/3 | |
| Desktop darkroom | Scientex | B-S8 | |
| Digital still camera | SONY | RX100II | |
| EGTA | Dojindo | G002 | |
| Fiji | https://fiji.sc/ | ||
| Green LED plate | CCS | ISLM-150X150-GG | |
| HCl | Fujifilm WAKO | 080-01066 | |
| HEPES | Dojindo | GB70 | |
| KCl | Nakalai tesque | 238514-75 | |
| Lightbox (Flat viewer) | ATTO | 2196160 | |
| Microscope | Olympus | BX-53 | |
| Petri dish (φ3.5 cm) | IWAKI | 1000-035 | |
| Pottasium acetate | Nakalai tesque | 28434-25 | |
| Power supply for the green LED plate | CCS | ISC-201-2 | |
| Red filter | Shibuya Optical | S-RG630 |
Referenzen
- Demmig-Adams, B., Adams, W. W. Photoprotection and other responses of plants to high light stress. Annual Reviews Plant Physiology and Plant Molecular Biology. 43, 599-626 (1992).
- Wada, M. Chloroplast movement. Plant Science. 210, 177-182 (2013).
- Sgarbossa, A., Checcucci, G., Lenci, F. Photoreception and photomovements of microorganisms. Photochemical & Photobiological Sciences. 1 (7), 459-467 (2002).
- Ueki, N., et al. Eyespot-dependent determination of the phototactic sign in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 113 (19), 5299-5304 (2016).
- Foster, K. W., Smyth, R. D. Light antennas in phototactic algae. Microbiological Reviews. 44 (4), 572-630 (1980).
- Nagel, G., et al. Channelrhodopsin-1: a light-gated proton channel in green algae. Science. 296 (5577), 2395-2398 (2002).
- Nagel, G., et al. Channelrhodopsin-2, a directly light-gated cation-selective membrane channel. Proceedings of the National Academy of Sciences of the United States of America. 100 (24), 13940-13945 (2003).
- Sineshchekov, O. A., Jung, K. -H., Spudich, J. L. Two rhodopsins mediate phototaxis to low- and high-intensity light in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 99 (13), 8689-8694 (2002).
- Suzuki, T., et al. Archaeal-type rhodopsins in Chlamydomonas: model structure and intracellular localization. Biochemical and Biophysical Research Communications. 301 (3), 711-717 (2003).
- Berthold, P., et al. Channelrhodopsin-1 initiates phototaxis and photophobic responses in Chlamydomonas by immediate light-induced depolarization. Plant Cell. 20 (6), 1665-1677 (2008).
- Deisseroth, K., et al. Next-generation optical technologies for illuminating genetically targeted brain circuits. Journal of Neuroscience. 26 (41), 10380-10386 (2006).
- Wakabayashi, K., Isu, A., Ueki, N. Channelrhodopsin-dependent photo-behavioral responses in the unicellular green alga Chlamydomonas reinhardtii. Optogenetics (Advances in Experimental Medicine and Biology), 2nd ed. , Springer. 21-33 (2021).
- Rüffer, U., Nultsch, W. Flagellar photoresponses of Chlamydomonas cells held on micropipettes: II. Change in flagellar beat pattern. Cell Motility and the Cytoskeleton. 18 (4), 269-278 (1991).
- Kamiya, R., Hasegawa, E. Intrinsic difference in beat frequency between the two flagella of Chlamydomonas reinhardtii. Experimental Cell Research. 173, 299-304 (1987).
- Kamiya, R., Witman, G. B. Submicromolar levels of calcium control the balance of beating between the two flagella in demembranated models of Chlamydomonas. Journal of Cell Biology. 98 (1), 97-107 (1984).
- Okita, N., Isogai, N., Hirono, M., Kamiya, R., Yoshimura, K. Phototactic activity in Chlamydomonas 'non-phototactic' mutants deficient in Ca2+-dependent control of flagellar dominance or in inner-arm dynein. Journal of Cell Science. 118, 529-537 (2005).
- Horst, C. J., Witman, G. B. ptx1, a nonphototactic mutant of Chlamydomonas, lacks control of flagellar dominance. Journal of Cell Biology. 120 (3), 733-741 (1993).
- Hyams, J. S., Borisy, G. G. Isolated flagellar apparatus of Chlamydomonas: characterization of forward swimming and alteration of waveform and reversal of motion by calcium ions in vitro. Journal of Cell Science. 33, 235-253 (1978).
- Bessen, M., Fay, R. B., Witman, G. B. Calcium control of waveform in isolated flagellar axonemes of Chlamydomonas. Journal of Cell Biology. 86 (2), 446-455 (1980).
- Reiter, J. F., Leroux, M. R. Genes and molecular pathways underpinning ciliopathies. Nature Reviews Molecular Cell Biology. 18 (9), 533-547 (2017).
- Ide, T., et al. Identification of the agg1 mutation responsible for negative phototaxis in a "wild-type" strain of Chlamydomonas reinhardtii. Biochemistry and Biophysics Reports. 7, 379-385 (2016).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Finst, R. J., Kim, P. J., Quarmby, L. M. Genetics of the deflagellation pathway in Chlamydomonas. Genetics. 149 (2), 927-936 (1998).
- Morel-Laurens, N. Calcium control of phototactic orientation in Chlamydomonas reinhardtii: sign and strength of response. Photochemistry and Photobiology. 45 (1), 119-128 (1987).
- Wakabayashi, K., King, S. M. Modulation of Chlamydomonas reinhardtii flagellar motility by redox poise. Journal of Cell Biology. 173 (5), 743-754 (2006).
- Wakabayashi, K., Misawa, Y., Mochiji, S., Kamiya, R. Reduction-oxidation poise regulates the sign of phototaxis in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11280-11284 (2011).
- Harris, E. H. in The Chlamydomonas Sourcebook Second Edition. 1, Academic Press. Ch. 2 25-64 (2009).
- Mergenhagen, D. Circadian clock: genetic characterization of a short period mutant of Chlamydomonas reinhardii. European Journal of Cell Biology. 33 (1), 13-18 (1984).
- Ozasa, K., Lee, J., Song, S., Hara, M., Maeda, M. Two-dimensional optical feedback control of Euglena confined in closed-type microfluidic channels. Lab on a Chip. 11 (11), 1933-1940 (2011).
- Tanno, A., et al. The four-celled Volvocales green alga Tetrabaena socialis exhibits weak photobehavior and high-photoprotection ability. PLoS One. 16 (10), 0259138(2021).
- Ueno, Y., Aikawa, S., Kondo, A., Akimoto, S. Adaptation of light-harvesting functions of unicellular green algae to different light qualities. Photosynthesis Research. 139 (1-3), 145-154 (2019).
- Takahashi, T., Watanabe, M. Photosynthesis modulates the sign of phototaxis of wild-type Chlamydomonas reinhardtii. Effects of red background illumination and 3-(3',4'-dichlorophenyl)-1,1-dimethylurea. FEBS Letters. 336 (3), 516-520 (1993).
- Morishita, J., Tokutsu, R., Minagawa, J., Hisabori, T., Wakabayashi, K. I. Characterization of Chlamydomonas reinhardtii mutants that exhibit strong positive phototaxis. Plants (Basel). 10 (7), (2021).
- Fujiu, K., Nakayama, Y., Yanagisawa, A., Sokabe, M., Yoshimura, K. Chlamydomonas CAV2 encodes a voltage- dependent calcium channel required for the flagellar waveform conversion. Current Biology. 19 (2), 133-139 (2009).
- Inaba, K. Calcium sensors of ciliary outer arm dynein: functions and phylogenetic considerations for eukaryotic evolution. Cilia. 4 (1), 6(2015).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten