È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Osservazione del comportamento fotografico in Chlamydomonas reinhardtii
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
La maggior parte degli organismi fotoautotrofici che nuotano mostrano cambiamenti comportamentali fotoindotti (comportamento fotografico). Il presente protocollo osserva il suddetto comportamento fotografico nell'organismo modello Chlamydomonas reinhardtii.
Abstract
Per la sopravvivenza dei microrganismi fototrofici mobili, essere in condizioni di luce adeguate è fondamentale. Di conseguenza, mostrano comportamenti fotoindotti (o fotocomportamenti) e alterano la loro direzione di movimento in risposta alla luce. I comportamenti fotografici tipici includono la risposta al fotoshock (o fotofobico) e la fototassi. Il photoshock è una risposta a un improvviso cambiamento dell'intensità della luce (ad esempio, l'illuminazione del flash), in cui gli organismi smettono temporaneamente di muoversi o si muovono all'indietro. Durante la fototaxi, gli organismi si muovono verso la sorgente luminosa o nella direzione opposta (chiamata fototassi positiva o negativa, rispettivamente). L'alga verde unicellulare Chlamydomonas reinhardtii è un organismo eccellente per studiare il comportamento fotografico perché cambia rapidamente il suo modello di nuoto modulando il battito delle ciglia (alias flagelli) dopo la fotoricezione. Qui, vengono mostrati vari metodi semplici per osservare i comportamenti fotografici in C. reinhardtii. La ricerca sui fotocomportamenti di C. reinhardtii ha portato alla scoperta di meccanismi regolatori comuni tra ciglia eucariotiche e channelrhodopsins, che possono contribuire a una migliore comprensione delle ciliopatie e allo sviluppo di nuovi metodi optogenetici.
Introduzione
La luce è una fonte di energia indispensabile per gli organismi fotosintetici, ma troppa luce può causare danni fotoossidanti. Pertanto, gli organismi fototrofici devono sopravvivere sotto una luce di intensità moderata, dove possono fotosintetizzare ma non subire danni foto-ossidativi1. Nelle piante terrestri, i cloroplasti non possono uscire dalla foglia e mostrare movimenti fotografici nella cellula; i cloroplasti si spostano alla periferia della cellula in condizioni di luce intensa e la superficie cellulare in condizioni di scarsa illuminazione2, mentre molte alghe mobili mostrano fotocomportamenti che consentono loro di trovare condizioni di luce adeguate per la fotosintesi e, quindi, facilitare la loro sopravvivenza3.
Chlamydomonas reinhardtii è un'alga verde unicellulare considerata un organismo modello in campi di ricerca come le ciglia (alias flagelli), la fotosintesi e il fotocomportamento. C. reinhardtii si presenta con una macchia oculare e due ciglia per cellula, utilizzate rispettivamente per la fotoricezione e il nuoto. La macchia oculare ha due componenti: le channelrhodopsins (ChRs), i canali ionici light-gated nella membrana plasmatica e gli strati di granuli ricchi di carotenoidi situati proprio dietro i ChRs. La macchia oculare agisce come un recettore della luce direzionale poiché gli strati di granuli ricchi di carotenoidi funzionano come un riflettore di luce 4,5.
I ChR sono stati inizialmente identificati come fotorecettori che causano fotocomportamenti in C. reinhardtii 6,7,8,9. Sebbene due isoforme, ChR1 e ChR2, si trovino nella macchia oculare, esperimenti di abbattimento hanno dimostrato che ChR1 è il fotorecettore primario per i fotocomportamenti10. Nonostante ciò, ChR2 ha ricevuto maggiore attenzione e ha svolto un ruolo centrale nello sviluppo dell'optogenetica, una tecnica per controllare l'eccitazione cellulare mediante la luce11. Pertanto, lo studio dei meccanismi regolatori che regolano i fotocomportamentatori in C. reinhardtii favorirà la comprensione della funzione ChR e migliorerà l'optogenetica.
Dopo la fotoricezione, le cellule di C. reinhardtii mostrano due tipi di fotocomportamenti: fototaxi e risposta al fotoshock12. La fototaxi è il comportamento delle cellule che nuotano nella direzione della sorgente luminosa o nella direzione opposta, chiamata fototassi positiva o negativa, rispettivamente. La risposta al fotoshock è un comportamento che le cellule mostrano dopo aver rilevato un improvviso cambiamento nell'intensità della luce, ad esempio quando illuminato da un flash. Le cellule smettono di nuotare o nuotano all'indietro (cioè nuotano con il corpo cellulare in avanti) per un breve periodo, in genere <1 s.
I movimenti ciliari in C. reinhardtii sono coinvolti nei suoi fotocomportamenti. Due ciglia di solito battono come la rana di un essere umano che nuota, e questo è modulato per i fotocomportamenti. Per la fototassi, le forze generate dalle due ciglia sono sbilanciate dalla modulazione della frequenza di battitura e dall'ampiezza della forma d'onda di ciascun cilio13. Il cilio più vicino alla macchia oculare è chiamato cis cilium e l'altro è chiamato trans cilium. Queste due ciglia differiscono su vari punti. Ad esempio, la frequenza di battitura ciliare del transcilio in vitro è del 30%-40% superiorea 14. Inoltre, la loro sensibilità Ca2+ è diversa. La riattivazione dei modelli di cellule demembranate15 ha mostrato che il cis cilium batte più fortemente del trans cilium per Ca2+ <1 x 10−8 M, mentre il contrario è vero per Ca2+ >1 x 10−7 M. Questa asimmetria nella sensibilità di Ca2+ è probabilmente importante per i turni fototattici poiché i mutanti privi di questa asimmetria non presentano una normale fototassi16,17. Al contrario, la conversione della forma d'onda è necessaria per il photoshock. La forma d'onda ciliare si trasforma dalla forma d'onda asimmetrica nel nuoto in avanti alla forma d'onda simmetrica nel nuoto all'indietro. Questa conversione della forma d'onda è regolata anche da Ca2+, ad una soglia di 1 x 10−4 M18,19. Poiché i difetti nella regolazione dei movimenti ciliari causano discinesia ciliare primaria negli esseri umani, lo studio dei fotocomportamentatori in C. reinhardtii potrebbe aiutare a comprendere meglio queste malattie e gli sviluppi terapeutici20.
Qui, vengono dimostrati quattro semplici metodi per osservare i fotocomportamentatori in C. reinhardtii . In primo luogo, viene mostrato un test di fototassi utilizzando piastre di Petri e, in secondo luogo, un test di fototassi contro le goccioline di sospensione cellulare. Il fenomeno osservato in entrambi i casi non è strettamente fototaxi ma fotoaccumulo, dove le cellule tendono ad accumularsi vicino al lato della sorgente luminosa o al lato opposto. In C. reinhardtii, il fotoaccumulo è causato principalmente dalla fototassi in un modo che può essere usato come approssimazione alla fototassi. In terzo luogo, viene mostrato un test più rigoroso per la fototassi al microscopio, e l'ultimo è un test di fotoshock al microscopio.
Protocollo
Nel presente studio, un ceppo wild-type di Chlamydomonas reinhardtii, una progenie della croce CC-124 x CC-125 con agg1+mt-, è stato utilizzato21. CC-124 e CC-125 sono stati ottenuti dal Chlamydomonas Resource Center (vedi Tabella dei materiali) e mantenuti su un mezzo tris-acetato-fosfato (TAP)22, 1,5% di agarosio a 20-25 °C. Qualsiasi ceppo mobile può essere utilizzato per questo protocollo.
1. Coltura cellulare
- Coltivare un ceppo di interesse di Chlamydomonas reinhardtii in mezzo liquido TAP con aerazione facendo gorgogliare aria sterile in un periodo chiaro-scuro di 12 h/12 h (periodo di luce, ~50 μmol fotoni·m−2·s−1 luce bianca) a 20-25 °C per 2 giorni.
NOTA: è necessario utilizzare cellule in una fase di crescita logaritmica media. La coltura lunga (>3 giorni, nella fase di crescita tardo-logaritmica) rende le cellule meno sensibili allo stimolo luminoso e aumenta il numero di cellule morte in coltura, ostacolando la lettura dei risultati.

Figura 1: Coltura liquida dopo 2 giorni di coltivazione. Da una piastra di agar TAP-1,5%, un pezzo di cellule selvatiche che riempivano il loop di platino è stato inoculato in ~ 150 ml di mezzo liquido TAP in un pallone. La densità cellulare dopo coltura di 2 giorni era ~ 5,0 x 106 cellule / ml. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Pretrattamento delle cellule
- Mescolare ~ 10 μL della coltura con un volume uguale di soluzione di deciliazione per impedire alle cellule di nuotare e misurare la densità cellulare usando un contatore cellulare o un emocitometro.
NOTA: ci sono circa 1-5 x 106 cellule/ml dopo una coltura di 2 giorni (Figura 1). La composizione della soluzione di deciliazione è la seguente23: 40 mM acetato di potassio, 1 mM CaCl2, pH 4,5 regolato con HCl. - Centrifugare la quantità necessaria di coltura liquida a 1000 x g per 3 minuti a temperatura ambiente.
NOTA: Un esperimento richiede 3 mL di 2 x 107 celle/mL per le condizioni standard per il test della fototassi in una capsula di Petri. Se verranno eseguiti due esperimenti con 1 x 106 cellule/ mL di coltura, è necessario centrifugare 120 mL di coltura. - Sospendere il pellet cellulare con la quantità richiesta di soluzione sperimentale di fotocomportamento a ~2 x 107 cellule/mL (per il saggio di fototassi a piatto) o 1 x 106 cellule/mL (per il saggio di fototassi a livello cellulare o il test di risposta al fotoshock) e mettere la sospensione cellulare in un tubo conico.
NOTA: la densità cellulare non ha bisogno di essere controllata in modo molto rigoroso per una stima approssimativa dei fotocomportamenti o può cambiare a seconda dello scopo del test. Soluzione sperimentale di fotocomportamento14: 5 mM HEPES (pH 7,4), 0,2 mM EGTA, 1 mM KCl, 0,3 mM CaCl2. Questa fase di scambio del buffer può essere omessa per un semplice test sulla capacità di esibire fotocomportamenti e l'esperimento può essere eseguito con il terreno di coltura. Tuttavia, poiché la composizione ionica della soluzione influisce sui segni fototattici24 e la composizione ionica del mezzo dopo la coltura potrebbe non essere costante, si raccomanda l'uso di questa soluzione per un test più rigoroso. La sostituzione con un mezzo TAP fresco può essere un'opzione. - Posizionare il tubo sotto una luce rossa fioca (10-30 μmol fotoni·m−2·s−1) per ~1 h (Figura 2).
NOTA: Questo passaggio aumenta la sensibilità delle cellule allo stimolo luminoso.

Figura 2: Sospensione cellulare sotto semaforo rosso. Una normale luce bianca fluorescente ricoperta da un foglio di cellophane rosso. Un tubo contenente la sospensione cellulare è posto sotto ~10 μmol fotoni·m−2·s−1 luce rossa. Fare clic qui per visualizzare una versione più grande di questa figura.
3. Test di fototatassi con la capsula di Petri (il cosiddetto "saggio del piatto")
- Mettere 2-3 ml di sospensione cellulare in una capsula di Petri (3,5 cm), posizionarla su una lightbox (in alternativa, un foglio di carta bianco o una piastra di plastica bianca), agitare delicatamente per distribuire uniformemente le cellule e acquisire un'immagine prima dell'illuminazione.
NOTA: la dimensione del piatto può cambiare a seconda dello scopo. A seconda del ceppo e dello stato della coltura, le cellule potrebbero attaccarsi al fondo della capsula di Petri. In tal caso, le cellule bloccate devono essere rimosse mediante pipettaggio prima del test. - Illuminare la parabola da un lato con un diodo a emissione di luce verde (LED, vedere Tabella dei materiali) in una camera oscura desktop (Figura 3).
NOTA: Le condizioni di luce tipiche sono λ = 525 nm e 50-100 μmol fotoni·m−2·s−1. Vedere la discussione sulla selezione della lunghezza d'onda della sorgente luminosa nella sezione di discussione. Coprire sia i piatti che il LED con una scatola o un panno nero quando si utilizza un piccolo LED. - Lasciarli per ≥5 minuti e poi acquisire le immagini.
NOTA: il tempo per l'illuminazione della luce può essere modificato a seconda delle dimensioni o dello scopo della capsula di Petri (Figura 4). - Importare il file di immagine nelle Figi (vedere Tabella dei materiali) per la quantificazione.
NOTA: Fiji è una distribuzione di ImageJ2, che raggruppa molti plugin. - Modificare l'immagine a colori in scala di grigi tramite Image > Digitare > a 8 bit (Figura 5).
- Inverti il bianco e nero tramite Modifica > Inverti.
- Circonda l'intero piatto come regione di interesse (ROI) e misura la densità attraverso Analizza > Misura.
NOTA: la densità del piatto totale può essere calcolata come (Area) x (Media). - Circonda la metà della parabola più vicina alla sorgente luminosa come ROI e misura la densità.
- Calcolare l'indice fototattico come (densità delle cellule fototattiche) per (densità delle cellule totali).

Figura 3: Illuminazione laterale per il saggio della parabola della fototaxi. (A) Una capsula di Petri contenente una sospensione cellulare posta su una lightbox in una camera oscura desktop. Luce verde (piastra LED da 525 nm, ~100 μmol fotoni·m−2·s−1) illuminata lateralmente. (B) Metodo di illuminazione alternativo. Un LED da 5 mm a palla di cannone. (C) Per bloccare la luce dall'esterno, è possibile utilizzare una scatola con un panno nero all'interno al posto di una camera oscura desktop. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Esempio di fototassi negativa dopo un'illuminazione laterale di 5 minuti. (A) Sospensione cellulare wild-type in una capsula di Petri illuminata per 5 min. La maggior parte delle cellule si è accumulata sul lato opposto della sorgente luminosa. Questi dati possono essere interpretati come fototaxi negativi. (B) Immagine del piatto dall'alto. Fare clic qui per visualizzare una versione più grande di questa figura.
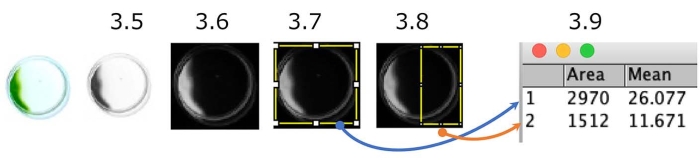
Figura 5: Quantificazione del saggio di fototassi del piatto. Un esempio di celle che mostrano fototassi negativa (la fonte di luce è sul lato destro). L'immagine a colori viene convertita in scala di grigi (passaggio 3.5.) e quindi invertita (passaggio 3.6.). Le regioni di interesse (ROI), l'intero piatto (passaggio 3.7.) e la metà lato sorgente luminosa del piatto (passaggio 3.8.) sono stati delimitati. È stata misurata la densità di ciascun ROI (Step 3.9.). In questo caso, l'indice fototattico (PI) è di circa 0,18 ([1.512 x 11,671] / [2.970 x 26,077]). PI è 1 o 0 quando tutte le cellule mostrano rispettivamente fototassi positiva o negativa. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Saggio di fototassi con goccioline di coltura cellulare
- Posizionare 25 μL di goccioline di sospensione cellulare (fase 2.4.) direttamente su una piastra di plastica bianca utilizzando una micropipetta.
- Illuminare le goccioline da un lato con un LED verde in una camera oscura desktop (Figura 6).
NOTA: Le condizioni di luce tipiche sono λ = 525 nm e 50-100 μmol fotoni m−2 s−1. Coprire sia le piastre che il LED con una scatola o un panno nero quando si utilizza un piccolo LED. - Lasciarli per 3 minuti e poi acquisire le immagini.
NOTA: Questo test è adatto per un rapido controllo della fototassi di molti campioni contemporaneamente, come nello screening mutante o nell'analisi tetradica. Una coltura cellulare nella fase mid-log cresciuta in una piastra a 96 pozzetti può essere illuminata direttamente per prestazioni più facili. In entrambi i casi, la fase 2 (pretrattamento cellulare) può essere omessa.

Figura 6: Saggio di fototassi a goccia. (A) Nove goccioline di una sospensione cellulare da 25 μL poste su un foglio di plastica bianca e illuminate lateralmente da un LED verde. (B) Dopo 3 minuti di illuminazione. In ogni goccia, le cellule si sono accumulate sul lato della sorgente luminosa (fototassi positiva), accumulate sul lato opposto (fototaxi negativi) o diffuse nella goccia (senza fototaxi). Barra della scala = 1 cm. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Analisi della fototassi al microscopio
- Prelevare ~30 μL di sospensione cellulare (1 x 106 celle/mL in soluzione sperimentale fotocomportamentale) su un vetrino e posizionare un coperchio (18 mm x 18 mm) con distanziali in cima (Figura 7).
NOTA: I distanziali possono essere realizzati con petrolio bianco o nastri biadesivi su due lati opposti di un coperchio. La luce deve provenire da una direzione in cui non ci sono distanziali. - Illuminare il campione da un lato del coperchio senza un distanziatore con un LED verde e osservare le cellule sotto un microscopio a campo scuro con una lente obiettivo 10x in penombra rossa (λ > 630 nm, ~5 μmol fotoni·m−2·s−1, Figura 8).
NOTA: evitare di esporre le celle sul palco a una luce diversa dai LED o dalla luce di osservazione, come l'illuminazione della stanza o la luce di un monitor del PC. L'ingrandimento adeguato di un obiettivo dipende dall'angolo di visione della fotocamera. Selezionare un obiettivo con un ingrandimento che consenta alla cella di nuotare per ~ 2 s senza lasciare il campo visivo. Per tracciare le celle, è auspicabile un'immagine ad alto contrasto; pertanto, l'uso di un condensatore a campo oscuro è raccomandato per l'osservazione microscopica. Tuttavia, altri condensatori, come il campo luminoso, possono essere utilizzati per osservare la fototassi. Assicurarsi che la luce del LED splenda sulle celle. - Registra il movimento cellulare per ~ 20 s dopo l'illuminazione della luce utilizzando un microscopio dotato di fotocamera.
NOTA: Pochi secondi dopo l'inizio dell'illuminazione LED verde, potrebbe verificarsi una risposta fotoshock e/o l'orientamento fototattico (o segno) non è stabile; pertanto, si consiglia una registrazione di 20 s per la stabilizzazione della risposta (Movie 1).

Figura 7: Realizzazione di distanziali sui bordi delle coperture. (A) Un sottile strato di vaselina è stato applicato al palmo di una mano. Una piccola quantità di petrolio bianco è stata raschiata via con il bordo di una copertura. (B) Un distanziatore sul bordo di una copertura. (C) Un altro distanziatore sul bordo opposto. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 8: Illuminazione laterale al microscopio. (A) Configurazione di un LED verde. Un LED verde a palla di cannone è fissato al manicotto e fissato al supporto accanto al microscopio. Le cellule sono state osservate al microscopio a campo scuro con un filtro a taglio acuto (λ > 630 nm). (B) Illuminazione laterale tramite LED verde. Fare clic qui per visualizzare una versione più grande di questa figura.
Filmato 1: Analisi della fototaxi al microscopio. Luce verde illuminata a ~0 s da destra. A quel punto, le cellule tendevano a nuotare in una direzione casuale. Dopo 0 s nel contatore del tempo, le cellule nuotavano a destra oa sinistra, mostrando fototassi positiva o negativa. La luce è stata spenta a ~ 15 s quando le cellule hanno iniziato a nuotare di nuovo in una direzione casuale. Barra della scala = 100 μm. Fare clic qui per scaricare questo filmato.
6. Tracciamento delle cellule fototattiche e disegno dell'istogramma polare
- Estrarre 1,5 s dal video registrato, a partire da 15 s dopo l'illuminazione.
NOTA: la durata della traiettoria può variare a seconda della velocità di nuoto della cellula. Un video da 1,5 s contiene 46 fotogrammi a 30 fps di registrazione. - Importare il file nelle Figi come sequenza di immagini Tag Image File Format tramite File > Import > Image Sequence.
- Esegui il plugin "Manual Tracking" per il monitoraggio delle celle utilizzando Plugin > Tracking > Manual Tracking.
- Fate clic su Aggiungi traccia e viene visualizzato il primo fotogramma.
- Fare clic su una cella di interesse e viene visualizzato il secondo fotogramma.
- Spostate il dispositivo di scorrimento sull'ultimo fotogramma e fate clic sulla stessa cella del passaggio 6.5.
NOTA: non è necessario tenere traccia delle celle in ogni fotogramma. Solo l'angolo tra l'asse della luce e il segmento di linea formato dall'inizio e dal punto finale è essenziale. - Ripetere i passaggi 6.4.-6.6. per ~30 celle.
NOTA: selezionare le celle che rimangono all'interno dell'angolo di visione per l'intero 1,5 s. Se la densità cellulare è sufficientemente bassa e nessuna traiettoria di nuoto cellulare si interseca per 1,5 s, è possibile eseguire il tracciamento automatico. In tal caso, scegli il plugin "MTrack2" (impostazioni tipiche: Dimensione minima dell'oggetto = 1; Dimensione massima dell'oggetto = 10.000; Velocità massima = 100; Lunghezza minima tack [Fotogrammi] = 46). Si consiglia di verificare la corrispondenza tra le celle del video e i risultati del tracciamento ottenendo i dati selezionando Mostra testo? > Sovrapposizione punti e linee. - Copia e incolla i risultati nel software per fogli di calcolo (ad esempio, Excel).
NOTA: qui viene mostrato come disegnare un istogramma polare con Excel. - Misurare l'angolo tra l'asse della luce e la direzione di nuoto di una cella di "=gradi(atan2(x2-x1),-(y2-y1))", dove la posizione iniziale della cella (fetta #1) è (x1, y1) e l'ultima posizione (fetta #46) è (x2, y1).
NOTA: in un'immagine delle Figi, l'angolo in alto a sinistra è l'origine (0, 0). - Ripetere il passaggio 6.9. per tutte le cellule misurate (tipicamente ~30 celle).
- Preparare una tabella di distribuzione della frequenza per tutti i dati ottenuti con i contenitori da -180° a +180°, suddivisa in 15° ad entrambe le estremità e 30° per il resto dell'intervallo utilizzando la funzione "FREQUENZA".
NOTA: dopo aver selezionato immediatamente le colonne nelle colonne bin, immettere quanto segue nella colonna superiore =FREQUENZA (data_array, bins_array) e premere MAIUSC + CTRL + INVIO (Figura supplementare 1). - Ricreare una tabella di distribuzione delle frequenze con i valori calcolati nel passaggio 6.8. ruotato di -90° per impostare il lato destro su 0° (cioè, convertire il numero nell'intervallo da -15° a 15°, nell'intervallo da -105° a -75°), poiché, con il metodo di cui sopra, le cellule che nuotano verso l'alto sono considerate nuotare con un angolo di 0° rispetto alla luce (cioè, mostrando fototassi positiva), sebbene la luce provenga da destra (Figura supplementare 2).
- Immettere il valore dell'angolo nel mezzo dell'intervallo di un contenitore di 30° (ad esempio, 30° per 15°-45°), saltando una colonna, e inserire il conteggio dei campioni corrispondente alla sua destra (Figura supplementare 3).
- Convertire ogni valore di conteggio del campione in percentuale e immettere 0 nella colonna corrispondente alla colonna vuota tra i raccoglitori.
- Disegnare un grafico radar utilizzando i valori angolari come etichette degli assi orizzontali e i valori percentuali (%) come legenda (Figura supplementare 4).
- Calcola l'indice fototattico (PI) per un'ulteriore quantificazione facendo la media del cosθ (θ = l'angolo tra l'asse della luce e la direzione del nuoto)16.
NOTA: PI è 0 quando le cellule nuotano in una direzione casuale e 1 o -1 quando il 100% delle cellule mostra rispettivamente fototassi positiva o negativa.
7. Test di risposta al fotoshock al microscopio
- Posizionare ~ 30 μL di sospensione cellulare (1 x 106 celle / mL nella soluzione sperimentale di fotocomportamento) su un vetrino e posizionare un coperchio (18 mm x 18 mm) con distanziali in cima, come nel passaggio 4.1.
- Osservare le cellule al microscopio con luce rossa fioca (λ > 630 nm, ~5 μmol fotoni·m−2·s−1).
- Applicare l'illuminazione del flash utilizzando un flash della fotocamera (Filmato 2,3).
NOTA: un altro modo per indurre una risposta fotoshock è quello di rimuovere rapidamente un filtro rosso a mano dal percorso della luce di osservazione. Tuttavia, questo metodo è più variabile in quanto la velocità di rimozione del filtro varia da persona a persona (Film 4,5). - Registra il movimento cellulare utilizzando un microscopio dotato di fotocamera.
- Per la quantificazione, eseguire una o entrambe le seguenti operazioni: (1) calcolare il rapporto tra le cellule che mostrano la risposta al fotoshock per le cellule totali16; (2) per le cellule che mostrano la risposta al fotoshock, misurare il tempo dallo stimolo del fotoshock al recupero del nuoto in avanti25.
Filmato 2: Illuminazione Photoshock da un flash della fotocamera. Il flash della fotocamera è stato tenuto fino allo stadio del microscopio e acceso. Clicca qui per scaricare questo film.
Filmato 3: Risposta al photoshock causata da un flash al microscopio. Le cellule sono state osservate sotto una debole luce rossa. Un flash è stato emesso a ~ 0 s. Quasi tutte le cellule hanno smesso di nuotare in avanti, hanno nuotato all'indietro per un breve periodo e hanno recuperato il nuoto in avanti. Barra della scala = 100 μm. Fare clic qui per scaricare questo filmato.
Filmato 4: Risposta al photoshock causata dalla rimozione di un filtro rosso al microscopio. Le cellule sono state osservate sotto una debole luce rossa. Il filtro rosso è stato rimosso a ~ 5 s. Quasi tutte le cellule hanno smesso di nuotare in avanti, hanno nuotato all'indietro per un breve periodo e hanno recuperato il nuoto in avanti. Barra della scala = 100 μm. Fare clic qui per scaricare questo filmato.
Filmato 5: Rimozione di un filtro rosso. Rimozione rapida di un filtro rosso impostato nel percorso della luce per fornire photoshock. Clicca qui per scaricare questo film.
Risultati
I tipici saggi di fototassi e risposta al fotoshock di C. reinhardtii sono mostrati qui. Dopo la stima della densità cellulare, la coltura cellulare wild-type (una progenie della croce CC-124 × CC-125 con agg1+ mt -)23 è stata lavata con una soluzione sperimentale di fotocomportamentare per il test della piastra di fototaxi. La sospensione cellulare è stata posta sotto una debole luce rossa per ~ 1 ora. Una sospensione cellulare da 2 ml è stata collocata in una capsula di Petri da 3,...
Discussione
Il presente protocollo è facile e non richiede molto tempo. Se si sospetta che un mutante di C. reinhardtii presenti difetti nella fotoricezione o nel moto ciliare, questo metodo potrebbe servire come analisi fenotipica primaria.
Tuttavia, esistono alcuni passaggi critici. Uno è quello di utilizzare le cellule nell'esperimento nella fase di crescita da iniziale a metà registro. Dopo aver coltivato per lunghi periodi, le cellule diventano meno mobili, meno sensibili alla luce e form...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo studio è stato supportato da sovvenzioni della Japan Society for the Promotion of Science KAKENHI (https://www.jsps.go.jp/english/index.html) a NU (19K23758, 21K06295), TH (16H06556) e KW (19H03242, 20K21420, 21H00420), dalla Ohsumi Frontier Science Foundation (https://www.ofsf.or.jp/en/) a KW, e dalla Dynamic Alliance for Open Innovation Bridging Human, Environment and Materials (http://alliance.tagen.tohoku.ac.jp/english/) a NU, TH e KW.
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL conical tube | SARSTEDT | 62.554.502 | |
| 5 mm Cannonball green LED | Optosupply | OSPG5161P | |
| 50 mL conical tube | SARSTEDT | 62.547.254 | |
| AC adaptor for the light box | ATTO | 2196161 | |
| Auto cell counter | DeNovix | CellDrop BF | |
| CaCl2 | Nakalai tesque | 06731-05 | |
| Camera flash | NEWWER | TT560 | |
| Centrifuge | KUBOTA | 2800 | |
| Chlamydomonas strains CC-124 and CC-125 | Chlamydomonas Resource Center | https://www.chlamycollection.org/ | |
| C-mout CCD camera | Wraymer | 1129HMN1/3 | |
| Desktop darkroom | Scientex | B-S8 | |
| Digital still camera | SONY | RX100II | |
| EGTA | Dojindo | G002 | |
| Fiji | https://fiji.sc/ | ||
| Green LED plate | CCS | ISLM-150X150-GG | |
| HCl | Fujifilm WAKO | 080-01066 | |
| HEPES | Dojindo | GB70 | |
| KCl | Nakalai tesque | 238514-75 | |
| Lightbox (Flat viewer) | ATTO | 2196160 | |
| Microscope | Olympus | BX-53 | |
| Petri dish (φ3.5 cm) | IWAKI | 1000-035 | |
| Pottasium acetate | Nakalai tesque | 28434-25 | |
| Power supply for the green LED plate | CCS | ISC-201-2 | |
| Red filter | Shibuya Optical | S-RG630 |
Riferimenti
- Demmig-Adams, B., Adams, W. W. Photoprotection and other responses of plants to high light stress. Annual Reviews Plant Physiology and Plant Molecular Biology. 43, 599-626 (1992).
- Wada, M. Chloroplast movement. Plant Science. 210, 177-182 (2013).
- Sgarbossa, A., Checcucci, G., Lenci, F. Photoreception and photomovements of microorganisms. Photochemical & Photobiological Sciences. 1 (7), 459-467 (2002).
- Ueki, N., et al. Eyespot-dependent determination of the phototactic sign in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 113 (19), 5299-5304 (2016).
- Foster, K. W., Smyth, R. D. Light antennas in phototactic algae. Microbiological Reviews. 44 (4), 572-630 (1980).
- Nagel, G., et al. Channelrhodopsin-1: a light-gated proton channel in green algae. Science. 296 (5577), 2395-2398 (2002).
- Nagel, G., et al. Channelrhodopsin-2, a directly light-gated cation-selective membrane channel. Proceedings of the National Academy of Sciences of the United States of America. 100 (24), 13940-13945 (2003).
- Sineshchekov, O. A., Jung, K. -. H., Spudich, J. L. Two rhodopsins mediate phototaxis to low- and high-intensity light in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 99 (13), 8689-8694 (2002).
- Suzuki, T., et al. Archaeal-type rhodopsins in Chlamydomonas: model structure and intracellular localization. Biochemical and Biophysical Research Communications. 301 (3), 711-717 (2003).
- Berthold, P., et al. Channelrhodopsin-1 initiates phototaxis and photophobic responses in Chlamydomonas by immediate light-induced depolarization. Plant Cell. 20 (6), 1665-1677 (2008).
- Deisseroth, K., et al. Next-generation optical technologies for illuminating genetically targeted brain circuits. Journal of Neuroscience. 26 (41), 10380-10386 (2006).
- Wakabayashi, K., Isu, A., Ueki, N. Channelrhodopsin-dependent photo-behavioral responses in the unicellular green alga Chlamydomonas reinhardtii. Optogenetics (Advances in Experimental Medicine and Biology), 2nd ed. , 21-33 (2021).
- Rüffer, U., Nultsch, W. Flagellar photoresponses of Chlamydomonas cells held on micropipettes: II. Change in flagellar beat pattern. Cell Motility and the Cytoskeleton. 18 (4), 269-278 (1991).
- Kamiya, R., Hasegawa, E. Intrinsic difference in beat frequency between the two flagella of Chlamydomonas reinhardtii. Experimental Cell Research. 173, 299-304 (1987).
- Kamiya, R., Witman, G. B. Submicromolar levels of calcium control the balance of beating between the two flagella in demembranated models of Chlamydomonas. Journal of Cell Biology. 98 (1), 97-107 (1984).
- Okita, N., Isogai, N., Hirono, M., Kamiya, R., Yoshimura, K. Phototactic activity in Chlamydomonas 'non-phototactic' mutants deficient in Ca2+-dependent control of flagellar dominance or in inner-arm dynein. Journal of Cell Science. 118, 529-537 (2005).
- Horst, C. J., Witman, G. B. ptx1, a nonphototactic mutant of Chlamydomonas, lacks control of flagellar dominance. Journal of Cell Biology. 120 (3), 733-741 (1993).
- Hyams, J. S., Borisy, G. G. Isolated flagellar apparatus of Chlamydomonas: characterization of forward swimming and alteration of waveform and reversal of motion by calcium ions in vitro. Journal of Cell Science. 33, 235-253 (1978).
- Bessen, M., Fay, R. B., Witman, G. B. Calcium control of waveform in isolated flagellar axonemes of Chlamydomonas. Journal of Cell Biology. 86 (2), 446-455 (1980).
- Reiter, J. F., Leroux, M. R. Genes and molecular pathways underpinning ciliopathies. Nature Reviews Molecular Cell Biology. 18 (9), 533-547 (2017).
- Ide, T., et al. Identification of the agg1 mutation responsible for negative phototaxis in a "wild-type" strain of Chlamydomonas reinhardtii. Biochemistry and Biophysics Reports. 7, 379-385 (2016).
- Gorman, D. S., Levine, R. P. Cytochrome f and plastocyanin: their sequence in the photosynthetic electron transport chain of Chlamydomonas reinhardi. Proceedings of the National Academy of Sciences of the United States of America. 54 (6), 1665-1669 (1965).
- Finst, R. J., Kim, P. J., Quarmby, L. M. Genetics of the deflagellation pathway in Chlamydomonas. Genetics. 149 (2), 927-936 (1998).
- Morel-Laurens, N. Calcium control of phototactic orientation in Chlamydomonas reinhardtii: sign and strength of response. Photochemistry and Photobiology. 45 (1), 119-128 (1987).
- Wakabayashi, K., King, S. M. Modulation of Chlamydomonas reinhardtii flagellar motility by redox poise. Journal of Cell Biology. 173 (5), 743-754 (2006).
- Wakabayashi, K., Misawa, Y., Mochiji, S., Kamiya, R. Reduction-oxidation poise regulates the sign of phototaxis in Chlamydomonas reinhardtii. Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11280-11284 (2011).
- Harris, E. H. . in The Chlamydomonas Sourcebook Second Edition. 1, 25-64 (2009).
- Mergenhagen, D. Circadian clock: genetic characterization of a short period mutant of Chlamydomonas reinhardii. European Journal of Cell Biology. 33 (1), 13-18 (1984).
- Ozasa, K., Lee, J., Song, S., Hara, M., Maeda, M. Two-dimensional optical feedback control of Euglena confined in closed-type microfluidic channels. Lab on a Chip. 11 (11), 1933-1940 (2011).
- Tanno, A., et al. The four-celled Volvocales green alga Tetrabaena socialis exhibits weak photobehavior and high-photoprotection ability. PLoS One. 16 (10), 0259138 (2021).
- Ueno, Y., Aikawa, S., Kondo, A., Akimoto, S. Adaptation of light-harvesting functions of unicellular green algae to different light qualities. Photosynthesis Research. 139 (1-3), 145-154 (2019).
- Takahashi, T., Watanabe, M. Photosynthesis modulates the sign of phototaxis of wild-type Chlamydomonas reinhardtii. Effects of red background illumination and 3-(3',4'-dichlorophenyl)-1,1-dimethylurea. FEBS Letters. 336 (3), 516-520 (1993).
- Morishita, J., Tokutsu, R., Minagawa, J., Hisabori, T., Wakabayashi, K. I. Characterization of Chlamydomonas reinhardtii mutants that exhibit strong positive phototaxis. Plants (Basel). 10 (7), (2021).
- Fujiu, K., Nakayama, Y., Yanagisawa, A., Sokabe, M., Yoshimura, K. Chlamydomonas CAV2 encodes a voltage- dependent calcium channel required for the flagellar waveform conversion. Current Biology. 19 (2), 133-139 (2009).
- Inaba, K. Calcium sensors of ciliary outer arm dynein: functions and phylogenetic considerations for eukaryotic evolution. Cilia. 4 (1), 6 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon