Method Article
Monitoreo de respuestas de señalización en el objetivo en larvas de pez cebra - Z-REX desenmascara mecanismos precisos de fármacos electrofílicos y metabolitos
En este artículo
Resumen
El pez cebra dirigido a electrófilos y oxidantes reactivos (Z-REX) es un método basado en la biología química para la investigación de la señalización reactiva de moléculas pequeñas. Esta técnica se puede aplicar a peces vivos de diferentes etapas de desarrollo. Aquí, combinamos ensayos estándar en pez cebra con Z-REX para el análisis de la vía de señalización.
Resumen
Los metabolitos reactivos y los fármacos electrofílicos relacionados se encuentran entre las moléculas pequeñas más difíciles de estudiar. Los enfoques convencionales para deconstruir el modo de acción (MOA) de tales moléculas aprovechan el tratamiento a granel de especímenes experimentales con un exceso de una especie reactiva específica. En este enfoque, la alta reactividad de los electrófilos hace que el etiquetado no discriminatorio del proteoma dependa del tiempo y el contexto; Las proteínas y procesos sensibles a redox también pueden verse afectados indirectamente y, a menudo, irreversiblemente. En este contexto de innumerables objetivos potenciales y efectos secundarios indirectos, vincular el fenotipo con el compromiso objetivo específico sigue siendo una tarea compleja. El pez cebra dirigido a electrófilos y oxidantes reactivos (Z-REX), una plataforma de administración de electrófilos reactivos a pedido adaptada para su uso en larvas de pez cebra, está diseñada para administrar electrófilos a una proteína específica de interés (POI) en embriones de peces vivos imperturbables. Las características clave de esta técnica incluyen un bajo nivel de invasividad, junto con la administración de electrófilos de precisión controlada por dosificación, quimiotipo y espaciotemporalmente. Por lo tanto, junto con un conjunto único de controles, esta técnica evita los efectos fuera del objetivo y la toxicidad sistémica, observada de otro modo después de la exposición masiva incontrolada de animales a electrófilos reactivos y fármacos electrofílicos pleiotrópicos. Aprovechando Z-REX, los investigadores pueden establecer un punto de apoyo en la comprensión de cómo las respuestas individuales al estrés y las salidas de señalización se alteran como resultado del compromiso específico del ligando reactivo con un POI específico, en condiciones casi fisiológicas en animales vivos intactos.
Introducción
Una miríada de eventos de señalización celular implican reacciones entre pequeñas moléculas reactivas (producidas endógenamente en la célula o xenobióticos / xenometabolitos, como los medicamentos) y su proteína diana. En muchos casos, un nivel subestequiométrico de tales eventos de unión covalente puede desencadenar respuestas celulares, lo que lleva a cambios en, por ejemplo, el desarrollo, el metabolismo, la apoptosis y / o la respuesta inmune1. Sin embargo, deconstruir el modo de acción (MOA) vinculando eventos de unión específicos a sus consecuencias fenotípicas ha demostrado ser un desafío. Los métodos tradicionales de dosificación en bolo que implican la introducción de altas concentraciones de las especies reactivas a menudo resultan en una multitud de proteínas modificadas, así como una toxicidad excesiva para el organismo modelo2. Tales condiciones están lejos de ser ideales. Se desarrolló un método para resolver estos problemas en cultivo celular utilizando la administración de electrófilos localizados de precisión en un contexto celular nativo, llamado T-REX (electrófilos y oxidantes reactivos dirigibles)3. En los años intermedios, la atención se ha centrado en experimentos en organismos completos, que permiten la oportunidad de estudiar proteínas en contextos celulares específicos en células no transformadas. Así, hemos ampliado la técnica para que sea compatible con varios modelos, incluidos los modelos de embriones de Danio rerio . Aquí, presentamos Z-REX (pez cebra dirigido a electrófilos y oxidantes reactivos) (Figura 1).
Para comprender Z-REX, este artículo primero presenta las tecnologías REX y sus conceptos subyacentes. En esencia, estas técnicas modelan la señalización de especies electrofílicas reactivas fisiológicas endógenas (RES) imitando cómo los electrófilos naturales se producen localmente in vivo con precisión espaciotemporal. La proteína de interés (POI) se expresa como una construcción de fusión a Halo; este último ancla la sonda inerte y permeable al tejido que soporta la RES fotoenjaulada en una estequiometría 1:1. Una de estas RES endógenas es 4-hidroxinonenal (HNE en adelante), que está fotoenjaulada en la sonda Ht-PreHNE. En muchos casos, utilizamos una versión funcionalizada de alquino de HNE [es decir, HNE (alquino)], que tiene propiedades biológicas esencialmente idénticas a HNE, pero puede ser etiquetada por química de clic. La sonda, que también está funcionalizada con un cloroalcano para la reactividad con Halo, se conoce como Ht-PreHNE (alquino). El complejo de la fusión Halo-POI y la sonda así formada permite la entrega proximal de RES a la POI fusionada tras la irradiación con luz UV. Si el POI reacciona rápidamente con el RES liberado, el etiquetado covalente resultante del POI con RES nos permite identificar cisteínas cinéticamente privilegiadas.
Z-REX toma las ventajas antes mencionadas de las tecnologías REX y las aplica ampliamente para estudiar vías de señalización específicas en peces vivos. Este protocolo ha sido optimizado para el pez cebra (D. rerio), ya que son organismos vertebrados genéticamente tratables que son transparentes durante el desarrollo y, por lo tanto, ideales para técnicas optoquímicas / genéticas como las tecnologías REX. Sin embargo, es probable que una estrategia similar también funcione bien en otras especies de peces genéticamente tratables, ya que la amplia aplicabilidad del método se debe al mecanismo pseudo-intramolecular de la administración de electrófilos derivados de lípidos (LDE). De hecho, el procedimiento es altamente biocompatible, ya que los peces pueden ser tratados con el electrófilo fotoenjaulado Z-REX [por ejemplo, Ht-PreHNE (alquino)] durante al menos 48 h sin ningún impacto notable en el desarrollo. Un protocolo similar funciona en C. elegans 4,5.
El protocolo describe primero el uso de la inyección de ARNm para producir una expresión transitoria de una construcción de fusión Halo-POI no nativa en modelos embrionarios de pez cebra, 1-1.5 días después de la fertilización (dpf). Esto da como resultado la expresión de la proteína ectópica en la mayoría de las células dentro del pez (en lo sucesivo denominadas "ubicuas"), en lugar de en tejidos o lugares específicos; Sin embargo, los datos muestran que se pueden observar efectos específicos de las células en ciertos casos. Después de la inyección, los embriones se incuban con una baja concentración [0.3-5 μM Ht-PreHNE (alquino)] de la sonda hasta 30.5 h después de la fertilización (hpf). Luego, en un momento prescrito por el usuario, la entrega del RES al POI dentro de los peces se logra mediante fotounjaula durante 2-5 min. Después del fotouncado de la RES, se pueden realizar una variedad de ensayos fenotípicos aguas abajo durante las próximas 2-10 h: 1) imágenes en vivo de las líneas del reportero (Figura 2A); 2) evaluación del etiquetado de objetivos mediante análisis de Western blot (Figura 3); 3) análisis transcriptómico (Figura 4); o 4) inmunofluorescencia de montaje completo (Figura 5).
Como ejemplo de imágenes en vivo de líneas reporteras, Z-REX se demuestra en conjunto con imágenes en vivo de líneas de peces, Tg (lyz: TagRFP) y Tg (mpeg1: eGFP), para medir cómo la modificación RES de un POI específico del sensor electrófilo (a saber, Keap1) disminuye los niveles de neutrófilos y macrófagos, respectivamente, sin efectos observables en otras células de los peces. Sin embargo, hemos demostrado previamente que el marcado de POI y la consiguiente señalización de la vía de los estudios T-REX se pueden reproducir utilizando Z-REX para varias proteínas: Akt3 6, Keap17 y Ube2v26. En general, con Z-REX, los científicos pueden estudiar las consecuencias de la modificación covalente de los POI por RES en el contexto de varias vías redox complejas. Esta técnica está preparada para identificar objetivos y sus residuos funcionales para el diseño de fármacos covalentes y nuevos mecanismos farmacológicos en un modelo animal completo más relevante contextualmente.
Protocolo
Los procedimientos de cría y manejo del pez cebra en la Universidad de Cornell (Estados Unidos) se realizaron siguiendo las directrices de los Institutos Nacionales de Salud (NIH) y aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC). Los procedimientos de cría y manipulación del pez cebra en la unidad de pez cebra del Instituto Federal Suizo de Tecnología de Lausana (EPFL, Suiza) se realizaron siguiendo la Ley de Bienestar Animal SR 455 y la Ordenanza de Bienestar Animal SR 455.1, con autorización veterinaria cantonal VD-H23.
NOTA: En este protocolo, las líneas de peces Tg( lyz:TagRFP ) y Tg(mpeg1:EGFP) que expresan Halo-TeV-Keap1 se utilizan para demostrar Z-REX. El método se puede extender a otras proteínas de interés, líneas de peces reporteros transgénicos y ensayos biológicos aguas abajo. Consulte la Tabla suplementaria 1 para conocer los tampones utilizados en este estudio. Todos los reactivos, instrumentos, equipos, anticuerpos, plásmidos, cepas de pez cebra y equipos se enumeran en la Tabla de materiales.
1. Preparación del ARNm
- Prepare el ARNm Halo-TeV-Keap1-2xHA y Halo-2xHA-P2A-Keap1-2xHA utilizando el kit de transcripción in vitro mMessage mMachine SP6.
NOTA: Siga las instrucciones del fabricante y realice reacciones de escala de 40 μL para cada ARNm. Vuelva a disolver el gránulo de ARNm en 10 μL de agua libre de nucleasa. - Evaluar la calidad del ARNm y medir la concentración mediante espectrofotómetro de microvolumen y electroforesis en gel de agarosa. Un ARNm de buena calidad debe tener una relación A260/A280 de alrededor o por encima de 2.0.
- Diluir el ARNm a 1-1,5 mg/ml con agua libre de nucleasas.
- Alícuota la solución de ARNm (1-2 μL por tubo) y conservar las alícuotas a -80 °C.
2. Producción de embriones de peces
- Opción 1: Producir embriones de peces de tipo silvestre (WT).
- Establezca 10 pares de cruce de peces en 10 tanques separados, y cada tanque contiene un divisor entre los peces padres machos y hembras.
NOTA: Un total de 10 pares de cruce típicamente proporciona un número suficiente de embriones para los ensayos. El número de pares de cruce se puede ajustar de acuerdo con el diseño / necesidad del experimento y la fecundidad de los peces parentales. - A la mañana siguiente, después de instalar el inyector, retire los divisores en cinco de los tanques. Espere 30 minutos para que los peces se apareen.
- Mueva el pez padre a otro tanque, recolecte embriones pasando el agua del tanque a través de un colador y luego enjuague los embriones del colador en una placa de Petri de 10 cm. Estos embriones se utilizarán para la primera ronda de inyecciones.
NOTA: Si la calidad del huevo de un determinado lote es pobre (por ejemplo, los huevos son opacos debido a la agregación de proteínas), no los agrupe con los otros embriones. - (Opcional) Realizar procedimientos similares a los descritos en los pasos 2.1.2-2.1.3 en los otros cinco tanques para la siguiente ronda de inyección.
NOTA: Lo mejor es realizar solo una ronda de inyección, para minimizar la diferencia de edad entre embriones. Sin embargo, si se requieren más embriones de los que se pueden inyectar en un lote, se recomienda realizar dos rondas de inyección para garantizar que los embriones permanezcan en una etapa de 1-4 células durante la inyección de ARNm. El número de rondas de inyección se puede ajustar de acuerdo con la habilidad de inyección del operador y el diseño del experimento. Sin embargo, se sugiere realizar todo el procedimiento (desde la extracción del primer divisor hasta la inyección del último embrión) dentro de las 2 h. Una gran diferencia de edad entre los embriones puede afectar la fiabilidad y reproducibilidad de los resultados del experimento.
- Establezca 10 pares de cruce de peces en 10 tanques separados, y cada tanque contiene un divisor entre los peces padres machos y hembras.
- Opción 2: Producir embriones de peces reporteros de neutrófilos/macrófagos transgénicos heterocigotos.
- Configure 10 pares de cruce de peces en 10 tanques separados, con cada tanque insertado con un divisor entre peces padres machos y hembras: peces WT versus Tg(lyz: TagRFP), o peces WT versus Tg (mpeg1: eGFP).
NOTA: Evite los cruces entre peces transgénicos heterocigotos, que pueden afectar las lecturas fluorescentes aguas abajo, ya que los peces reporteros homocigotos muestran una señal fluorescente más alta en comparación con los peces heterocigotos. Las líneas reporteras transgénicas y los embriones WT se pueden distinguir fácilmente cuando se toman imágenes. Tener una mezcla de embriones WT y heterocigotos en el mismo grupo no es un problema. lyz:TagRFP informa de neutrófilos, y mpeg:eGFP informa de macrófagos. Este protocolo también se puede aplicar a otras líneas de peces reporteras. - Siga los pasos 2.1.2-2.1.4.
- Configure 10 pares de cruce de peces en 10 tanques separados, con cada tanque insertado con un divisor entre peces padres machos y hembras: peces WT versus Tg(lyz: TagRFP), o peces WT versus Tg (mpeg1: eGFP).
3. Configuración del microinyector
- Encienda la fuente de aire, ajuste la contrapresión a 0.2-0.5 psi y establezca la presión de inyección a 25-30 psi. Por lo general, se recomienda el rango específico de presión que se muestra.
NOTA: Es esencial tener una contrapresión estable para evitar que los medios de pescado se refluyan hacia la aguja. Al calibrar el volumen de inyección en el paso 3.8, sólo debe cambiarse el tiempo de inyección. No cambie la presión de inyección en los siguientes pasos; La baja presión de inyección puede conducir a fallas de inyección debido a la tensión superficial e interfacial, mientras que la alta presión de inyección puede dañar los embriones. - Limpie el equipo y la plataforma de inyección con la solución de descontaminación RNasa.
NOTA: La RNasa, que degrada el ARNm, podría provenir del operador o del equipo. Es necesario realizar la limpieza antes del experimento y usar guantes. - (Opcional) Si se inyectan conjuntamente ARNm y morfolino, premezcle los dos en un tubo de 0,2 ml.
NOTA: Z-REX funciona bien utilizando la solución de ARNm Halo-TeV-Keap1-2xHA con una concentración de 250-1500 ng / μL. Varios morfolinos también han sido utilizados en el pez cebra, y las concentraciones óptimas han sido reportadas7. Si se utiliza un morfolino con una secuencia no publicada, el operador debe evaluar primero la toxicidad y la eficiencia de eliminación de genes del morfolino antes de usarlo en Z-REX. - Transfiera 1-2 μL de ARNm (y/o morfolino cuando corresponda7) a una aguja de inyección con una punta de pipeta de microcargador.
NOTA: Si prepara agujas con un extractor de micropipeta Flaming/Brown, la configuración es la siguiente. Calor: 520 unidades; fuerza de tracción: 60 unidades; velocidad: 70 unidades; retardo: 155 unidades; presión: 550 unidades; Rampa: 530 unidades. - Instale la aguja en el manipulador de microinyección.
NOTA: La contrapresión de la fuente de aire debe empujar la solución de ARNm (/ morfolino) a la punta de la aguja. - Use pinzas afiladas o una cuchilla de afeitar para romper la punta de la aguja, creando una abertura adecuada para la inyección.
- Sumerja la punta de la aguja en aceite mineral en un micrómetro de etapa.
- Aplique dos o tres pulsos de inyección para eliminar las burbujas de aire en la punta.
- Calibre el tamaño de la gota a 2 nL cambiando el tiempo de inyección.
NOTA: Esto se realiza mejor inyectando en aceite mineral (que imita la viscosidad del saco vitelino) colocado sobre un hemocitómetro. Usando un microscopio, use las líneas de cuadrícula del hemocitómetro para estimar el tamaño de la gota formada durante la inyección y ajuste el tiempo de inyección en consecuencia. Aunque a veces se usa colorante rojo fenol, no se ha observado su necesidad en el procedimiento de inyección de ARNm descrito aquí.
4. Microinyección
- Llene una placa de inyección con medio fresco de solución salina balanceada (HBSS) de Hank al 10% y alinee los embriones en las ranuras de la placa con pinzas romas.
NOTA: La placa de inyección se prepara con agarosa al 2% en medio HBSS al 10%; Las ranuras se forman utilizando un molde de plástico. - Sumerja la punta de la aguja en un medio HBSS al 10% en la placa de inyección.
- Aplique dos o tres pulsos de inyección para eliminar las burbujas de aire en la punta.
- Para cada inyección, penetre el corion y el saco vitelino en un solo movimiento y aplique un pulso de inyección. Este líquido inyectado se puede ver inmediatamente después de la inyección como un pequeño esferoide dentro del saco vitelino. Este pequeño esferoide se disipa con relativa rapidez. Repita este paso para otros embriones, hasta que se haya obtenido un número suficiente de embriones inyectados.
NOTA: La tasa de supervivencia de los embriones (inyectados y no inyectados) varía típicamente del 50% al 90%. Intente inyectar el doble del número de embriones necesarios para cada grupo de control / experimental. En el ensayo de extracción de biotina, se requieren 100-140 embriones viables para cada condición. En el ensayo qRT-PCR, se recomiendan cinco embriones viables para cada condición. El tamaño de la muestra para imágenes de peces vivos y ensayo de tinción de inmunofluorescencia de montaje completo está definido por el usuario; Se recomienda tener al menos 20 embriones viables por condición para obtener un buen poder estadístico en el análisis. - Enjuague los embriones inyectados en una nueva placa de Petri de 10 cm que contenga medios frescos al 10% de HBSS.
NOTA: Los embriones se pueden enjuagar fácilmente de las ranuras con una botella de chorro. - Agrupe los embriones no inyectados en otra placa.
NOTA: Los embriones no inyectados pueden servir como controles de calidad para la salud de los peces, la expresión proteica basal, los niveles de fluorescencia de fondo, etc., según sea necesario. Si el procedimiento de inyección funcionó bien y el ARNm/morfolino inyectado no es letal para los embriones, los embriones inyectados y no inyectados deben tener una viabilidad similar.
5. Z-REX
- Distribuya los embriones inyectados en platos de 10 cm, de acuerdo con la configuración del experimento (es decir, el número de grupos de control / experimento).
- En una habitación oscura con iluminación de luz roja, reemplace el medio con 30 ml de HBSS al 10% con o sin 1 μM Ht-PreHNE (alquino).
NOTA: Ht-PreHNE (alquino) es un compuesto lábil ligero. Los embriones deben mantenerse en la oscuridad en los siguientes pasos. - Incubar los embriones a 28,5 °C en la oscuridad.
- A 30.5 hpf, en una habitación oscura, reemplace el medio con 30 ml frescos de HBSS al 10%.
NOTA: Al reemplazar el medio, retire la mayor cantidad posible del medio antiguo. Esto es crítico para eliminar la cantidad no unida / excesiva de Ht-PreHNE (alquino) de los embriones. - Incubar los embriones a 28,5 °C en la oscuridad durante 30 min.
- Repita los pasos 5.4-5.5 dos veces más.
- Encienda la lámpara UV (365 nm, 3 mW/cm2) durante 5 min para precalentar la lámpara.
NOTA: El paso de precalentamiento de la lámpara debe realizarse antes del paso 5.8. La potencia de la lámpara es menor/inestable en los primeros minutos después de encenderse. La potencia de la lámpara debe medirse regularmente con un medidor UV. - A 32 hpf, exponga los embriones a la luz UV.
- Opción 1: Para lecturas posteriores, como imágenes de peces vivos, tinción de inmunofluorescencia de montaje completo, análisis de fluorescencia en gel (acoplamiento de clic con azida Cy5), RNA-seq o qRT-PCR, exponga los embriones a la luz UV durante 3 minutos, girando las placas cada 30 s.
- Opción 2: Para lecturas posteriores, como el ensayo de extracción de biotina, exponga los embriones a la luz UV durante un máximo de 5 minutos (y un mínimo de 3 minutos), agitando las placas cada 30 s y enfríe las placas en hielo durante 1 minuto.
NOTA: Si se utilizan diferentes sondas, el tiempo de exposición a la luz debe optimizarse, dependiendo de la t 1/2 de fotouncaging para una sonda de electrófilo fotojaula dada y la fuente de luz desplegada. t1/2fotouncaging puede determinarse utilizando procedimientos conocidos8. Para Ht-PreHNE(alquino), t 1/2 es < 1 min3; Por lo tanto, el tiempo indicado anteriormente es suficiente.
6. Ensayos posteriores
- Opción 1: Ensayo fenotípico. Imágenes en vivo de un reportero transgénico de neutrófilos/macrófagos
líneas de pescado, Tg( lyz:TagRFP ) y Tg(mpeg1:eGFP ) (Figura 2)- Anestesiar los embriones por incubación a 4 °C durante 10 min.
NOTA: Se recomienda tener al menos 20 embriones viables por condición. - Retire los embriones no fertilizados/muertos de la placa.
NOTA: Los embriones no fertilizados/muertos son turbios/no transparentes, y pueden ser identificados visualmente. Si se observa una tasa de mortalidad alta, vuelva al procedimiento de inyección o intente reducir la concentración de ARNm o morfolino. - Decorionar los embriones con fórceps afilados. Sostenga el corion con un par de fórceps, sin tocar el pez larval, y use el otro par de fórceps para arrancar el corion. El embrión es frágil. Solo toque el corion cuando realice la decorionación.
NOTA: Es común, especialmente en principiantes, dañar algunos embriones durante la decorionación. Por lo tanto, siempre tenga más embriones que el mínimo necesario. - Monte los embriones en una placa de agarosa al 2% (preparada con medio HBSS al 10%) y obtenga imágenes de los embriones con un microscopio estereoscópico (campo brillante y canales fluorescentes respectivos) (Figura 2A, C, G).
NOTA: Después de Z-REX [combinación de inyección de ARNm Halo-TeV-Keap1-2xHA y tratamiento con Ht-PreHNE(alquino)], se encontró que el agotamiento de neutrófilos (lyz: TagRFP) era más significativo a 36 hpf (4 h después de Z-REX), mientras que la reducción de macrófagos (mpeg1: eGFP) fue más significativa a 34 hpf (2 h después de Z-REX) (Figura 2E, F). Se pueden usar otros puntos de tiempo si se usan diferentes líneas reporteras, ARNm/morfolino o sondas. El tiempo de exposición y/o la ganancia deben optimizarse para visualizar células individuales o las (ultra)estructuras específicas deseadas. - Contar el número de neutrófilos/macrófagos de cada pez por ImageJ (NIH) (Figura 2B, D-F, H). Utilice la herramienta Selección a mano alzada en ImageJ para rodear el pez entero y utilice la opción Buscar máximos para contar las células fluorescentes.
- Anestesiar los embriones por incubación a 4 °C durante 10 min.
- Opción 2: Evaluación del etiquetado de objetivos. Acoplamiento azida-clic de biotina y ensayo de extracción de biotina (Figura 3)
- Anestesiar los embriones por incubación a 4 °C. Esto generalmente toma 10 minutos.
NOTA: Para obtener suficiente lisado de peces, se requieren 100-140 embriones viables para cada condición. - Retire los embriones no fertilizados/muertos de la placa.
- Realizar dechorionation y deyolking. Realice las dos manipulaciones sosteniendo el corion con un par de pinzas afiladas, usando el otro par de pinzas para penetrar el saco vitelino y luego separando el saco vitelino mientras retira el corion para permitir que salgan las proteínas de la yema.
NOTA: Es fundamental eliminar las proteínas de la yema en este paso. Las abundantes proteínas de la yema en la muestra interfieren con el análisis posterior. - Transfiera los embriones desyemas a un tubo de 1,5 ml.
NOTA: Agite la placa para centrar los embriones desyemas, lo que facilita la recolección. Los restos de corion más ligeros se eliminan durante este proceso. - Después de que los embriones se asienten en el fondo del tubo, retire el sobrenadante y agregue 1 ml de solución salina tamponada con HEPES refrigerada (pH 7.6).
- Repita el paso 6.2.5 dos veces más.
- (Opcional) Si no tiene la intención de continuar con el siguiente paso inmediatamente, retire el tampón, congele rápidamente las muestras en nitrógeno líquido y guárdelas a -80 ° C.
NOTA: Los pellets de pescado desyomados pueden congelarse rápidamente en nitrógeno líquido y almacenarse a -80 °C. Las muestras congeladas pueden almacenarse a -80 °C durante un máximo de 2 semanas. - Resuspender los pellets de pescado en tampón de lisis.
NOTA: El tampón de lisis (pH 7.6) está compuesto por 50 mM HEPES, 100 mM NaCl, 1% Triton X-100, 0.3 mM TCEP, 2x Roche cOmplete Mini inhibidores de proteasa libre de EDTA y 0.1 mg / ml inhibidor de tripsina de soja. Un embrión da alrededor de 2 μg de lisado. Use 100 μL de tampón de lisis por cada 120 embriones. Dos inhibidores de la proteasa libres de EDTA Roche cOmplete Mini e inhibidores de tripsina de soja deben agregarse al tampón de lisis justo antes de su uso. - Agregue perlas de zirconia al 20% v/v al tubo.
- Vórtice durante 20 s, congelación instantánea en nitrógeno líquido y descongelación en un baño maría a 37 °C.
- Repita el paso 6.2.10 otras dos veces.
- Centrifugar la solución a 21.000 g a 4 °C durante 10 min.
- Transfiera el sobrenadante a un nuevo tubo preenfriado de 1,5 ml.
- Mida la concentración de proteína mediante el ensayo de Bradford.
- Diluir el lisado a 1 mg/ml.
- Para cada condición, transfiera 170 μg de lisado a un tubo de 2 ml.
- Mezclar el lisado del paso 6.2.16 con 0,2 mg/ml de proteasa TeV (S219V) e incubar la solución a 37 °C durante 30 min.
NOTA: Para grupos no tratados con proteasa TeV, simplemente mezcle el lisado con un volumen igual de tampón de lisis a la solución de proteasa TeV utilizada en otros grupos. - Prepare una mezcla maestra 10x para la reacción de clic biotina-azida: 10% p/v SDS, 10 mM CuSO4, 1 mM Cu(TBTA), 1 mM biotina-azida y 20 mM TCEP.
NOTA: Agregue TCEP a la mezcla justo antes del paso 6.2.19. - Añadir 8,5 μL de t-BuOH y 17 μL de mezcla maestra de reacción de clic 10x al lisado (digerido por proteasa TeV) del paso 6.2.17. Vortex, centrifugar e incubar la solución a 37 °C durante 15 min.
- Agregue otro TCEP de 1 mM a la solución, y luego vórtice, centrifugar e incubar la solución a 37 ° C durante otros 15 minutos. El tiempo de incubación para los pasos 6.2.19-6.2.20 es de 30 min en total.
NOTA: Este suplemento de TCEP, un reactivo reductor para generar Cu(I), mejora la eficiencia de la reacción de clic. - Añadir 600 μL de etanol a -20 °C a cada tubo, vorátice la solución e incubarla a -80 °C durante la noche.
NOTA: Las muestras pueden almacenarse a -80 °C durante 1 semana; Si no es así, continúe con el siguiente paso inmediatamente. - Centrifugar la solución a 21.000 g a 4 °C durante 1 h.
NOTA: Se debe formar un pellet en el fondo del tubo después de la centrifugación, que es la fracción deseada. - Retire el sobrenadante, agregue 1 ml de etanol a -20 °C, vórtice, y centrifugar la solución a 21,000 x g a 4 °C durante 10 min.
- Repita el paso 6.2.23.
- Retirar el sobrenadante, añadir 1 ml de acetona a -20 °C, vórtice, y centrifugar la solución a 21.000 x g a 4 °C durante 10 min.
- Retire el sobrenadante. Permita que el exceso de acetona residual se evapore, aunque no completamente a la sequedad.
- Resuspender el pellet en 100 μL de tampón de resuspensión (8% p/v dodecil sulfato de litio [LDS], 1 mM EDTA en solución salina HEPES de 50 mM, pH 7.6), vórtice durante 15 s y sonicar hasta que el pellet se disuelva.
- Centrifugar la solución a 21.000 g a temperatura ambiente (RT) durante 5 min.
- Transfiera el sobrenadante a un nuevo tubo de 2 ml y agregue 1,5 ml de solución salina HEPES de 50 mM, pH 7,6.
NOTA: La concentración final de LDS en este paso es de 0.5%. Las concentraciones más altas de LDS pueden socavar la eficiencia de descenso. Por lo tanto, aunque el aumento de la concentración LDS puede ayudar a la reducción de la unión no específica, también puede reducir la eficiencia de la distracción. En consecuencia, se recomienda no cambiar la concentración LDS en este paso. - Recolectar la muestra de "entrada" (Figura 3): transferir 30 μL de lisado de 1 mg/ml a un nuevo tubo de 1,5 ml y añadir 10 μL de tampón de muestra de Laemmli 4x que contiene 6% de β-mercaptoetanol (BME). Congele rápidamente la solución y guárdela a -80 °C.
- Transfiera 100 μL de resina de alta capacidad de estreptavidina de volumen en cama a un nuevo tubo de 2 ml. Añadir 1 ml de LDS al 0,5% en solución salina HEPES de 50 mM (pH 7,6), centrifugar a 1.500 x g a RT durante 2 min y retirar el sobrenadante. Repita el lavado con otro 1 ml de LDS al 0,5% en solución salina HEPES de 50 mM (pH 7,6).
- Transfiera la solución del paso 6.2.29 al tubo que contiene resina de alta capacidad de estreptavidina prelavada del paso 6.2.31 e incube la solución en un mezclador de extremo a extremo a RT durante 4-6 h.
- Centrifugar la mezcla a 1.500 x g a RT durante 2 min, tomar 30 μL del sobrenadante y mezclar con 10 μL de tampón de muestra Laemmli 4x que contenga 6% de BME para la muestra de "flujo continuo". Luego, retire el sobrenadante restante.
NOTA: Las muestras de "flujo continuo" se pueden analizar mediante Western Blot para verificar la eficiencia de extracción de estreptavidina. Es esencial eliminar la mayor cantidad posible de sobrenadante para eliminar las proteínas no unidas. Primero, retire la mayor parte del sobrenadante con una pipeta P-1000 y luego retire el sobrenadante restante con una pipeta P-20 con una punta de carga de gel. - Agregue 1 ml de LDS al 0,5% en solución salina HEPES de 50 mM (pH 7,6) a la resina, e incube la mezcla durante 30 minutos en RT con rotación de extremo a extremo.
- Centrifugar la mezcla a 1.500 x g a RT durante 2 min, y retirar el sobrenadante.
NOTA: Por lo general, 0.5% LDS es suficiente para eliminar la mayoría de las proteínas de unión no específicas. Si todavía se observan señales de unión no específicas en el análisis posterior, se puede aumentar la concentración LDS en el tampón de lavado. - Repita los pasos 6.2.34-6.2.35 dos veces más.
- Añadir 40 μL de 2 tampones de muestra Laemmli que contengan un 6% de BME a la resina.
- Eluya las proteínas unidas incubando la mezcla a 98 °C durante 5 min.
- Centrifugar la mezcla a 21.000 x g a RT durante 5 min, y transferir el sobrenadante a un nuevo tubo de 1,5 ml. Esta es la muestra "elute".
NOTA: La carga directa de la solución que contiene resina del paso 6.2.38 en el gel puede afectar al análisis SDS-PAGE. - Cargue 20 μL en cada pocillo de gel de poliacrilamida al 10% de 10 carriles y ejecute electroforesis en gel.
NOTA: Haga correr el gel a un voltaje más bajo (120 V) hasta que el frente del tinte alcance el gel de resolución y cambie el voltaje a 170 V. Detenga el programa después de que salga el frente del tinte. - Realice Western blot con anticuerpos anti-HA, anti-Halo u otros anticuerpos que detecten proteínas de mantenimiento (Figura 3).
- Anestesiar los embriones por incubación a 4 °C. Esto generalmente toma 10 minutos.
- Opción 3: Análisis transcriptómico. RNA-seq y qRT-PCR (Figura 4)
NOTA: Se recomienda encarecidamente utilizar embriones colocados dentro de los 15 minutos uno del otro para este ensayo. La diferencia de edad de los embriones afecta significativamente los resultados del ensayo.- Anestesiar los embriones incubándolos a 4 °C durante 10 min, 2 h después de Z-REX.
- Realizar la decorionación con fórceps afilados (paso 6.1.3).
- (Opcional) Realice la segmentación con fórceps (por ejemplo, separe la cabeza de la cola) si se van a analizar diferentes segmentos por separado.
- Si extrae ARN de un embrión completo, transfiera de tres a cinco embriones a un tubo de 1,5 ml. Si extrae ARN de la cabeza o la cola, transfiera 10-12 segmentos disecados a un tubo de 1,5 ml.
NOTA: Se recomienda realizar el experimento con tres a cinco réplicas biológicas. - Agregue 1 ml de reactivo TRIzol y perlas de vidrio al tubo.
NOTA: Se encontró que las perlas de vidrio funcionan mejor que las perlas de zirconia para la extracción de ARN. - Vortex la mezcla durante 30 s.
NOTA: Si no se continúa con el siguiente paso inmediatamente, la solución se puede almacenar a -80 °C durante 1-3 semanas. - Extraiga el ARN de acuerdo con las instrucciones del fabricante.
- Evaluar la calidad y concentración de ARN mediante espectrofotómetro de microvolumen y electroforesis en gel de agarosa.
NOTA: El ARN de buena calidad debe tener una relación A260/A280 de alrededor o por encima de 2.0. - Enviar el ARN para secuenciación, o tratar 1 μg de ARN con DNasa I de grado de amplificación, y transcribir inversamente usando transcriptasa inversa superíndice III y oligo-(dT)20. Realice este paso de acuerdo con las instrucciones del fabricante.
- Realizar qRT-PCR y analizar los datos por el método ΔΔCT9 (Figura 4B-D).
- Opción 4: Análisis de expresión y colocalización de POI. Ensayo de tinción de inmunofluorescencia de montaje completo (Figura 5)
NOTA: Los embriones fijados en formaldehído son frágiles. Evite las sacudidas vigorosas y manéjelas con cuidado.- Decorionar los embriones siguiendo los pasos 6.1.1-6.1.3.
- Transfiera los embriones a un tubo de 1,5 ml.
NOTA: Cada tubo debe tener un número igual de embriones, y no más de 40 embriones. - Después de que los embriones se asienten en el fondo del tubo, retire el sobrenadante y agregue 1 ml de solución salina tamponada con fosfato (PBS) (pH 7.6).
- Repita el paso 6.4.3 una vez más.
- Retire el sobrenadante, agregue 1 ml de formaldehído al 4% en PBS (pH 7.6) e incube el tubo a 4 °C durante la noche con un balanceo suave.
NOTA: Las muestras en solución de formaldehído pueden conservarse a 4 °C durante 1 semana. - Retire el sobrenadante, agregue 1 ml de metanol a -20 °C e incube el tubo de lado a -20 °C durante al menos 18 h.
NOTA: Las muestras pueden almacenarse a -20 °C durante 1 mes o más. - Retire el sobrenadante y agregue 1 ml de tampón PDT (0,3% v/v Triton X-100, 0,1% v/v Tween-20 y 1% v/v dimetilsulfóxido [DMSO] en tampón PBS).
- Repita el paso 6.4.7 e incube el tubo a RT durante 30 minutos con un balanceo suave.
- Retire el sobrenadante, agregue 1 ml de tampón de bloqueo (10% v/v suero fetal bovino inactivado por calor [FBS], 2% p/v albúmina sérica bovina [BSA] y 0.1% v/v Tween-20 en tampón PBS), e incube el tubo a RT durante 1 h con balanceo suave.
- Retire el sobrenadante y agregue 200 μL de solución de anticuerpos primarios (diluidos en tampón de bloqueo).
- Retirar el sobrenadante, añadir 500 μL de solución de anticuerpos primarios (diluidos en tampón de bloqueo) e incubar el tubo a 4 °C durante la noche con un balanceo suave.
NOTA: Si se utiliza un nuevo anticuerpo primario, vale la pena incluir algunas muestras sin tinción de anticuerpos primarios para que sirvan como controles negativos. Sin embargo, idealmente, los embriones con eliminación de morfolino o modificados con genes knockout, o embriones en los que se ha estimulado la expresión de la proteína diana, son medios más confiables para validar el anticuerpo. - Retire el sobrenadante, agregue 1 ml de tampón PDT e incube el tubo a RT durante 30 minutos con un balanceo suave.
- Repita el paso 6.4.12.
- Retire el sobrenadante, agregue 1 ml de tampón de bloqueo e incube el tubo en RT durante 1 h con un balanceo suave.
NOTA: Las muestras deben protegerse de la luz después de este paso, para evitar el fotoblanqueo del fluoróforo conjugado en el anticuerpo secundario. - Retire el sobrenadante y agregue 200 μL de solución de anticuerpos secundarios (diluidos en tampón de bloqueo).
- Retire el sobrenadante, agregue 500 μL de solución de anticuerpos secundarios (diluidos en tampón de bloqueo) e incube el tubo a RT durante 1,5 h con un balanceo suave.
- Retire el sobrenadante, agregue 1 ml de tampón PDT e incube el tubo a RT durante 30 minutos con un balanceo suave.
- Repita el paso 6.4.17.
- Monte los embriones en una placa de agarosa al 2% (hecha con PBS, pH 7.6) y obtenga una imagen de los embriones con un microscopio estereoscópico (campo brillante y canales fluorescentes respectivos) (Figura 5A, B, D, F).
NOTA: Si utiliza el estereomicroscopio de fluorescencia Leica M165 FC, utilice un aumento de 25x para obtener imágenes con buena resolución. - Cuantificar/analizar la intensidad de la señal fluorescente por ImageJ (NIH). Utilice la herramienta Selección a mano alzada de ImageJ para cuantificar la señal en la región de interés.
Resultados
Imágenes en vivo de peces reporteros transgénicos de neutrófilos/macrófagos tratados con Z-REX, Tg(lyz:TagRFP) y Tg(mpeg1:EGFP). Inducción de la apoptosis de neutrófilos/macrófagos mediante HNEilación de Keap1. (Ver también Figura 2). El efecto del marcaje electrofilo de Keap1 sobre los niveles de neutrófilos y macrófagos se evaluó inyectando embriones transgénicos heterocigotos derivados de Tg(lyz:TagRFP) o Tg(mpeg1:EGFP) con ARNm que codifica Halo-Keap1, y luego tratando con Ht-PreHNE(alquino). Siguiendo los procedimientos para el paso 6.1-ensayo aguas abajo, se liberó la opción 1-HNE(alquino) y se etiquetó Keap1. Los niveles de neutrófilos y macrófagos se evaluaron mediante imágenes en vivo de líneas reporteras, Tg(lyz:TagRFP) y Tg(mpeg1:eGFP), respectivamente. El nivel de ambos tipos de células disminuyó en un 30% -40% después del tratamiento con Z-REX, en el que HNE se administró a Keap1. Por el contrario, no se observó pérdida de neutrófilos o macrófagos en los grupos de control técnico Z-REX [sin luz y Ht-PreHNE(alquino), luz sola, o Ht-PreHNE(alquino) solo] (Figura 1D y Figura 2A-D).
La inducción de la apoptosis de neutrófilos/macrófagos indicó una entrega exitosa de HNE a Keap1 a través de Z-REX. Se han publicado detalles para el análisis de la vía y el mecanismo de apoptosis5. Para tener en cuenta los efectos fuera del objetivo de HNE (alquino), se utilizaron varios controles. (1) En las mismas condiciones experimentales, en lugar de ARNm de Halo-TeV-Keap1, se inyectaron embriones con ARNm de Halo-P2A-Keap1. El enlazador P2A permitió que las proteínas Halo y Keap1 se expresaran de forma independiente. En este escenario, HNE(alquino) liberado de Halo no pudo etiquetar a Keap1, ya que ya no era proximal a Halo (Figura 1D); Por lo tanto, la vía de señalización de la apoptosis no se activó. No se observaron cambios en los niveles de macrófagos o neutrófilos en este grupo (Figura 2A, B). (2) Las mismas condiciones experimentales se realizaron utilizando ARNm que codifica Halo-TeV-Keap1(C151S,C273W,C288E), un mutante de Keap1 que no responde al HNE(alquino) (Figura 1D). No se observaron cambios en los niveles de macrófagos o neutrófilos (Figura 2G, H).
Acoplamiento azida-clic de biotina y ensayo pull-down de biotina. Evaluación del etiquetado de objetivos. (Ver también Figura 3). La evaluación del etiquetado objetivo se llevó a cabo utilizando embriones WT, inyectados con ARNm que codifica Halo-TeV-Keap1-2xHA (construcción de fusión Halo-POI) o Halo-2xHA-P2A-Keap1-2xHA (construcción dividida P2A, en la que Halo y Keap1 no están fusionados; Figura 1D). La proteína Keap1 marcada solo se redujo en el grupo que expresaba proteína de fusión y se trató con Z-REX (segundo carril en el blot anti-HA superior), pero no en otros grupos de control (sin inyección de ARNm, construcción de fusión sin Z-REX o construcción dividida en P2A). Los resultados indican que el HNE (alquino) se entregó con éxito a Keap1, y el Keap1 modificado se conjugó posteriormente con biotina a través de la reacción de clic, y el Keap1 marcado con biotina fue bajado por resina de estreptavidina.
Análisis transcripcional. RNA-seq y qRT-PCR. (Ver también Figura 4). El cambio transcripcional después del tratamiento con Z-REX se evaluó mediante RNA-seq y qRT-PCR. En RNA-seq, varios genes relacionados con el sistema inmune fueron regulados a la baja después de Z-REX. En contraste, muchos genes relacionados con la respuesta antioxidante (AR) fueron regulados al alza después de Z-REX, que resultó de la inducción de la vía Keap1-Nrf2-AR sobre HNEylation en Keap110 (Figura 4A). En el análisis qRT-PCR, se encontraron resultados similares al analizar tres genes relacionados con el sistema inmunitario (lyz, mpeg1.1 y coro1a) (Figura 4B). La regulación ascendente y descendente de los genes respectivos mostró la inducción exitosa de vías mediadas por la HNEilación de Keap1.
Ensayo de tinción de (co)inmunofluorescencia de montaje completo y análisis de colocalización. (Ver también Figura 5). La expresión exógena de Halo-TeV-Keap1-2xHA y Halo-2xHA-P2A-Keap1-2xHA se evaluó mediante tinción de inmunofluorescencia (IF) de montaje completo (Figura 5A, B). La construcción dividida P2A tenía dos veces el número de etiquetas HA que la construcción de fusión TeV, lo que corresponde a una señal anti-HA dos veces mayor en el grupo P2A-split-construct-mRNA-inyectado que la otra, lo que indica que el nivel de expresión de las dos construcciones fue similar (Figura 5C). Los niveles de expresión de Halo-TeV-Keap1 (wt) y Halo-TeV-Keap1(C151S,C273W,C288E) también se encontraron similares al sondear con anti-Halo (Figura 5D,E). La colocalización de neutrófilos y caspasa activa 3 en Tg tratada con Z-REX (lyz: TagRFP) se observó mediante co-inmunotinción con anti-RFP y anti-active-Caspasa 3 (Figura 5F). La Caspasa 3 activa es un indicador de eventos de apoptosis.
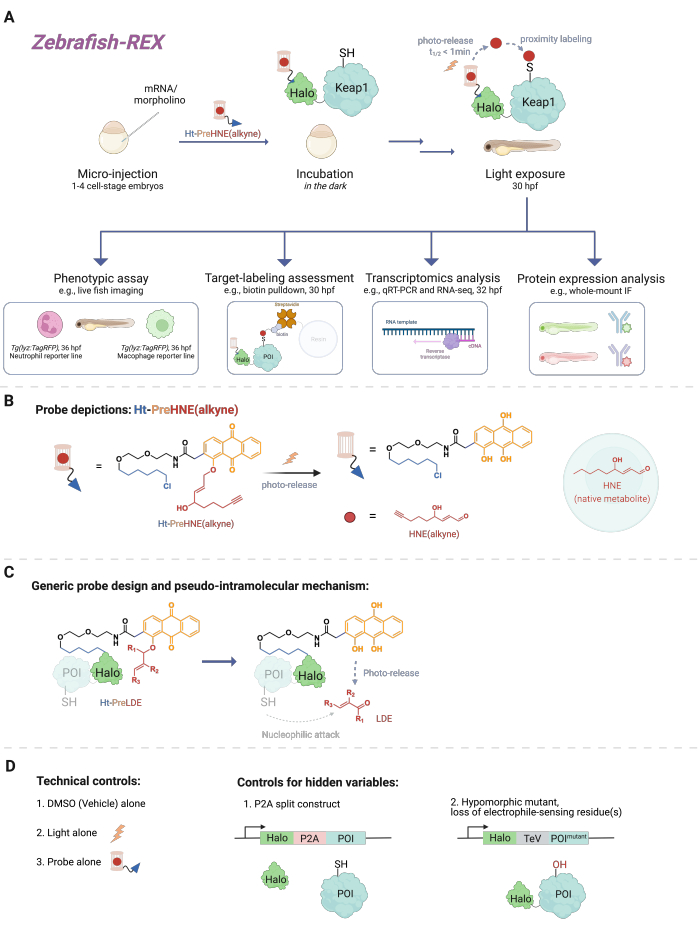
Figura 1: Flujo de trabajo de Z-REX. (A,B) Un embrión de pez cebra en estadio de 1-4 células se inyecta con (morfolino y) ARNm que codifica Halo-POI (por ejemplo, Halo-Keap1). Los embriones inyectados se tratan con una sonda compuesta por un ligando HaloTag y un electrófilo fotoenjaulado anexado con un grupo funcional alquino, como Ht-PreHNE (alquino) en B. Después de eliminar la cantidad excesiva de sonda, el embrión se expone a la luz para liberar al electrófilo de interés [por ejemplo, HNE o su análogo, HNE (alquino)]. El análisis posterior se realiza en un punto de tiempo determinado/definido por el usuario. (C) Diseño y mecanismo de la sonda Ht-PreLDE, que es aplicable a diferentes electrófilos derivados de lípidos (LDE). (D) Grupos de control negativos/técnicos para Z-REX. Haga clic aquí para ver una versión más grande de esta figura.
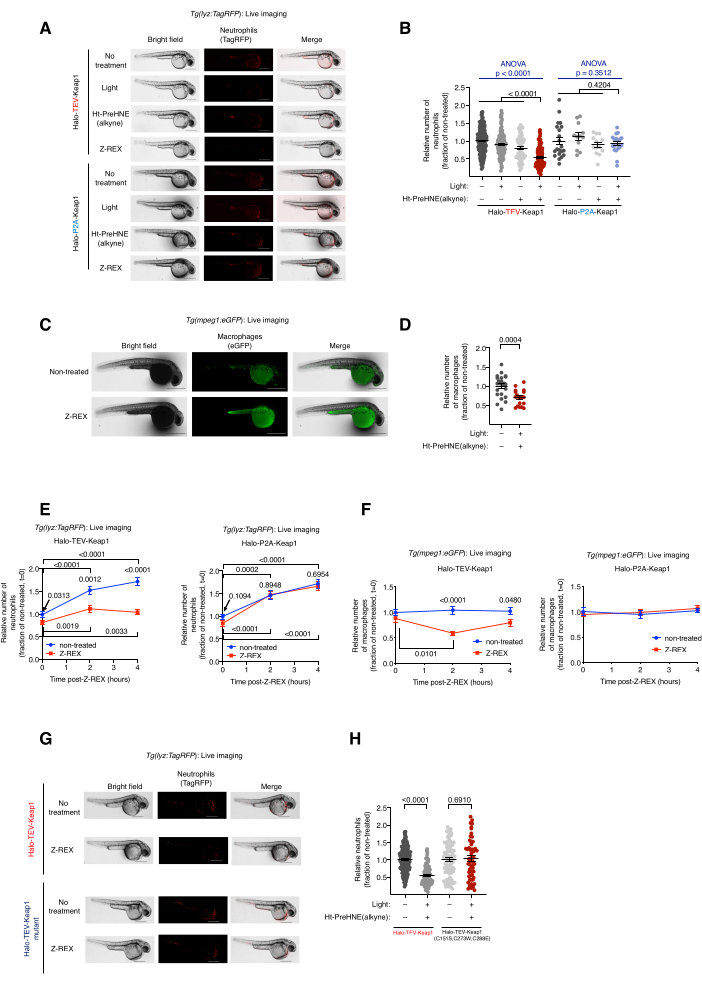
Figura 2: Imágenes en vivo de peces reporteros transgénicos de neutrófilos/macrófagos sometidos a Z-REX. La HNEilación de Keap1 mediada por Z-REX induce la apoptosis de neutrófilos/macrófagos. (A) Imágenes representativas de peces Tg(lyz:TagRFP ) que expresan Halo-TeV-Keap1 (construcción de fusión) o Halo-P2A-Keap1 (construcción dividida), y se someten a condiciones de control negativo [sin tratamiento, luz sola, o Ht-PreHNE (alquino) solo o Z-REX]. Edad embrionaria: 36 hpf. (B) Cuantificación de los niveles de neutrófilos en A. (C) Imágenes representativas de peces Tg(mpeg1:eGFP) que expresan Halo-TeV-Keap1 con o sin tratamiento Z-REX. Edad embrionaria: 34 hpf. (D) Cuantificación de los niveles de macrófagos en C. (E,F) Medición del curso temporal de (E) neutrófilos y (F) macrófagos después del tratamiento con Z-REX. (G) Experimento similar al de A en peces que expresan Halo-TeV-Keap1 (WT) o Halo-TeV-Keap1 (C151S, C273W, C288E), un mutante que no tiene capacidad de detección de HNE. (H) Cuantificación de los niveles de neutrófilos en G. Barras de escala: 500 μm. Todos los gráficos se presentan con media ± SEM. Los valores de p se calcularon con ANOVA unidireccional (azul) y prueba t de Student de dos colas (negro). Esta cifra ha sido modificada de Poganik et al.7. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Ensayo de extracción de biotina. Los embriones WT que expresan Halo-TeV-Keap1-2XHA o Halo-2XHA-P2A-Keap1-2XHA fueron tratados con Z-REX o condiciones de control negativo respectivas (sin tratamiento de sonda en este caso). Después de la cosecha, los embriones fueron lisados y tratados con proteasa TeV antes del ensayo de extracción de biotina. Los resultados fueron analizados por Western blotting. Esta cifra ha sido modificada de Huang et al. Z-REX: guiar a los electrófilos reactivos a proteínas específicas expresadas específicamente en el tejido o en la ubicua, y registrar las respuestas redox inducidas por electrófilos funcionales resultantes en peces larvarios. Esta cifra ha sido modificada de Huang et al.11. Haga clic aquí para ver una versión más grande de esta figura.
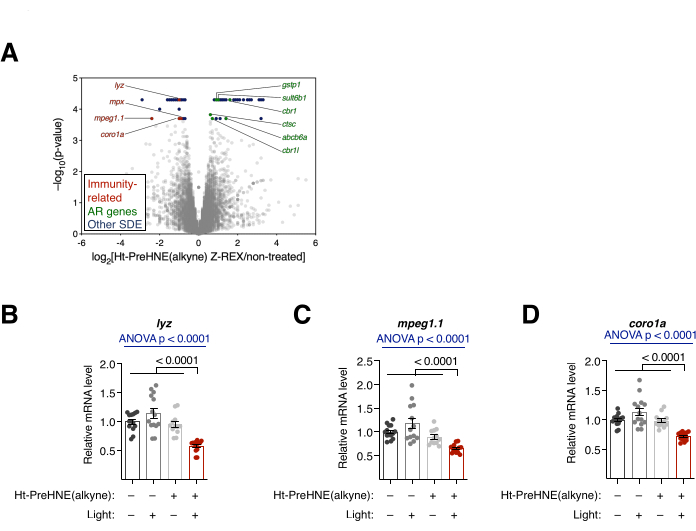
Figura 4: Análisis transcripcional. (A) Resultados RNA-seq de embriones tratados con Z-REX versus embriones no tratados. Se destacan los genes estadísticamente significativos expresados diferencialmente (SDE). Los genes SDE relacionados con la inmunidad son de color rojo. Los genes relacionados con la respuesta antioxidante (AR) son de color verde. Otros genes SDE son de color azul. Todos los valores de p se calcularon con CuffDiff. (B-D) Tres genes SDE relacionados con la inmunidad de A: (B) lyz, (C) mpeg1.1 y (D) coro1a se analizaron más a fondo con qRT-PCR, y solo los embriones tratados con Z-REX mostraron la supresión de estas transcripciones. Todos los gráficos se presentan con media ± SEM. Los valores de p se calcularon con ANOVA unidireccional (azul) y prueba t de Student de dos colas (negro). Esta cifra ha sido modificada de Poganik et al.7. Haga clic aquí para ver una versión más grande de esta figura.
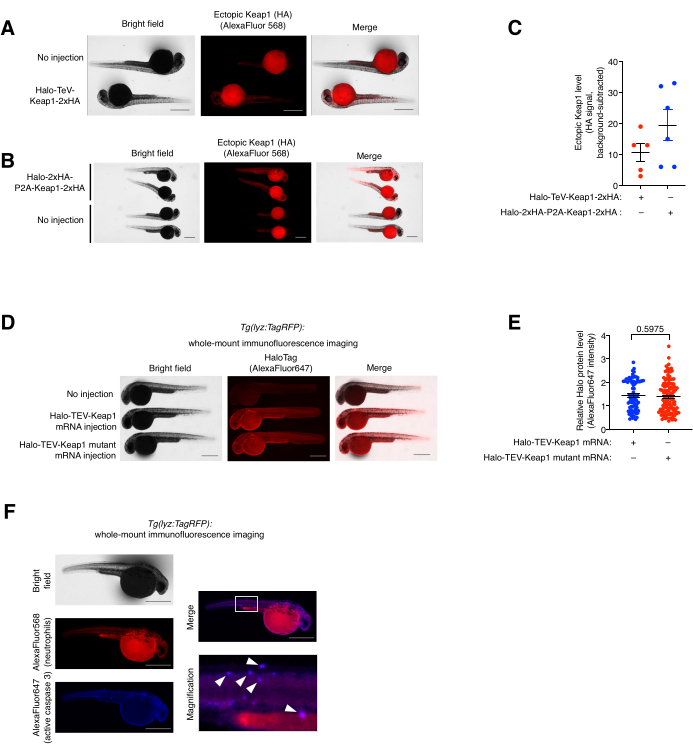
Figura 5: Ensayo de tinción de inmunofluorescencia de montaje completo . (A,B) Imágenes representativas de embriones que expresan (A) Halo-TeV-Keap1-2xHA o (B) Halo-2xHA-P2A-Keap1-2xHA inmunoteñidos con anti-HA y anticuerpo secundario conjugado con AlexaFluor568. Los peces inyectados con ARNm se compararon con peces no inyectados de la misma edad. (C) Cuantificación de la señal anti-HA en (A,B). (D) Imágenes representativas de embriones que expresan Halo-TeV-Keap1 (WT) o Halo-TeV-Keap1 (C151S, C273W, C288E) inmunoteñidos con anti-Halo y anticuerpos secundarios conjugados con AlexaFluor647. Los peces inyectados con ARNm se compararon con peces no inyectados de la misma edad. (E) Cuantificación de la señal anti-Halo en D. Los valores de p se calcularon con la prueba t de Student de dos colas. (F) Los embriones Tg(lyz:TagRFP) sometidos a Z-REX fueron co-inmunoteñidos con anti-RFP y anti-activa Caspasa 3, y los respectivos anticuerpos secundarios conjugados con fluoróforos. El cuadro blanco marca el área ampliada. Las flechas blancas indican colocalizaciones de neutrófilos y Caspasa 3 activa. Barras de escala: 500 μm. Todos los gráficos se presentan con media ± SEM. Esta cifra ha sido modificada de Poganik et al.7. y Huang et al.11. Haga clic aquí para ver una versión más grande de esta figura.
Tabla complementaria 1: Lista de tampones utilizados en este estudio. Haga clic aquí para descargar este archivo.
Discusión
Z-REX descrito en este protocolo demuestra una estrategia robusta para la investigación de pares electrófilos-objetivo y la deconvolución de la vía de señalización en peces vivos. La administración dirigida por proximidad permite la dosificación y el control espacial del tratamiento con compuestos electrofílicos. A diferencia de los métodos convencionales de dosificación en bolo, en los que las concentraciones suprafisiológicas de electrófilos desplegados a menudo conducen a problemas fuera del objetivo, la cantidad relativamente menor de electrófilo liberado al sistema hace que Z-REX sea en gran medida no invasivo. Hemos utilizado 0,1-6 μM Ht-PreHNE(alquino) en embriones de pez cebra, y los resultados mostraron que el tratamiento no es perjudicial para el desarrollo embrionario11.
El procedimiento Z-REX es generalmente más largo que T-REX, una técnica para detectar / estudiar proteínas sensibles a electrófilos en células cultivadas. Supongamos que el propósito del experimento es detectar interacciones electrófilo-objetivo; sugerimos primero realizar un cribado extensivo por T-REX en células cultivadas y usar Z-REX para la validación in vivo y el análisis fenotípico/vísta. En comparación con el cultivo celular, los requisitos para realizar Z-REX son técnicas básicas de cría de peces, además de las habilidades experimentales bioquímicas requeridas por T-REX. Un marco de tiempo típico para Z-REX (desde el cruce de peces hasta la administración de electrófilos inducibles por luz) es de 2-3 días, que no es más de 1 día más que el tiempo típico para un experimento T-REX en células vivas transfectadas. Las imágenes en vivo para el análisis fenotípico se pueden realizar 2-10 h después de la iluminación de la luz; el acoplamiento de clic con biotina-azida para el ensayo pull-down toma 3 días; qRT-PCR para analizar la respuesta transcripcional tarda 3 días; La tinción IF tarda 5 días. Estos pasos son más o menos similares a sus equivalentes de cultivo celular, aunque la interpretación de los datos requiere una comprensión de la fisiología de los peces y las cepas reporteras.
Como procedimiento de variable múltiple12, son necesarios varios grupos de control para que Z-REX excluya incertidumbres en los resultados (Figura 1D). Los grupos de control comunes son: (1) DMSO/tratamiento del vehículo solamente; (2) tratamiento de sonda, pero sin iluminación de luz; (3) iluminación ligera, pero sin tratamiento de sonda; (4) Construcción P2A-split, en la que Halo y el POI se expresan por separado, por lo que la entrega de proximidad se ablabla; y (5) mutantes hipomórficos, cuyos residuos de detección de electrófilos están mutados, como Akt3 (C119S)6 y Keap1 (C151S, C273W, C288E)5, que hemos utilizado en estudios anteriores.
Si los ensayos aguas abajo implican análisis de Western blot, el desyolking debe realizarse antes de la cosecha. Las proteínas de la yema reducen la fidelidad de las evaluaciones de concentración de lisado y pueden unirse no específicamente a los anticuerpos. Al realizar imágenes de peces vivos o tinción de FI de montaje completo, también hemos observado señales fluorescentes no específicas en el saco vitelino, probablemente como resultado de proteínas autofluorescentes en el saco vitelino o unión no específica de los propios anticuerpos. Si la señal de autofluorescencia interfiere con la señal, sugerimos excluir el saco vitelino de la cuantificación o cuantificar diferentes regiones por separado. La dechorinación es necesaria para obtener imágenes de peces vivos y para el ensayo de tinción de FI de montaje completo. El corion puede interferir con las imágenes y, más tarde, con la cuantificación / recuento celular. Sin embargo, la decorinación solo es aplicable a embriones mayores de 1 dpf; Los embriones más jóvenes en etapas de blastulación/gastrulación/segmentación son demasiado frágiles para ser descorionados.
El protocolo Z-REX descrito aquí se basa en la expresión de POI ectópica impulsada por ARNm. El procedimiento es rápido en comparación con el uso/generación de líneas de peces transgénicas. La expresión impulsada por ARNm es ubicua y transitoria, y dura al menos 2 días para los ARNm utilizados en este protocolo. Sin embargo, es probable que la duración de la expresión varíe en otros casos. Por lo tanto, este enfoque proporciona una ventana de investigación rápida y más global sobre los efectos de un evento específico de etiquetado de electrófilos, compatible con varios ensayos de alto rendimiento / alto contenido. Las líneas transgénicas con expresión estable de Halo-POI en tejidos específicos también son compatibles con Z-REX11. Estas líneas se utilizan mejor cuando es necesario hacer una pregunta más precisa, por ejemplo, cuando se predice un fenotipo en un órgano específico a partir de datos de cultivo celular, o cuando el cribado de experimentos de inyección de ARNm predice que un órgano específico es sensible a un evento de marcado de electrófilos. Se demostró una inducción de respuesta antioxidante específica del corazón a través de Z-REX utilizando peces Tg(gstp1: GFP; DsRed-P2A-myl7: Halo-TeV-Keap1) en nuestra publicación anterior11. También puede ser posible realizar Z-REX en peces transgénicos mayores de 2 dpf.
Divulgaciones
Los inhibidores de quinasa de moléculas pequeñas específicas de isoforma, cuyo descubrimiento fue posible gracias a las tecnologías REX, se han presentado para la solicitud de patente
Agradecimientos
Financiación: Novartis FreeNovation, NCCR y EPFL.
Materiales
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol (BME) | Sigma-Aldrich | M6250 | |
| 1.5-mL microcentrifuge tube | Axygen | MCT-150-C-S | |
| 10-cm Petri dishes | Celltreat | 229692 | |
| 2-mL microcentrifuge tube | Axygen | MCT-200-C-S | |
| 30% Acrylamide and bis-acrylamide solution | BioRad | 1610154 | |
| 6-well plate | Celltreat | 229506 | |
| Acetone | Fisher | A/0600/15 | |
| Agarose | GoldBio | A201-100 | |
| All Blue Prestained Protein Standards | BioRad | 1610373 | |
| Ammonium persulfate | Sigma | A3678 | |
| Biotin-dPEG11-azide | Quanta Biodesign | 102856-142 | |
| Bradford Dye | BioRad | 5000205 | |
| BSA | Fisher | BP1600-100 | |
| Capillary tubes | VWR | HARV30-00200 | |
| Chloroform | Supelco, Inc | 1.02445.1000 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Cu(TBTA) | Lumiprobe | 21050 | |
| CuSO4 | Sigma | 209198 | |
| DMSO | Fisher | D128-500 | |
| Donkey anti-mouse-Alexa Fluor 647 | Abcam | ab150107 | 1:800 (IF) |
| Donkey anti-rabbit-Alexa Fluor 647 | Abcam | ab150075 | 1:1000 (IF) |
| Donkey anti-rat-AlexaFluor 568 | Abcam | ab175475 | 1:1000 (IF) |
| ECL substrate | Thermo Fisher Scientific | 32106 | |
| ECL-Plus substrate | Thermo Fisher Scientific | 32132 | |
| End-to-end rotator | FinePCR | Rotator AG | |
| Ethanol | Fisher | E/0650DF/15 | |
| Ethidium bromide | Sigma | E1510 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument Co. | P-97 | |
| Fluorescence stereomicroscope | Leica | M165 FC | |
| Forceps (blunt) | self made | self made by blunting sharp forceps (Fine Science Tools, Dumont #5, 11252-40) | |
| Forceps (sharp) | Fine Science Tools | Dumont #5, 11252-40 | |
| Gel loading tip | Fisher | 02-707-181 | |
| Gel/blot imager | Vilber | Fusion FX imager | |
| Glass beads | Sigma | 45-G1145 | |
| Glass stage micrometer | Meiji Techno. | MA285 | |
| Heat inactivated FBS | Sigma | F2442 | |
| Heated ultrasonic bath | VWR | 89375-470 | |
| HEPES | Fisher | BP310-1 | |
| High capacity streptavidin agarose | Thermo Fisher Scientific | 20359 | |
| Ht-PreHNE alkyne probe | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Imaging plate (10% HBSS buffer, for live embryos) | Made with Petri dish, and 2% agarose in 10% HBSS buffer | ||
| Imaging plate (PBS, for formaldehyde-fixed embryos) | Made with Petri dish, and 2% agarose in PBS | ||
| Injection plate | Made with microinjection mold, Petri dish, and 2% agarose in 10% HBSS buffer | ||
| LDS | Apollo | APOBI3331 | |
| Methanol | VWR | 20864.32 | |
| Microinjection mold | Adaptive Science Tools | TU-1 | |
| Microloader tips | Eppendorf | 930001007 | |
| Micromanipulator | Narishige | MN-153 | |
| Microscope for micro-injection | Olympus | SZ61 | |
| Microscope light source | Olympus | KL 1600 LED | |
| Mineral oil | Sigma | M3516 | |
| mMessage mMachine SP6 Transcription Kit | Ambion | AM1340 | |
| Mouse anti- β-actin-HRP | Sigma | A3854 | 1:20000 (WB) |
| Mouse anti-HaloTag | Promega | G921A | 1:500 (IF) |
| Multi-mode reader | BioTek Instruments | Cytation 5 | |
| Nucleic acid agarose gel electrophoresis apparatus | Biorad | Mini-Sub Cell GT Systems | |
| Oligo(dT)20 | Integrated DNA Technologies | customized: (dT)20 | |
| Orange G | Sigma | O3756 | |
| Paraformaldehyde | Sigma | P6148 | |
| PBS | Gibco | 14190144 | |
| pCS2+8 Halo-2XHA-P2A-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| pCS2+8 Halo-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| Pneumatic PicoPump | WPI | SYS-PV820 | |
| Protein electrophoresis equipment and supplies | Biorad | Mini-PROTEAN Tetra Vertical Electrophoresis | |
| Rabbit anti-active Caspase-3 | BD Pharmingen | 559565 | 1:800 (IF) |
| Rat anti-HA-HRP | Sigma | H3663 | 1:500 (IF and WB) |
| Rat anti-RFP | ChromoTek | 5F8 | 1:800 (IF) |
| Refrigerated centrifuge | Eppendorf | 5417R | |
| RNAseOUT recombinant ribonuclease inhibitor | ThermoFisher Scientific | 10777019 | |
| RnaseZap RNA decontamination solution | ThermoFisher Scientific | AM9780 | |
| Rocking Shaker | DLAB | SK-R1807-S | |
| SDS | Teknova | S9974 | |
| SuperScript III reverse transcriptase | ThermoFisher Scientific | 18080085 | |
| t-Butanol | Sigma | 471712 | |
| TCEP-HCl | Gold Biotechnology | TCEP1 | |
| TeV protease (S219V) | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Tg(lyz:TagRFP) | Zebrafish International Resource Center (ZIRC) | uwm11Tg (ZFIN) | - |
| Tg(mpeg1:eGFP) | Zebrafish International Resource Center (ZIRC) | gl22Tg (ZFIN) | - |
| Thermal cycler | Analytik Jana | 846-x-070-280 | |
| TMEDA | Sigma | T7024 | |
| Transfer pipets | Fisher | 13-711-9D | |
| Tris | Apollo | APOBI2888 | |
| Triton X-100 | Fisher | BP151-100 | |
| TRIzol reagent | Thermo Fisher Scientific | 15596018 | |
| Trypsin inhibitor from Glycine max (soybean) | Sigma | T9003 | |
| Tween 20 | Fisher | BP337-500 | |
| UV lamp with 365-nm light | Spectroline | ENF 240C | |
| UV meter | Spectroline | XS-365 | |
| Vortexer | Scientific Industries, Inc. | Vortex-Genie 2 | |
| Western Blotting Transfer equipment and supplies | Biorad | Mini Trans-Blot or Criterion Blotter | |
| Zebrafish husbandry and breeding equipment | in house | ||
| Zirconia beads | BioSpec | 11079107zx |
Referencias
- Long, M. J. C., Ly, P., Aye, Y. A primer on harnessing non-enzymatic post-translational modifications for drug design. RSC Medicinal Chemistry. 12 (11), 1797-1807 (2021).
- Parvez, S., Long, M. J. C., Poganik, J. R., Aye, Y. Redox signaling by reactive electrophiles and oxidants. Chemical Reviews. 118 (18), 8798-8888 (2018).
- Parvez, S., et al. T-REX on-demand redox targeting in live cells. Nature Protocols. 11 (12), 2328-2356 (2016).
- Long, M. J. C., et al. Precision electrophile tagging in Caenorhabditis elegans. Biochemistry. 57 (2), 216-220 (2018).
- Van Hall-Beauvais, A., Zhao, Y., Urul, D. A., Long, M. J. C., Aye, Y. Single-protein-specific redox targeting in live mammalian cells and C. elegans. Current Protocols in Chemical Biology. 10 (3), 43 (2018).
- Long, M. J. C., et al. Akt3 is a privileged first responder in isozyme-specific electrophile response. Nature Chemical Biology. 13 (3), 333-338 (2017).
- Poganik, J. R., et al. Wdr1 and cofilin are necessary mediators of immune-cell-specific apoptosis triggered by Tecfidera. Nature Communications. 12 (1), 5736 (2021).
- Lin, H. -. Y., Haegele, J. A., Disare, M. T., Lin, Q., Aye, Y. A generalizable platform for interrogating target- and signal-specific consequences of electrophilic modifications in redox-dependent cell signaling. Journal of the American Chemical Society. 137 (19), 6232-6244 (2015).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative CT method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Wakabayashi, N., et al. Keap1-null mutation leads to postnatal lethality due to constitutive Nrf2 activation. Nature Genetics. 35 (3), 238-245 (2003).
- Huang, K. -. T., et al. Z-REX: Shepherding reactive electrophiles to specific proteins expressed either tissue-specifically or ubiquitously, and recording the resultant functional electrophile-induced redox responses in larval fish. Nature Protocols. , (2023).
- Long, M. J. C., Assari, M., Aye, Y. Hiding in plain sight: The issue of hidden variables. ACS Chemical Biology. 17 (6), 1285-1292 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados