Method Article
Monitoramento de respostas de sinalização no alvo em larvas de peixe-zebra - Z-REX desmascara mecanismos precisos de drogas eletrofílicas e metabólitos
Neste Artigo
Resumo
Zebrafish visando eletrófilos reativos e oxidantes (Z-REX) é um método baseado em biologia química para a investigação da sinalização de pequenas moléculas reativas. Esta técnica pode ser aplicada a peixes vivos de diferentes estágios de desenvolvimento. Aqui, acoplamos ensaios padrão em zebrafish com Z-REX para análise de vias de sinalização.
Resumo
Metabólitos reativos e drogas eletrofílicas relacionadas estão entre as pequenas moléculas mais desafiadoras para estudar. Abordagens convencionais para desconstruir o modo de ação (MOA) de tais moléculas alavancam o tratamento em massa de espécimes experimentais com um excesso de uma espécie reativa específica. Nessa abordagem, a alta reatividade dos eletrófilos torna a marcação não discriminada do proteoma de maneira dependente do tempo e do contexto; Proteínas e processos sensíveis à redox também podem ser afetados indiretamente e muitas vezes de forma irreversível. Em um cenário de inúmeros alvos potenciais e efeitos secundários indiretos, vincular o fenótipo ao engajamento de alvos específicos continua sendo uma tarefa complexa. Zebrafish visando eletrófilos reativos e oxidantes (Z-REX) - uma plataforma de entrega de eletrófilos reativos sob demanda adaptada para uso em larvas de peixes-zebra - é projetado para fornecer eletrófilos a uma proteína específica de interesse (POI) em embriões de peixes vivos não perturbados. As principais características desta técnica incluem um baixo nível de invasividade, juntamente com a dosagem - quimiotipo - e liberação eletrofílica de precisão controlada espaço-temporalmente. Assim, em conjunto com um conjunto único de controles, essa técnica evita efeitos fora do alvo e toxicidade sistêmica, observados após exposição descontrolada em massa de animais a eletrófilos reativos e drogas eletrofílicas pleiotrópicas. Aproveitando o Z-REX, os pesquisadores podem estabelecer um ponto de apoio na compreensão de como as respostas individuais ao estresse e as saídas de sinalização são alteradas como resultado do envolvimento específico do ligante reativo com um POI específico, sob condições quase fisiológicas em animais vivos intactos.
Introdução
Uma miríade de eventos de sinalização celular envolve reações entre pequenas moléculas reativas (produzidas endogenamente na célula ou xenobióticos/xenometabolitas, como drogas) e seu alvo proteico. Em muitos casos, um nível subestequiométrico desses eventos de ligação covalente pode desencadear respostas celulares, levando a alterações no desenvolvimento, metabolismo, apoptose e/ou resposta imune, por exemplo1. No entanto, a desconstrução do modo de ação (MOA) ligando eventos de ligação específicos às suas consequências fenotípicas tem se mostrado desafiadora. Métodos tradicionais de dosagem em bolus que envolvem a introdução de altas concentrações das espécies reativas frequentemente resultam em uma infinidade de proteínas sendo modificadas, bem como toxicidade excessiva para o organismo modelo2. Tais condições estão longe de ser as ideais. Um método foi desenvolvido para resolver esses problemas em cultura celular usando liberação de eletrófilos localizados de precisão em um contexto celular nativo, denominado T-REX (targetable reactive electrophiles and oxidants)3. Nos anos seguintes, o foco voltou-se para experimentos em organismos inteiros, que permitem a oportunidade de estudar proteínas em contextos celulares específicos em células não transformadas. Assim, ampliamos a técnica para ser compatível com vários modelos, incluindo modelos embrionários de Danio rerio . Neste trabalho, apresentamos o Z-REX (zebrafish visando eletrófilos reativos e oxidantes) (Figura 1).
Para entender o Z-REX, este artigo primeiro apresenta as tecnologias REX e seus conceitos subjacentes. Em sua essência, essas técnicas modelam a sinalização de espécies eletrofílicas reativas fisiológicas endógenas (RES) mimetizando como os eletrófilos naturais são produzidos localmente in vivo com precisão espaço-temporal. A proteína de interesse (POI) é expressa como um construto de fusão para Halo; este último ancora a sonda inerte e permeável ao tecido com o ER fotoengaiolado em uma estequiometria 1:1. Um desses RES endógenos é o 4-hidroxinonenal (HNE doravante), que é fotocageado na sonda Ht-PreHNE. Em muitos casos, usamos uma versão funcionalizada com alquina do HNE [i.e., HNE(alquino)], que tem propriedades biológicas essencialmente idênticas à HNE, mas pode ser rotulada pela química do clique. A sonda, que também é funcionalizada com um cloroalcano para reatividade com Halo, é referida como Ht-PreHNE (alquino). O complexo da fusão Halo-POI e a sonda assim formada permite a entrega proximal de RES ao POI fundido após irradiação com luz UV. Se o POI reage rapidamente com o RES, a marcação covalente resultante do POI com o ER permite identificar cisteínas cineticamente privilegiadas.
Z-REX pega as vantagens acima mencionadas das tecnologias REX e as aplica amplamente para estudar vias de sinalização específicas em peixes vivos. Este protocolo foi otimizado para zebrafish (D. rerio), uma vez que são organismos vertebrados geneticamente tratáveis que são transparentes durante o desenvolvimento e, portanto, ideais para técnicas optoquímicas/genéticas como as tecnologias REX. No entanto, uma estratégia semelhante também provavelmente funcionará bem em outras espécies de peixes geneticamente tratáveis, uma vez que a ampla aplicabilidade do método é devida ao mecanismo pseudo-intramolecular de liberação de eletrófilos derivados de lipídios (LDE). De fato, o procedimento é altamente biocompatível, uma vez que os peixes podem ser tratados com o Z-REX fotocaged-electrophile [por exemplo, Ht-PreHNE(alkyne)] por pelo menos 48 h sem impactos perceptíveis no desenvolvimento. Protocolo semelhante funciona em C. elegans 4,5.
O protocolo descreve primeiramente o uso de injeção de RNAm para produzir uma expressão transitória de uma construção de fusão Halo-POI não nativa em modelos embrionários de zebrafish, 1-1,5 dias pós-fertilização (dpf). Isto resulta na expressão da proteína ectópica na maioria das células do peixe (doravante designada por «ubíqua»), e não em tecidos ou locais específicos; no entanto, os dados mostram que efeitos específicos de células podem ser observados em certos casos. Após a injeção, os embriões são incubados com uma baixa concentração [0,3-5 μM Ht-PreHNE(alquina)] da sonda por até 30,5 h pós-fertilização (hpf). Então, em um tempo prescrito pelo usuário, a entrega do RES ao POI dentro dos peixes é alcançada por fotouncaging por 2-5 min. Após a fotouncaging do RES, uma variedade de ensaios fenotípicos a jusante pode ser realizada nas próximas 2-10 h: 1) imagens ao vivo das linhas do repórter (Figura 2A); 2) avaliação da marcação de alvos por análise de western blot (Figura 3); 3) análise transcriptômica (Figura 4); ou 4) imunofluorescência total (Figura 5).
Como um exemplo de imagem ao vivo de linhas de repórteres, Z-REX é demonstrado em conjunto com imagens ao vivo de linhagens de peixes, Tg(lyz:TagRFP) e Tg(mpeg1:eGFP), para medir como a modificação de RES de um sensor eletrófilo específico POI (ou seja, Keap1) diminui os níveis de neutrófilos e macrófagos, respectivamente, sem efeitos observáveis em outras células nos peixes. No entanto, mostramos anteriormente que a marcação de POI e a consequente sinalização da via de estudos de T-REX podem ser reproduzidas usando Z-REX para várias proteínas: Akt3 6, Keap17 e Ube2v26. Em geral, com o Z-REX, os cientistas podem estudar a(s) consequência(s) da modificação covalente de POIs por RES no contexto de várias vias redox complexas. Esta técnica é preparada para identificar alvos e seus resíduos funcionais para o planejamento covalente de fármacos e novos mecanismos de fármacos em um modelo animal inteiro mais contextualmente relevante.
Protocolo
Os procedimentos de criação e manejo de zebrafish na Universidade de Cornell (Estados Unidos) foram realizados seguindo as diretrizes do National Institutes of Health (NIH) e aprovados pelo Institutional Animal Care and Use Committee (IACUC). Os procedimentos de criação e manejo de peixes-zebra na unidade de peixe-zebra do Instituto Federal Suíço de Tecnologia de Lausanne (EPFL, Suíça) foram realizados seguindo a Lei de Bem-Estar Animal SR 455 e a Portaria de Bem-Estar Animal SR 455.1, com autorização veterinária cantonal VD-H23.
NOTA: Neste protocolo, as linhas de peixes Tg( lyz:TagRFP ) e Tg(mpeg1:EGFP) expressando Halo-TeV-Keap1 são usadas para demonstrar Z-REX. O método pode ser estendido a outras proteínas de interesse, linhagens de peixes repórteres transgênicos e ensaios biológicos a jusante. Consulte a Tabela Suplementar 1 para os buffers utilizados neste estudo. Todos os reagentes, instrumentos, equipamentos, anticorpos, plasmídeos, cepas de peixe-zebra e equipamentos estão listados na Tabela de Materiais.
1. Preparação de mRNA
- Preparar o mRNA Halo-TeV-Keap1-2xHA e Halo-2xHA-P2A-Keap1-2xHA usando o kit de transcrição in vitro mMessage mMachine SP6.
NOTA: Siga as instruções do fabricante e execute reações em escala de 40 μL para cada mRNA. Redissolver a pastilha de RNAm em 10 μL de água livre de nucleases. - Avaliar a qualidade do RNAm e medir a concentração por espectrofotômetro de microvolume e eletroforese em gel de agarose. Um mRNA de boa qualidade deve ter uma relação A260/A280 em torno ou acima de 2,0.
- Diluir o mRNA para 1-1,5 mg/mL com água livre de nucleases.
- Aliquot a solução de mRNA (1-2 μL por tubo) e armazenar as alíquotas a -80 °C.
2. Produção de embriões de peixe
- Opção 1: Produção de embriões de peixes selvagens (WT).
- Configure 10 pares de cruzamento de peixes em 10 tanques separados, com cada tanque contendo um divisor entre os peixes machos e fêmeas.
NOTA: Um total de 10 pares cruzados normalmente fornece um número suficiente de embriões para os ensaios. O número de pares de cruzamentos pode ser ajustado de acordo com o desenho/necessidade do experimento e a fecundidade do peixe progenitor. - Na manhã seguinte, após a instalação do injetor, retire as divisórias em cinco dos tanques. Aguarde 30 min para o peixe acasalar.
- Mova os peixes-mãe para outro tanque, colete embriões passando a água do tanque através de um filtro e, em seguida, enxágue os embriões do filtro em uma placa de Petri de 10 cm. Esses embriões serão usados para a primeira rodada de injeções.
NOTA: Se a qualidade do ovo de um determinado lote for ruim (por exemplo, os ovos são opacos devido à agregação de proteínas), não os agrupe com os outros embriões. - (Opcional) Efectuar procedimentos semelhantes aos descritos nos passos 2.1.2-2.1.3 nos outros cinco tanques para a próxima injecção.
NOTA: É melhor realizar apenas uma rodada de injeção, para minimizar a diferença de idade entre os embriões. No entanto, se forem necessários mais embriões do que podem ser injetados em um lote, recomenda-se a realização de duas rodadas de injeção para garantir que os embriões permaneçam em um estágio de 1-4 células durante a injeção de mRNA. O número de tiros de injeção pode ser ajustado de acordo com a habilidade de injeção do operador e o design do experimento. No entanto, sugere-se realizar todo o procedimento (desde a remoção do primeiro divisor até a injeção do último embrião) dentro de 2 h. Uma grande diferença de idade entre os embriões pode prejudicar a confiabilidade e a reprodutibilidade dos resultados do experimento.
- Configure 10 pares de cruzamento de peixes em 10 tanques separados, com cada tanque contendo um divisor entre os peixes machos e fêmeas.
- Opção 2: Produção de embriões de peixe repórter neutrófilos/macrófagos heterozigotos transgênicos.
- Configure 10 pares de cruzamento de peixes em 10 tanques separados, com cada tanque inserido com um divisor entre peixes pais machos e fêmeas: peixes WT versus Tg( lyz:TagRFP) ou peixes WT versus Tg(mpeg1:eGFP).
NOTA: Evite cruzamentos entre peixes transgênicos heterozigotos, que podem afetar as leituras fluorescentes a jusante, pois os peixes repórteres homozigotos mostram um sinal fluorescente mais alto em comparação com os peixes heterozigotos. Linhas de repórteres transgênicos e embriões WT podem ser facilmente distinguidos durante a obtenção de imagens. Ter uma mistura de WT e embriões heterozigotos no mesmo pool não é um problema. lyz:TagRFP relata neutrófilos, e mpeg:eGFP relata macrófagos. Esse protocolo também pode ser aplicado a outras linhas de peixes. - Siga as etapas 2.1.2-2.1.4.
- Configure 10 pares de cruzamento de peixes em 10 tanques separados, com cada tanque inserido com um divisor entre peixes pais machos e fêmeas: peixes WT versus Tg( lyz:TagRFP) ou peixes WT versus Tg(mpeg1:eGFP).
3. Configuração do microinjetor
- Ligue a fonte de ar, ajuste a contrapressão para 0,2-0,5 psi e ajuste a pressão de injeção para 25-30 psi. A faixa de pressão específica mostrada é normalmente recomendada.
NOTA: É essencial ter uma contrapressão estável para evitar o refluxo do meio de peixe para a agulha. Ao calibrar o volume de injeção na etapa 3.8, apenas o tempo de injeção deve ser alterado. Não altere a pressão de injeção nos seguintes passos; A baixa pressão de injeção pode levar à falha da injeção devido à tensão superficial e interfacial, enquanto a alta pressão de injeção pode danificar os embriões. - Limpe o equipamento e a plataforma de injeção com solução de descontaminação RNase.
NOTA: A RNase, que degrada o mRNA, pode vir do operador ou do equipamento. É necessário realizar a limpeza antes do experimento, e usar luvas. - (Opcional) Se co-injetar mRNA e morfolino, premix os dois em um tubo de 0,2 mL.
NOTA: Z-REX funciona bem usando solução de mRNA Halo-TeV-Keap1-2xHA com uma concentração de 250-1500 ng/μL. Vários morfolínos também têm sido utilizados em zebrafish, e as concentrações ótimas têm sido relatadas7. Se estiver usando um morfolino com uma sequência não publicada, o operador deve primeiro avaliar a toxicidade e a eficiência de knockdown do gene do morpholino antes de usá-lo em Z-REX. - Transfira 1-2 μL de mRNA (e/ou morfolino, quando aplicável7) para uma agulha de injeção com uma ponta de pipeta de microcarregador.
NOTA: Se preparar agulhas com um puxador de micropipeta Flaming/Brown, a configuração é a seguinte. Calor: 520 unidades; força de tração: 60 unidades; velocidade: 70 unidades; atraso: 155 unidades; pressão: 550 unidades; rampa: 530 unidades. - Instale a agulha no manipulador de microinjeção.
NOTA: A contrapressão da fonte de ar deve empurrar a solução de mRNA(/morpholino) para a ponta da agulha. - Use pinças afiadas ou uma lâmina de barbear para quebrar a ponta da agulha, criando uma abertura adequada para injeção.
- Submergir a ponta da agulha em óleo mineral em um micrômetro de estágio.
- Aplique dois ou três pulsos de injeção para remover bolhas de ar na ponta.
- Calibre o tamanho da gota para 2 nL alterando o tempo de injeção.
NOTA: Isso é melhor realizado injetando em óleo mineral (que imita a viscosidade do saco vitelino) colocado sobre um hemocitômetro. Usando um microscópio, use as linhas de grade do hemocitômetro para estimar o tamanho da gotícula formada durante a injeção e ajustar o tempo de injeção de acordo. Embora o corante vermelho de fenol às vezes seja usado, a necessidade dele no procedimento de injeção de RNAm descrito aqui não foi observada.
4. Microinjeção
- Encha uma placa de injeção com meio fresco de solução salina balanceada de Hank a 10% (HBSS) e alinhe os embriões nos sulcos da placa com pinças rombas.
NOTA: A placa de injeção é preparada com agarose a 2% em meio HBSS a 10%; Os sulcos são formados usando um molde plástico. - Submergir a ponta da agulha em meio HBSS a 10% na placa de injeção.
- Aplique dois ou três pulsos de injeção para remover bolhas de ar na ponta.
- Para cada injeção, penetre no córion e no saco vitelínico em um único movimento e aplique um pulso de injeção. Este líquido injetado pode ser visto logo após a injeção como um pequeno esferoide dentro do saco vitelino. Este pequeno esferoide dissipa-se relativamente rapidamente. Repita este passo para outros embriões, até que um número suficiente de embriões injetados tenha sido obtido.
NOTA: A taxa de sobrevivência dos embriões (injetados e não injetados) normalmente varia de 50% a 90%. Objetivar injetar o dobro do número de embriões necessários para cada grupo controle/experimental. No ensaio pull-down de biotina, 100-140 embriões viáveis são necessários para cada condição. No ensaio de qRT-PCR, cinco embriões viáveis são recomendados para cada condição. O tamanho da amostra para imagens de peixes vivos e ensaio de coloração de imunofluorescência de montagem inteira é definido pelo usuário; Recomenda-se ter pelo menos 20 embriões viáveis por condição para produzir bom poder estatístico na análise. - Enxaguar os embriões injetados em uma nova placa de Petri de 10 cm contendo meio HBSS fresco a 10%.
NOTA: Os embriões podem ser facilmente enxaguados dos sulcos usando um frasco de esguicho. - Junte os embriões não injetados em outra placa.
NOTA: Os embriões não injetados podem servir como controles de qualidade para a saúde dos peixes, expressão de proteínas basais, níveis de fluorescência de fundo, etc., conforme necessário. Se o procedimento de injeção funcionou bem, e o mRNA/morfolino injetado não for letal para os embriões, embriões injetados e não injetados devem ter viabilidade semelhante.
5. Z-REX
- Distribuir os embriões injetados em placas de 10 cm, de acordo com a configuração do experimento (i.e., número de grupos controle/experimento).
- Em uma sala escura com iluminação de luz vermelha, substitua a mídia por 30 mL de HBSS a 10% com ou sem 1 μM de Ht-PreHNE (alquino).
NOTA: Ht-PreHNE (alquino) é um composto leve-lábil. Os embriões devem ser mantidos no escuro nas etapas seguintes. - Incubar os embriões a 28,5 °C no escuro.
- A 30,5 hpf, em sala escura, substitua o meio por 30 mL frescos de HBSS a 10%.
NOTA: Ao substituir o meio, remova o máximo possível do meio antigo. Isso é fundamental para remover a quantidade não ligada/excessiva de Ht-PreHNE (alquino) dos embriões. - Incubar os embriões a 28,5 °C no escuro durante 30 minutos.
- Repita as etapas 5.4-5.5 mais duas vezes.
- Ligue a lâmpada UV (365 nm, 3 mW/cm2) por 5 min para pré-aquecer a lâmpada.
NOTA: A etapa de pré-aquecimento da lâmpada deve ser executada antes da etapa 5.8. A potência da lâmpada é menor/instável nos primeiros minutos após ser ligada. A potência da lâmpada deve ser medida por um medidor UV regularmente. - A 32 hpf, expor os embriões à luz UV.
- Opção 1: Para leituras a jusante, como imagens de peixes vivos, coloração de imunofluorescência de montagem inteira, análise de fluorescência em gel (acoplamento de clique com azida Cy5), RNA-seq ou qRT-PCR, exponha os embriões à luz UV por 3 min, girando as placas a cada 30 s.
- Opção 2: Para leituras a jusante, como o ensaio pull-down de biotina, exponha os embriões à luz UV por no máximo 5 min (e mínimo de 3 min), rodopiando as placas a cada 30 s e resfriando as placas no gelo por 1 min.
NOTA: Se estiver usando sondas diferentes, o tempo de exposição à luz precisa ser otimizado, dependendo do t1/2 de fotouncaging para uma determinada sonda eletrofílica fotocaged e fonte de luz implantada. t1/2fotouncaging pode ser determinado usando procedimentos conhecidos8. Para Ht-PreHNE (alquino), t 1/2 é < 1 min3; Assim, o tempo indicado acima é suficiente.
6. Ensaios a jusante
- Opção 1: Ensaio fenotípico. Imagem ao vivo do repórter transgênico de neutrófilos/macrófagos
linhas de peixes, Tg(lyz:TagRFP) e Tg(mpeg1:eGFP) (Figura 2)- Anestesiar os embriões por incubação a 4 °C por 10 min.
NOTA: Recomenda-se ter pelo menos 20 embriões viáveis por condição. - Retire os embriões não fertilizados/mortos da placa.
NOTA: Os embriões não fertilizados/mortos são turvos/não transparentes, e podem ser identificados visualmente. Se uma alta taxa de mortalidade for observada, volte ao procedimento de injeção ou tente reduzir a concentração de mRNA ou morfolino. - Descorionar os embriões com pinças afiadas. Segure o córion com um par de pinças, sem tocar nas larvas de peixes, e use o outro par de pinças para arrancar o córion. O embrião é frágil. Só toque no córion ao executar a descorionação.
NOTA: É comum, especialmente em iniciantes, danificar alguns embriões durante a descorionação. Portanto, tenha sempre mais embriões do que o mínimo necessário. - Montar os embriões em placa de agarose a 2% (preparada com meio HBSS a 10%) e fotografar os embriões com estereomicroscópio (campo claro e respectivos canais fluorescentes) (Figura 2A, C, G).
NOTA: Após Z-REX [combinação de injeção de mRNA Halo-TeV-Keap1-2xHA e tratamento com Ht-PreHNE(alquino)], a depleção de neutrófilos (lyz:TagRFP) foi mais significativa em 36 hpf (4 h pós-Z-REX), enquanto a redução de macrófagos (mpeg1:eGFP) foi mais significativa em 34 hpf (2 h pós-Z-REX) (Figura 2E,F). Outros pontos de tempo podem ser usados se usar diferentes linhas de repórter, mRNA/morfolino ou sondas. O tempo de exposição e/ou ganho deve ser otimizado para visualização de células isoladas ou das (ultra)estruturas específicas desejadas. - Conte o número de neutrófilos/macrófagos de cada peixe por ImageJ (NIH) (Figura 2B, D-F, H). Use a ferramenta Seleção à mão livre no ImageJ para circundar os peixes inteiros e use a opção Find Maxima para contar as células fluorescentes.
- Anestesiar os embriões por incubação a 4 °C por 10 min.
- Opção 2: Avaliação da rotulagem de destino. Acoplamento azida-clique de biotina e ensaio pull-down de biotina (Figura 3)
- Anestesiar os embriões por incubação a 4 °C. Isso geralmente leva 10 minutos.
NOTA: Para obter lisado de peixe suficiente, 100-140 embriões viáveis são necessários para cada condição. - Retire os embriões não fertilizados/mortos da placa.
- Realizar decorionação e deyolking. Execute as duas manipulações segurando o córion com um par de pinças afiadas, usando o outro par de pinças para penetrar no saco vitelínico e, em seguida, separando o saco vitelínico enquanto remove o córion para permitir que as proteínas da gema saiam.
OBS: É fundamental remover as proteínas da gema nesta etapa. As proteínas vitelínicas abundantes na amostra interferem nas análises posteriores. - Transfira os embriões desgemados para um tubo de 1,5 mL.
OBS: Gire a placa para centralizar os embriões desgemidos, o que facilita a coleta. Os detritos mais leves do córion vencem durante esse processo. - Após os embriões se acomodarem no fundo do tubo, retirar o sobrenadante e adicionar 1 mL de solução salina tamponada com HEPES resfriada (pH 7,6).
- Repita a etapa 6.2.5 mais duas vezes.
- (Opcional) Se não pretender prosseguir com o próximo passo imediatamente, remova o tampão, congele rapidamente as amostras em azoto líquido e armazene-as a -80 °C.
NOTA: Os pellets de peixe desgemados podem ser congelados em azoto líquido e armazenados a -80 °C. As amostras congeladas por flash podem ser armazenadas a -80 °C por até 2 semanas. - Ressuspenda os pellets de peixe em tampão de lise.
NOTA: O tampão de lise (pH 7,6) é composto por 50 mM HEPES, 100 mM NaCl, 1% Triton X-100, 0,3 mM TCEP, 2x Roche cOmplete Mini inibidores de protease livres de EDTA e 0,1 mg/mL inibidor de tripsina de soja. Um embrião dá cerca de 2 μg de lisado. Use 100 μL de tampão de lise para cada 120 embriões. Dois inibidores de protease Roche cOmplete Mini livres de EDTA e inibidores de tripsina de soja devem ser adicionados ao tampão de lise imediatamente antes do uso. - Adicione 20% v/v de contas de zircônia ao tubo.
- Vórtice por 20 s, congelamento instantâneo em nitrogênio líquido e descongelamento em banho-maria a 37 °C.
- Repita a etapa 6.2.10 mais duas vezes.
- Centrifugar a solução a 21.000 x g a 4 °C durante 10 min.
- Transfira o sobrenadante para um novo tubo pré-resfriado de 1,5 mL.
- Medir a concentração de proteína pelo ensaio de Bradford.
- Diluir o lisado para 1 mg/ml.
- Para cada condição, transferir 170 μg de lisado para um tubo de 2 mL.
- Misturar o lisado do passo 6.2.16 com 0,2 mg/ml de protease TeV (S219V) e incubar a solução a 37 °C durante 30 minutos.
NOTA: Para grupos não tratados com protease TeV, basta misturar o lisado com um voloume igual de tampão de lise à solução de protease TeV usada em outros grupos. - Prepare uma mistura mestre de 10x para a reação de clique de biotina-azida: 10% p/v SDS, 10 mM CuSO4, 1 mM(TBTA), 1 mM biotina-azida e 20 mM TCEP.
NOTA: Adicione TCEP à mistura imediatamente antes do passo 6.2.19. - Adicionar 8,5 μL de t-BuOH e 17 μL de mistura mestra de reação de clique 10x ao lisado (digerido por protease TeV) da etapa 6.2.17. Vórtice, centrifugar e incubar a solução a 37 °C durante 15 minutos.
- Adicione mais 1 mM de TCEP à solução e, em seguida, vórtice, centrífuga e incube a solução a 37 °C por mais 15 min. O tempo de incubação para as etapas 6.2.19-6.2.20 é de 30 min no total.
NOTA: Este suplemento de TCEP, um reagente redutor para gerar(I), melhora a eficiência da reação de clique. - Adicionar 600 μL de etanol -20 °C a cada tubo, agitar a solução e incubá-la a -80 °C durante a noite.
NOTA: As amostras podem ser armazenadas a -80 °C por 1 semana; Caso contrário, prossiga com a próxima etapa imediatamente. - Centrifugar a solução a 21.000 x g a 4 °C durante 1 h.
OBS: Um pellet deve se formar no fundo do tubo após a centrifugação, que é a fração desejada. - Retire o sobrenadante, adicione 1 mL de etanol -20 °C, vórtice e centrifugar a solução a 21.000 x g a 4 °C por 10 min.
- Repita a etapa 6.2.23.
- Retire o sobrenadante, adicione 1 ml de acetona -20 °C, vórtice e centrifugar a solução a 21.000 x g a 4 °C durante 10 minutos.
- Remova o sobrenadante. Deixe o excesso de acetona residual evaporar, embora não completamente para secar.
- Ressuspender o pellet em 100 μL de tampão de ressuspensão (8% p/v dodecil sulfato de lítio [LDS], 1 mM EDTA em solução salina HEPES 50 mM, pH 7,6), vórtice por 15 s e sonicate até que o pellet esteja dissolvido.
- Centrifugar a solução a 21.000 x g à temperatura ambiente (TR) durante 5 min.
- Transferir o sobrenadante para um novo tubo de 2 mL e adicionar 1,5 mL de solução salina HEPES 50 mM, pH 7,6.
NOTA: A concentração final de LDS nesta etapa é de 0,5%. Concentrações mais altas de LDS podem prejudicar a eficiência de pull-down. Assim, embora o aumento da concentração de LDS possa auxiliar na redução da ligação não específica, também pode reduzir a eficiência do pulldown. Assim, recomenda-se não alterar a concentração de LDS nesta etapa. - Coletar a amostra de "entrada" (Figura 3): transferir 30 μL de lisado de 1 mg/mL para um novo tubo de 1,5 mL e adicionar 10 μL de tampão de amostra Laemmli 4x contendo 6% de β-Mercaptoetanol (BME). Congele a solução e armazene-a a -80 °C.
- Transferir 100 μL de resina de alta capacidade de estreptavidina de volume leito para um novo tubo de 2 mL. Adicionar 1 mL de LDS a 0,5% em solução salina HEPES 50 mM (pH 7,6), centrifugar a 1.500 x g em TR por 2 min e retirar o sobrenadante. Repetir a lavagem com mais 1 mL de solução fisiológica a 0,5% LDS em solução salina HEPES 50 mM (pH 7,6).
- Transferir a solução do passo 6.2.29 para o tubo contendo resina de alta capacidade de estreptavidina pré-lavada a partir do passo 6.2.31 e incubar a solução num misturador de ponta a ponta em RT durante 4-6 horas.
- Centrifugar a mistura a 1.500 x g em RT durante 2 min, tomar 30 μL do sobrenadante e misturá-la com 10 μL de tampão de amostra Laemmli 4x contendo 6% de BME para a amostra de "flowthrough". Em seguida, remova o sobrenadante restante.
NOTA: As amostras de "Flowthrough" podem ser analisadas por western blotting para verificar a eficiência de pull-down da estreptavidina. É essencial remover o máximo possível do sobrenadante para lavar as proteínas não ligadas. Primeiro, remova a maior parte do sobrenadante usando uma pipeta P-1000 e, em seguida, remova o sobrenadante restante usando uma pipeta P-20 com uma ponta de carregamento de gel. - Adicionar 1 mL de LDS a 0,5% em solução salina HEPES 50 mM (pH 7,6) à resina e incubar a mistura por 30 min no TR com rotação término-sobre-extremo.
- Centrifugar a mistura a 1.500 x g em TR por 2 min e retirar o sobrenadante.
NOTA: Normalmente, 0,5% LDS é suficiente para remover a maioria das proteínas de ligação não específicas. Se sinais de ligação não específicos ainda forem vistos na análise posterior, a concentração de LDS no tampão de lavagem pode ser aumentada. - Repita a etapa 6.2.34-6.2.35 mais duas vezes.
- Adicionar 40 μL de tampão de amostra 2x Laemmli contendo 6% de BME à resina.
- Eluir as proteínas ligadas incubando a mistura a 98 °C durante 5 min.
- Centrifugar a mistura a 21.000 x g em TR por 5 min e transferir o sobrenadante para um novo tubo de 1,5 mL. Esta é a amostra "eluída".
NOTA: O carregamento directo da solução contendo resina do passo 6.2.38 no gel pode afectar a análise SDS-PAGE. - Carregar 20 μL em cada poço de gel de poliacrilamida a 10% de 10 pistas e executar eletroforese em gel.
NOTA: Execute o gel em uma tensão mais baixa (120 V) até que a frente do corante atinja o gel de resolução e altere a tensão para 170 V. Pare o programa depois que a frente do corante sair. - Realizar western blotting com anti-HA, anti-Halo ou outros anticorpos que detectem proteínas housekeeping (Figura 3).
- Anestesiar os embriões por incubação a 4 °C. Isso geralmente leva 10 minutos.
- Opção 3: Análise transcriptômica. RNA-seq e qRT-PCR (Figura 4)
NOTA: Recomenda-se vivamente a utilização de embriões colocados dentro de 15 minutos um do outro para este ensaio. A diferença de idade dos embriões afeta significativamente os resultados do ensaio.- Anestesiar os embriões incubando-os a 4 °C por 10 min, 2 h após Z-REX.
- Realizar a descorionação com pinça afiada (passo 6.1.3).
- (Opcional) Realizar segmentação com pinça (por exemplo, separar a cabeça da cauda) se diferentes segmentos forem analisados separadamente.
- Se extrair RNA de um embrião inteiro, transfira de três a cinco embriões para um tubo de 1,5 mL. Se extrair RNA da cabeça ou cauda, transferir 10-12 segmentos dissecados para um tubo de 1,5 mL.
OBS: Recomenda-se a realização do experimento com três a cinco repetições biológicas. - Adicionar 1 mL do reagente TRIzol e esferas de vidro ao tubo.
NOTA: Verificou-se que as esferas de vidro funcionam melhor do que as contas de zircônia para extração de RNA. - Vórtice a mistura por 30 s.
NOTA: Se não prosseguir com o próximo passo imediatamente, a solução pode ser armazenada a -80 °C por 1 a 3 semanas. - Extraia o RNA de acordo com as instruções do fabricante.
- Avaliar a qualidade e concentração do RNA por espectrofotômetro de microvolume e eletroforese em gel de agarose.
NOTA: RNA de boa qualidade deve ter uma relação A260/A280 em torno ou acima de 2,0. - Submeter o RNA para sequenciamento, ou tratar 1 μg de RNA com DNase I grau de amplificação, e transcrever reversamente usando transcriptase reversa sobrescrita III e oligo-(dT)20. Execute esta etapa de acordo com as instruções do fabricante.
- Realizar qRT-PCR e analisar os dados pelo método ΔΔCT9 (Figura 4B-D).
- Opção 4: Análise de expressão e colocalização de POI. Ensaio de imunofluorescência total (Figura 5)
NOTA: Os embriões fixados em formaldeído são frágeis. Evite tremores vigorosos e manuseie com cuidado.- Descorionar os embriões seguindo os passos 6.1.1-6.1.3.
- Transferir os embriões para um tubo de 1,5 mL.
NOTA: Cada trompa deve ter um número igual de embriões, e não mais de 40 embriões. - Após os embriões se acomodarem no fundo do tubo, retirar o sobrenadante e adicionar 1 mL de solução salina tamponada com fosfato (PBS) (pH 7,6).
- Repita a etapa 6.4.3 mais uma vez.
- Retire o sobrenadante, adicione 1 mL de formaldeído a 4% em PBS (pH 7,6) e incube o tubo a 4 °C durante a noite com balanço suave.
NOTA: As amostras em solução de formaldeído podem ser armazenadas a 4 °C durante 1 semana. - Retire o sobrenadante, adicione 1 ml de metanol -20 °C e incube o tubo de lado a -20 °C durante, pelo menos, 18 horas.
NOTA: As amostras podem ser armazenadas a -20 °C por 1 mês ou mais. - Remova o sobrenadante e adicione 1 mL de tampão PDT (0,3% v/v Triton X-100, 0,1% v/v Tween-20 e 1% v/v dimetilsulfóxido [DMSO] em tampão PBS).
- Repetir o passo 6.4.7 e incubar o tubo em RT durante 30 minutos com um balanço suave.
- Remover o sobrenadante, adicionar 1 mL de tampão de bloqueio (10% v/v de soro fetal bovino [SFB] inativado pelo calor, 2% p/v de albumina de soro bovino [BSA] e 0,1% v/v de Tween-20 em tampão PBS) e incubar o tubo em TR por 1 h com balanço suave.
- Remover o sobrenadante e adicionar 200 μL de solução de anticorpos primários (diluída em tampão de bloqueio).
- Retire o sobrenadante, adicione 500 μL de solução de anticorpos primários (diluída em tampão de bloqueio) e incube o tubo a 4 °C durante a noite com balanço suave.
NOTA: Se usar um novo anticorpo primário, vale a pena incluir algumas amostras sem coloração de anticorpos primários para servir como controles negativos. No entanto, idealmente, embriões knockdown morfolino ou nocaute genético modificado, ou embriões em que a expressão da proteína-alvo foi estimulada, são meios mais confiáveis para validar o anticorpo. - Retire o sobrenadante, adicione 1 mL de tampão PDT e incube o tubo em TR por 30 min com balanço suave.
- Repita a etapa 6.4.12.
- Retire o sobrenadante, adicione 1 mL de tampão de bloqueio e incube o tubo em TR por 1 h com balanço suave.
NOTA: As amostras devem ser protegidas da luz após esta etapa, para evitar o fotobranqueamento do fluoróforo conjugado no anticorpo secundário. - Remover o sobrenadante e adicionar 200 μL de solução de anticorpos secundários (diluída em tampão de bloqueio).
- Remover o sobrenadante, adicionar 500 μL de solução de anticorpos secundários (diluída em tampão de bloqueio) e incubar o tubo em TR durante 1,5 h com balanço suave.
- Retire o sobrenadante, adicione 1 mL de tampão PDT e incube o tubo em TR por 30 min com balanço suave.
- Repita a etapa 6.4.17.
- Montar os embriões em placa de agarose a 2% (confeccionada com PBS, pH 7,6) e obter imagens dos embriões com estereomicroscópio (campo claro e respectivos canais fluorescentes) (Figura 5A,B,D,F).
NOTA: Se estiver usando o estereomicroscópio de fluorescência Leica M165 FC, use uma ampliação de 25x para obter imagens com boa resolução. - Quantificar/analisar a intensidade do sinal fluorescente pelo ImageJ (NIH). Use a ferramenta Freehand Selection no ImageJ para quantificar o sinal na região de interesse.
Resultados
Imagem ao vivo de peixes repórteres transgênicos de neutrófilos/macrófagos tratados com Z-REX, Tg(lyz:TagRFP) e Tg(mpeg1:EGFP). Indução de apoptose de neutrófilos/macrófagos através da HNEylation Keap1. (Veja também a Figura 2). O efeito da marcação eletrofílica de Keap1 sobre os níveis de neutrófilos e macrófagos foi avaliado injetando-se embriões transgênicos heterozigotos derivados de Tg(lyz:TagRFP) ou Tg(mpeg1:EGFP) com RNAm codificando Halo-Keap1 e, em seguida, tratando com Ht-PreHNE(alquino). Após os procedimentos para o ensaio passo 6.1-downstream, a opção 1-HNE (alquino) foi liberada e Keap1 foi rotulado. Os níveis de neutrófilos e macrófagos foram avaliados por imagens ao vivo das linhas repórteres, Tg(lyz:TagRFP) e Tg(mpeg1:eGFP), respectivamente. O nível de ambos os tipos celulares diminuiu em 30%-40% após o tratamento com Z-REX, no qual HNE foi entregue a Keap1. Ao contrário, nenhuma perda de neutrófilos ou macrófagos foi observada nos grupos controle técnico Z-REX [sem luz e Ht-PreHNE(alquino), luz isoladamente, ou Ht-PreHNE(alquino) isoladamente] (Figura 1D e Figura 2A-D).
A indução de apoptose de neutrófilos/macrófagos indicou sucesso na liberação de HNE para Keap1 através do Z-REX. Detalhes para a análise da via e mecanismo de apoptose foram publicados5. Para explicar os efeitos fora do alvo de HNE (alquino), vários controles foram usados. (1) Sob as mesmas condições experimentais, em vez de halo-TeV-Keap1 mRNA, embriões foram injetados com halo-P2A-Keap1 mRNA. O ligante P2A permitiu que as proteínas Halo e Keap1 fossem expressas de forma independente. Nesse cenário, HNE(alquino) liberado do Halo não poderia rotular Keap1, pois não era mais proximal ao Halo (Figura 1D); portanto, a via de sinalização da apoptose não foi desencadeada. Não foram observadas alterações nos níveis de macrófagos e neutrófilos neste grupo (Figura 2A,B). (2) As mesmas condições experimentais foram realizadas utilizando RNAm que codifica Halo-TeV-Keap1(C151S,C273W,C288E), um mutante de Keap1 que não responde ao HNE(alquino) (Figura 1D). Não foram observadas alterações nos níveis de macrófagos ou neutrófilos (Figura 2G,H).
Acoplamento azida-clique de biotina e ensaio pull-down de biotina. Avaliação de rotulagem de alvos. (Veja também a Figura 3). A avaliação da marcação de alvos foi realizada usando embriões WT, injetados com RNAm codificando Halo-TeV-Keap1-2xHA (construção de fusão Halo-POI) ou Halo-2xHA-P2A-Keap1-2xHA (construção P2A-split, na qual Halo e Keap1 não são fundidos; Figura 1D). A proteína Keap1 marcada só foi puxada para baixo no grupo que expressa a proteína de fusão e tratada com Z-REX (segunda faixa no blot anti-HA superior), mas não em outros grupos de controle (sem injeção de mRNA, construção de fusão sem Z-REX ou construção de divisão P2A). Os resultados indicam que o HNE (alquino) foi entregue com sucesso a Keap1, e o Keap1 modificado foi subsequentemente conjugado com biotina através da reação de clique, e o Keap1 marcado com biotina foi puxado para baixo pela resina estreptavidina.
Análise transcricional. RNA-seq e qRT-PCR. (Veja também a Figura 4). A alteração transcricional após o tratamento com Z-REX foi avaliada por RNA-seq e qRT-PCR. No RNA-seq, vários genes relacionados à imunidade foram regulados negativamente após o Z-REX. Em contraste, muitos genes relacionados à resposta antioxidante (AR) foram upregulated após Z-REX, o que resultou da indução da via Keap1-Nrf2-AR na HNEylation em Keap110 (Figura 4A). Na análise por qRT-PCR, resultados semelhantes foram encontrados ao analisar três genes relacionados à imunidade (lyz, mpeg1.1 e coro1a) (Figura 4B). A up- e down-regulation dos respectivos genes mostrou o sucesso da indução de vias mediadas por Keap1 HNEylation.
Ensaio de coloração de (co)imunofluorescência de montagem total e análise de colocalização. (Veja também a Figura 5). A expressão exógena de Halo-TeV-Keap1-2xHA e Halo-2xHA-P2A-Keap1-2xHA foi avaliada pela coloração de imunofluorescência total (IF) (Figura 5A,B). O P2A-split-construct apresentou duas vezes o número de tags HA do que o TeV-fusion-construct, o que corresponde a um sinal anti-HA duas vezes maior no grupo P2A-split-construct-mRNA-injectado do que o outro, indicando que o nível de expressão dos dois construtos foi semelhante (Figura 5C). Os níveis de expressão do Halo-TeV-Keap1 (wt) e Halo-TeV-Keap1(C151S,C273W,C288E) também foram encontrados semelhantes quando sondados com anti-Halo (Figura 5D,E). A colocalização de neutrófilos e caspase 3 ativa em Tg (lyz:TagRFP) tratada com Z-REX foi observada por co-imunomarcação com anti-RFP e anti-Caspase 3 ativa (Figura 5F). A Caspase Ativa 3 é um indicador de eventos de apoptose.
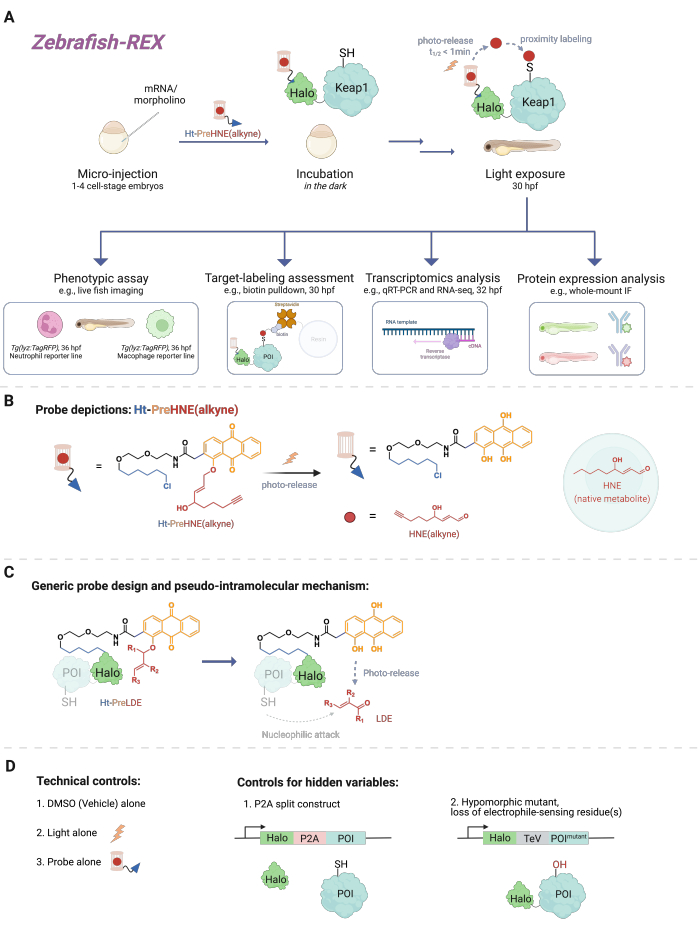
Figura 1: Fluxo de trabalho do Z-REX. (A,B) Um embrião de peixe-zebra em estágio celular 1-4 é injetado com (morfolino e) RNAm que codifica Halo-POI (por exemplo, Halo-Keap1). Os embriões injetados são então tratados com uma sonda composta por um ligante HaloTag e um eletrófilo fotocagelado anexado com um grupo funcional alquino, como Ht-PreHNE (alquino) em B. Depois de remover a quantidade excessiva de sonda, o embrião é exposto à luz para liberar o eletrófilo de interesse [por exemplo, HNE ou seu análogo, HNE (alquino)]. A análise a jusante é realizada em um determinado momento/definido pelo usuário. (C) Projeto e mecanismo da sonda Ht-PreLDE, que é aplicável a diferentes eletrófilos derivados de lipídios (LDE). (D) Grupos de controle negativo/técnico para Z-REX. Clique aqui para ver uma versão maior desta figura.
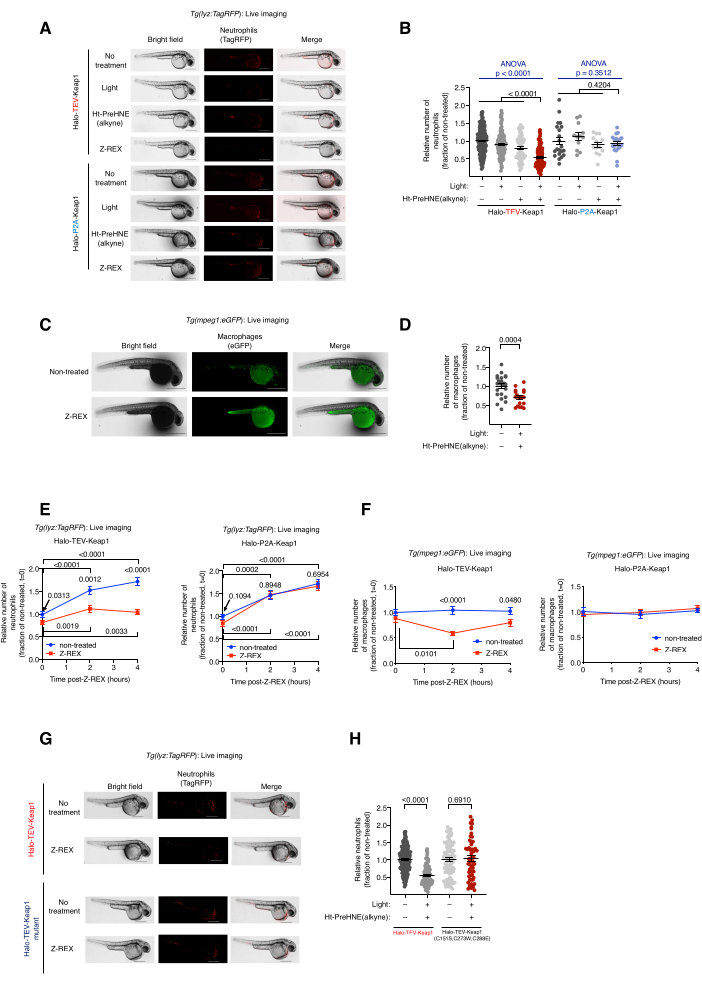
Figura 2: Imagem ao vivo de peixes repórteres neutrófilos/macrófagos transgênicos submetidos ao Z-REX. Keap1 HNEylation mediada por Z-REX-induz apoptose de neutrófilos/macrófagos. (A) Imagens representativas de peixes Tg(lyz:TagRFP ) expressando Halo-TeV-Keap1 (construção de fusão) ou Halo-P2A-Keap1 (construção dividida), e submetidos a condições de controle negativo [sem tratamento, luz isoladamente, ou Ht-PreHNE(alquino) sozinho ou Z-REX]. Idade do embrião: 36 hpf. (B) Quantificação dos níveis de neutrófilos em A. (C) Imagens representativas de peixes Tg(mpeg1:eGFP) expressando Halo-TeV-Keap1 com ou sem tratamento Z-REX. Idade do embrião: 34 hpf. (D) Quantificação dos níveis de macrófagos em C. (E,F) Medição ao longo do tempo dos níveis de (E) neutrófilos e (F) macrófagos após o tratamento com Z-REX. (G) Experimento semelhante ao de A em peixes que expressam Halo-TeV-Keap1 (WT) ou Halo-TeV-Keap1 (C151S, C273W, C288E), um mutante que não tem capacidade de detecção de HNE. (H) Quantificação dos níveis de neutrófilos em G. Barras de escala: 500 μm. Todos os gráficos são apresentados com média ± EPM. Os valores de p foram calculados com ANOVA one-way (azul) e teste t de Student bicaudal (preto). Esse valor foi modificado a partir de Poganik et al.7. Clique aqui para ver uma versão maior desta figura.

Figura 3: Ensaio pull-down de biotina. Embriões WT expressando Halo-TeV-Keap1-2XHA ou Halo-2XHA-P2A-Keap1-2XHA foram tratados com Z-REX ou respectivas condições de controle negativo (nenhum tratamento com sonda neste caso). Após a colheita, os embriões foram lisados e tratados com protease TeV antes do ensaio de puxamento da biotina. Os resultados foram analisados por western blotting. Este valor foi modificado de Huang et al. Z-REX: conduzindo eletrófilos reativos a proteínas específicas expressas de forma específica ou ubíqua do tecido, e registrando as respostas redox induzidas por eletrófilos funcionais resultantes em larvas de peixes. Este valor foi modificado de Huang et al.11. Clique aqui para ver uma versão maior desta figura.
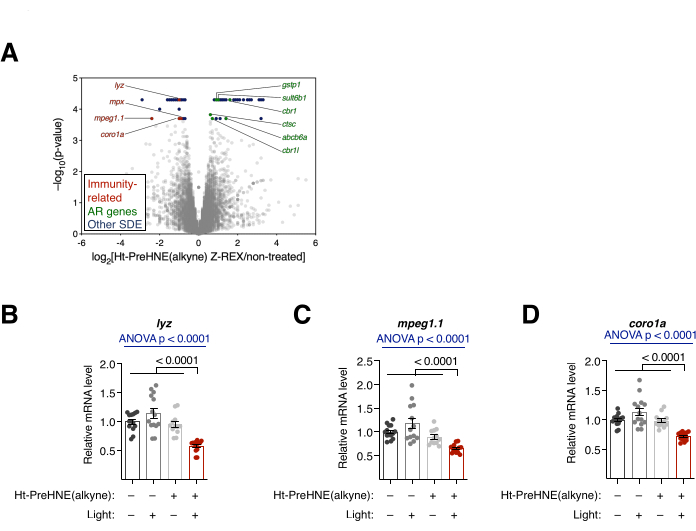
Figura 4: Análise transcricional. (A) Resultados RNA-seq de embriões tratados com Z-REX versus não tratados. Genes diferencialmente expressos estatisticamente significativos (SDE) são destacados. Os genes SDE relacionados à imunidade são coloridos de vermelho. Os genes relacionados à resposta antioxidante (RA) são coloridos de verde. Outros genes SDE são coloridos de azul. Todos os valores de p foram calculados com CuffDiff. (B-D) Três genes SDE relacionados à imunidade de A: (B) lyz, (C) mpeg1.1 e (D) coro1a foram analisados com qRT-PCR, e apenas os embriões tratados com Z-REX mostraram a supressão desses transcritos. Todos os gráficos são apresentados com média ± EPM. Os valores de p foram calculados com ANOVA one-way (azul) e teste t de Student bicaudal (preto). Esse valor foi modificado a partir de Poganik et al.7. Clique aqui para ver uma versão maior desta figura.
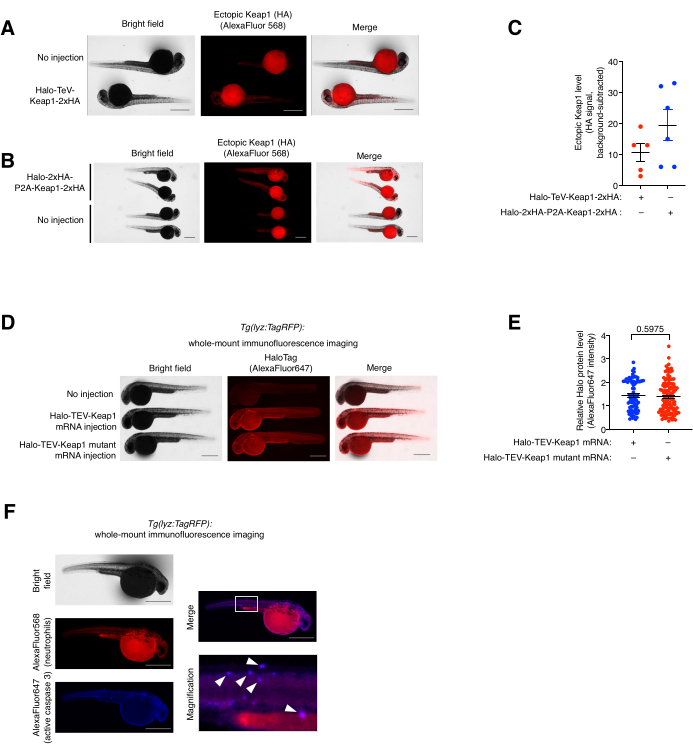
Figura 5: Ensaio de imunofluorescência total . (A,B) Imagens representativas de embriões expressando (A) Halo-TeV-Keap1-2xHA ou (B) Halo-2xHA-P2A-Keap1-2xHA imunomarcados com anti-HA e anticorpo secundário conjugado com AlexaFluor568. Peixes injetados com mRNA foram comparados com peixes não injetados pareados por idade. (C) Quantificação do sinal anti-AH em (A,B). (D) Imagens representativas de embriões expressando Halo-TeV-Keap1 (WT) ou Halo-TeV-Keap1 (C151S, C273W, C288E) imunomarcados com anti-Halo e anticorpo secundário conjugado com AlexaFluor647. Peixes injetados com mRNA foram comparados com peixes não injetados pareados por idade. (E) Quantificação do sinal anti-Halo em D. Os valores de p foram calculados com o teste t de Student bicaudal. (F) Embriões Tg(lyz:TagRFP) submetidos a Z-REX foram co-imunomarcados com anti-RFP e anti-Caspase 3 anti-ativa, e respectivos anticorpos secundários conjugados a fluoróforos. A caixa branca marca a área ampliada. As setas brancas indicam colocalizações de neutrófilos e caspase ativa 3. Barras de escala: 500 μm. Todos os gráficos são apresentados com média ± EPM. Esse valor foi modificado a partir de Poganik et al.7. e Huang et al.11. Clique aqui para ver uma versão maior desta figura.
Tabela Suplementar 1: Lista dos buffers utilizados neste estudo. Clique aqui para baixar este arquivo.
Discussão
Z-REX descrito neste protocolo demonstra uma estratégia robusta para investigação de pares eletrófilos-alvo e deconvolução de vias de sinalização em peixes vivos. A entrega direcionada à proximidade permite a dosagem e o controle espacial do tratamento com compostos eletrofílicos. Ao contrário dos métodos convencionais de dosagem em bolus, nos quais as concentrações suprafisiológicas de eletrófilos implantados muitas vezes levam a problemas fora do alvo, a quantidade relativamente pequena de eletrófilo liberada para o sistema torna o Z-REX amplamente não invasivo. Utilizamos 0,1-6 μM de Ht-PreHNE (alquino) em embriões de zebrafish, e os resultados mostraram que o tratamento não é prejudicial ao desenvolvimento embrionário11.
O procedimento Z-REX é geralmente mais longo do que o T-REX, uma técnica para triagem/estudo de proteínas sensíveis a eletrófilos em células cultivadas. Suponha que o propósito do experimento seja rastrear interações eletrophile-target; sugerimos primeiramente realizar uma triagem extensiva por T-REX em células cultivadas e usar Z-REX para validação in vivo e análise fenotípica/via. Em comparação com a cultura celular, os requisitos para a realização do Z-REX são técnicas básicas de criação de peixes, além das habilidades experimentais bioquímicas exigidas pelo T-REX. Um período de tempo típico para Z-REX (do cruzamento de peixes à entrega de eletrófilos induzíveis por luz) é de 2-3 dias, que não é mais do que 1 dia a mais do que o tempo típico para um experimento de T-REX em células vivas transfectadas. Imagens ao vivo para análise fenotípica podem ser realizadas 2-10 h após a iluminação da luz; o acoplamento de clique com biotina-azida para ensaio pull-down leva 3 dias; qRT-PCR para dosagem de resposta transcricional leva 3 dias; A coloração IF leva 5 dias. Essas etapas são aproximadamente semelhantes aos seus equivalentes de cultura celular, embora a interpretação dos dados exija uma compreensão da fisiologia dos peixes e das cepas repórteres.
Como procedimento de múltiplas variáveis12, vários grupos de controle são necessários para que o Z-REX exclua incertezas nos resultados (Figura 1D). Os grupos de controle comuns são: (1) DMSO/tratamento do veículo apenas; (2) tratamento com sonda, mas sem iluminação luminosa; (3) iluminação luminosa, mas sem tratamento com sonda; (4) construção P2A-split, na qual Halo e o POI são expressos separadamente, de modo que a entrega de proximidade é ablida; e (5) mutantes hipomórficos, cujos resíduos sensíveis a eletrófilos são/são mutados, como Akt3 (C119S)6 e Keap1 (C151S, C273W, C288E)5, que já utilizamos em estudos anteriores.
Se os ensaios a jusante envolverem análise de western blot, a desidratação deve ser realizada antes da colheita. As proteínas da gema reduzem a fidelidade das avaliações da concentração de lisado e podem ligar-se não especificamente aos anticorpos. Ao realizar imagens de peixes vivos ou coloração de FI de montagem inteira, também observamos sinais fluorescentes inespecíficos no saco vitelino, provavelmente resultantes de proteínas autofluorescentes no saco vitelino, ou ligação inespecífica dos próprios anticorpos. Se o sinal de autofluorescência interferir com o sinal, sugerimos excluir o saco vitelínico da quantificação, ou quantificar diferentes regiões separadamente. A descorinação é necessária para imagens de peixes vivos e ensaio de coloração de FI de montagem inteira. O córion pode interferir na imagem e, posteriormente, na quantificação/contagem celular. No entanto, a descorinação só é aplicável a embriões com mais de 1 dpf; embriões mais jovens nos estágios de blastulação/gastrulação/segmentação são frágeis demais para serem descorionados.
O protocolo Z-REX descrito aqui é baseado na expressão de POI ectópica impulsionada por mRNA. O procedimento é rápido em comparação com o uso/geração de linhas de peixes transgênicos. A expressão impulsionada por mRNA é ubíqua e transitória, e dura pelo menos 2 dias para mRNAs usados neste protocolo. No entanto, é provável que a duração da expressão varie em outros casos. Assim, essa abordagem fornece uma janela investigativa rápida e mais global sobre os efeitos de um evento específico de marcação de eletrófilos, compatível com vários ensaios de alto rendimento/alto conteúdo. Linhagens transgênicas com expressão estável de Halo-POI em tecidos específicos também são compatíveis com Z-REX11. Tais linhas são melhor usadas quando uma pergunta mais precisa precisa ser feita, por exemplo, quando um fenótipo em um órgão específico é previsto a partir de dados de cultura celular, ou quando a triagem de experimentos de injeção de RNAm prevê que um órgão específico é sensível a um evento de marcação de letrófilos. Uma indução de resposta antioxidante cardíaca específica através de Z-REX foi demonstrada usando peixes Tg(gstp1:GFP;DsRed-P2A-myl7:Halo-TeV-Keap1) em nossa publicação anterior11. Também pode ser possível realizar Z-REX em peixes transgênicos com mais de 2 dpf.
Divulgações
Inibidores de pequenas moléculas quinases específicos para isoformas, cuja descoberta foi possibilitada pelas tecnologias REX, foram depositados para pedido de patente
Agradecimentos
Financiamento: Novartis FreeNovation, NCCR e EPFL.
Materiais
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol (BME) | Sigma-Aldrich | M6250 | |
| 1.5-mL microcentrifuge tube | Axygen | MCT-150-C-S | |
| 10-cm Petri dishes | Celltreat | 229692 | |
| 2-mL microcentrifuge tube | Axygen | MCT-200-C-S | |
| 30% Acrylamide and bis-acrylamide solution | BioRad | 1610154 | |
| 6-well plate | Celltreat | 229506 | |
| Acetone | Fisher | A/0600/15 | |
| Agarose | GoldBio | A201-100 | |
| All Blue Prestained Protein Standards | BioRad | 1610373 | |
| Ammonium persulfate | Sigma | A3678 | |
| Biotin-dPEG11-azide | Quanta Biodesign | 102856-142 | |
| Bradford Dye | BioRad | 5000205 | |
| BSA | Fisher | BP1600-100 | |
| Capillary tubes | VWR | HARV30-00200 | |
| Chloroform | Supelco, Inc | 1.02445.1000 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Cu(TBTA) | Lumiprobe | 21050 | |
| CuSO4 | Sigma | 209198 | |
| DMSO | Fisher | D128-500 | |
| Donkey anti-mouse-Alexa Fluor 647 | Abcam | ab150107 | 1:800 (IF) |
| Donkey anti-rabbit-Alexa Fluor 647 | Abcam | ab150075 | 1:1000 (IF) |
| Donkey anti-rat-AlexaFluor 568 | Abcam | ab175475 | 1:1000 (IF) |
| ECL substrate | Thermo Fisher Scientific | 32106 | |
| ECL-Plus substrate | Thermo Fisher Scientific | 32132 | |
| End-to-end rotator | FinePCR | Rotator AG | |
| Ethanol | Fisher | E/0650DF/15 | |
| Ethidium bromide | Sigma | E1510 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument Co. | P-97 | |
| Fluorescence stereomicroscope | Leica | M165 FC | |
| Forceps (blunt) | self made | self made by blunting sharp forceps (Fine Science Tools, Dumont #5, 11252-40) | |
| Forceps (sharp) | Fine Science Tools | Dumont #5, 11252-40 | |
| Gel loading tip | Fisher | 02-707-181 | |
| Gel/blot imager | Vilber | Fusion FX imager | |
| Glass beads | Sigma | 45-G1145 | |
| Glass stage micrometer | Meiji Techno. | MA285 | |
| Heat inactivated FBS | Sigma | F2442 | |
| Heated ultrasonic bath | VWR | 89375-470 | |
| HEPES | Fisher | BP310-1 | |
| High capacity streptavidin agarose | Thermo Fisher Scientific | 20359 | |
| Ht-PreHNE alkyne probe | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Imaging plate (10% HBSS buffer, for live embryos) | Made with Petri dish, and 2% agarose in 10% HBSS buffer | ||
| Imaging plate (PBS, for formaldehyde-fixed embryos) | Made with Petri dish, and 2% agarose in PBS | ||
| Injection plate | Made with microinjection mold, Petri dish, and 2% agarose in 10% HBSS buffer | ||
| LDS | Apollo | APOBI3331 | |
| Methanol | VWR | 20864.32 | |
| Microinjection mold | Adaptive Science Tools | TU-1 | |
| Microloader tips | Eppendorf | 930001007 | |
| Micromanipulator | Narishige | MN-153 | |
| Microscope for micro-injection | Olympus | SZ61 | |
| Microscope light source | Olympus | KL 1600 LED | |
| Mineral oil | Sigma | M3516 | |
| mMessage mMachine SP6 Transcription Kit | Ambion | AM1340 | |
| Mouse anti- β-actin-HRP | Sigma | A3854 | 1:20000 (WB) |
| Mouse anti-HaloTag | Promega | G921A | 1:500 (IF) |
| Multi-mode reader | BioTek Instruments | Cytation 5 | |
| Nucleic acid agarose gel electrophoresis apparatus | Biorad | Mini-Sub Cell GT Systems | |
| Oligo(dT)20 | Integrated DNA Technologies | customized: (dT)20 | |
| Orange G | Sigma | O3756 | |
| Paraformaldehyde | Sigma | P6148 | |
| PBS | Gibco | 14190144 | |
| pCS2+8 Halo-2XHA-P2A-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| pCS2+8 Halo-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| Pneumatic PicoPump | WPI | SYS-PV820 | |
| Protein electrophoresis equipment and supplies | Biorad | Mini-PROTEAN Tetra Vertical Electrophoresis | |
| Rabbit anti-active Caspase-3 | BD Pharmingen | 559565 | 1:800 (IF) |
| Rat anti-HA-HRP | Sigma | H3663 | 1:500 (IF and WB) |
| Rat anti-RFP | ChromoTek | 5F8 | 1:800 (IF) |
| Refrigerated centrifuge | Eppendorf | 5417R | |
| RNAseOUT recombinant ribonuclease inhibitor | ThermoFisher Scientific | 10777019 | |
| RnaseZap RNA decontamination solution | ThermoFisher Scientific | AM9780 | |
| Rocking Shaker | DLAB | SK-R1807-S | |
| SDS | Teknova | S9974 | |
| SuperScript III reverse transcriptase | ThermoFisher Scientific | 18080085 | |
| t-Butanol | Sigma | 471712 | |
| TCEP-HCl | Gold Biotechnology | TCEP1 | |
| TeV protease (S219V) | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Tg(lyz:TagRFP) | Zebrafish International Resource Center (ZIRC) | uwm11Tg (ZFIN) | - |
| Tg(mpeg1:eGFP) | Zebrafish International Resource Center (ZIRC) | gl22Tg (ZFIN) | - |
| Thermal cycler | Analytik Jana | 846-x-070-280 | |
| TMEDA | Sigma | T7024 | |
| Transfer pipets | Fisher | 13-711-9D | |
| Tris | Apollo | APOBI2888 | |
| Triton X-100 | Fisher | BP151-100 | |
| TRIzol reagent | Thermo Fisher Scientific | 15596018 | |
| Trypsin inhibitor from Glycine max (soybean) | Sigma | T9003 | |
| Tween 20 | Fisher | BP337-500 | |
| UV lamp with 365-nm light | Spectroline | ENF 240C | |
| UV meter | Spectroline | XS-365 | |
| Vortexer | Scientific Industries, Inc. | Vortex-Genie 2 | |
| Western Blotting Transfer equipment and supplies | Biorad | Mini Trans-Blot or Criterion Blotter | |
| Zebrafish husbandry and breeding equipment | in house | ||
| Zirconia beads | BioSpec | 11079107zx |
Referências
- Long, M. J. C., Ly, P., Aye, Y. A primer on harnessing non-enzymatic post-translational modifications for drug design. RSC Medicinal Chemistry. 12 (11), 1797-1807 (2021).
- Parvez, S., Long, M. J. C., Poganik, J. R., Aye, Y. Redox signaling by reactive electrophiles and oxidants. Chemical Reviews. 118 (18), 8798-8888 (2018).
- Parvez, S., et al. T-REX on-demand redox targeting in live cells. Nature Protocols. 11 (12), 2328-2356 (2016).
- Long, M. J. C., et al. Precision electrophile tagging in Caenorhabditis elegans. Biochemistry. 57 (2), 216-220 (2018).
- Van Hall-Beauvais, A., Zhao, Y., Urul, D. A., Long, M. J. C., Aye, Y. Single-protein-specific redox targeting in live mammalian cells and C. elegans. Current Protocols in Chemical Biology. 10 (3), 43 (2018).
- Long, M. J. C., et al. Akt3 is a privileged first responder in isozyme-specific electrophile response. Nature Chemical Biology. 13 (3), 333-338 (2017).
- Poganik, J. R., et al. Wdr1 and cofilin are necessary mediators of immune-cell-specific apoptosis triggered by Tecfidera. Nature Communications. 12 (1), 5736 (2021).
- Lin, H. -. Y., Haegele, J. A., Disare, M. T., Lin, Q., Aye, Y. A generalizable platform for interrogating target- and signal-specific consequences of electrophilic modifications in redox-dependent cell signaling. Journal of the American Chemical Society. 137 (19), 6232-6244 (2015).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative CT method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Wakabayashi, N., et al. Keap1-null mutation leads to postnatal lethality due to constitutive Nrf2 activation. Nature Genetics. 35 (3), 238-245 (2003).
- Huang, K. -. T., et al. Z-REX: Shepherding reactive electrophiles to specific proteins expressed either tissue-specifically or ubiquitously, and recording the resultant functional electrophile-induced redox responses in larval fish. Nature Protocols. , (2023).
- Long, M. J. C., Assari, M., Aye, Y. Hiding in plain sight: The issue of hidden variables. ACS Chemical Biology. 17 (6), 1285-1292 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados