Method Article
Überwachung von On-Target-Signalantworten in Zebrafischlarven - Z-REX entschlüsselt präzise Mechanismen elektrophiler Wirkstoffe und Metaboliten
In diesem Artikel
Zusammenfassung
Zebrafish targeting reactive electrophiles and oxidants (Z-REX) ist eine auf chemischer Biologie basierende Methode zur Untersuchung reaktiver niedermolekularer Signalwege. Diese Technik kann auf lebende Fische in verschiedenen Entwicklungsstadien angewendet werden. Hier koppeln wir Standard-Assays im Zebrafisch mit Z-REX für die Analyse des Signalwegs.
Zusammenfassung
Reaktive Metaboliten und verwandte elektrophile Wirkstoffe gehören zu den am schwierigsten zu untersuchenden kleinen Molekülen. Konventionelle Ansätze zur Dekonstruktion der Wirkungsweise (MOA) solcher Moleküle nutzen die Massenbehandlung von Versuchsproben mit einem Überschuss an einer bestimmten reaktiven Spezies. Bei diesem Ansatz ermöglicht die hohe Reaktivität von Elektrophilen eine nicht-diskriminierende Markierung des Proteoms in einer zeit- und kontextabhängigen Weise; Auch redoxsensitive Proteine und Prozesse können indirekt und oft irreversibel beeinflusst werden. Vor dem Hintergrund unzähliger potenzieller Targets und indirekter Sekundäreffekte bleibt die Verknüpfung des Phänotyps mit dem spezifischen Target-Engagement eine komplexe Aufgabe. Zebrafische, die auf reaktive Elektrophile und Oxidationsmittel (Z-REX) abzielen - eine On-Demand-Plattform für die reaktiv-elektrophile Verabreichung, die für den Einsatz in Zebrafischlarven geeignet ist - wurde entwickelt, um Elektrophile an ein bestimmtes Protein von Interesse (POI) in ansonsten ungestörten lebenden Fischembryonen zu liefern. Zu den wichtigsten Merkmalen dieser Technik gehören ein geringes Maß an Invasivität sowie eine dosis-, chemotyp- und raumzeitgesteuerte elektrophile Präzisionsabgabe. In Verbindung mit einer einzigartigen Reihe von Kontrollen umgeht diese Technik somit Off-Target-Effekte und systemische Toxizität, die sonst nach unkontrollierter Massenexposition von Tieren gegenüber reaktiven Elektrophilen und pleiotropen elektrophilen Medikamenten beobachtet werden. Mit Hilfe von Z-REX können Forscher verstehen, wie sich individuelle Stressreaktionen und Signalausgänge als Ergebnis einer spezifischen reaktiven Ligandenbindung an einen bestimmten POI unter nahezu physiologischen Bedingungen in intakten lebenden Tieren verändern.
Einleitung
Eine Vielzahl von zellulären Signalereignissen umfasst Reaktionen zwischen kleinen reaktiven Molekülen (endogen in der Zelle produziert oder Xenobiotika/Xenometaboliten, wie z. B. Medikamente) und ihrem Proteinziel. In vielen Fällen kann ein substöchiometrisches Niveau solcher kovalenten Bindungsereignisse zelluläre Reaktionen auslösen, die zu Veränderungen z. B. in der Entwicklung, im Stoffwechsel, in der Apoptose und/oder in der Immunantwort führen1. Die Dekonstruktion der Wirkungsweise (MOA) durch die Verknüpfung spezifischer Bindungsereignisse mit ihren phänotypischen Konsequenzen hat sich jedoch als schwierig erwiesen. Herkömmliche Bolusdosierungsmethoden, bei denen hohe Konzentrationen der reaktiven Spezies eingebracht werden, führen häufig zu einer Vielzahl von Proteinen, die modifiziert werden, sowie zu einer übermäßigen Toxizität für den Modellorganismus2. Solche Bedingungen sind alles andere als ideal. Es wurde eine Methode entwickelt, um diese Probleme in der Zellkultur zu lösen, indem eine präzise lokalisierte elektrophile Verabreichung in einem nativen zellulären Kontext verwendet wird, genannt T-REX (targetable reactive electrophiles and oxidants)3. In der Zwischenzeit hat sich der Fokus auf Experimente an ganzen Organismen verlagert, die die Möglichkeit bieten, Proteine in bestimmten zellulären Kontexten in nicht transformierten Zellen zu untersuchen. Daher haben wir die Technik so erweitert, dass sie mit mehreren Modellen kompatibel ist, einschließlich Danio-Rerio-Embryomodellen . In dieser Arbeit stellen wir Z-REX (Zebrafische gegen reaktive Elektrophile und Oxidationsmittel) vor (Abbildung 1).
Um Z-REX zu verstehen, werden in diesem Artikel zunächst REX-Technologien und die ihnen zugrunde liegenden Konzepte vorgestellt. Im Kern modellieren diese Techniken die Signalübertragung endogener physiologischer reaktiver elektrophiler Spezies (RES), indem sie nachahmen, wie natürliche Elektrophile lokal in vivo mit raumzeitlicher Präzision produziert werden. Das interessierende Protein (POI) wird als Fusionskonstrukt zu Halo exprimiert; Letzteres verankert die gewebedurchlässige und inerte Sonde, die das photocaged RES trägt, in einer 1:1-Stöchiometrie. Ein solches endogenes RES ist 4-Hydroxynonenal (im Folgenden HNE), das in der Sonde Ht-PreHNE photocaged ist. In vielen Fällen verwenden wir eine alkinfunktionalisierte Version von HNE [d.h. HNE(alkin)], die im Wesentlichen identische biologische Eigenschaften wie HNE hat, aber durch Click-Chemie markiert werden kann. Die Sonde, die ebenfalls mit einem Chloralkan für die Reaktivität mit Halo funktionalisiert ist, wird als Ht-PreHNE (Alkin) bezeichnet. Der Komplex der Halo-POI-Fusion und der so gebildeten Sonde ermöglicht die proximale Abgabe von RES an den fusionierten POI bei Bestrahlung mit UV-Licht. Reagiert der POI schnell mit dem freigesetzten RES, ermöglicht die daraus resultierende kovalente Markierung des POI mit RES die Identifizierung kinetisch privilegierter Cysteine.
Z-REX nutzt die oben genannten Vorteile der REX-Technologien und wendet sie breit an, um spezifische Signalwege in lebenden Fischen zu untersuchen. Dieses Protokoll wurde für Zebrafische (D. rerio) optimiert, da es sich um genetisch handhabbare Wirbeltierorganismen handelt, die während der Entwicklung transparent sind und sich daher ideal für optochemische/-genetische Techniken wie REX-Technologien eignen. Nichtsdestotrotz dürfte eine ähnliche Strategie auch bei anderen genetisch beherrschbaren Fischarten gut funktionieren, da die breite Anwendbarkeit der Methode auf den pseudo-intramolekularen Mechanismus der Lipid-abgeleiteten elektrophilen (LDE) Verabreichung zurückzuführen ist. In der Tat ist das Verfahren sehr biokompatibel, da Fische mindestens 48 Stunden lang mit dem photokäfig-elektrophilen Z-REX [z. B. Ht-PreHNE(alkin)] behandelt werden können, ohne dass die Entwicklung spürbar beeinträchtigt wird. Ein ähnliches Protokoll funktioniert in C. elegans 4,5.
Das Protokoll beschreibt zunächst die Verwendung der mRNA-Injektion zur Erzeugung einer transienten Expression eines nicht-nativen Halo-POI-Fusionskonstrukts in embryonalen Zebrafischmodellen, 1-1,5 Tage nach der Befruchtung (dpf). Dies führt dazu, dass das ektopische Protein in den meisten Zellen innerhalb des Fisches (im Folgenden als "ubiquitär" bezeichnet) und nicht in bestimmten Geweben oder Lokalitäten exprimiert wird. Die Daten zeigen jedoch, dass in bestimmten Fällen zellspezifische Effekte beobachtet werden können. Nach der Injektion werden die Embryonen mit einer niedrigen Konzentration [0,3-5 μM Ht-PreHNE(alkin)] der Sonde bis zu 30,5 h nach der Befruchtung (hpf) inkubiert. Dann, zu einer vom Benutzer vorgeschriebenen Zeit, wird die Abgabe des RES an den POI innerhalb des Fisches durch Photouncaging für 2-5 Minuten erreicht. Nach dem Photouncaging des RES kann in den nächsten 2-10 Stunden eine Vielzahl von nachgeschalteten phänotypischen Assays durchgeführt werden: 1) Live-Bildgebung von Reporterlinien (Abbildung 2A); 2) Bewertung der Zielkennzeichnung durch Western-Blot-Analyse (Abbildung 3); 3) transkriptomische Analyse (Abbildung 4); oder 4) Whole-Mount-Immunfluoreszenz (Abbildung 5).
Als Beispiel für die Live-Bildgebung von Reporterlinien wird Z-REX zusammen mit der Live-Bildgebung von Fischlinien, Tg(lyz:TagRFP) und Tg(mpeg1:eGFP), demonstriert, um zu messen, wie die RES-Modifikation eines bestimmten elektrophilen Sensor-POI (nämlich Keap1) die Neutrophilen- bzw. Makrophagenkonzentration senkt, ohne beobachtbare Auswirkungen auf andere Zellen im Fisch. Wir haben jedoch bereits gezeigt, dass die POI-Markierung und der daraus resultierende Signalweg aus T-REX-Studien mit Hilfe von Z-REX für mehrere Proteine reproduziert werden können: Akt3 6, Keap17 und Ube2v26. Insgesamt können Wissenschaftler mit Z-REX die Konsequenz(en) der kovalenten Modifikation von POIs durch RES im Kontext mehrerer komplexer Redoxwege untersuchen. Diese Technik ist darauf ausgelegt, Targets und ihre funktionellen Reste für das kovalente Wirkstoffdesign und neuartige Wirkstoffmechanismen in einem kontextuell relevanteren Ganztiermodell zu lokalisieren.
Protokoll
Die Zebrafischhaltung und der Umgang mit Zebrafischen an der Cornell University (USA) wurden nach den Richtlinien der National Institutes of Health (NIH) durchgeführt und vom Institutional Animal Care and Use Committee (IACUC) genehmigt. Die Zebrafischhaltung und der Umgang mit Zebrafischen in der Abteilung Zebrafische der Eidgenössischen Technischen Hochschule Lausanne (EPFL, Schweiz) wurden nach dem Tierschutzgesetz SR 455 und der Tierschutzverordnung SR 455.1 mit kantonaler Veterinärbewilligung VD-H23 durchgeführt.
HINWEIS: In diesem Protokoll werden Tg(lyz:TagRFP ) und Tg(mpeg1:EGFP) Fischlinien, die Halo-TeV-Keap1 exprimieren, verwendet, um Z-REX zu demonstrieren. Die Methode kann auf andere Proteine von Interesse, transgene Reporterfischlinien und nachgeschaltete biologische Assays ausgeweitet werden. In der ergänzenden Tabelle 1 finden Sie Informationen zu den in dieser Studie verwendeten Puffern. Alle Reagenzien, Instrumente, Geräte, Antikörper, Plasmide, Zebrafischstämme und Geräte sind in der Materialtabelle aufgeführt.
1. mRNA-Präparation
- Halo-TeV-Keap1-2xHA und Halo-2xHA-P2A-Keap1-2xHA mRNA werden mit dem mMessage mMachine SP6 In-vitro-Transkriptionskit hergestellt.
Anmerkungen: Befolgen Sie die Anweisungen des Herstellers und führen Sie für jede mRNA Reaktionen im 40-μl-Maßstab durch. Lösen Sie das mRNA-Pellet in 10 μl nukleasefreiem Wasser auf. - Beurteilung der mRNA-Qualität und Messung der Konzentration mittels Mikrovolumen-Spektralphotometer und Agarose-Gelelektrophorese. Eine mRNA von guter Qualität sollte ein A-260/A-280-Verhältnis von etwa oder mehr von 2,0 aufweisen.
- Verdünnen Sie die mRNA auf 1-1,5 mg/ml mit nukleasefreiem Wasser.
- Aliquotieren Sie die mRNA-Lösung (1-2 μl pro Röhrchen) und lagern Sie die Aliquots bei -80 °C.
2. Produktion von Fischembryonen
- Option 1: Produktion von Wildtyp-Fischembryonen (WT).
- Richten Sie 10 Fischkreuzungspaare in 10 separaten Becken ein, wobei jedes Becken eine Trennwand zwischen dem männlichen und dem weiblichen Elternfisch enthält.
HINWEIS: Insgesamt 10 Kreuzungspaare liefern in der Regel eine ausreichende Anzahl von Embryonen für die Assays. Die Anzahl der Kreuzungspaare kann je nach Versuchsplanung/Bedarf und Fruchtbarkeit der Elternfische angepasst werden. - Entfernen Sie am nächsten Morgen nach dem Einrichten des Injektors die Trennwände in fünf der Tanks. Warten Sie 30 Minuten, bis sich der Fisch paart.
- Bringen Sie die Elternfische in ein anderes Becken, sammeln Sie die Embryonen, indem Sie das Aquarienwasser durch ein Sieb geben, und spülen Sie die Embryonen dann aus dem Sieb in eine 10 cm große Petrischale. Diese Embryonen werden für die erste Runde der Injektionen verwendet.
HINWEIS: Wenn die Qualität der Eizellen aus einer bestimmten Charge schlecht ist (z. B. wenn die Eizellen aufgrund der Aggregation von Proteinen undurchsichtig sind), sollten Sie sie nicht mit den anderen Embryonen kombinieren. - (Optional) Führen Sie ähnliche Verfahren wie in den Schritten 2.1.2 bis 2.1.3 beschrieben an den anderen fünf Tanks für die nächste Injektionsrunde durch.
HINWEIS: Es ist am besten, nur eine Injektionsrunde durchzuführen, um den Altersunterschied zwischen den Embryonen zu minimieren. Sollten jedoch mehr Embryonen benötigt werden, als in einer Charge injiziert werden können, wird empfohlen, zwei Injektionsrunden durchzuführen, um sicherzustellen, dass die Embryonen während der mRNA-Injektion in einem 1-4-Zell-Stadium verbleiben. Die Anzahl der Injektionsrunden kann je nach Injektionsgeschick und Versuchsplanung des Bedieners angepasst werden. Es wird jedoch empfohlen, den gesamten Eingriff (von der Entfernung des ersten Teilers bis zur Injektion des letzten Embryos) innerhalb von 2 Stunden durchzuführen. Ein großer Altersunterschied zwischen den Embryonen kann die Zuverlässigkeit und Reproduzierbarkeit der Versuchsergebnisse beeinträchtigen.
- Richten Sie 10 Fischkreuzungspaare in 10 separaten Becken ein, wobei jedes Becken eine Trennwand zwischen dem männlichen und dem weiblichen Elternfisch enthält.
- Option 2: Herstellung heterozygoter transgener Neutrophilen/Makrophagen-Reporterfischembryonen.
- Richten Sie 10 Fischkreuzungspaare in 10 separaten Becken ein, wobei jedes Becken mit einer Trennwand zwischen männlichen und weiblichen Elternfischen ausgestattet ist: WT-Fisch gegen Tg(lyz:TagRFP) oder WT-Fisch gegen Tg(mpeg1:eGFP).
HINWEIS: Vermeiden Sie In-Kreuzungen zwischen heterozygoten transgenen Fischen, die die nachgeschalteten Fluoreszenzanzeigen beeinträchtigen können, da homozygote Reporterfische im Vergleich zu heterozygoten Fischen ein höheres Fluoreszenzsignal aufweisen. Transgene Reporterlinien und WT-Embryonen können bei der Bildgebung leicht unterschieden werden. Eine Mischung aus WT und heterozygoten Embryonen im selben Pool ist kein Problem. lyz:TagRFP berichtet über neutrophile Granulozyten und mpeg:eGFP über Makrophagen. Dieses Protokoll kann auch auf andere Reporter-Fischlinien angewendet werden. - Führen Sie die Schritte 2.1.2-2.1.4 aus.
- Richten Sie 10 Fischkreuzungspaare in 10 separaten Becken ein, wobei jedes Becken mit einer Trennwand zwischen männlichen und weiblichen Elternfischen ausgestattet ist: WT-Fisch gegen Tg(lyz:TagRFP) oder WT-Fisch gegen Tg(mpeg1:eGFP).
3. Einrichtung des Mikroinjektors
- Schalten Sie die Luftquelle ein, stellen Sie den Gegendruck auf 0,2-0,5 psi und den Einspritzdruck auf 25-30 psi ein. In der Regel wird der angegebene spezifische Druckbereich empfohlen.
Anmerkungen: Es ist wichtig, einen stabilen Gegendruck zu haben, um zu verhindern, dass Fischmedien in die Nadel zurückfließen. Bei der Kalibrierung des Einspritzvolumens in Schritt 3.8 sollte nur der Einspritzzeitpunkt geändert werden. Ändern Sie den Einspritzdruck in den folgenden Schritten nicht. Ein niedriger Injektionsdruck kann aufgrund der Oberflächen- und Grenzflächenspannung zu einem Versagen der Injektion führen, während ein hoher Injektionsdruck die Embryonen schädigen kann. - Reinigen Sie das Gerät und die Injektionsplattform mit RNase-Dekontaminationslösung.
HINWEIS: Die RNase, die mRNA abbaut, kann vom Bediener oder vom Gerät stammen. Es ist notwendig, die Reinigung vor dem Experiment durchzuführen und Handschuhe zu tragen. - (Optional) Wenn Sie mRNA und Morpholino gleichzeitig injizieren, mischen Sie die beiden in einem 0,2-ml-Röhrchen vor.
HINWEIS: Z-REX funktioniert gut mit Halo-TeV-Keap1-2xHA mRNA-Lösung mit einer Konzentration von 250-1500 ng/μL. Mehrere Morpholinos wurden auch in Zebrafischen verwendet, und die optimalen Konzentrationen wurden berichtet7. Bei Verwendung eines Morpholinos mit einer unveröffentlichten Sequenz sollte der Anwender zunächst die Toxizität und die Gen-Knockdown-Effizienz des Morpholinos bewerten, bevor er es in Z-REX verwendet. - Übertragen Sie 1-2 μl mRNA (und/oder ggf.Morpholino 7) in eine Injektionsnadel mit einer Micro-Loader-Pipettenspitze.
Anmerkungen: Wenn Sie Nadeln mit einem Flaming/Brown-Mikropipettenabzieher vorbereiten, gehen Sie wie folgt vor. Hitze: 520 Einheiten; Zugkraft: 60 Einheiten; Geschwindigkeit: 70 Einheiten; Verzögerung: 155 Einheiten; Druck: 550 Einheiten; Rampe: 530 Einheiten. - Setzen Sie die Nadel auf den Mikroinjektionsmanipulator.
Anmerkungen: Der Gegendruck von der Luftquelle sollte die mRNA(/morpholino)-Lösung zur Nadelspitze drücken. - Verwenden Sie eine scharfe Pinzette oder eine Rasierklinge, um die Nadelspitze zu brechen und eine geeignete Öffnung für die Injektion zu schaffen.
- Tauchen Sie die Nadelspitze in Mineralöl auf einer Tischmikrometerschraube.
- Wenden Sie zwei oder drei Injektionsimpulse an, um Luftblasen in der Spitze zu entfernen.
- Kalibrieren Sie die Tropfengröße auf 2 nL, indem Sie die Injektionszeit ändern.
Anmerkungen: Dies geschieht am besten durch Injektion in Mineralöl (das die Viskosität des Dottersacks nachahmt), das auf ein Hämozytometer aufgetragen wird. Verwenden Sie mit einem Mikroskop die Gitterlinien des Hämozytometers, um die Größe des während der Injektion gebildeten Tröpfchens abzuschätzen, und passen Sie die Injektionszeit entsprechend an. Obwohl manchmal Phenolrotfarbstoff verwendet wird, wurde die Notwendigkeit dafür bei dem hier beschriebenen mRNA-Injektionsverfahren nicht beobachtet.
4. Mikroinjektion
- Füllen Sie eine Injektionsplatte mit frischem 10%igem HBSS-Medium (Hank's Balanced Salt Solution) und richten Sie die Embryonen mit einer stumpfen Pinzette in den Rillen der Platte aus.
HINWEIS: Die Injektionsplatte wird mit 2 % Agarose in 10 % HBSS-Medium hergestellt. Die Nuten werden mit einer Kunststoffform geformt. - Tauchen Sie die Nadelspitze in 10%iges HBSS-Medium in die Injektionsplatte.
- Wenden Sie zwei oder drei Injektionsimpulse an, um Luftblasen in der Spitze zu entfernen.
- Bei jeder Injektion wird das Chorion und der Dottersack mit einer einzigen Bewegung durchdrungen und ein Injektionsimpuls angewendet. Diese injizierte Flüssigkeit ist direkt nach der Injektion als kleines Sphäroid im Dottersack zu sehen. Dieses kleine Sphäroid löst sich relativ schnell auf. Wiederholen Sie diesen Schritt für andere Embryonen, bis eine ausreichende Anzahl von injizierten Embryonen erhalten wurde.
HINWEIS: Die Überlebensrate von Embryonen (sowohl injiziert als auch nicht injiziert) variiert in der Regel zwischen 50% und 90%. Ziel ist es, die doppelte Anzahl von Embryonen zu injizieren, die für jede Kontroll-/Versuchsgruppe benötigt wird. Im Biotin-Pull-Down-Assay werden für jede Erkrankung 100-140 lebensfähige Embryonen benötigt. Im qRT-PCR-Assay werden für jede Erkrankung fünf lebensfähige Embryonen empfohlen. Die Stichprobengröße für die Bildgebung lebender Fische und den Whole-Mount-Immunfluoreszenz-Färbe-Assay ist benutzerdefiniert. Es wird empfohlen, mindestens 20 lebensfähige Embryonen pro Erkrankung zu haben, um eine gute statistische Aussagekraft in der Analyse zu erzielen. - Spülen Sie die injizierten Embryonen in eine neue 10-cm-Petrischale, die frisches 10%iges HBSS-Medium enthält.
HINWEIS: Die Embryonen können leicht mit einer Spritzflasche aus den Rillen gespült werden. - Fassen Sie die nicht injizierten Embryonen in einer anderen Platte zusammen.
HINWEIS: Die nicht injizierten Embryonen können je nach Bedarf als Qualitätskontrollen für die Gesundheit der Fische, die Ausgangsproteinexpression, die Hintergrundfluoreszenz usw. dienen. Wenn das Injektionsverfahren gut funktioniert hat und injizierte mRNA/Morpholino für die Embryonen nicht tödlich ist, sollten injizierte und nicht injizierte Embryonen eine ähnliche Lebensfähigkeit aufweisen.
5. Z-REX
- Verteilen Sie die injizierten Embryonen in 10-cm-Schalen, entsprechend dem Versuchsaufbau (d.h. Anzahl der Kontroll-/Versuchsgruppen).
- Ersetzen Sie das Medium in einem dunklen Raum mit Rotlichtbeleuchtung durch 30 ml 10%iges HBSS mit oder ohne 1 μM Ht-PreHNE (Alkin).
HINWEIS: Ht-PreHNE (Alkin) ist eine lichtlabile Verbindung. Die Embryonen sollten in den folgenden Schritten im Dunkeln aufbewahrt werden. - Inkubieren Sie die Embryonen bei 28,5 °C im Dunkeln.
- Ersetzen Sie bei 30,5 hpf in einem dunklen Raum das Medium durch frische 30 ml mit 10 % HBSS.
Anmerkungen: Entfernen Sie beim Ersetzen des Mediums so viel wie möglich vom alten Medium. Dies ist entscheidend, um die ungebundene/überschüssige Menge an Ht-PreHNE (Alkin) aus den Embryonen zu entfernen. - Inkubieren Sie die Embryonen bei 28,5 °C im Dunkeln für 30 Minuten.
- Wiederholen Sie die Schritte 5.4-5.5 noch zweimal.
- Schalten Sie die UV-Lampe (365 nm, 3 mW/cm2) für 5 Minuten ein, um die Lampe vorzuwärmen.
Anmerkungen: Der Schritt zum Vorwärmen der Lampe muss vor Schritt 5.8 durchgeführt werden. Die Lampenleistung ist in den ersten Minuten nach dem Einschalten geringer/instabil. Die Lampenleistung sollte regelmäßig mit einem UV-Messgerät gemessen werden. - Bei 32 hpf werden die Embryonen UV-Licht ausgesetzt.
- Option 1: Für nachgeschaltete Auslesungen, wie z. B. die Bildgebung lebender Fische, die Immunfluoreszenzfärbung im gesamten Mount, die In-Gel-Fluoreszenzanalyse (Klick-Kopplung mit Cy5-azid), RNA-seq oder qRT-PCR, werden die Embryonen 3 Minuten lang UV-Licht ausgesetzt und die Platten alle 30 s verwirbelt.
- Option 2: Für nachgeschaltete Auslesungen, wie z. B. den Biotin-Pull-Down-Assay, setzen Sie die Embryonen maximal 5 Minuten (und mindestens 3 Minuten) UV-Licht aus, schwenken Sie die Platten alle 30 s und kühlen Sie die Platten 1 Minute lang auf Eis.
Anmerkungen: Wenn Sie verschiedene Sonden verwenden, muss die Belichtungszeit optimiert werden, abhängig von der Hälftedes Photouncaging für eine bestimmte photogekäfige elektrophile Sonde und eingesetzte Lichtquelle. T1/2Photouncaging kann unter Verwendung bekannter Verfahren8 bestimmt werden. Für Ht-PreHNE (Alkin) ist t 1/2 < 1 min3; Somit ist die oben angegebene Zeit ausreichend.
6. Downstream-Analysen
- Option 1: Phänotypischer Assay. Live-Bildgebung von transgenen Neutrophilen/Makrophagen-Reportern
Fischschnüre, Tg(lyz:TagRFP) und Tg(mpeg1:eGFP) (Abbildung 2)- Betäuben Sie die Embryonen durch Inkubation bei 4 °C für 10 Minuten.
HINWEIS: Es wird empfohlen, mindestens 20 lebensfähige Embryonen pro Erkrankung zu haben. - Entfernen Sie die unbefruchteten/toten Embryonen von der Platte.
HINWEIS: Die unbefruchteten/toten Embryonen sind trüb/undurchsichtig und können visuell identifiziert werden. Wenn eine hohe Sterblichkeitsrate festgestellt wird, überprüfen Sie das Injektionsverfahren oder versuchen Sie, die Konzentration von mRNA oder Morpholino zu reduzieren. - Dechorionieren Sie die Embryonen mit einer scharfen Pinzette. Halten Sie die Chorion mit einer Pinzette, ohne die Fischlarven zu berühren, und reißen Sie die Chorion mit der anderen Pinzette ab. Der Embryo ist zerbrechlich. Berühren Sie den Chorion nur, wenn Sie eine Dechorionation durchführen.
HINWEIS: Es ist üblich, besonders bei Anfängern, einige Embryonen während der Dechorionation zu beschädigen. Haben Sie daher immer mehr Embryonen als das erforderliche Minimum. - Die Embryonen werden auf eine 2%ige Agaroseplatte (mit 10%igem HBSS-Medium präpariert) montiert und mit einem Stereomikroskop (Hellfeld und entsprechende Fluoreszenzkanäle) abgebildet (Abbildung 2A, C, G).
ANMERKUNG: Nach Z-REX [Kombination aus Halo-TeV-Keap1-2xHA mRNA-Injektion und Ht-PreHNE(alkin)-Behandlung] war die Depletion der neutrophilen Granulozyten (lyz:TagRFP) bei 36 hpf (4 h nach Z-REX) am signifikantesten, während die Reduktion von Makrophagen (mpeg1:eGFP) bei 34 hpf (2 h nach Z-REX) am signifikantesten war (Abbildung 2E,F). Andere Zeitpunkte können verwendet werden, wenn verschiedene Reporterlinien, mRNA/Morpholino oder Sonden verwendet werden. Die Belichtungszeit und/oder -verstärkung muss für die Visualisierung einzelner Zellen oder der gewünschten spezifischen (Ultra-)Strukturen optimiert werden. - Zählen Sie die Anzahl der Neutrophilen/Makrophagen jedes Fisches anhand von ImageJ (NIH) (Abbildung 2B, D-F, H). Verwenden Sie das Freihandauswahl-Werkzeug in ImageJ, um den ganzen Fisch einzukreisen, und verwenden Sie die Option Maxima suchen, um die fluoreszierenden Zellen zu zählen.
- Betäuben Sie die Embryonen durch Inkubation bei 4 °C für 10 Minuten.
- Option 2: Bewertung der Zielkennzeichnung. Biotin-Azid-Klick-Kopplung und Biotin-Pull-Down-Assay (Abbildung 3)
- Betäuben Sie die Embryonen durch Inkubation bei 4 °C. Dies dauert in der Regel 10 Minuten.
HINWEIS: Um genügend Fischlysat zu erhalten, sind für jede Erkrankung 100-140 lebensfähige Embryonen erforderlich. - Entfernen Sie die unbefruchteten/toten Embryonen von der Platte.
- Führen Sie Dechorionation und Deyolking durch. Führen Sie die beiden Manipulationen durch, indem Sie das Chorion mit einer scharfen Pinzette halten, mit der anderen Pinzette in den Dottersack eindringen und dann den Dottersack trennen, während Sie den Chorion entfernen, damit die Dotterproteine herauskommen können.
HINWEIS: Es ist wichtig, die Eigelbproteine in diesem Schritt zu entfernen. Die reichlich vorhandenen Dotterproteine in der Probe stören die spätere Analyse. - Übertragen Sie die entyolkten Embryonen in ein 1,5-ml-Röhrchen.
Anmerkungen: Schwenken Sie die Platte, um die entyolkten Embryonen zu zentrieren, was die Entnahme erleichtert. Der leichtere Chorion-Schmutz löst sich während dieses Prozesses ab. - Nachdem sich die Embryonen auf dem Röhrchenboden niedergelassen haben, entfernen Sie den Überstand und fügen Sie 1 ml gekühlte HEPES-gepufferte Kochsalzlösung (pH 7,6) hinzu.
- Wiederholen Sie Schritt 6.2.5 noch zweimal.
- (Optional) Wenn Sie nicht sofort mit dem nächsten Schritt fortfahren möchten, entfernen Sie den Puffer, frieren Sie die Proben in flüssigem Stickstoff ein und lagern Sie sie bei -80 °C.
HINWEIS: Entyolkte Fischpellets können in flüssigem Stickstoff schockgefroren und bei -80 °C gelagert werden. Schockgefrorene Proben können bis zu 2 Wochen bei -80 °C gelagert werden. - Die Fischpellets in Lysepuffer resuspendieren.
HINWEIS: Der Lysepuffer (pH 7,6) besteht aus 50 mM HEPES, 100 mM NaCl, 1 % Triton X-100, 0,3 mM TCEP, 2x Roche cOmplete Mini EDTA-freien Proteasehemmern und 0,1 mg/ml Trypsin-Inhibitor aus Sojabohnen. Ein Embryo liefert etwa 2 μg Lysat. Verwenden Sie 100 μl Lysepuffer pro 120 Embryonen. Zwei Roche cOmplete Mini EDTA-freie Proteasehemmer und Trypsininhibitoren aus Sojabohnen sollten kurz vor der Anwendung in den Lysepuffer gegeben werden. - Geben Sie 20% v/v Zirkoniaperlen in das Röhrchen.
- 20 s vortexen, in flüssigem Stickstoff einfrieren und im 37 °C warmen Wasserbad auftauen.
- Wiederholen Sie Schritt 6.2.10 noch zwei weitere Male.
- Zentrifugieren Sie die Lösung bei 21.000 x g bei 4 °C für 10 Minuten.
- Übertragen Sie den Überstand in ein neues, vorgekühltes 1,5-ml-Röhrchen.
- Messen Sie die Proteinkonzentration mit dem Bradford-Assay.
- Verdünnen Sie das Lysat auf 1 mg/ml.
- Für jede Bedingung werden 170 μg Lysat in ein 2-ml-Röhrchen überführt.
- Mischen Sie das Lysat aus Schritt 6.2.16 mit 0,2 mg/ml TeV-Protease (S219V) und inkubieren Sie die Lösung bei 37 °C für 30 min.
HINWEIS: Mischen Sie bei nicht mit TeV-Protease behandelten Gruppen einfach das Lysat mit einem gleichen Voloum an Lysepuffer wie die TeV-Proteaselösung, die in anderen Gruppen verwendet wird. - Bereiten Sie eine 10-fache Mastermischung für die Biotin-Azid-Click-Reaktion vor: 10 % w/v SDS, 10 mM CuSO4, 1 mM Cu(TBTA), 1 mM Biotinazid und 20 mM TCEP.
Anmerkungen: Fügen Sie TCEP kurz vor Schritt 6.2.19 hinzu. - 8,5 μl t-BuOH und 17 μl 10x click-Reaktionsmastermix werden dem (TeV-Protease-verdauten) Lysat aus Schritt 6.2.17 zugegeben. Vortex, zentrifugieren und inkubieren Sie die Lösung bei 37 °C für 15 Minuten.
- Fügen Sie der Lösung weitere 1 mM TCEP hinzu, wirbeln, zentrifugieren Sie sie und inkubieren Sie die Lösung bei 37 °C für weitere 15 Minuten. Die Inkubationszeit für die Schritte 6.2.19-6.2.20 beträgt insgesamt 30 min.
HINWEIS: Diese Ergänzung von TCEP, einem reduzierenden Reagenz zur Erzeugung von Cu(I), verbessert die Effizienz der Click-Reaktion. - Geben Sie 600 μl -20 °C Ethanol in jedes Röhrchen, wirbeln Sie die Lösung und inkubieren Sie sie über Nacht bei -80 °C.
HINWEIS: Die Proben können 1 Woche lang bei -80 °C gelagert werden. Wenn nicht, fahren Sie sofort mit dem nächsten Schritt fort. - Zentrifugieren Sie die Lösung bei 21.000 x g bei 4 °C für 1 h.
Anmerkungen: Nach dem Zentrifugieren sollte sich am Boden des Röhrchens ein Pellet bilden, das die gewünschte Fraktion ist. - Entfernen Sie den Überstand, fügen Sie 1 ml -20 °C Ethanol hinzu, wirbeln Sie vor und zentrifugieren Sie die Lösung bei 21.000 x g bei 4 °C für 10 Minuten.
- Wiederholen Sie Schritt 6.2.23.
- Entfernen Sie den Überstand, fügen Sie 1 ml -20 °C Aceton hinzu, wirbeln Sie vor und zentrifugieren Sie die Lösung bei 21.000 x g bei 4 °C für 10 Minuten.
- Entfernen Sie den Überstand. Lassen Sie überschüssiges Restaceton verdunsten, wenn auch nicht vollständig bis zur Trockenheit.
- Resuspendieren Sie das Pellet in 100 μl Resuspensionspuffer (8 % w/v Lithiumdodecylsulfat [LDS], 1 mM EDTA in 50 mM HEPES-Kochsalzlösung, pH 7,6), wirbeln Sie es 15 s lang und beschallen Sie, bis sich das Pellet aufgelöst hat.
- Zentrifugieren Sie die Lösung bei 21.000 x g bei Raumtemperatur (RT) für 5 Minuten.
- Übertragen Sie den Überstand in ein neues 2-ml-Röhrchen und fügen Sie 1,5 ml 50 mM HEPES-Kochsalzlösung mit einem pH-Wert von 7,6 hinzu.
HINWEIS: Die Endkonzentration von LDS in diesem Schritt beträgt 0,5 %. Höhere LDS-Konzentrationen können die Pull-Down-Effizienz untergraben. Obwohl eine Erhöhung der LDS-Konzentration zur Verringerung der unspezifischen Bindung beitragen kann, kann sie auch die Effizienz des Pulldowns verringern. Dementsprechend wird empfohlen, die LDS-Konzentration in diesem Schritt nicht zu verändern. - Entnahme der "Eingangsprobe" (Abbildung 3): 30 μl 1 mg/ml Lysat in ein neues 1,5-ml-Röhrchen überführen und 10 μl 4x Laemmli-Probenpuffer mit 6 % β-Mercaptoethanol (BME) hinzufügen. Schockfrosten Sie die Lösung ein und lagern Sie sie bei -80 °C.
- Übertragen Sie 100 μl Streptavidin-Hochleistungsharz mit Bettvolumen in ein neues 2-ml-Röhrchen. Fügen Sie 1 ml 0,5 % LDS in 50 mM HEPES-Kochsalzlösung (pH 7,6) hinzu, zentrifugieren Sie 2 Minuten lang bei 1.500 x g RT und entfernen Sie den Überstand. Wiederholen Sie die Wäsche mit weiteren 1 ml 0,5 % LDS in 50 mM HEPES-Kochsalzlösung (pH 7,6).
- Übertragen Sie die Lösung aus Schritt 6.2.29 in das Röhrchen mit vorgewaschenem Streptavidin-Hochleistungsharz aus Schritt 6.2.31 und inkubieren Sie die Lösung 4-6 h lang auf einem End-over-End-Mischer bei RT.
- Zentrifugieren Sie das Gemisch bei 1.500 x g bei RT für 2 min, nehmen Sie 30 μl des Überstands und mischen Sie es mit 10 μl 4x Laemmli-Probenpuffer mit 6 % BME für die "Durchfluss"-Probe. Entfernen Sie dann den restlichen Überstand.
HINWEIS: "Flowthrough"-Proben können mittels Western Blot analysiert werden, um die Streptavidin-Pulldown-Effizienz zu überprüfen. Es ist wichtig, so viel Überstand wie möglich zu entfernen, um die ungebundenen Proteine auszuwaschen. Entfernen Sie zuerst den größten Teil des Überstands mit einer P-1000-Pipette und entfernen Sie dann den verbleibenden Überstand mit einer P-20-Pipette mit einer Gelladespitze. - Fügen Sie dem Harz 1 ml 0,5 % LDS in 50 mM HEPES-Kochsalzlösung (pH 7,6) hinzu und inkubieren Sie die Mischung 30 Minuten lang bei RT mit End-over-End-Rotation.
- Zentrifugieren Sie die Mischung bei 1.500 x g RT für 2 Minuten und entfernen Sie den Überstand.
HINWEIS: In der Regel reichen 0,5 % LDS aus, um die meisten unspezifischen Bindungsproteine zu entfernen. Wenn in der späteren Analyse noch unspezifische Bindungssignale zu sehen sind, kann die LDS-Konzentration im Waschpuffer erhöht werden. - Wiederholen Sie die Schritte 6.2.34-6.2.35 noch zweimal.
- Fügen Sie dem Harz 40 μl 2x Laemmli-Probenpuffer mit 6% BME hinzu.
- Elute die gebundenen Proteine, indem die Mischung bei 98 °C für 5 min inkubiert wird.
- Zentrifugieren Sie das Gemisch bei 21.000 x g RT für 5 Minuten und überführen Sie den Überstand in ein neues 1,5-ml-Röhrchen. Dies ist das "Elute"-Beispiel.
HINWEIS: Das direkte Einbringen von harzhaltiger Lösung aus Schritt 6.2.38 in das Gel kann die SDS-PAGE-Analyse beeinträchtigen. - Laden Sie 20 μl in jede Vertiefung mit 10-bahnigem Gel aus 10 % Polyacrylamid und führen Sie eine Gelelektrophorese durch.
Anmerkungen: Lassen Sie das Gel mit einer niedrigeren Spannung (120 V) laufen, bis die Farbstofffront das auflösende Gel erreicht, und ändern Sie die Spannung auf 170 V. Stoppen Sie das Programm, nachdem die Farbstofffront herausgekommen ist. - Führen Sie ein Western-Blot mit Anti-HA-, Anti-Halo- oder anderen Antikörpern durch, die Haushaltsproteine erkennen (Abbildung 3).
- Betäuben Sie die Embryonen durch Inkubation bei 4 °C. Dies dauert in der Regel 10 Minuten.
- Option 3: Transkriptomanalyse. RNA-seq und qRT-PCR (Abbildung 4)
HINWEIS: Es wird dringend empfohlen, für diesen Test Embryonen zu verwenden, die innerhalb von 15 Minuten voneinander entfernt liegen. Der Altersunterschied der Embryonen hat einen erheblichen Einfluss auf die Testergebnisse.- Betäuben Sie die Embryonen, indem Sie sie bei 4 °C für 10 min, 2 h nach Z-REX inkubieren.
- Führen Sie die Dechorionation mit einer scharfen Pinzette durch (Schritt 6.1.3).
- (Optional) Führen Sie eine Segmentierung mit einer Pinzette durch (z. B. den Kopf vom Schwanz trennen), wenn verschiedene Segmente separat analysiert werden sollen.
- Wenn Sie RNA aus einem ganzen Embryo extrahieren, übertragen Sie drei bis fünf Embryonen in ein 1,5-ml-Röhrchen. Wenn Sie RNA aus dem Kopf oder Schwanz extrahieren, übertragen Sie 10-12 präzierte Segmente in ein 1,5-ml-Röhrchen.
HINWEIS: Es wird empfohlen, das Experiment mit drei bis fünf biologischen Wiederholungen durchzuführen. - Geben Sie 1 ml TRIzol-Reagenz und Glasperlen in das Röhrchen.
HINWEIS: Es wurde festgestellt, dass Glasperlen für die RNA-Extraktion besser geeignet sind als Zirkonoxidperlen. - Wirbeln Sie die Mischung 30 s lang.
Anmerkungen: Wenn Sie nicht sofort mit dem nächsten Schritt fortfahren, kann die Lösung 1−3 Wochen lang bei -80 °C gelagert werden. - Extrahieren Sie die RNA gemäß den Anweisungen des Herstellers.
- Beurteilung der RNA-Qualität und -Konzentration mittels Mikrovolumen-Spektralphotometer und Agarose-Gelelektrophorese.
HINWEIS: RNA von guter Qualität sollte ein A-260/A-280-Verhältnis von etwa oder mehr von 2,0 aufweisen. - Entweder wird die RNA zur Sequenzierung eingereicht oder 1 μg RNA mit DNase I in Amplifikationsqualität behandelt und mit hochgestellter Reverse-Transkriptase III und Oligo-(dT)20 reverse transkribiert. Führen Sie diesen Schritt gemäß den Anweisungen des Herstellers aus.
- Führen Sie eine qRT-PCR durch und analysieren Sie die Daten mit der ΔΔCT-Methode9 (Abbildung 4B-D).
- Option 4: POI-Ausdruck und Kolokalisationsanalyse. Whole-Mount-Immunfluoreszenz-Färbeassay (Abbildung 5)
HINWEIS: Formaldehyd-fixierte Embryonen sind zerbrechlich. Vermeiden Sie starkes Schütteln und gehen Sie vorsichtig damit um.- Dechorionieren Sie die Embryonen in den Schritten 6.1.1-6.1.3.
- Übertragen Sie die Embryonen in ein 1,5-ml-Röhrchen.
HINWEIS: Jedes Röhrchen sollte die gleiche Anzahl von Embryonen und nicht mehr als 40 Embryonen enthalten. - Nachdem sich die Embryonen am Boden des Röhrchens niedergelassen haben, entfernen Sie den Überstand und fügen Sie 1 ml phosphatgepufferte Kochsalzlösung (PBS) (pH 7,6) hinzu.
- Wiederholen Sie Schritt 6.4.3 noch einmal.
- Entfernen Sie den Überstand, fügen Sie 1 ml 4%iges Formaldehyd in PBS (pH 7,6) hinzu und inkubieren Sie das Röhrchen über Nacht bei 4 °C unter leichtem Schaukeln.
HINWEIS: Die Proben in Formaldehydlösung können 1 Woche lang bei 4 °C gelagert werden. - Entfernen Sie den Überstand, fügen Sie 1 ml -20 °C Methanol hinzu und inkubieren Sie das Röhrchen auf der Seite bei -20 °C für mindestens 18 h.
HINWEIS: Die Proben können 1 Monat oder länger bei -20 °C gelagert werden. - Entfernen Sie den Überstand und fügen Sie 1 ml PDT-Puffer hinzu (0,3 % v/v Triton X-100, 0,1 % v/v Tween-20 und 1 % v/v Dimethylsulfoxid [DMSO] in PBS-Puffer).
- Wiederholen Sie Schritt 6.4.7 und inkubieren Sie das Röhrchen 30 Minuten lang bei RT unter leichtem Schaukeln.
- Entfernen Sie den Überstand, fügen Sie 1 ml Blockierungspuffer hinzu (10 % v/v hitzeinaktiviertes fötales Kälberserum [FBS], 2 % w/v Rinderserumalbumin [BSA] und 0,1 % v/v Tween-20 in PBS-Puffer) und inkubieren Sie das Röhrchen bei RT für 1 h unter leichtem Schaukeln.
- Entfernen Sie den Überstand und fügen Sie 200 μl primäre Antikörperlösung (verdünnt in Blockierungspuffer) hinzu.
- Entfernen Sie den Überstand, fügen Sie 500 μl primäre Antikörperlösung (verdünnt in Blockierungspuffer) hinzu und inkubieren Sie das Röhrchen über Nacht bei 4 °C unter leichtem Schaukeln.
HINWEIS: Wenn Sie einen neuen Primärantikörper verwenden, lohnt es sich, einige Proben ohne primäre Antikörperfärbung als Negativkontrolle einzuschließen. Im Idealfall sind jedoch Morpholino-Knockdown- oder Gen-Knockout-Embryonen oder Embryonen, bei denen die Expression des Zielproteins stimuliert wurde, zuverlässigere Mittel zur Validierung des Antikörpers. - Entfernen Sie den Überstand, fügen Sie 1 ml PDT-Puffer hinzu und inkubieren Sie das Röhrchen 30 Minuten lang bei RT unter leichtem Schaukeln.
- Wiederholen Sie Schritt 6.4.12.
- Entfernen Sie den Überstand, fügen Sie 1 ml Blockierungspuffer hinzu und inkubieren Sie das Röhrchen 1 h lang bei RT unter leichtem Schaukeln.
HINWEIS: Die Proben sollten nach diesem Schritt vor Licht geschützt werden, um eine Photobleichung des auf dem Sekundärantikörper konjugierten Fluorophors zu verhindern. - Entfernen Sie den Überstand und fügen Sie 200 μl sekundäre Antikörperlösung (verdünnt mit Blockierungspuffer) hinzu.
- Entfernen Sie den Überstand, fügen Sie 500 μl sekundäre Antikörperlösung (verdünnt in Blockierungspuffer) hinzu und inkubieren Sie das Röhrchen bei RT für 1,5 h unter leichtem Schaukeln.
- Entfernen Sie den Überstand, fügen Sie 1 ml PDT-Puffer hinzu und inkubieren Sie das Röhrchen 30 Minuten lang bei RT unter leichtem Schaukeln.
- Wiederholen Sie Schritt 6.4.17.
- Montieren Sie die Embryonen auf eine 2%ige Agaroseplatte (hergestellt mit PBS, pH 7,6) und bilden Sie die Embryonen mit einem Stereomikroskop (Hellfeld und entsprechende Fluoreszenzkanäle) ab (Abbildung 5A,B,D,F).
HINWEIS: Wenn Sie das Fluoreszenz-Stereomikroskop Leica M165 FC verwenden, verwenden Sie eine 25-fache Vergrößerung, um Bilder mit guter Auflösung zu erhalten. - Quantifizierung/Analyse der Intensität des Fluoreszenzsignals mit ImageJ (NIH). Verwenden Sie das Freihandauswahl-Werkzeug in ImageJ, um das Signal im Interessenbereich zu quantifizieren.
Ergebnisse
Live-Bildgebung von Z-REX-behandelten transgenen Neutrophilen/Makrophagen-Reporterfischen, Tg(lyz:TagRFP) und Tg(mpeg1:EGFP). Induktion der Apoptose von Neutrophilen/Makrophagen durch Keap1 HNEylierung. (Siehe auch Abbildung 2). Die Wirkung der elektrophilen Markierung von Keap1 auf die Konzentrationen von Neutrophilen und Makrophagen wurde durch die Injektion heterozygoter transgener Embryonen, die von Tg(lyz:TagRFP) oder Tg(mpeg1:EGFP) abstammen, mit mRNA, die für Halo-Keap1 kodiert, und die anschließende Behandlung mit Ht-PreHNE(alkin) untersucht. Im Anschluss an die Verfahren für Schritt 6.1-Downstream-Assay wurde Option 1-HNE (Alkin) freigesetzt und Keap1 markiert. Die Konzentrationen von Neutrophilen und Makrophagen wurden durch Live-Bildgebung der Reporterlinien Tg(lyz:TagRFP) bzw. Tg(mpeg1:eGFP) bestimmt. Der Spiegel beider Zelltypen sank um 30%-40% nach der Z-REX-Behandlung, bei der HNE an Keap1 abgegeben wurde. Im Gegensatz dazu wurde in den technischen Kontrollgruppen von Z-REX kein Verlust von Neutrophilen oder Makrophagen beobachtet [ohne Licht und Ht-PreHNE (Alkin), Licht allein oder Ht-PreHNE (Alkin) allein] (Abbildung 1D und Abbildung 2A-D).
Die Induktion der Apoptose von Neutrophilen/Makrophagen deutete auf eine erfolgreiche HNE-Abgabe an Keap1 durch Z-REX hin. Details zur Signalwegsanalyse und zum Apoptosemechanismus wurden veröffentlicht5. Um Off-Target-Effekte von HNE (Alkin) zu berücksichtigen, wurden mehrere Kontrollen verwendet. (1) Unter den gleichen experimentellen Bedingungen wurde den Embryonen anstelle von Halo-TeV-Keap1 mRNA Halo-P2A-Keap1 mRNA injiziert. Der P2A-Linker ermöglichte es, die Proteine Halo und Keap1 unabhängig voneinander zu exprimieren. In diesem Szenario konnte HNE(alkin), das von Halo freigesetzt wurde, Keap1 nicht markieren, da es sich nicht mehr proximal zu Halo befand (Abbildung 1D); Der Apoptose-Signalweg wurde also nicht ausgelöst. In dieser Gruppe wurden keine Veränderungen der Makrophagen- oder Neutrophilenkonzentration beobachtet (Abbildung 2A,B). (2) Die gleichen experimentellen Bedingungen wurden mit mRNA durchgeführt, die für Halo-TeV-Keap1(C151S,C273W,C288E) kodiert, eine Mutante von Keap1, die nicht auf HNE(Alkin) reagiert (Abbildung 1D). Es wurden keine Veränderungen der Makrophagen- oder Neutrophilenkonzentration beobachtet (Abbildung 2G,H).
Biotin-Azid-Klick-Kopplung und Biotin-Pull-Down-Assay. Bewertung der Zielkennzeichnung. (Siehe auch Abbildung 3). Die Target-Marking-Bewertung wurde mit WT-Embryonen durchgeführt, die mit mRNA injiziert wurden, die entweder für Halo-TeV-Keap1-2xHA (Halo-POI-Fusionskonstrukt) oder Halo-2xHA-P2A-Keap1-2xHA (P2A-Split-Konstrukt, in dem Halo und Keap1 nicht fusioniert sind; Abbildung 1D). Das markierte Keap1-Protein wurde nur in der Gruppe, die das Fusionsprotein exprimierte, heruntergezogen und mit Z-REX (zweite Spur im oberen Anti-HA-Blot) behandelt, nicht aber in anderen Kontrollgruppen (keine mRNA-Injektion, Fusionskonstrukt ohne Z-REX oder P2A-Split-Konstrukt). Die Ergebnisse deuten darauf hin, dass das HNE (Alkin) erfolgreich an Keap1 geliefert wurde, und das modifizierte Keap1 anschließend durch Click-Reaktion mit Biotin konjugiert wurde und das Biotin-markierte Keap1 durch Streptavidinharz nach unten gezogen wurde.
Transkriptionsanalyse. RNA-seq und qRT-PCR. (Siehe auch Abbildung 4). Die transkriptionelle Veränderung nach Z-REX-Behandlung wurde mittels RNA-seq und qRT-PCR untersucht. In RNA-seq wurden mehrere immunrelevante Gene nach Z-REX herunterreguliert. Im Gegensatz dazu waren viele Gene, die mit der antioxidativen Antwort (AR) in Verbindung stehen, nach Z-REX hochreguliert, was aus der Induktion des Keap1-Nrf2-AR-Signalwegs bei HNEylierung auf Keap1resultierte 10 (Abbildung 4A). In der qRT-PCR-Analyse wurden ähnliche Ergebnisse bei der Analyse von drei immunrelevanten Genen (lyz, mpeg1.1 und coro1a) gefunden (Abbildung 4B). Die Hoch- und Herunterregulierung der jeweiligen Gene zeigte die erfolgreiche Induktion von Signalwegen, die durch Keap1 HNEylierung vermittelt werden.
Whole-Mount (Co-)Immunfluoreszenz-Färbe-Assay und Kolokalisationsanalyse. (Siehe auch Abbildung 5). Die exogene Expression von Halo-TeV-Keap1-2xHA und Halo-2xHA-P2A-Keap1-2xHA wurde mittels Whole-Mount Immunfluoreszenz (IF) Färbung bestimmt (Abbildung 5A,B). Das P2A-Split-Konstrukt hatte doppelt so viele HA-Tags wie das TeV-Fusions-Konstrukt, was einem doppelt höheren Anti-HA-Signal in der P2A-Split-Construct-mRNA-injizierten Gruppe entspricht als die andere, was darauf hindeutet, dass das Expressionsniveau der beiden Konstrukte ähnlich war (Abbildung 5C). Die Expressionsniveaus von Halo-TeV-Keap1 (wt) und Halo-TeV-Keap1 (C151S,C273W,C288E) waren auch bei der Sondierung mit Anti-Halo ähnlich (Abbildung 5D,E). Die Kolokalisation von neutrophilen Granulozyten und aktiver Caspase 3 in Z-REX-behandelter Tg(lyz:TagRFP) wurde durch Co-Immunfärbung mit Anti-RFP und Anti-Aktiv-Caspase 3 beobachtet (Abbildung 5F). Active Caspase 3 ist ein Indikator für Apoptoseereignisse.
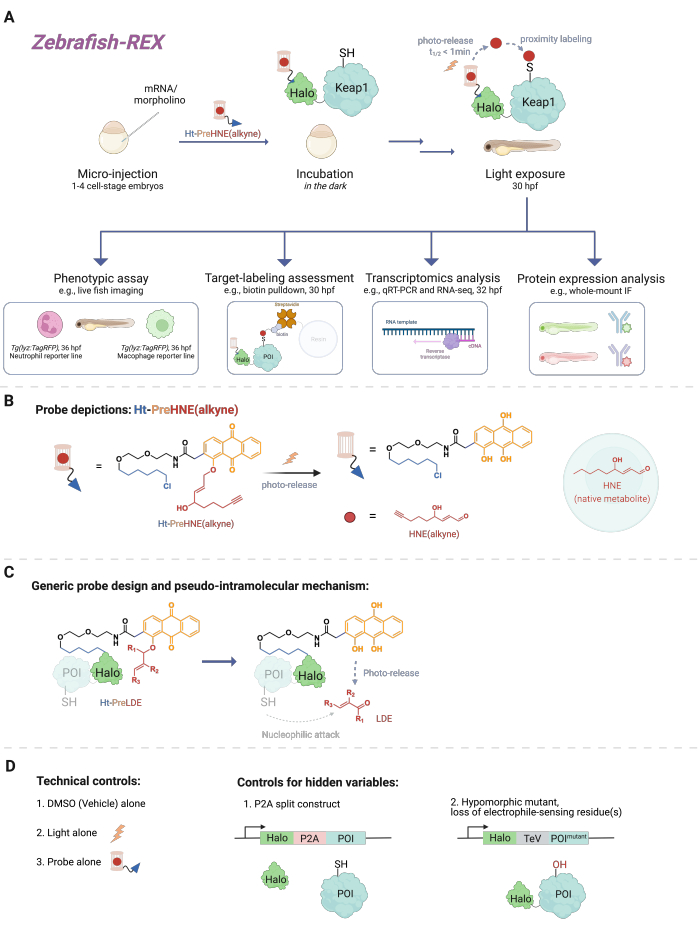
Abbildung 1: Z-REX-Arbeitsablauf. (A,B) Ein Zebrafischembryo im 1-4-Zellstadium wird mit (morpholino und) mRNA injiziert, die für Halo-POI kodiert (z. B. Halo-Keap1). Injizierte Embryonen werden dann mit einer Sonde behandelt, die aus einem HaloTag-Liganden und einem Photokäfig-Elektrophil besteht, an das eine funktionelle Alkingruppe angehängt ist, wie z. B. Ht-PreHNE (Alkin) in B. Nach dem Entfernen der überschüssigen Sondenmenge wird der Embryo Licht ausgesetzt, um das interessierende Elektrophil freizusetzen [z. B. HNE oder sein Analogon HNE (Alkin)]. Die nachgelagerte Analyse wird zu einem gegebenen/benutzerdefinierten Zeitpunkt durchgeführt. (C) Design und Mechanismus der Ht-PreLDE-Sonde, die auf verschiedene Lipid-abgeleitete Elektrophile (LDE) anwendbar ist. (D) Negative/technische Kontrollgruppen für Z-REX. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
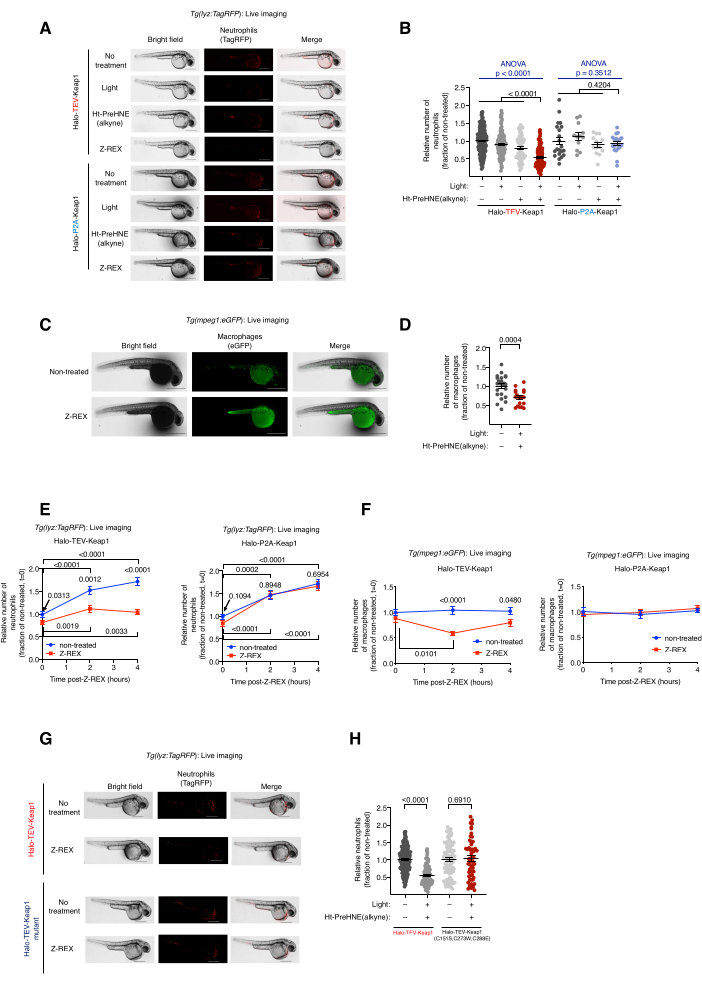
Abbildung 2: Live-Bildgebung von transgenen Neutrophilen/Makrophagen-Reporterfischen, die Z-REX ausgesetzt waren. Z-REX-vermittelte Keap1 HNEylierung induziert Neutrophilen/Makrophagen-Apoptose. (A) Repräsentative Bilder von Tg(lyz:TagRFP)- Fischen, die entweder Halo-TeV-Keap1 (Fusionskonstrukt) oder Halo-P2A-Keap1 (Split-Konstrukt) exprimieren und negativen Kontrollbedingungen ausgesetzt wurden [keine Behandlung, Licht allein oder Ht-PreHNE (Alkin) allein oder Z-REX]. Alter des Embryos: 36 hpf. (B) Quantifizierung der Neutrophilenkonzentration in A. (C) Repräsentative Bilder von Tg(mpeg1:eGFP)- Fischen, die Halo-TeV-Keap1 exprimieren, mit oder ohne Z-REX-Behandlung. Alter der Embryonen: 34 hpf. (D) Quantifizierung der Makrophagenkonzentration in C. (E,F) Zeitverlaufsmessung der (E) Neutrophilen- und (F) Makrophagenspiegel nach Z-REX-Behandlung. (G) Ähnliches Experiment wie in A an Fischen, die entweder Halo-TeV-Keap1 (WT) oder Halo-TeV-Keap1 (C151S, C273W, C288E) exprimieren, eine Mutante, die keine HNE-Sensorfähigkeit besitzt. (H) Quantifizierung der neutrophilen Granulozyten in G. Maßstabsbalken: 500 μm. Alle Graphen werden mit Mittelwert ± REM dargestellt. Die p-Werte wurden mit einer einfaktoriellen ANOVA (blau) und einem zweiseitigen Student-t-Test (schwarz) berechnet. Diese Abbildung wurde von Poganik et al.7 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 3: Biotin-Pull-Down-Assay. WT-Embryonen, die Halo-TeV-Keap1-2XHA oder Halo-2XHA-P2A-Keap1-2XHA exprimierten, wurden mit Z-REX oder entsprechenden Negativkontrollbedingungen behandelt (in diesem Fall keine Sondenbehandlung). Nach der Entnahme wurden die Embryonen lysiert und mit TeV-Protease behandelt, bevor der Biotin-Pull-Down-Assay durchgeführt wurde. Die Ergebnisse wurden mittels Western Blot analysiert. Diese Abbildung wurde von Huang et al. Z-REX: Lenkung reaktiver Elektrophile zu spezifischen Proteinen, die entweder gewebespezifisch oder ubiquitär exprimiert werden, und Aufzeichnung der daraus resultierenden funktionellen elektrophil-induzierten Redoxantworten in Fischlarven. Diese Abbildung wurde von Huang et al.11. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
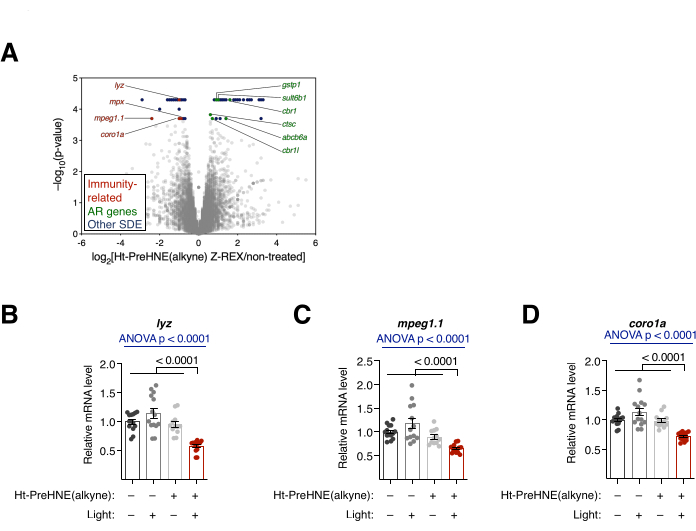
Abbildung 4: Transkriptionsanalyse. (A) RNA-seq-Ergebnisse von Z-REX-behandelten und unbehandelten Embryonen. Statistisch signifikante differentiell exprimierte (SDE) Gene werden hervorgehoben. Immunitätsbezogene SDE-Gene sind rot eingefärbt. Gene, die mit der antioxidativen Reaktion (AR) in Verbindung stehen, sind grün eingefärbt. Andere SDE-Gene sind blau gefärbt. Alle p-Werte wurden mit CuffDiff berechnet. (B-D) Drei immunitätsrelevante SDE-Gene aus A: (B) lyz, (C) mpeg1.1 und (D) coro1a wurden mittels qRT-PCR weiter analysiert, und nur die Z-REX-behandelten Embryonen zeigten die Suppression dieser Transkripte. Alle Graphen werden mit Mittelwert ± REM dargestellt. Die p-Werte wurden mit einer einfaktoriellen ANOVA (blau) und einem zweiseitigen Student-t-Test (schwarz) berechnet. Diese Abbildung wurde von Poganik et al.7 modifiziert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
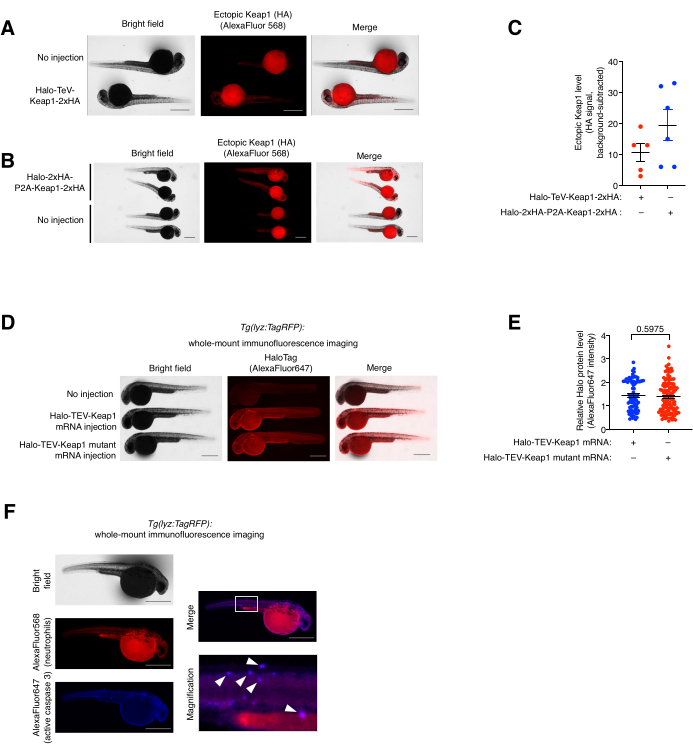
Abbildung 5: Whole-Mount-Immunfluoreszenz-Färbeassay. (A,B) Repräsentative Bilder von Embryonen, die entweder (A) Halo-TeV-Keap1-2xHA oder (B) Halo-2xHA-P2A-Keap1-2xHA exprimieren, immungefärbt mit Anti-HA und sekundärem Antikörper, der mit AlexaFluor568 konjugiert ist. mRNA-injizierte Fische wurden mit altersgleichen, nicht injizierten Fischen verglichen. (C) Quantifizierung des Anti-HA-Signals in (A,B). (D) Repräsentative Bilder von Embryonen, die Halo-TeV-Keap1 (WT) oder Halo-TeV-Keap1 (C151S, C273W, C288E) exprimieren, immungefärbt mit Anti-Halo und sekundären Antikörpern, die mit AlexaFluor647 konjugiert sind. mRNA-injizierte Fische wurden mit altersgleichen, nicht injizierten Fischen verglichen. (E) Quantifizierung des Anti-Halo-Signals in D. p-Werte wurden mit dem zweiseitigen Student-t-Test berechnet. (F) Tg(lyz:TagRFP)-Embryonen, die Z-REX ausgesetzt waren, wurden mit Anti-RFP und antiaktiver Caspase 3 sowie entsprechenden Fluorophor-konjugierten Sekundärantikörpern co-immungefärbt. Der weiße Kasten markiert den vergrößerten Bereich. Weiße Pfeile zeigen Kolokalisationen von neutrophilen Granulozyten und aktiver Caspase 3 an. Maßstabsbalken: 500 μm. Alle Graphen werden mit Mittelwert ± REM dargestellt. Diese Abbildung wurde von Poganik et al.7 modifiziert. und Huang et al.11. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Tabelle 1: Liste der in dieser Studie verwendeten Puffer. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Z-REX, das in diesem Protokoll beschrieben wird, demonstriert eine robuste Strategie für die Untersuchung von Elektrophil-Target-Paaren und die Entfaltung von Signalwegen in lebenden Fischen. Die näherungsgerichtete Abgabe ermöglicht die Dosierung und räumliche Kontrolle der elektrophilen Substanzbehandlung. Im Gegensatz zu herkömmlichen Bolusdosierungsmethoden, bei denen die supraphysiologischen Konzentrationen des eingesetzten Elektrophils oft zu Off-Target-Problemen führen, macht die relativ geringe Menge an Elektrophil, die an das System abgegeben wird, Z-REX weitgehend nicht-invasiv. Wir haben 0,1-6 μM Ht-PreHNE (Alkin) in Zebrafischembryonen verwendet, und die Ergebnisse zeigten, dass die Behandlung der Embryonalentwicklung nicht abträglich ist11.
Das Z-REX-Verfahren ist im Allgemeinen länger als das T-REX-Verfahren, eine Technik zum Screenen/Untersuchen elektrophiler Sensorproteine in kultivierten Zellen. Angenommen, der Zweck des Experiments besteht darin, nach Wechselwirkungen zwischen Elektrophilen und Zielmolekülen zu suchen. Wir empfehlen, zunächst ein umfangreiches Screening mit T-REX in kultivierten Zellen durchzuführen und Z-REX für die In-vivo-Validierung und die phänotypische/Signalweganalyse zu verwenden. Im Vergleich zur Zellkultur sind die Voraussetzungen für die Durchführung von Z-REX neben den für T-REX erforderlichen biochemischen Versuchsfähigkeiten grundlegende Fischhaltungstechniken. Ein typischer Zeitrahmen für Z-REX (von der Fischkreuzung bis zur lichtinduzierbaren elektrophilen Abgabe) beträgt 2-3 Tage, was nicht mehr als 1 Tag länger ist als die typische Zeit für ein T-REX-Experiment an transfizierten lebenden Zellen. Die Live-Bildgebung für die phänotypische Analyse kann 2-10 h nach der Beleuchtung des Lichts durchgeführt werden. Die Klick-Kopplung mit Biotin-Azid für den Pull-Down-Assay dauert 3 Tage; Die qRT-PCR zur Bestimmung des transkriptionellen Ansprechens dauert 3 Tage; Die IF-Färbung dauert 5 Tage. Diese Schritte ähneln in etwa ihren Zellkultur-Äquivalenten, obwohl die Interpretation der Daten ein Verständnis der Fischphysiologie und der Reporterstämme erfordert.
Da es sich um ein Multiple-Variablen-Verfahren12 handelt, sind für Z-REX mehrere Kontrollgruppen notwendig, um Unsicherheiten in den Ergebnissen auszuschließen (Abbildung 1D). Gängige Kontrollgruppen sind: (1) nur DMSO/Fahrzeugbehandlung; (2) Sondenbehandlung, jedoch ohne Lichtbeleuchtung; (3) Lichtbeleuchtung, jedoch ohne Sondenbehandlung; (4) P2A-Split-Konstrukt, in dem Halo und der POI separat ausgedrückt werden, so dass die Proximity-Lieferung abgetragen wird; und (5) hypomorphe Mutanten, deren elektrophil-sensible(r) Rest(e) mutiert ist/sind, wie z.B. Akt3 (C119S)6 und Keap1 (C151S, C273W, C288E)5, die wir in früheren Studien verwendet haben.
Wenn nachgelagerte Analysen eine Western-Blot-Analyse beinhalten, muss vor der Ernte eine Deyolking durchgeführt werden. Die Dotterproteine verringern die Genauigkeit der Beurteilung der Lysatkonzentration und können unspezifisch an Antikörper binden. Bei der Bildgebung lebender Fische oder der IF-Färbung im Whole-Mount haben wir auch unspezifische Fluoreszenzsignale im Dottersack beobachtet, die wahrscheinlich auf autofluoreszierende Proteine im Dottersack oder eine unspezifische Bindung der Antikörper selbst zurückzuführen sind. Wenn das Autofluoreszenzsignal das Signal stört, empfehlen wir, den Dottersack von der Quantifizierung auszuschließen oder verschiedene Regionen separat zu quantifizieren. Die Dechorinierung ist für die Bildgebung lebender Fische und den Whole-Mount-IF-Färbetest erforderlich. Das Chorion kann die Bildgebung und später die Quantifizierung/Zellzählung beeinträchtigen. Die Dechorinierung ist jedoch nur bei Embryonen anwendbar, die älter als 1 dpf sind. Jüngere Embryonen in Blastulations-/Gastrulations-/Segmentierungsstadien sind zu zerbrechlich, um dechorioniert zu werden.
Das hier beschriebene Z-REX-Protokoll basiert auf der mRNA-gesteuerten ektopischen POI-Expression. Das Verfahren ist im Vergleich zur Verwendung/Erzeugung transgener Fischlinien schnell. Die mRNA-gesteuerte Expression ist ubiquitär und vorübergehend und hält für mRNAs, die in diesem Protokoll verwendet werden, mindestens 2 Tage an. Die Dauer der Expression kann jedoch in anderen Fällen variieren. Somit bietet dieser Ansatz ein schnelles und globaleres Untersuchungsfenster für die Auswirkungen eines bestimmten elektrophilen Markierungsereignisses, das mit mehreren Hochdurchsatz-/High-Content-Assays kompatibel ist. Transgene Linien mit stabiler Halo-POI-Expression in bestimmten Geweben sind ebenfalls mit Z-REX11 kompatibel. Solche Zeilen werden am besten verwendet, wenn eine genauere Frage gestellt werden muss, z. B. wenn ein Phänotyp in einem bestimmten Organ aus Zellkulturdaten vorhergesagt wird oder wenn das Screening aus mRNA-Injektionsexperimenten vorhersagt, dass ein bestimmtes Organ empfindlich auf ein elektrophiles Markierungsereignis reagiert. In unserer vorherigen Publikation11 wurde eine herzspezifische Induktion einer antioxidativen Reaktion durch Z-REX anhand von Tg(gstp1:GFP;DsRed-P2A-myl7:Halo-TeV-Keap1)-Fischen demonstriert. Möglicherweise ist es auch möglich, Z-REX bei transgenen Fischen durchzuführen, die älter als 2 dpf sind.
Offenlegungen
Isoform-spezifische niedermolekulare Kinase-Inhibitoren, deren Entdeckung durch REX-Technologien ermöglicht wurde, wurden zum Patent angemeldet
Danksagungen
Finanzierung: Novartis FreeNovation, NFS und EPFL.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol (BME) | Sigma-Aldrich | M6250 | |
| 1.5-mL microcentrifuge tube | Axygen | MCT-150-C-S | |
| 10-cm Petri dishes | Celltreat | 229692 | |
| 2-mL microcentrifuge tube | Axygen | MCT-200-C-S | |
| 30% Acrylamide and bis-acrylamide solution | BioRad | 1610154 | |
| 6-well plate | Celltreat | 229506 | |
| Acetone | Fisher | A/0600/15 | |
| Agarose | GoldBio | A201-100 | |
| All Blue Prestained Protein Standards | BioRad | 1610373 | |
| Ammonium persulfate | Sigma | A3678 | |
| Biotin-dPEG11-azide | Quanta Biodesign | 102856-142 | |
| Bradford Dye | BioRad | 5000205 | |
| BSA | Fisher | BP1600-100 | |
| Capillary tubes | VWR | HARV30-00200 | |
| Chloroform | Supelco, Inc | 1.02445.1000 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Cu(TBTA) | Lumiprobe | 21050 | |
| CuSO4 | Sigma | 209198 | |
| DMSO | Fisher | D128-500 | |
| Donkey anti-mouse-Alexa Fluor 647 | Abcam | ab150107 | 1:800 (IF) |
| Donkey anti-rabbit-Alexa Fluor 647 | Abcam | ab150075 | 1:1000 (IF) |
| Donkey anti-rat-AlexaFluor 568 | Abcam | ab175475 | 1:1000 (IF) |
| ECL substrate | Thermo Fisher Scientific | 32106 | |
| ECL-Plus substrate | Thermo Fisher Scientific | 32132 | |
| End-to-end rotator | FinePCR | Rotator AG | |
| Ethanol | Fisher | E/0650DF/15 | |
| Ethidium bromide | Sigma | E1510 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument Co. | P-97 | |
| Fluorescence stereomicroscope | Leica | M165 FC | |
| Forceps (blunt) | self made | self made by blunting sharp forceps (Fine Science Tools, Dumont #5, 11252-40) | |
| Forceps (sharp) | Fine Science Tools | Dumont #5, 11252-40 | |
| Gel loading tip | Fisher | 02-707-181 | |
| Gel/blot imager | Vilber | Fusion FX imager | |
| Glass beads | Sigma | 45-G1145 | |
| Glass stage micrometer | Meiji Techno. | MA285 | |
| Heat inactivated FBS | Sigma | F2442 | |
| Heated ultrasonic bath | VWR | 89375-470 | |
| HEPES | Fisher | BP310-1 | |
| High capacity streptavidin agarose | Thermo Fisher Scientific | 20359 | |
| Ht-PreHNE alkyne probe | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Imaging plate (10% HBSS buffer, for live embryos) | Made with Petri dish, and 2% agarose in 10% HBSS buffer | ||
| Imaging plate (PBS, for formaldehyde-fixed embryos) | Made with Petri dish, and 2% agarose in PBS | ||
| Injection plate | Made with microinjection mold, Petri dish, and 2% agarose in 10% HBSS buffer | ||
| LDS | Apollo | APOBI3331 | |
| Methanol | VWR | 20864.32 | |
| Microinjection mold | Adaptive Science Tools | TU-1 | |
| Microloader tips | Eppendorf | 930001007 | |
| Micromanipulator | Narishige | MN-153 | |
| Microscope for micro-injection | Olympus | SZ61 | |
| Microscope light source | Olympus | KL 1600 LED | |
| Mineral oil | Sigma | M3516 | |
| mMessage mMachine SP6 Transcription Kit | Ambion | AM1340 | |
| Mouse anti- β-actin-HRP | Sigma | A3854 | 1:20000 (WB) |
| Mouse anti-HaloTag | Promega | G921A | 1:500 (IF) |
| Multi-mode reader | BioTek Instruments | Cytation 5 | |
| Nucleic acid agarose gel electrophoresis apparatus | Biorad | Mini-Sub Cell GT Systems | |
| Oligo(dT)20 | Integrated DNA Technologies | customized: (dT)20 | |
| Orange G | Sigma | O3756 | |
| Paraformaldehyde | Sigma | P6148 | |
| PBS | Gibco | 14190144 | |
| pCS2+8 Halo-2XHA-P2A-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| pCS2+8 Halo-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| Pneumatic PicoPump | WPI | SYS-PV820 | |
| Protein electrophoresis equipment and supplies | Biorad | Mini-PROTEAN Tetra Vertical Electrophoresis | |
| Rabbit anti-active Caspase-3 | BD Pharmingen | 559565 | 1:800 (IF) |
| Rat anti-HA-HRP | Sigma | H3663 | 1:500 (IF and WB) |
| Rat anti-RFP | ChromoTek | 5F8 | 1:800 (IF) |
| Refrigerated centrifuge | Eppendorf | 5417R | |
| RNAseOUT recombinant ribonuclease inhibitor | ThermoFisher Scientific | 10777019 | |
| RnaseZap RNA decontamination solution | ThermoFisher Scientific | AM9780 | |
| Rocking Shaker | DLAB | SK-R1807-S | |
| SDS | Teknova | S9974 | |
| SuperScript III reverse transcriptase | ThermoFisher Scientific | 18080085 | |
| t-Butanol | Sigma | 471712 | |
| TCEP-HCl | Gold Biotechnology | TCEP1 | |
| TeV protease (S219V) | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Tg(lyz:TagRFP) | Zebrafish International Resource Center (ZIRC) | uwm11Tg (ZFIN) | - |
| Tg(mpeg1:eGFP) | Zebrafish International Resource Center (ZIRC) | gl22Tg (ZFIN) | - |
| Thermal cycler | Analytik Jana | 846-x-070-280 | |
| TMEDA | Sigma | T7024 | |
| Transfer pipets | Fisher | 13-711-9D | |
| Tris | Apollo | APOBI2888 | |
| Triton X-100 | Fisher | BP151-100 | |
| TRIzol reagent | Thermo Fisher Scientific | 15596018 | |
| Trypsin inhibitor from Glycine max (soybean) | Sigma | T9003 | |
| Tween 20 | Fisher | BP337-500 | |
| UV lamp with 365-nm light | Spectroline | ENF 240C | |
| UV meter | Spectroline | XS-365 | |
| Vortexer | Scientific Industries, Inc. | Vortex-Genie 2 | |
| Western Blotting Transfer equipment and supplies | Biorad | Mini Trans-Blot or Criterion Blotter | |
| Zebrafish husbandry and breeding equipment | in house | ||
| Zirconia beads | BioSpec | 11079107zx |
Referenzen
- Long, M. J. C., Ly, P., Aye, Y. A primer on harnessing non-enzymatic post-translational modifications for drug design. RSC Medicinal Chemistry. 12 (11), 1797-1807 (2021).
- Parvez, S., Long, M. J. C., Poganik, J. R., Aye, Y. Redox signaling by reactive electrophiles and oxidants. Chemical Reviews. 118 (18), 8798-8888 (2018).
- Parvez, S., et al. T-REX on-demand redox targeting in live cells. Nature Protocols. 11 (12), 2328-2356 (2016).
- Long, M. J. C., et al. Precision electrophile tagging in Caenorhabditis elegans. Biochemistry. 57 (2), 216-220 (2018).
- Van Hall-Beauvais, A., Zhao, Y., Urul, D. A., Long, M. J. C., Aye, Y. Single-protein-specific redox targeting in live mammalian cells and C. elegans. Current Protocols in Chemical Biology. 10 (3), 43 (2018).
- Long, M. J. C., et al. Akt3 is a privileged first responder in isozyme-specific electrophile response. Nature Chemical Biology. 13 (3), 333-338 (2017).
- Poganik, J. R., et al. Wdr1 and cofilin are necessary mediators of immune-cell-specific apoptosis triggered by Tecfidera. Nature Communications. 12 (1), 5736 (2021).
- Lin, H. -. Y., Haegele, J. A., Disare, M. T., Lin, Q., Aye, Y. A generalizable platform for interrogating target- and signal-specific consequences of electrophilic modifications in redox-dependent cell signaling. Journal of the American Chemical Society. 137 (19), 6232-6244 (2015).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative CT method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Wakabayashi, N., et al. Keap1-null mutation leads to postnatal lethality due to constitutive Nrf2 activation. Nature Genetics. 35 (3), 238-245 (2003).
- Huang, K. -. T., et al. Z-REX: Shepherding reactive electrophiles to specific proteins expressed either tissue-specifically or ubiquitously, and recording the resultant functional electrophile-induced redox responses in larval fish. Nature Protocols. , (2023).
- Long, M. J. C., Assari, M., Aye, Y. Hiding in plain sight: The issue of hidden variables. ACS Chemical Biology. 17 (6), 1285-1292 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten