Method Article
Monitoraggio delle risposte di segnalazione on-target nel pesce zebra larvale - Z-REX smaschera meccanismi precisi di farmaci elettrofili e metaboliti
In questo articolo
Riepilogo
Zebrafish targeting reactive electrophiles and oxidants (Z-REX) è un metodo basato sulla biologia chimica per lo studio della segnalazione reattiva di piccole molecole. Questa tecnica può essere applicata a pesci vivi di diversi stadi di sviluppo. Qui, abbiniamo saggi standard nel pesce zebra con Z-REX per l'analisi del percorso di segnalazione.
Abstract
I metaboliti reattivi e i farmaci elettrofili correlati sono tra le piccole molecole più difficili da studiare. Gli approcci convenzionali per decostruire la modalità d'azione (MOA) di tali molecole sfruttano il trattamento di massa di campioni sperimentali con un eccesso di una specifica specie reattiva. In questo approccio, l'elevata reattività degli elettrofili rende l'etichettatura non discriminatoria del proteoma in modo dipendente dal tempo e dal contesto; Anche le proteine e i processi redox-sensibili possono essere influenzati indirettamente e spesso in modo irreversibile. In tale contesto di innumerevoli potenziali bersagli ed effetti secondari indiretti, collegare il fenotipo all'impegno specifico del bersaglio rimane un compito complesso. Il pesce zebra che prende di mira gli elettrofili reattivi e gli ossidanti (Z-REX) - una piattaforma di somministrazione elettrofila reattiva su richiesta adattata per l'uso nel pesce zebra larvale - è progettato per fornire elettrofili a una specifica proteina di interesse (POI) in embrioni di pesci vivi altrimenti imperturbati. Le caratteristiche chiave di questa tecnica includono un basso livello di invasività, insieme a dosaggio, chemiotipo e somministrazione elettrofila di precisione controllata spaziotemporalmente. Pertanto, in combinazione con una serie unica di controlli, questa tecnica elude gli effetti fuori bersaglio e la tossicità sistemica, altrimenti osservati a seguito di un'esposizione incontrollata di massa di animali a elettrofili reattivi e farmaci elettrofili pleiotropici. Sfruttando Z-REX, i ricercatori possono stabilire un punto d'appoggio nella comprensione di come le risposte individuali allo stress e gli output di segnalazione sono alterati a seguito di specifici impegni del ligando reattivo con uno specifico POI, in condizioni quasi fisiologiche in animali viventi intatti.
Introduzione
Una miriade di eventi di segnalazione cellulare coinvolgono reazioni tra piccole molecole reattive (prodotte endogenamente nella cellula o xenobiotici / xenomeboliti, come i farmaci) e il loro bersaglio proteico. In molti casi, un livello substechiometrico di tali eventi di legame covalente può innescare risposte cellulari, portando a cambiamenti, ad esempio, nello sviluppo, nel metabolismo, nell'apoptosi e / o nella risposta immunitaria1. Tuttavia, decostruire la modalità d'azione (MOA) collegando specifici eventi di legame alle loro conseguenze fenotipiche si è dimostrato difficile. I metodi tradizionali di dosaggio in bolo che comportano l'introduzione di alte concentrazioni delle specie reattive spesso comportano una moltitudine di proteine modificate, nonché un'eccessiva tossicità per l'organismo modello2. Tali condizioni sono tutt'altro che ideali. È stato sviluppato un metodo per risolvere questi problemi in coltura cellulare utilizzando la somministrazione elettrofila localizzata di precisione in un contesto cellulare nativo, chiamato T-REX (targetable reactive electrophiles and oxidants)3. Negli anni successivi, l'attenzione si è rivolta agli esperimenti in organismi interi, che consentono l'opportunità di studiare proteine in specifici contesti cellulari in cellule non trasformate. Pertanto, abbiamo esteso la tecnica per essere compatibile con diversi modelli, inclusi i modelli di embrioni Danio rerio . Qui presentiamo Z-REX (zebrafish targeting reactive electrophiles and oxidants) (Figura 1).
Per comprendere Z-REX, questo articolo presenta innanzitutto le tecnologie REX e i loro concetti sottostanti. Al loro interno, queste tecniche modellano la segnalazione endogena delle specie elettrofile reattive fisiologiche (RES) imitando il modo in cui gli elettrofili naturali sono prodotti localmente in vivo con precisione spaziotemporale. La proteina di interesse (POI) è espressa come costrutto di fusione ad Halo; quest'ultimo ancora, in una stechiometria 1:1, la sonda permeabile ai tessuti e inerte che porta il RES fotogabbiato. Uno di questi RES endogeni è il 4-idrossinonenale (HNE di seguito), che viene fotoingabbiato nella sonda Ht-PreHNE. In molti casi, usiamo una versione alchini-funzionalizzata di HNE [cioè HNE(alchini)], che ha proprietà biologiche essenzialmente identiche a HNE, ma può essere etichettata con la chimica del clic. La sonda, che è anche funzionalizzata con un cloroalcano per la reattività con Halo, è indicata come Ht-PreHNE (alchino). Il complesso della fusione Halo-POI e la sonda così formata consentono l'erogazione prossimale di RES al POI fuso dopo irradiazione con luce UV. Se il POI reagisce rapidamente con il RES liberato, la conseguente etichettatura covalente del POI con RES ci consente di identificare cisteine cineticamente privilegiate.
Z-REX prende i suddetti vantaggi delle tecnologie REX e li applica ampiamente per studiare specifiche vie di segnalazione nei pesci vivi. Questo protocollo è stato ottimizzato per i pesci zebra (D. rerio), poiché sono organismi vertebrati geneticamente trattabili che sono trasparenti durante lo sviluppo, e quindi ideali per tecniche opto-chimiche / genetiche come le tecnologie REX. Tuttavia, è probabile che una strategia simile funzioni bene anche su altre specie di pesci geneticamente trattabili, poiché l'ampia applicabilità del metodo è dovuta al meccanismo pseudo-intramolecolare della somministrazione elettrofila derivata dai lipidi (LDE). In effetti, la procedura è altamente biocompatibile, poiché i pesci possono essere trattati con l'elettrofilo fotocagato Z-REX [ad esempio, Ht-PreHNE (alchino)] per almeno 48 ore senza alcun impatto evidente sullo sviluppo. Un protocollo simile funziona in C. elegans 4,5.
Il protocollo descrive innanzitutto l'uso dell'iniezione di mRNA per produrre un'espressione transitoria di un costrutto di fusione Halo-POI non nativo in modelli embrionali di zebrafish, 1-1,5 giorni dopo la fecondazione (dpf). Ciò si traduce nell'espressione della proteina ectopica nella maggior parte delle cellule all'interno del pesce (di seguito denominata "ubiquitaria"), piuttosto che in tessuti o locali specifici; Tuttavia, i dati mostrano che in alcuni casi si possono osservare effetti specifici delle cellule. Dopo l'iniezione, gli embrioni vengono incubati con una bassa concentrazione [0,3-5 μM Ht-PreHNE(alchini)] della sonda fino a 30,5 ore dopo la fecondazione (hpf). Quindi, in un momento prescritto dall'utente, la consegna del RES al POI all'interno del pesce si ottiene fotosbloccando per 2-5 minuti. Dopo il fotosblocco del RES, è possibile eseguire una varietà di saggi fenotipici a valle nelle successive 2-10 ore: 1) imaging dal vivo delle linee reporter (Figura 2A); 2) valutazione dell'etichettatura del target mediante analisi western blot (Figura 3); 3) analisi trascrittomica (Figura 4); o 4) immunofluorescenza a montaggio intero (Figura 5).
Come esempio di imaging dal vivo delle linee reporter, Z-REX è dimostrato in tandem con l'imaging dal vivo delle linee di pesce, Tg (lyz: TagRFP) e Tg (mpeg1: eGFP), per misurare come la modifica RES di uno specifico POI del sensore elettrofilo (vale a dire Keap1) diminuisce i livelli di neutrofili e macrofagi, rispettivamente, senza effetti osservabili su altre cellule nel pesce. Tuttavia, abbiamo dimostrato in precedenza che la marcatura POI e la conseguente segnalazione del pathway dagli studi T-REX possono essere riprodotte utilizzando Z-REX per diverse proteine: Akt3 6, Keap17 e Ube2v26. Nel complesso, con Z-REX, gli scienziati possono studiare le conseguenze della modificazione covalente dei POI da parte delle FER nel contesto di diversi percorsi redox complessi. Questa tecnica è innescata per individuare bersagli e i loro residui funzionali per la progettazione di farmaci covalenti e nuovi meccanismi farmacologici in un modello animale intero più rilevante dal punto di vista contestuale.
Protocollo
Le procedure di allevamento e manipolazione del pesce zebra presso la Cornell University (Stati Uniti) sono state eseguite seguendo le linee guida del National Institutes of Health (NIH) e approvate dall'Institutional Animal Care and Use Committee (IACUC). Le procedure di allevamento e manipolazione del pesce zebra presso l'unità zebrafish del Politecnico federale di Losanna (EPFL, Svizzera) sono state eseguite secondo la legge sulla protezione degli animali RS 455 e l'ordinanza sulla protezione degli animali RS 455.1, con autorizzazione veterinaria cantonale VD-H23.
NOTA: In questo protocollo, le linee di pesce Tg( lyz:TagRFP ) e Tg(mpeg1:EGFP) che esprimono Halo-TeV-Keap1 sono utilizzate per dimostrare Z-REX. Il metodo può essere esteso ad altre proteine di interesse, linee di pesci reporter transgenici e saggi biologici a valle. Fare riferimento alla tabella supplementare 1 per i buffer utilizzati in questo studio. Tutti i reagenti, gli strumenti, le attrezzature, gli anticorpi, i plasmidi, i ceppi e le attrezzature del pesce zebra sono elencati nella tabella dei materiali.
1. Preparazione dell'mRNA
- Preparare l'mRNA Halo-TeV-Keap1-2xHA e Halo-2xHA-P2A-Keap1-2xHA utilizzando il kit di trascrizione in vitro mMessage mMachine SP6.
NOTA: Seguire le istruzioni del produttore ed eseguire reazioni su scala 40 μL per ciascun mRNA. Risciogliere il pellet di mRNA in 10 μL di acqua priva di nucleasi. - Valutare la qualità dell'mRNA e misurare la concentrazione mediante spettrofotometro a microvolume ed elettroforesi su gel di agarosio. Un mRNA di buona qualità dovrebbe avere un rapporto A260/A280 di circa o superiore a 2,0.
- Diluire l'mRNA a 1-1,5 mg/ml con acqua priva di nucleasi.
- Aliquotare la soluzione di mRNA (1-2 μL per tubo) e conservare le aliquote a -80 °C.
2. Produzione di embrioni di pesce
- Opzione 1: produzione di embrioni di pesci wild-type (WT).
- Organizza 10 coppie di pesci che attraversano in 10 vasche separate, con ogni vasca contenente un divisorio tra il pesce genitore maschio e femmina.
NOTA: Un totale di 10 coppie incrociate fornisce in genere un numero sufficiente di embrioni per i saggi. Il numero di coppie incrociate può essere regolato in base al progetto/necessità dell'esperimento e alla fecondità del pesce genitore. - La mattina seguente, dopo aver installato l'iniettore, rimuovere i divisori in cinque dei serbatoi. Attendere 30 minuti affinché il pesce si accoppia.
- Spostare il pesce genitore in un altro acquario, raccogliere gli embrioni facendo passare l'acqua dell'acquario attraverso un colino, quindi sciacquare gli embrioni dal colino in una capsula di Petri di 10 cm. Questi embrioni saranno utilizzati per il primo ciclo di iniezioni.
NOTA: Se la qualità delle uova di un determinato lotto è scarsa (ad esempio, le uova sono opache a causa dell'aggregazione delle proteine), non metterle in comune con gli altri embrioni. - (Facoltativo) Eseguire procedure simili a quelle descritte nei punti 2.1.2-2.1.3 sugli altri cinque serbatoi per il successivo ciclo di iniezione.
NOTA: È meglio eseguire solo un ciclo di iniezione, per ridurre al minimo la differenza di età tra gli embrioni. Tuttavia, se sono necessari più embrioni di quelli che possono essere iniettati in un lotto, si raccomanda di eseguire due cicli di iniezione per garantire che gli embrioni rimangano in uno stadio di 1-4 cellule durante l'iniezione di mRNA. Il numero di cicli di iniezione può essere regolato in base all'abilità di iniezione dell'operatore e al design dell'esperimento. Tuttavia, si consiglia di eseguire l'intera procedura (dalla rimozione del primo divisore all'iniezione dell'ultimo embrione) entro 2 ore. Una grande differenza di età tra gli embrioni può compromettere l'affidabilità e la riproducibilità dei risultati dell'esperimento.
- Organizza 10 coppie di pesci che attraversano in 10 vasche separate, con ogni vasca contenente un divisorio tra il pesce genitore maschio e femmina.
- Opzione 2: Produrre embrioni di pesce reporter eterozigoti transgenici di neutrofili/macrofagi.
- Impostare 10 coppie di pesci incrociati in 10 vasche separate, con ogni vasca inserita con un divisorio tra pesci genitori maschi e femmine: pesce WT contro Tg (lyz: TagRFP) o pesce WT contro Tg (mpeg1: eGFP).
NOTA: Evitare incroci tra pesci transgenici eterozigoti, che possono influenzare le letture fluorescenti a valle poiché i pesci reporter omozigoti mostrano un segnale fluorescente più elevato rispetto ai pesci eterozigoti. Le linee di reporter transgeniche e gli embrioni WT possono essere facilmente distinti durante l'imaging. Avere una miscela di WT ed embrioni eterozigoti nello stesso pool non è un problema. lyz:TagRFP riporta neutrofili e mpeg:eGFP riporta macrofagi. Questo protocollo può essere applicato anche ad altre linee di pesci reporter. - Seguire i passaggi 2.1.2-2.1.4.
- Impostare 10 coppie di pesci incrociati in 10 vasche separate, con ogni vasca inserita con un divisorio tra pesci genitori maschi e femmine: pesce WT contro Tg (lyz: TagRFP) o pesce WT contro Tg (mpeg1: eGFP).
3. Configurazione del microiniettore
- Accendere la fonte d'aria, impostare la contropressione su 0,2-0,5 psi, quindi impostare la pressione di iniezione su 25-30 psi. L'intervallo specifico di pressione mostrato è in genere raccomandato.
NOTA: È essenziale avere una contropressione stabile per evitare il riflusso dei mezzi di pesce nell'ago. Quando si calibra il volume di iniezione nella fase 3.8, deve essere modificato solo il tempo di iniezione. Non modificare la pressione di iniezione nei passaggi successivi; Una bassa pressione di iniezione può portare al fallimento dell'iniezione a causa della tensione superficiale e interfacciale, mentre un'alta pressione di iniezione può danneggiare gli embrioni. - Pulire l'apparecchiatura e la piattaforma di iniezione con la soluzione di decontaminazione della RNasi.
NOTA: la RNasi, che degrada l'mRNA, potrebbe provenire dall'operatore o dall'apparecchiatura. È necessario eseguire la pulizia prima dell'esperimento e indossare i guanti. - (Facoltativo) Se si co-iniettano mRNA e morfolino, premiscelare i due in una provetta da 0,2 ml.
NOTA: Z-REX funziona bene utilizzando la soluzione di mRNA Halo-TeV-Keap1-2xHA con una concentrazione di 250-1500 ng/μL. Diversi morpholinos sono stati utilizzati anche nel pesce zebra e le concentrazioni ottimali sono state riportate7. Se si utilizza un morfolino con una sequenza non pubblicata, l'operatore deve prima valutare la tossicità e l'efficienza di knockdown genico del morfolino prima di utilizzarlo in Z-REX. - Trasferire 1-2 μL di mRNA (e/o morfolino ove applicabile7) in un ago per iniezione con punta di pipetta micro-caricatore.
NOTA: Se si preparano aghi con un estrattore di micropipette Flaming/Brown, la configurazione è la seguente. Calore: 520 unità; forza di trazione: 60 unità; velocità: 70 unità; ritardo: 155 unità; pressione: 550 unità; rampa: 530 unità. - Installare l'ago sul manipolatore di microiniezione.
NOTA: La contropressione dalla sorgente d'aria dovrebbe spingere la soluzione di mRNA (/ morfolino) alla punta dell'ago. - Utilizzare una pinza affilata o una lama di rasoio per rompere la punta dell'ago, creando un'apertura adatta per l'iniezione.
- Immergere la punta dell'ago in olio minerale su un micrometro di stadio.
- Applicare due o tre impulsi di iniezione per rimuovere le bolle d'aria nella punta.
- Calibrare la dimensione della goccia a 2 nL modificando il tempo di iniezione.
NOTA: Questo è meglio eseguire iniettando in olio minerale (che imita la viscosità del sacco vitellino) posto su un emocitometro. Utilizzando un microscopio, utilizzare le linee della griglia dell'emocitometro per stimare la dimensione della goccia formata durante l'iniezione e regolare il tempo di iniezione di conseguenza. Sebbene a volte venga usato il colorante rosso fenolo, la necessità di esso nella procedura di iniezione di mRNA qui descritta non è stata osservata.
4. Microiniezione
- Riempire una piastra di iniezione con una soluzione salina bilanciata fresca al 10% di Hank (HBSS) e allineare gli embrioni nelle scanalature della piastra con una pinza smussata.
NOTA: La piastra di iniezione è preparata con agarosio al 2% in mezzo HBSS al 10%; Le scanalature sono formate usando uno stampo di plastica. - Immergere la punta dell'ago nel mezzo HBSS al 10% nella piastra di iniezione.
- Applicare due o tre impulsi di iniezione per rimuovere le bolle d'aria nella punta.
- Per ogni iniezione, penetrare nel corion e nel sacco vitellino in una sola mossa e applicare un impulso di iniezione. Questo liquido iniettato può essere visto subito dopo l'iniezione come un piccolo sferoide all'interno del sacco vitellino. Questo piccolo sferoide si dissipa relativamente rapidamente. Ripetere questo passaggio per gli altri embrioni, fino a ottenere un numero sufficiente di embrioni iniettati.
NOTA: Il tasso di sopravvivenza degli embrioni (sia iniettati che non iniettati) varia tipicamente dal 50% al 90%. Obiettivo di iniettare il doppio del numero di embrioni necessari per ciascun gruppo di controllo/sperimentale. Nel test di pull-down della biotina, sono necessari 100-140 embrioni vitali per ogni condizione. Nel test qRT-PCR, sono raccomandati cinque embrioni vitali per ogni condizione. La dimensione del campione per l'imaging di pesci vivi e il test di colorazione con immunofluorescenza a montaggio intero è definita dall'utente; Si raccomanda di avere almeno 20 embrioni vitali per condizione per produrre un buon potere statistico nell'analisi. - Risciacquare gli embrioni iniettati in una nuova capsula di Petri da 10 cm contenente terreni freschi al 10% di HBSS.
NOTA: Gli embrioni possono essere facilmente risciacquati dalle scanalature usando un flacone a spruzzo. - Raggruppare gli embrioni non iniettati in un'altra piastra.
NOTA: Gli embrioni non iniettati possono servire come controlli di qualità per la salute del pesce, l'espressione proteica di base, i livelli di fluorescenza di fondo, ecc., Se necessario. Se la procedura di iniezione ha funzionato bene e l'mRNA/morfolino iniettato non è letale per gli embrioni, gli embrioni iniettati e non iniettati dovrebbero avere una vitalità simile.
5. Z-REX
- Distribuire gli embrioni iniettati in piatti di 10 cm, secondo la configurazione dell'esperimento (cioè il numero di gruppi di controllo / esperimento).
- In una stanza buia con illuminazione a luce rossa, sostituire il fluido con 30 mL di HBSS al 10% con o senza 1 μM Ht-PreHNE (alchino).
NOTA: Ht-PreHNE (alchino) è un composto leggero-labile. Gli embrioni dovrebbero essere tenuti al buio nei seguenti passaggi. - Incubare gli embrioni a 28,5 °C al buio.
- A 30,5 hpf, in una stanza buia, sostituire il fluido con un nuovo 30 ml di HBSS al 10%.
NOTA: quando si sostituisce il supporto, rimuovere il più possibile il vecchio supporto. Questo è fondamentale per rimuovere la quantità non legata / in eccesso di Ht-PreHNE (alchini) dagli embrioni. - Incubare gli embrioni a 28,5 °C al buio per 30 minuti.
- Ripetere i passaggi 5.4-5.5 altre due volte.
- Accendere la lampada UV (365 nm, 3 mW/cm2) per 5 minuti per preriscaldare la lampada.
NOTA: la fase di preriscaldamento della lampada deve essere eseguita prima del punto 5.8. La potenza della lampada è inferiore / instabile nei primi minuti dopo l'accensione. La potenza della lampada deve essere misurata regolarmente da un misuratore UV. - A 32 hpf, esporre gli embrioni alla luce UV.
- Opzione 1: Per le letture a valle, come l'imaging di pesci vivi, la colorazione con immunofluorescenza a montaggio intero, l'analisi di fluorescenza in gel (accoppiamento a clic con azide Cy5), RNA-seq o qRT-PCR, esporre gli embrioni alla luce UV per 3 minuti, ruotando le piastre ogni 30 s.
- Opzione 2: Per le letture a valle, come il saggio di pull-down della biotina, esporre gli embrioni alla luce UV per un massimo di 5 minuti (e minimo 3 minuti), ruotando le piastre ogni 30 secondi e raffreddare le piastre sul ghiaccio per 1 minuto.
NOTA: se si utilizzano sonde diverse, il tempo di esposizione alla luce deve essere ottimizzato, a seconda del t1/2 di fotouncaging per una determinata sonda elettrofila fotoingabbiata e sorgente luminosa dispiegata. t1/2il fotouncaging può essere determinato utilizzando procedure note8. Per Ht-PreHNE(alchino), t 1/2 è < 1 min3; Pertanto, il tempo indicato sopra è sufficiente.
6. Saggi a valle
- Opzione 1: Saggio fenotipico. Live imaging del reporter transgenico di neutrofili/macrofagi
linee di pesce, Tg(lyz:TagRFP) e Tg(mpeg1:eGFP) (Figura 2)- Anestetizzare gli embrioni mediante incubazione a 4 °C per 10 min.
NOTA: Si raccomanda di avere almeno 20 embrioni vitali per condizione. - Rimuovere gli embrioni non fecondati / morti dalla piastra.
NOTA: Gli embrioni non fecondati / morti sono torbidi / non trasparenti e possono essere identificati visivamente. Se si osserva un alto tasso di mortalità, controllare nuovamente la procedura di iniezione o cercare di ridurre la concentrazione di mRNA o morfolino. - Dechorionate gli embrioni con una pinza affilata. Tenere il corion con un paio di pinze, senza toccare il pesce larvale, e usare l'altro paio di pinze per strappare il corion. L'embrione è fragile. Toccare il corion solo quando si esegue la dechorionation.
NOTA: È comune, soprattutto nei principianti, danneggiare alcuni embrioni durante la dechorionation. Pertanto, avere sempre più embrioni del minimo necessario. - Montare gli embrioni su una piastra di agarosio al 2% (preparata con terreno HBSS al 10%) e visualizzare gli embrioni con uno stereomicroscopio (campo luminoso e rispettivi canali fluorescenti) (Figura 2A, C, G).
NOTA: Dopo Z-REX [combinazione di iniezione di mRNA Halo-TeV-Keap1-2xHA e trattamento con Ht-PreHNE(alchino)], la deplezione dei neutrofili (lyz:TagRFP) è risultata più significativa a 36 hpf (4 ore dopo Z-REX), mentre la riduzione dei macrofagi (mpeg1:eGFP) è stata più significativa a 34 hpf (2 h dopo Z-REX) (Figura 2E,F). Altri punti temporali possono essere utilizzati se si utilizzano diverse linee reporter, mRNA / morfolino o sonde. Il tempo di esposizione e/o il guadagno devono essere ottimizzati per visualizzare singole celle o le specifiche (ultra)strutture desiderate. - Contare il numero di neutrofili/macrofagi di ciascun pesce in base a ImageJ (NIH) (Figura 2B, D-F, H). Usate lo strumento selezione a mano libera in ImageJ per cerchiare l'intero pesce e usate l'opzione Trova Maxima per contare le celle fluorescenti.
- Anestetizzare gli embrioni mediante incubazione a 4 °C per 10 min.
- Opzione 2: valutazione dell'etichettatura target. Accoppiamento a clic di biotina e saggio di pull-down della biotina (Figura 3)
- Anestetizzare gli embrioni mediante incubazione a 4 °C. Questo di solito richiede 10 minuti.
NOTA: Per ottenere abbastanza lisato di pesce, sono necessari 100-140 embrioni vitali per ogni condizione. - Rimuovere gli embrioni non fecondati / morti dalla piastra.
- Eseguire dechorionation e deyolking. Eseguire le due manipolazioni tenendo il corion con un paio di pinze affilate, usando l'altro paio di pinze per penetrare nel sacco vitellino, quindi separando il sacco vitellino mentre si rimuove il corion per consentire alle proteine del tuorlo di uscire.
NOTA: È fondamentale rimuovere le proteine del tuorlo in questo passaggio. Le abbondanti proteine del tuorlo nel campione interferiscono con le analisi successive. - Trasferire gli embrioni distolati in una provetta da 1,5 ml.
NOTA: Ruotare la piastra per centrare gli embrioni disaccoppiati, facilitando la raccolta. I detriti del corion più leggero si allontanano durante questo processo. - Dopo che gli embrioni si sono depositati sul fondo del tubo, rimuovere il surnatante e aggiungere 1 ml di soluzione salina refrigerata tamponata HEPES (pH 7,6).
- Ripetere il passaggio 6.2.5 altre due volte.
- (Facoltativo) Se non si intende procedere immediatamente con la fase successiva, rimuovere il tampone, congelare i campioni in azoto liquido e conservarli a -80 °C.
NOTA: I pellet di pesce Deyolked possono essere congelati in azoto liquido e conservati a -80 °C. I campioni congelati possono essere conservati a -80 °C per un massimo di 2 settimane. - Risospendere i pellet di pesce nel tampone di lisi.
NOTA: Il tampone di lisi (pH 7,6) è composto da 50 mM HEPES, 100 mM NaCl, 1% Triton X-100, 0,3 mM TCEP, 2x inibitori della proteasi Roche cOmplete Mini EDTA-free e 0,1 mg/mL di inibitore della tripsina da soia. Un embrione dà circa 2 μg di lisato. Utilizzare 100 μL di tampone di lisi per ogni 120 embrioni. Due inibitori della proteasi privi di EDTA e inibitori della tripsina della soia devono essere aggiunti al tampone di lisi appena prima dell'uso. - Aggiungere il 20% di perle di zirconia v / v al tubo.
- Vortice per 20 s, congelamento flash in azoto liquido e scongelamento a bagnomaria a 37 °C.
- Ripetere il passaggio 6.2.10 altre due volte.
- Centrifugare la soluzione a 21.000 x g a 4 °C per 10 minuti.
- Trasferire il surnatante in una nuova provetta prerefrigerata da 1,5 ml.
- Misurare la concentrazione proteica mediante il saggio di Bradford.
- Diluire il lisato a 1 mg/ml.
- Per ogni condizione, trasferire 170 μg di lisato in una provetta da 2 ml.
- Mescolare il lisato del punto 6.2.16 con 0,2 mg/mL di proteasi TeV (S219V) e incubare la soluzione a 37 °C per 30 minuti.
NOTA: Per i gruppi non trattati con proteasi TeV, è sufficiente miscelare il lisato con un uguale volume di tampone di lisi alla soluzione di proteasi TeV utilizzata in altri gruppi. - Preparare una miscela master 10x per la reazione click biotina-azide: 10% w/v SDS, 10 mM CuSO4, 1 mM Cu(TBTA), 1 mM biotina-azide e 20 mM TCEP.
NOTA: aggiungere TCEP al mix appena prima del punto 6.2.19. - Aggiungere 8,5 μL di t-BuOH e 17 μL di miscela master di reazione a clic 10x al lisato (digerito con proteasi TeV) del punto 6.2.17. Vortice, centrifugare e incubare la soluzione a 37 °C per 15 minuti.
- Aggiungere un altro 1 mM TCEP alla soluzione, quindi vortice, centrifugare e incubare la soluzione a 37 °C per altri 15 minuti. Il tempo di incubazione per le fasi 6.2.19-6.2.20 è di 30 minuti in totale.
NOTA: Questo integratore di TCEP, un reagente riducente per la generazione di Cu(I), migliora l'efficienza della reazione del clic. - Aggiungere 600 μL di etanolo da -20 °C a ciascun tubo, vortice la soluzione e incubarla a -80 °C durante la notte.
NOTA: I campioni possono essere conservati a -80 °C per 1 settimana; In caso contrario, procedere immediatamente con il passaggio successivo. - Centrifugare la soluzione a 21.000 x g a 4 °C per 1 ora.
NOTA: Un pellet dovrebbe formarsi sul fondo del tubo dopo la centrifugazione, che è la frazione desiderata. - Rimuovere il surnatante, aggiungere 1 mL di etanolo da -20 °C, vortice e centrifugare la soluzione a 21.000 x g a 4 °C per 10 minuti.
- Ripetere il passaggio 6.2.23.
- Rimuovere il surnatante, aggiungere 1 mL di acetone a -20 °C, vortice e centrifugare la soluzione a 21.000 x g a 4 °C per 10 minuti.
- Rimuovere il surnatante. Lasciare evaporare l'acetone residuo in eccesso, anche se non completamente asciutto.
- Risospendere il pellet in 100 μL di tampone di risospensione (8% p/v di litio dodecilsolfato [LDS], 1 mM EDTA in 50 mM HEPES soluzione salina, pH 7,6), vortice per 15 s e sonicare fino a quando il pellet non si scioglie.
- Centrifugare la soluzione a 21.000 x g a temperatura ambiente (RT) per 5 minuti.
- Trasferire il surnatante in una nuova provetta da 2 mL e aggiungere 1,5 mL di soluzione salina HEPES da 50 mM, pH 7,6.
NOTA: La concentrazione finale di LDS in questa fase è dello 0,5%. Concentrazioni più elevate di LDS possono minare l'efficienza di pull-down. Pertanto, sebbene l'aumento della concentrazione LDS possa aiutare la riduzione del legame non specifico, può anche ridurre l'efficienza del pulldown. Di conseguenza, si raccomanda di non modificare la concentrazione LDS in questa fase. - Raccogliere il campione "in ingresso" (Figura 3): trasferire 30 μL di lisato da 1 mg/mL in una nuova provetta da 1,5 mL e aggiungere 10 μL di tampone 4x Laemmli contenente il 6% di β-mercaptoetanolo (BME). Flash congelare la soluzione e conservarla a -80 °C.
- Trasferire 100 μL di resina ad alta capacità di streptavidina volumetrica del letto in un nuovo tubo da 2 ml. Aggiungere 1 mL di LDS allo 0,5% in soluzione salina HEPES da 50 mM (pH 7,6), centrifugare a 1.500 x g a RT per 2 minuti e rimuovere il surnatante. Ripetere il lavaggio con un altro 1 mL di LDS allo 0,5% in soluzione salina HEPES 50 mM (pH 7,6).
- Trasferire la soluzione dal punto 6.2.29 al tubo contenente la resina prelavata ad alta capacità di streptavidina a partire dal punto 6.2.31 e incubare la soluzione su un miscelatore end-over-end a RT per 4-6 ore.
- Centrifugare la miscela a 1.500 x g a RT per 2 minuti, prelevare 30 μL di surnatante e mescolarla con 10 μL di 4x tampone di campionamento Laemmli contenente il 6% di BME per il campione "flowthrough". Quindi, rimuovere il supernatante rimanente.
NOTA: I campioni "Flowthrough" possono essere analizzati mediante western blotting per verificare l'efficienza di pull-down della streptavidina. È essenziale rimuovere il più possibile il surnatante per eliminare le proteine non legate. Innanzitutto, rimuovere la maggior parte del surnatante utilizzando una pipetta P-1000, quindi rimuovere il surnatante rimanente utilizzando una pipetta P-20 con una punta di caricamento del gel. - Aggiungere 1 mL di LDS allo 0,5% in soluzione salina HEPES 50 mM (pH 7,6) alla resina e incubare la miscela per 30 minuti a RT con rotazione end-over-end.
- Centrifugare la miscela a 1.500 x g a RT per 2 minuti e rimuovere il surnatante.
NOTA: In genere, lo 0,5% di LDS è sufficiente per rimuovere la maggior parte delle proteine leganti non specifiche. Se nell'analisi successiva si osservano ancora segnali di legame non specifici, la concentrazione di LDS nel tampone di lavaggio può essere aumentata. - Ripetere i passaggi 6.2.34-6.2.35 altre due volte.
- Aggiungere 40 μL di 2x tampone campione Laemmli contenente il 6% di BME alla resina.
- Eluire le proteine legate incubando la miscela a 98 °C per 5 minuti.
- Centrifugare la miscela a 21.000 x g a RT per 5 minuti e trasferire il surnatante in una nuova provetta da 1,5 ml. Questo è il campione "eluito".
NOTA: il caricamento diretto della soluzione contenente resina dal punto 6.2.38 nel gel può influire sull'analisi SDS-PAGE. - Caricare 20 μL in ciascun pozzetto di gel di poliacrilammide al 10% a 10 corsie ed eseguire l'elettroforesi su gel.
NOTA: eseguire il gel a una tensione inferiore (120 V) fino a quando il fronte del colorante raggiunge il gel risolvente e modificare la tensione a 170 V. Interrompere il programma dopo l'uscita del fronte del colorante. - Eseguire il western blotting con anti-HA, anti-Halo o altri anticorpi che rilevano le proteine domestiche (Figura 3).
- Anestetizzare gli embrioni mediante incubazione a 4 °C. Questo di solito richiede 10 minuti.
- Opzione 3: Analisi trascrittomica. RNA-seq e qRT-PCR (Figura 4)
NOTA: Si raccomanda vivamente di utilizzare embrioni deposti entro 15 minuti l'uno dall'altro per questo test. La differenza di età degli embrioni influisce significativamente sui risultati del test.- Anestetizzare gli embrioni incubandoli a 4 °C per 10 min, 2 ore dopo Z-REX.
- Eseguire la dechorionazione con pinze acuminate (punto 6.1.3).
- (Facoltativo) Eseguire la segmentazione con una pinza (ad esempio, separare la testa dalla coda) se diversi segmenti devono essere analizzati separatamente.
- Se si estrae l'RNA da un intero embrione, trasferire da tre a cinque embrioni in una provetta da 1,5 ml. Se si estrae l'RNA dalla testa o dalla coda, trasferire 10-12 segmenti sezionati in un tubo da 1,5 ml.
NOTA: Si raccomanda di eseguire l'esperimento con da tre a cinque repliche biologiche. - Aggiungere 1 mL di reagente TRIzol e perle di vetro al tubo.
NOTA: È stato scoperto che le perle di vetro funzionano meglio delle perle di zirconia per l'estrazione dell'RNA. - Vortice la miscela per 30 s.
NOTA: Se non si procede immediatamente con la fase successiva, la soluzione può essere conservata a -80 °C per 1-3 settimane. - Estrarre l'RNA secondo le istruzioni del produttore.
- Valutare la qualità e la concentrazione dell'RNA mediante spettrofotometro a microvolume ed elettroforesi su gel di agarosio.
NOTA: L'RNA di buona qualità dovrebbe avere un rapporto A260/A280 di circa o superiore a 2,0. - Sottoporre l'RNA al sequenziamento o trattare 1 μg di RNA con DNasi I di grado di amplificazione e trascrivere inversamente utilizzando la trascrittasi inversa in apice III e oligo-(dT)20. Eseguire questo passaggio secondo le istruzioni del produttore.
- Eseguire qRT-PCR e analizzare i dati con il metodo ΔΔCT9 (Figura 4B-D).
- Opzione 4: analisi dell'espressione dei POI e della colocalizzazione. Saggio di colorazione con immunofluorescenza a montaggio intero (Figura 5)
NOTA: Gli embrioni fissati con formaldeide sono fragili. Evitare agitazioni vigorose e maneggiare con cura.- Dechorionare gli embrioni seguendo le fasi 6.1.1-6.1.3.
- Trasferire gli embrioni in una provetta da 1,5 ml.
NOTA: Ogni provetta deve avere un numero uguale di embrioni e non più di 40 embrioni. - Dopo che gli embrioni si sono depositati sul fondo del tubo, rimuovere il surnatante e aggiungere 1 ml di soluzione salina tamponata fosfato (PBS) (pH 7,6).
- Ripetere il passaggio 6.4.3 ancora una volta.
- Rimuovere il surnatante, aggiungere 1 mL di formaldeide al 4% in PBS (pH 7,6) e incubare il tubo a 4 °C durante la notte con un leggero oscillo.
NOTA: I campioni in soluzione di formaldeide possono essere conservati a 4 °C per 1 settimana. - Rimuovere il surnatante, aggiungere 1 mL di metanolo a -20 °C e incubare il tubo su un lato a -20 °C per almeno 18 ore.
NOTA: I campioni possono essere conservati a -20 °C per 1 mese o più. - Rimuovere il surnatante e aggiungere 1 mL di tampone PDT (0,3% v/v Triton X-100, 0,1% v/v Tween-20 e 1% v/v dimetil solfossido [DMSO] nel tampone PBS).
- Ripetere il punto 6.4.7 e incubare il tubo a RT per 30 minuti con un leggero dondolio.
- Rimuovere il surnatante, aggiungere 1 mL di tampone bloccante (siero bovino fetale inattivato dal calore [FBS] 10% v/v, albumina sierica bovina 2% p/v [BSA] e 0,1% v/v Tween-20 nel tampone PBS) e incubare il tubo a RT per 1 ora con un leggero dondolio.
- Rimuovere il surnatante e aggiungere 200 μL di soluzione anticorpale primaria (diluita in tampone bloccante).
- Rimuovere il surnatante, aggiungere 500 μL di soluzione anticorpale primaria (diluita in tampone bloccante) e incubare il tubo a 4 °C durante la notte con un leggero oscillo.
NOTA: Se si utilizza un nuovo anticorpo primario, vale la pena includere alcuni campioni senza colorazione anticorpale primaria per fungere da controlli negativi. Tuttavia, idealmente, gli embrioni morpholino-knockdown o ingegnerizzati gene-knockout, o gli embrioni in cui è stata stimolata l'espressione della proteina bersaglio, sono mezzi più affidabili per convalidare l'anticorpo. - Rimuovere il surnatante, aggiungere 1 mL di tampone PDT e incubare il tubo a RT per 30 minuti con un leggero oscillo.
- Ripetere il passaggio 6.4.12.
- Rimuovere il surnatante, aggiungere 1 mL di tampone bloccante e incubare il tubo a RT per 1 ora con un leggero dondolio.
NOTA: I campioni devono essere protetti dalla luce dopo questo passaggio, per evitare il fotosbiancamento del fluoroforo coniugato sull'anticorpo secondario. - Rimuovere il surnatante e aggiungere 200 μL di soluzione anticorpale secondaria (diluita nel tampone bloccante).
- Rimuovere il surnatante, aggiungere 500 μL di soluzione anticorpale secondaria (diluita in tampone bloccante) e incubare il tubo a RT per 1,5 ore con un leggero oscillo.
- Rimuovere il surnatante, aggiungere 1 mL di tampone PDT e incubare il tubo a RT per 30 minuti con un leggero oscillo.
- Ripetere il punto 6.4.17.
- Montare gli embrioni su una piastra di agarosio al 2% (fatta con PBS, pH 7,6) e fotografare gli embrioni con uno stereomicroscopio (campo luminoso e rispettivi canali fluorescenti) (Figura 5A,B,D,F).
NOTA: Se si utilizza lo stereomicroscopio a fluorescenza Leica M165 FC, utilizzare un ingrandimento di 25x per ottenere immagini con una buona risoluzione. - Quantificare/analizzare l'intensità del segnale fluorescente mediante ImageJ (NIH). Utilizzare lo strumento Selezione a mano libera in ImageJ per quantificare il segnale nella regione di interesse.
Risultati
Live imaging di pesci reporter transgenici di neutrofili/macrofagi trattati con Z-REX, Tg(lyz:TagRFP) e Tg(mpeg1:EGFP). Induzione dell'apoptosi dei neutrofili/macrofagi mediante HNEilazione di Keap1. (Vedi anche Figura 2). L'effetto della marcatura elettrofila di Keap1 sui livelli di neutrofili e macrofagi è stato valutato iniettando embrioni transgenici eterozigoti derivati da Tg(lyz:TagRFP) o Tg(mpeg1:EGFP) con mRNA codificante Halo-Keap1, e quindi trattando con Ht-PreHNE(alchino). Seguendo le procedure per il saggio fase 6.1-downstream, l'opzione 1-HNE(alchino) è stata liberata ed è stato marcato Keap1. I livelli di neutrofili e macrofagi sono stati valutati mediante imaging dal vivo delle linee reporter, rispettivamente Tg(lyz:TagRFP) e Tg(mpeg1:eGFP). Il livello di entrambi i tipi di cellule è diminuito del 30%-40% dopo il trattamento con Z-REX, in cui HNE è stato somministrato a Keap1. Al contrario, nessuna perdita di neutrofili o macrofagi è stata osservata nei gruppi di controllo tecnico Z-REX [senza luce e Ht-PreHNE (alchino), luce da sola, o Ht-PreHNE (alchino) da solo] (Figura 1D e Figura 2A-D).
L'induzione dell'apoptosi dei neutrofili/macrofagi ha indicato il successo della somministrazione di HNE a Keap1 attraverso Z-REX. Sono stati pubblicati i dettagli per l'analisi del percorso e il meccanismo di apoptosi5. Per tenere conto degli effetti fuori bersaglio dell'HNE(alchino), sono stati utilizzati diversi controlli. (1) Nelle stesse condizioni sperimentali, invece dell'mRNA Halo-TeV-Keap1, gli embrioni sono stati iniettati con l'mRNA Halo-P2A-Keap1. Il linker P2A ha permesso alle proteine Halo e Keap1 di essere espresse in modo indipendente. In questo scenario, HNE(alchino) rilasciato da Halo non poteva etichettare Keap1, poiché non era più prossimale ad Halo (Figura 1D); Quindi, la via di segnalazione dell'apoptosi non è stata attivata. In questo gruppo non sono stati osservati cambiamenti nei livelli di macrofagi o neutrofili (Figura 2A,B). (2) Le stesse condizioni sperimentali sono state eseguite utilizzando mRNA codificante Halo-TeV-Keap1 (C151S, C273W, C288E), un mutante di Keap1 che non risponde a HNE (alchino) (Figura 1D). Non sono state osservate variazioni nei livelli di macrofagi o neutrofili (Figura 2G,H).
Accoppiamento a clic di biotina e saggio di pull-down della biotina. Valutazione dell'etichettatura del target. (Vedi anche Figura 3). La valutazione dell'etichettatura target è stata effettuata utilizzando embrioni WT, iniettati con mRNA codificante Halo-TeV-Keap1-2xHA (costrutto di fusione Halo-POI) o Halo-2xHA-P2A-Keap1-2xHA (costrutto diviso P2A, in cui Halo e Keap1 non sono fusi; Figura 1D). La proteina Keap1 marcata è stata abbassata solo nel gruppo che esprime la proteina di fusione e trattata con Z-REX (seconda corsia nella macchia anti-HA superiore), ma non in altri gruppi di controllo (nessuna iniezione di mRNA, costrutto di fusione senza Z-REX o costrutto P2A-split). I risultati indicano che l'HNE (alchino) è stato consegnato con successo a Keap1, e il Keap1 modificato è stato successivamente coniugato con biotina attraverso la reazione del clic, e il Keap1 marcato con biotina è stato tirato giù dalla resina streptavidina.
Analisi trascrizionale. RNA-seq e qRT-PCR. (Vedi anche Figura 4). La variazione trascrizionale dopo il trattamento con Z-REX è stata valutata mediante RNA-seq e qRT-PCR. Nell'RNA-seq, diversi geni immuno-correlati sono stati sottoregolati dopo Z-REX. Al contrario, molti geni correlati alla risposta antiossidante (AR) sono stati sovraregolati dopo Z-REX, che è risultato dall'induzione della via Keap1-Nrf2-AR su HNEilazione su Keap110 (Figura 4A). Nell'analisi qRT-PCR, risultati simili sono stati trovati analizzando tre geni immuno-correlati (lyz, mpeg1.1 e coro1a) (Figura 4B). La regolazione verso l'alto e verso il basso dei rispettivi geni ha mostrato l'induzione di successo di percorsi mediati dalla HNEilazione di Keap1.
Saggio di colorazione a (co-)immunofluorescenza a montaggio intero e analisi di colocalizzazione. (Cfr. anche figura 5). L'espressione esogena di Halo-TeV-Keap1-2xHA e Halo-2xHA-P2A-Keap1-2xHA è stata valutata mediante colorazione con immunofluorescenza (IF) a montaggio intero (Figura 5A,B). Il costrutto P2A-split-construct aveva due volte il numero di tag HA rispetto al TeV-fusion-construct, che corrisponde a un segnale anti-HA due volte più alto nel gruppo P2A-split-construct-mRNA-injected rispetto all'altro, indicando che il livello di espressione dei due costrutti era simile (Figura 5C). Anche i livelli di espressione di Halo-TeV-Keap1 (wt) e Halo-TeV-Keap1 (C151S,C273W,C288E) sono stati trovati simili durante il sondaggio con anti-Halo (Figura 5D,E). La colocalizzazione dei neutrofili e della caspasi attiva 3 in Tg(lyz:TagRFP) trattati con Z-REX è stata osservata mediante co-immunocolorazione con anti-RFP e anti-attiva-caspasi 3 (Figura 5F). La caspasi attiva 3 è un indicatore di eventi di apoptosi.
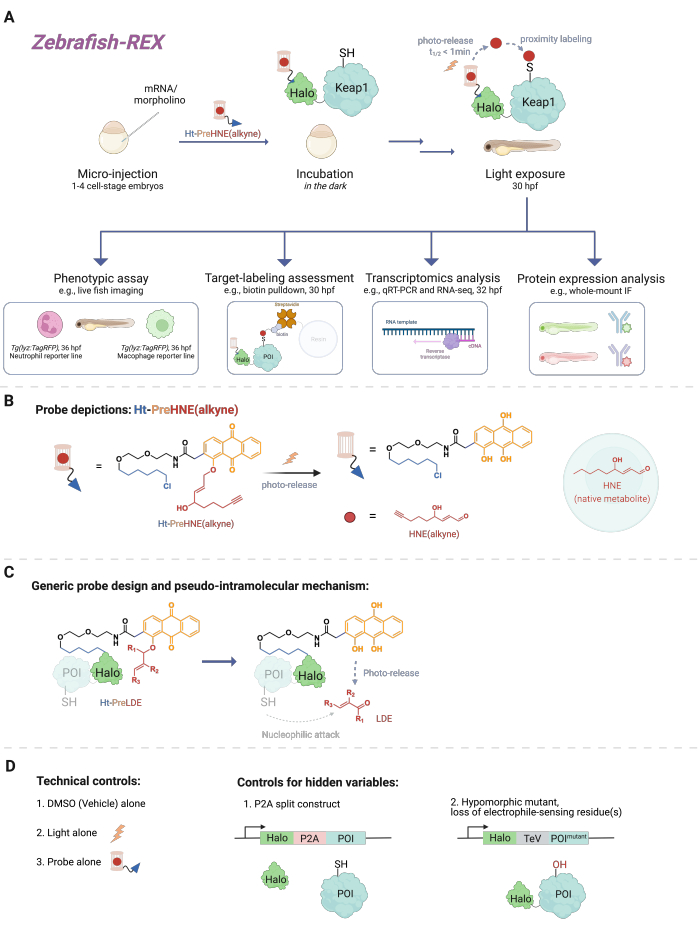
Figura 1: Flusso di lavoro Z-REX. (A,B) Un embrione di zebrafish allo stadio di 1-4 cellule viene iniettato con (morfolino e) mRNA codificante Halo-POI (ad esempio, Halo-Keap1). Gli embrioni iniettati vengono quindi trattati con una sonda composta da un ligando HaloTag e un elettrofilo fotocaged aggiunto con un gruppo funzionale alchino, come Ht-PreHNE (alchino) in B. Dopo aver rimosso la quantità eccessiva di sonda, l'embrione viene esposto alla luce per rilasciare l'elettrofilo di interesse [ad esempio, HNE o il suo analogo, HNE (alchino)]. L'analisi a valle viene eseguita in un determinato punto temporale definito dall'utente. (C) Progettazione e meccanismo della sonda Ht-PreLDE, applicabile a diversi elettrofili derivati dai lipidi (LDE). (D) Gruppi di controllo negativi/tecnici per Z-REX. Fare clic qui per visualizzare una versione ingrandita di questa figura.
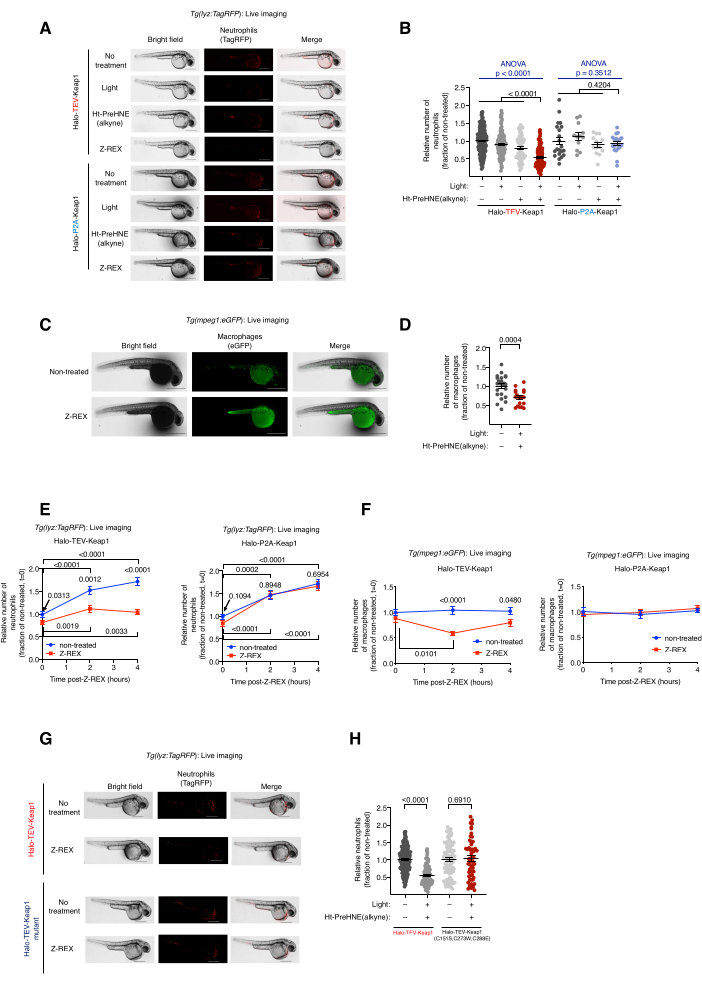
Figura 2: Live imaging di pesci reporter transgenici di neutrofili/macrofagi sottoposti a Z-REX. La HNEilazione Keap1 mediata da Z-REX induce l'apoptosi dei neutrofili/macrofagi. (A) Immagini rappresentative di pesci Tg(lyz:TagRFP ) che esprimono Halo-TeV-Keap1 (costrutto di fusione) o Halo-P2A-Keap1 (costrutto diviso) e sottoposti a condizioni di controllo negative [nessun trattamento, luce da sola, o Ht-PreHNE(alchino) da solo o Z-REX]. Età embrionale: 36 hpf. (B) Quantificazione dei livelli di neutrofili in A. (C) Immagini rappresentative di pesci Tg(mpeg1:eGFP) che esprimono Halo-TeV-Keap1 con o senza trattamento Z-REX. Età embrionale: 34 hpf. (D) Quantificazione dei livelli di macrofagi in C. (E,F) Misurazione nel tempo dei livelli di (E) neutrofili e (F) macrofagi dopo trattamento con Z-REX. (G) Esperimento simile a quello di A in pesci che esprimono Halo-TeV-Keap1 (WT) o Halo-TeV-Keap1 (C151S, C273W, C288E), un mutante che non ha capacità di rilevamento HNE. (H) Quantificazione dei livelli di neutrofili in G. Barre scala: 500 μm. Tutti i grafici sono presentati con valori medi ± SEM. p sono stati calcolati con ANOVA unidirezionale (blu) e test t di Student a due code (nero). Questa cifra è stata modificata da Poganik et al.7. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 3: Saggio di pull-down della biotina. Gli embrioni WT che esprimono Halo-TeV-Keap1-2XHA o Halo-2XHA-P2A-Keap1-2XHA sono stati trattati con Z-REX o rispettive condizioni di controllo negativo (nessun trattamento con sonda in questo caso). Dopo il prelievo, gli embrioni sono stati lisati e trattati con proteasi TeV prima del test di pull-down della biotina. I risultati sono stati analizzati mediante western blotting. Questa cifra è stata modificata da Huang et al. Z-REX: guida elettrofili reattivi a proteine specifiche espresse in modo specifico tissutale o ubiquitario e registrazione delle risultanti risposte redox funzionali indotte da elettrofili nei pesci larvali. Questa cifra è stata modificata da Huang et al.11. Fare clic qui per visualizzare una versione ingrandita di questa figura.
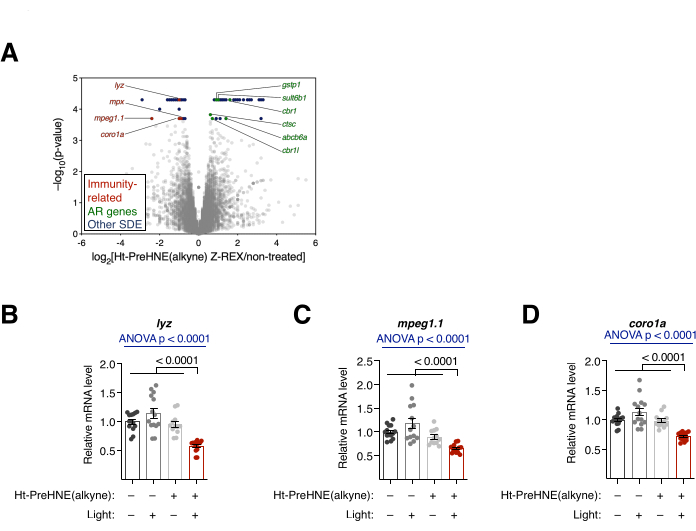
Figura 4: Analisi trascrizionale. (A) Risultati con RNA-seq di embrioni trattati con Z-REX rispetto a embrioni non trattati. Vengono evidenziati i geni statisticamente significativi differenzialmente espressi (SDE). I geni SDE correlati all'immunità sono colorati di rosso. I geni correlati alla risposta antiossidante (AR) sono colorati di verde. Altri geni SDE sono colorati di blu. Tutti i valori p sono stati calcolati con CuffDiff. (B-D) Tre geni SDE correlati all'immunità da A: (B) lyz, (C) mpeg1.1 e (D) coro1a sono stati ulteriormente analizzati con qRT-PCR e solo gli embrioni trattati con Z-REX hanno mostrato la soppressione di questi trascritti. Tutti i grafici sono presentati con valori medi ± SEM. p sono stati calcolati con ANOVA unidirezionale (blu) e test t di Student a due code (nero). Questa cifra è stata modificata da Poganik et al.7. Fare clic qui per visualizzare una versione ingrandita di questa figura.
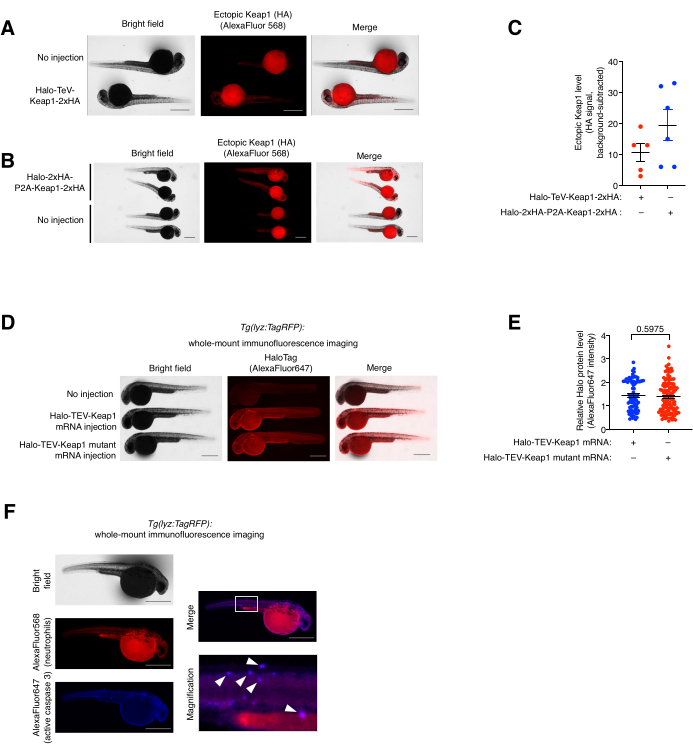
Figura 5: Saggio di colorazione con immunofluorescenza a montaggio intero . (A,B) Immagini rappresentative di embrioni che esprimono (A) Halo-TeV-Keap1-2xHA o (B) Halo-2xHA-P2A-Keap1-2xHA immunocolorato con anti-HA e anticorpo secondario coniugato con AlexaFluor568. I pesci iniettati con mRNA sono stati confrontati con pesci non iniettati di età corrispondente. (C) Quantificazione del segnale anti-HA in (A,B). (D) Immagini rappresentative di embrioni che esprimono Halo-TeV-Keap1 (WT) o Halo-TeV-Keap1 (C151S, C273W, C288E) immunocolorati con anti-alone e anticorpi secondari coniugati con AlexaFluor647. I pesci iniettati con mRNA sono stati confrontati con pesci non iniettati di età corrispondente. (E) Quantificazione del segnale anti-Halo in D. I valori p sono stati calcolati con il t-test di Student a due code. (F) Gli embrioni di Tg(lyz:TagRFP) sottoposti a Z-REX sono stati co-immunocolorati con anti-RFP e anti-attivo Caspase 3 e rispettivi anticorpi secondari coniugati con fluorofori. La casella bianca segna l'area ingrandita. Le frecce bianche indicano la colocalizzazione dei neutrofili e della caspasi attiva 3. Barre scala: 500 μm. Tutti i grafici sono presentati con media ± SEM. Questa cifra è stata modificata da Poganik et al.7. e Huang et al.11. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella supplementare 1: Elenco delle riserve utilizzate in questo studio. Clicca qui per scaricare questo file.
Discussione
Z-REX descritto in questo protocollo dimostra una solida strategia per lo studio della coppia elettrofilo-bersaglio e la deconvoluzione della via di segnalazione nei pesci vivi. La somministrazione diretta alla prossimità consente il dosaggio e il controllo spaziale del trattamento elettrofilo composto. A differenza dei metodi convenzionali di dosaggio in bolo, in cui le concentrazioni soprafisiologiche di elettrofilo dispiegate spesso portano a problemi fuori bersaglio, la quantità relativamente minore di elettrofilo rilasciata al sistema rende Z-REX in gran parte non invasivo. Abbiamo usato 0,1-6 μM Ht-PreHNE (alchini) in embrioni di zebrafish e i risultati hanno mostrato che il trattamento non è dannoso per lo sviluppo embrionale11.
La procedura Z-REX è generalmente più lunga di T-REX, una tecnica per lo screening/studio delle proteine elettrofile in cellule in coltura. Supponiamo che lo scopo dell'esperimento sia quello di schermare le interazioni elettrofilo-bersaglio; suggeriamo innanzitutto di eseguire uno screening approfondito mediante T-REX in cellule in coltura e di utilizzare Z-REX per la validazione in vivo e l'analisi fenotipica/pathway. Rispetto alla coltura cellulare, i requisiti per l'esecuzione di Z-REX sono tecniche di allevamento ittico di base oltre alle competenze sperimentali biochimiche richieste da T-REX. Un lasso di tempo tipico per Z-REX (dall'incrocio dei pesci alla consegna elettrofila inducibile dalla luce) è di 2-3 giorni, che non è più di 1 giorno in più rispetto al tempo tipico per un esperimento T-REX su cellule vive trasfettate. L'imaging dal vivo per l'analisi fenotipica può essere eseguito 2-10 ore dopo l'illuminazione della luce; l'accoppiamento a clic con biotina-azide per il test pull-down richiede 3 giorni; qRT-PCR per il dosaggio della risposta trascrizionale richiede 3 giorni; La colorazione IF richiede 5 giorni. Questi passaggi sono approssimativamente simili ai loro equivalenti di coltura cellulare, sebbene l'interpretazione dei dati richieda una comprensione della fisiologia dei pesci e dei ceppi reporter.
Come procedura a variabili multiple12, sono necessari diversi gruppi di controllo affinché Z-REX escluda incertezze nei risultati (Figura 1D). I gruppi di controllo comuni sono: (1) solo trattamento DMSO/veicolo; (2) trattamento della sonda, ma senza illuminazione luminosa; (3) illuminazione della luce, ma senza trattamento della sonda; (4) Costrutto P2A-split, in cui Halo e il POI sono espressi separatamente, quindi la consegna di prossimità viene ablata; e (5) mutanti ipomorfici, i cui residui elettrofili sono/sono mutati, come Akt3 (C119S)6 e Keap1 (C151S, C273W, C288E)5, che abbiamo usato in studi precedenti.
Se i saggi a valle comportano l'analisi western blot, la disidratazione deve essere eseguita prima del raccolto. Le proteine del tuorlo riducono la fedeltà delle valutazioni della concentrazione di lisato e possono legarsi in modo non specifico agli anticorpi. Durante l'esecuzione di immagini di pesci vivi o colorazione IF a montaggio intero, abbiamo anche osservato segnali fluorescenti non specifici nel sacco vitellino, probabilmente derivanti da proteine autofluorescenti nel sacco vitellino, o legame non specifico degli anticorpi stessi. Se il segnale di autofluorescenza interferisce con il segnale, suggeriamo di escludere il sacco vitellino dalla quantificazione o di quantificare separatamente le diverse regioni. La decorinazione è necessaria per l'imaging di pesci vivi e il test di colorazione IF a montaggio intero. Il corion può interferire con l'imaging e successivamente con la quantificazione / conteggio delle cellule. Tuttavia, la decorinazione è applicabile solo agli embrioni di età superiore a 1 dpf; Gli embrioni più giovani nelle fasi di blastulazione/gastrulazione/segmentazione sono troppo fragili per essere decorionati.
Il protocollo Z-REX qui descritto si basa sull'espressione di POI ectopici guidati da mRNA. La procedura è rapida rispetto all'utilizzo/generazione di lenze transgeniche. L'espressione guidata da mRNA è ubiquitaria e transitoria e dura almeno 2 giorni per gli mRNA utilizzati in questo protocollo. Tuttavia, è probabile che la durata dell'espressione vari in altri casi. Pertanto, questo approccio fornisce una finestra investigativa rapida e più globale sugli effetti di uno specifico evento di etichettatura elettrofila, compatibile con diversi saggi ad alto rendimento / alto contenuto. Le linee transgeniche con espressione stabile di Halo-POI in tessuti specifici sono compatibili anche con Z-REX11. Tali linee sono meglio utilizzate quando è necessario porre una domanda più precisa, ad esempio, quando un fenotipo in un organo specifico è previsto dai dati di coltura cellulare, o quando lo screening da esperimenti di iniezione di mRNA prevede che un organo specifico è sensibile a un evento di marcatura elettrofila. Un'induzione della risposta antiossidante specifica per il cuore attraverso Z-REX è stata dimostrata usando pesci Tg (gstp1: GFP; DsRed-P2A-myl7: Halo-TeV-Keap1) nella nostra precedente pubblicazione11. Potrebbe anche essere possibile eseguire Z-REX su pesci transgenici di età superiore a 2 dpf.
Divulgazioni
Gli inibitori delle chinasi a piccole molecole isoforma-specifici, la cui scoperta è stata resa possibile dalle tecnologie REX, sono stati depositati per la domanda di brevetto
Riconoscimenti
Finanziamenti: Novartis FreeNovation, NCCR e EPFL.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-Mercaptoethanol (BME) | Sigma-Aldrich | M6250 | |
| 1.5-mL microcentrifuge tube | Axygen | MCT-150-C-S | |
| 10-cm Petri dishes | Celltreat | 229692 | |
| 2-mL microcentrifuge tube | Axygen | MCT-200-C-S | |
| 30% Acrylamide and bis-acrylamide solution | BioRad | 1610154 | |
| 6-well plate | Celltreat | 229506 | |
| Acetone | Fisher | A/0600/15 | |
| Agarose | GoldBio | A201-100 | |
| All Blue Prestained Protein Standards | BioRad | 1610373 | |
| Ammonium persulfate | Sigma | A3678 | |
| Biotin-dPEG11-azide | Quanta Biodesign | 102856-142 | |
| Bradford Dye | BioRad | 5000205 | |
| BSA | Fisher | BP1600-100 | |
| Capillary tubes | VWR | HARV30-00200 | |
| Chloroform | Supelco, Inc | 1.02445.1000 | |
| cOmplete, Mini, EDTA-free Protease Inhibitor Cocktail | Roche | 11836170001 | |
| Cu(TBTA) | Lumiprobe | 21050 | |
| CuSO4 | Sigma | 209198 | |
| DMSO | Fisher | D128-500 | |
| Donkey anti-mouse-Alexa Fluor 647 | Abcam | ab150107 | 1:800 (IF) |
| Donkey anti-rabbit-Alexa Fluor 647 | Abcam | ab150075 | 1:1000 (IF) |
| Donkey anti-rat-AlexaFluor 568 | Abcam | ab175475 | 1:1000 (IF) |
| ECL substrate | Thermo Fisher Scientific | 32106 | |
| ECL-Plus substrate | Thermo Fisher Scientific | 32132 | |
| End-to-end rotator | FinePCR | Rotator AG | |
| Ethanol | Fisher | E/0650DF/15 | |
| Ethidium bromide | Sigma | E1510 | |
| Flaming/Brown Micropipette Puller | Sutter Instrument Co. | P-97 | |
| Fluorescence stereomicroscope | Leica | M165 FC | |
| Forceps (blunt) | self made | self made by blunting sharp forceps (Fine Science Tools, Dumont #5, 11252-40) | |
| Forceps (sharp) | Fine Science Tools | Dumont #5, 11252-40 | |
| Gel loading tip | Fisher | 02-707-181 | |
| Gel/blot imager | Vilber | Fusion FX imager | |
| Glass beads | Sigma | 45-G1145 | |
| Glass stage micrometer | Meiji Techno. | MA285 | |
| Heat inactivated FBS | Sigma | F2442 | |
| Heated ultrasonic bath | VWR | 89375-470 | |
| HEPES | Fisher | BP310-1 | |
| High capacity streptavidin agarose | Thermo Fisher Scientific | 20359 | |
| Ht-PreHNE alkyne probe | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Imaging plate (10% HBSS buffer, for live embryos) | Made with Petri dish, and 2% agarose in 10% HBSS buffer | ||
| Imaging plate (PBS, for formaldehyde-fixed embryos) | Made with Petri dish, and 2% agarose in PBS | ||
| Injection plate | Made with microinjection mold, Petri dish, and 2% agarose in 10% HBSS buffer | ||
| LDS | Apollo | APOBI3331 | |
| Methanol | VWR | 20864.32 | |
| Microinjection mold | Adaptive Science Tools | TU-1 | |
| Microloader tips | Eppendorf | 930001007 | |
| Micromanipulator | Narishige | MN-153 | |
| Microscope for micro-injection | Olympus | SZ61 | |
| Microscope light source | Olympus | KL 1600 LED | |
| Mineral oil | Sigma | M3516 | |
| mMessage mMachine SP6 Transcription Kit | Ambion | AM1340 | |
| Mouse anti- β-actin-HRP | Sigma | A3854 | 1:20000 (WB) |
| Mouse anti-HaloTag | Promega | G921A | 1:500 (IF) |
| Multi-mode reader | BioTek Instruments | Cytation 5 | |
| Nucleic acid agarose gel electrophoresis apparatus | Biorad | Mini-Sub Cell GT Systems | |
| Oligo(dT)20 | Integrated DNA Technologies | customized: (dT)20 | |
| Orange G | Sigma | O3756 | |
| Paraformaldehyde | Sigma | P6148 | |
| PBS | Gibco | 14190144 | |
| pCS2+8 Halo-2XHA-P2A-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| pCS2+8 Halo-TeV-Keap1-2xHA | self-cloned | - | Available from Prof. Yimon AYE's group at EPFL |
| Pneumatic PicoPump | WPI | SYS-PV820 | |
| Protein electrophoresis equipment and supplies | Biorad | Mini-PROTEAN Tetra Vertical Electrophoresis | |
| Rabbit anti-active Caspase-3 | BD Pharmingen | 559565 | 1:800 (IF) |
| Rat anti-HA-HRP | Sigma | H3663 | 1:500 (IF and WB) |
| Rat anti-RFP | ChromoTek | 5F8 | 1:800 (IF) |
| Refrigerated centrifuge | Eppendorf | 5417R | |
| RNAseOUT recombinant ribonuclease inhibitor | ThermoFisher Scientific | 10777019 | |
| RnaseZap RNA decontamination solution | ThermoFisher Scientific | AM9780 | |
| Rocking Shaker | DLAB | SK-R1807-S | |
| SDS | Teknova | S9974 | |
| SuperScript III reverse transcriptase | ThermoFisher Scientific | 18080085 | |
| t-Butanol | Sigma | 471712 | |
| TCEP-HCl | Gold Biotechnology | TCEP1 | |
| TeV protease (S219V) | self-made | - | Parvez, S. et al. T-REX on-demand redox targeting in live cells. Nat Protoc. 11 (12), 2328-2356, (2016). |
| Tg(lyz:TagRFP) | Zebrafish International Resource Center (ZIRC) | uwm11Tg (ZFIN) | - |
| Tg(mpeg1:eGFP) | Zebrafish International Resource Center (ZIRC) | gl22Tg (ZFIN) | - |
| Thermal cycler | Analytik Jana | 846-x-070-280 | |
| TMEDA | Sigma | T7024 | |
| Transfer pipets | Fisher | 13-711-9D | |
| Tris | Apollo | APOBI2888 | |
| Triton X-100 | Fisher | BP151-100 | |
| TRIzol reagent | Thermo Fisher Scientific | 15596018 | |
| Trypsin inhibitor from Glycine max (soybean) | Sigma | T9003 | |
| Tween 20 | Fisher | BP337-500 | |
| UV lamp with 365-nm light | Spectroline | ENF 240C | |
| UV meter | Spectroline | XS-365 | |
| Vortexer | Scientific Industries, Inc. | Vortex-Genie 2 | |
| Western Blotting Transfer equipment and supplies | Biorad | Mini Trans-Blot or Criterion Blotter | |
| Zebrafish husbandry and breeding equipment | in house | ||
| Zirconia beads | BioSpec | 11079107zx |
Riferimenti
- Long, M. J. C., Ly, P., Aye, Y. A primer on harnessing non-enzymatic post-translational modifications for drug design. RSC Medicinal Chemistry. 12 (11), 1797-1807 (2021).
- Parvez, S., Long, M. J. C., Poganik, J. R., Aye, Y. Redox signaling by reactive electrophiles and oxidants. Chemical Reviews. 118 (18), 8798-8888 (2018).
- Parvez, S., et al. T-REX on-demand redox targeting in live cells. Nature Protocols. 11 (12), 2328-2356 (2016).
- Long, M. J. C., et al. Precision electrophile tagging in Caenorhabditis elegans. Biochemistry. 57 (2), 216-220 (2018).
- Van Hall-Beauvais, A., Zhao, Y., Urul, D. A., Long, M. J. C., Aye, Y. Single-protein-specific redox targeting in live mammalian cells and C. elegans. Current Protocols in Chemical Biology. 10 (3), 43 (2018).
- Long, M. J. C., et al. Akt3 is a privileged first responder in isozyme-specific electrophile response. Nature Chemical Biology. 13 (3), 333-338 (2017).
- Poganik, J. R., et al. Wdr1 and cofilin are necessary mediators of immune-cell-specific apoptosis triggered by Tecfidera. Nature Communications. 12 (1), 5736 (2021).
- Lin, H. -. Y., Haegele, J. A., Disare, M. T., Lin, Q., Aye, Y. A generalizable platform for interrogating target- and signal-specific consequences of electrophilic modifications in redox-dependent cell signaling. Journal of the American Chemical Society. 137 (19), 6232-6244 (2015).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative CT method. Nature Protocols. 3 (6), 1101-1108 (2008).
- Wakabayashi, N., et al. Keap1-null mutation leads to postnatal lethality due to constitutive Nrf2 activation. Nature Genetics. 35 (3), 238-245 (2003).
- Huang, K. -. T., et al. Z-REX: Shepherding reactive electrophiles to specific proteins expressed either tissue-specifically or ubiquitously, and recording the resultant functional electrophile-induced redox responses in larval fish. Nature Protocols. , (2023).
- Long, M. J. C., Assari, M., Aye, Y. Hiding in plain sight: The issue of hidden variables. ACS Chemical Biology. 17 (6), 1285-1292 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon