Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Identificación y análisis de progenitores miogénicos in vivo durante la lesión aguda del músculo esquelético mediante citometría de masa unicelular de alta dimensión
En este artículo
Resumen
El protocolo aquí presentado permite la identificación y el análisis de alta dimensión de células madre y progenitoras musculares mediante citometría de masas unicelulares y su purificación por FACS para estudios en profundidad de su función. Este enfoque se puede aplicar para estudiar la dinámica de regeneración en modelos de enfermedad y probar la eficacia de las intervenciones farmacológicas.
Resumen
La regeneración del músculo esquelético es un proceso dinámico impulsado por las células madre musculares adultas y su progenie. En su mayoría inactivas en un estado estacionario, las células madre musculares adultas se activan tras una lesión muscular. Después de la activación, proliferan, y la mayoría de su progenie se diferencia para generar células musculares competentes para la fusión, mientras que el resto se renueva para reponer la reserva de células madre. Si bien la identidad de las células madre musculares se definió hace más de una década, sobre la base de la coexpresión de marcadores de superficie celular, los progenitores miogénicos se identificaron solo recientemente utilizando enfoques unicelulares de alta dimensión. Aquí, presentamos un método de citometría de masa unicelular (citometría por tiempo de vuelo [CyTOF]) para analizar células madre y células progenitoras en lesiones musculares agudas para resolver la dinámica celular y molecular que se despliega durante la regeneración muscular. Este enfoque se basa en la detección simultánea de nuevos marcadores de superficie celular y factores de transcripción miogénicos clave cuya expresión dinámica permite la identificación de células madre activadas y poblaciones de células progenitoras que representan hitos de miogénesis. Es importante destacar que se describe una estrategia de clasificación basada en la detección de los marcadores de superficie celular CD9 y CD104, que permite el aislamiento prospectivo de células madre y progenitoras musculares mediante clasificación celular activada por fluorescencia (FACS) para estudios en profundidad de su función. Las células progenitoras musculares proporcionan un eslabón perdido crítico para estudiar el control del destino de las células madre musculares, identificar nuevos objetivos terapéuticos para las enfermedades musculares y desarrollar aplicaciones de terapia celular para la medicina regenerativa. El enfoque presentado aquí se puede aplicar para estudiar las células madre y progenitoras musculares in vivo en respuesta a perturbaciones, como las intervenciones farmacológicas dirigidas a vías de señalización específicas. También se puede utilizar para investigar la dinámica de las células madre y progenitoras musculares en modelos animales de enfermedades musculares, avanzando en nuestra comprensión de las enfermedades de las células madre y acelerando el desarrollo de terapias.
Introducción
El músculo esquelético constituye el tejido más grande por masa en el cuerpo y regula múltiples funciones, desde la vista hasta la respiración, desde la postura hasta el movimiento, así como el metabolismo1. Por lo tanto, mantener la integridad y la función del músculo esquelético es fundamental para la salud. El tejido muscular esquelético, que consiste en haces apretados de miofibras multinucleadas rodeadas por una compleja red de nervios y vasos sanguíneos, exhibe un notable potencial regenerativo 1,2.
Los principales impulsores de la regeneración del músculo esquelético son las células madre musculares adultas (MuSC). También conocidas como células satélite, debido a su ubicación anatómica única adyacente a la membrana plasmática de la miofibra y debajo de la lámina basal, se identificaron por primera vez en 19613. Las MuSC expresan un marcador molecular único, el factor de transcripción emparejado box 7 (Pax7)4. En su mayoría inactivos en adultos sanos, se activan con una lesión muscular y proliferan para dar lugar a una progenie que (i) se diferenciará en células musculares competentes para la fusión que formarán nuevas miofibras para reparar el daño muscular o (ii) se autorrenovarán para reponer el grupo de células madre5.
A nivel celular y molecular, el proceso de regeneración es bastante dinámico e involucra transiciones de estado celular, caracterizadas por la expresión coordinada de factores de transcripción miogénicos clave, también conocidos como factores reguladores miogénicos (MRF)6,7. Los estudios previos de desarrollo in vivo, los experimentos de rastreo de linaje y el trabajo de cultivo celular con mioblastos han demostrado que la expresión secuencial de estos factores de transcripción impulsa la miogénesis, con el factor miogénico 5 (Myf5) expresándose al activarse, la expresión de diferenciación miogénica 1 (MyoD1) marcando el compromiso con el programa miogénico y la expresión de miogenina (MyoG) marcando la diferenciación 8,9,10,11, 12,13,14. A pesar de este conocimiento y del descubrimiento de marcadores de superficie celular para purificar MuSCs, han faltado estrategias y herramientas para identificar y aislar poblaciones discretas a lo largo de la ruta de diferenciación miogénica y resolver una progresión miogénica in vivo 15,16,17,18.
En este trabajo presentamos un método novedoso, basado en investigaciones recientemente publicadas, que permite la identificación de células madre y progenitoras en el músculo esquelético y el análisis de su dinámica celular, molecular y de proliferación en el contexto de la lesión muscular aguda19. Este enfoque se basa en la citometría de masas de una sola célula (también conocida como citometría por tiempo de vuelo [CyTOF]) para detectar simultáneamente marcadores clave de la superficie celular (α7 integrina, CD9, CD44, CD98 y CD104), factores de transcripción miogénicos intracelulares (Pax7, Myf5, MyoD y MyoG) y un análogo de nucleósido (5-Iodo-2'-desoxiuridina, IdU), para monitorear células en faseS 19,20, 21,22,23. Además, el protocolo presenta una estrategia basada en la detección de dos marcadores de superficie celular, CD9 y CD104, para purificar estas poblaciones celulares mediante clasificación celular activada por fluorescencia (FACS), lo que permite futuros estudios en profundidad de su función en el contexto de lesiones y enfermedades musculares. Si bien los mioblastos primarios se han utilizado ampliamente en el pasado para estudiar las etapas tardías de la diferenciación miogénica in vitro, no se sabe si recapitulan el estado molecular de las células progenitoras musculares encontradas in vivo 24,25,26,27,28,29,30 . La producción de mioblastos es laboriosa y requiere mucho tiempo, y el estado molecular de este cultivo primario cambia rápidamente al pasar31. Por lo tanto, los progenitores miogénicos recién aislados purificados con este método proporcionarán un sistema más fisiológico para estudiar la miogénesis y el efecto de las manipulaciones genéticas o farmacológicas ex vivo.
El protocolo presentado aquí se puede aplicar para abordar una variedad de preguntas de investigación, por ejemplo, para estudiar la dinámica del compartimento miogénico in vivo en modelos animales de enfermedades musculares, en respuesta a manipulaciones genéticas agudas o en intervenciones farmacológicas, profundizando así nuestra comprensión de la disfunción de las células madre musculares en diferentes contextos biológicos y facilitando el desarrollo de nuevas intervenciones terapéuticas.
Protocolo
Los procedimientos con animales fueron aprobados por la inspección danesa de experimentos con animales (protocolo # 2022-15-0201-01293), y los experimentos se realizaron de acuerdo con las directrices institucionales de la Universidad de Aarhus. Se proporciona analgesia (buprenorfina) en el agua potable 24 horas antes de la lesión para que los ratones se adapten al sabor. El suministro de buprenorfina en el agua potable se continúa durante 24 horas después de la lesión. Junto con una inyección subcutánea (s.c.) de buprenorfina en el momento de la lesión muscular aguda, la buprenorfina en el agua potable después de la inyección de notexina aliviará el dolor asociado con la lesión. Si bien se recomienda administrar una inyección s.c. de buprenorfina en el momento de la lesión muscular aguda, seguida de buprenorfina en el agua potable, la buprenorfina en el agua potable antes de la lesión es opcional. Sin embargo, los investigadores deben seguir las normas y directrices de bienestar animal establecidas por la agencia reguladora correspondiente.
NOTA: Para los experimentos de citometría de masa unicelular (CyTOF) de músculos lesionados de las extremidades posteriores, comience en la sección 1: Analgesia en agua 24 h antes de la lesión muscular hasta 24 h después de la lesión. Para clasificar las células madre y progenitoras musculares de ratones no lesionados, realice las secciones 5 y 6: Eutanasia + Disección y disociación del músculo esquelético, y continúe con la sección 11: Tinción con anticuerpos conjugados con fluoróforos para FACS. En la Figura 1 se muestra una descripción general de la configuración experimental y el protocolo.
1. Analgesia en agua desde 24 h antes de la lesión muscular hasta 24 h después de la lesión
- A una botella oscura o envuelta en papel de aluminio, agregue 3 mL de buprenorfina (0.3 mg/mL) y llene hasta 100 mL con agua filtrada para alcanzar una concentración final de 0.009 mg/mL 24 h antes de la lesión. Conéctalo a la jaula del ratón.
- Retire la botella 24 horas después de la lesión y vuelva a conectar la jaula del ratón al sistema de válvulas para beber.
2. Preparación para el procedimiento de lesión aguda
NOTA: Use etanol al 70% para desinfectar el banco de trabajo, la configuración del cono de la nariz y la caja de inducción.
- Prepare un tubo de 1,5 mL con notexina diluida en PBS (5 μg/mL). Diluya buprenorfina (0,3 mg/mL) en solución salina estéril al 0,9% a 0,015 mg/mL en un tubo de 1,5 mL seguro para la luz. Mantener en hielo. Prepare jeringas de insulina para inyecciones de buprenorfina (28 G, 0,5 mL) y notexina (29 G, 0,3 mL).
- Configuración de las unidades de anestésico a base de isoflurano:
- Para la anestesia en una caja de inducción, utilice isoflurano al 3% con un caudal de 1,5 L/min (50% O2, 50% aire atmosférico).
- Para el mantenimiento utilizando una configuración de cono de nariz, use isoflurano al 1,5% con un flujo de 0,6 L/min (50% O2, 50% aire atmosférico).
3. Lesión aguda por inyección de notexina
PRECAUCIÓN: Notexin tiene actividad de fosfolipasa A2 y es el componente principal del veneno de la serpiente tigre australiana (Notechis scutatus), con una LDintravenosa 50 de 5-17 mg de notexina/kg en ratones32,33. En el presente protocolo, el músculo tibial anterior (TA) de cada miembro posterior se inyecta con 10 μL de 5 mg/mL de notexina, y el músculo gastrocnemio (GA) de cada miembro posterior se inyecta dos veces (una en cada cabeza del músculo) con 15 μL de 5 mg/mL de notexina. Es importante realizar correctamente las inyecciones intramusculares (i.m.) para limitar el daño e inspeccionar con frecuencia los animales inyectados para garantizar un dolor mínimo.
- Anestesiar al ratón en la caja de inducción (isoflurano al 3%). Cuando el ratón esté inconsciente, baje el nivel de isoflurano al 1,5% en la caja de inducción.
- Pesar y marcar individualmente a los ratones de acuerdo con los procedimientos aprobados por la institución. Debajo de la configuración del cono de la nariz, afeite las extremidades traseras con una recortadora y transfiéralas nuevamente a la caja de inducción.
- Calcule el volumen de buprenorfina para cada ratón:
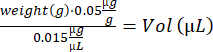
- Mezcle la solución de notexina pipeteando hacia arriba y hacia abajo. Prepare 2 jeringas de insulina (29 G, 0,3 mL) cargadas con 10 μL de solución de notexina (5 μg/mL) para inyecciones de TA, y 4 jeringas de insulina cargadas con 15 μL de solución de notexina (5 μg/mL) para inyección de GA.
NOTA: El músculo tibial anterior (TA) se encuentra en la parte anterior de la parte inferior de la pierna del ratón, extendiéndose desde la rodilla hasta el tobillo. El músculo gastrocnemio (GA) es un músculo bicéfalo que se encuentra en la parte posterior de la parte inferior de la pierna, superficial al sóleo. Va desde sus dos cabezas (medial y lateral) justo por encima de la rodilla hasta el talón, extendiéndose a través de un total de tres articulaciones (rodilla, tobillo y articulaciones subastragalina). - Realizar inyecciones intramusculares
- Inyección de TA (2 inyecciones en total)
- Coloque el ratón en posición supina debajo de la configuración del cono de la nariz y desinfecte el lugar de la inyección con toallitas estériles con alcohol (etanol al 70% o isopropanol). Inserte la aguja, con el bisel hacia abajo, en el vientre del AT (distal a la mitad del vientre) en un ángulo de 30°. Avance la aguja a lo largo del músculo, moviéndose paralelamente a la tibia, para llegar a la mitad del vientre del TA. Inyecte la notexina lenta y continuamente, dejando la aguja en su lugar durante 10 segundos antes de retirarla. No inserte la aguja demasiado profundamente inicialmente (para evitar inyectar notexina por debajo del TA en el músculo extensor largo de los dedos), y no demasiado proximalmente al avanzar la aguja (para evitar inyectar demasiado cerca de la rodilla).
- Inyección de GA (4 inyecciones en total)
- Inserte la aguja, biselada hacia abajo, en un ángulo de ~45° en la mitad del abdomen de la cabeza lateral del AG. Inyecte la notexina lenta y continuamente, dejando la aguja en su lugar durante 10 segundos antes de retirarla. Gire la extremidad posterior e inyecte notexina en la cabeza medial del músculo GA como se indicó anteriormente. Se debe tener cuidado de no insertar la aguja demasiado profundamente.
- Inyección de TA (2 inyecciones en total)
- Dé la vuelta al ratón e inyecte buprenorfina s.c. con una jeringa de insulina (28 G, 0,5 ml). Transfiera el mouse a una jaula de recuperación vacía en una placa calefactora. Solo la mitad del fondo de la jaula debe colocarse sobre la placa calefactora para permitir que el ratón se termorregule durante la recuperación34. Repita los pasos 3.3 a 3.6 para los ratones restantes.
- Cuando el ratón de la jaula de recuperación esté completamente despierto, devuélvalo a la jaula original. Devuélvelo al establo y compleméntalo con comida mojada. Retire el frasco de buprenorfina 24 h después de la inyección.
- Monitoree a los ratones durante 6 h después de la inyección de notexina y luego cada 12 h durante 2 días para detectar signos de dolor, movilidad deteriorada y disminución del consumo de alimentos35,36.
4. Inyección de 5-Iodo-2'-desoxiuridina
PRECAUCIÓN: Se sospecha que la 5-yodo-2'-desoxiuridina (IdU) causa defectos genéticos y daña la fertilidad del feto. Lea la hoja de datos de seguridad (SDS) antes de manipularlo. Se debe usar equipo de protección personal durante la manipulación. Utilice una campana extractora cuando pese el polvo IdU. Los materiales que hayan estado en contacto con IdU deben desecharse de acuerdo con las normas de seguridad locales.
NOTA: El marcaje de IdU in vivo se utiliza para controlar la división celular durante el curso del tiempo de lesión porque IdU, un análogo de la timidina yodada, se incorpora al ADN de las células en la fase S. El IdU se inyecta por vía intraperitoneal (i.p.) a 20 mg/kg de peso corporal 8 h antes del sacrificio del ratón.
- Utilice un sonicador a 37 °C para preparar una solución madre de 2 mg/ml de IdU en PBS estéril. IdU es sensible a la luz; úselo el mismo día o congele a -20 °C durante un máximo de 3 meses. Si se utiliza IdU congelado: descongelar, vórtice, centrifugar a 10.000 x g durante 30 s y utilizar el sobrenadante para las inyecciones a continuación.
- Anestesiar al ratón en la caja de inducción (isoflurano al 3%). Tenga en cuenta la asignación de fondos, pese los ratones y calcule el volumen de la solución IdU:
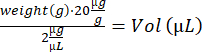
- Realice la inyección i.p. con una jeringa de insulina (28 G, 0,5 mL) y transfiera el ratón a una jaula de recuperación vacía en una placa calefactora. Cuando esté despierto, mueva el ratón de vuelta a su jaula original. Repita los pasos 4.2 a 4.3 para los ratones restantes y devuélvalos al establo.
5. Eutanasia
NOTA: Consulte la Tabla 1 para ver las recetas de tampón. Prepare los medios de lavado (mezcla de nutrientes F-10 (Jamón), 10% de suero de caballo, 1x Pen/Strep) y filtre a través de una membrana de polietersulfona (PES) en un recipiente de poliestireno. Prepare un tampón de disociación (medios de lavado suplementados con 650 U/mL de colagenasa, tipo II) y manténgalo en hielo. Las mediciones de citometría de masas CyTOF son muy sensibles a los contaminantes. Por esta razón, es esencial utilizar reactivos del más alto grado analítico para el procesamiento de muestras. Para evitar la contaminación por metales, se recomienda encarecidamente utilizar utensilios de plástico estériles y cristalería nueva que nunca se haya lavado con detergente porque muchos jabones de laboratorio contienen altos niveles de bario. Se recomienda utilizar agua desionizada, destilada y doble filtrada para la preparación de reactivos. La solución salina tamponada con fosfato (PBS) se prepara internamente. Diluya las 10x culatas a 1x y filtre las 1x PBS con filtros de 0,2 μm. Vuelva a filtrar el 1x PBS al comienzo de cada experimento. Las herramientas de disección no deben limpiarse con detergente debido a la presencia de bario.
- Prepare la báscula de pesaje, el recipiente y la unidad de anestesia a base de isoflurano con la caja de inducción. Desinfecte las herramientas de disección adecuadas.
- Prepare y marque tubos de 50 ml para cada ratón con 5 ml de tampón de disociación y una placa de Petri. Mantenga el tampón de disociación en hielo.
- Anestesiar al ratón en la caja de inducción (3% de isoflurano, aumentar al 5% cuando está inconsciente) y realizar la luxación cervical.
6. Disección y disociación del músculo esquelético
- Disecciona los músculos TA y GA de ambas extremidades traseras y transfiérelos a la tapa de una placa de Petri. Use unas tijeras para cortar el pañuelo en una lechada picada (~ 1 mm3 piezas). Transfiera a un tubo de 50 mL que contenga 5 mL de tampón de disociación helado. Mantener en hielo.
- Repita el procedimiento de eutanasia (paso 5.3) y el procedimiento de disección y disociación del músculo esquelético (paso 6.1) hasta que se hayan procesado todos los ratones.
- Precalentar todos los tubos de muestra en un baño de agua a 37 °C (2 min) e incubar durante 45 min en rotación en una incubadora (37 °C).
- Vértice y lave añadiendo 10 ml de medios de lavado. Centrífuga (380 x g, 10 min, temperatura ambiente [RT]) y aspire hasta 4 mL.
- Añadir 0,5 mL de colagenasa (1000 U/mL) y 0,5 mL de dispasa (11 U/mL). Agite e incube durante 20 minutos en rotación a 37 °C (por ejemplo, en una incubadora). Centrifugar (380 x g, 1 min, RT) y resuspender con una pipeta de 5 mL.
- Agregue filtros de celdas de 40 μm a tubos de 50 ml y humedezca previamente con 5 ml de medio de lavado. Aspire y expulse la suspensión celular 10 veces con una jeringa de 5 ml con una aguja de 20 g. Colar a través del filtro de células de 40 μm previamente humedecido. Lave el tubo de 50 mL con medios de lavado de 10 mL, transfiéralo al filtro de celdas de 40 μm y centrifugue (380 x g, 10 min, 4 °C) la suspensión de celdas.
7. Tinción de vivos/muertos con fijación de cisplatino y paraformaldehído
PRECAUCIÓN: El cisplatino y el paraformaldehído (PFA) son cancerígenos. Lea la SDS antes de manipularla. El paraformaldehído (PFA; 16%) es un irritante de la piel, los ojos y las vías respiratorias. Use equipo de protección personal y manipule estas sustancias debajo de una campana extractora. Durante la fijación de las células, la concentración final de PFA será del 1,6%. Se deben tomar las medidas de protección correctas y los desechos deben manejarse de acuerdo con las regulaciones locales.
NOTA: Prepare DMEM sin suero frío (4 °C) y tibio (37 °C). Prepare DMEM suplementado con un 10% de FBS, filtre a través de una membrana de PES en un recipiente de poliestireno y manténgalo en hielo. Prepare PBS y medios de tinción celular (CSM; PBS, 0,5% BSA, 0,02% azida de sodio) en un frasco de vidrio dedicado a CyTOF y filtra a través de una membrana de PES. El CSM puede almacenarse a 4 °C durante un máximo de 6 meses.
- Aspire el sobrenadante, agite el pellet y vuelva a suspender en 1 ml de DMEM frío sin suero. Cuente las células y ajuste la densidad celular a 1 x 106 células/mL en DMEM sin suero.
- Añadir caldo de cisplatino (25 mM) hasta una concentración final de 25 μM. Vortex 10 s e incubar durante exactamente 1 min (la reacción es muy sensible al tiempo). Apague la reacción con DMEM+10% FBS helado (3 veces el volumen de la muestra) y manténgalo en hielo. Centrífuga (380 x g, 10 min, 4 °C), aspirar el sobrenadante y resuspender el pellet a fondo (hasta 10 x 106 células/mL en CSM). Filtre la suspensión a través de un filtro de celdas de 35 μm.
- Fije la suspensión celular con una solución madre de PFA filtrada (16%) y pipetee hacia arriba y hacia abajo (realice bajo una campana extractora) para alcanzar la concentración final de PFA (1,6%) (p. ej., agregue 100 μL de PFA al 16% a 900 μL de suspensión celular). Vórtice durante 30 s, incubar durante 10 min en hielo y lavar dos veces con 2 mL de CSM por centrifugación (800 x g, 5 min, RT).
NOTA: En este punto, las muestras pueden congelarse rápidamente en hielo seco y almacenarse a -80 °C o usarse directamente para la tinción de anticuerpos. Si las células se van a congelar, transfiéralas a tubos de polipropileno de 5 ml, ya que el poliestireno puede agrietarse a bajas temperaturas. Si se utilizan células recién fijadas para la tinción, continúe con el paso 8.2.
8. Tinción con anticuerpos conjugados con metales
PRECAUCIÓN: El metanol (MeOH) es altamente inflamable y corrosivo para las vías respiratorias. Lea la SDS antes de manipularla. Use equipo de protección personal y manipule esta sustancia debajo de una campana extractora. Manejar los residuos de acuerdo con las regulaciones locales.
NOTA: La lista de anticuerpos (Ab) dirigidos a marcadores de superficie y marcadores intracelulares se puede encontrar en la Tabla 2.
Conjugación de anticuerpos: La mayoría de los anticuerpos utilizados en este protocolo se conjugaron internamente porque no estaban disponibles comercialmente. Los protocolos para la conjugación de metales de anticuerpos han sido publicados previamente, y los kits de conjugación ya están disponibles comercialmente37,38. La inmunoglobulina tipo G (IgG) es compatible con los protocolos de conjugación disponibles. Es de gran importancia que la formulación de anticuerpos utilizada para la conjugación de metales esté libre de proteínas transportadoras que contengan cisteína (por ejemplo, albúmina sérica bovina (BSA)), que pueden afectar la eficiencia de la conjugación al competir por los grupos de maleimida libres del polímero y pueden interferir con la cuantificación del anticuerpo conjugado con metales. El contenido de cisteína de la gelatina es mucho menor que el de la BSA. Sin embargo, se recomienda que si la formulación del anticuerpo contiene proteínas transportadoras, dichas proteínas se eliminen antes de la conjugación. Ahora es posible solicitar al fabricante anticuerpos sin BSA ni gelatina. Los conservantes de moléculas pequeñas (por ejemplo, azida de sodio, glicerol y trehalosa) son compatibles con los protocolos de conjugación de metales 37,38.
Titulación de anticuerpos: Después de cada conjugación de metales, los anticuerpos deben valorarse para determinar la concentración óptima de anticuerpos que proporciona la máxima relación señal-ruido. Para la valoración de anticuerpos, se realiza una dilución en serie doble en 6 pasos y se tiñen las muestras que se sabe que expresan (por ejemplo, células musculares, controles positivos) y carecen (controles negativos) de la proteína de interés 19,21,37,38.
Prepare una solución de trabajo fresca Cell-ID Intercalator-Ir (stock = 500 μM; solución intercaladora-ir) diluyendo el stock a 0,1 μM en PBS/1,6% PFA.
- Descongele las muestras congeladas durante 5 min en RT y centrifugue (800 x g, 1 min, RT). Lavar con 2 mL de CSM por centrifugación (800 x g, 5 min, RT).
- Prepare una mezcla de tinción de anticuerpos de superficie (Ab) 2,5x en CSM. Aspire el sobrenadante a ~60 μL y vuelva a suspender el pellet a fondo. Teñir las células añadiendo 40 μL de mezcla de tinción Ab superficial e incubarla durante 1 h a RT.
- Pasa las muestras cada 20 minutos para mezclarlas. Lavar dos veces con 1 mL de CSM por centrifugación (800 x g, 5 min, RT). Aspire el sobrenadante y agite la bolita.
NOTA: Si no se necesita tinción intracelular, no se requiere permeabilización de metanol. Sin embargo, si solo se realiza la tinción superficial, la solución intercaladora-ir debe diluirse en un tampón que contenga un agente permeable (por ejemplo, Maxpar fix y perm buffer) para penetrar en la membrana nuclear. Continúe con el paso 8.5 y diluya el Cell-ID Intercalator-Ir en un tampón que contenga un agente permeable y un 1,6% de PFA. De lo contrario, continúe con la tinción intracelular a continuación.
PRECAUCIÓN: El iridio es peligroso y se deben tomar precauciones para un manejo seguro. El iridio es inflamable y es irritante para los ojos y la piel. Evite crear y respirar polvo o vapores. Evite el contacto con la piel y los ojos. Sin embargo, la mezcla de solución intercaladora-ir se proporciona a una concentración del <1% en agua, que se considera no peligrosa según el sistema globalmente armonizado (GHS) de clasificación y etiquetado de productos químicos. Lea la SDS de Standard Biotools antes de manipularlo. Se debe usar equipo de protección personal durante la manipulación. Los materiales que hayan estado en contacto con la solución intercaladora-ir deben desecharse de acuerdo con las normas de seguridad locales. - Para permeabilizar las células, agregue 0,5 mL de MeOH helado gota a gota mientras se agita el vórtice. Incubar durante 15 minutos en hielo bajo una campana extractora. Centrifugar (800 x g, 5 min, RT) y luego lavar dos veces con 1 mL de CSM por centrifugación (800 x g, 5 min, RT). Después del último lavado, aspire el sobrenadante a ~60 μL y vuelva a suspender bien el pellet.
- Prepare una mezcla de tinción de Ab intracelular 2,5x en CSM. Teñir las células añadiendo 40 μL de mezcla de tinción Ab intracelular e incubar durante 1 h en RT. Frotar las muestras cada 20 min para mezclar.
- Lavar dos veces con 1 mL de CSM por centrifugación (800 x g, 5 min, RT). Aspire el sobrenadante y agite la bolita. Vuelva a suspender las muestras en 0,5 mL de solución intercaladora-iridio (Tabla 1) y vórtice. Incubar las muestras durante 1 h a RT o durante la noche (O/N) a 4 °C (ver nota abajo).
NOTA: Las muestras pueden almacenarse en una solución intercaladora de iridio a 4 °C durante un máximo de 48 h. El protocolo de tinción utilizado en este estudio se ha desarrollado en base al trabajo pionero del laboratorio Nolan{C}23. Difiere de los protocolos estándar de Biotools, ya que (i) se utilizan tampones preparados internamente para el lavado y la tinción, (ii) las células se fijan antes de la tinción superficial y (iii) las células se permeabilizan con metanol para la tinción intracelular con anticuerpos contra factores de transcripción o moléculas de señalización. Los investigadores que desarrollen un nuevo protocolo deben probar exhaustivamente la compatibilidad de su panel de anticuerpos con el metanol como agente de permeabilización. Al agregar nuevos anticuerpos a un panel, se recomienda probar la especificidad de los anticuerpos mediante citometría de flujo utilizando muestras de control positivas y negativas antes de la conjugación y valoración de metales.
9. Preparación de la muestra para la carga en el citómetro de masa
NOTA: Los gránulos de celda están muy sueltos cuando están en el tampón CAS (Tabla de materiales). Durante los lavados con tampón CAS, no aspire hasta la sequedad. En su lugar, mantenga un volumen residual como se describe a continuación.
- Vórtice y centrífuga (800 x g, 10 min, RT) las muestras. Vierta el sobrenadante (manéjelo como residuo de PFA) y el vórtice.
- Lavar con 1 mL de CSM por centrifugación (800 x g, 10 min, RT). Aspire el sobrenadante y agite la bolita.
- Lavar con 1 mL de tampón CAS por centrifugación (800 x g, 10 min, RT). Aspire a ~200 μL. Vortex y agregue 1 mL de tampón CAS. Saque 5 μL de alícuota para el recuento de células. Centrifugar (800 x g, 10 min, RT) y aspirar cuidadosamente hasta ≤50 μL. No moleste el pellet.
- Vuelva a suspender el pellet a 1–2 x 106 células/ml en tampón CAS y agregue perlas de calibración (1x; Tabla de Materiales) hasta una concentración final de 0,1x (por ejemplo, añadir 100 μL de perlas de calibración 1x a 900 μL de suspensión celular).
- Cargue la muestra en el citómetro de masas y recopile datos utilizando un caudal de 400-500 células/s.
- Realizar la normalización de datos de archivos FCS utilizando el software CyTOF o herramientas de normalización desarrolladas previamente39.
NOTA: El funcionamiento de los citómetros de masa CyTOF es específico del instrumento40,41. Se recomienda consultar el manual del usuario de CyTOF antes de la operación. Las perlas de calibración son gránulos de normalización de poliestireno incrustados en metal que contienen concentraciones conocidas de los isótopos metálicos cerio (140/142Ce), europio (151/153Eu), holmio (165Ho) y lutecio (175/176Lu). Las perlas de calibración permiten controlar la sensibilidad de la máquina, que puede variar con el tiempo, principalmente debido a la acumulación de material biológico y a las variaciones en la ionización del plasma a lo largo del tiempo.
10. Análisis de datos CyTOF
NOTA: Para el análisis posterior, los archivos FCS normalizados se pueden analizar localmente o cargar en soluciones de software basadas en la nube como Cytobank, Cell Engine, OMIQ o FCS Express42.
- Concatene los archivos FCS individuales de cada muestra en un solo archivo, si es necesario.
- Identifique las células individuales mediante la activación de eventos positivos del intercalador de iridio, lo que permite la discriminación de células nucleadas individuales de desechos o dobletes.
- Puerta para células vivas seleccionando eventos negativos de cisplatino. El cisplatino se une covalentemente a las proteínas celulares y marca las células moribundas y muertas con membranas comprometidas en mayor medida que las células vivas43.
- Determinar la población de interés, por ejemplo, el compartimento miogénico (Vivo/CD45-/CD31-/Sca1-/α7integrina+/CD9+) (Figura 2A) y cuantificar la proporción relativa de células madre y progenitoras. Este enfoque requiere un conocimiento previo de la superficie celular o de la expresión de marcadores intracelulares para definir poblaciones individuales.
- Realizar análisis de alta dimensión que permitan la identificación de subconjuntos de células raras previamente no reconocidos dentro de una población compleja, exportar la población de interés y utilizar algoritmos de agrupamiento que se han desarrollado específicamente para el análisis de datos de CyTOF44.
NOTA: En trabajos anteriores se empleó el algoritmo de desplazamiento X, que utiliza la estimación ponderada de la densidad del vecino k-más cercano (kNN-DE) en un espacio de alta dimensión para realizar agrupamientos no supervisados basados en parámetros definidos19. Se ha demostrado que X-shift es muy eficaz en la identificación de poblaciones de células raras45. - Para el análisis de desplazamiento X, descargue el paquete de software vortex (de la página de GitHub del laboratorio de Nolan [https://github.com/nolanlab/vortex]) y Java de 64 bits46. Las instrucciones se pueden encontrar aquí: https://github.com/nolanlab/vortex/wiki/Getting-Started.
- Cargue las poblaciones de celdas exportadas en una base de datos local y defina los parámetros de agrupación en clústeres. Entre los parámetros definidos por el usuario se encuentran (i) los marcadores utilizados para la agrupación, (ii) el rango de k valores (por ejemplo, de 5 a 150) y (iii) el número de pasos de agrupación.
NOTA: En trabajos anteriores se utilizó, como marcadores de agrupamiento, una combinación de marcadores de superficie que se sabe que se expresan en células madre musculares, nuevos marcadores de superficie identificados en un cribado citométrico de flujo de alto rendimiento de células musculares y mioblastos, y factores de transcripción miogénicos (TF) que se sabe que definen diferentes etapas de la miogénesis. Este enfoque permitió la identificación de dos marcadores de superficie celular, CD9 y CD104, cuyo patrón de coexpresión distingue poblaciones de células progenitoras previamente no reconocidas19. La elección cuidadosa de los marcadores de agrupamiento permitirá al investigador responder preguntas de investigación específicas. Por ejemplo, si se desea la identificación y el análisis a lo largo del tiempo de los subconjuntos de células en división, se recomienda incluir IdU como uno de los marcadores de agrupamiento. X-shift puede realizar la agrupación en clústeres a múltiples valores k e identificar automáticamente, mediante el cálculo del "punto de codo", el valor k que da como resultado un número óptimo de clústeres, evitando así la agrupación insuficiente o la fragmentación excesiva. Se recomienda calcular el punto de codo y utilizar el análisis de agrupación en clústeres definido por el valor k óptimo para la visualización de datos y las aplicaciones posteriores. - Para visualizar las relaciones espaciales entre las poblaciones de células dentro de los clústeres de desplazamiento X, realice un diseño dirigido por fuerza, que generará un mapa 2D donde la distancia entre los clústeres indica su similitud en la expresión de marcadores en un espacio fenotípico de alta dimensión. Al colorear el mapa usando un marcador a la vez, es posible descubrir nuevas poblaciones celulares y seguir su dinámica. Se recomienda la cuantificación de seguimiento mediante compuerta manual en software, como Cytobank y Cell Engine.
- Realice análisis de mapas de calor para cuantificar la expresión dinámica de múltiples proteínas de superficie e intracelulares a lo largo del tiempo y revelar nuevas tendencias.
NOTA: Al realizar un curso de tiempo de lesión, es posible agrupar los datos de todos los puntos de tiempo para generar un mapa de regeneración. Además, es posible diseccionar dicho mapa en los puntos de tiempo individuales para seguir la dinámica celular y molecular de la regeneración a lo largo del tiempo19.
11. Tinción con anticuerpos conjugados con fluoróforos para FACS
NOTA: Las celdas utilizadas para los controles de un solo color sin teñir y los controles de fluorescencia menos uno (FMO) pueden originarse a partir del conjunto de TA y GA de un mouse adicional si está disponible. Alternativamente, el cuádriceps (músculo anterior superior del muslo) se puede diseccionar y digerir en una suspensión de una sola célula, siguiendo el mismo procedimiento que para el TA + GA establecido anteriormente y utilizado para los controles. Prepare el tampón FACS (PBS, 2,5% de suero de cabra, 2 mM de EDTA), filtre a través de una membrana de PES en un recipiente de poliestireno y manténgalo en hielo. El tampón FACS puede almacenarse a 4 °C durante un máximo de 1 mes. En la Tabla 3 se puede encontrar una lista de anticuerpos utilizados para el FACS.
- Prepara una mezcla de linajes. Teñir cada control en 50 μL (que contiene aprox. 3-5 x 105 células). Calcule la cantidad de mezcla de linaje necesaria. Aumente el volumen entre un 20% y un 30%. La concentración de anticuerpos utilizados para la mezcla de linaje es de 0,2 mg/mL.
- En un tubo de 0,5 mL, agregue anti-CD45 APC-Cy7, anti-CD31 APC-Cy7, anti-Sca1 APC-Cy7 y anti-CD11b APC-Cy7 para alcanzar concentraciones finales de 1 μg/mL, 2,5 μg/mL, 2,5 μg/mL y 0,63 μg/mL, respectivamente.
- Prepara la mezcla para todas las manchas. Teñir cada muestra totalmente teñida en 500 μL (que contenga aprox. 3–5 x 106 células). Haga un exceso de volumen del 10% de la mezcla para todas las manchas.
- En un tubo de 1,5 mL, agregue anticuerpos de mezcla de linaje como los anteriores + integrina PE anti-α7 , anti-CD9 APC y anti-CD104 FITC para alcanzar concentraciones finales de 2 μg/mL, 1,2 μg/mL y 3 μg/mL, respectivamente.
- Preparación de tampón FACS + DAPI: Para 10 mL de tampón FACS, agregue 1 μL de DAPI (stock = 1 mg/mL) para alcanzar una concentración final de DAPI de 100 ng/mL.
PRECAUCIÓN: DAPI (diclorhidrato de 4',6-diamidino-2-fenilindol) está clasificado como un posible irritante cutáneo y respiratorio. Sin embargo, la solución de DAPI a una concentración del <1% se considera no peligrosa, según el GHS. Lea la SDS antes de manipularla. Se debe usar equipo de protección personal durante la manipulación. Los materiales que hayan estado en contacto con DAPI deben desecharse de acuerdo con las normas de seguridad locales. - Volver a suspender las células de la sección 6: Disección y disociación del músculo esquelético:
- En el caso de todas las muestras teñidas, vuelva a suspender los conjuntos de TA y GA de un solo ratón en 500 μl de tampón FACS y transfiéralo a tubos de 15 ml para la tinción.
- Para los controles, use conjuntos TA y GA o conjuntos cuádruples. Si utiliza 1 ratón para los controles, vuelva a suspender las células en 750 μL de tampón FACS. Si se trata de varios ratones, vuelva a suspender cada conjunto de TA y GA o cuádriceps en 500 μL de tampón FACS y combine los conjuntos en una sola muestra de 1 mL tomando una fracción de cada muestra. Añada 50 μl de pilas de control (3–5 x 105 células) a tubos de polipropileno de 5 ml para la tinción.
- Manchar como se describe en la Tabla 4 durante 45 min a 4 °C en la oscuridad.
- Lave todas las muestras manchadas añadiendo 5 mL de tampón FACS. Lave los controles añadiendo 1 ml de tampón FACS y centrífuga (380 x g, 10 min, 4 °C).
- Vuelva a suspender todas las muestras teñidas en 1 mL de tampón FACS + DAPI. Vuelva a suspender los controles en 300 μL de tampón FACS con o sin DAPI.
- Mantenga las células a 4 °C en la oscuridad hasta que se clasifiquen en un citómetro de flujo con 4 láseres (405 nm, 488 nm, 561 nm, 633 nm).
- Ejecute los controles y las muestras en el clasificador y cree puertas de clasificación utilizando el software correspondiente. En el caso de los controles sin manchar/de un solo color y los controles FMO, registre los eventos 1 x 104 y 0,3–1 x 105 , respectivamente. En el caso de las muestras totalmente teñidas, registre hasta 1 x 106 eventos. Clasifique todas las muestras manchadas, o tantas como sea necesario, en función de los requisitos del ensayo posterior. Para el citómetro de flujo utilizado aquí (FACSAria III), clasifique en modo de pureza con una boquilla de 70 mm.
- Consulte la Figura 4 para ver la estrategia de compuertas.
- Utilice los controles sin manchar y de un solo color para ajustar el voltaje de todos los detectores.
- Utilice los controles de un solo color para configurar la matriz de compensación.
- Utilice los controles FMO para establecer puertas de clasificación.
- Las distintas poblaciones de células madre y progenitoras musculares se clasifican en un tampón FACS helado.
- Centrifugar (380 x g, 10 min, 4 °C) las poblaciones celulares. Vuelva a suspender en el búfer adecuado, cuente y continúe con el análisis posterior.
Resultados
Aquí presentamos una descripción general de la configuración experimental para usar este enfoque combinado que incluye (i) análisis CyTOF de alta dimensión del curso del tiempo de una lesión aguda mediante inyección de notexina para estudiar la dinámica celular y molecular de las células madre y progenitoras en el músculo esquelético (Figura 1, esquema superior); y (ii) FACS de células madre y progenitoras utilizando dos marcadores de superficie ...
Discusión
La regeneración del músculo esquelético es un proceso dinámico que depende de la función de las células madre adultas. Si bien los estudios previos se han centrado en el papel de las células madre musculares durante la regeneración, su progenie in vivo ha sido poco estudiada, principalmente debido a la falta de herramientas para identificar y aislar estas poblaciones celulares 15,16,17,18.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Agradecemos a los miembros del Centro Principal de FACS en el Departamento de Biomedicina de la Universidad de Aarhus por su apoyo técnico. Agradecemos a Alexander Schmitz, gerente de la Unidad de Citometría de Masas del Departamento de Biomedicina, por la discusión y el apoyo. Las ilustraciones científicas se crearon utilizando Biorender.com. Este trabajo fue financiado por una beca inicial de Aarhus Universitets Forskningsfond (AUFF) y una subvención del paquete Start (0071113) de la Fundación Novo Nordisk a E.P.
Materiales
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Fisher Scientific | 07-200-886 | |
| 20 G needle | KDM | KD-fine 900123 | |
| 28 G, 0.5 mL insulin syringe | BD | 329461 | |
| 29 G, 0.3 mL insulin syringe | BD | 324702 | |
| 3 mL syringes | Terumo medical | MDSS03SE | |
| 40 µm cell strainers | Fisher Scientific | 11587522 | |
| 5 mL polypropylene tubes | Fisher Scientific | 352002 | |
| 5 mL polystyrene test tubes with 35 µm cell strainer | Falcon | 352235 | |
| 5 mL syringes | Terumo medical | SS05LE1 | |
| 50 mL centrifuge tube | Fisher Scientific | 05-539-13 | |
| 5-Iodo-2-deoxyuridine (IdU) | Merck | I7125-5g | |
| anti-CD104 FITC (clone: 346-11A) | Biolegend | 123605 | Stock = 0.5 mg/mL |
| anti-CD11b APC-Cy7 (Clone: M1/70) | Biolegend | 101226 | Stock = 0.2 mg/mL |
| anti-CD31 APC-Cy7 (clone: 390) | Biolegend | 102440 | Stock = 0.2 mg/mL |
| anti-CD45 APC-Cy7 (Clone: 30-F11) | Biolegend | 103116 | Stock = 0.2 mg/mL |
| anti-CD9 APC (clone: KMC8) | ThermoFisher Scientific | 17-0091-82 | Stock = 0.2 mg/mL |
| anti-Sca1 (Ly6A/E) APC-Cy7 (clone: D7) | Biolegend | 108126 | Stock = 0.2 mg/mL |
| anti-α7 integrin PE (clone: R2F2)) | UBC AbLab | 67-0010-05 | Stock = 1 mg/mL |
| BD FACS Aria III (4 laser) instrument | BD Biosciences | N/A | 405, 488, 561, and 633 nm laser |
| Bovine Serum Albumin | Sigma Aldrich | A7030-50G | |
| Buprenorphine 0.3 mg/mL | Ceva | Vnr 054594 | |
| CD104 (Clone: 346-11A) | BD Biosciences | 553745 | Dy162; In-house conjugated |
| CD106/VCAM-1 (Clone: 429 MVCAM.A) | Biolegend | 105701 | Er170; In-house conjugated |
| CD11b (Clone: M1/70) | BD Biosciences | 553308 | Nd148; In-house conjugated |
| CD29/Integrin β1 (Clone: 9EG7) | BD Biosciences | 553715 | Tm169; In-house conjugated |
| CD31 (Clone: MEC 13.3) | BD Biosciences | 557355 | Sm154; In-house conjugated |
| CD34 (Clone: RAM34) | BD Biosciences | 551387 | Lu175; In-house conjugated |
| CD44 (Clone: IM7) | BD Biosciences | 550538 | Yb171; In-house conjugated |
| CD45 (Clone: MEC 30-F11) | BD Biosciences | 550539 | Sm147; In-house conjugated |
| CD9 (Clone: KMC8) | Thermo Fisher Scientific | 14-0091-85 | Yb174; In-house conjugated |
| CD90.2/Thy1.2 (Clone: 30-H12) | BD Biosciences | 553009 | Nd144; In-house conjugated |
| CD98 (Clone: H202-141) | BD Biosciences | 557479 | Pr141; In-house conjugated |
| Cell Acquisition Solution/Maxpar CAS-buffer | Standard Biotools | 201240 | |
| Cell-ID Intercalator-Iridium | Standard Biotools | 201192B | cationic nucleic acid intercalator |
| Cisplatin | Merck | P4394 | Pt195 |
| Cisplatin (cis-Diammineplatinum(II) dichloride) | Merck | P4394 | |
| Clear 1.5 mL tube | Fisher Scientific | 11926955 | |
| Collagenase, Type II | Worthington Biochemical Corporation | LS004177 | |
| Counting chamber | Merck | BR718620-1EA | |
| CXCR4/SDF1 (Clone: 2B11/CXCR4 ) | BD Biosciences | 551852 | Gd158; In-house conjugated |
| DAPI (1 mg/mL) | BD Biosciences | 564907 | |
| Dark 1.5 mL tube | Fisher Scientific | 15386548 | |
| Dispase II | Thermo Fisher Scientific | 17105041 | |
| Dissection Scissors | Fine Science Tools | 14568-09 | |
| DMEM (low glucose, with pyruvate) | Thermo Fisher Scientific | 11885-092 | |
| EDTA (Ethylenediaminetetraacetic acid disodium salt) | Merck | E5134 | Na2EDTA-2H20 |
| EQ Four Element Calibration Beads (EQ beads) | Standard Biotools | 201078 | Calibration beads |
| Fetal Bovine Serum, qualified, Brazil origin | Thermo Fisher Scientific | 10270106 | |
| Forceps Dumont #5SF | Fine Science Tools | 11252-00 | |
| Forceps Dumont #7 | Hounisen.com | 1606.3350 | |
| Goat serum | Thermo Fisher Scientific | 16210-072 | |
| Helios CyTOF system | Standard Biotools | N/A | |
| Horse Serum, heat inactivated, New Zealand origin | Thermo Fisher Scientific | 26-050-088 | |
| IdU | Merck | I7125 | I127 |
| Iridium-Intercalator | Standard Biotools | 201240 | Ir191/193 |
| Isoflurane/Attane Vet | ScanVet | Vnr 055226 | |
| Methanol | Fisher Scientific | M/3900/17 | |
| Myf5 (Clone: C-20) | Santa Cruz Biotechnology | Sc-302 | Yb173; In-house conjugated |
| MyoD (Clone: 5.8A) | BD Biosciences | 554130 | Dy164; In-house conjugated |
| MyoG (Clone: F5D) | BD Biosciences | 556358 | Gd160; In-house conjugated |
| Nalgene Rapid-Flow Sterile Disposable Bottle Top 0.20 μM PES Filters | Thermo Fisher Scientific | 595-4520 | |
| Notexin | Latoxan | L8104 | Resuspend to 50 µg/ml in sterile PBS. Keep stocks (e.g. 50 µl) at -20 °C |
| Nutrient mixture F-10 (Ham's) | Thermo Fisher Scientific | 31550031 | |
| pAkt (Clone: D9E) | Standard Biotools | 3152005A | Sm152 |
| Pax7 (Clone: PAX7) | Santa Cruz Biotechnology | Sc-81648 | Eu153; In-house conjugated |
| Penicillin-Streptomycin (10,000 U/mL) (Pen/Strep) | Thermo Fisher Scientific | 15140122 | |
| PES Filter Units 0.20 μM | Fisher Scientific | 15913307 | |
| PES Syringe Filter | Fisher Scientific | 15206869 | |
| Petri dish | Sarstedt | 82.1472.001 | |
| PFA 16% EM grade | MP Biomedicals | 219998320 | |
| Potassium chloride (KCl) | Fisher Scientific | 10375810 | |
| Potassium phosphate, monobasic, anhydrous (KH2PO4) | Fisher Scientific | 10573181 | |
| pRb (Clone: J112-906) | Standard Biotools | 3166011A | Er166 |
| pS6 kinase (Clone: N7-548) | Standard Biotools | 3172008A | Yb172 |
| Sca-1 (Clone: E13-161.7) | BD Biosciences | 553333 | Nd142; In-house conjugated |
| Sodium Azide | Sigma Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | 10553515 | |
| Sodium phosphate, dibasic, heptahydrate (Na2HPO4-6H2O) | Merck | S9390 | |
| Sterile saline solution 0.9% | Fresenius | B306414/02 | |
| α7 integrin (Clone: 3C12) | MBL international | K0046-3 | Ho165; In-house conjugated |
Referencias
- Mukund, K., Subramaniam, S. Skeletal muscle: A review of molecular structure and function in health and disease. Wiley Interdiscip Rev Syst Biol Med. 12 (1), e1462 (2020).
- Feige, P., Brun, C. E., Ritso, M., Rudnicki, M. A. Orienting muscle stem cells for regeneration in homeostasis, aging, and disease. Cell Stem Cell. 23 (5), 653-664 (2018).
- Mauro, A. Satellite cell of skeletal muscle fibers. J Biophys Biochem Cytol. 9 (2), 493-495 (1961).
- Seale, P., et al. Pax7 is required for the specification of myogenic satellite cells. Cell. 102 (6), 777-786 (2000).
- Fuchs, E., Blau, H. M. Tissue stem cells: Architects of their niches. Cell Stem Cell. 27 (4), 532-556 (2020).
- Hernández-hernández, J. M., et al. The myogenic regulatory factors, determinants of muscle development, cell identity and regeneration. Semin Cell Dev Biol. 72, 10-18 (2017).
- Zammit, P. S. Function of the myogenic regulatory factors Myf5, MyoD, Myogenin and MRF4 in skeletal muscle, satellite cells and regenerative myogenesis. Semin Cell Dev Biol. 72, 19-32 (2017).
- Sabourin, L. A. The molecular regulation of myogenesis. Clin Genet. 57 (1), 16-25 (2000).
- Cooper, R. N., et al. In vivo satellite cell activation via Myf5 and MyoD in regenerating mouse skeletal muscle. J Cell Sci. 112 (17), 2895-2901 (1999).
- Rudnicki, M. A., Jaenisch, R. The MyoD family of transcription factors and skeletal myogenesis. Bioessays. 17 (3), 203-209 (1995).
- Braun, T., Arnold, H. H. Inactivation of Myf-6 and Myf-5 genes in mice leads to alterations in skeletal muscle development. EMBO J. 14 (6), 1176-1186 (1995).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Braun, T., et al. MyoD expression marks the onset of skeletal myogenesis in Myf-5 mutant mice. Development. 120 (11), 3083-3092 (1994).
- Rudnicki, M. A., et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell. 75 (7), 1351-1359 (1993).
- Montarras, D., et al. Developmental biology: Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Sacco, A., Doyonnas, R., Kraft, P., Vitorovic, S., Blau, H. M. Self-renewal and expansion of single transplanted muscle stem cells. Nature. 456 (7221), 502-506 (2008).
- Cerletti, M., et al. Highly efficient, functional engraftment of skeletal muscle stem cells in dystrophic muscles. Cell. 134 (1), 37-47 (2008).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nat Protoc. 10 (10), 1612-1624 (2015).
- Porpiglia, E., et al. High-resolution myogenic lineage mapping by single-cell mass cytometry. Nat Cell Biol. 19 (5), 558-567 (2017).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry Part A. 81 (7), 552-566 (2012).
- Hartmann, F. J., et al. . Mass Cytometry: Methods and Protocols. , (2019).
- Devine, R. D., Behbehani, G. K. Use of the pyrimidine analog, 5-iodo-2'-deoxyuridine (IdU) with cell cycle markers to establish cell cycle phases in a mass cytometry platform. J Vis Exp. (176), e60556 (2021).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Nag, A. C., Foster, J. D. Myogenesis in adult mammalian skeletal muscle in vitro. J Anat. 132, 1-18 (1981).
- Le Moigne, A., et al. Characterization of myogenesis from adult satellite cells cultured in vitro). Int J Dev Biol. 34, 171-180 (1990).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Chu, C., Cogswell, J., Kohtz, D. S. MyoD functions as a transcriptional repressor in proliferating myoblasts. J Biol Chem. 272 (6), 3145-3148 (1997).
- Shah, B., Hyde-Dunn, J., Jones, G. E. Proliferation of murine myoblasts as measured by bromodeoxyuridine incorporation. Methods in Mol Biol. 75, 349-355 (1997).
- Springer, M. L., Blau, H. M. High-efficiency retroviral infection of primary myoblasts. Somat Cell Mol Genet. 23 (3), 203-209 (1997).
- Rando, T. A., Blau, H. M. Primary mouse myoblast purification, characterization, and transplantation for cell-mediated gene therapy. J Cell Biol. 125 (6), 1275-1287 (1994).
- Springer, M. L., Rando, T. A., Blau, H. M. Gene delivery to muscle. Curr Protoc Hum Genet. , (2002).
- Cull-Candy, S. G., Fohlman, J., Gustavsson, D., Lullmann-Rauch, R., Thesleff, S. The effects of taipoxin and notexin on the function and fine structure of the murine neuromuscular junction. Neuroscience. 1 (3), 175-180 (1976).
- Francis, B., John, T. R., Seebart, C., Kaiser, I. I. New toxins from the venom of the common tiger snake (Notechis scutatus scutatus). Toxicon. 29 (1), 85-96 (1991).
- Navarro, K. L., Huss, M., Smith, J. C., Sharp, P., Marx, J. O., Pacharinsak, C. Mouse Anesthesia: The Art and Science. ILAR Journal. 62, 238-273 (2021).
- Langford, D., Bailey, A., Chanda, M., et al. Coding of facial expressions of pain in the laboratory mouse. Nat Methods. 7, 447-449 (2010).
- Matsumiya, L. C., Sorge, R. E., Sotocinal, S. G., Tabaka, J. M., Wieskopf, J. S., Zaloum, A., King, O. D., Mogil, J. S. Using the Mouse Grimace Scale to reevaluate the efficacy of postoperative analgesics in laboratory mice. J Am Assoc Lab Anim Sci. 2012 (1), 42-49 (2012).
- Gonzalez, V. D., et al. High-grade serous ovarian tumor cells modulate NK cell function to create an immune-tolerant microenvironment. Cell Rep. 36 (9), 109632 (2021).
- Delgado-Gonzalez, A., et al. Measuring trogocytosis between ovarian tumor and natural killer cells. STAR Protoc. 3 (2), 101425 (2022).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry Part A. 83 (5), 483-494 (2013).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. J Vis Exp. (69), e4398 (2012).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample preparation for mass cytometry analysis. J Vis Exp. (122), e54394 (2017).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Curr Protoc Cytom. , (2010).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry Part A. 81 (6), 467-475 (2012).
- Kimball, A. K., et al. A beginner's guide to analyzing and visualizing mass cytometry data. J Immunol. 200 (1), 3-22 (2018).
- Weber, L. M., Robinson, M. D. Comparison of clustering methods for high-dimensional single-cell flow and mass cytometry data. Cytometry Part A. 89 (12), 1084-1096 (2016).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nat Methods. 13 (6), 493-496 (2016).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Anal Chem. 80 (7), 2539-2547 (2008).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nat Commun. 12 (1), 692 (2021).
- de Morree, A., et al. Staufen1 inhibits MyoD translation to actively maintain muscle stem cell quiescence. Proc Natl Acad Sci U S A. 114 (43), E8996-E9005 (2017).
- Luo, D., et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c. Proc Natl Acad Sci U S A. 114 (15), E3071-E3080 (2017).
- Wersto, R. P., et al. Doublet discrimination in DNA cell-cycle analysis. Cytometry. 46 (5), 296-306 (2001).
- Porpiglia, E., Blau, H. M. Plasticity of muscle stem cells in homeostasis and aging. Curr Opin Genet Dev. 77, 101999 (2022).
- Porpiglia, E., et al. Elevated CD47 is a hallmark of dysfunctional aged muscle stem cells that can be targeted to augment regeneration. Cell Stem Cell. 29 (12), 1653-1668 (2022).
- Brunet, A., Goodell, M. A., Rando, T. A. Ageing and rejuvenation of tissue stem cells and their niches. Nat Rev Mol Cell Biol. 24 (1), 45-62 (2022).
- Danielli, S. G., et al. Single-cell profiling of alveolar rhabdomyosarcoma reveals RAS pathway inhibitors as cell-fate hijackers with therapeutic relevance. Sci Adv. 9 (6), (2023).
- de Morree, A., Rando, T. A. Regulation of adult stem cell quiescence and its functions in the maintenance of tissue integrity. Nat Rev Mol Cell Biol. 24 (5), 334-354 (2023).
- Yucel, N., et al. Glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell function. Cell Rep. 27 (13), 3939-3955 (2019).
- Tierney, M. T., Sacco, A. Inducing and evaluating skeletal muscle injury by notexin and barium chloride. Methods Mol Biol. 1460, 53-60 (2016).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198 (2016).
- Call, J. A., Lowe, D. A. Eccentric contraction-induced muscle injury: Reproducible, quantitative, physiological models to impair skeletal muscle's capacity to generate force. Methods Mol Biol. 1460, 3-18 (2016).
- Garry, G. A., Antony, M. L., Garry, D. J. Cardiotoxin Induced Injury and Skeletal Muscle Regeneration. Methods Mol Biol. 1460, 61-71 (2016).
- Le, G., Lowe, D. A., Kyba, M. Freeze injury of the tibialis anterior muscle. Methods Mol Biol. 1460, 33-41 (2016).
- Borok, M., et al. Progressive and coordinated mobilization of the skeletal muscle niche throughout tissue repair revealed by single-cell proteomic analysis. Cells. 10 (4), 744 (2021).
- Petrilli, L. L., et al. High-dimensional single-cell quantitative profiling of skeletal muscle cell population dynamics during regeneration. Cells. 9 (7), 1723 (2020).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Mol Cell. 74 (3), 609-621 (2019).
- Hartmann, F. J., et al. Scalable conjugation and characterization of immunoglobulins with stable mass isotope reporters for single-cell mass cytometry analysis. Methods Mol Biol. 1989, 55-81 (2019).
- Frimand, Z., Das Barman, S., Kjær, T. R., Porpiglia, E., de Morrée, A. Isolation of quiescent stem cell populations from individual skeletal muscles. J Vis Exp. (190), e64557 (2022).
- Krutzik, P. O., Nolan, G. P. Intracellular phospho-protein staining techniques for flow cytometry: monitoring single cell signaling events. Cytometry A. 55 (2), 61-70 (2003).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat Biotechnol. 30 (9), 858-867 (2012).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Curr Protoc Immunol. , 11-20 (2012).
- Krutzik, P. O., Clutter, M. R., Nolan, G. P. Coordinate analysis of murine immune cell surface markers and intracellular phosphoproteins by flow cytometry. J Immunol. 175 (4), 2357-2365 (2005).
- Krutzik, P. O., Irish, J. M., Nolan, G. P., Perez, O. D. Analysis of protein phosphorylation and cellular signaling events by flow cytometry: techniques and clinical applications. Clin Immunol. 110 (3), 206-221 (2004).
- Han, G., Spitzer, M. H., Bendall, S. C., Fantl, W. J., Nolan, G. P. Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry. Nat Protoc. 13 (10), 2121-2148 (2018).
- Chevrier, S., et al. Compensation of signal spillover in suspension and imaging mass cytometry. Cell Syst. 6 (5), 612-620 (2018).
- Bjornson, Z. B., Nolan, G. P., Fantl, W. J. Single-cell mass cytometry for analysis of immune system functional states. Curr Opin Immunol. 25 (4), 484-494 (2013).
- Kalina, T., Lundsten, K., Engel, P. Relevance of antibody validation for flow cytometry. Cytometry A. 97 (2), 126-136 (2020).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J Immunol Methods. 243 (1-2), 77-97 (2000).
- Roederer, M. Spectral compensation for flow cytometry: visualization artifacts, limitations, and caveats. Cytometry. 45 (3), 194-205 (2001).
- Tung, J. W., Parks, D. R., Moore, W. A., Herzenberg, L. A., Herzenberg, L. A. New approaches to fluorescence compensation and visualization of FACS data. Clin Immunol. 110 (3), 277-283 (2004).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados