Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification et analyse de progéniteurs myogéniques in vivo au cours d’une lésion aiguë des muscles squelettiques par cytométrie de masse unicellulaire de grande dimension
Dans cet article
Résumé
Le protocole présenté ici permet l’identification et l’analyse de haute dimension des cellules souches musculaires et progénitrices par cytométrie de masse unicellulaire et leur purification par FACS pour des études approfondies de leur fonction. Cette approche peut être appliquée pour étudier la dynamique de régénération dans des modèles de maladies et tester l’efficacité d’interventions pharmacologiques.
Résumé
La régénération des muscles squelettiques est un processus dynamique piloté par les cellules souches musculaires adultes et leur progéniture. La plupart du temps au repos à l’état d’équilibre, les cellules souches musculaires adultes s’activent lors d’une lésion musculaire. Après l’activation, ils prolifèrent et la plupart de leur progéniture se différencie pour générer des cellules musculaires compétentes en matière de fusion, tandis que le reste s’auto-renouvelle pour reconstituer le pool de cellules souches. Alors que l’identité des cellules souches musculaires a été définie il y a plus d’une décennie, sur la base de la co-expression de marqueurs de surface cellulaire, les progéniteurs myogéniques n’ont été identifiés que récemment à l’aide d’approches unicellulaires de grande dimension. Ici, nous présentons une méthode de cytométrie de masse unicellulaire (cytométrie par temps de vol [CyTOF]) pour analyser les cellules souches et les cellules progénitrices dans les lésions musculaires aiguës afin de résoudre la dynamique cellulaire et moléculaire qui se déroule pendant la régénération musculaire. Cette approche est basée sur la détection simultanée de nouveaux marqueurs de surface cellulaire et de facteurs clés de transcription myogénique dont l’expression dynamique permet l’identification de cellules souches activées et de populations de cellules progénitrices qui représentent des jalons de la myogenèse. Il est important de noter qu’une stratégie de tri basée sur la détection des marqueurs de surface cellulaire CD9 et CD104 est décrite, permettant l’isolement prospectif des cellules souches musculaires et des cellules progénitrices à l’aide du tri cellulaire activé par fluorescence (FACS) pour des études approfondies de leur fonction. Les cellules progénitrices musculaires constituent un chaînon manquant essentiel pour étudier le contrôle du destin des cellules souches musculaires, identifier de nouvelles cibles thérapeutiques pour les maladies musculaires et développer des applications de thérapie cellulaire pour la médecine régénérative. L’approche présentée ici peut être appliquée à l’étude des cellules souches musculaires et des cellules progénitrices in vivo en réponse à des perturbations, telles que des interventions pharmacologiques ciblant des voies de signalisation spécifiques. Il peut également être utilisé pour étudier la dynamique des cellules souches musculaires et des cellules progénitrices dans des modèles animaux de maladies musculaires, faisant progresser notre compréhension des maladies des cellules souches et accélérant le développement de thérapies.
Introduction
Le muscle squelettique constitue le plus grand tissu en masse du corps et régule de multiples fonctions, de la vue à la respiration, de la posture au mouvement, ainsi que le métabolisme1. Par conséquent, le maintien de l’intégrité et de la fonction des muscles squelettiques est essentiel à la santé. Le tissu musculaire squelettique, qui se compose de faisceaux serrés de myofibres multinucléées entourés d’un réseau complexe de nerfs et de vaisseaux sanguins, présente un potentiel de régénération remarquable 1,2.
Les principaux moteurs de la régénération des muscles squelettiques sont les cellules souches musculaires adultes (MuSC). Également connues sous le nom de cellules satellites, en raison de leur emplacement anatomique unique adjacent à la membrane plasmique de la myofibre et sous la lame basale, elles ont été identifiées pour la première fois en 19613. Les MuSC expriment un marqueur moléculaire unique, le facteur de transcription apparié case 7 (Pax7)4. Principalement quiescents chez les adultes en bonne santé, ils s’activent lors d’une blessure musculaire et prolifèrent pour donner naissance à une progéniture qui (i) se différenciera en cellules musculaires compétentes en fusion qui formeront de nouvelles myofibres pour réparer les dommages musculaires ou (ii) s’auto-renouvelleront pour reconstituer le pool de cellules souches5.
Au niveau cellulaire et moléculaire, le processus de régénération est assez dynamique et implique des transitions d’état cellulaire, caractérisées par l’expression coordonnée de facteurs de transcription myogéniques clés, également appelés facteurs de régulation myogéniques (MRF)6,7. Des études antérieures de développement in vivo, des expériences de traçage de la lignée et des travaux de culture cellulaire utilisant des myoblastes ont montré que l’expression séquentielle de ces facteurs de transcription entraîne la myogenèse, le facteur myogénique 5 (Myf5) étant exprimé lors de l’activation, la différenciation myogénique 1 (MyoD1) marquant l’engagement envers le programme myogénique et l’expression de la myogénine (MyoG) marquant la différenciation 8,9,10,11, 12,13,14. Malgré ces connaissances et la découverte de marqueurs de surface cellulaire pour purifier les MuSCs, les stratégies et les outils permettant d’identifier et d’isoler des populations discrètes le long de la voie de différenciation myogénique et de résoudre une progression myogénique in vivo ont fait défaut 15,16,17,18.
Nous présentons ici une nouvelle méthode, basée sur des recherches récemment publiées, qui permet d’identifier les cellules souches et progénitrices dans le muscle squelettique et d’analyser leur dynamique cellulaire, moléculaire et de prolifération dans le contexte d’une lésion musculaire aiguë19. Cette approche s’appuie sur la cytométrie de masse unicellulaire (également connue sous le nom de cytométrie par temps de vol [CyTOF]) pour détecter simultanément les principaux marqueurs de surface cellulaire (intégrine α7, CD9, CD44, CD98 et CD104), les facteurs de transcription myogénique intracellulaires (Pax7, Myf5, MyoD et MyoG) et un analogue nucléosidique (5-Iodo-2′-désoxyuridine, IdU), pour surveiller les cellules en phase S19,20, 21,22,23. De plus, le protocole présente une stratégie basée sur la détection de deux marqueurs de surface cellulaire, CD9 et CD104, pour purifier ces populations cellulaires par tri cellulaire activé par fluorescence (FACS), permettant ainsi de futures études approfondies de leur fonction dans le contexte des lésions et des maladies musculaires. Bien que les myoblastes primaires aient été largement utilisés dans le passé pour étudier les stades tardifs de la différenciation myogénique in vitro, on ne sait pas s’ils récapitulent l’état moléculaire des cellules progénitrices musculaires trouvées in vivo 24,25,26,27,28,29,30 . La production de myoblastes est laborieuse et prend du temps, et l’état moléculaire de cette culture primaire change rapidement lors du passage de31. Par conséquent, des progéniteurs myogéniques fraîchement isolés purifiés avec cette méthode fourniront un système plus physiologique pour étudier la myogenèse et l’effet des manipulations génétiques ou pharmacologiques ex-vivo.
Le protocole présenté ici peut être appliqué pour répondre à une variété de questions de recherche, par exemple, pour étudier la dynamique du compartiment myogénique in vivo dans des modèles animaux de maladies musculaires, en réponse à des manipulations génétiques aiguës ou à des interventions pharmacologiques, approfondissant ainsi notre compréhension du dysfonctionnement des cellules souches musculaires dans différents contextes biologiques et facilitant le développement de nouvelles interventions thérapeutiques.
Access restricted. Please log in or start a trial to view this content.
Protocole
Les procédures sur les animaux ont été approuvées par l’inspection danoise des expériences sur les animaux (protocole # 2022-15-0201-01293), et les expériences ont été réalisées conformément aux directives institutionnelles de l’Université d’Aarhus. L’analgésie (buprénorphine) est fournie dans l’eau de boisson 24 h avant la blessure pour que les souris s’adaptent au goût. L’apport de buprénorphine dans l’eau potable est maintenu pendant 24 heures après la blessure. Associée à une injection sous-cutanée (s.c.) de buprénorphine au moment de la lésion musculaire aiguë, la buprénorphine dans l’eau de boisson après l’injection de notexine soulagera la douleur associée à la blessure. Bien qu’il soit recommandé d’administrer une injection de buprénorphine au moment de la blessure musculaire aiguë, suivie de buprénorphine dans l’eau potable, la buprénorphine dans l’eau potable avant la blessure est facultative. Cependant, les chercheurs doivent suivre les normes et les lignes directrices en matière de bien-être animal établies par l’organisme de réglementation approprié.
REMARQUE : Pour les expériences de cytométrie de masse unicellulaire (CyTOF) des muscles des membres postérieurs blessés, commencer à la section 1 : Analgésie dans l’eau 24 h avant la blessure musculaire jusqu’à 24 h après la blessure. Pour le tri des cellules souches musculaires et des cellules progénitrices de souris non blessées, effectuez les sections 5 et 6 : Euthanasie + dissection et dissociation des muscles squelettiques, et passez à la section 11 : Coloration avec des anticorps conjugués aux fluorophores pour le FACS. La figure 1 donne un aperçu du dispositif expérimental et du protocole.
1. Analgésie dans l’eau 24 h avant la blessure musculaire jusqu’à 24 h après la blessure
- Dans une bouteille foncée ou enveloppée d’une feuille d’aluminium, ajoutez 3 mL de buprénorphine (0,3 mg/mL) et remplissez jusqu’à 100 mL d’eau filtrée pour atteindre une concentration finale de 0,009 mg/mL 24 h avant la blessure. Fixez-le à la cage de la souris.
- Retirez la gourde 24 h après la blessure et reconnectez la cage de la souris au système de valve d’abreuvement.
2. Préparation à la procédure de blessure aiguë
REMARQUE : Utilisez de l’éthanol à 70% pour désinfecter l’établi, la configuration du cône de nez et la boîte d’induction.
- Préparez un tube de 1,5 mL avec de la notexine diluée dans du PBS (5 μg/mL). Diluer la buprénorphine (0,3 mg/mL) dans une solution saline stérile à 0,9 % jusqu’à 0,015 mg/mL dans un tube de 1,5 mL sans danger pour la lumière. Restez sur la glace. Préparez des seringues à insuline pour les injections de buprénorphine (28 g, 0,5 ml) et de notexine (29 g, 0,3 ml).
- Configurer les unités d’anesthésie à base d’isoflurane :
- Pour l’anesthésie dans une boîte à induction, utilisez 3% d’isoflurane avec un débit de 1,5 L/min (50% O2, 50% air atmosphérique).
- Pour l’entretien à l’aide d’une configuration à cône de nez, utilisez 1,5 % d’isoflurane avec un débit de 0,6 L/min (50 % O2, 50 % d’air atmosphérique).
3. Blessure aiguë par injection de notexine
ATTENTION : La notexine a une activité de phospholipase A2 et est le principal composant du venin de la couleuvre tigrée australienne (Notechis scutatus), avec une DL50 intraveineuse de 5 à 17 mg de notexine/kg chez la souris32,33. Dans le protocole actuel, le muscle tibial antérieur (TA) de chaque membre postérieur est injecté avec 10 μL de notexine à 5 mg/mL, et le muscle gastrocnémien (GA) de chaque membre postérieur est injecté deux fois (une fois dans chaque tête du muscle) avec 15 μL de notexine à 5 mg/mL. Il est important d’effectuer correctement les injections intramusculaires (i.m.) pour limiter les dommages et d’inspecter fréquemment les animaux injectés pour s’assurer de minimiser la douleur.
- Anesthésier la souris dans la boîte d’induction (3% d’isoflurane). Lorsque la souris est inconsciente, abaissez le taux d’isoflurane à 1,5 % dans la boîte à induction.
- Pesez et marquez individuellement les souris conformément aux procédures approuvées par l’établissement. Sous la configuration du cône nasal, rasez les membres postérieurs avec une tondeuse et transférez-les dans la boîte d’induction.
- Calculer le volume de buprénorphine pour chaque souris :
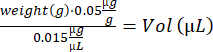
- Mélangez la solution de notexine en pipetant de haut en bas. Préparez 2 seringues à insuline (29 g, 0,3 ml) chargées de 10 μL de solution de notexine (5 μg/mL) pour les injections d’AT, et 4 seringues à insuline chargées de 15 μL de solution de notexine (5 μg/mL) pour l’injection de GA.
REMARQUE : Le muscle tibial antérieur (TA) est situé sur la face antérieure de la jambe inférieure de la souris, s’étendant du genou à la cheville. Le muscle gastrocnémien (GA) est un muscle à deux têtes situé à l’arrière de la jambe, superficiellement par rapport au soléaire. Il s’étend de ses deux têtes (médiale et latérale) juste au-dessus du genou jusqu’au talon, s’étendant sur un total de trois articulations (genou, cheville et articulations sous-taliennes). - Effectuer des injections intramusculaires
- Injection TA (2 injections au total)
- Placez la souris en position couchée sous le cône nasal et désinfectez le site d’injection avec des lingettes imbibées d’alcool stériles (éthanol à 70 % ou isopropanol). Insérez l’aiguille, biseau vers le bas, dans le ventre du TA (distal au milieu du ventre) à un angle de 30°. Avancez l’aiguille le long du muscle, parallèlement au tibia, pour atteindre le milieu du ventre de l’AT. Injectez la notexine lentement et continuellement, en laissant l’aiguille en place pendant 10 secondes avant de la retirer. N’insérez pas l’aiguille trop profondément au départ (pour éviter d’injecter de la notexine sous le TA dans le muscle long extenseur des doigts), et pas trop loin en proximal lors de l’avancement de l’aiguille (pour éviter d’injecter trop près du genou).
- Injection GA (4 injections au total)
- Insérez l’aiguille, biseau vers le bas, à un angle de ~45° au milieu du ventre de la tête latérale du GA. Injectez la notexine lentement et continuellement, en laissant l’aiguille en place pendant 10 secondes avant de la retirer. Faites pivoter le membre postérieur et injectez de la notexine dans la tête médiale du muscle GA comme ci-dessus. Il faut veiller à ne pas insérer l’aiguille trop profondément.
- Injection TA (2 injections au total)
- Retournez la souris et injectez la buprénorphine s.c. à l’aide d’une seringue à insuline (28 G, 0,5 ml). Transférez la souris dans une cage de récupération vide sur une plaque chauffante. Seule la moitié du fond de la cage doit être placée sur la plaque chauffante pour permettre à la souris de se thermoréguler pendant la récupération34. Répétez les étapes 3.3 à 3.6 pour les souris restantes.
- Lorsque la souris dans la cage de récupération est complètement réveillée, remettez-la dans la cage d’origine. Remettez-le à l’étable et complétez-le avec de la nourriture mouillée. Retirer la bouteille de buprénorphine 24 h après l’injection.
- Surveillez les souris pendant 6 h après l’injection de notexin, puis toutes les 12 h pendant 2 jours pour détecter des signes de douleur, une mobilité réduite et une diminution de la consommation alimentaire35,36.
4. Injection de 5-iodo-2'-désoxyuridine
ATTENTION : La 5-iodo-2'-désoxyuridine (IdU) est soupçonnée de provoquer des anomalies génétiques et de nuire à la fertilité de l’enfant à naître. Lisez la fiche de données de sécurité (FDS) avant de la manipuler. Un équipement de protection individuelle doit être porté pendant la manipulation. Utilisez une hotte pour peser la poudre IdU. Les matériaux qui ont été en contact avec l’IdU doivent être jetés conformément aux réglementations de sécurité locales.
REMARQUE : Le marquage IdU in vivo est utilisé pour surveiller la division cellulaire pendant le temps de blessure, car l’IdU, un analogue iodé de la thymidine, est incorporé dans l’ADN des cellules en phase S. L’IdU est injecté par voie intrapéritonéale (i.p.) à raison de 20 mg/kg de poids corporel 8 h avant de sacrifier la souris.
- À l’aide d’un sonicateur à 37 °C, préparer une solution mère de 2 mg/mL d’IdU dans du PBS stérile. L’IdU est sensible à la lumière ; à utiliser dans la même journée ou à congeler à -20 °C jusqu’à 3 mois. En cas d’utilisation d’IdU congelé : décongélation, vortex, centrifugeuse à 10 000 x g pendant 30 s, et utilisation du surnageant pour les injections ci-dessous.
- Anesthésier la souris dans la boîte d’induction (3% d’isoflurane). Notez l’affectation, pesez les souris et calculez le volume de la solution IdU :
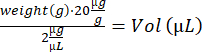
- Effectuez l’injection intraveineuse à l’aide d’une seringue à insuline (28 G, 0,5 ml) et transférez la souris dans une cage de récupération vide sur une plaque chauffante. Une fois réveillé, remettez la souris dans sa cage d’origine. Répétez les étapes 4.2 à 4.3 pour les souris restantes et renvoyez-les à l’étable.
5. Euthanasie
REMARQUE : Voir le Tableau 1 pour les recettes de tampons. Préparez le milieu de lavage (mélange nutritif F-10 (jambon), 10 % de sérum de cheval, 1 stylo/streptocoque) et filtrez à travers une membrane en polyéthersulfone (PES) dans un récipient en polystyrène. Préparez un tampon de dissociation (milieu de lavage complété par 650 U/mL de collagénase, type II) et conservez-le sur de la glace. Les mesures de cytométrie de masse CyTOF sont très sensibles aux contaminants. Pour cette raison, il est essentiel d’utiliser des réactifs de la plus haute qualité analytique pour le traitement des échantillons. Pour éviter la contamination métallique, il est fortement recommandé d’utiliser des articles en plastique stériles et de la verrerie neuve qui n’a jamais été lavée avec un détergent, car de nombreux savons de laboratoire contiennent des niveaux élevés de baryum. Il est recommandé d’utiliser de l’eau distillée déminéralisée doublement filtrée pour la préparation des réactifs. La solution saline tamponnée au phosphate (PBS) est préparée sur place. Diluez les crosses 10x à 1x et filtrez le PBS 1x avec des filtres de 0,2 μm. Filtrez à nouveau le 1x PBS au début de chaque expérience. Les outils de dissection ne doivent pas être nettoyés avec un détergent en raison de la présence de baryum.
- Préparez la balance, le récipient et l’unité d’anesthésie à base d’isoflurane avec la boîte à induction. Désinfectez les outils de dissection appropriés.
- Préparez et marquez des tubes de 50 ml pour chaque souris avec 5 ml de tampon de dissociation et une boîte de Pétri. Gardez le tampon de dissociation sur la glace.
- Anesthésier la souris dans la boîte d’induction (3% d’isoflurane, augmenter à 5% en cas d’inconscience) et effectuer une luxation cervicale.
6. Dissection et dissociation des muscles squelettiques
- Disséquez les muscles TA et GA des deux membres postérieurs et transférez-les dans le couvercle d’une boîte de Pétri. À l’aide de ciseaux, coupez le tissu en une bouillie hachée (~1 mm,3 morceaux). Transférer dans un tube de 50 mL contenant 5 mL de tampon de dissociation glacé. Restez sur la glace.
- Répétez la procédure d’euthanasie (étape 5.3) et la procédure de dissection et de dissociation des muscles squelettiques (étape 6.1) jusqu’à ce que toutes les souris aient été traitées.
- Préchauffez tous les tubes d’échantillon dans un bain-marie à 37 °C (2 min) et incubez pendant 45 min en rotation dans un incubateur (37 °C).
- Vortex et laver en ajoutant 10 mL de produit de lavage. Centrifuger (380 x g, 10 min, température ambiante [RT]) et aspirer à 4 mL.
- Ajouter 0,5 mL de collagénase (1000 U/mL) et 0,5 mL de dispase (11 U/mL). Vortex et incuber pendant 20 min en rotation à 37 °C (par exemple, dans un incubateur). Centrifuger (380 x g, 1 min, RT) et remettre en suspension avec une pipette de 5 mL.
- Ajouter des crépines à cellules de 40 μm dans des tubes de 50 mL et pré-mouiller avec 5 mL de produit de lavage. Aspirer et éjecter la suspension cellulaire 10 fois à l’aide d’une seringue de 5 mL avec une aiguille de 20 G. Filtrer à travers la crépine de 40 μm pré-humidifiée. Lavez le tube de 50 ml avec un produit de lavage de 10 ml, transférez-le dans la crépine à cellules de 40 μm et centrifugez (380 x g, 10 min, 4 °C) la suspension cellulaire.
7. Coloration vivante/morte avec fixation au cisplatine et au paraformaldéhyde
ATTENTION : Le cisplatine et le paraformaldéhyde (PFA) sont cancérigènes. Lisez la FDS avant de la manipuler. Le paraformaldéhyde (PFA ; 16 %) est un irritant pour la peau, les yeux et les voies respiratoires. Portez un équipement de protection individuelle et manipulez ces substances sous une hotte. Lors de la fixation des cellules, la concentration finale de PFA sera de 1,6%. Des mesures de protection appropriées doivent être prises et les déchets doivent être traités conformément aux réglementations locales.
REMARQUE : Préparez du DMEM sans sérum froid (4 °C) et chaud (37 °C). Préparez le DMEM complété par 10 % de FBS, filtrez à travers une membrane PES dans un récipient en polystyrène et conservez-le sur de la glace. Préparer le PBS et les milieux de coloration cellulaire (CSM ; PBS, 0,5 % BSA, 0,02 % d’azoture de sodium) dans une bouteille en verre dédiée au CyTOF et filtrer à travers une membrane PES. Le CSM peut être conservé à 4 °C jusqu’à 6 mois.
- Aspirez le surnageant, agissez la pastille et mettez-la en suspension dans 1 ml de DMEM froid sans sérum. Comptez les cellules et ajustez la densité cellulaire à 1 x 106 cellules/mL dans du DMEM sans sérum.
- Ajouter le stock de cisplatine (25 mM) à une concentration finale de 25 μM. Vortex 10 s et incuber pendant exactement 1 min (la réaction est très sensible au temps). Éteignez la réaction avec du DMEM glacé + 10% FBS (3 fois le volume de l’échantillon) et maintenez-le sur de la glace. Centrifuger (380 x g, 10 min, 4 °C), aspirer le surnageant et remettre complètement la pastille en suspension (jusqu’à 10 x 106 cellules/mL dans le CSM). Filtrer la suspension à travers une crépine à cellules de 35 μm.
- Fixez la suspension cellulaire avec une solution mère de PFA filtrée (16 %) et pipetez de haut en bas (sous une hotte) pour atteindre la concentration finale de PFA (1,6 %) (par exemple, ajoutez 100 μL de PFA à 16 % à 900 μL de suspension cellulaire). Vortex pendant 30 s, incuber 10 min sur glace et laver deux fois avec 2 mL de CSM par centrifugation (800 x g, 5 min, RT).
REMARQUE : À ce stade, les échantillons peuvent être soit congelés sur de la glace sèche et stockés à -80 °C, soit utilisés directement pour la coloration des anticorps. Si les cellules doivent être congelées, transférez-les dans des tubes de polypropylène de 5 ml, car le polystyrène peut se fissurer à basse température. Si des cellules fraîchement fixées sont utilisées pour la coloration, passez à l’étape 8.2.
8. Coloration avec des anticorps conjugués aux métaux
ATTENTION : Le méthanol (MeOH) est hautement inflammable et corrosif pour les voies respiratoires. Lisez la FDS avant de la manipuler. Portez un équipement de protection individuelle et manipulez cette substance sous une hotte. Manipulez les déchets conformément aux réglementations locales.
REMARQUE : Le tableau 2 présente la liste des anticorps (Ab) ciblant les marqueurs de surface et les marqueurs intracellulaires.
Conjugaison des anticorps : La plupart des anticorps utilisés dans ce protocole ont été conjugués à l’interne car ils n’étaient pas disponibles dans le commerce. Des protocoles de conjugaison des métaux des anticorps ont déjà été publiés, et des kits de conjugaison sont maintenant disponibles dans le commerce37,38. L’immunoglobuline de type G (IgG) est compatible avec les protocoles de conjugaison disponibles. Il est très important que la formulation d’anticorps utilisée pour la conjugaison des métaux soit exempte de protéines porteuses contenant de la cystéine (p. ex., l’albumine sérique bovine (BSA)), qui peuvent affecter l’efficacité de la conjugaison en rivalisant pour les groupes maléimides libres du polymère, et peuvent interférer avec la quantification de l’anticorps conjugué aux métaux. La teneur en cystéine de la gélatine est beaucoup plus faible que celle de la BSA. Cependant, il est recommandé que si la formulation de l’anticorps contient des protéines porteuses, ces protéines soient éliminées avant la conjugaison. Il est désormais possible de demander des anticorps sans BSA ni gélatine auprès du fabricant. Les conservateurs à petites molécules (par exemple, l’azoture de sodium, le glycérol et le tréhalose) sont compatibles avec les protocoles de conjugaison des métaux37,38.
Titrage des anticorps : Après chaque conjugaison métallique, les anticorps doivent être titrés pour déterminer la concentration optimale d’anticorps qui fournit le rapport signal/bruit maximal. Pour le titrage des anticorps, effectuez une dilution en série en deux étapes et colorez les deux échantillons connus pour exprimer (par exemple, cellules musculaires, contrôles positifs) et manquer (contrôles négatifs) la protéine d’intérêt 19,21,37,38.
Préparez une solution de travail fraîche de Cell-ID Intercalator-Ir (stock = 500 μM ; solution d’intercalator-ir) en diluant le stock à 0,1 μM dans PBS/1,6 % de PFA.
- Décongeler les échantillons congelés pendant 5 min à l’heure RT et centrifuger (800 x g, 1 min, RT). Laver avec 2 mL de CSM par centrifugation (800 x g, 5 min, RT).
- Préparez un mélange de coloration d’anticorps de surface (Ab) 2,5x dans CSM. Aspirer le surnageant à ~60 μL et remettre la pastille en suspension à fond. Teindre les cellules en ajoutant 40 μL de mélange de coloration Ab de surface et en l’incubant pendant 1 h à RT.
- Feuilletez les échantillons toutes les 20 minutes pour les mélanger. Laver deux fois avec 1 mL de CSM par centrifugation (800 x g, 5 min, RT). Aspirez le surnageant et secouez la pastille.
REMARQUE : Si aucune coloration intracellulaire n’est nécessaire, la perméabilisation au méthanol n’est pas nécessaire. Cependant, si seule une coloration de surface est effectuée, la solution intercalator-ir doit être diluée dans un tampon contenant un agent de perméabilisation (par exemple, Maxpar fix et tampon permanent) pour pénétrer dans la membrane nucléaire. Passez à l’étape 8.5 et diluez le Cell-ID Intercalator-Ir dans un tampon contenant un agent perméabilisant et 1,6 % de PFA. Sinon, continuez avec la coloration intracellulaire ci-dessous.
ATTENTION : L’iridium est dangereux et des précautions pour une manipulation en toute sécurité doivent être prises. L’iridium est inflammable et irritant pour les yeux et la peau. Évitez de créer et de respirer de la poussière ou des fumées. Évitez tout contact avec la peau et les yeux. Cependant, le mélange de solution intercalator-ir est fourni à une concentration de <1 % dans l’eau, ce qui est considéré comme non dangereux selon le Système général harmonisé (SGH) de classification et d’étiquetage des produits chimiques. Lisez la FDS de Standard Biotools avant de la manipuler. Un équipement de protection individuelle doit être porté pendant la manipulation. Les matériaux qui ont été en contact avec la solution intercalator-ir doivent être jetés conformément aux réglementations de sécurité locales. - Pour perméabiliser les cellules, ajoutez 0,5 mL de MeOH glacé goutte à goutte tout en tourbillonnant. Incuber 15 min sur glace sous une hotte. Centrifuger (800 x g, 5 min, RT) puis laver deux fois avec 1 mL de CSM par centrifugation (800 x g, 5 min, RT). Après le dernier lavage, aspirez le surnageant à ~60 μL et remettez complètement la pastille en suspension.
- Préparez un mélange de coloration Ab intracellulaire 2,5x dans CSM. Colorez les cellules en ajoutant 40 μL de mélange de coloration Ab intracellulaire et laissez incuber pendant 1 h à RT. Feuilletez les échantillons toutes les 20 minutes pour mélanger.
- Laver deux fois avec 1 mL de CSM par centrifugation (800 x g, 5 min, RT). Aspirez le surnageant et secouez la pastille. Remettre les échantillons en suspension dans 0,5 mL de solution d’intercalateur-iridium (tableau 1) et de vortex. Incuber les échantillons pendant 1 h à RT ou toute la nuit (O/N) à 4 °C (voir note ci-dessous).
REMARQUE : Les échantillons peuvent être conservés dans une solution d’intercalateur-iridium à 4 °C pendant une période allant jusqu’à 48 h. Le protocole de coloration utilisé dans cette étude a été développé sur la base des travaux pionniers du laboratoire Nolan{C}23. Il diffère des protocoles Standard Biotools, car (i) des tampons préparés en interne sont utilisés pour le lavage et la coloration, (ii) les cellules sont fixées avant la coloration de surface, et (iii) les cellules sont perméabilisées au méthanol pour la coloration intracellulaire avec des anticorps contre les facteurs de transcription ou les molécules de signalisation. Les chercheurs qui élaborent un nouveau protocole doivent tester minutieusement la compatibilité de leur panel d’anticorps avec le méthanol en tant qu’agent de perméabilisation. Lors de l’ajout de nouveaux anticorps à un panel, il est recommandé de tester la spécificité des anticorps par cytométrie en flux à l’aide d’échantillons de contrôle positifs et négatifs avant la conjugaison et le titrage des métaux.
9. Préparation de l’échantillon pour le chargement dans le cytomètre de masse
REMARQUE : Les granulés de cellules sont très lâches lorsqu’ils sont dans le tampon CAS (Table of Materials). Lors des lavages avec tampon CAS, ne pas aspirer à sec. Au lieu de cela, conservez un volume résiduel comme décrit ci-dessous.
- Vortex et centrifugeuse (800 x g, 10 min, RT) les échantillons. Videz le surnageant (poignée comme déchet PFA) et vortex.
- Laver avec 1 mL de CSM par centrifugation (800 x g, 10 min, RT). Aspirez le surnageant et secouez la pastille.
- Laver avec 1 mL de tampon CAS par centrifugation (800 x g, 10 min, RT). Aspirer à ~200 μL. Vortex et ajouter 1 mL de tampon CAS. Prélever 5 μL d’aliquote pour la numération cellulaire. Centrifuger (800 x g, 10 min, RT) et aspirer soigneusement à ≤50 μL. Ne dérangez pas le granulé.
- Remettre la pastille en suspension à 1–2 x 106 cellules/mL dans le tampon CAS et ajouter des billes d’étalonnage (1x ; Table des matériaux) jusqu’à une concentration finale de 0,1x (p. ex., ajouter 100 μL de billes d’étalonnage 1x à 900 μL de suspension cellulaire).
- Chargez l’échantillon dans le cytomètre de masse et collectez les données en utilisant un débit de 400 à 500 cellules/s.
- Effectuer la normalisation des données des fichiers FCS à l’aide du logiciel CyTOF ou des outils de normalisation précédemment développés39.
REMARQUE : Le fonctionnement des cytomètres de masse CyTOF est spécifique à l’instrument40,41. Il est recommandé de consulter le manuel d’utilisation de CyTOF avant l’utilisation. Les billes d’étalonnage sont des étalons de normalisation en polystyrène incorporé dans du métal contenant des concentrations connues des isotopes métalliques cérium (140/142Ce), europium (151/153Eu), holmium (165Ho) et lutécium (175/176Lu). Les billes d’étalonnage permettent de contrôler la sensibilité de la machine, qui peut varier au fil du temps, principalement en raison de l’accumulation de matière biologique et des variations de l’ionisation du plasma au fil du temps.
10. Analyse des données CyTOF
REMARQUE : Pour l’analyse en aval, les fichiers FCS normalisés peuvent être analysés localement ou téléchargés vers des solutions logicielles basées sur le cloud telles que Cytobank, Cell Engine, OMIQ ou FCS Express42.
- Concaténez des fichiers FCS individuels pour chaque échantillon dans un seul fichier, si nécessaire.
- Identifiez les cellules uniques en tenant compte des événements positifs de l’intercalateur d’iridium, ce qui permet de distinguer les cellules nucléées simples des débris ou des doublets.
- Gate pour les cellules vivantes en sélectionnant les événements négatifs au cisplatine. Le cisplatine se lie de manière covalente aux protéines cellulaires et marque les cellules mourantes et mortes dont les membranes sont compromises dans une plus grande mesure que les cellules vivantes43.
- Repérez la population d’intérêt, par exemple, le compartiment myogénique (Live/CD45-/CD31-/Sca1-/α7integrin+/CD9+) (Figure 2A) et quantifiez la proportion relative de cellules souches et de cellules progénitrices. Cette approche nécessite une connaissance préalable de la surface cellulaire ou de l’expression des marqueurs intracellulaires pour définir les populations individuelles.
- Effectuer une analyse de grande dimension qui permettra d’identifier des sous-ensembles de cellules rares jusqu’alors non reconnus au sein d’une population complexe, exporter la population d’intérêt et utiliser des algorithmes de clustering qui ont été développés spécifiquement pour l’analyse des données CyTOF44.
REMARQUE : Des travaux antérieurs ont utilisé l’algorithme X-shift, qui utilise l’estimation pondérée de la densité du voisin le plus proche (kNN-DE) dans l’espace de grande dimension pour effectuer un clustering non supervisé basé sur des paramètres définis19. Le décalage X s’est avéré très efficace pour identifier des populations de cellules rares45. - Pour l’analyse X-shift, téléchargez le progiciel vortex (à partir de la page GitHub du laboratoire Nolan [https://github.com/nolanlab/vortex]) et Java 64-bit46. Vous trouverez les instructions ici : https://github.com/nolanlab/vortex/wiki/Getting-Started.
- Téléchargez les populations de cellules exportées dans une base de données locale et définissez les paramètres de clustering. Parmi les paramètres définis par l’utilisateur figurent (i) les marqueurs utilisés pour le clustering, (ii) la plage de valeurs k (par exemple, 5 à 150) et (iii) le nombre d’étapes de clustering.
REMARQUE : Des travaux antérieurs ont utilisé, comme marqueurs de regroupement, une combinaison de marqueurs de surface connus pour être exprimés dans les cellules souches musculaires, de nouveaux marqueurs de surface identifiés dans un criblage cytométrique en flux à haut débit de cellules musculaires et de myoblastes, et de facteurs de transcription myogéniques (TF) connus pour définir différentes étapes de la myogenèse. Cette approche a permis d’identifier deux marqueurs de surface cellulaire, CD9 et CD104, dont le modèle de co-expression distingue des populations de cellules progénitrices jusqu’alors non reconnues19. Le choix minutieux des marqueurs de regroupement permettra au chercheur de répondre à des questions de recherche spécifiques. Par exemple, si l’identification et l’analyse au fil du temps de sous-ensembles de cellules en division sont souhaitées, il est recommandé d’inclure IdU comme l’un des marqueurs de clustering. X-shift peut effectuer un clustering à plusieurs valeurs k et identifier automatiquement, en calculant le « point de coude », la valeur k qui aboutit au nombre optimal de clusters, évitant ainsi le sous-clustering ou la sur-fragmentation. Il est recommandé de calculer le point de coude et d’utiliser l’analyse de clustering définie par la valeur k optimale pour la visualisation de données et les applications en aval. - Pour visualiser les relations spatiales entre les populations cellulaires au sein des grappes à décalage X, effectuez une mise en page dirigée par la force, qui générera une carte 2D où la distance entre les grappes indique leur similitude dans l’expression des marqueurs dans l’espace phénotypique de grande dimension. En coloriant la carte à l’aide d’un marqueur à la fois, il est possible de découvrir de nouvelles populations cellulaires et de suivre leur dynamique. Il est recommandé d’effectuer une quantification ultérieure par contrôle manuel dans un logiciel tel que Cytobank et Cell Engine.
- Effectuez une analyse de carte thermique pour quantifier l’expression dynamique de plusieurs protéines de surface et intracellulaires tout au long du temps et révéler de nouvelles tendances.
REMARQUE : Lors de l’exécution d’une course au temps de blessure, il est possible de regrouper les données de tous les points temporels pour générer une carte de régénération. De plus, il est possible de disséquer une telle carte en points temporels individuels pour suivre la dynamique cellulaire et moléculaire de la régénération au fil du temps19.
11. Coloration avec des anticorps conjugués aux fluorophores pour le FACS
REMARQUE : Les cellules utilisées pour les contrôles unicolores non colorés et les contrôles de fluorescence moins un (FMO) peuvent provenir de l’ensemble TA et GA d’une souris supplémentaire si disponible. Alternativement, le quadriceps (muscle antérieur supérieur de la cuisse) peut être disséqué et digéré en une suspension unicellulaire, en suivant la même procédure que pour le TA+GA défini ci-dessus et utilisé pour les contrôles. Préparez un tampon FACS (PBS, 2,5 % de sérum de chèvre, 2 mM d’EDTA), filtrez à travers une membrane PES dans un récipient en polystyrène et conservez-le sur de la glace. La mémoire tampon FACS peut être stockée à 4 °C jusqu’à 1 mois. Une liste des anticorps utilisés pour le FACS se trouve dans le tableau 3.
- Préparez un mélange de lignées. Colorer chaque témoin dans 50 μL (contenant environ 3 à 5 x 105 cellules). Calculez la quantité de mélange de lignage nécessaire. Faites un excès de volume de 20 à 30 %. La concentration de base des anticorps utilisés pour le mélange de lignées est de 0,2 mg/mL.
- Dans un tube de 0,5 mL, ajouter l’APC-Cy7 anti-CD45, l’APC-Cy7 anti-CD31, l’APC-Cy7 anti-Sca1 et l’APC-Cy7 anti-CD11b pour atteindre des concentrations finales de 1 μg/mL, 2,5 μg/mL, 2,5 μg/mL et 0,63 μg/mL, respectivement.
- Préparez un mélange pour toutes les taches. Colorer chaque échantillon entièrement coloré dans 500 μL (contenant environ 3 à 5 x 106 cellules). Faire 10% d’excès de volume de mélange toutes teintures.
- Dans un tube de 1,5 mL, ajouter les anticorps du mélange de lignées comme ci-dessus + l’anti-α7 intégrine PE, l’APC anti-CD9 et l’anti-CD104 FITC pour atteindre des concentrations finales de 2 μg/mL, 1,2 μg/mL et 3 μg/mL, respectivement.
- Préparer le tampon FACS + DAPI : Pour 10 mL de tampon FACS, ajouter 1 μL de DAPI (stock = 1 mg/mL) pour atteindre une concentration finale de 100 ng/mL de DAPI.
MISE EN GARDE : Le DAPI (dichlorhydrate de 4',6-diamidino-2-phénylindole) est classé comme un irritant possible pour la peau et les voies respiratoires. Cependant, la solution DAPI à une concentration de <1 % est considérée comme non dangereuse, selon le SGH. Lisez la FDS avant de la manipuler. Un équipement de protection individuelle doit être porté pendant la manipulation. Les matériaux qui ont été en contact avec le DAPI doivent être jetés conformément aux réglementations de sécurité locales. - Remise en suspension des cellules de la section 6 : Dissection et dissociation des muscles squelettiques :
- Pour les échantillons entièrement colorés, remettre en suspension les ensembles TA et GA d’une seule souris dans 500 μL de tampon FACS et les transférer dans des tubes de 15 mL pour la coloration.
- Pour les contrôles, utilisez des séries TA et GA ou des séries de quadriceps. Si vous utilisez 1 souris pour les commandes, résuspendez les cellules dans 750 μL de tampon FACS. S’il s’agit de plusieurs souris, remettre en suspension chaque ensemble TA et GA ou quadriceps dans 500 μL de tampon FACS et combiner les ensembles en un seul échantillon de 1 mL en prélevant une fraction de chaque échantillon. Ajouter 50 μL de cellules témoins (3 à 5 x 105 cellules) dans des tubes en polypropylène de 5 mL pour la coloration.
- Colorer comme décrit dans le tableau 4 pendant 45 min à 4 °C dans l’obscurité.
- Lavez tous les échantillons tachés en ajoutant 5 ml de tampon FACS. Lavez les commandes en ajoutant 1 ml de tampon FACS et centrifugez (380 x g, 10 min, 4 °C).
- Remettez en suspension les échantillons entièrement colorés dans 1 mL de tampon FACS + DAPI. Remettre les commandes en suspension dans 300 μL de tampon FACS avec ou sans DAPI.
- Maintenir les cellules à 4 °C dans l’obscurité jusqu’au tri sur un cytomètre en flux avec 4 lasers (405 nm, 488 nm, 561 nm, 633 nm).
- Exécutez les commandes et les échantillons sur le trieur et créez des portes de tri à l’aide du logiciel approprié. Pour les commandes non tachées/monochromes et les commandes FMO, enregistrez respectivement 1 x 104 et 0,3 à 1 x 105 . Pour les échantillons entièrement colorés, enregistrez jusqu’à 1 x 106 événements. Triez tous les échantillons entièrement colorés, ou autant que nécessaire, en fonction des exigences de test en aval. Pour le cytomètre en flux utilisé ici (FACSAria III), trier en mode Pureté à l’aide d’une buse de 70 mm.
- Voir la figure 4 pour la stratégie de contrôle.
- Utilisez les commandes non colorées et unicolores pour régler la tension de tous les détecteurs.
- Utilisez les commandes monochromes pour configurer la matrice de compensation.
- Utilisez les commandes FMO pour établir les portes de tri.
- Des populations distinctes de cellules souches musculaires et de cellules progénitrices sont triées dans un tampon FACS glacial.
- Centrifuger (380 x g, 10 min, 4 °C) les populations cellulaires. Résuspendez dans la mémoire tampon appropriée, comptez et poursuivez l’analyse en aval.
Access restricted. Please log in or start a trial to view this content.
Résultats
Nous présentons ici un aperçu du dispositif expérimental d’utilisation de cette approche combinée, qui comprend : (i) l’analyse CyTOF de haute dimension de l’évolution temporelle d’une lésion aiguë par injection de notexine pour étudier la dynamique cellulaire et moléculaire des cellules souches et progénitrices dans le muscle squelettique (Figure 1, schéma du haut) ; et (ii) FACS de cellules souches et progénitrices à l’aide de deux m...
Access restricted. Please log in or start a trial to view this content.
Discussion
La régénération des muscles squelettiques est un processus dynamique qui repose sur la fonction des cellules souches adultes. Alors que des études antérieures se sont concentrées sur le rôle des cellules souches musculaires lors de la régénération, leur descendance in vivo a été sous-étudiée, principalement en raison d’un manque d’outils pour identifier et isoler ces populations cellulaires 15,16,17,18....
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous remercions les membres de la plateforme FACS du Département de biomédecine de l’Université d’Aarhus pour leur soutien technique. Nous remercions Alexander Schmitz, directeur de l’unité de cytométrie de masse du Département de biomédecine, pour ses discussions et son soutien. Les illustrations scientifiques ont été créées à l’aide de Biorender.com. Ce travail a été financé par une bourse de démarrage de l’Aarhus Universitets Forskningsfond (AUFF) et une subvention Start Package (0071113) de la Fondation Novo Nordisk à E.P.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Fisher Scientific | 07-200-886 | |
| 20 G needle | KDM | KD-fine 900123 | |
| 28 G, 0.5 mL insulin syringe | BD | 329461 | |
| 29 G, 0.3 mL insulin syringe | BD | 324702 | |
| 3 mL syringes | Terumo medical | MDSS03SE | |
| 40 µm cell strainers | Fisher Scientific | 11587522 | |
| 5 mL polypropylene tubes | Fisher Scientific | 352002 | |
| 5 mL polystyrene test tubes with 35 µm cell strainer | Falcon | 352235 | |
| 5 mL syringes | Terumo medical | SS05LE1 | |
| 50 mL centrifuge tube | Fisher Scientific | 05-539-13 | |
| 5-Iodo-2-deoxyuridine (IdU) | Merck | I7125-5g | |
| anti-CD104 FITC (clone: 346-11A) | Biolegend | 123605 | Stock = 0.5 mg/mL |
| anti-CD11b APC-Cy7 (Clone: M1/70) | Biolegend | 101226 | Stock = 0.2 mg/mL |
| anti-CD31 APC-Cy7 (clone: 390) | Biolegend | 102440 | Stock = 0.2 mg/mL |
| anti-CD45 APC-Cy7 (Clone: 30-F11) | Biolegend | 103116 | Stock = 0.2 mg/mL |
| anti-CD9 APC (clone: KMC8) | ThermoFisher Scientific | 17-0091-82 | Stock = 0.2 mg/mL |
| anti-Sca1 (Ly6A/E) APC-Cy7 (clone: D7) | Biolegend | 108126 | Stock = 0.2 mg/mL |
| anti-α7 integrin PE (clone: R2F2)) | UBC AbLab | 67-0010-05 | Stock = 1 mg/mL |
| BD FACS Aria III (4 laser) instrument | BD Biosciences | N/A | 405, 488, 561, and 633 nm laser |
| Bovine Serum Albumin | Sigma Aldrich | A7030-50G | |
| Buprenorphine 0.3 mg/mL | Ceva | Vnr 054594 | |
| CD104 (Clone: 346-11A) | BD Biosciences | 553745 | Dy162; In-house conjugated |
| CD106/VCAM-1 (Clone: 429 MVCAM.A) | Biolegend | 105701 | Er170; In-house conjugated |
| CD11b (Clone: M1/70) | BD Biosciences | 553308 | Nd148; In-house conjugated |
| CD29/Integrin β1 (Clone: 9EG7) | BD Biosciences | 553715 | Tm169; In-house conjugated |
| CD31 (Clone: MEC 13.3) | BD Biosciences | 557355 | Sm154; In-house conjugated |
| CD34 (Clone: RAM34) | BD Biosciences | 551387 | Lu175; In-house conjugated |
| CD44 (Clone: IM7) | BD Biosciences | 550538 | Yb171; In-house conjugated |
| CD45 (Clone: MEC 30-F11) | BD Biosciences | 550539 | Sm147; In-house conjugated |
| CD9 (Clone: KMC8) | Thermo Fisher Scientific | 14-0091-85 | Yb174; In-house conjugated |
| CD90.2/Thy1.2 (Clone: 30-H12) | BD Biosciences | 553009 | Nd144; In-house conjugated |
| CD98 (Clone: H202-141) | BD Biosciences | 557479 | Pr141; In-house conjugated |
| Cell Acquisition Solution/Maxpar CAS-buffer | Standard Biotools | 201240 | |
| Cell-ID Intercalator-Iridium | Standard Biotools | 201192B | cationic nucleic acid intercalator |
| Cisplatin | Merck | P4394 | Pt195 |
| Cisplatin (cis-Diammineplatinum(II) dichloride) | Merck | P4394 | |
| Clear 1.5 mL tube | Fisher Scientific | 11926955 | |
| Collagenase, Type II | Worthington Biochemical Corporation | LS004177 | |
| Counting chamber | Merck | BR718620-1EA | |
| CXCR4/SDF1 (Clone: 2B11/CXCR4 ) | BD Biosciences | 551852 | Gd158; In-house conjugated |
| DAPI (1 mg/mL) | BD Biosciences | 564907 | |
| Dark 1.5 mL tube | Fisher Scientific | 15386548 | |
| Dispase II | Thermo Fisher Scientific | 17105041 | |
| Dissection Scissors | Fine Science Tools | 14568-09 | |
| DMEM (low glucose, with pyruvate) | Thermo Fisher Scientific | 11885-092 | |
| EDTA (Ethylenediaminetetraacetic acid disodium salt) | Merck | E5134 | Na2EDTA-2H20 |
| EQ Four Element Calibration Beads (EQ beads) | Standard Biotools | 201078 | Calibration beads |
| Fetal Bovine Serum, qualified, Brazil origin | Thermo Fisher Scientific | 10270106 | |
| Forceps Dumont #5SF | Fine Science Tools | 11252-00 | |
| Forceps Dumont #7 | Hounisen.com | 1606.3350 | |
| Goat serum | Thermo Fisher Scientific | 16210-072 | |
| Helios CyTOF system | Standard Biotools | N/A | |
| Horse Serum, heat inactivated, New Zealand origin | Thermo Fisher Scientific | 26-050-088 | |
| IdU | Merck | I7125 | I127 |
| Iridium-Intercalator | Standard Biotools | 201240 | Ir191/193 |
| Isoflurane/Attane Vet | ScanVet | Vnr 055226 | |
| Methanol | Fisher Scientific | M/3900/17 | |
| Myf5 (Clone: C-20) | Santa Cruz Biotechnology | Sc-302 | Yb173; In-house conjugated |
| MyoD (Clone: 5.8A) | BD Biosciences | 554130 | Dy164; In-house conjugated |
| MyoG (Clone: F5D) | BD Biosciences | 556358 | Gd160; In-house conjugated |
| Nalgene Rapid-Flow Sterile Disposable Bottle Top 0.20 μM PES Filters | Thermo Fisher Scientific | 595-4520 | |
| Notexin | Latoxan | L8104 | Resuspend to 50 µg/ml in sterile PBS. Keep stocks (e.g. 50 µl) at -20 °C |
| Nutrient mixture F-10 (Ham's) | Thermo Fisher Scientific | 31550031 | |
| pAkt (Clone: D9E) | Standard Biotools | 3152005A | Sm152 |
| Pax7 (Clone: PAX7) | Santa Cruz Biotechnology | Sc-81648 | Eu153; In-house conjugated |
| Penicillin-Streptomycin (10,000 U/mL) (Pen/Strep) | Thermo Fisher Scientific | 15140122 | |
| PES Filter Units 0.20 μM | Fisher Scientific | 15913307 | |
| PES Syringe Filter | Fisher Scientific | 15206869 | |
| Petri dish | Sarstedt | 82.1472.001 | |
| PFA 16% EM grade | MP Biomedicals | 219998320 | |
| Potassium chloride (KCl) | Fisher Scientific | 10375810 | |
| Potassium phosphate, monobasic, anhydrous (KH2PO4) | Fisher Scientific | 10573181 | |
| pRb (Clone: J112-906) | Standard Biotools | 3166011A | Er166 |
| pS6 kinase (Clone: N7-548) | Standard Biotools | 3172008A | Yb172 |
| Sca-1 (Clone: E13-161.7) | BD Biosciences | 553333 | Nd142; In-house conjugated |
| Sodium Azide | Sigma Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | 10553515 | |
| Sodium phosphate, dibasic, heptahydrate (Na2HPO4-6H2O) | Merck | S9390 | |
| Sterile saline solution 0.9% | Fresenius | B306414/02 | |
| α7 integrin (Clone: 3C12) | MBL international | K0046-3 | Ho165; In-house conjugated |
Références
- Mukund, K., Subramaniam, S. Skeletal muscle: A review of molecular structure and function in health and disease. Wiley Interdiscip Rev Syst Biol Med. 12 (1), e1462(2020).
- Feige, P., Brun, C. E., Ritso, M., Rudnicki, M. A. Orienting muscle stem cells for regeneration in homeostasis, aging, and disease. Cell Stem Cell. 23 (5), 653-664 (2018).
- Mauro, A. Satellite cell of skeletal muscle fibers. J Biophys Biochem Cytol. 9 (2), 493-495 (1961).
- Seale, P., et al. Pax7 is required for the specification of myogenic satellite cells. Cell. 102 (6), 777-786 (2000).
- Fuchs, E., Blau, H. M. Tissue stem cells: Architects of their niches. Cell Stem Cell. 27 (4), 532-556 (2020).
- Hernández-hernández, J. M., et al. The myogenic regulatory factors, determinants of muscle development, cell identity and regeneration. Semin Cell Dev Biol. 72, 10-18 (2017).
- Zammit, P. S. Function of the myogenic regulatory factors Myf5, MyoD, Myogenin and MRF4 in skeletal muscle, satellite cells and regenerative myogenesis. Semin Cell Dev Biol. 72, 19-32 (2017).
- Sabourin, L. A. The molecular regulation of myogenesis. Clin Genet. 57 (1), 16-25 (2000).
- Cooper, R. N., et al. In vivo satellite cell activation via Myf5 and MyoD in regenerating mouse skeletal muscle. J Cell Sci. 112 (17), 2895-2901 (1999).
- Rudnicki, M. A., Jaenisch, R. The MyoD family of transcription factors and skeletal myogenesis. Bioessays. 17 (3), 203-209 (1995).
- Braun, T., Arnold, H. H. Inactivation of Myf-6 and Myf-5 genes in mice leads to alterations in skeletal muscle development. EMBO J. 14 (6), 1176-1186 (1995).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Braun, T., et al. MyoD expression marks the onset of skeletal myogenesis in Myf-5 mutant mice. Development. 120 (11), 3083-3092 (1994).
- Rudnicki, M. A., et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell. 75 (7), 1351-1359 (1993).
- Montarras, D., et al. Developmental biology: Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Sacco, A., Doyonnas, R., Kraft, P., Vitorovic, S., Blau, H. M. Self-renewal and expansion of single transplanted muscle stem cells. Nature. 456 (7221), 502-506 (2008).
- Cerletti, M., et al. Highly efficient, functional engraftment of skeletal muscle stem cells in dystrophic muscles. Cell. 134 (1), 37-47 (2008).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nat Protoc. 10 (10), 1612-1624 (2015).
- Porpiglia, E., et al. High-resolution myogenic lineage mapping by single-cell mass cytometry. Nat Cell Biol. 19 (5), 558-567 (2017).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry Part A. 81 (7), 552-566 (2012).
- Hartmann, F. J., et al. Mass Cytometry: Methods and Protocols. , Springer, Humana. New York, NY. (2019).
- Devine, R. D., Behbehani, G. K. Use of the pyrimidine analog, 5-iodo-2'-deoxyuridine (IdU) with cell cycle markers to establish cell cycle phases in a mass cytometry platform. J Vis Exp. (176), e60556(2021).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Nag, A. C., Foster, J. D. Myogenesis in adult mammalian skeletal muscle in vitro. J Anat. 132, Pt 1 1-18 (1981).
- Le Moigne, A., et al. Characterization of myogenesis from adult satellite cells cultured in vitro). Int J Dev Biol. 34, 171-180 (1990).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Chu, C., Cogswell, J., Kohtz, D. S. MyoD functions as a transcriptional repressor in proliferating myoblasts. J Biol Chem. 272 (6), 3145-3148 (1997).
- Shah, B., Hyde-Dunn, J., Jones, G. E. Proliferation of murine myoblasts as measured by bromodeoxyuridine incorporation. Methods in Mol Biol. 75, 349-355 (1997).
- Springer, M. L., Blau, H. M. High-efficiency retroviral infection of primary myoblasts. Somat Cell Mol Genet. 23 (3), 203-209 (1997).
- Rando, T. A., Blau, H. M. Primary mouse myoblast purification, characterization, and transplantation for cell-mediated gene therapy. J Cell Biol. 125 (6), 1275-1287 (1994).
- Springer, M. L., Rando, T. A., Blau, H. M. Gene delivery to muscle. Curr Protoc Hum Genet. , Chapter 13, Unit13.4 (2002).
- Cull-Candy, S. G., Fohlman, J., Gustavsson, D., Lullmann-Rauch, R., Thesleff, S. The effects of taipoxin and notexin on the function and fine structure of the murine neuromuscular junction. Neuroscience. 1 (3), 175-180 (1976).
- Francis, B., John, T. R., Seebart, C., Kaiser, I. I. New toxins from the venom of the common tiger snake (Notechis scutatus scutatus). Toxicon. 29 (1), 85-96 (1991).
- Navarro, K. L., Huss, M., Smith, J. C., Sharp, P., Marx, J. O., Pacharinsak, C. Mouse Anesthesia: The Art and Science. ILAR Journal. 62, Issue 1-2, 2021 238-273 (2021).
- Langford, D., Bailey, A., Chanda, M., et al. Coding of facial expressions of pain in the laboratory mouse. Nat Methods. 7, 447-449 (2010).
- Matsumiya, L. C., Sorge, R. E., Sotocinal, S. G., Tabaka, J. M., Wieskopf, J. S., Zaloum, A., King, O. D., Mogil, J. S. Using the Mouse Grimace Scale to reevaluate the efficacy of postoperative analgesics in laboratory mice. J Am Assoc Lab Anim Sci. 2012 (1), PMID: 22330867; PMCID: PMC3276965 42-49 (2012).
- Gonzalez, V. D., et al. High-grade serous ovarian tumor cells modulate NK cell function to create an immune-tolerant microenvironment. Cell Rep. 36 (9), 109632(2021).
- Delgado-Gonzalez, A., et al. Measuring trogocytosis between ovarian tumor and natural killer cells. STAR Protoc. 3 (2), 101425(2022).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry Part A. 83 (5), 483-494 (2013).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. J Vis Exp. (69), e4398(2012).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample preparation for mass cytometry analysis. J Vis Exp. (122), e54394(2017).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Curr Protoc Cytom. , Chapter 10., Uni10.17 (2010).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry Part A. 81 (6), 467-475 (2012).
- Kimball, A. K., et al. A beginner's guide to analyzing and visualizing mass cytometry data. J Immunol. 200 (1), 3-22 (2018).
- Weber, L. M., Robinson, M. D. Comparison of clustering methods for high-dimensional single-cell flow and mass cytometry data. Cytometry Part A. 89 (12), 1084-1096 (2016).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nat Methods. 13 (6), 493-496 (2016).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Anal Chem. 80 (7), 2539-2547 (2008).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nat Commun. 12 (1), 692(2021).
- de Morree, A., et al. Staufen1 inhibits MyoD translation to actively maintain muscle stem cell quiescence. Proc Natl Acad Sci U S A. 114 (43), E8996-E9005 (2017).
- Luo, D., et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c. Proc Natl Acad Sci U S A. 114 (15), E3071-E3080 (2017).
- Wersto, R. P., et al. Doublet discrimination in DNA cell-cycle analysis. Cytometry. 46 (5), 296-306 (2001).
- Porpiglia, E., Blau, H. M. Plasticity of muscle stem cells in homeostasis and aging. Curr Opin Genet Dev. 77, 101999(2022).
- Porpiglia, E., et al. Elevated CD47 is a hallmark of dysfunctional aged muscle stem cells that can be targeted to augment regeneration. Cell Stem Cell. 29 (12), e1658 1653-1668 (2022).
- Brunet, A., Goodell, M. A., Rando, T. A. Ageing and rejuvenation of tissue stem cells and their niches. Nat Rev Mol Cell Biol. 24 (1), 45-62 (2022).
- Danielli, S. G., et al. Single-cell profiling of alveolar rhabdomyosarcoma reveals RAS pathway inhibitors as cell-fate hijackers with therapeutic relevance. Sci Adv. 9 (6), eade9238 (2023).
- de Morree, A., Rando, T. A. Regulation of adult stem cell quiescence and its functions in the maintenance of tissue integrity. Nat Rev Mol Cell Biol. 24 (5), 334-354 (2023).
- Yucel, N., et al. Glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell function. Cell Rep. 27 (13), 3939-3955 (2019).
- Tierney, M. T., Sacco, A. Inducing and evaluating skeletal muscle injury by notexin and barium chloride. Methods Mol Biol. 1460, 53-60 (2016).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198(2016).
- Call, J. A., Lowe, D. A. Eccentric contraction-induced muscle injury: Reproducible, quantitative, physiological models to impair skeletal muscle's capacity to generate force. Methods Mol Biol. 1460, 3-18 (2016).
- Garry, G. A., Antony, M. L., Garry, D. J. Cardiotoxin Induced Injury and Skeletal Muscle Regeneration. Methods Mol Biol. 1460, 61-71 (2016).
- Le, G., Lowe, D. A., Kyba, M. Freeze injury of the tibialis anterior muscle. Methods Mol Biol. 1460, 33-41 (2016).
- Borok, M., et al. Progressive and coordinated mobilization of the skeletal muscle niche throughout tissue repair revealed by single-cell proteomic analysis. Cells. 10 (4), 744(2021).
- Petrilli, L. L., et al. High-dimensional single-cell quantitative profiling of skeletal muscle cell population dynamics during regeneration. Cells. 9 (7), 1723(2020).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Mol Cell. 74 (3), e606 609-621 (2019).
- Hartmann, F. J., et al. Scalable conjugation and characterization of immunoglobulins with stable mass isotope reporters for single-cell mass cytometry analysis. Methods Mol Biol. 1989, 55-81 (2019).
- Frimand, Z., Das Barman, S., Kjær, T. R., Porpiglia, E., de Morrée, A. Isolation of quiescent stem cell populations from individual skeletal muscles. J Vis Exp. (190), e64557(2022).
- Krutzik, P. O., Nolan, G. P. Intracellular phospho-protein staining techniques for flow cytometry: monitoring single cell signaling events. Cytometry A. 55 (2), 61-70 (2003).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat Biotechnol. 30 (9), 858-867 (2012).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Curr Protoc Immunol. , Chapter 8 11-20 (2012).
- Krutzik, P. O., Clutter, M. R., Nolan, G. P. Coordinate analysis of murine immune cell surface markers and intracellular phosphoproteins by flow cytometry. J Immunol. 175 (4), 2357-2365 (2005).
- Krutzik, P. O., Irish, J. M., Nolan, G. P., Perez, O. D. Analysis of protein phosphorylation and cellular signaling events by flow cytometry: techniques and clinical applications. Clin Immunol. 110 (3), 206-221 (2004).
- Han, G., Spitzer, M. H., Bendall, S. C., Fantl, W. J., Nolan, G. P. Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry. Nat Protoc. 13 (10), 2121-2148 (2018).
- Chevrier, S., et al. Compensation of signal spillover in suspension and imaging mass cytometry. Cell Syst. 6 (5), e5 612-620 (2018).
- Bjornson, Z. B., Nolan, G. P., Fantl, W. J. Single-cell mass cytometry for analysis of immune system functional states. Curr Opin Immunol. 25 (4), 484-494 (2013).
- Kalina, T., Lundsten, K., Engel, P. Relevance of antibody validation for flow cytometry. Cytometry A. 97 (2), 126-136 (2020).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J Immunol Methods. 243 (1-2), 77-97 (2000).
- Roederer, M. Spectral compensation for flow cytometry: visualization artifacts, limitations, and caveats. Cytometry. 45 (3), 194-205 (2001).
- Tung, J. W., Parks, D. R., Moore, W. A., Herzenberg, L. A., Herzenberg, L. A. New approaches to fluorescence compensation and visualization of FACS data. Clin Immunol. 110 (3), 277-283 (2004).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon