È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Identificazione e analisi di progenitori miogenici in vivo durante la lesione acuta del muscolo scheletrico mediante citometria di massa unicellulare ad alta dimensionalità
In questo articolo
Riepilogo
Il protocollo qui presentato consente l'identificazione e l'analisi ad alta dimensionalità delle cellule staminali muscolari e delle cellule progenitrici mediante citometria di massa a singola cellula e la loro purificazione mediante FACS per studi approfonditi della loro funzione. Questo approccio può essere applicato per studiare le dinamiche di rigenerazione in modelli di malattia e testare l'efficacia degli interventi farmacologici.
Abstract
La rigenerazione muscolare scheletrica è un processo dinamico guidato dalle cellule staminali muscolari adulte e dalla loro progenie. Per lo più quiescenti in uno stato stazionario, le cellule staminali muscolari adulte si attivano in seguito a una lesione muscolare. Dopo l'attivazione, proliferano e la maggior parte della loro progenie si differenzia per generare cellule muscolari competenti per la fusione, mentre le rimanenti si autorinnovano per ricostituire il pool di cellule staminali. Mentre l'identità delle cellule staminali muscolari è stata definita più di un decennio fa, sulla base della co-espressione di marcatori di superficie cellulare, i progenitori miogenici sono stati identificati solo di recente utilizzando approcci unicellulari ad alta dimensione. Qui, presentiamo un metodo di citometria di massa a singola cellula (citometria a tempo di volo [CyTOF]) per analizzare le cellule staminali e le cellule progenitrici in lesioni muscolari acute per risolvere le dinamiche cellulari e molecolari che si svolgono durante la rigenerazione muscolare. Questo approccio si basa sulla rilevazione simultanea di nuovi marcatori di superficie cellulare e fattori chiave di trascrizione miogenica la cui espressione dinamica consente l'identificazione di cellule staminali attivate e popolazioni di cellule progenitrici che rappresentano punti di riferimento della miogenesi. È importante sottolineare che viene descritta una strategia di smistamento basata sul rilevamento dei marcatori di superficie cellulare CD9 e CD104, che consente l'isolamento prospettico delle cellule staminali muscolari e delle cellule progenitrici utilizzando la selezione cellulare attivata dalla fluorescenza (FACS) per studi approfonditi della loro funzione. Le cellule progenitrici muscolari forniscono un anello mancante critico per studiare il controllo del destino delle cellule staminali muscolari, identificare nuovi bersagli terapeutici per le malattie muscolari e sviluppare applicazioni di terapia cellulare per la medicina rigenerativa. L'approccio qui presentato può essere applicato allo studio delle cellule staminali muscolari e delle cellule progenitrici in vivo in risposta a perturbazioni, come gli interventi farmacologici mirati a specifiche vie di segnalazione. Può anche essere utilizzato per studiare le dinamiche delle cellule staminali muscolari e delle cellule progenitrici in modelli animali di malattie muscolari, facendo progredire la nostra comprensione delle malattie delle cellule staminali e accelerando lo sviluppo di terapie.
Introduzione
Il muscolo scheletrico costituisce il tessuto più grande per massa del corpo e regola molteplici funzioni, dalla vista alla respirazione, dalla postura al movimento, nonché al metabolismo1. Pertanto, mantenere l'integrità e la funzione del muscolo scheletrico è fondamentale per la salute. Il tessuto muscolare scheletrico, costituito da fasci stretti di miofibre multinucleate circondate da una complessa rete di nervi e vasi sanguigni, mostra un notevole potenziale rigenerativo 1,2.
I principali motori della rigenerazione del muscolo scheletrico sono le cellule staminali muscolari adulte (MuSC). Conosciute anche come cellule satelliti, a causa della loro posizione anatomica unica adiacente alla membrana plasmatica della miofibra e al di sotto della lamina basale, sono state identificate per la prima volta nel 19613. Le muSC esprimono un marcatore molecolare unico, il fattore di trascrizione accoppiato box 7 (Pax7)4. Per lo più quiescenti negli adulti sani, si attivano in seguito a lesioni muscolari e proliferano per dare origine a una progenie che (i) si differenzierà in cellule muscolari competenti per la fusione che formeranno nuove miofibre per riparare il danno muscolare o (ii) si auto-rinnoveranno per ricostituire il pool di cellule staminali5.
A livello cellulare e molecolare, il processo di rigenerazione è piuttosto dinamico e coinvolge transizioni di stato cellulare, caratterizzate dall'espressione coordinata di fattori chiave di trascrizione miogenica, noti anche come fattori regolatori miogenici (MRFs)6,7. Precedenti studi sullo sviluppo in vivo, esperimenti di tracciamento del lignaggio e lavori di coltura cellulare utilizzando mioblasti hanno dimostrato che l'espressione sequenziale di questi fattori di trascrizione guida la miogenesi, con il fattore miogenico 5 (Myf5) espresso dopo l'attivazione, l'espressione della differenziazione miogenica 1 (MyoD1) che segna l'impegno nel programma miogenico e l'espressione della miogenina (MyoG) che segna la differenziazione 8,9,10,11, 12,13,14. Nonostante queste conoscenze e la scoperta di marcatori di superficie cellulare per purificare le MuSC, le strategie e gli strumenti per identificare e isolare popolazioni discrete lungo il percorso di differenziamento miogenico e risolvere una progressione miogenica in vivo sono stati carenti 15,16,17,18.
Qui, presentiamo un nuovo metodo, basato su ricerche pubblicate di recente, che consente l'identificazione di cellule staminali e progenitrici nel muscolo scheletrico e l'analisi delle loro dinamiche cellulari, molecolari e di proliferazione nel contesto di una lesione muscolare acuta19. Questo approccio si basa sulla citometria di massa a singola cellula (nota anche come citometria a tempo di volo [CyTOF]) per rilevare simultaneamente i marcatori chiave della superficie cellulare (α7 integrina, CD9, CD44, CD98 e CD104), fattori di trascrizione miogenica intracellulare (Pax7, Myf5, MyoD e MyoG) e un analogo nucleosidico (5-Iodo-2'-deossiuridina, IdU), per monitorare le cellule nella fase S19,20, 21,22,23. Inoltre, il protocollo presenta una strategia basata sulla rilevazione di due marcatori di superficie cellulare, CD9 e CD104, per purificare queste popolazioni cellulari mediante smistamento cellulare attivato dalla fluorescenza (FACS), consentendo così futuri studi approfonditi della loro funzione nel contesto di lesioni e malattie muscolari. Mentre i mioblasti primari sono stati ampiamente utilizzati in passato per studiare gli stadi tardivi della differenziazione miogenica in vitro, non è noto se ricapitolino lo stato molecolare delle cellule progenitrici muscolari presenti in vivo 24,25,26,27,28,29,30 . La produzione di mioblasti è laboriosa e richiede molto tempo, e lo stato molecolare di questa coltura primaria cambia rapidamente al passaggio31. Quindi, i progenitori miogenici appena isolati e purificati con questo metodo forniranno un sistema più fisiologico per studiare la miogenesi e l'effetto di manipolazioni genetiche o farmacologiche ex-vivo.
Il protocollo qui presentato può essere applicato per affrontare una varietà di questioni di ricerca, ad esempio, per studiare la dinamica del compartimento miogenico in vivo in modelli animali di malattie muscolari, in risposta a manipolazioni genetiche acute o su interventi farmacologici, approfondendo così la nostra comprensione della disfunzione delle cellule staminali muscolari in diversi contesti biologici e facilitando lo sviluppo di nuovi interventi terapeutici.
Protocollo
Le procedure sugli animali sono state approvate dall'ispettorato danese per gli esperimenti sugli animali (protocollo # 2022-15-0201-01293) e gli esperimenti sono stati eseguiti in conformità con le linee guida istituzionali dell'Università di Aarhus. L'analgesia (buprenorfina) viene fornita in acqua potabile 24 ore prima dell'infortunio affinché i topi si adattino al gusto. La fornitura di buprenorfina nell'acqua potabile viene continuata per 24 ore dopo l'infortunio. Insieme a un'iniezione sottocutanea (s.c.) di buprenorfina al momento della lesione muscolare acuta, la buprenorfina nell'acqua potabile dopo l'iniezione di notexin allevierà il dolore associato alla lesione. Mentre si raccomanda di somministrare un'iniezione s.c. di buprenorfina al momento della lesione muscolare acuta, seguita da buprenorfina nell'acqua potabile, la buprenorfina nell'acqua potabile prima dell'infortunio è facoltativa. Tuttavia, i ricercatori devono seguire gli standard e le linee guida sul benessere degli animali stabiliti dall'agenzia di regolamentazione appropriata.
NOTA: Per gli esperimenti di citometria di massa unicellulare (CyTOF) dei muscoli degli arti posteriori lesionati, iniziare dalla sezione 1: Analgesia in acqua 24 ore prima della lesione muscolare fino a 24 ore dopo la lesione. Per lo smistamento delle cellule staminali muscolari e delle cellule progenitrici da topi illesi, eseguire le sezioni 5 e 6: Eutanasia + Dissezione e dissociazione del muscolo scheletrico e continuare con la sezione 11: Colorazione con anticorpi coniugati con fluorofori per FACS. Una panoramica della configurazione sperimentale e del protocollo è mostrata nella Figura 1.
1. Analgesia in acqua 24 ore prima della lesione muscolare fino a 24 ore dopo la lesione
- A una borraccia scura o avvolta in un foglio di alluminio, aggiungere 3 ml di buprenorfina (0,3 mg/mL) e riempire fino a 100 ml con acqua filtrata per raggiungere una concentrazione finale di 0,009 mg/mL 24 ore prima dell'infortunio. Attaccalo alla gabbia del mouse.
- Rimuovere la borraccia 24 ore dopo l'infortunio e ricollegare la gabbia del topo al sistema di valvole per bere.
2. Preparazione per la procedura di lesione acuta
NOTA: Utilizzare etanolo al 70% per disinfettare il banco di lavoro, la configurazione del cono anteriore e la scatola di induzione.
- Preparare una provetta da 1,5 mL con notexin diluita in PBS (5 μg/mL). Diluire la buprenorfina (0,3 mg/mL) in soluzione fisiologica sterile allo 0,9% a 0,015 mg/mL in una provetta da 1,5 mL sicura per la luce. Mantenere il ghiaccio. Preparare siringhe da insulina per iniezioni di buprenorfina (28 G, 0,5 ml) e notesxin (29 g, 0,3 ml).
- Configurazione delle unità anestesiologiche a base di isoflurano:
- Per l'anestesia in una scatola di induzione, utilizzare isoflurano al 3% con un flusso di 1,5 L/min (50% O2, 50% aria atmosferica).
- Per la manutenzione che utilizza una configurazione a ogiva, utilizzare isoflurano all'1,5% con un flusso di 0,6 L/min (50% O2, 50% aria atmosferica).
3. Lesione acuta da iniezione di notexin
ATTENZIONE: Notexin ha attività di fosfolipasi A2 ed è il componente principale del veleno del serpente tigre australiano (Notechis scutatus), con una LD50 per via endovenosa di 5-17 mg di notexin/kg nei topi32,33. Nel presente protocollo, il muscolo tibiale anteriore (TA) di ciascun arto posteriore viene iniettato con 10 μL di 5 mg/mL di notexina e il muscolo gastrocnemio (GA) di ciascun arto posteriore viene iniettato due volte (una volta in ciascuna testa del muscolo) con 15 μL di 5 mg/mL di notexina. È importante eseguire correttamente le iniezioni intramuscolari (i.m.) per limitare i danni e ispezionare frequentemente gli animali iniettati per garantire il minimo dolore.
- Anestetizzare il topo nella scatola di induzione (isoflurano al 3%). Quando il topo è incosciente, abbassare il livello di isoflurano all'1,5% nella scatola di induzione.
- Pesare e marcare individualmente i topi secondo le procedure approvate dall'istituto. Sotto la configurazione del cono del naso, radere gli arti posteriori con un rifinitore e trasferirli nuovamente nella scatola di induzione.
- Calcola il volume di buprenorfina per ogni topo :
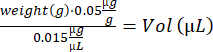
- Mescolare la soluzione di notexin pipettando su e giù. Preparare 2 siringhe da insulina (29 G, 0,3 ml) caricate con 10 μl di soluzione di notexina (5 μg/mL) per iniezioni di TA e 4 siringhe da insulina caricate con 15 μl di soluzione di notexin (5 μg/mL) per l'iniezione di GA.
NOTA: Il muscolo tibiale anteriore (TA) si trova sul lato anteriore della parte inferiore della gamba del topo, estendendosi dal ginocchio alla caviglia. Il muscolo gastrocnemio (GA) è un muscolo a due teste che si trova nella parte posteriore della parte inferiore della gamba, superficiale al soleo. Corre dalle sue due teste (mediale e laterale) appena sopra il ginocchio fino al tallone, estendendosi su un totale di tre articolazioni (ginocchio, caviglia e articolazioni sottoastragaliche). - Eseguire iniezioni intramuscolari
- Iniezione di TA (2 iniezioni in totale)
- Posizionare il mouse in posizione supina sotto la configurazione del cono del naso e disinfettare il sito di iniezione con salviettine imbevute di alcol sterili (70% di etanolo o isopropanolo). Inserire l'ago, smussato verso il basso, nella pancia del TA (distale a metà pancia) con un angolo di 30°. Far avanzare l'ago lungo il muscolo, muovendosi parallelamente alla tibia, per raggiungere la metà del ventre del TA. Iniettare la notexin lentamente e continuamente, lasciando l'ago in posizione per 10 secondi prima di ritirarlo. Non inserire l'ago troppo in profondità inizialmente (per evitare di iniettare notexin sotto l'AT nel muscolo estensore lungo delle dita) e non troppo prossimalmente quando si avanza l'ago (per evitare di iniettare troppo vicino al ginocchio).
- Iniezione di GA (4 iniezioni in totale)
- Inserire l'ago, smussato verso il basso, con un angolo di ~45° a metà pancia della testa laterale del GA. Iniettare la notexin lentamente e continuamente, lasciando l'ago in posizione per 10 s prima di ritirarlo. Ruotare l'arto posteriore e iniettare la notexin nella testa mediale del muscolo GA come sopra. Bisogna fare attenzione a non inserire l'ago troppo in profondità.
- Iniezione di TA (2 iniezioni in totale)
- Girare il mouse e iniettare buprenorfina s.c. utilizzando una siringa da insulina (28 G, 0,5 mL). Trasferisci il mouse in una gabbia di recupero vuota su una piastra riscaldante. Solo metà del fondo della gabbia deve essere posizionato sopra la piastra riscaldante per consentire al mouse di termoregolarsi durante il recupero34. Ripetere i passaggi 3.3-3.6 per i mouse rimanenti.
- Quando il mouse nella gabbia di recupero è completamente sveglio, spostalo di nuovo nella gabbia originale. Rimettilo nella stalla e integralo con cibo bagnato. Rimuovere il flacone di buprenorfina 24 ore dopo l'iniezione.
- Monitorare i topi per 6 ore dopo l'iniezione di notexin e poi ogni 12 ore per 2 giorni per segni di dolore, mobilità compromessa e diminuzione del consumo di cibo35,36.
4. Iniezione di 5-iodo-2'-deossiuridina
ATTENZIONE: Si sospetta che la 5-iodo-2'-deossiuridina (IdU) causi difetti genetici e danneggi la fertilità del nascituro. Leggere la scheda di sicurezza (SDS) prima di maneggiarla. Durante la manipolazione devono essere indossati dispositivi di protezione individuale. Utilizzare una cappa aspirante quando si pesa la polvere IdU. I materiali che sono stati a contatto con IdU devono essere smaltiti secondo le norme di sicurezza locali.
NOTA: La marcatura IdU in vivo viene utilizzata per monitorare la divisione cellulare durante il decorso del tempo di lesione perché IdU, un analogo iodato della timidina, viene incorporato nel DNA delle cellule in fase S. L'IdU viene iniettato per via intraperitoneale (i.p.) a 20 mg/kg di peso corporeo 8 ore prima di sacrificare il topo.
- Utilizzare un sonicatore a 37 °C per preparare una soluzione madre di 2 mg/mL di IdU in PBS sterile. IdU è sensibile alla luce; utilizzare entro lo stesso giorno o congelare a -20 °C per un massimo di 3 mesi. Se si utilizza IdU congelato: scongelare, vortice, centrifugare a 10.000 x g per 30 s e utilizzare il surnatante per le iniezioni sottostanti.
- Anestetizzare il topo nella scatola di induzione (isoflurano al 3%). Annota l'assegnazione, pesa i topi e calcola il volume della soluzione IdU:
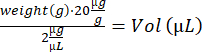
- Eseguire l'iniezione i.p. utilizzando una siringa da insulina (28 G, 0,5 mL) e trasferire il topo in una gabbia di recupero vuota su una piastra riscaldante. Quando è sveglio, riporta il mouse nella sua gabbia originale. Ripetere i passaggi 4.2-4.3 per i topi rimanenti e rimetterli nella stalla.
5. Eutanasia
NOTA: Vedere la Tabella 1 per le ricette del buffer. Preparare il terreno di lavaggio (miscela nutritiva F-10 (prosciutto), siero di cavallo al 10%, 1x Pen/Streptococco) e filtrare attraverso una membrana di polietersulfone (PES) in un contenitore di polistirene. Preparare il tampone di dissociazione (terreno di lavaggio integrato con 650 U/mL di collagenasi, tipo II) e conservare in ghiaccio. Le misure di citometria di massa CyTOF sono molto sensibili ai contaminanti. Per questo motivo, è essenziale utilizzare reagenti di altissimo grado analitico per l'elaborazione dei campioni. Per prevenire la contaminazione da metalli, si consiglia vivamente di utilizzare plastica sterile e vetreria nuova che non è mai stata lavata con detersivo perché molti saponi da laboratorio contengono alti livelli di bario. Si consiglia di utilizzare acqua a doppio filtro, distillata e deionizzata per la preparazione dei reagenti. La soluzione salina tamponata con fosfato (PBS) viene preparata internamente. Diluire i supporti 10x a 1x e filtrare il PBS 1x con filtri da 0,2 μm. Filtra nuovamente il PBS 1x all'inizio di ogni esperimento. Gli strumenti di dissezione non devono essere puliti con detergente a causa della presenza di bario.
- Preparare la bilancia, il contenitore e l'unità di anestesia a base di isoflurano con la scatola di induzione. Disinfettare gli strumenti di dissezione appropriati.
- Preparare e marcare provette da 50 mL per ciascun topo con 5 mL di tampone di dissociazione e una capsula di Petri. Mantenere il tampone di dissociazione sul ghiaccio.
- Anestetizzare il topo nella scatola di induzione (3% di isoflurano, aumentare al 5% quando si è incoscienti) ed eseguire la lussazione cervicale.
6. Dissezione e dissociazione del muscolo scheletrico
- Sezionare i muscoli TA e GA da entrambi gli arti posteriori e trasferirli nel coperchio di una capsula di Petri. Usa le forbici per tagliare il tessuto in un impasto tritato (~1 mm3 pezzi). Trasferire in una provetta da 50 mL contenente 5 mL di tampone di dissociazione ghiacciato. Mantenere il ghiaccio.
- Ripetere la procedura di eutanasia (passaggio 5.3) e la procedura di dissezione e dissociazione del muscolo scheletrico (passaggio 6.1) fino a quando tutti i topi non sono stati processati.
- Preriscaldare tutte le provette in un bagno d'acqua a 37 °C (2 min) e incubare per 45 min a rotazione in un incubatore (37 °C).
- Agitare e lavare aggiungendo 10 ml di terreno di lavaggio. Centrifugare (380 x g, 10 min, temperatura ambiente [RT]) e aspirare a 4 mL.
- Aggiungere 0,5 mL di collagenasi (1000 U/mL) e 0,5 mL di dispasi (11 U/mL). Agitare e incubare per 20 minuti a rotazione a 37 °C (ad es. in un incubatore). Centrifugare (380 x g, 1 min, RT) e risospendere con una pipetta da 5 mL.
- Aggiungere filtri cellulari da 40 μm a provette da 50 mL e pre-bagnare con 5 mL di terreno di lavaggio. Aspirare ed espellere la sospensione cellulare 10 volte utilizzando una siringa da 5 ml con un ago da 20 G. Filtrare attraverso il filtro cellulare da 40 μm pre-bagnato. Lavare la provetta da 50 mL con 10 mL di terreno di lavaggio, trasferirla nel filtro cellulare da 40 μm e centrifugare (380 x g, 10 min, 4 °C) la sospensione cellulare.
7. Colorazione viva/morta con fissazione con cisplatino e paraformaldeide
ATTENZIONE: Il cisplatino e la paraformaldeide (PFA) sono cancerogeni. Leggere la SDS prima di maneggiarla. La paraformaldeide (PFA; 16%) è irritante per la pelle, gli occhi e le vie respiratorie. Indossare dispositivi di protezione individuale e maneggiare queste sostanze sotto una cappa aspirante. Durante la fissazione delle cellule, la concentrazione finale di PFA sarà dell'1,6%. Devono essere prese le corrette misure di protezione e i rifiuti devono essere gestiti secondo le normative locali.
NOTA: Preparare DMEM freddo (4 °C) e caldo (37 °C) senza siero. Preparare il DMEM integrato con il 10% di FBS, filtrare attraverso una membrana in PES in un contenitore di polistirolo e conservare in ghiaccio. Preparare PBS e terreni di colorazione cellulare (CSM; PBS, 0,5% BSA, 0,02% sodio azide) in un flacone di vetro dedicato CyTOF e filtrare attraverso una membrana in PES. Il CSM può essere conservato a 4 °C per un massimo di 6 mesi.
- Aspirare il surnatante, azionare il pellet e risospendere in 1 mL di DMEM freddo senza siero. Contare le cellule e regolare la densità cellulare a 1 x 106 cellule/mL in DMEM senza siero.
- Aggiungere il cisplatin stock (25 mM) a una concentrazione finale di 25 μM. Vortex 10 s e incubare per esattamente 1 minuto (la reazione è molto sensibile al tempo). Estinguete la reazione con DMEM+10% FBS ghiacciato (3 volte il volume del campione) e mantenetelo in ghiaccio. Centrifugare (380 x g, 10 min, 4 °C), aspirare il surnatante e risospendere accuratamente il pellet (fino a 10 x 106 cellule/mL in CSM). Filtrare la sospensione attraverso un filtro cellulare da 35 μm.
- Fissare la sospensione cellulare con la soluzione madre di PFA filtrata (16%) e pipettare su e giù (eseguire sotto una cappa aspirante) per raggiungere la concentrazione finale di PFA (1,6%) (ad esempio, aggiungere 100 μl di PFA al 16% a 900 μl di sospensione cellulare). Agitare per 30 s, incubare per 10 minuti con ghiaccio e lavare due volte con 2 mL di CSM per centrifugazione (800 x g, 5 min, RT).
NOTA: A questo punto, i campioni possono essere congelati su ghiaccio secco e conservati a -80 °C o utilizzati direttamente per la colorazione degli anticorpi. Se le cellule devono essere congelate, trasferirle in provette di polipropilene da 5 mL, poiché il polistirene può rompersi a basse temperature. Se per la colorazione si utilizzano celle appena fissate, procedere al passaggio 8.2.
8. Colorazione con anticorpi metallo-coniugati
ATTENZIONE: Il metanolo (MeOH) è altamente infiammabile e corrosivo per le vie respiratorie. Leggere la SDS prima di maneggiarla. Indossare dispositivi di protezione individuale e maneggiare questa sostanza sotto una cappa aspirante. Gestire i rifiuti in conformità con le normative locali.
NOTA: L'elenco degli anticorpi (Ab) che hanno come bersaglio i marcatori di superficie e i marcatori intracellulari è riportato nella Tabella 2.
Coniugazione degli anticorpi: la maggior parte degli anticorpi utilizzati in questo protocollo sono stati coniugati internamente perché non erano disponibili in commercio. I protocolli per la coniugazione metallica degli anticorpi sono stati precedentemente pubblicati e i kit di coniugazione sono ora disponibili in commercio37,38. L'immunoglobulina di tipo G (IgG) è compatibile con i protocolli di coniugazione disponibili. È di grande importanza che la formulazione dell'anticorpo utilizzata per la coniugazione del metallo sia priva di proteine trasportatrici contenenti cisteina (ad esempio, l'albumina sierica bovina (BSA)), che possono influenzare l'efficienza della coniugazione competendo per i gruppi maleimmidici liberi del polimero e possono interferire con la quantificazione dell'anticorpo coniugato con il metallo. Il contenuto di cisteina della gelatina è molto inferiore a quello della BSA. Tuttavia, si raccomanda che se la formulazione anticorpale contiene proteine trasportatrici, tali proteine vengano rimosse prima della coniugazione. Ora è possibile richiedere al produttore anticorpi privi di BSA e gelatina. I conservanti di piccole molecole (ad es. sodio azide, glicerolo e trealosio) sono compatibili con i protocolli di coniugazione dei metalli37,38.
Titolazione degli anticorpi: dopo ogni coniugazione del metallo, gli anticorpi devono essere titolati per determinare la concentrazione ottimale di anticorpi che fornisce il massimo rapporto segnale/rumore. Per la titolazione degli anticorpi, eseguire una diluizione seriale in 6 fasi e colorare sia i campioni noti per esprimere (ad esempio, cellule muscolari, controlli positivi) che la mancanza (controlli negativi) della proteina di interesse 19,21,37,38.
Preparare la soluzione di lavoro fresca Cell-ID Intercalator-Ir (stock = 500 μM; soluzione intercalator-ir) diluendo il brodo a 0,1 μM in PBS/1,6% PFA.
- Scongelare i campioni congelati per 5 minuti a RT e centrifugare (800 x g, 1 min, RT). Lavare con 2 mL di CSM per centrifugazione (800 x g, 5 min, RT).
- Preparare una miscela di colorazione con anticorpi di superficie (Ab) 2,5x in CSM. Aspirare il surnatante a ~60 μl e risospendere accuratamente il pellet. Colorare le cellule aggiungendo 40 μl di miscela di colorazione per Ab di superficie e incubarla per 1 ora a RT.
- Sfogliare i campioni ogni 20 minuti per mescolare. Lavare due volte con 1 mL di CSM per centrifugazione (800 x g, 5 min, RT). Aspirare il surnatante e azionare il pellet.
NOTA: Se non è necessaria alcuna colorazione intracellulare, non è necessaria la permeabilizzazione del metanolo. Tuttavia, se viene eseguita solo la colorazione superficiale, la soluzione intercalator-ir deve essere diluita in un tampone contenente un agente permeabilizzante (ad esempio, Maxpar fix e tampone perm) per penetrare nella membrana nucleare. Procedere al passaggio 8.5 e diluire il Cell-ID Intercalator-Ir in un tampone contenente un agente permeabilizzante e PFA all'1,6%. In caso contrario, continuare con la colorazione intracellulare riportata di seguito.
ATTENZIONE: L'iridio è pericoloso ed è necessario prendere precauzioni per una manipolazione sicura. L'iridio è infiammabile ed è irritante per gli occhi e la pelle. Evitare di creare e respirare polvere o fumi. Evitare il contatto con la pelle e gli occhi. Tuttavia, la miscela di soluzione intercalator-ir viene fornita a una concentrazione del <1% in acqua, che è considerata non pericolosa secondo il sistema globale armonizzato (GHS) di classificazione ed etichettatura delle sostanze chimiche. Leggere la scheda di sicurezza di Standard Biotools prima di maneggiarla. Durante la manipolazione devono essere indossati dispositivi di protezione individuale. I materiali che sono stati a contatto con la soluzione intercalator-ir devono essere smaltiti secondo le norme di sicurezza locali. - Per permeabilizzare le cellule, aggiungere 0,5 ml di MeOH ghiacciato goccia a goccia durante il vortex. Incubare per 15 minuti su ghiaccio sotto una cappa aspirante. Centrifugare (800 x g, 5 min, RT) e quindi lavare due volte con 1 mL di CSM mediante centrifugazione (800 x g, 5 min, RT). Dopo l'ultimo lavaggio, aspirare il surnatante a ~60 μL e risospendere accuratamente il pellet.
- Preparare una miscela di colorazione Ab intracellulare 2,5x in CSM. Colorare le cellule aggiungendo 40 μl di miscela di colorazione Ab intracellulare e incubando per 1 ora a RT. Scorrere i campioni ogni 20 minuti per mescolare.
- Lavare due volte con 1 mL di CSM per centrifugazione (800 x g, 5 min, RT). Aspirare il surnatante e azionare il pellet. Risospendere i campioni in 0,5 mL di soluzione intercalator-iridio (Tabella 1) e vortex. Incubare i campioni per 1 ora a RT o durante la notte (O/N) a 4 °C (vedere la nota seguente).
NOTA: I campioni possono essere conservati in soluzione intercalator-iridio a 4 °C per un massimo di 48 ore. Il protocollo di colorazione utilizzato in questo studio è stato sviluppato sulla base del lavoro pionieristico del laboratorio Nolan{C}23. Si differenzia dai protocolli standard di Biotools, in quanto (i) i tamponi preparati internamente vengono utilizzati per il lavaggio e la colorazione, (ii) le cellule vengono fissate prima della colorazione superficiale e (iii) le cellule vengono permeabilizzate con metanolo per la colorazione intracellulare con anticorpi contro fattori di trascrizione o molecole di segnalazione. I ricercatori che sviluppano un nuovo protocollo devono testare a fondo la compatibilità del loro pannello di anticorpi con il metanolo come agente di permeabilizzazione. Quando si aggiungono nuovi anticorpi a un pannello, si consiglia di testare la specificità degli anticorpi mediante citometria a flusso utilizzando campioni di controllo positivi e negativi prima della coniugazione e della titolazione dei metalli.
9. Preparazione del campione per il caricamento nel citometro di massa
NOTA: I pellet delle celle sono molto sciolti quando si trovano nel buffer CAS (Table of Materials). Durante i lavaggi con tampone CAS, non aspirare fino all'asciutto. Invece, mantenere un volume residuo come descritto di seguito.
- Vortice e centrifuga (800 x g, 10 min, RT) i campioni. Versare il surnatante (maneggiare come rifiuto di PFA) e vortice.
- Lavare con 1 mL di CSM per centrifugazione (800 x g, 10 min, RT). Aspirare il surnatante e azionare il pellet.
- Lavare con 1 mL di tampone CAS per centrifugazione (800 x g, 10 min, RT). Aspirare a ~200 μL. Vortex e aggiungere 1 mL di tampone CAS. Estrarre 5 μL di aliquota per la conta cellulare. Centrifugare (800 x g, 10 min, RT) e aspirare con cura a ≤50 μl. Non disturbare il pellet.
- Risospendere il pellet a 1–2 x 106 cellule/mL in tampone CAS e aggiungere le perle di calibrazione (1x; Tabella dei materiali) a una concentrazione finale di 0,1x (ad esempio, aggiungere 100 μl di perle di calibrazione 1x a 900 μl di sospensione cellulare).
- Caricare il campione nel citometro di massa e raccogliere i dati utilizzando una velocità di flusso di 400-500 cellule/s.
- Eseguire la normalizzazione dei dati dei file FCS utilizzando il software CyTOF o strumenti di normalizzazione sviluppati in precedenza39.
NOTA: Il funzionamento dei citometri di massa CyTOF è specifico per lo strumento40,41. Si consiglia di consultare il manuale utente CyTOF prima dell'uso. Le perle di calibrazione sono standard di perline di normalizzazione in polistirene incorporato in metallo contenenti concentrazioni note degli isotopi metallici cerio (140/142Ce), europio (151/153Eu), olmio (165Ho) e lutezio (175/176Lu). Le perline di calibrazione consentono di controllare la sensibilità della macchina, che può variare nel tempo, principalmente a causa dell'accumulo di materiale biologico e delle variazioni della ionizzazione del plasma nel tempo.
10. Analisi dei dati CyTOF
NOTA: Per l'analisi a valle, i file FCS normalizzati possono essere analizzati localmente o caricati su soluzioni software basate su cloud come Cytobank, Cell Engine, OMIQ o FCS Express42.
- Concatenare singoli file FCS per ogni campione in un singolo file, se necessario.
- Identifica le singole cellule controllando gli eventi positivi dell'intercalatore di iridio, che consente la discriminazione delle singole cellule nucleate da detriti o doppietti.
- Gate per cellule vive selezionando eventi negativi al cisplatino. Il cisplatino si lega in modo covalente alle proteine cellulari e marcasce le cellule morte e morenti con membrane compromesse in misura maggiore rispetto alle cellule vive43.
- Effettuare il gate sulla popolazione di interesse, ad esempio, il compartimento miogenico (Live/CD45-/CD31-/Sca1-/α7integrina+/CD9+) (Figura 2A) e quantificare la proporzione relativa di cellule staminali e progenitrici. Questo approccio richiede una conoscenza preliminare della superficie cellulare o dell'espressione di marcatori intracellulari per definire le singole popolazioni.
- Eseguire analisi ad alta dimensionalità che consentiranno l'identificazione di sottoinsiemi di cellule rare precedentemente non riconosciuti all'interno di una popolazione complessa, esportare la popolazione di interesse e utilizzare algoritmi di clustering sviluppati specificamente per l'analisi dei dati CyTOF44.
NOTA: Il lavoro precedente ha impiegato l'algoritmo X-shift, che utilizza la stima ponderata della densità del k-nearest neighbor (kNN-DE) in uno spazio ad alta dimensionalità per eseguire il clustering non supervisionato basato su parametri definiti19. X-shift ha dimostrato di essere altamente efficace nell'identificare popolazioni cellulari rare45. - Per l'analisi X-shift, scaricare il pacchetto software vortex (dalla pagina GitHub del laboratorio Nolan [https://github.com/nolanlab/vortex]) e Java 64-bit46. Le istruzioni sono disponibili qui: https://github.com/nolanlab/vortex/wiki/Getting-Started.
- Caricare le popolazioni di celle esportate in un database locale e definire i parametri di clustering. Tra i parametri definiti dall'utente ci sono (i) i marcatori utilizzati per il clustering, (ii) l'intervallo di valori k (ad esempio, da 5 a 150) e (iii) il numero di passaggi di clustering.
NOTA: Lavori precedenti hanno utilizzato, come marcatori di clustering, una combinazione di marcatori di superficie noti per essere espressi nelle cellule staminali muscolari, nuovi marcatori di superficie identificati in uno screening citometrico a flusso ad alto rendimento di cellule muscolari e mioblasti e fattori di trascrizione miogenica (TF) noti per definire diversi stadi della miogenesi. Questo approccio ha permesso l'identificazione di due marcatori di superficie cellulare, CD9 e CD104, il cui modello di co-espressione distingue popolazioni di cellule progenitrici precedentemente non riconosciute19. L'attenta scelta dei marcatori di clustering permetterà al ricercatore di rispondere a specifiche domande di ricerca. Ad esempio, se si desidera l'identificazione e l'analisi nel tempo di sottoinsiemi di cellule in divisione, si consiglia di includere IdU come uno dei marcatori di clustering. X-shift è in grado di eseguire il clustering a più valori k e di identificare automaticamente, calcolando il "punto a gomito", il valore k che risulta in un numero ottimale di cluster, evitando quindi il sotto-clustering o la sovra-frammentazione. Si consiglia di calcolare il punto del gomito e di utilizzare l'analisi di clustering definita dal valore k ottimale per la visualizzazione dei dati e le applicazioni a valle. - Per visualizzare le relazioni spaziali tra le popolazioni cellulari all'interno dei cluster X-shift, eseguire un layout diretto dalla forza, che genererà una mappa 2D in cui la distanza tra i cluster indica la loro somiglianza nell'espressione dei marcatori nello spazio fenotipico ad alta dimensione. Colorando la mappa utilizzando un pennarello alla volta, è possibile scoprire nuove popolazioni cellulari e seguirne le dinamiche. Si raccomanda la quantificazione di follow-up mediante gating manuale in software, come Cytobank e Cell Engine.
- Esegui l'analisi della heatmap per quantificare l'espressione dinamica di più proteine di superficie e intracellulari nel corso del tempo e rivelare nuove tendenze.
NOTA: Quando si esegue un andamento del tempo di infortunio, è possibile raggruppare i dati di tutti i punti temporali per generare una mappa della rigenerazione. Inoltre, è possibile sezionare tale mappa nei singoli punti temporali per seguire le dinamiche cellulari e molecolari della rigenerazione nel tempo19.
11. Colorazione con anticorpi coniugati con fluorofori per FACS
NOTA: le celle utilizzate per i controlli monocolore non colorati e per i controlli FMO (fluorescenza meno uno) possono provenire dal set TA e GA da un mouse aggiuntivo, se disponibile. In alternativa, i quadricipiti (muscolo anteriore superiore della coscia) possono essere sezionati e digeriti in una sospensione a singola cellula, seguendo la stessa procedura del set TA+GA sopra e utilizzato per i controlli. Preparare il tampone FACS (PBS, 2,5% siero di capra, 2 mM EDTA), filtrare attraverso una membrana PES in un contenitore di polistirene e conservare in ghiaccio. Il tampone FACS può essere conservato a 4 °C per un massimo di 1 mese. Un elenco degli anticorpi utilizzati per la FACS è riportato nella Tabella 3.
- Prepara un mix di lignaggio. Colorare ogni controllo in 50 μl (contenente circa 3-5 x 105 cellule). Calcola la quantità di mix di lignaggio necessaria. Effettuare un eccesso di volume del 20%-30%. La concentrazione stock di anticorpi utilizzati per la miscela di lignaggio è di 0,2 mg/mL.
- In una provetta da 0,5 mL, aggiungere l'anti-CD45 APC-Cy7, l'anti-CD31 APC-Cy7, l'anti-Sca1 APC-Cy7 e l'anti-CD11b APC-Cy7 per raggiungere le concentrazioni finali di 1 μg/mL, 2,5 μg/mL, 2,5 μg/mL e 0,63 μg/mL, rispettivamente.
- Preparare il mix di tutte le macchie. Colorare ogni campione completamente colorato in 500 μl (contenente circa 3-5 x 106 cellule). Versare il 10% in eccesso di volume di all-stain-mix.
- In una provetta da 1,5 mL, aggiungere gli anticorpi di base come sopra + anti-α7 integrina PE, anti-CD9 APC e anti-CD104 FITC per raggiungere concentrazioni finali di 2 μg/mL, 1,2 μg/mL e 3 μg/mL, rispettivamente.
- Preparare il tampone FACS + DAPI: per 10 mL di tampone FACS, aggiungere 1 μL di DAPI (stock = 1 mg/mL) per raggiungere una concentrazione finale di DAPI di 100 ng/mL.
ATTENZIONE: Il DAPI (4',6-diamidino-2-fenilindolo dicloridrato) è classificato come possibile irritante per la pelle e le vie respiratorie. Tuttavia, la soluzione DAPI a una concentrazione del <1% è considerata non pericolosa, secondo il GHS. Leggere la SDS prima di maneggiarla. Durante la manipolazione devono essere indossati dispositivi di protezione individuale. I materiali che sono stati a contatto con DAPI devono essere smaltiti secondo le norme di sicurezza locali. - Risospendere le cellule della sezione 6: Dissezione e dissociazione del muscolo scheletrico:
- Per i campioni tutti colorati, risospendere i set TA e GA da un singolo topo in 500 μl di tampone FACS e trasferirlo in provette da 15 mL per la colorazione.
- Per i controlli, utilizzare i set TA e GA o i set di quadricipiti. Se si utilizza 1 mouse per i controlli, risospendere le cellule in 750 μL di tampone FACS. Se si utilizzano più topi, risospendere ciascun set di TA e GA o quadricipite in 500 μL di tampone FACS e combinare i set in un singolo campione da 1 mL prelevando una frazione da ciascun campione. Aggiungere 50 μl di celle di controllo (3-5 x 105 cellule) a provette in polipropilene da 5 ml per la colorazione.
- Colorare come descritto nella Tabella 4 per 45 minuti a 4 °C al buio.
- Lavare tutti i campioni colorati aggiungendo 5 mL di tampone FACS. Lavare i controlli aggiungendo 1 mL di tampone FACS e centrifugare (380 x g, 10 min, 4 °C).
- Risospendere tutti i campioni colorati in 1 mL di tampone FACS + DAPI. Risospendere i controlli in 300 μL di tampone FACS con o senza DAPI.
- Mantenere le cellule a 4 °C al buio fino alla selezione su un citometro a flusso con 4 laser (405 nm, 488 nm, 561 nm, 633 nm).
- Esegui i controlli e i campioni sullo smistatore e crea i cancelli di smistamento utilizzando il software pertinente. Per i controlli a colore singolo non colorati e i controlli FMO, registrare rispettivamente 1 x 104 e 0,3–1 x 105 eventi. Per i campioni tutti colorati, registrare fino a 1 x 106 eventi. Ordinate tutti i campioni completamente colorati, o quanto necessario, a seconda dei requisiti del saggio a valle. Per il citometro a flusso utilizzato qui (FACSAria III), selezionare in modalità di purezza utilizzando un ugello da 70 mm.
- Vedere la Figura 4 per la strategia di gating.
- Utilizzare i controlli non colorati e monocolore per impostare la tensione per tutti i rilevatori.
- Utilizzare i controlli monocolore per impostare la matrice di compensazione.
- Utilizzare i controlli FMO per stabilire i cancelli di smistamento.
- Popolazioni distinte di cellule staminali muscolari e progenitrici sono ordinate in un tampone FACS ghiacciato.
- Centrifugare (380 x g, 10 min, 4 °C) le popolazioni cellulari. Risospendere nel buffer appropriato, contare e continuare con l'analisi a valle.
Risultati
Qui presentiamo una panoramica della configurazione sperimentale per l'utilizzo di questo approccio combinato che include (i) l'analisi CyTOF ad alta dimensionalità di un decorso temporale di lesione acuta mediante iniezione di notexin per studiare la dinamica cellulare e molecolare delle cellule staminali e progenitrici nel muscolo scheletrico (Figura 1, schema in alto); e (ii) FACS di cellule staminali e progenitrici utilizzando due marcatori di superfici...
Discussione
La rigenerazione muscolare scheletrica è un processo dinamico che si basa sulla funzione delle cellule staminali adulte. Mentre studi precedenti si sono concentrati sul ruolo delle cellule staminali muscolari durante la rigenerazione, la loro progenie in vivo è stata poco studiata, principalmente a causa della mancanza di strumenti per identificare e isolare queste popolazioni cellulari 15,16,17,18.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Ringraziamo i membri della FACS Core Facility del Dipartimento di Biomedicina dell'Università di Aarhus per il supporto tecnico. Ringraziamo Alexander Schmitz, direttore dell'Unità di Citometria di Massa presso il Dipartimento di Biomedicina, per il confronto e il supporto. Le illustrazioni scientifiche sono state create utilizzando Biorender.com. Questo lavoro è stato finanziato da una sovvenzione Starting Grant dell'Aarhus Universitets Forskningsfond (AUFF) e da una sovvenzione Start Package (0071113) della Novo Nordisk Foundation a E.P.
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Fisher Scientific | 07-200-886 | |
| 20 G needle | KDM | KD-fine 900123 | |
| 28 G, 0.5 mL insulin syringe | BD | 329461 | |
| 29 G, 0.3 mL insulin syringe | BD | 324702 | |
| 3 mL syringes | Terumo medical | MDSS03SE | |
| 40 µm cell strainers | Fisher Scientific | 11587522 | |
| 5 mL polypropylene tubes | Fisher Scientific | 352002 | |
| 5 mL polystyrene test tubes with 35 µm cell strainer | Falcon | 352235 | |
| 5 mL syringes | Terumo medical | SS05LE1 | |
| 50 mL centrifuge tube | Fisher Scientific | 05-539-13 | |
| 5-Iodo-2-deoxyuridine (IdU) | Merck | I7125-5g | |
| anti-CD104 FITC (clone: 346-11A) | Biolegend | 123605 | Stock = 0.5 mg/mL |
| anti-CD11b APC-Cy7 (Clone: M1/70) | Biolegend | 101226 | Stock = 0.2 mg/mL |
| anti-CD31 APC-Cy7 (clone: 390) | Biolegend | 102440 | Stock = 0.2 mg/mL |
| anti-CD45 APC-Cy7 (Clone: 30-F11) | Biolegend | 103116 | Stock = 0.2 mg/mL |
| anti-CD9 APC (clone: KMC8) | ThermoFisher Scientific | 17-0091-82 | Stock = 0.2 mg/mL |
| anti-Sca1 (Ly6A/E) APC-Cy7 (clone: D7) | Biolegend | 108126 | Stock = 0.2 mg/mL |
| anti-α7 integrin PE (clone: R2F2)) | UBC AbLab | 67-0010-05 | Stock = 1 mg/mL |
| BD FACS Aria III (4 laser) instrument | BD Biosciences | N/A | 405, 488, 561, and 633 nm laser |
| Bovine Serum Albumin | Sigma Aldrich | A7030-50G | |
| Buprenorphine 0.3 mg/mL | Ceva | Vnr 054594 | |
| CD104 (Clone: 346-11A) | BD Biosciences | 553745 | Dy162; In-house conjugated |
| CD106/VCAM-1 (Clone: 429 MVCAM.A) | Biolegend | 105701 | Er170; In-house conjugated |
| CD11b (Clone: M1/70) | BD Biosciences | 553308 | Nd148; In-house conjugated |
| CD29/Integrin β1 (Clone: 9EG7) | BD Biosciences | 553715 | Tm169; In-house conjugated |
| CD31 (Clone: MEC 13.3) | BD Biosciences | 557355 | Sm154; In-house conjugated |
| CD34 (Clone: RAM34) | BD Biosciences | 551387 | Lu175; In-house conjugated |
| CD44 (Clone: IM7) | BD Biosciences | 550538 | Yb171; In-house conjugated |
| CD45 (Clone: MEC 30-F11) | BD Biosciences | 550539 | Sm147; In-house conjugated |
| CD9 (Clone: KMC8) | Thermo Fisher Scientific | 14-0091-85 | Yb174; In-house conjugated |
| CD90.2/Thy1.2 (Clone: 30-H12) | BD Biosciences | 553009 | Nd144; In-house conjugated |
| CD98 (Clone: H202-141) | BD Biosciences | 557479 | Pr141; In-house conjugated |
| Cell Acquisition Solution/Maxpar CAS-buffer | Standard Biotools | 201240 | |
| Cell-ID Intercalator-Iridium | Standard Biotools | 201192B | cationic nucleic acid intercalator |
| Cisplatin | Merck | P4394 | Pt195 |
| Cisplatin (cis-Diammineplatinum(II) dichloride) | Merck | P4394 | |
| Clear 1.5 mL tube | Fisher Scientific | 11926955 | |
| Collagenase, Type II | Worthington Biochemical Corporation | LS004177 | |
| Counting chamber | Merck | BR718620-1EA | |
| CXCR4/SDF1 (Clone: 2B11/CXCR4 ) | BD Biosciences | 551852 | Gd158; In-house conjugated |
| DAPI (1 mg/mL) | BD Biosciences | 564907 | |
| Dark 1.5 mL tube | Fisher Scientific | 15386548 | |
| Dispase II | Thermo Fisher Scientific | 17105041 | |
| Dissection Scissors | Fine Science Tools | 14568-09 | |
| DMEM (low glucose, with pyruvate) | Thermo Fisher Scientific | 11885-092 | |
| EDTA (Ethylenediaminetetraacetic acid disodium salt) | Merck | E5134 | Na2EDTA-2H20 |
| EQ Four Element Calibration Beads (EQ beads) | Standard Biotools | 201078 | Calibration beads |
| Fetal Bovine Serum, qualified, Brazil origin | Thermo Fisher Scientific | 10270106 | |
| Forceps Dumont #5SF | Fine Science Tools | 11252-00 | |
| Forceps Dumont #7 | Hounisen.com | 1606.3350 | |
| Goat serum | Thermo Fisher Scientific | 16210-072 | |
| Helios CyTOF system | Standard Biotools | N/A | |
| Horse Serum, heat inactivated, New Zealand origin | Thermo Fisher Scientific | 26-050-088 | |
| IdU | Merck | I7125 | I127 |
| Iridium-Intercalator | Standard Biotools | 201240 | Ir191/193 |
| Isoflurane/Attane Vet | ScanVet | Vnr 055226 | |
| Methanol | Fisher Scientific | M/3900/17 | |
| Myf5 (Clone: C-20) | Santa Cruz Biotechnology | Sc-302 | Yb173; In-house conjugated |
| MyoD (Clone: 5.8A) | BD Biosciences | 554130 | Dy164; In-house conjugated |
| MyoG (Clone: F5D) | BD Biosciences | 556358 | Gd160; In-house conjugated |
| Nalgene Rapid-Flow Sterile Disposable Bottle Top 0.20 μM PES Filters | Thermo Fisher Scientific | 595-4520 | |
| Notexin | Latoxan | L8104 | Resuspend to 50 µg/ml in sterile PBS. Keep stocks (e.g. 50 µl) at -20 °C |
| Nutrient mixture F-10 (Ham's) | Thermo Fisher Scientific | 31550031 | |
| pAkt (Clone: D9E) | Standard Biotools | 3152005A | Sm152 |
| Pax7 (Clone: PAX7) | Santa Cruz Biotechnology | Sc-81648 | Eu153; In-house conjugated |
| Penicillin-Streptomycin (10,000 U/mL) (Pen/Strep) | Thermo Fisher Scientific | 15140122 | |
| PES Filter Units 0.20 μM | Fisher Scientific | 15913307 | |
| PES Syringe Filter | Fisher Scientific | 15206869 | |
| Petri dish | Sarstedt | 82.1472.001 | |
| PFA 16% EM grade | MP Biomedicals | 219998320 | |
| Potassium chloride (KCl) | Fisher Scientific | 10375810 | |
| Potassium phosphate, monobasic, anhydrous (KH2PO4) | Fisher Scientific | 10573181 | |
| pRb (Clone: J112-906) | Standard Biotools | 3166011A | Er166 |
| pS6 kinase (Clone: N7-548) | Standard Biotools | 3172008A | Yb172 |
| Sca-1 (Clone: E13-161.7) | BD Biosciences | 553333 | Nd142; In-house conjugated |
| Sodium Azide | Sigma Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | 10553515 | |
| Sodium phosphate, dibasic, heptahydrate (Na2HPO4-6H2O) | Merck | S9390 | |
| Sterile saline solution 0.9% | Fresenius | B306414/02 | |
| α7 integrin (Clone: 3C12) | MBL international | K0046-3 | Ho165; In-house conjugated |
Riferimenti
- Mukund, K., Subramaniam, S. Skeletal muscle: A review of molecular structure and function in health and disease. Wiley Interdiscip Rev Syst Biol Med. 12 (1), e1462 (2020).
- Feige, P., Brun, C. E., Ritso, M., Rudnicki, M. A. Orienting muscle stem cells for regeneration in homeostasis, aging, and disease. Cell Stem Cell. 23 (5), 653-664 (2018).
- Mauro, A. Satellite cell of skeletal muscle fibers. J Biophys Biochem Cytol. 9 (2), 493-495 (1961).
- Seale, P., et al. Pax7 is required for the specification of myogenic satellite cells. Cell. 102 (6), 777-786 (2000).
- Fuchs, E., Blau, H. M. Tissue stem cells: Architects of their niches. Cell Stem Cell. 27 (4), 532-556 (2020).
- Hernández-hernández, J. M., et al. The myogenic regulatory factors, determinants of muscle development, cell identity and regeneration. Semin Cell Dev Biol. 72, 10-18 (2017).
- Zammit, P. S. Function of the myogenic regulatory factors Myf5, MyoD, Myogenin and MRF4 in skeletal muscle, satellite cells and regenerative myogenesis. Semin Cell Dev Biol. 72, 19-32 (2017).
- Sabourin, L. A. The molecular regulation of myogenesis. Clin Genet. 57 (1), 16-25 (2000).
- Cooper, R. N., et al. In vivo satellite cell activation via Myf5 and MyoD in regenerating mouse skeletal muscle. J Cell Sci. 112 (17), 2895-2901 (1999).
- Rudnicki, M. A., Jaenisch, R. The MyoD family of transcription factors and skeletal myogenesis. Bioessays. 17 (3), 203-209 (1995).
- Braun, T., Arnold, H. H. Inactivation of Myf-6 and Myf-5 genes in mice leads to alterations in skeletal muscle development. EMBO J. 14 (6), 1176-1186 (1995).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Braun, T., et al. MyoD expression marks the onset of skeletal myogenesis in Myf-5 mutant mice. Development. 120 (11), 3083-3092 (1994).
- Rudnicki, M. A., et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell. 75 (7), 1351-1359 (1993).
- Montarras, D., et al. Developmental biology: Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Sacco, A., Doyonnas, R., Kraft, P., Vitorovic, S., Blau, H. M. Self-renewal and expansion of single transplanted muscle stem cells. Nature. 456 (7221), 502-506 (2008).
- Cerletti, M., et al. Highly efficient, functional engraftment of skeletal muscle stem cells in dystrophic muscles. Cell. 134 (1), 37-47 (2008).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nat Protoc. 10 (10), 1612-1624 (2015).
- Porpiglia, E., et al. High-resolution myogenic lineage mapping by single-cell mass cytometry. Nat Cell Biol. 19 (5), 558-567 (2017).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry Part A. 81 (7), 552-566 (2012).
- Hartmann, F. J., et al. . Mass Cytometry: Methods and Protocols. , (2019).
- Devine, R. D., Behbehani, G. K. Use of the pyrimidine analog, 5-iodo-2'-deoxyuridine (IdU) with cell cycle markers to establish cell cycle phases in a mass cytometry platform. J Vis Exp. (176), e60556 (2021).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Nag, A. C., Foster, J. D. Myogenesis in adult mammalian skeletal muscle in vitro. J Anat. 132, 1-18 (1981).
- Le Moigne, A., et al. Characterization of myogenesis from adult satellite cells cultured in vitro). Int J Dev Biol. 34, 171-180 (1990).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Chu, C., Cogswell, J., Kohtz, D. S. MyoD functions as a transcriptional repressor in proliferating myoblasts. J Biol Chem. 272 (6), 3145-3148 (1997).
- Shah, B., Hyde-Dunn, J., Jones, G. E. Proliferation of murine myoblasts as measured by bromodeoxyuridine incorporation. Methods in Mol Biol. 75, 349-355 (1997).
- Springer, M. L., Blau, H. M. High-efficiency retroviral infection of primary myoblasts. Somat Cell Mol Genet. 23 (3), 203-209 (1997).
- Rando, T. A., Blau, H. M. Primary mouse myoblast purification, characterization, and transplantation for cell-mediated gene therapy. J Cell Biol. 125 (6), 1275-1287 (1994).
- Springer, M. L., Rando, T. A., Blau, H. M. Gene delivery to muscle. Curr Protoc Hum Genet. , (2002).
- Cull-Candy, S. G., Fohlman, J., Gustavsson, D., Lullmann-Rauch, R., Thesleff, S. The effects of taipoxin and notexin on the function and fine structure of the murine neuromuscular junction. Neuroscience. 1 (3), 175-180 (1976).
- Francis, B., John, T. R., Seebart, C., Kaiser, I. I. New toxins from the venom of the common tiger snake (Notechis scutatus scutatus). Toxicon. 29 (1), 85-96 (1991).
- Navarro, K. L., Huss, M., Smith, J. C., Sharp, P., Marx, J. O., Pacharinsak, C. Mouse Anesthesia: The Art and Science. ILAR Journal. 62, 238-273 (2021).
- Langford, D., Bailey, A., Chanda, M., et al. Coding of facial expressions of pain in the laboratory mouse. Nat Methods. 7, 447-449 (2010).
- Matsumiya, L. C., Sorge, R. E., Sotocinal, S. G., Tabaka, J. M., Wieskopf, J. S., Zaloum, A., King, O. D., Mogil, J. S. Using the Mouse Grimace Scale to reevaluate the efficacy of postoperative analgesics in laboratory mice. J Am Assoc Lab Anim Sci. 2012 (1), 42-49 (2012).
- Gonzalez, V. D., et al. High-grade serous ovarian tumor cells modulate NK cell function to create an immune-tolerant microenvironment. Cell Rep. 36 (9), 109632 (2021).
- Delgado-Gonzalez, A., et al. Measuring trogocytosis between ovarian tumor and natural killer cells. STAR Protoc. 3 (2), 101425 (2022).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry Part A. 83 (5), 483-494 (2013).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. J Vis Exp. (69), e4398 (2012).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample preparation for mass cytometry analysis. J Vis Exp. (122), e54394 (2017).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Curr Protoc Cytom. , (2010).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry Part A. 81 (6), 467-475 (2012).
- Kimball, A. K., et al. A beginner's guide to analyzing and visualizing mass cytometry data. J Immunol. 200 (1), 3-22 (2018).
- Weber, L. M., Robinson, M. D. Comparison of clustering methods for high-dimensional single-cell flow and mass cytometry data. Cytometry Part A. 89 (12), 1084-1096 (2016).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nat Methods. 13 (6), 493-496 (2016).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Anal Chem. 80 (7), 2539-2547 (2008).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nat Commun. 12 (1), 692 (2021).
- de Morree, A., et al. Staufen1 inhibits MyoD translation to actively maintain muscle stem cell quiescence. Proc Natl Acad Sci U S A. 114 (43), E8996-E9005 (2017).
- Luo, D., et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c. Proc Natl Acad Sci U S A. 114 (15), E3071-E3080 (2017).
- Wersto, R. P., et al. Doublet discrimination in DNA cell-cycle analysis. Cytometry. 46 (5), 296-306 (2001).
- Porpiglia, E., Blau, H. M. Plasticity of muscle stem cells in homeostasis and aging. Curr Opin Genet Dev. 77, 101999 (2022).
- Porpiglia, E., et al. Elevated CD47 is a hallmark of dysfunctional aged muscle stem cells that can be targeted to augment regeneration. Cell Stem Cell. 29 (12), 1653-1668 (2022).
- Brunet, A., Goodell, M. A., Rando, T. A. Ageing and rejuvenation of tissue stem cells and their niches. Nat Rev Mol Cell Biol. 24 (1), 45-62 (2022).
- Danielli, S. G., et al. Single-cell profiling of alveolar rhabdomyosarcoma reveals RAS pathway inhibitors as cell-fate hijackers with therapeutic relevance. Sci Adv. 9 (6), (2023).
- de Morree, A., Rando, T. A. Regulation of adult stem cell quiescence and its functions in the maintenance of tissue integrity. Nat Rev Mol Cell Biol. 24 (5), 334-354 (2023).
- Yucel, N., et al. Glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell function. Cell Rep. 27 (13), 3939-3955 (2019).
- Tierney, M. T., Sacco, A. Inducing and evaluating skeletal muscle injury by notexin and barium chloride. Methods Mol Biol. 1460, 53-60 (2016).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198 (2016).
- Call, J. A., Lowe, D. A. Eccentric contraction-induced muscle injury: Reproducible, quantitative, physiological models to impair skeletal muscle's capacity to generate force. Methods Mol Biol. 1460, 3-18 (2016).
- Garry, G. A., Antony, M. L., Garry, D. J. Cardiotoxin Induced Injury and Skeletal Muscle Regeneration. Methods Mol Biol. 1460, 61-71 (2016).
- Le, G., Lowe, D. A., Kyba, M. Freeze injury of the tibialis anterior muscle. Methods Mol Biol. 1460, 33-41 (2016).
- Borok, M., et al. Progressive and coordinated mobilization of the skeletal muscle niche throughout tissue repair revealed by single-cell proteomic analysis. Cells. 10 (4), 744 (2021).
- Petrilli, L. L., et al. High-dimensional single-cell quantitative profiling of skeletal muscle cell population dynamics during regeneration. Cells. 9 (7), 1723 (2020).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Mol Cell. 74 (3), 609-621 (2019).
- Hartmann, F. J., et al. Scalable conjugation and characterization of immunoglobulins with stable mass isotope reporters for single-cell mass cytometry analysis. Methods Mol Biol. 1989, 55-81 (2019).
- Frimand, Z., Das Barman, S., Kjær, T. R., Porpiglia, E., de Morrée, A. Isolation of quiescent stem cell populations from individual skeletal muscles. J Vis Exp. (190), e64557 (2022).
- Krutzik, P. O., Nolan, G. P. Intracellular phospho-protein staining techniques for flow cytometry: monitoring single cell signaling events. Cytometry A. 55 (2), 61-70 (2003).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat Biotechnol. 30 (9), 858-867 (2012).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Curr Protoc Immunol. , 11-20 (2012).
- Krutzik, P. O., Clutter, M. R., Nolan, G. P. Coordinate analysis of murine immune cell surface markers and intracellular phosphoproteins by flow cytometry. J Immunol. 175 (4), 2357-2365 (2005).
- Krutzik, P. O., Irish, J. M., Nolan, G. P., Perez, O. D. Analysis of protein phosphorylation and cellular signaling events by flow cytometry: techniques and clinical applications. Clin Immunol. 110 (3), 206-221 (2004).
- Han, G., Spitzer, M. H., Bendall, S. C., Fantl, W. J., Nolan, G. P. Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry. Nat Protoc. 13 (10), 2121-2148 (2018).
- Chevrier, S., et al. Compensation of signal spillover in suspension and imaging mass cytometry. Cell Syst. 6 (5), 612-620 (2018).
- Bjornson, Z. B., Nolan, G. P., Fantl, W. J. Single-cell mass cytometry for analysis of immune system functional states. Curr Opin Immunol. 25 (4), 484-494 (2013).
- Kalina, T., Lundsten, K., Engel, P. Relevance of antibody validation for flow cytometry. Cytometry A. 97 (2), 126-136 (2020).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J Immunol Methods. 243 (1-2), 77-97 (2000).
- Roederer, M. Spectral compensation for flow cytometry: visualization artifacts, limitations, and caveats. Cytometry. 45 (3), 194-205 (2001).
- Tung, J. W., Parks, D. R., Moore, W. A., Herzenberg, L. A., Herzenberg, L. A. New approaches to fluorescence compensation and visualization of FACS data. Clin Immunol. 110 (3), 277-283 (2004).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon