Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Identifizierung und Analyse myogener Vorläuferzellen in vivo während akuter Skelettmuskelverletzung mittels hochdimensionaler Einzelzell-Massenzytometrie
In diesem Artikel
Zusammenfassung
Das hier vorgestellte Protokoll ermöglicht die Identifizierung und hochdimensionale Analyse von Muskelstamm- und Vorläuferzellen mittels Einzelzell-Massenzytometrie und deren Aufreinigung durch FACS für vertiefte Studien ihrer Funktion. Dieser Ansatz kann angewendet werden, um die Regenerationsdynamik in Krankheitsmodellen zu untersuchen und die Wirksamkeit pharmakologischer Interventionen zu testen.
Zusammenfassung
Die Regeneration der Skelettmuskulatur ist ein dynamischer Prozess, der von adulten Muskelstammzellen und ihren Nachkommen gesteuert wird. Adulte Muskelstammzellen, die meist in einem stationären Zustand ruhen, werden bei Muskelverletzungen aktiviert. Nach der Aktivierung vermehren sie sich, und die meisten ihrer Nachkommen differenzieren sich, um fusionskompetente Muskelzellen zu erzeugen, während sich der Rest selbst erneuert, um den Stammzellpool aufzufüllen. Während die Identität von Muskelstammzellen vor mehr als einem Jahrzehnt auf der Grundlage der Co-Expression von Zelloberflächenmarkern definiert wurde, wurden myogene Vorläuferzellen erst vor kurzem mit Hilfe hochdimensionaler Einzelzellansätze identifiziert. Hier stellen wir eine Einzelzell-Massenzytometrie-Methode (Cytometry by Time of Flight [CyTOF]) vor, um Stammzellen und Vorläuferzellen bei akuten Muskelverletzungen zu analysieren, um die zelluläre und molekulare Dynamik aufzuklären, die sich während der Muskelregeneration entfaltet. Dieser Ansatz basiert auf der gleichzeitigen Detektion neuartiger Zelloberflächenmarker und wichtiger myogener Transkriptionsfaktoren, deren dynamische Expression die Identifizierung von aktivierten Stammzellen und Vorläuferzellpopulationen ermöglicht, die Meilensteine der Myogenese darstellen. Wichtig ist, dass eine Sortierstrategie beschrieben wird, die auf dem Nachweis der Zelloberflächenmarker CD9 und CD104 basiert und die prospektive Isolierung von Muskelstamm- und Vorläuferzellen mittels Fluoreszenz-aktivierter Zellsortierung (FACS) für eingehende Studien ihrer Funktion ermöglicht. Muskelvorläuferzellen stellen ein entscheidendes fehlendes Bindeglied dar, um die Kontrolle des Schicksals von Muskelstammzellen zu untersuchen, neue therapeutische Ziele für Muskelerkrankungen zu identifizieren und zelltherapeutische Anwendungen für die regenerative Medizin zu entwickeln. Der hier vorgestellte Ansatz kann angewendet werden, um Muskelstamm- und Vorläuferzellen in vivo als Reaktion auf Störungen zu untersuchen, wie z.B. pharmakologische Interventionen, die auf bestimmte Signalwege abzielen. Es kann auch verwendet werden, um die Dynamik von Muskelstamm- und Vorläuferzellen in Tiermodellen von Muskelerkrankungen zu untersuchen, unser Verständnis von Stammzellerkrankungen zu verbessern und die Entwicklung von Therapien zu beschleunigen.
Einleitung
Die Skelettmuskulatur stellt das massemäßig größte Gewebe des Körpers dar und reguliert mehrere Funktionen, vom Sehvermögen bis zur Atmung, von der Haltung bis zur Bewegung sowie den Stoffwechsel1. Daher ist die Aufrechterhaltung der Integrität und Funktion der Skelettmuskulatur entscheidend für die Gesundheit. Das Skelettmuskelgewebe, das aus dicht gepackten Bündeln mehrkerniger Myofasern besteht, die von einem komplexen Netzwerk von Nerven und Blutgefäßen umgeben sind, weist ein bemerkenswertes regeneratives Potenzialauf 1,2.
Die Haupttreiber der Regeneration der Skelettmuskulatur sind adulte Muskelstammzellen (MuSCs). Sie sind aufgrund ihrer einzigartigen anatomischen Lage neben der Plasmamembran der Myofaser und unter der Basallamina auch als Satellitenzellen bekannt und wurden erstmals 1961identifiziert 3. MuSCs exprimieren einen einzigartigen molekularen Marker, die transkriptionsfaktorgepaarte Box 7 (Pax7)4. Bei gesunden Erwachsenen sind sie meist ruhig, werden bei Muskelverletzungen aktiviert und vermehren sich, um Nachkommen hervorzubringen, die sich (i) in fusionskompetente Muskelzellen differenzieren, die neue Myofasern bilden, um Muskelschäden zu reparieren, oder (ii) sich selbst erneuern, um den Stammzellpool aufzufüllen5.
Auf zellulärer und molekularer Ebene ist der Prozess der Regeneration recht dynamisch und beinhaltet Zellzustandsübergänge, die durch die koordinierte Expression wichtiger myogener Transkriptionsfaktoren, auch bekannt als myogene regulatorische Faktoren (MRFs), gekennzeichnet sind6,7. Frühere in vivo-Entwicklungsstudien, Experimente zur Abstammungsverfolgung und Zellkulturarbeiten mit Myoblasten haben gezeigt, dass die sequentielle Expression dieser Transkriptionsfaktoren die Myogenese antreibt, wobei der myogene Faktor 5 (Myf5) bei der Aktivierung exprimiert wird, die Expression der myogenen Differenzierung 1 (MyoD1) die Bindung an das myogene Programm markiert und die Expression von Myogenin (MyoG) die Differenzierungmarkiert 8,9,10,11. 12,13,14. Trotz dieses Wissens und der Entdeckung von Zelloberflächenmarkern zur Reinigung von MuSCs fehlten Strategien und Werkzeuge zur Identifizierung und Isolierung diskreter Populationen entlang des myogenen Differenzierungspfads und zur Auflösung einer myogenen Progression in vivo 15,16,17,18.
Hier stellen wir eine neuartige Methode vor, die auf kürzlich veröffentlichten Forschungsergebnissen basiert und die Identifizierung von Stamm- und Vorläuferzellen in der Skelettmuskulatur und die Analyse ihrer zellulären, molekularen und Proliferationsdynamik im Rahmen akuter Muskelverletzungen ermöglicht19. Dieser Ansatz beruht auf Einzelzell-Massenzytometrie (auch bekannt als Cytometry by Time of Flight [CyTOF]), um gleichzeitig wichtige Zelloberflächenmarker (α7 Integrin, CD9, CD44, CD98 und CD104), intrazelluläre myogene Transkriptionsfaktoren (Pax7, Myf5, MyoD und MyoG) und ein Nukleosidanalogon (5-Iod-2'-Desoxyuridin, IdU) zu detektieren, um Zellen in der S-Phase19,20 zu überwachen. 21,22,23. Darüber hinaus stellt das Protokoll eine Strategie vor, die auf dem Nachweis von zwei Zelloberflächenmarkern, CD9 und CD104, basiert, um diese Zellpopulationen durch fluoreszenzaktivierte Zellsortierung (FACS) zu reinigen und so zukünftige eingehende Studien ihrer Funktion im Zusammenhang mit Verletzungen und Muskelerkrankungen zu ermöglichen. Während primäre Myoblasten in der Vergangenheit ausgiebig verwendet wurden, um die späten Stadien der myogenen Differenzierung in vitro zu untersuchen, ist nicht bekannt, ob sie den molekularen Zustand von Muskelvorläuferzellen rekapitulieren, die in vivo gefunden wurden 24,25,26,27,28,29,30 . Die Herstellung von Myoblasten ist mühsam und zeitaufwändig, und der molekulare Zustand dieser Primärkultur ändert sich nach dem Passieren schnell31. Daher werden frisch isolierte myogene Vorläuferzellen, die mit dieser Methode gereinigt wurden, ein physiologischeres System zur Untersuchung der Myogenese und der Auswirkungen genetischer oder pharmakologischer Manipulationen ex vivo bieten.
Das hier vorgestellte Protokoll kann angewendet werden, um eine Vielzahl von Forschungsfragen zu beantworten, z.B. um die Dynamik des myogenen Kompartiments in vivo in Tiermodellen von Muskelerkrankungen zu untersuchen, als Reaktion auf akute genetische Manipulationen oder bei pharmakologischen Interventionen, wodurch unser Verständnis der Dysfunktion von Muskelstammzellen in verschiedenen biologischen Kontexten vertieft und die Entwicklung neuartiger therapeutischer Interventionen erleichtert wird.
Protokoll
Die Tierversuche wurden von der dänischen Tierversuchsaufsichtsbehörde genehmigt (Protokoll # 2022-15-0201-01293), und die Versuche wurden in Übereinstimmung mit den institutionellen Richtlinien der Universität Aarhus durchgeführt. Die Analgesie (Buprenorphin) wird 24 Stunden vor der Verletzung im Trinkwasser verabreicht, damit sich die Mäuse an den Geschmack anpassen können. Die Zufuhr von Buprenorphin ins Trinkwasser wird für 24 Stunden nach der Verletzung fortgesetzt. Zusammen mit einer subkutanen (s.c.) Injektion von Buprenorphin zum Zeitpunkt einer akuten Muskelverletzung lindert Buprenorphin im Trinkwasser nach Notexin-Injektion die mit der Verletzung verbundenen Schmerzen. Während es empfohlen wird, zum Zeitpunkt einer akuten Muskelverletzung eine s.c.-Injektion von Buprenorphin zu verabreichen, gefolgt von Buprenorphin im Trinkwasser, ist Buprenorphin im Trinkwasser vor der Verletzung optional. Forscher müssen jedoch die Tierschutzstandards und -richtlinien befolgen, die von der zuständigen Regulierungsbehörde festgelegt wurden.
HINWEIS: Für Einzelzell-Massenzytometrie-Experimente (CyTOF) an verletzten Muskeln der Hintergliedmaßen beginnen Sie bei Abschnitt 1: Analgesie in Wasser 24 Stunden vor der Muskelverletzung bis 24 Stunden nach der Verletzung. Für die Sortierung von Muskelstamm- und Vorläuferzellen von unverletzten Mäusen führen Sie die Abschnitte 5 und 6: Euthanasie + Skelettmuskeldissektion und -dissoziation durch und fahren Sie mit Abschnitt 11: Färbung mit Fluorophor-konjugierten Antikörpern für FACS fort. Eine Übersicht über den Versuchsaufbau und das Protokoll ist in Abbildung 1 dargestellt.
1. Analgesie in Wasser 24 h vor der Muskelverletzung bis 24 h nach der Verletzung
- Geben Sie in eine dunkle oder in Aluminiumfolie eingewickelte Trinkflasche 3 ml Buprenorphin (0,3 mg/ml) und füllen Sie sie auf 100 mL mit gefiltertem Wasser, um eine Endkonzentration von 0,009 mg/ml 24 Stunden vor der Verletzung zu erreichen. Befestigen Sie es am Mauskäfig.
- Entfernen Sie die Trinkflasche 24 Stunden nach der Verletzung und schließen Sie den Mauskäfig wieder an das Trinkventilsystem an.
2. Vorbereitung auf den Eingriff bei akuten Verletzungen
HINWEIS: Verwenden Sie 70% Ethanol, um die Werkbank, die Einrichtung des Nasenkonus und die Induktionsbox zu desinfizieren.
- Bereiten Sie ein 1,5-ml-Röhrchen mit verdünntem Notexin in PBS (5 μg/ml) vor. Verdünnen Sie Buprenorphin (0,3 mg/ml) in steriler 0,9%iger Kochsalzlösung auf 0,015 mg/ml in einem lichtsicheren 1,5-ml-Röhrchen. Bleiben Sie auf Eis. Bereiten Sie Insulinspritzen für Buprenorphin (28 G, 0,5 ml) und Notexin-Injektionen (29 G, 0,3 ml) vor.
- Konfigurieren Sie Isofluran-basierte Anästhesieeinheiten:
- Für die Anästhesie in einer Induktionsbox verwenden Sie 3 % Isofluran mit einem Durchfluss von 1,5 l/min (50 % O2, 50 % atmosphärische Luft).
- Verwenden Sie für die Wartung mit einem Nasenkonus-Setup 1,5 % Isofluran mit einem Durchfluss von 0,6 l/min (50 % O2, 50 % atmosphärische Luft).
3. Akute Verletzung durch Notexin-Injektion
VORSICHT: Notexin hat eine Phospholipase A2-Aktivität und ist der Hauptbestandteil des Giftes der australischen Tigernatter (Notechis scutatus) mit einer intravenösen LD50 von 5–17 mg Notexin/kg bei Mäusen32,33. Im vorliegenden Protokoll wird der Musculus tibialis anterior (TA) jeder Hintergliedmaße mit 10 μl 5 mg/ml Notexin injiziert, und der Gastrocnemius (GA)-Muskel jeder Hintergliedmaße wird zweimal (einmal in jeden Kopf des Muskels) mit 15 μl 5 mg/ml Notexin injiziert. Es ist wichtig, die intramuskulären (i.m.) Injektionen korrekt durchzuführen, um Schäden zu begrenzen, und die injizierten Tiere häufig zu inspizieren, um minimale Schmerzen zu gewährleisten.
- Betäuben Sie die Maus in der Induktionsbox (3% Isofluran). Wenn die Maus bewusstlos ist, senken Sie den Isofluranspiegel in der Induktionsbox auf 1,5 %.
- Wiegen und markieren Sie die Mäuse einzeln nach institutionell anerkannten Verfahren. Unter dem Nasenkegel die Hinterbeine mit einem Trimmer rasieren und zurück in die Induktionsbox geben.
- Berechnen Sie das Buprenorphin-Volumen für jede Maus :
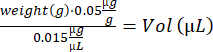
- Mischen Sie die Notexin-Lösung, indem Sie auf und ab pipettieren. Bereiten Sie 2 Insulinspritzen (29 g, 0,3 ml) mit 10 μl Notexin-Lösung (5 μg/ml) für TA-Injektionen und 4 Insulinspritzen mit 15 μl Notexin-Lösung (5 μg/ml) für die GA-Injektion vor.
HINWEIS: Der Musculus Tibialis anterior (TA) befindet sich an der vorderen Seite des unteren Beins der Maus und erstreckt sich vom Knie bis zum Knöchel. Der Musculus Gastrocnemius (GA) ist ein zweiköpfiger Muskel, der sich im hinteren Teil des Unterschenkels oberflächlich zum Soleus befindet. Er verläuft von seinen beiden Köpfen (medial und lateral) knapp über dem Knie bis zur Ferse und erstreckt sich über insgesamt drei Gelenke (Knie-, Sprunggelenk- und Subtalargelenk). - Intramuskuläre Injektionen durchführen
- TA-Injektion (insgesamt 2 Injektionen)
- Platzieren Sie die Maus in Rückenlage unter dem Nasenkonus und desinfizieren Sie die Injektionsstelle mit sterilen Alkoholtüchern (70 % Ethanol oder Isopropanol). Führen Sie die Nadel mit der Abschrägung nach unten in den Bauch des TA (distal zum Mittelbauch) in einem Winkel von 30° ein. Schieben Sie die Nadel entlang des Muskels und bewegen Sie sich parallel zur Tibia, um den Mittelbauch des TA zu erreichen. Injizieren Sie das Notexin langsam und kontinuierlich und lassen Sie die Nadel 10 s lang an Ort und Stelle, bevor Sie sie zurückziehen. Führen Sie die Nadel anfangs nicht zu tief ein (um zu vermeiden, dass Notexin unterhalb des TA in den Musculus extensor digitorum longus injiziert wird) und nicht zu weit proximal, wenn Sie die Nadel vorschieben (um eine zu nahe am Knie zu injizieren).
- GA-Injektion (insgesamt 4 Injektionen)
- Führen Sie die Nadel mit einer Abschrägung nach unten in einem Winkel von ~45° in den Mittelbauch des seitlichen Kopfes des GA ein. Injizieren Sie das Notexin langsam und kontinuierlich, wobei Sie die Nadel 10 s lang an Ort und Stelle lassen, bevor Sie sie zurückziehen. Drehen Sie die hintere Extremität und injizieren Sie Notexin in den medialen Kopf des GA-Muskels wie oben. Es sollte darauf geachtet werden, die Nadel nicht zu tief einzuführen.
- TA-Injektion (insgesamt 2 Injektionen)
- Drehen Sie die Maus um und injizieren Sie Buprenorphin s.c. mit einer Insulinspritze (28 G, 0,5 ml). Übertragen Sie die Maus in einen leeren Auffangkäfig auf einer Heizplatte. Es sollte nur die Hälfte des Käfigbodens über die Heizplatte gelegt werden, damit die Maus während der Wiederherstellung34 thermoregulieren kann. Wiederholen Sie die Schritte 3.3 bis 3.6 für die verbleibenden Mäuse.
- Wenn die Maus im Bergungskäfig vollständig aktiviert ist, setzen Sie sie wieder in den ursprünglichen Käfig ein. Bringen Sie es zurück in den Stall und ergänzen Sie es mit benetztem Futter. Entfernen Sie die Buprenorphin-Trinkflasche 24 h nach der Injektion.
- Überwachen Sie die Mäuse 6 Stunden nach der Notexin-Injektion und dann alle 12 Stunden für 2 Tage auf Anzeichen von Schmerzen, eingeschränkter Mobilität und verminderter Nahrungsaufnahme35,36.
4. 5-Iod-2'-Desoxyuridin-Injektion
ACHTUNG: 5-Iod-2'-Desoxyuridin (IdU) steht im Verdacht, genetische Defekte zu verursachen und die Fruchtbarkeit oder das ungeborene Kind zu schädigen. Lesen Sie vor der Handhabung das Sicherheitsdatenblatt (SDB). Während der Handhabung sollte persönliche Schutzausrüstung getragen werden. Verwenden Sie beim Wiegen des IdU-Pulvers einen Abzug. Materialien, die mit IdU in Berührung gekommen sind, sollten gemäß den örtlichen Sicherheitsvorschriften entsorgt werden.
HINWEIS: Die IdU-Markierung in vivo wird verwendet, um die Zellteilung während des Verlaufs der Verletzungszeit zu überwachen, da IdU, ein jodiertes Thymidin-Analogon, in die DNA von Zellen in der S-Phase eingebaut wird. IdU wird intraperitoneal (i.p.) mit 20 mg/kg Körpergewicht 8 h vor der Schädigung der Maus injiziert.
- Verwenden Sie ein Ultraschallgerät bei 37 °C, um eine 2 mg/ml-Stammlösung von IdU in sterilem PBS herzustellen. IdU ist lichtempfindlich; Am selben Tag verwenden oder bei -20 °C bis zu 3 Monate einfrieren. Bei Verwendung von gefrorenem IdU: Auftauen, Vortexen, 30 s lang bei 10.000 x g zentrifugieren und den Überstand für die Injektionen unten verwenden.
- Betäuben Sie die Maus in der Induktionsbox (3% Isofluran). Notieren Sie die Ohrmarkierung, wiegen Sie die Mäuse und berechnen Sie das Volumen der IdU-Lösung:
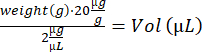
- Führen Sie eine i.p. Injektion mit einer Insulinspritze (28 G, 0,5 ml) durch und übertragen Sie die Maus in einen leeren Auffangkäfig auf einer Heizplatte. Wenn die Maus aktiviert ist, bewegen Sie sie zurück in ihren ursprünglichen Käfig. Wiederholen Sie die Schritte 4.2 bis 4.3 für die verbleibenden Mäuse und bringen Sie sie in den Stall zurück.
5. Euthanasie
HINWEIS: Siehe Tabelle 1 für Pufferrezepte. Waschmedium (Nährstoffmischung F-10 (Schinken), 10% Pferdeserum, 1x Pen/Strepto) vorbereiten und durch eine Polyethersulfon (PES)-Membran in einen Styroporbehälter filtrieren. Dissoziationspuffer vorbereiten (Waschmedium ergänzt mit 650 U/ml Kollagenase, Typ II) und auf Eis legen. CyTOF-Massenzytometrie-Messungen sind sehr empfindlich gegenüber Verunreinigungen. Aus diesem Grund ist es unerlässlich, für die Probenaufbereitung Reagenzien von höchster analytischer Qualität zu verwenden. Um eine Kontamination mit Metallen zu vermeiden, wird dringend empfohlen, sterile Kunststoffartikel und neue Glaswaren zu verwenden, die noch nie mit Reinigungsmittel gewaschen wurden, da viele Laborseifen einen hohen Bariumgehalt aufweisen. Es wird empfohlen, für die Reagenzienvorbereitung doppelt gefiltertes, destilliertes, deionisiertes Wasser zu verwenden. Phosphatgepufferte Kochsalzlösung (PBS) wird im eigenen Haus hergestellt. Verdünnen Sie die 10x Vorräte auf 1x und filtrieren Sie die 1x PBS mit 0,2 μm Filtern. Filtern Sie die 1x PBS zu Beginn jedes Experiments erneut. Sezierwerkzeuge dürfen aufgrund des Vorhandenseins von Barium nicht mit Reinigungsmittel gereinigt werden.
- Bereiten Sie die Waage, den Behälter und das Anästhesiegerät auf Isofluranbasis mit der Induktionsbox vor. Desinfizieren Sie geeignete Sezierwerkzeuge.
- Bereiten Sie 50-ml-Röhrchen für jede Maus vor und markieren Sie sie mit 5 mL Dissoziationspuffer und einer Petrischale. Bewahren Sie den Dissoziationspuffer auf Eis auf.
- Betäuben Sie die Maus in der Induktionsbox (3 % Isofluran, bei Bewusstlosigkeit auf 5 % erhöhen) und führen Sie eine Zervixluxation durch.
6. Dissektion und Dissoziation der Skelettmuskulatur
- Präparieren Sie TA- und GA-Muskeln von beiden Hintergliedmaßen und übertragen Sie sie in den Deckel einer Petrischale. Schneiden Sie das Gewebe mit einer Schere in eine zerkleinerte Aufschlämmung (~1 mm3 Stücke). In ein 50-ml-Röhrchen mit 5 mL eiskaltem Dissoziationspuffer umfüllen. Bleiben Sie auf Eis.
- Wiederholen Sie das Euthanasieverfahren (Schritt 5.3) und das Verfahren der Skelettmuskeldissektion und -dissoziation (Schritt 6.1), bis alle Mäuse bearbeitet wurden.
- Alle Probenröhrchen in einem 37 °C warmen Wasserbad (2 min) vorheizen und 45 min im Wechsel in einem Inkubator (37 °C) inkubieren.
- Vortex und Waschen durch Zugabe von 10 mL Waschmedium. Zentrifugieren (380 x g, 10 min, Raumtemperatur [RT]) und auf 4 mL aspirieren.
- Fügen Sie 0,5 ml Kollagenase (1000 U/ml) und 0,5 mL Dispase (11 U/ml) hinzu. Vortexen und 20 min in Rotation bei 37 °C inkubieren (z. B. in einem Inkubator). Zentrifugieren (380 x g, 1 min, RT) und mit einer 5 mL Pipette resuspendieren.
- Geben Sie 40 μm Zellsiebe in 50 ml-Röhrchen und befeuchten Sie sie mit 5 mL Waschmedium. Aspirieren und ausstoßen Sie die Zellsuspension 10 Mal mit einer 5-ml-Spritze und einer 20-g-Nadel. Durch das vorbenetzte 40 μm Zellsieb abseihen. Waschen Sie das 50-ml-Röhrchen mit 10 mL-Waschmedium, geben Sie es in das 40-μm-Zellsieb und zentrifugieren Sie die Zellsuspension (380 x g, 10 min, 4 °C).
7. Lebend-/Totfärbung mit Cisplatin- und Paraformaldehyd-Fixierung
ACHTUNG: Cisplatin und Paraformaldehyd (PFA) sind krebserregend. Lesen Sie das Sicherheitsdatenblatt, bevor Sie es verwenden. Paraformaldehyd (PFA; 16%) ist ein Reizstoff für Haut, Augen und Atemwege. Tragen Sie persönliche Schutzausrüstung und handhaben Sie diese Substanzen unter einem Abzug. Während der Fixierung der Zellen beträgt die Endkonzentration von PFA 1,6 %. Es sollten die richtigen Schutzmaßnahmen ergriffen werden, und der Abfall sollte gemäß den örtlichen Vorschriften behandelt werden.
HINWEIS: Bereiten Sie kaltes (4 °C) und warmes (37 °C) serumfreies DMEM vor. Bereiten Sie DMEM zu, das mit 10 % FBS angereichert ist, filtrieren Sie durch eine PES-Membran in einen Styroporbehälter und bewahren Sie es auf Eis auf. Bereiten Sie PBS und Zellfärbemedien (CSM; PBS, 0,5 % BSA, 0,02 % Natriumazid) in einer CyTOF-speziellen Glasflasche und filtrieren Sie durch eine PES-Membran. CSM kann bis zu 6 Monate bei 4 °C gelagert werden.
- Aspirieren Sie den Überstand, schnippen Sie das Pellet und resuspendieren Sie es in 1 ml kaltem, serumfreiem DMEM. Zählen Sie die Zellen und stellen Sie die Zelldichte auf 1 x 106 Zellen/ml in serumfreiem DMEM ein.
- Geben Sie Cisplatin-Stamm (25 mM) bis zu einer Endkonzentration von 25 μM. Vortex 10 s und inkubieren Sie genau 1 min lang (die Reaktion ist sehr zeitkritisch). Quaktieren Sie die Reaktion mit eiskaltem DMEM + 10 % FBS (3-faches Probenvolumen) und halten Sie sie auf Eis. Zentrifugieren (380 x g, 10 min, 4 °C), den Überstand absaugen und das Pellet gründlich resuspendieren (bis zu 10 x 106 Zellen/ml in CSM). Die Suspension wird durch ein 35 μm Zellsieb filtriert.
- Fixieren Sie die Zellsuspension mit gefilterter PFA-Stammlösung (16 %) und pipettieren Sie sie auf und ab (unter einem Abzug), um die endgültige PFA-Konzentration (1,6 %) zu erreichen (z. B. fügen Sie 100 μl 16 % PFA zu 900 μl Zellsuspension hinzu). 30 s vortexen, 10 min auf Eis inkubieren und zweimal mit 2 mL CSM durch Zentrifugation waschen (800 x g, 5 min, RT).
HINWEIS: Zu diesem Zeitpunkt können die Proben entweder auf Trockeneis schockgefroren und bei -80 °C gelagert oder direkt für die Antikörperfärbung verwendet werden. Wenn Zellen eingefroren werden sollen, füllen Sie die Zellen in 5-ml-Polypropylen-Röhrchen um, da Polystyrol bei niedrigen Temperaturen reißen kann. Wenn frisch fixierte Zellen für die Färbung verwendet werden, fahren Sie mit Schritt 8.2 fort.
8. Färbung mit metallkonjugierten Antikörpern
ACHTUNG: Methanol (MeOH) ist leicht entzündlich und ätzend für die Atemwege. Lesen Sie das Sicherheitsdatenblatt, bevor Sie es verwenden. Tragen Sie persönliche Schutzausrüstung und handhaben Sie diesen Stoff unter einem Abzug. Behandeln Sie Abfälle in Übereinstimmung mit den örtlichen Vorschriften.
HINWEIS: Eine Liste der Antikörper (Ab), die auf Oberflächenmarker und intrazelluläre Marker abzielen, finden Sie in Tabelle 2.
Antikörper-Konjugation: Die meisten der in diesem Protokoll verwendeten Antikörper wurden intern konjugiert, da sie nicht im Handel erhältlich waren. Protokolle für die Metallkonjugation von Antikörpern wurden bereits veröffentlicht, und Konjugationskits sind jetzt kommerziell erhältlich37,38. Immunglobulin Typ G (IgG) ist mit den verfügbaren Konjugationsprotokollen kompatibel. Es ist von großer Bedeutung, dass die für die Metallkonjugation verwendete Antikörperformulierung frei von Cystein-haltigen Trägerproteinen (z. B. Rinderserumalbumin (BSA)) ist, die die Konjugationseffizienz beeinflussen können, indem sie um die freien Maleimidgruppen des Polymers konkurrieren, und die Quantifizierung des metallkonjugierten Antikörpers beeinträchtigen können. Der Cysteingehalt von Gelatine ist viel niedriger als der von BSA. Es wird jedoch empfohlen, dass, wenn die Antikörperformulierung Trägerproteine enthält, diese Proteine vor der Konjugation entfernt werden. Es ist nun möglich, BSA- und gelatinefreie Antikörper beim Hersteller anzufordern. Konservierungsmittel für kleine Moleküle (z. B. Natriumazid, Glycerin und Trehalose) sind mit Metallkonjugationsprotokollen kompatibel37,38.
Antikörpertitration: Nach jeder Metallkonjugation sollten Antikörper titriert werden, um die optimale Antikörperkonzentration zu bestimmen, die das maximale Signal-Rausch-Verhältnis bietet. Für die Antikörpertitration führen Sie eine 6-stufige zweifache serielle Verdünnung durch und färben Sie sowohl Proben, von denen bekannt ist, dass sie das interessierende Protein exprimieren (z. B. Muskelzellen, Positivkontrollen) als auch fehlen (Negativkontrollen) 19,21,37,38.
Bereiten Sie frische Cell-ID Intercalator-Ir (Stamm = 500 μM; Interkalator-ir-Lösung) Arbeitslösung vor, indem Sie die Rohlösung auf 0,1 μM in PBS/1,6 % PFA verdünnen.
- Gefrorene Proben 5 min bei RT auftauen und zentrifugieren (800 x g, 1 min, RT). Mit 2 mL CSM durch Zentrifugation waschen (800 x g, 5 min, RT).
- Bereiten Sie eine 2,5-fache Mischung aus Oberflächenantikörpern (Ab) in CSM vor. Aspirieren Sie den Überstand auf ~60 μL und resuspendieren Sie das Pellet gründlich. Färben Sie die Zellen, indem Sie 40 μl Oberflächen-Ab-Färbemischung hinzufügen und 1 h lang bei RT inkubieren.
- Streichen Sie die Samples alle 20 Minuten, um sie zu mischen. Zweimal mit 1 mL CSM durch Zentrifugation waschen (800 x g, 5 min, RT). Sauge den Überstand an und schnippe das Kügelchen.
HINWEIS: Wenn keine intrazelluläre Färbung erforderlich ist, ist keine Methanolpermeabilisierung erforderlich. Wenn jedoch nur eine Oberflächenfärbung durchgeführt wird, muss die Interkalator-ir-Lösung in einem Puffer verdünnt werden, der ein Permeabilisierungsmittel (z. B. Maxpar fix und perm Puffer) enthält, um die Kernmembran zu durchdringen. Fahren Sie mit Schritt 8.5 fort und verdünnen Sie den Cell-ID Intercalator-Ir in einem Puffer, der ein Permeabilisierungsmittel und 1,6 % PFA enthält. Andernfalls fahren Sie mit der intrazellulären Färbung unten fort.
ACHTUNG: Iridium ist gefährlich und es müssen Vorsichtsmaßnahmen für eine sichere Handhabung getroffen werden. Iridium ist brennbar und reizt die Augen/die Haut. Vermeiden Sie es, Staub oder Dämpfe zu erzeugen und einzuatmen. Vermeiden Sie den Kontakt mit Haut und Augen. Das Interkalator-IR-Lösungsgemisch wird jedoch in einer Konzentration von <1 % in Wasser bereitgestellt, das nach dem global harmonisierten System (GHS) zur Einstufung und Kennzeichnung von Chemikalien als ungefährlich gilt. Lesen Sie vor der Handhabung das Sicherheitsdatenblatt von Standard Biotools. Während der Handhabung sollte persönliche Schutzausrüstung getragen werden. Materialien, die mit der Interkalator-IR-Lösung in Berührung gekommen sind, sollten gemäß den örtlichen Sicherheitsvorschriften entsorgt werden. - Um die Zellen zu permeabilisieren, fügen Sie während des Vortexens tropfenweise 0,5 mL eiskaltes MeOH hinzu. 15 Minuten auf Eis unter einem Abzug inkubieren. Zentrifugieren (800 x g, 5 min, RT) und dann zweimal mit 1 mL CSM durch Zentrifugation waschen (800 x g, 5 min, RT). Nach der letzten Wäsche den Überstand auf ~60 μL ansaugen und das Pellet gründlich resuspendieren.
- Bereiten Sie eine 2,5x intrazelluläre Ab-Färbemischung in CSM vor. Färben Sie die Zellen, indem Sie 40 μl intrazelluläre Ab-Färbemischung hinzufügen und 1 h bei RT inkubieren. Streichen Sie die Proben alle 20 Minuten, um sie zu mischen.
- Zweimal mit 1 mL CSM durch Zentrifugation waschen (800 x g, 5 min, RT). Sauge den Überstand an und schnippe das Kügelchen. Die Proben werden in 0,5 ml Interkalator-Iridium-Lösung (Tabelle 1) resuspendiert und vortext. Die Proben werden 1 h lang bei RT oder über Nacht (O/N) bei 4 °C inkubiert (siehe Hinweis unten).
HINWEIS: Die Proben können in einer Interkalator-Iridium-Lösung bei 4 °C bis zu 48 Stunden gelagert werden. Das in dieser Studie verwendete Färbeprotokoll wurde auf der Grundlage der Pionierarbeit des Nolan-Labors{C}23 entwickelt. Es unterscheidet sich von den Standardprotokollen von Biotools dadurch, dass (i) hauseigene hergestellte Puffer zum Waschen und Färben verwendet werden, (ii) die Zellen vor der Oberflächenfärbung fixiert werden und (iii) die Zellen mit Methanol für die intrazelluläre Färbung mit Antikörpern gegen Transkriptionsfaktoren oder Signalmoleküle permeabilisiert werden. Forscher, die ein neues Protokoll entwickeln, müssen die Verträglichkeit ihres Antikörper-Panels mit Methanol als Permeabilisierungsmittel gründlich testen. Beim Hinzufügen neuer Antikörper zu einem Panel wird empfohlen, die Antikörperspezifität durch Durchflusszytometrie mit positiven und negativen Kontrollproben vor der Metallkonjugation und Titration zu testen.
9. Probenvorbereitung für das Laden in das Massenzytometer
HINWEIS: Zellpellets sind sehr locker, wenn sie sich in CAS-Puffer befinden (Materialtabelle). Bei Wäschen mit CAS-Puffer nicht bis zur Trockenheit ansaugen. Behalten Sie stattdessen ein Restvolumen bei, wie unten beschrieben.
- Vortex und zentrifugieren (800 x g, 10 min, RT) die Proben. Gießen Sie den Überstand ab (Griff als PFA-Abfall) und wirbeln Sie ihn ein.
- Mit 1 mL CSM durch Zentrifugation (800 x g, 10 min, RT) waschen. Sauge den Überstand an und schnippe das Kügelchen.
- Mit 1 mL CAS-Puffer durch Zentrifugation waschen (800 x g, 10 min, RT). Aspirieren Sie auf ~200 μl. Vortex und fügen Sie 1 mL CAS-Puffer hinzu. 5 μl aliquot für die Zellzahl herausnehmen. Zentrifugieren (800 x g, 10 min, RT) und vorsichtig auf ≤50 μl aspirieren. Stören Sie das Pellet nicht.
- Resuspendieren Sie das Pellet auf 1–2 x 106 Zellen/ml in CAS-Puffer und fügen Sie Kalibrierperlen hinzu (1x; Tabelle der Materialien) auf eine Endkonzentration von 0,1x (z. B. 100 μl 1x Kalibrierkugeln zu 900 μl Zellsuspension hinzufügen).
- Laden Sie die Probe in das Massenzytometer und sammeln Sie Daten mit einer Flussrate von 400–500 Zellen/s.
- Führen Sie eine Datennormalisierung von FCS-Dateien mit der CyTOF-Software oder zuvor entwickelten Normalisierungswerkzeugendurch 39.
HINWEIS: Der Betrieb von CyTOF-Massenzytometern ist gerätespezifisch40,41. Es wird empfohlen, vor dem Betrieb die Bedienungsanleitung von CyTOF zu konsultieren. Bei den Kalibrierkugeln handelt es sich um metalleingebettete Polystyrol-Normalisierungskugelstandards, die bekannte Konzentrationen der Metallisotope Cer (140/142Ce), Europium (151/153Eu), Holmium (165Ho) und Lutetium (175/176Lu) enthalten. Die Kalibrierkügelchen ermöglichen die Kontrolle der Maschinenempfindlichkeit, die im Laufe der Zeit variieren kann, vor allem aufgrund der Ansammlung von biologischem Material und Schwankungen der Plasmaionisation im Laufe der Zeit.
10. CyTOF-Datenanalyse
HINWEIS: Für die nachgelagerte Analyse können normalisierte FCS-Dateien lokal analysiert oder in Cloud-basierte Softwarelösungen wie Cytobank, Cell Engine, OMIQ oder FCS Express42 hochgeladen werden.
- Verketten Sie bei Bedarf einzelne FCS-Dateien für jedes Beispiel in einer einzigen Datei.
- Identifizierung einzelner Zellen durch Gating auf Iridium-Interkalator-positive Ereignisse, was die Unterscheidung von einzelkernigen Zellen von Trümmern oder Dubletten ermöglicht.
- Gate für lebende Zellen durch Selektion von Cisplatin-negativen Ereignissen. Cisplatin bindet kovalent an zelluläre Proteine und markiert sterbende und tote Zellen mit kompromittierten Membranen in größerem Maße als lebende Zellen43.
- Gate auf die interessierende Population, z. B. das myogene Kompartiment (Lebend/CD45-/CD31-/Sca1-/α7Integrin+/CD9+) (Abbildung 2A), und quantifizieren Sie den relativen Anteil von Stamm- und Vorläuferzellen. Dieser Ansatz erfordert Vorkenntnisse über die Expression von Zelloberflächen- oder intrazellulären Markern, um einzelne Populationen zu definieren.
- Durchführung einer hochdimensionalen Analyse, die die Identifizierung bisher unerkannter seltener Zelluntergruppen innerhalb einer komplexen Population ermöglicht, Export der interessierenden Population und Verwendung von Clustering-Algorithmen, die speziell für die CyTOF-Datenanalyse entwickelt wurden44.
HINWEIS: Frühere Arbeiten verwendeten den X-Shift-Algorithmus, der die gewichtete k-Nächste-Nachbarn-Dichteschätzung (kNN-DE) im hochdimensionalen Raum verwendet, um unüberwachtes Clustering auf der Grundlage definierter Parameterdurchzuführen 19. Es hat sich gezeigt, dass die X-Verschiebung bei der Identifizierung seltener Zellpopulationen sehr effektiv ist45. - Laden Sie für die X-Shift-Analyse das Vortex-Softwarepaket (von der GitHub-Seite des Nolan-Labors [https://github.com/nolanlab/vortex]) und Java 64-Bit46 herunter. Eine Anleitung finden Sie hier: https://github.com/nolanlab/vortex/wiki/Getting-Started.
- Laden Sie die exportierten Zellpopulationen in eine lokale Datenbank hoch und definieren Sie die Clustering-Parameter. Zu den benutzerdefinierten Parametern gehören (i) die Marker, die für das Clustering verwendet werden, (ii) der Bereich der k-Werte (z. B. 5 bis 150) und (iii) die Anzahl der Clustering-Schritte.
HINWEIS: Frühere Arbeiten verwendeten als Clustering-Marker eine Kombination von Oberflächenmarkern, von denen bekannt ist, dass sie in Muskelstammzellen exprimiert werden, neuartigen Oberflächenmarkern, die in einem durchflusszytometrischen Hochdurchsatz-Screening von Muskelzellen und Myoblasten identifiziert wurden, und myogenen Transkriptionsfaktoren (TFs), von denen bekannt ist, dass sie verschiedene Stadien der Myogenese definieren. Dieser Ansatz ermöglichte die Identifizierung von zwei Zelloberflächenmarkern, CD9 und CD104, deren Co-Expressionsmuster bisher unerkannte Vorläuferzellpopulationen unterscheidet19. Die sorgfältige Auswahl der Clustering-Marker ermöglicht es dem Forscher, spezifische Forschungsfragen zu beantworten. Wenn z. B. die Identifizierung und Analyse von sich teilenden Zelluntergruppen im Zeitverlauf gewünscht wird, wird empfohlen, IdU als einen der Clustering-Marker einzubeziehen. X-Shift kann Clustering bei mehreren k-Werten durchführen und durch Berechnung des "Ellbogenpunkts" automatisch den k-Wert identifizieren, der zu einer optimalen Cluster-Anzahl führt, wodurch ein Unter-Cluster-Wachstum oder eine übermäßige Fragmentierung vermieden wird. Es wird empfohlen, den Ellbogenpunkt zu berechnen und die Clustering-Analyse zu verwenden, die durch den optimalen k-Wert für die Datenvisualisierung und nachgelagerte Anwendungen definiert ist. - Um die räumlichen Beziehungen zwischen Zellpopulationen innerhalb der X-Shift-Cluster zu visualisieren, führen Sie ein kraftgerichtetes Layout durch, das eine 2D-Karte erzeugt, in der der Abstand zwischen den Clustern ihre Ähnlichkeit in der Markerexpression im hochdimensionalen phänotypischen Raum anzeigt. Durch das Einfärben der Karte mit jeweils einem Marker ist es möglich, neue Zellpopulationen zu entdecken und ihre Dynamik zu verfolgen. Eine anschließende Quantifizierung durch manuelles Gating in Software wie Cytobank und Cell Engine wird empfohlen.
- Führen Sie Heatmap-Analysen durch, um die dynamische Expression mehrerer Oberflächen- und intrazellulärer Proteine im Laufe der Zeit zu quantifizieren und neue Trends aufzudecken.
HINWEIS: Bei der Durchführung eines Unfallzeitkurses ist es möglich, Daten von allen Zeitpunkten zu gruppieren, um eine Karte der Regeneration zu erstellen. Darüber hinaus ist es möglich, eine solche Karte in die einzelnen Zeitpunkte zu zerlegen, um die zelluläre und molekulare Dynamik der Regeneration im Zeitverlauf zu verfolgen19.
11. Färbung mit Fluorophor-konjugierten Antikörpern für FACS
HINWEIS: Zellen, die für ungefärbte, einfarbige Steuerungen und Fluoreszenz-Minus-Eins-Kontrollen (FMO) verwendet werden, können aus dem TA- und GA-Set einer zusätzlichen Maus stammen, falls verfügbar. Alternativ kann der Quadrizeps (Oberschenkelmuskel) präpariert und in eine Einzelzellsuspension verdaut werden, wobei das gleiche Verfahren wie bei der oben genannten TA+GA angewendet und für Kontrollen verwendet wird. FACS-Puffer (PBS, 2,5 % Ziegenserum, 2 mM EDTA) vorbereiten, durch eine PES-Membran in einen Polystyrolbehälter filtrieren und auf Eis legen. FACS-Puffer kann bis zu 1 Monat bei 4 °C gelagert werden. Eine Liste der Antikörper, die für FACS verwendet werden, ist in Tabelle 3 zu finden.
- Bereiten Sie eine Abstammungsmischung vor. Färben Sie jede Kontrolle in 50 μL (enthält ca. 3–5 x 105 Zellen). Berechnen Sie die Menge der benötigten Abstammungsmischung. Machen Sie 20 % bis 30 % Volumenüberschuss. Die Stammkonzentration der Antikörper, die für den Lineage-Mix verwendet werden, beträgt 0,2 mg/ml.
- Fügen Sie in einem 0,5-ml-Röhrchen Anti-CD45 APC-Cy7, Anti-CD31 APC-Cy7, Anti-Sca1 APC-Cy7 und Anti-CD11b APC-Cy7 hinzu, um Endkonzentrationen von 1 μg/ml, 2,5 μg/ml, 2,5 μg/ml bzw. 0,63 μg/ml zu erreichen.
- Bereiten Sie eine Mischung aus reiner Beize vor. Färben Sie jede vollständig gefärbte Probe in 500 μl (enthält ca. 3–5 x 106 Zellen). Machen Sie 10 % Volumenüberschuss mit All-Stain-Mix.
- Fügen Sie in einem 1,5-ml-Röhrchen Antikörper wie oben + Anti-α 7-Integrin PE, Anti-CD9 APC und Anti-CD104 FITC hinzu, um Endkonzentrationen von 2 μg/ml, 1,2 μg/ml bzw. 3 μg/ml zu erreichen.
- FACS-Puffer + DAPI vorbereiten: Für 10 ml FACS-Puffer 1 μl DAPI (Stamm = 1 mg/ml) hinzufügen, um eine DAPI-Endkonzentration von 100 ng/ml zu erreichen.
ACHTUNG: DAPI (4',6-Diamidino-2-phenylindoldihydrochlorid) wird als mögliches Reizmittel für die Haut und die Atemwege eingestuft. DAPI-Lösung in einer Konzentration von <1 % gilt jedoch laut GHS als ungefährlich. Lesen Sie das Sicherheitsdatenblatt, bevor Sie es verwenden. Während der Handhabung sollte persönliche Schutzausrüstung getragen werden. Materialien, die mit DAPI in Berührung gekommen sind, sollten gemäß den örtlichen Sicherheitsvorschriften entsorgt werden. - Resuspendierung von Zellen aus Abschnitt 6: Dissektion und Dissoziation der Skelettmuskulatur:
- Bei vollständig gefärbten Proben resuspendieren Sie TA- und GA-Sets von einer einzigen Maus in 500 μl FACS-Puffer und übertragen Sie sie zur Färbung in 15-ml-Röhrchen.
- Verwenden Sie für Steuerelemente entweder TA- und GA-Sets oder Quadrizeps-Sets. Wenn Sie 1 Maus zur Steuerung verwenden, resuspendieren Sie die Zellen in 750 μl FACS-Puffer. Bei mehreren Mäusen resuspendieren Sie jedes TA- und GA- oder Quadrizeps-Set in 500 μl FACS-Puffer und kombinieren Sie die Sets zu einer einzigen 1-ml-Probe, indem Sie von jeder Probe einen Bruchteil entnehmen. Geben Sie 50 μl Kontrollzellen (3–5 x 105 Zellen) zur Färbung in 5 ml-Polypropylenröhrchen.
- Wie in Tabelle 4 beschrieben 45 min bei 4 °C im Dunkeln färben.
- Waschen Sie alle gefärbten Proben, indem Sie 5 ml FACS-Puffer hinzufügen. Waschen Sie die Steuerungen durch Zugabe von 1 ml FACS-Puffer und Zentrifuge (380 x g, 10 min, 4 °C).
- Resuspendieren Sie alle gefärbten Proben in 1 ml FACS-Puffer + DAPI. Die Kontrollen werden in 300 μl FACS-Puffer mit oder ohne DAPI resuspendiert.
- Halten Sie die Zellen bis zur Sortierung auf einem Durchflusszytometer mit 4 Lasern (405 nm, 488 nm, 561 nm, 633 nm) bei 4 °C im Dunkeln.
- Führen Sie die Kontrollen und Proben auf dem Sorter aus und erstellen Sie Sortiertore mit der entsprechenden Software. Zeichnen Sie für nicht gefärbte/einfarbige Steuerelemente und FMO-Steuerelemente 1 x 104 bzw. 0,3–1 x 105 Ereignisse auf. Bei vollständig gefärbten Proben können bis zu 1 x 106 Ereignisse aufgezeichnet werden. Sortieren Sie alle vollständig gefärbten Proben oder so viel wie nötig, je nach den Anforderungen des nachgelagerten Assays. Für das hier verwendete Durchflusszytometer (FACSAria III) sortieren Sie im Reinheitsmodus mit einer 70-mm-Düse.
- In Abbildung 4 finden Sie die Gating-Strategie.
- Verwenden Sie die ungefärbten und einfarbigen Regler, um die Spannung für alle Detektoren einzustellen.
- Verwenden Sie die einfarbigen Steuerelemente, um die Kompensationsmatrix einzurichten.
- Verwenden Sie die FMO-Steuerungen, um Sortiergatter einzurichten.
- Unterschiedliche Muskelstamm- und Vorläuferzellpopulationen werden in eiskalten FACS-Puffer sortiert.
- Zentrifugieren Sie die Zellpopulationen (380 x g, 10 min, 4 °C). Unterbrechen Sie die Unterbrechung im entsprechenden Puffer erneut, zählen Sie und fahren Sie mit der nachgelagerten Analyse fort.
Ergebnisse
Hier präsentieren wir einen Überblick über den experimentellen Aufbau für die Verwendung dieses kombinierten Ansatzes, der Folgendes umfasst: (i) hochdimensionale CyTOF-Analyse eines akuten Verletzungszeitverlaufs durch Notexin-Injektion zur Untersuchung der zellulären und molekularen Dynamik von Stamm- und Vorläuferzellen im Skelettmuskel (Abbildung 1, oberes Schema); und (ii) FACS von Stamm- und Vorläuferzellen unter Verwendung von zwei Zelloberflä...
Diskussion
Die Regeneration der Skelettmuskulatur ist ein dynamischer Prozess, der auf der Funktion adulter Stammzellen beruht. Während sich frühere Studien auf die Rolle von Muskelstammzellen während der Regeneration konzentriert haben, wurden ihre Nachkommen in vivo zu wenig untersucht, vor allem aufgrund eines Mangels an Instrumenten zur Identifizierung und Isolierung dieser Zellpopulationen 15,16,17,18.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Wir danken den Mitgliedern der FACS Core Facility in der Abteilung für Biomedizin der Universität Aarhus für die technische Unterstützung. Wir danken Alexander Schmitz, dem Leiter der Abteilung Massenzytometrie an der Klinik für Biomedizin, für das Gespräch und die Unterstützung. Wissenschaftliche Illustrationen wurden mit Biorender.com erstellt. Diese Arbeit wurde durch einen Starting Grant des Aarhus Universitets Forskningsfond (AUFF) und einen Start Package Grant (0071113) der Novo Nordisk Foundation an E.P. finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 15 mL centrifuge tube | Fisher Scientific | 07-200-886 | |
| 20 G needle | KDM | KD-fine 900123 | |
| 28 G, 0.5 mL insulin syringe | BD | 329461 | |
| 29 G, 0.3 mL insulin syringe | BD | 324702 | |
| 3 mL syringes | Terumo medical | MDSS03SE | |
| 40 µm cell strainers | Fisher Scientific | 11587522 | |
| 5 mL polypropylene tubes | Fisher Scientific | 352002 | |
| 5 mL polystyrene test tubes with 35 µm cell strainer | Falcon | 352235 | |
| 5 mL syringes | Terumo medical | SS05LE1 | |
| 50 mL centrifuge tube | Fisher Scientific | 05-539-13 | |
| 5-Iodo-2-deoxyuridine (IdU) | Merck | I7125-5g | |
| anti-CD104 FITC (clone: 346-11A) | Biolegend | 123605 | Stock = 0.5 mg/mL |
| anti-CD11b APC-Cy7 (Clone: M1/70) | Biolegend | 101226 | Stock = 0.2 mg/mL |
| anti-CD31 APC-Cy7 (clone: 390) | Biolegend | 102440 | Stock = 0.2 mg/mL |
| anti-CD45 APC-Cy7 (Clone: 30-F11) | Biolegend | 103116 | Stock = 0.2 mg/mL |
| anti-CD9 APC (clone: KMC8) | ThermoFisher Scientific | 17-0091-82 | Stock = 0.2 mg/mL |
| anti-Sca1 (Ly6A/E) APC-Cy7 (clone: D7) | Biolegend | 108126 | Stock = 0.2 mg/mL |
| anti-α7 integrin PE (clone: R2F2)) | UBC AbLab | 67-0010-05 | Stock = 1 mg/mL |
| BD FACS Aria III (4 laser) instrument | BD Biosciences | N/A | 405, 488, 561, and 633 nm laser |
| Bovine Serum Albumin | Sigma Aldrich | A7030-50G | |
| Buprenorphine 0.3 mg/mL | Ceva | Vnr 054594 | |
| CD104 (Clone: 346-11A) | BD Biosciences | 553745 | Dy162; In-house conjugated |
| CD106/VCAM-1 (Clone: 429 MVCAM.A) | Biolegend | 105701 | Er170; In-house conjugated |
| CD11b (Clone: M1/70) | BD Biosciences | 553308 | Nd148; In-house conjugated |
| CD29/Integrin β1 (Clone: 9EG7) | BD Biosciences | 553715 | Tm169; In-house conjugated |
| CD31 (Clone: MEC 13.3) | BD Biosciences | 557355 | Sm154; In-house conjugated |
| CD34 (Clone: RAM34) | BD Biosciences | 551387 | Lu175; In-house conjugated |
| CD44 (Clone: IM7) | BD Biosciences | 550538 | Yb171; In-house conjugated |
| CD45 (Clone: MEC 30-F11) | BD Biosciences | 550539 | Sm147; In-house conjugated |
| CD9 (Clone: KMC8) | Thermo Fisher Scientific | 14-0091-85 | Yb174; In-house conjugated |
| CD90.2/Thy1.2 (Clone: 30-H12) | BD Biosciences | 553009 | Nd144; In-house conjugated |
| CD98 (Clone: H202-141) | BD Biosciences | 557479 | Pr141; In-house conjugated |
| Cell Acquisition Solution/Maxpar CAS-buffer | Standard Biotools | 201240 | |
| Cell-ID Intercalator-Iridium | Standard Biotools | 201192B | cationic nucleic acid intercalator |
| Cisplatin | Merck | P4394 | Pt195 |
| Cisplatin (cis-Diammineplatinum(II) dichloride) | Merck | P4394 | |
| Clear 1.5 mL tube | Fisher Scientific | 11926955 | |
| Collagenase, Type II | Worthington Biochemical Corporation | LS004177 | |
| Counting chamber | Merck | BR718620-1EA | |
| CXCR4/SDF1 (Clone: 2B11/CXCR4 ) | BD Biosciences | 551852 | Gd158; In-house conjugated |
| DAPI (1 mg/mL) | BD Biosciences | 564907 | |
| Dark 1.5 mL tube | Fisher Scientific | 15386548 | |
| Dispase II | Thermo Fisher Scientific | 17105041 | |
| Dissection Scissors | Fine Science Tools | 14568-09 | |
| DMEM (low glucose, with pyruvate) | Thermo Fisher Scientific | 11885-092 | |
| EDTA (Ethylenediaminetetraacetic acid disodium salt) | Merck | E5134 | Na2EDTA-2H20 |
| EQ Four Element Calibration Beads (EQ beads) | Standard Biotools | 201078 | Calibration beads |
| Fetal Bovine Serum, qualified, Brazil origin | Thermo Fisher Scientific | 10270106 | |
| Forceps Dumont #5SF | Fine Science Tools | 11252-00 | |
| Forceps Dumont #7 | Hounisen.com | 1606.3350 | |
| Goat serum | Thermo Fisher Scientific | 16210-072 | |
| Helios CyTOF system | Standard Biotools | N/A | |
| Horse Serum, heat inactivated, New Zealand origin | Thermo Fisher Scientific | 26-050-088 | |
| IdU | Merck | I7125 | I127 |
| Iridium-Intercalator | Standard Biotools | 201240 | Ir191/193 |
| Isoflurane/Attane Vet | ScanVet | Vnr 055226 | |
| Methanol | Fisher Scientific | M/3900/17 | |
| Myf5 (Clone: C-20) | Santa Cruz Biotechnology | Sc-302 | Yb173; In-house conjugated |
| MyoD (Clone: 5.8A) | BD Biosciences | 554130 | Dy164; In-house conjugated |
| MyoG (Clone: F5D) | BD Biosciences | 556358 | Gd160; In-house conjugated |
| Nalgene Rapid-Flow Sterile Disposable Bottle Top 0.20 μM PES Filters | Thermo Fisher Scientific | 595-4520 | |
| Notexin | Latoxan | L8104 | Resuspend to 50 µg/ml in sterile PBS. Keep stocks (e.g. 50 µl) at -20 °C |
| Nutrient mixture F-10 (Ham's) | Thermo Fisher Scientific | 31550031 | |
| pAkt (Clone: D9E) | Standard Biotools | 3152005A | Sm152 |
| Pax7 (Clone: PAX7) | Santa Cruz Biotechnology | Sc-81648 | Eu153; In-house conjugated |
| Penicillin-Streptomycin (10,000 U/mL) (Pen/Strep) | Thermo Fisher Scientific | 15140122 | |
| PES Filter Units 0.20 μM | Fisher Scientific | 15913307 | |
| PES Syringe Filter | Fisher Scientific | 15206869 | |
| Petri dish | Sarstedt | 82.1472.001 | |
| PFA 16% EM grade | MP Biomedicals | 219998320 | |
| Potassium chloride (KCl) | Fisher Scientific | 10375810 | |
| Potassium phosphate, monobasic, anhydrous (KH2PO4) | Fisher Scientific | 10573181 | |
| pRb (Clone: J112-906) | Standard Biotools | 3166011A | Er166 |
| pS6 kinase (Clone: N7-548) | Standard Biotools | 3172008A | Yb172 |
| Sca-1 (Clone: E13-161.7) | BD Biosciences | 553333 | Nd142; In-house conjugated |
| Sodium Azide | Sigma Aldrich | S2002 | |
| Sodium chloride (NaCl) | Fisher Scientific | 10553515 | |
| Sodium phosphate, dibasic, heptahydrate (Na2HPO4-6H2O) | Merck | S9390 | |
| Sterile saline solution 0.9% | Fresenius | B306414/02 | |
| α7 integrin (Clone: 3C12) | MBL international | K0046-3 | Ho165; In-house conjugated |
Referenzen
- Mukund, K., Subramaniam, S. Skeletal muscle: A review of molecular structure and function in health and disease. Wiley Interdiscip Rev Syst Biol Med. 12 (1), e1462 (2020).
- Feige, P., Brun, C. E., Ritso, M., Rudnicki, M. A. Orienting muscle stem cells for regeneration in homeostasis, aging, and disease. Cell Stem Cell. 23 (5), 653-664 (2018).
- Mauro, A. Satellite cell of skeletal muscle fibers. J Biophys Biochem Cytol. 9 (2), 493-495 (1961).
- Seale, P., et al. Pax7 is required for the specification of myogenic satellite cells. Cell. 102 (6), 777-786 (2000).
- Fuchs, E., Blau, H. M. Tissue stem cells: Architects of their niches. Cell Stem Cell. 27 (4), 532-556 (2020).
- Hernández-hernández, J. M., et al. The myogenic regulatory factors, determinants of muscle development, cell identity and regeneration. Semin Cell Dev Biol. 72, 10-18 (2017).
- Zammit, P. S. Function of the myogenic regulatory factors Myf5, MyoD, Myogenin and MRF4 in skeletal muscle, satellite cells and regenerative myogenesis. Semin Cell Dev Biol. 72, 19-32 (2017).
- Sabourin, L. A. The molecular regulation of myogenesis. Clin Genet. 57 (1), 16-25 (2000).
- Cooper, R. N., et al. In vivo satellite cell activation via Myf5 and MyoD in regenerating mouse skeletal muscle. J Cell Sci. 112 (17), 2895-2901 (1999).
- Rudnicki, M. A., Jaenisch, R. The MyoD family of transcription factors and skeletal myogenesis. Bioessays. 17 (3), 203-209 (1995).
- Braun, T., Arnold, H. H. Inactivation of Myf-6 and Myf-5 genes in mice leads to alterations in skeletal muscle development. EMBO J. 14 (6), 1176-1186 (1995).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Braun, T., et al. MyoD expression marks the onset of skeletal myogenesis in Myf-5 mutant mice. Development. 120 (11), 3083-3092 (1994).
- Rudnicki, M. A., et al. MyoD or Myf-5 is required for the formation of skeletal muscle. Cell. 75 (7), 1351-1359 (1993).
- Montarras, D., et al. Developmental biology: Direct isolation of satellite cells for skeletal muscle regeneration. Science. 309 (5743), 2064-2067 (2005).
- Sacco, A., Doyonnas, R., Kraft, P., Vitorovic, S., Blau, H. M. Self-renewal and expansion of single transplanted muscle stem cells. Nature. 456 (7221), 502-506 (2008).
- Cerletti, M., et al. Highly efficient, functional engraftment of skeletal muscle stem cells in dystrophic muscles. Cell. 134 (1), 37-47 (2008).
- Liu, L., Cheung, T. H., Charville, G. W., Rando, T. A. Isolation of skeletal muscle stem cells by fluorescence-activated cell sorting. Nat Protoc. 10 (10), 1612-1624 (2015).
- Porpiglia, E., et al. High-resolution myogenic lineage mapping by single-cell mass cytometry. Nat Cell Biol. 19 (5), 558-567 (2017).
- Behbehani, G. K., Bendall, S. C., Clutter, M. R., Fantl, W. J., Nolan, G. P. Single-cell mass cytometry adapted to measurements of the cell cycle. Cytometry Part A. 81 (7), 552-566 (2012).
- Hartmann, F. J., et al. . Mass Cytometry: Methods and Protocols. , (2019).
- Devine, R. D., Behbehani, G. K. Use of the pyrimidine analog, 5-iodo-2'-deoxyuridine (IdU) with cell cycle markers to establish cell cycle phases in a mass cytometry platform. J Vis Exp. (176), e60556 (2021).
- Bendall, S. C., et al. Single-cell mass cytometry of differential immune and drug responses across a human hematopoietic continuum. Science. 332 (6030), 687-696 (2011).
- Nag, A. C., Foster, J. D. Myogenesis in adult mammalian skeletal muscle in vitro. J Anat. 132, 1-18 (1981).
- Le Moigne, A., et al. Characterization of myogenesis from adult satellite cells cultured in vitro). Int J Dev Biol. 34, 171-180 (1990).
- Yablonka-Reuveni, Z. Development and postnatal regulation of adult myoblasts. Microsc Res Tech. 30 (5), 366-380 (1995).
- Chu, C., Cogswell, J., Kohtz, D. S. MyoD functions as a transcriptional repressor in proliferating myoblasts. J Biol Chem. 272 (6), 3145-3148 (1997).
- Shah, B., Hyde-Dunn, J., Jones, G. E. Proliferation of murine myoblasts as measured by bromodeoxyuridine incorporation. Methods in Mol Biol. 75, 349-355 (1997).
- Springer, M. L., Blau, H. M. High-efficiency retroviral infection of primary myoblasts. Somat Cell Mol Genet. 23 (3), 203-209 (1997).
- Rando, T. A., Blau, H. M. Primary mouse myoblast purification, characterization, and transplantation for cell-mediated gene therapy. J Cell Biol. 125 (6), 1275-1287 (1994).
- Springer, M. L., Rando, T. A., Blau, H. M. Gene delivery to muscle. Curr Protoc Hum Genet. , (2002).
- Cull-Candy, S. G., Fohlman, J., Gustavsson, D., Lullmann-Rauch, R., Thesleff, S. The effects of taipoxin and notexin on the function and fine structure of the murine neuromuscular junction. Neuroscience. 1 (3), 175-180 (1976).
- Francis, B., John, T. R., Seebart, C., Kaiser, I. I. New toxins from the venom of the common tiger snake (Notechis scutatus scutatus). Toxicon. 29 (1), 85-96 (1991).
- Navarro, K. L., Huss, M., Smith, J. C., Sharp, P., Marx, J. O., Pacharinsak, C. Mouse Anesthesia: The Art and Science. ILAR Journal. 62, 238-273 (2021).
- Langford, D., Bailey, A., Chanda, M., et al. Coding of facial expressions of pain in the laboratory mouse. Nat Methods. 7, 447-449 (2010).
- Matsumiya, L. C., Sorge, R. E., Sotocinal, S. G., Tabaka, J. M., Wieskopf, J. S., Zaloum, A., King, O. D., Mogil, J. S. Using the Mouse Grimace Scale to reevaluate the efficacy of postoperative analgesics in laboratory mice. J Am Assoc Lab Anim Sci. 2012 (1), 42-49 (2012).
- Gonzalez, V. D., et al. High-grade serous ovarian tumor cells modulate NK cell function to create an immune-tolerant microenvironment. Cell Rep. 36 (9), 109632 (2021).
- Delgado-Gonzalez, A., et al. Measuring trogocytosis between ovarian tumor and natural killer cells. STAR Protoc. 3 (2), 101425 (2022).
- Finck, R., et al. Normalization of mass cytometry data with bead standards. Cytometry Part A. 83 (5), 483-494 (2013).
- Leipold, M. D., Maecker, H. T. Mass cytometry: protocol for daily tuning and running cell samples on a CyTOF mass cytometer. J Vis Exp. (69), e4398 (2012).
- McCarthy, R. L., Duncan, A. D., Barton, M. C. Sample preparation for mass cytometry analysis. J Vis Exp. (122), e54394 (2017).
- Kotecha, N., Krutzik, P. O., Irish, J. M. Web-based analysis and publication of flow cytometry experiments. Curr Protoc Cytom. , (2010).
- Fienberg, H. G., Simonds, E. F., Fantl, W. J., Nolan, G. P., Bodenmiller, B. A platinum-based covalent viability reagent for single-cell mass cytometry. Cytometry Part A. 81 (6), 467-475 (2012).
- Kimball, A. K., et al. A beginner's guide to analyzing and visualizing mass cytometry data. J Immunol. 200 (1), 3-22 (2018).
- Weber, L. M., Robinson, M. D. Comparison of clustering methods for high-dimensional single-cell flow and mass cytometry data. Cytometry Part A. 89 (12), 1084-1096 (2016).
- Samusik, N., Good, Z., Spitzer, M. H., Davis, K. L., Nolan, G. P. Automated mapping of phenotype space with single-cell data. Nat Methods. 13 (6), 493-496 (2016).
- Ornatsky, O. I., et al. Study of cell antigens and intracellular DNA by identification of element-containing labels and metallointercalators using inductively coupled plasma mass spectrometry. Anal Chem. 80 (7), 2539-2547 (2008).
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nat Commun. 12 (1), 692 (2021).
- de Morree, A., et al. Staufen1 inhibits MyoD translation to actively maintain muscle stem cell quiescence. Proc Natl Acad Sci U S A. 114 (43), E8996-E9005 (2017).
- Luo, D., et al. Deltex2 represses MyoD expression and inhibits myogenic differentiation by acting as a negative regulator of Jmjd1c. Proc Natl Acad Sci U S A. 114 (15), E3071-E3080 (2017).
- Wersto, R. P., et al. Doublet discrimination in DNA cell-cycle analysis. Cytometry. 46 (5), 296-306 (2001).
- Porpiglia, E., Blau, H. M. Plasticity of muscle stem cells in homeostasis and aging. Curr Opin Genet Dev. 77, 101999 (2022).
- Porpiglia, E., et al. Elevated CD47 is a hallmark of dysfunctional aged muscle stem cells that can be targeted to augment regeneration. Cell Stem Cell. 29 (12), 1653-1668 (2022).
- Brunet, A., Goodell, M. A., Rando, T. A. Ageing and rejuvenation of tissue stem cells and their niches. Nat Rev Mol Cell Biol. 24 (1), 45-62 (2022).
- Danielli, S. G., et al. Single-cell profiling of alveolar rhabdomyosarcoma reveals RAS pathway inhibitors as cell-fate hijackers with therapeutic relevance. Sci Adv. 9 (6), (2023).
- de Morree, A., Rando, T. A. Regulation of adult stem cell quiescence and its functions in the maintenance of tissue integrity. Nat Rev Mol Cell Biol. 24 (5), 334-354 (2023).
- Yucel, N., et al. Glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell glucose metabolism drives histone acetylation landscape transitions that dictate muscle stem cell function. Cell Rep. 27 (13), 3939-3955 (2019).
- Tierney, M. T., Sacco, A. Inducing and evaluating skeletal muscle injury by notexin and barium chloride. Methods Mol Biol. 1460, 53-60 (2016).
- Hardy, D., et al. Comparative study of injury models for studying muscle regeneration in mice. PLoS One. 11 (1), e0147198 (2016).
- Call, J. A., Lowe, D. A. Eccentric contraction-induced muscle injury: Reproducible, quantitative, physiological models to impair skeletal muscle's capacity to generate force. Methods Mol Biol. 1460, 3-18 (2016).
- Garry, G. A., Antony, M. L., Garry, D. J. Cardiotoxin Induced Injury and Skeletal Muscle Regeneration. Methods Mol Biol. 1460, 61-71 (2016).
- Le, G., Lowe, D. A., Kyba, M. Freeze injury of the tibialis anterior muscle. Methods Mol Biol. 1460, 33-41 (2016).
- Borok, M., et al. Progressive and coordinated mobilization of the skeletal muscle niche throughout tissue repair revealed by single-cell proteomic analysis. Cells. 10 (4), 744 (2021).
- Petrilli, L. L., et al. High-dimensional single-cell quantitative profiling of skeletal muscle cell population dynamics during regeneration. Cells. 9 (7), 1723 (2020).
- Giordani, L., et al. High-dimensional single-cell cartography reveals novel skeletal muscle-resident cell populations. Mol Cell. 74 (3), 609-621 (2019).
- Hartmann, F. J., et al. Scalable conjugation and characterization of immunoglobulins with stable mass isotope reporters for single-cell mass cytometry analysis. Methods Mol Biol. 1989, 55-81 (2019).
- Frimand, Z., Das Barman, S., Kjær, T. R., Porpiglia, E., de Morrée, A. Isolation of quiescent stem cell populations from individual skeletal muscles. J Vis Exp. (190), e64557 (2022).
- Krutzik, P. O., Nolan, G. P. Intracellular phospho-protein staining techniques for flow cytometry: monitoring single cell signaling events. Cytometry A. 55 (2), 61-70 (2003).
- Bodenmiller, B., et al. Multiplexed mass cytometry profiling of cellular states perturbed by small-molecule regulators. Nat Biotechnol. 30 (9), 858-867 (2012).
- Schulz, K. R., Danna, E. A., Krutzik, P. O., Nolan, G. P. Single-cell phospho-protein analysis by flow cytometry. Curr Protoc Immunol. , 11-20 (2012).
- Krutzik, P. O., Clutter, M. R., Nolan, G. P. Coordinate analysis of murine immune cell surface markers and intracellular phosphoproteins by flow cytometry. J Immunol. 175 (4), 2357-2365 (2005).
- Krutzik, P. O., Irish, J. M., Nolan, G. P., Perez, O. D. Analysis of protein phosphorylation and cellular signaling events by flow cytometry: techniques and clinical applications. Clin Immunol. 110 (3), 206-221 (2004).
- Han, G., Spitzer, M. H., Bendall, S. C., Fantl, W. J., Nolan, G. P. Metal-isotope-tagged monoclonal antibodies for high-dimensional mass cytometry. Nat Protoc. 13 (10), 2121-2148 (2018).
- Chevrier, S., et al. Compensation of signal spillover in suspension and imaging mass cytometry. Cell Syst. 6 (5), 612-620 (2018).
- Bjornson, Z. B., Nolan, G. P., Fantl, W. J. Single-cell mass cytometry for analysis of immune system functional states. Curr Opin Immunol. 25 (4), 484-494 (2013).
- Kalina, T., Lundsten, K., Engel, P. Relevance of antibody validation for flow cytometry. Cytometry A. 97 (2), 126-136 (2020).
- Baumgarth, N., Roederer, M. A practical approach to multicolor flow cytometry for immunophenotyping. J Immunol Methods. 243 (1-2), 77-97 (2000).
- Roederer, M. Spectral compensation for flow cytometry: visualization artifacts, limitations, and caveats. Cytometry. 45 (3), 194-205 (2001).
- Tung, J. W., Parks, D. R., Moore, W. A., Herzenberg, L. A., Herzenberg, L. A. New approaches to fluorescence compensation and visualization of FACS data. Clin Immunol. 110 (3), 277-283 (2004).
- Cossarizza, A., et al. Guidelines for the use of flow cytometry and cell sorting in immunological studies (third edition). Eur J Immunol. 51 (12), 2708-3145 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten