Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Determinación de la estabilidad sérica de la enzima adenosina desaminasa 1 humana
En este artículo
Resumen
En este artículo, detallamos métodos para caracterizar la capacidad de una enzima para retener la función cuando se incuba a 37 °C en suero humano, una propiedad farmacológica conocida como su estabilidad sérica. Esta capacidad puede ser un factor clave en la predicción del perfil farmacocinético de una enzima y su idoneidad para uso terapéutico.
Resumen
El concepto de estabilidad enzimática se utiliza normalmente para referirse a la termoestabilidad de una enzima, es decir, su capacidad para retener la estructura y la actividad a medida que aumenta la temperatura. En el caso de una enzima terapéutica, otras medidas de estabilidad también pueden ser críticas, en particular su capacidad para mantener la función en el suero humano a 37 °C, lo que denominamos estabilidad sérica. Aquí, describimos un ensayo in vitro para evaluar la estabilidad sérica de la enzima Homo sapiens adenosina desaminasa I (HsADA1) de tipo salvaje utilizando un procedimiento de microplaca basado en absorbancia. En concreto, este manuscrito describe la preparación de tampones y reactivos, un método que prevé la coincubación de HsADA1 en suero, un método para analizar las muestras de prueba utilizando un lector de microplacas y un análisis complementario para determinar la fracción de actividad que una enzima HsADA1 retiene en el suero en función del tiempo. Además, discutimos las consideraciones para adaptar este protocolo a otras enzimas, utilizando un ejemplo de una enzima Homo sapiens kynureninasa, para ayudar a la adaptación del protocolo a otras enzimas donde la estabilidad sérica es de interés.
Introducción
El siguiente método permite al usuario evaluar cuantitativamente la capacidad de una enzima para retener su actividad cuando se expone a condiciones que imitan lo que encontrará después de una inyección intravenosa. El método in vitro imita estas condiciones in vivo y consiste en la incubación de la enzima en suero humano combinado a 37 °C y análisis de la retención de la actividad enzimática en el tiempo. Nos referimos a la capacidad de una enzima para retener la actividad en estas condiciones como su estabilidad sérica, y el método de análisis de la actividad enzimática aprovecha las diferencias en la absorbancia entre el sustrato de una enzima y el producto resultante. El concepto de estabilidad sérica no es solo específico de la enzima, sino que también se ha aplicado a otras modalidades de tratamiento. Por ejemplo, la estabilidad sérica de los aptámeros de ARN dirigidos a las proteínas de la espícula de la COVID se ha evaluado previamente mediante el seguimiento de su degradación tras la incubación con suero fetal de ternero1. Los péptidos antibacterianos también se han evaluado por su capacidad para suprimir el crecimiento bacteriano después de la incubación con suero humano combinado2.
HsADA1 es una enzima que cataliza la conversión de adenosina o 2-desoxiadenosina en inosina o 2-desoxiinosina, respectivamente3. La adenosina tiene un pico de absorción de 260 nm, mientras que la inosina se absorbe más fuerte a 250 nm4. Este cambio en los picos de absorción se puede detectar en un lector de microplacas mediante una disminución en la intensidad de absorción a 260 nm cuando se agrega HsADA1 a la adenosina. La enzima HsADA1 tiene importantes implicaciones en el cuerpo humano, y su deficiencia causa una inmunodeficiencia combinada severa (ADA-SCID)5. El homólogo bovino de HsADA1, BtADA1, puede utilizarse como un reemplazo enzimático terapéutico para el tratamiento de ADA-SCID, y hemos demostrado previamente que HsADA1 de tipo salvaje pierde su actividad cuando se incuba con suero humano combinado, lo que podría dificultar su uso como terapéutico6. Por lo tanto, seleccionamos la enzima HsADA1 de tipo salvaje para demostrar un procedimiento para determinar la estabilidad sérica de una enzima. Un método de purificación detallado para HsADA1 ha sido descrito previamente6.
En el siguiente protocolo (como se detalla en la Figura 1), demostramos cómo co-incubar HsADA1 de tipo salvaje en suero humano combinado a 37 °C. Durante este tiempo, las muestras de prueba se toman en puntos de tiempo definidos y se congelan rápidamente para análisis futuros. Una vez que se han tomado todas las muestras, se realiza un ensayo de microplaca en el que cada muestra se combina con el sustrato, siendo la disminución de la absorbancia resultante una correlación de la actividad retenida de la enzima. Se muestran resultados representativos que ilustran la estabilidad sérica de HsADA1, y debido a que esta métrica puede ser relevante para determinar el valor terapéutico potencial de otras enzimas, también discutimos las consideraciones para adaptar este protocolo a una enzima Homo sapiens kynureninase modificada (HsKYNasa) y a cualquier enzima en general. La HsKYNasa es una enzima implicada en el metabolismo del triptófano y es capaz de degradar los subproductos del triptófano quinurenina e hidroxiquinurenina (OH-Kyn) en ácido antranílico y ácido hidroxi-antranílico (OH-AA), respectivamente. La modulación enzimática del metabolismo del triptófano puede ser de relevancia terapéutica7.
Protocolo
1. Incubación del suero
- Prepare un stock de 10x HsADA1 en 1x PBS pH 7.4 (1x PBS) a una concentración final de 10 μM. Descongele una alícuota de 15 mL de suero humano agrupado y precaliéntelo a 37 °C. Precalentar una alícuota de 50 mL de 1x PBS a 37 °C.
- Prepare las mezclas de incubación enzima-suero añadiendo 100 μL de la cepa 10x HsADA1 a 900 μL de suero humano agrupado en un tubo de microcentrífuga de baja unión, denominado mezcla de enzima + suero. En un tubo de microcentrífuga de baja unión separado, agregue 100 μL de la culata 10x HsADA1 a 900 μL de 1x PBS para compararlo con un control sin suero, denominado mezcla de enzima + 1x PBS. La concentración final de HsADA1 en cada mezcla será de 1 μM.
- Prepare un control adicional en un tubo de microcentrífuga de baja unión que consta de 100 μL de 1x PBS mezclado con 900 μL de suero humano combinado. Esto servirá como control no enzimático para la mezcla de enzima + suero. Además, realice un control que conste de 1 mL de 1x PBS. Esto servirá como control no enzimático para la mezcla de enzima + 1x PBS.
- Aliquotar 100 μL de cada mezcla del paso 1.2 en tubos separados de baja adherencia y diluir 1:1 con 100 μL de glicerol al 30% (v/v) en 1x PBS. Congele rápidamente con nitrógeno líquido y almacene a -80 °C. Estas muestras serán el punto de tiempo del Día 0, y la concentración de HsADA1 en ellas será de 500 nM. Repita este paso para las mezclas de control no enzimáticas de 1.3. Selle todas las enzimas y mezclas de control con una película transparente y colóquelas en una incubadora a 37 °C.
- Después de 24 h, alícuota 100 μL de cada muestra incubada y diluir 1:1 con 100 μL de glicerol al 30% (v/v) en 1x PBS. Congele rápidamente con nitrógeno líquido y almacene a -80 °C. Estas muestras serán el punto de tiempo del Día 1. Regrese todas las mezclas de enzimas y controles a la incubadora a 37 °C. Repita este paso a las 72 h y a las 120 h.
NOTA: No es necesario que las muestras alícuotas se congelen rápidamente. Alternativamente, el usuario puede realizar el ensayo basado en microplacas inmediatamente después de tomar cada muestra, aunque se deben preparar curvas estándar frescas y existencias de sustrato de adenosina cada vez que se analiza una muestra.
2. Ensayo basado en microplacas
- Prepare el lector de microplacas configurando un protocolo de lectura cinética (Figura 2) para medir la absorbancia a 260 nm con el intervalo de lectura más corto durante 30 minutos y precalentar el lector a un punto de ajuste de 37 °C.
- Descongele todas las muestras recolectadas en hielo. Diluir cada muestra descongelada 1:10 utilizando 1x PBS precalentado a 37 °C como diluyente en tubos de microcentrífuga de baja fijación. La nueva concentración de HsADA1 en estas muestras será de 50 nM.
- Realice también una dilución 10x en 1x PBS a partir de la muestra de control de suero no enzimático. Además, prepare un caldo de adenosina de 5 mM añadiendo 66,8 mg de adenosina a 50 mL de PBS 1x. Prepare una dilución de 250 μM de adenosina añadiendo 400 μL de caldo de adenosina a 7.600 μL de 1x PBS y precaliéntelo a 37 °C en una incubadora.
- A una microplaca de 96 pocillos compatible con rayos UV, agregue 160 μL de la dilución de adenosina de 250 μM y 40 μL de cada muestra de proteína diluida por triplicado para un volumen total de 200 μL. Esto producirá una concentración final de adenosina de 200 μM y una concentración final de enzima de 10 nM.
- Repita la dilución para las muestras de control añadiendo 40 μL del suero diluido o 1x controles de PBS y 160 μL de la dilución de adenosina de 250 μM por triplicado. Esto también producirá una concentración final de adenosina de 200 μM pero sin enzima.
NOTA: Puede ser beneficioso agregar primero todas las muestras de enzima y control diluidas individualmente a la microplaca y luego agregar el sustrato de adenosina con una micropipeta de 12 canales para reducir el tiempo de carga total. Además, en este paso se deben utilizar placas transparentes a los rayos UV para evitar que la propia placa confunda la señal de absorbancia medida. - Mida la absorbancia de cada pocillo a 260 nm durante 30 min a 37 °C. Una vez hecho esto, exporte los datos a una hoja de cálculo para su posterior análisis.
3. Análisis de los datos del lector de microplacas
- Determine la pendiente de la región lineal de los datos de absorbancia en función del tiempo durante los primeros minutos de los datos de la microplaca. Para las muestras diluidas en suero, esto correspondió a los primeros ~120 s. Para las muestras diluidas con PBS 1x, esto correspondió a los primeros ~270 s. Esta pendiente de valores de absorbancia decrecientes tendrá un valor numérico negativo y tendrá unidades de cambio en unidades de absorbancia por segundo (ΔA260/s).
- Reste la pendiente del control negativo que consiste en suero humano agrupado de la pendiente de los datos de enzima + suero. Realice la misma operación en las mezclas de enzima + 1x PBS restando la pendiente del control 1x PBS.
- Normalice todas las pendientes ajustadas a la pendiente original a t=0 h para obtener la fracción de actividad restante utilizando la Ecuación 1.
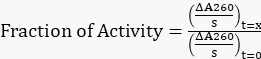 Ecuación 1
Ecuación 1 - Representa la fracción de actividad restante en función del tiempo.
Resultados
Las figuras muestran los resultados de la ejecución del ensayo cuando se realizó con HsADA1 de tipo salvaje. La figura 3A, B ilustra las curvas de disminución de la absorbancia a 260 nm de las muestras que se originan a partir de las mezclas 1x de PBS/suero-enzima para HsADA1 de tipo salvaje después de la adición de adenosina. Esta disminución de la absorbancia en función de los datos de tiempo es lo que el usuario puede esperar al co...
Discusión
Este protocolo utiliza el cambio de absorbancia a medida que el sustrato se convierte en el producto para medir la actividad de una enzima. Como tal, el sustrato y el producto deben tener perfiles espectrales distintos. Este es el caso de la adenosina y la inosina, ambas con perfiles espectrales distintos y coeficientes de extinción entre 260-265 nm 6,8,12,13....
Divulgaciones
JB y MRJ son inventores de patentes o solicitudes de patentes relacionadas con las enzimas adenosina desaminasa y/o quinureninasa. Todos los demás autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo contó con el apoyo de los Institutos Nacionales de Salud [1DP2CA280622-01] y la financiación de Biolocity. Agradecemos a la Dra. Maria Jennings y Andrea Fox por proporcionar los vectores de expresión HsADA1 y HsKYNase.
Materiales
| Name | Company | Catalog Number | Comments |
| Adenosine | Sigma Aldrich | A9251-25G | 25 g |
| BioTek Synergy HT Microplate Reader | |||
| Eppendorf LoBind Microcentrifuge Tubes: Protein | Fisher Scientific | 13-698-795 | 2 mL |
| Glycerol | Fisher Scientific | G33-4 | 4 L |
| HsKYNase66-W102H-T333N | In-house | ||
| Human Serum, Pooled | MP Biomedicals | 92931149 | 100 mL |
| Hydroxy-kynurenine | Cayman Chemicals | 27778 | |
| Inosine | TCI | I0037 | 25 g |
| PBS, 1x pH 7.4+/- 0.1 | Corning | 21-040-CM | |
| Pyridoxal 5-phosphate monohydrate, 99% | Thermo Scientifc | 228170010 | 1 g |
| UV-STAR MICROPLATE, 96 WELL, COC, F-BOTTOM | Greiner Bio | 655801 | |
| Wildtype Human Adenosine Deaminase 1 | In-house |
Referencias
- Valero, J., et al. A serum-stable RNA aptamer specific for SARS-COV-2 neutralizes viral entry. Proc Natl Acad Sci U S A. 118 (50), e2112942118 (2021).
- Iannuzo, N., et al. High-throughput screening identifies synthetic peptides with antibacterial activity against mycobacterium abscessus and serum stability. ACS Omega. 7 (27), 23967-23977 (2022).
- Ma, M. T., Jennings, M. R., Blazeck, J., Lieberman, R. L. Catalytically active holo homo sapiens adenosine deaminase i adopts a closed conformation. Acta Crystallograph Sect D. 78 (1), 91-103 (2022).
- Li, W., et al. Determination of 4 nucleosides via one reference compound in chinese cordyceps by hplc-uv at equal absorption wavelength. Natural Prod Comm. 18 (3), 1934578X231161410 (2023).
- Whitmore, K. V., Gaspar, H. B. Adenosine deaminase deficiency - more than just an immunodeficiency. Front Immunol. 7, 314 (2016).
- Jennings, M. R., et al. Optimized expression and purification of a human adenosine deaminase in E. coli and characterization of its asp8asn variant. Prot Express Purificat. 213, 106362 (2024).
- Blazeck, J., et al. Bypassing evolutionary dead ends and switching the rate-limiting step of a human immunotherapeutic enzyme. Nat Catalysis. 5 (10), 952-967 (2022).
- Lu, J., Grenache, D. G. Development of a rapid, microplate-based kinetic assay for measuring adenosine deaminase activity in body fluids. Clinica Chimica Acta. 413 (19), 1637-1640 (2012).
- Maciel, L. G., Dos Anjos, J. V., Soares, T. A. Fast and low-cost evaluation of hydroxykynurenine activity. MethodsX. 7, 100982 (2020).
- Bokman, A. H., Schweigert, B. S. 3-hydroxyanthranilic acid metabolism. Iv. Spectrophotometric evidence for the formation of an intermediate. Arch Biochem Biophys. 33 (2), 270-276 (1951).
- Tanford, C. . Advances in protein chemistry. 23, 121-282 (1968).
- Tritsch, G. L. Validity of the continuous spectrophotometric assay of kalckar for adenosine deaminase activity. Anal Biochem. 129 (1), 207-209 (1983).
- Gracia, E., et al. The catalytic site structural gate of adenosine deaminase allosterically modulates ligand binding to adenosine receptors. FASEB J. 27 (3), 1048-1061 (2013).
- Kalckar, H. M. Differential spectrophotometry of purine compounds by means of specific enzymes: Iii. Studies of the enzymes of purine metabolism. J Bio Chem. 167 (2), 461-475 (1947).
- Hartwick, R., Jeffries, A., Krstulovic, A., Brown, P. R. An optimized assay for adenosine deaminase using reverse phase high pressure liquid chromatography. J Chromatographic Sci. 16 (9), 427-435 (1978).
- Paul, M. K., Grover, V., Mukhopadhyay, A. K. Merits of hplc-based method over spectrophotometric method for assessing the kinetics and inhibition of mammalian adenosine deaminase. J Chromatography B. 822 (1), 146-153 (2005).
- Ubbink, J. B., Vermaak, W. J. H., Bissbort, S. H. High-performance liquid chromatographic assay of human lymphocyte kynureninase activity levels. J Chromatography B: Biomed Sci Appl. 566 (2), 369-375 (1991).
- Tsentalovich, Y. P., Snytnikova, O. A., Forbes, M. D. E., Chernyak, E. I., Morozov, S. V. Photochemical and thermal reactivity of kynurenine. Exp Eye Res. 83 (6), 1439-1445 (2006).
- Demain, A. L., Vaishnav, P. Production of recombinant proteins by microbes and higher organisms. Biotechnol Adv. 27 (3), 297-306 (2009).
- Gräslund, S., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Faber, M. S., Whitehead, T. A. Data-driven engineering of protein therapeutics. Curr Opin Biotechnol. 60, 104-110 (2019).
- Das, S., Zhao, L., Elofson, K., Finn, M. G. Enzyme stabilization by virus-like particles. Biochemistry. 59 (31), 2870-2881 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados