Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détermination de la stabilité sérique de l’enzyme adénosine désaminase 1 humaine
Dans cet article
Résumé
Dans cet article, nous détaillons des méthodes pour caractériser la capacité d’une enzyme à conserver sa fonction lorsqu’elle est incubée à 37 °C dans le sérum humain, une propriété pharmacologique appelée stabilité sérique. Cette capacité peut être un facteur clé dans la prédiction du profil pharmacocinétique d’une enzyme et de son aptitude à un usage thérapeutique.
Résumé
Le concept de stabilité enzymatique est généralement utilisé pour faire référence à la thermostabilité d’une enzyme, c’est-à-dire à sa capacité à conserver sa structure et son activité lorsque la température augmente. Pour une enzyme thérapeutique, d’autres mesures de stabilité peuvent également être critiques, en particulier sa capacité à conserver sa fonction dans le sérum humain à 37 °C, ce que nous appelons la stabilité sérique. Ici, nous décrivons un test in vitro pour évaluer la stabilité sérique de l’enzyme sauvage Homo sapiens adénosine désaminase I (HsADA1) à l’aide d’une procédure de microplaque basée sur l’absorbance. Plus précisément, ce manuscrit décrit la préparation de tampons et de réactifs, une méthode d’organisation de la coïncubation de HsADA1 dans le sérum, une méthode d’analyse des échantillons de test à l’aide d’un lecteur de microplaques et une analyse d’accompagnement pour déterminer la fraction d’activité qu’une enzyme HsADA1 conserve dans le sérum en fonction du temps. Nous discutons plus en détail des considérations visant à adapter ce protocole à d’autres enzymes, en utilisant l’exemple d’une enzyme kynurénase Homo sapiens , afin d’aider à l’adaptation du protocole à d’autres enzymes où la stabilité sérique est intéressante.
Introduction
La méthode suivante permet à un utilisateur d’évaluer quantitativement la capacité d’une enzyme à conserver son activité lorsqu’elle est exposée à des conditions qui imitent ce qu’elle rencontrera après une injection intraveineuse. La méthode in vitro imite de telles conditions in vivo et consiste en l’incubation de l’enzyme dans du sérum humain groupé à 37 °C et des analyses chronologiques de la rétention de l’activité enzymatique. Nous nous référons à la capacité d’une enzyme à conserver son activité dans ces conditions comme sa stabilité sérique, et la méthode d’analyse de l’activité enzymatique tire parti des différences d’absorbance entre le substrat d’une enzyme et le produit résultant. Le concept de stabilité sérique n’est pas seulement spécifique à une enzyme et a également été appliqué à plusieurs autres modalités de traitement. Par exemple, la stabilité sérique des aptamères d’ARN ciblant les protéines de pointe COVID a déjà été évaluée en surveillant leur dégradation après l’incubation avec le sérum de veau fœtal1. Des peptides antibactériens ont également été évalués pour leur capacité à supprimer la croissance bactérienne après l’incubation avec du sérum humain2 mélangé.
HsADA1 est une enzyme qui catalyse la conversion de l’adénosine ou de la 2-désoxyadénosine en inosine ou en 2-désoxyinosine, respectivement3. L’adénosine a un pic d’absorption de 260 nm, tandis que l’inosine absorbe le plus fort à 250 nm4. Ce décalage des pics d’absorption peut être détecté sur un lecteur de microplaques par une diminution de l’intensité d’absorption à 260 nm lorsque HsADA1 est ajouté à l’adénosine. L’enzyme HsADA1 a des implications importantes dans le corps humain, et sa carence provoque une immunodéficience combinée sévère (ADA-SCID)5. L’homologue bovin de HsADA1, BtADA1, peut être utilisé comme thérapie de remplacement enzymatique pour le traitement de l’ADA-SCID, et nous avons précédemment montré que le HsADA1 de type sauvage perd son activité lorsqu’il est incubé avec du sérum humain mélangé, ce qui pourrait entraver son utilisation en tant que thérapeutique6. Par conséquent, nous avons sélectionné l’enzyme HsADA1 de type sauvage pour démontrer une procédure permettant de déterminer la stabilité sérique d’une enzyme. Une méthode de purification détaillée de HsADA1 a été décrite précédemment6.
Dans le protocole suivant (tel que détaillé dans la figure 1), nous démontrons comment co-incuber HsADA1 de type sauvage dans du sérum humain mélangé à 37 °C. Pendant ce temps, des échantillons d’essai sont prélevés à des moments définis et congelés pour une analyse future. Une fois que tous les échantillons ont été prélevés, un test sur microplaque est effectué dans lequel chaque échantillon est combiné avec le substrat, la diminution de l’absorbance résultante étant une corrélation avec l’activité conservée de l’enzyme. Des résultats représentatifs illustrant la stabilité sérique de HsADA1 sont présentés, et parce que cette métrique peut être pertinente pour déterminer la valeur thérapeutique potentielle d’autres enzymes, nous discutons également des considérations pour adapter ce protocole à une enzyme kynurénase Homo sapiens modifiée (HsKYNase) et à toute enzyme plus généralement. La HsKYNase est une enzyme impliquée dans le métabolisme du tryptophane et est capable de dégrader les sous-produits du tryptophane, la kynurénine et l’hydroxy-kynurénine (OH-Kyn), respectivement en acide anthranilique et en acide hydroxy-anthranilique (OH-AA). La modulation du métabolisme du tryptophane par l’intermédiaire enzymatique peut être d’une pertinence thérapeutique7.
Protocole
1. Incubation sérique
- Préparez une quantité 10x de HsADA1 dans 1x PBS pH 7,4 (1x PBS) à une concentration finale de 10 μM. Décongelez une aliquote de 15 mL de sérum humain mélangé et préchauffez-la à 37 °C. Préchauffer une aliquote de 50 mL de 1x PBS à 37 °C.
- Préparez les mélanges d’incubation enzyme-sérum en ajoutant 100 μL de la matrice 10x HsADA1 à 900 μL de sérum humain mélangé dans un tube de microcentrifugation à faible liaison, appelé mélange enzyme + sérum. Dans un tube de microcentrifugation séparé à faible liaison, ajoutez 100 μL de la matrice 10x HsADA1 à 900 μL de 1x PBS pour comparaison avec un témoin non sérum, appelé mélange enzyme + 1x PBS. La concentration finale de HsADA1 dans chaque mélange sera de 1 μM.
- Préparez un témoin supplémentaire dans un tube de microcentrifugation à faible liaison composé de 100 μL de 1x PBS mélangé à 900 μL de sérum humain mélangé. Cela servira de contrôle non enzymatique pour le mélange enzyme + sérum. De plus, faites un contrôle composé de 1 mL de 1x PBS. Cela servira de contrôle non enzymatique pour le mélange enzyme + 1x PBS.
- Aliquote 100 μL de chaque mélange à partir de l’étape 1.2 dans des tubes séparés à faible liaison et diluer 1:1 avec 100 μL de glycérol à 30 % (v/v) dans 1x PBS. Congeler rapidement avec de l’azote liquide et conserver à -80 °C. Ces échantillons seront le point temporel du jour 0, et la concentration de HsADA1 dans ceux-ci sera de 500 nM. Répétez cette étape pour les mélanges de contrôle non enzymatiques à partir de 1.3. Scellez toutes les enzymes et les mélanges de contrôle avec un film transparent et placez-les dans un incubateur à 37 °C.
- Après 24 h, aliquote 100 μL de chaque échantillon incubé et diluer 1:1 avec 100 μL de glycérol à 30 % (v/v) dans 1x PBS. Congeler rapidement avec de l’azote liquide et conserver à -80 °C. Ces échantillons seront le point temporel du jour 1. Remettre tous les mélanges d’enzymes et de témoins dans l’incubateur à 37 °C. Répétez cette étape à 72 h et 120 h.
REMARQUE : Les échantillons aliquotes n’ont pas nécessairement besoin d’être surgelés. L’utilisateur peut également effectuer le test sur microplaques immédiatement après le prélèvement de chaque échantillon, bien que de nouvelles courbes standard et des stocks de substrat d’adénosine doivent être préparés chaque fois qu’un échantillon est testé.
2. Dosage sur microplaque
- Préparez le lecteur de microplaques en mettant en place un protocole de lecture cinétique (Figure 2) pour mesurer l’absorbance à 260 nm avec l’intervalle de lecture le plus court pendant 30 min et préchauffer le lecteur à un point de consigne de 37 °C.
- Décongeler tous les échantillons prélevés sur de la glace. Diluer chaque échantillon décongelé à l’aide de 1x PBS préchauffé à 37 °C comme diluant dans des tubes de microcentrifugation à faible liaison. La nouvelle concentration de HsADA1 dans ces échantillons sera de 50 nM.
- Faites une dilution 10x dans 1x PBS à partir de l’échantillon de contrôle sérique non enzymatique également. De plus, préparez un stock d’adénosine de 5 mM en ajoutant 66,8 mg d’adénosine à 50 mL de 1x PBS. Préparez une dilution de 250 μM d’adénosine en ajoutant 400 μL de la pâte d’adénosine à 7 600 μL de 1x PBS et préchauffez-la à 37 °C dans un incubateur.
- À une microplaque compatible avec les UV à 96 puits, ajoutez 160 μL de dilution d’adénosine de 250 μM et 40 μL de chaque échantillon de protéine diluée en trois exemplaires pour un volume total de 200 μL. Cela donnera une concentration finale d’adénosine de 200 μM et une concentration finale d’enzyme de 10 nM.
- Répéter la dilution pour les échantillons témoins en ajoutant 40 μL de sérum dilué ou 1 témoin PBS et 160 μL de la dilution de 250 μM d’adénosine en trois exemplaires. Cela donnera également une concentration finale d’adénosine de 200 μM mais sans enzyme.
REMARQUE : Il peut être avantageux d’ajouter d’abord tous les échantillons d’enzymes et de contrôle dilués individuellement à la microplaque, puis d’ajouter le substrat d’adénosine avec une micropipette à 12 canaux pour réduire le temps de chargement total. De plus, des plaques transparentes aux UV doivent être utilisées dans cette étape pour éviter que la plaque elle-même ne confonde le signal d’absorbance mesuré. - Mesurer l’absorbance de chaque puits à 260 nm pendant 30 min à 37 °C. Une fois cela fait, exportez les données vers une feuille de calcul pour une analyse plus approfondie.
3. Analyse des données des lecteurs de microplaques
- Déterminez la pente de la région linéaire des données d’absorbance en fonction du temps pendant les premières minutes des données de microplaques. Pour les échantillons dilués dans le sérum, cela correspondait aux premiers ~120 s. Pour les échantillons 1x dilués par PBS, cela correspondait aux premiers ~270 s. Cette pente de valeurs d’absorbance décroissantes aura une valeur numérique négative et aura des unités de variation en unités d’absorbance par seconde (ΔA260/s).
- Soustrayez la pente du témoin négatif constitué de sérum humain regroupé de la pente des données enzyme + sérum. Effectuez la même opération sur les mélanges enzyme + 1x PBS en soustrayant la pente du témoin 1x PBS.
- Normalisez toutes les pentes ajustées à la pente d’origine à t = 0 h pour obtenir la fraction d’activité restante à l’aide de l’équation 1.
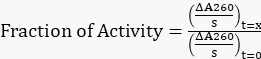 Équation 1
Équation 1 - Tracez la fraction d’activité restante en fonction du temps.
Résultats
Les figures montrent les résultats de l’analyse lorsqu’elle a été menée avec HsADA1 de type sauvage. Les figures 3A et B illustrent les courbes de baisse de l’absorbance à 260 nm des échantillons provenant des mélanges 1x PBS/sérum et enzymes pour HsADA1 de type sauvage après ajout d’adénosine. Cette absorbance décroissante en fonction des données temporelles est ce à quoi l’utilisateur peut s’attendre une fois le te...
Discussion
Ce protocole utilise le changement d’absorbance lorsque le substrat est converti en produit pour évaluer l’activité d’une enzyme. En tant que tels, le substrat et le produit doivent avoir des profils spectraux distincts. C’est le cas de l’adénosine et de l’inosine ayant toutes deux des profils spectraux distincts et des coefficients d’extinction compris entre 260 et 265 nm 6,8,12,13....
Déclarations de divulgation
JB et MRJ sont des inventeurs de brevets ou de demandes de brevets liés aux enzymes adénosine désaminase et/ou kynurénase. Tous les autres auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Ce travail a été soutenu par les National Institutes of Health [1DP2CA280622-01] et financé par Biolocity. Nous remercions la Dre Maria Jennings et Andrea Fox d’avoir fourni les vecteurs d’expression HsADA1 et HsKYNase.
matériels
| Name | Company | Catalog Number | Comments |
| Adenosine | Sigma Aldrich | A9251-25G | 25 g |
| BioTek Synergy HT Microplate Reader | |||
| Eppendorf LoBind Microcentrifuge Tubes: Protein | Fisher Scientific | 13-698-795 | 2 mL |
| Glycerol | Fisher Scientific | G33-4 | 4 L |
| HsKYNase66-W102H-T333N | In-house | ||
| Human Serum, Pooled | MP Biomedicals | 92931149 | 100 mL |
| Hydroxy-kynurenine | Cayman Chemicals | 27778 | |
| Inosine | TCI | I0037 | 25 g |
| PBS, 1x pH 7.4+/- 0.1 | Corning | 21-040-CM | |
| Pyridoxal 5-phosphate monohydrate, 99% | Thermo Scientifc | 228170010 | 1 g |
| UV-STAR MICROPLATE, 96 WELL, COC, F-BOTTOM | Greiner Bio | 655801 | |
| Wildtype Human Adenosine Deaminase 1 | In-house |
Références
- Valero, J., et al. A serum-stable RNA aptamer specific for SARS-COV-2 neutralizes viral entry. Proc Natl Acad Sci U S A. 118 (50), e2112942118 (2021).
- Iannuzo, N., et al. High-throughput screening identifies synthetic peptides with antibacterial activity against mycobacterium abscessus and serum stability. ACS Omega. 7 (27), 23967-23977 (2022).
- Ma, M. T., Jennings, M. R., Blazeck, J., Lieberman, R. L. Catalytically active holo homo sapiens adenosine deaminase i adopts a closed conformation. Acta Crystallograph Sect D. 78 (1), 91-103 (2022).
- Li, W., et al. Determination of 4 nucleosides via one reference compound in chinese cordyceps by hplc-uv at equal absorption wavelength. Natural Prod Comm. 18 (3), 1934578X231161410 (2023).
- Whitmore, K. V., Gaspar, H. B. Adenosine deaminase deficiency - more than just an immunodeficiency. Front Immunol. 7, 314 (2016).
- Jennings, M. R., et al. Optimized expression and purification of a human adenosine deaminase in E. coli and characterization of its asp8asn variant. Prot Express Purificat. 213, 106362 (2024).
- Blazeck, J., et al. Bypassing evolutionary dead ends and switching the rate-limiting step of a human immunotherapeutic enzyme. Nat Catalysis. 5 (10), 952-967 (2022).
- Lu, J., Grenache, D. G. Development of a rapid, microplate-based kinetic assay for measuring adenosine deaminase activity in body fluids. Clinica Chimica Acta. 413 (19), 1637-1640 (2012).
- Maciel, L. G., Dos Anjos, J. V., Soares, T. A. Fast and low-cost evaluation of hydroxykynurenine activity. MethodsX. 7, 100982 (2020).
- Bokman, A. H., Schweigert, B. S. 3-hydroxyanthranilic acid metabolism. Iv. Spectrophotometric evidence for the formation of an intermediate. Arch Biochem Biophys. 33 (2), 270-276 (1951).
- Tanford, C. . Advances in protein chemistry. 23, 121-282 (1968).
- Tritsch, G. L. Validity of the continuous spectrophotometric assay of kalckar for adenosine deaminase activity. Anal Biochem. 129 (1), 207-209 (1983).
- Gracia, E., et al. The catalytic site structural gate of adenosine deaminase allosterically modulates ligand binding to adenosine receptors. FASEB J. 27 (3), 1048-1061 (2013).
- Kalckar, H. M. Differential spectrophotometry of purine compounds by means of specific enzymes: Iii. Studies of the enzymes of purine metabolism. J Bio Chem. 167 (2), 461-475 (1947).
- Hartwick, R., Jeffries, A., Krstulovic, A., Brown, P. R. An optimized assay for adenosine deaminase using reverse phase high pressure liquid chromatography. J Chromatographic Sci. 16 (9), 427-435 (1978).
- Paul, M. K., Grover, V., Mukhopadhyay, A. K. Merits of hplc-based method over spectrophotometric method for assessing the kinetics and inhibition of mammalian adenosine deaminase. J Chromatography B. 822 (1), 146-153 (2005).
- Ubbink, J. B., Vermaak, W. J. H., Bissbort, S. H. High-performance liquid chromatographic assay of human lymphocyte kynureninase activity levels. J Chromatography B: Biomed Sci Appl. 566 (2), 369-375 (1991).
- Tsentalovich, Y. P., Snytnikova, O. A., Forbes, M. D. E., Chernyak, E. I., Morozov, S. V. Photochemical and thermal reactivity of kynurenine. Exp Eye Res. 83 (6), 1439-1445 (2006).
- Demain, A. L., Vaishnav, P. Production of recombinant proteins by microbes and higher organisms. Biotechnol Adv. 27 (3), 297-306 (2009).
- Gräslund, S., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Faber, M. S., Whitehead, T. A. Data-driven engineering of protein therapeutics. Curr Opin Biotechnol. 60, 104-110 (2019).
- Das, S., Zhao, L., Elofson, K., Finn, M. G. Enzyme stabilization by virus-like particles. Biochemistry. 59 (31), 2870-2881 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon