È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Determinazione della stabilità sierica dell'enzima adenosina deaminasi 1 umana
In questo articolo
Riepilogo
In questo articolo, descriviamo in dettaglio i metodi per caratterizzare la capacità di un enzima di mantenere la funzione quando incubato a 37 °C nel siero umano, una proprietà farmacologica indicata come stabilità del siero. Questa capacità può essere un fattore chiave nel prevedere il profilo farmacocinetico di un enzima e la sua idoneità per l'uso terapeutico.
Abstract
Il concetto di stabilità enzimatica è tipicamente usato per riferirsi alla termostabilità di un enzima, la sua capacità di mantenere la struttura e l'attività all'aumentare della temperatura. Per un enzima terapeutico, possono essere fondamentali anche altre misure di stabilità, in particolare la sua capacità di mantenere la funzione nel siero umano a 37 °C, che chiamiamo stabilità del siero. Qui, descriviamo un test in vitro per valutare la stabilità sierica dell'enzima wildtype Homo sapiens adenosina deaminasi I (HsADA1) utilizzando una procedura di micropiastra basata sull'assorbanza. In particolare, questo manoscritto descrive la preparazione di tamponi e reagenti, un metodo che organizza la coincubazione di HsADA1 nel siero, un metodo per analizzare i campioni di prova utilizzando un lettore di micropiastre e un'analisi di accompagnamento per determinare la frazione di attività che un enzima HsADA1 mantiene nel siero in funzione del tempo. Discutiamo inoltre le considerazioni per adattare questo protocollo ad altri enzimi, utilizzando un esempio di un enzima chinurenina Homo sapiens , per aiutare l'adattamento del protocollo ad altri enzimi in cui la stabilità sierica è di interesse.
Introduzione
Il seguente metodo consente a un utente di valutare quantitativamente la capacità di un enzima di mantenere la sua attività quando esposto a condizioni che imitano ciò che incontrerà dopo l'iniezione endovenosa. Il metodo in vitro imita tali condizioni in vivo e consiste nell'incubazione dell'enzima in un pool di siero umano a 37 °C e nell'analisi a tempo determinato della ritenzione dell'attività enzimatica. Ci riferiamo alla capacità di un enzima di mantenere l'attività in queste condizioni come stabilità del suo siero e il metodo di analisi per l'attività enzimatica sfrutta le differenze di assorbanza tra il substrato di un enzima e il prodotto risultante. Il concetto di stabilità del siero non è solo enzimatico specifico ed è stato applicato anche a diverse altre modalità di trattamento. Ad esempio, la stabilità sierica degli aptameri dell'RNA che hanno come bersaglio le proteine spike del COVID è stata precedentemente valutata monitorando la loro degradazione post-incubazione con siero fetale di vitello1. I peptidi antibatterici sono stati anche valutati per la loro capacità di sopprimere la crescita batterica dopo l'incubazione con siero umano aggregato2.
HsADA1 è un enzima appartenente alla classe delle transferiche di adenosina o 2-deossiadenosina in inosina o 2-deossiinosina, rispettivamente3. L'adenosina ha un picco di assorbimento di 260 nm, mentre l'inosina assorbe più forte a 250 nm4. Questo cambiamento nei picchi di assorbimento può essere rilevato su un lettore di micropiastre da una diminuzione dell'intensità di assorbimento a 260 nm quando HsADA1 viene aggiunto all'adenosina. L'enzima HsADA1 ha importanti implicazioni nel corpo umano e la sua carenza provoca una grave immunodeficienza combinata (ADA-SCID)5. L'omologo bovino di HsADA1, BtADA1, può essere utilizzato come terapia enzimatica sostitutiva per il trattamento di ADA-SCID, e abbiamo precedentemente dimostrato che HsADA1 wildtype perde la sua attività quando incubato con siero umano in pool, ostacolando potenzialmente il suo uso come terapeutico6. Pertanto, abbiamo selezionato l'enzima HsADA1 wildtype per dimostrare una procedura per determinare la stabilità sierica di un enzima. Un metodo di purificazione dettagliato per HsADA1 è stato descritto in precedenza6.
Nel seguente protocollo (come dettagliato nella Figura 1), dimostriamo come co-incubare HsADA1 wildtype in siero umano aggregato a 37 °C. Durante questo periodo, i campioni di prova vengono prelevati in punti temporali definiti e congelati per analisi future. Una volta prelevati tutti i campioni, viene eseguito un saggio su micropiastra in cui ogni campione viene combinato con il substrato, con la diminuzione dell'assorbanza risultante che è una correlazione per l'attività mantenuta dell'enzima. Vengono mostrati risultati rappresentativi che illustrano la stabilità sierica di HsADA1 e, poiché questa metrica può essere rilevante per determinare il potenziale valore terapeutico di altri enzimi, discutiamo anche le considerazioni per adattare questo protocollo a un enzima chinureninasi ingegnerizzato di Homo sapiens (HsKYNase) e a qualsiasi enzima più in generale. La HsKYNasi è un enzima coinvolto nel metabolismo del triptofano ed è in grado di degradare i sottoprodotti del triptofano chinurenina e idrossi-chinurenina (OH-Kyn) in acido antranilico e acido idrossi-antranilico (OH-AA), rispettivamente. La modulazione enzimatica del metabolismo del triptofano può essere di rilevanza terapeutica7.
Protocollo
1. Incubazione del siero
- Preparare una dose 10x di HsADA1 in 1x PBS pH 7,4 (1x PBS) a una concentrazione finale di 10 μM. Scongelare un'aliquota di 15 mL di siero umano aggregato e preriscaldarla a 37 °C. Preriscaldare un'aliquota da 50 mL di 1x PBS a 37 °C.
- Preparare le miscele di incubazione enzima-siero aggiungendo 100 μl del stock 10x HsADA1 a 900 μl di siero umano aggregato in una provetta da microcentrifuga a basso legame, denominata miscela enzima + siero. In una provetta da microcentrifuga separata a basso legame, aggiungere 100 μl del 10x HsADA1 stock a 900 μl di 1x PBS per il confronto con un controllo non sierico, indicato come miscela enzima + 1x PBS. La concentrazione finale di HsADA1 in ciascuna miscela sarà di 1 μM.
- Preparare un controllo aggiuntivo in una provetta da microcentrifuga a basso legame composta da 100 μl di 1x PBS miscelato con 900 μl di siero umano aggregato. Questo servirà come controllo non enzimatico per la miscela enzima + siero. Inoltre, effettuare un controllo composto da 1 ml di 1x PBS. Questo servirà come controllo non enzimatico per la miscela enzimatica + 1x PBS.
- Aliquotare 100 μl di ciascuna miscela dalla fase 1.2 in provette separate a basso legame e diluire 1:1 con 100 μl di glicerolo al 30% (v/v) in 1x PBS. Congelare con azoto liquido e conservare a -80 °C. Questi campioni saranno il punto temporale del Giorno 0 e la concentrazione di HsADA1 in essi sarà di 500 nM. Ripetere questo passaggio per le miscele di controllo non enzimatiche dal punto 1.3. Sigillare tutti gli enzimi e le miscele di controllo con una pellicola trasparente e metterle in un incubatore a 37 °C.
- Dopo 24 ore, aliquotare 100 μL di ciascun campione incubato e diluire 1:1 con 100 μL di glicerolo al 30% (v/v) in 1x PBS. Congelare con azoto liquido e conservare a -80 °C. Questi campioni saranno il punto temporale del giorno 1. Riportare tutte le miscele enzimatiche e di controllo nell'incubatore a 37 °C. Ripetere questo passaggio a 72 ore e 120 ore.
NOTA: I campioni quotati in aliquote non devono necessariamente essere congelati all'istante. In alternativa, l'utente può eseguire il test basato su micropiastra immediatamente dopo il prelievo di ciascun campione, anche se ogni volta che viene testato un campione devono essere preparate nuove curve standard e stock di substrato di adenosina.
2. Saggio basato su micropiastra
- Preparare il lettore di micropiastre impostando un protocollo di lettura cinetica (Figura 2) per misurare l'assorbanza a 260 nm con l'intervallo di lettura più breve per 30 minuti e preriscaldare il lettore a un setpoint di 37 °C.
- Scongelare tutti i campioni raccolti sul ghiaccio. Diluire ogni campione scongelato 1:10 utilizzando 1x PBS preriscaldato a 37 °C come diluente in provette per microcentrifuga a basso legame. La nuova concentrazione di HsADA1 in questi campioni sarà di 50 nM.
- Effettuare una diluizione 10x in 1x PBS anche dal campione di controllo del siero non enzimatico. Inoltre, preparare un stock di adenosina da 5 mM aggiungendo 66,8 mg di adenosina a 50 mL di 1x PBS. Preparare una diluizione di 250 μM di adenosina aggiungendo 400 μL di adenosina stock a 7.600 μL di 1x PBS e preriscaldarla a 37 °C in un incubatore.
- A una micropiastra a 96 pozzetti compatibile con i raggi UV, aggiungere 160 μl di diluizione di adenosina da 250 μM e 40 μl di ciascun campione proteico diluito in triplicato per un volume totale di 200 μl. Ciò produrrà una concentrazione finale di adenosina di 200 μM e una concentrazione enzimatica finale di 10 nM.
- Ripetere la diluizione per i campioni di controllo aggiungendo 40 μl di siero diluito o 1x controlli PBS e 160 μl di diluizione di adenosina 250 μM in triplicato. Questo produrrà anche una concentrazione finale di adenosina di 200 μM ma senza enzima.
NOTA: Può essere utile aggiungere prima tutti i campioni di enzima diluito e di controllo singolarmente alla micropiastra e poi aggiungere il substrato di adenosina con una micropipetta a 12 canali per ridurre il tempo di caricamento totale. Inoltre, in questa fase è necessario utilizzare piastre trasparenti ai raggi UV per evitare che la piastra stessa confonda il segnale di assorbanza misurato. - Misurare l'assorbanza di ciascun pozzetto a 260 nm per 30 minuti a 37 °C. Una volta terminato, esporta i dati in un foglio di calcolo per ulteriori analisi.
3. Analisi dei dati del lettore di micropiastre
- Determinare la pendenza della regione lineare dei dati di assorbanza in funzione del tempo per i primi minuti dei dati della micropiastra. Per i campioni diluiti con siero, questo corrispondeva ai primi ~120 s. Per i campioni diluiti con PBS 1x, questo corrispondeva ai primi ~270 s. Questa pendenza dei valori di assorbanza decrescenti avrà un valore numerico negativo e avrà unità di variazione in unità di assorbanza al secondo (ΔA260/s).
- Sottrarre la pendenza del controllo negativo costituito da siero umano aggregato dalla pendenza dei dati enzima + siero. Eseguire la stessa operazione sulle miscele enzimatica + 1x PBS sottraendo la pendenza del controllo 1x PBS.
- Normalizzare tutte le pendenze regolate alla pendenza originale a t=0 h per ottenere la frazione di attività rimanente utilizzando l'equazione 1.
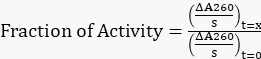 Equazione 1
Equazione 1 - Traccia la frazione di attività rimanente rispetto al tempo.
Risultati
Le cifre mostrano i risultati dell'esecuzione del test quando condotto con HsADA1 wildtype. Le figure 3A, B illustrano le curve di declino dell'assorbanza a 260 nm dei campioni originati dalle miscele 1x PBS/siero-enzima per HsADA1 wildtype dopo l'aggiunta di adenosina. Questa diminuzione dell'assorbanza in funzione dei dati temporali è ciò che l'utente può aspettarsi dopo aver completato con successo il test basato su micropiastra ed è ...
Discussione
Questo protocollo utilizza la variazione dell'assorbanza quando il substrato viene convertito nel prodotto per misurare l'attività di un enzima. Pertanto, il substrato e il prodotto devono avere profili spettrali distinti. Questo è il caso dell'adenosina e dell'inosina, entrambe con profili spettrali distinti e coefficienti di estinzione compresi tra 260-265 nm 6,8,12,13.
Divulgazioni
JB e MRJ sono inventori di brevetti o domande di brevetto relative agli enzimi adenosina deaminasi e/o chinureninasi. Tutti gli altri autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dal National Institutes of Health [1DP2CA280622-01] e finanziato da Biolocity. Ringraziamo la Dott.ssa Maria Jennings e Andrea Fox per aver fornito i vettori di espressione di HsADA1 e HsKYNase.
Materiali
| Name | Company | Catalog Number | Comments |
| Adenosine | Sigma Aldrich | A9251-25G | 25 g |
| BioTek Synergy HT Microplate Reader | |||
| Eppendorf LoBind Microcentrifuge Tubes: Protein | Fisher Scientific | 13-698-795 | 2 mL |
| Glycerol | Fisher Scientific | G33-4 | 4 L |
| HsKYNase66-W102H-T333N | In-house | ||
| Human Serum, Pooled | MP Biomedicals | 92931149 | 100 mL |
| Hydroxy-kynurenine | Cayman Chemicals | 27778 | |
| Inosine | TCI | I0037 | 25 g |
| PBS, 1x pH 7.4+/- 0.1 | Corning | 21-040-CM | |
| Pyridoxal 5-phosphate monohydrate, 99% | Thermo Scientifc | 228170010 | 1 g |
| UV-STAR MICROPLATE, 96 WELL, COC, F-BOTTOM | Greiner Bio | 655801 | |
| Wildtype Human Adenosine Deaminase 1 | In-house |
Riferimenti
- Valero, J., et al. A serum-stable RNA aptamer specific for SARS-COV-2 neutralizes viral entry. Proc Natl Acad Sci U S A. 118 (50), e2112942118 (2021).
- Iannuzo, N., et al. High-throughput screening identifies synthetic peptides with antibacterial activity against mycobacterium abscessus and serum stability. ACS Omega. 7 (27), 23967-23977 (2022).
- Ma, M. T., Jennings, M. R., Blazeck, J., Lieberman, R. L. Catalytically active holo homo sapiens adenosine deaminase i adopts a closed conformation. Acta Crystallograph Sect D. 78 (1), 91-103 (2022).
- Li, W., et al. Determination of 4 nucleosides via one reference compound in chinese cordyceps by hplc-uv at equal absorption wavelength. Natural Prod Comm. 18 (3), 1934578X231161410 (2023).
- Whitmore, K. V., Gaspar, H. B. Adenosine deaminase deficiency - more than just an immunodeficiency. Front Immunol. 7, 314 (2016).
- Jennings, M. R., et al. Optimized expression and purification of a human adenosine deaminase in E. coli and characterization of its asp8asn variant. Prot Express Purificat. 213, 106362 (2024).
- Blazeck, J., et al. Bypassing evolutionary dead ends and switching the rate-limiting step of a human immunotherapeutic enzyme. Nat Catalysis. 5 (10), 952-967 (2022).
- Lu, J., Grenache, D. G. Development of a rapid, microplate-based kinetic assay for measuring adenosine deaminase activity in body fluids. Clinica Chimica Acta. 413 (19), 1637-1640 (2012).
- Maciel, L. G., Dos Anjos, J. V., Soares, T. A. Fast and low-cost evaluation of hydroxykynurenine activity. MethodsX. 7, 100982 (2020).
- Bokman, A. H., Schweigert, B. S. 3-hydroxyanthranilic acid metabolism. Iv. Spectrophotometric evidence for the formation of an intermediate. Arch Biochem Biophys. 33 (2), 270-276 (1951).
- Tanford, C. . Advances in protein chemistry. 23, 121-282 (1968).
- Tritsch, G. L. Validity of the continuous spectrophotometric assay of kalckar for adenosine deaminase activity. Anal Biochem. 129 (1), 207-209 (1983).
- Gracia, E., et al. The catalytic site structural gate of adenosine deaminase allosterically modulates ligand binding to adenosine receptors. FASEB J. 27 (3), 1048-1061 (2013).
- Kalckar, H. M. Differential spectrophotometry of purine compounds by means of specific enzymes: Iii. Studies of the enzymes of purine metabolism. J Bio Chem. 167 (2), 461-475 (1947).
- Hartwick, R., Jeffries, A., Krstulovic, A., Brown, P. R. An optimized assay for adenosine deaminase using reverse phase high pressure liquid chromatography. J Chromatographic Sci. 16 (9), 427-435 (1978).
- Paul, M. K., Grover, V., Mukhopadhyay, A. K. Merits of hplc-based method over spectrophotometric method for assessing the kinetics and inhibition of mammalian adenosine deaminase. J Chromatography B. 822 (1), 146-153 (2005).
- Ubbink, J. B., Vermaak, W. J. H., Bissbort, S. H. High-performance liquid chromatographic assay of human lymphocyte kynureninase activity levels. J Chromatography B: Biomed Sci Appl. 566 (2), 369-375 (1991).
- Tsentalovich, Y. P., Snytnikova, O. A., Forbes, M. D. E., Chernyak, E. I., Morozov, S. V. Photochemical and thermal reactivity of kynurenine. Exp Eye Res. 83 (6), 1439-1445 (2006).
- Demain, A. L., Vaishnav, P. Production of recombinant proteins by microbes and higher organisms. Biotechnol Adv. 27 (3), 297-306 (2009).
- Gräslund, S., et al. Protein production and purification. Nat Methods. 5 (2), 135-146 (2008).
- Faber, M. S., Whitehead, T. A. Data-driven engineering of protein therapeutics. Curr Opin Biotechnol. 60, 104-110 (2019).
- Das, S., Zhao, L., Elofson, K., Finn, M. G. Enzyme stabilization by virus-like particles. Biochemistry. 59 (31), 2870-2881 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon