Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Caryotype spectral pour l'étude d'anomalies chromosomiques dans les humains et les souris de la maladie polykystique des reins
Dans cet article
Résumé
Caryotype spectral (SKY) est une technique avancée cytogénétique à identifier aberrations génomiques et chromosomiques. Cette technique tire avantage des sondes de peinture chromosomique, ce qui permet la classification de tous les chromosomes. SKY peut également identifier des aberrations chromosomiques complexes et les défauts d'isolement chez les souris et les humains atteints de diverses maladies, y compris la maladie polykystique des reins.
Résumé
Méthode conventionnelle pour identifier et classer des chromosomes individuels dépend de la configuration unique des bandes de chaque chromosome dans une espèce spécifique en cours d'analyse 1, 2. Cette technique classique de baguage, cependant, n'est pas fiable dans l'identification des aberrations chromosomiques complexes tels que ceux associés au cancer. Pour surmonter les limites de la technique de baguage, caryotype spectral (SKY) est introduite pour fournir beaucoup d'informations fiables sur les anomalies chromosomiques.
SKY est une fluorescence multicolore hybridation in situ (FISH) technique pour détecter les chromosomes en métaphase avec un microscope spectrale 3, 4. SKY a été prouvé être un outil précieux pour l'analyse cytogénétique d'un large éventail d'anomalies chromosomiques associées à un grand nombre de maladies génétiques et les tumeurs malignes 5, 6. SKY implique l'utilisation de sondes multicolores d'ADN par fluorescence-labellisés préparés à partir de l'oligonucle dégénéréeamorces otide par PCR. Ainsi, chaque chromosome a une couleur spectrale unique après dans-in situ hybridation avec des sondes, qui sont marquées de façon différentielle avec un mélange de colorants fluorescents (rhodamine, Texas Red, Cy5, FITC et Cy5.5). Les sondes utilisées pour SKY comporter jusqu'à 55 sondes chromosomiques spécifiques 7-10.
La procédure pour SKY comporte plusieurs étapes (Figure 1). SKY nécessite la disponibilité de cellules avec index mitotique élevé à partir de tissu normal ou malade ou de sang. Les chromosomes d'une cellule unique à partir d'une cellule fraîchement isolés primaire ou une lignée cellulaire sont étalées sur une lame de verre. Cette propagation chromosome est marqué avec une combinaison différente de colorants fluorescents spécifiques pour chaque chromosome. Pour sonde de détection et d'acquisition d'image, le système d'imagerie spectrale se compose de l'interféromètre de Sagnac et une caméra CCD. Ceci permet de mesurer le spectre de la lumière visible émise par l'échantillon et d'acquérir une image spectrale vientchromosomes m individuels. HiSKY, le logiciel utilisé pour analyser les résultats des images capturées, fournit une identification facile des anomalies chromosomiques. Le résultat final est une métaphase et une image de classification caryotype, dans lequel chaque paire de chromosomes a une couleur distincte (figure 2). Cela permet une identification facile des identités et des translocations chromosomiques. Pour plus de détails, s'il vous plaît visitez le site Web Applied Spectral Imaging ( http://www.spectral-imaging.com/ ).
SKY a été récemment utilisé pour l'identification des défauts de ségrégation des chromosomes et des anomalies chromosomiques chez l'homme et la souris avec rénale autosomique dominante polykystique (PKRAD), une maladie génétique caractérisée par un dysfonctionnement dans les cils primaires 11-13. En utilisant cette technique, nous avons démontré la présence de la ségrégation des chromosomes anormaux et anomalies chromosomiques chez les patients PKRAD et des modèles de souris 14. Des analyses plus poussées en utilisant SKY non seulement nous a permis d'identifier nombre chromosomique et de l'identité, mais aussi de détecter avec précision très complexes aberrations chromosomiques telles que des délétions chromosomiques et des translocations (figure 2).
Protocole
1. Prétraitement des cellules et la préparation métaphase
- Les cellules sont cultivées dans la modification de Dulbecco du milieu de Eagle (DMEM) contenant 10-15% de sérum bovin foetal (FBS) et 1% de pénicilline / streptomycine à 37 ° C avec 5% de CO 2 incubateur, jusqu'à ce qu'ils atteignent 70-80% de confluence.
- Traiter les cellules avec une solution colcémide à 0,05 pg / ml pendant 30-60 min.
- Recueillir le milieu contenant des cellules flottantes dans des tubes stériles de 50 ml à centrifuger faucon. Rincer les cellules sur la plaque avec sterile1X-PBS. Après une incubation avec de la trypsine stérile pendant 1-2 min, la récolte et de recueillir les cellules restantes dans le même tube.
- Faites tourner les tubes à 1000 rpm pendant 5 min, aspirer le surnageant en laissant 0,5 ml et desserrer culot en feuilletant avec le doigt uniquement.
- Selon la taille de granulés, ajoutez 5-10 ml de solution hypotonique de KCl 0,56% en dH 2 O et incuber la suspension à 37 ° C pendant 30-45 min.
- Ajouter une goutte de méthanol / acide acétique(03:01 vol / vol) par ml de suspension cellulaire hypotonique et inverser le tube avec précaution pour mélanger.
- Centrifuger à 1200 tours par minute pendant 5 minutes et recueillir des granules comme l'étape 1.4, puis ajoutez 5 ml de méthanol frais / acide acétique (3:1 vol / vol) goutte à goutte la solution de fixation tout en feuilletant le culot en continu. Cette procédure est essentielle afin que les étalements métaphasiques ne sera pas pris au piège dans les touffes des cellules qui pourraient compromettre l'expérience.
- Centrifuger de nouveau à 1200 rpm pendant 5 min et ajoutez 5-10 ml de méthanol glacé / fixateur acide acétique le long de la paroi du tube. A ce stade, si nécessaire, les cellules peuvent être stockées dans la solution de fixation dans un tube serré et scellé à -20 ° C à court terme ou -80 ° C pour le long terme (années) pour une utilisation future.
- Diapositives Propreté dans l'éthanol absolu, puis trempez la diapositive dans dH 2 O pendant approximately10X afin de former une gaine de l'eau sur la surface de la diapositive. Placer la lame sur une plaque de verre et de baisse de 15 20 pi de suspension cellulaire (de l'étape 1.8) De 10 "au dessus de la diapositive. Placez la lame dans un bain-marie réglé à 65-70 ° C pendant 1-2 min et laisser sécher.
- Vérifiez la lame sous un microscope optique 10x et 40x en utilisant un objectif à sec en s'assurant qu'il existe des chromosomes en métaphase et les pâtes à tartiner sont régulièrement espacées. Vérification de la présence cytoplasme entourant les chromosomes. Si cytoplasme est présente procéder à un prétraitement diapositive (digestion par la pepsine), si aucune cytoplasme est présente et les chromosomes ont une bonne morphologie, alors il n'ya pas besoin de prétraitement diapositive.
2. Prétraitement de diapositives (la pepsine de traitement)
- Appliquer 120 pi de 1:200 RNase solution (20 mg / ml) dissous dans 2X SSC-sur une lamelle de microscope 24 mm x 60 mm et inverser la surface de glissement vers le bas sur la métaphase de la lamelle, puis doucement la surface de glissement métaphase et incuber à 37 ° C pendant 45 min.
- Retirez délicatement la lamelle sans rayer la lame et laver dans 2X SSC-tampon dans une jarre Coplin toutes les 5 min pendant 15 minavec agitation.
- Ajouter 5-15 ul de la solution mère la pepsine (100 mg / ml dans dH 2 O) dans un bécher propre, puis ajouter 100 ml d'préchauffé (37 ° C) 0,01 M HCl. Il est important que la pepsine est ajouté dans un récipient propre bécher d'abord et non pas directement dans la solution de HCl, sinon il ne serait pas dissoudre dans une solution. Incuber la lame dans une jarre Coplin contenant la solution de HCl / pepsine à 37 ° C pendant 3-5 min. Cette étape est très critique que la digestion trop de provoquer des chromosomes à overdigested et trop peu sortir de la digestion du cytoplasme non digérés qui pourrait conduire à une liaison non spécifique de la sonde et interférer avec le signal d'hybridation.
- Laver la lame dans une jarre Coplin contenant 100 ml 1X-PBS pendant 5 min à deux reprises à la température ambiante.
- Laver la lame dans une jarre Coplin contenant 100 ml 1X-PBS/MgCl 2 pour 5 min à température ambiante (50 ml de 1M MgCl 2 dans 950 ml d'1X-PBS).
- Placez la lame dans un pot de containin Coplin100 g ml de formaldéhyde à 1% pendant 10 min à température ambiante (1,7 ml de formaldéhyde 37% dans 100 ml d'1X-PBS/MgCl 2).
- Laver la lame dans une jarre Coplin contenant 100 ml 1X-PBS pendant 5 min.
- Observer la lame sous un microscope optique à l'aide 40X objectif à sec afin de s'assurer que les lames sont digérés correctement et aucun cytoplasme est présent et la morphologie des chromosomes est conservé. Sélectionnez une zone pour l'hybridation à l'aide d'un stylo en diamant.
3. Dénaturation des chromosomes et de la sonde et l'hybridation
- Préparer une nouvelle solution de dénaturation (70% formamide/2X SSC, pH 7,0) et préchauffer à 70-80 ° C dans une jarre Coplin placée dans un bain d'eau Lieu de la diapositive dans la jarre Coplin contenant la solution dénaturante dans un bain d'eau à 70 ° C pour les chromosomes de souris et de 80 ° C pour les chromosomes de l'homme pour 30s-1.5 min.
- Immédiatement placer la lame dans la glace froide d'éthanol à 70% pendant 3 min puis 80% et éthanol à 100% pendant 3 min chacun et l'air sec. Examinez la lamepour la morphologie des chromosomes. La morphologie des chromosomes Bon est notée par les chromosomes sombres et pas "phase-légers" ou halo chromosomes.
- Réchauffez la sonde SkyPaint (kit de peinture SKY; flacon n ° 1) à 37 ° C avec agitation pendant 20 min, vortex et centrifuger brièvement à 1000 rpm pendant quelques secondes.
- Dénaturer la sonde dans un thermocycleur programmé pour un cycle de deux étapes à 85 ° C pendant 5 min du cycle suivi par 37 ° C pendant 60 min pour permettre marqué-sonde d'ADN pour prérecuit.
- Appliquer 10 pi de la sonde dénaturée sur la zone d'hybridation et de la couverture avec un 22 mm x 40 mm lamelle veillant à ne pas emprisonner des bulles d'air. Sceller les bords de la lamelle avec du ciment caoutchouc et incuber dans une chambre humide à 37 ° C pendant 48-72 heures.
4. Sonde de détection fluorescente
- Retirer la lamelle avec soin et placer la lame dans une jarre Coplin contenant préchauffé (45 ° C) une solution de lavage I (fraîchement préparé du formamide à 50% en 2X SSC). Laver pendant 5 mintrois fois à 45 ° C dans un bain-marie à agitation à 45 rpm
- Laver la lame dans la solution de lavage II (1X SSC) à 45 ° C pendant 5 min à deux reprises avec agitation.
- Laver la lame dans la solution de lavage III (4X SSC/0.1% de Tween 20) pendant 5 min à 45 ° C avec agitation.
- Appliquer 80 pi de réactif de blocage (kit de peinture SKY; flacon # 2), placer une lamelle et incuber à 37 ° C pendant 30 min.
- Retirer la lame et permettre au fluide de s'écouler. Appliquer 80 pi de Cy5 réactif de coloration (concentré Anticorps détection CAD kit; flacon n ° 3), appliquer une lamelle et incuber à 37 ° C pendant 40 min.
- Laver la lame avec la solution de lavage III à 45 ° C pendant 5 min trois fois avec agitation.
- Appliquer 80 pi de réactif de coloration Cy5.5 (concentré Anticorps détection CAD kit; flacon n ° 4), placer une lamelle et incuber à 37 ° C pendant 40 min.
- Laver la lame avec la solution de lavage III à 45 ° C pendant 5 min trois fois avec agitation.
- Inclinez la diapositive et de permettre au fluide de égouttez-les. Appliquer 20 ul du réactif anti-fade DAPI (kit de peinture SKY; flacon n ° 5) et placer un 24 mm lamelle de microscope x 60 mm. Retirez soigneusement les bulles d'air qui peuvent se former. Les diapositives peuvent être visualisés immédiatement ou conservés à 4 ° C dans l'obscurité pendant une durée n'excédant pas 1 semaine.
5. L'acquisition des images et l'analyse
- L'acquisition des images se fait en regardant des diapositives en métaphase en utilisant un microscope Olympus équipé d'une lentille à immersion d'huile 60X, un cube spectral (conçus sur mesure triple filtre passe-bande), un filtre DAPI et un module interféromètre de Sagnac avec une caméra CCD.
- Spectral-caryotypes ont été effectuées en utilisant le logiciel Sky View (Applied Version imagerie spectrale 1,62) suivant le manuel de l'utilisateur.
- Après avoir analysé les images, les chromosomes peuvent être considérées comme des images en couleur (avec des couleurs spécifiques fluorescents), les images couleur pseudo (avec des couleurs pour la classification) et des images renversées DAPI (spécifique motif de bandes).
Une procédure complète du ciel prend habituellement environ une fois par semaine (figure 1). Cela comprend l'acquisition d'images et d'analyse à condition que les cellules en métaphase sont en quantité suffisante. L'analyse du caryotype révèle un caryotype normal de la souris (40, XY) des cellules de souris de type sauvage (figure 2a). En revanche, les cellules de PKD1 - / - de la souris (modèle de souris PKD) montre une augmentation significative du nombre de chromosomes et des anomalies structurelles, telles que des délétions chromosomiques (chromosome n ° 8) et des translocations des chromosomes (n ° 11 et 19) (Figure 2b). Nous avons également analysé les tissus vasculaires des patients PKRAD. Une simple étude en comptant les nombres chromosomiques ont indiqué que la non-PKRAD et quelques échantillons PKRAD vasculaires étaient normaux nombres chromosomiques de 23 paires (figure 2c). En général, cependant, nous avons observé l'échec de la ségrégation chromosomique, résultant dans 46 paires de chromosomes dans uneÉchantillons DPKD au lieu de 23 paires (figure 2d).
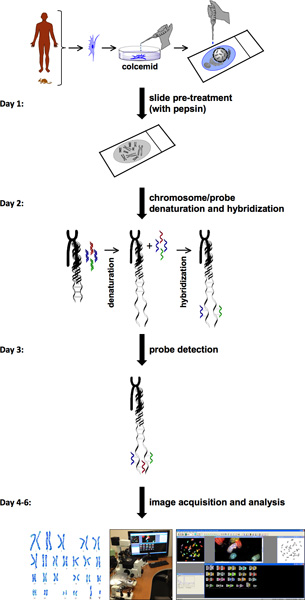
Figure 1. Organigramme protocole SKY. Diagramme du protocole SKY illustre les étapes pour mener à bien une expérience à partir de prétraitement des cellules et des préparations métaphasiques à l'acquisition de l'image et l'analyse. Un approximative d'une semaine calendrier est présenté sur la gauche à l'étape-par-étape des procédures pour chaque jour.

Figure 2. Caryotype spectral sur des lignées cellulaires immortalisées de souris fraîchement isolés et les cellules humaines primaires. Couleur et inversées images DAPI de chromosomes individuels ne soient présentées avant le tri chromosomique. Après le tri, les chromosomes sont présentés dans le "classement" de table. Une image du ciel) d'une propagation de la métaphase souris de type sauvage contient un caryotype normal (40, XY). B) d'une propagation de la métaphase PKD1 - / -. la souris indique le nombre anormal de chromosomes (68 au lieu de 40) et des anomalies chromosomiques telles que des délétions chromosomiques (chromosome n ° 8) et translocations chromosomiques (chromosomes n ° 11 et 19) c) SKY l'image d'une propagation métaphase à partir du tissu vasculaire d'un patient non-PKRAD a caryotype normal (46, XX). d) l'image d'une propagation SKY métaphase à partir du tissu vasculaire d'un patient PKRAD contient caryotype anormal (92, XXXX). Une partie des données ont déjà été signalés et réutilisé avec sa permission 14.
Discussion
Caryotype spectral (SKY) est une technique utilisée dans l'étude cytogénétique des compositions génomiques et chromosomiques. Cette technique tire avantage des sondes de peinture chromosomique, et la détection de ces sondes sont acquis par un interféromètre de Sagnac. Le processus complet SKY prend habituellement environ une semaine, et il comporte plusieurs étapes clés (figure 1). Le SKY utilise un protocole standard, qui a d'abord été décrit par Padilla Nash et al 3....
Déclarations de divulgation
Pas de conflits d'intérêt déclarés.
Remerciements
Auteurs tiens à remercier Brian Muntean, Shao Lo, Maki Takahashi et Blair Mell pour leur assistance technique. Ce travail a été soutenu par des prix du NIH (DK080640) et l'Université de Toledo & ProMedica de bourses de recherche translationnelle de stimulation à M. Surya Nauli.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Le numéro de catalogue | Commentaires |
| DMEM | Cellgro Mediatech Inc | 10-013-CV | |
| Sérum de veau fœtal (FBS) | Hyclone | SH30088-03 | |
| Penecillin / streptomycine | Thermo Scientific | SV30010 | |
| Colcemid | Roche Diagnostics | 10 295 892 001 | 10 pg / ml |
| HCl | Fisher Scientific | A144-500 | |
| KCl | Fisher Scientific | S77375-1 | |
| Phosphate de solution saline tamponnée au | Thermo Scientific | SH30256-01 | |
| SKY peinture sonde kit (Human) | Applied Spectral Imaging | SKY000028 | |
| SKY peinture sonde kit (souris) | Applied Spectral Imaging | SKY000030 | |
| Concentré kit de détection des anticorps | Applied Spectral Imaging | SKY000033 | |
| Trypsine | Thermo Scientific | SH30236.01 | |
| Le méthanol | Fisher Scientific | A433P-4 | |
| L'acide acétique | Fisher Scientific | A38-212 | |
| RNase A | Roche Diagnostics | 10 109 169 001 | |
| Pepsine | Sigma-Aldrich | P6887-5G | |
| MgCl 2 | Fluka | 63069-500ML | |
| Formaldéhyde 37% | Mallinckrodt Baker | 2106-02 | |
| 20X SSC | Promega | V4261 | |
| Le formamide | Fluka | 47671 | préparer juste avant utilisation |
| De Tween-20 | Fisher Scientific | BP337-500 | |
| Lames de verre de microscope | Fisher Scientific | 12-549 | |
| Microscope couvercle en verre 24x60mm | VWR Vista Vision | 16004-312 | |
| Colle de caoutchouc | Elmer (locales magasin de détail) | ||
| Hybridation / humidifiedchamber / Plateau | Simport | M920-2 | mettre des serviettes en papier humide au fond |
| Thermocycleur | Eppendorf | Epgradient S | |
| Secouant la plate-forme / Agitateur orbital | Bellco biotechnologie | ||
| Secouant salle de bain / eau | Précision scientifique | ||
| DAPI filtre cube | Technologie Chroma | ||
| SKY filtre cube | Technologie Chroma | ||
| SpectraCube | Applied Spectral Imaging | ||
| Microscope inversé culture cellulaire | Nikon | Nikon Eclipse TS100 | |
| Microscope à fluorescence | Olympe | IX70 | Huile de 60X |
Références
- Caspersson, T., Farber, S., Foley, G. E., Kudynowski, J., Modest, E. J., Simonsson, E., Wagh, U., Zech, L. Chemical differentiation along metaphase chromosomes. Experimental Cell Research. 49, 219-222 (1968).
- Seabright, M. A rapid banding technique for human chromosomes. Lancet. 2, 971-972 (1971).
- Padilla-Nash, H. M., Barenboim-Stapleton, L., Difilippantonio, M. J., Ried, T. Spectral karyotyping analysis of human and mouse chromosomes. Nature Protocols. 1, 3129-3142 (2006).
- Schrock, E., Zschieschang, P., O'Brien, P., Helmrich, A., Hardt, T., Matthaei, A., Stout-Weider, K. Spectral karyotyping of human, mouse, rat and ape chromosomes--applications for genetic diagnostics and research. Cytogenetic and Genome Research. 114, 199-221 (2006).
- Padilla-Nash, H. M., Heselmeyer-Haddad, K., Wangsa, D., Zhang, H., Ghadimi, B. M., Macville, M., Augustus, M., Schrock, E., Hilgenfeld, E., Ried, T. Jumping translocations are common in solid tumor cell lines and result in recurrent fusions of whole chromosome arms. Genes, Chromosomes & Cancer. 30, 349-363 (2001).
- Veldman, T., Vignon, C., Schrock, E., Rowley, J. D., Ried, T. Hidden chromosome abnormalities in haematological malignancies detected by multicolour spectral karyotyping. Nature Genetics. 15, 406-410 (1997).
- Karpf, A. R., Matsui, S. Genetic disruption of cytosine DNA methyltransferase enzymes induces chromosomal instability in human cancer cells. Cancer Research. 65, 8635-8639 (2005).
- Matsui, S., Faitar, S. L., Rossi, M. R., Cowell, J. K. Application of spectral karyotyping to the analysis of the human chromosome complement of interspecies somatic cell hybrids. Cancer Genetics and Cytogenetics. 142, 30-35 (2003).
- Nestor, A. L., Hollopeter, S. L., Matsui, S. I., Allison, D. A model for genetic complementation controlling the chromosomal abnormalities and loss of heterozygosity formation in cancer. Cytogenetic and Genome Research. 116, 235-247 (2007).
- Ried, T., Liyanage, M., du Manoir, S., Heselmeyer, K., Auer, K., Macville, M., Schrock, E. Tumor cytogenetics revisited: comparative genomic hybridization and spectral karyotyping. Journal of Molecular Medicine (Berlin, Germany). 75, 801-814 (1997).
- AbouAlaiwi, W. A., Takahashi, M., Mell, B. R., Jones, T. J., Ratnam, S., Kolb, R. J., Nauli, S. M. Ciliary polycystin-2 is a mechanosensitive calcium channel involved in nitric oxide signaling cascades. Circulation Research. 104, 860-869 (2009).
- Nauli, S. M., Alenghat, F. J., Luo, Y., Williams, E., Vassilev, P., Li, X., Elia, A. E., Lu, W., Brown, E. M., Quinn, S. J., Ingber, D. E., Zhou, J. Polycystins 1 and 2 mediate mechanosensation in the primary cilium of kidney cells. Nature Genetics. 33, 129-137 (2003).
- Nauli, S. M., Kawanabe, Y., Kaminski, J. J., Pearce, W. J., Ingber, D. E., Zhou, J. Endothelial cilia are fluid shear sensors that regulate calcium signaling and nitric oxide production through polycystin-1. Circulation. 117, 1161-1171 (2008).
- AbouAlaiwi, W. A., Ratnam, S., Booth, R. L., Shah, J. V., Nauli, S. M. Endothelial cells from humans and mice with polycystic kidney disease are characterized by polyploidy and chromosome segregation defects through survivin down-regulation. Human Molecular Genetics. 20, 354-367 (2011).
- Padilla-Nash, H. M., Nash, W. G., Padilla, G. M., Roberson, K. M., Robertson, C. N., Macville, M., Schrock, E., Ried, T. Molecular cytogenetic analysis of the bladder carcinoma cell line BK-10 by spectral karyotyping. Genes, Chromosomes & Cancer. 25, 53-59 (1999).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon