È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Cariotipo spettrale allo studio anomalie cromosomiche negli esseri umani e nei topi con malattia del rene policistico
In questo articolo
Riepilogo
Cariotipo spettrale (SKY) è una tecnica avanzata citogenetica per individuare aberrazioni genomiche e cromosomiche. Questa tecnica si avvale di sonde cromosomiche pittura, che permettono la classificazione di tutti i cromosomi. SKY può anche identificare aberrazioni cromosomiche complesse e difetti segregazione nei topi e nell'uomo con varie patologie, tra cui la malattia del rene policistico.
Abstract
Metodo convenzionale per identificare e classificare i singoli cromosomi dipende dal modello unico bande di ciascun cromosoma in una determinata specie in fase di analisi 1, 2. Questa tecnica classica banding, tuttavia, non è affidabile nell'identificare complesse aberrazioni cromosomiche come quelle associate con il cancro. Per superare i limiti della tecnica banding, cariotipo spettrale (SKY) viene introdotto per fornire molte informazioni affidabili sulle anomalie cromosomiche.
SKY è una fluorescenza multicolor ibridazione in situ (FISH), tecnica per rilevare cromosomi in metafase, con microscopio spettrale 3, 4. SKY ha dimostrato di essere uno strumento prezioso per l'analisi citogenetica di una vasta gamma di anomalie cromosomiche associate con un gran numero di malattie genetiche e tumori maligni 5, 6. SKY implica l'uso di sonde di DNA multicolor marcato in fluorescenza preparati dal degenerato oligonucleprimers otide mediante PCR. Così, ogni cromosoma ha un colore univoco spettrale dopo in-situ ibridazione con sonde, che sono differenzialmente etichettati con una miscela di coloranti fluorescenti (Rodamina, Texas Red, Cy5, FITC e Cy5.5). Le sonde utilizzate per SKY contenere fino a 55 sonde cromosoma specifiche 7-10.
La procedura per SKY prevede diversi passaggi (Figura 1). SKY richiede la disponibilità di cellule con elevato indice mitotico da tessuto normale o malato o di sangue. I cromosomi di una cellula singola da o una cella isolate di fresco primaria o una linea cellulare sono diffuse su un vetrino. Questo spread cromosoma è etichettato con una diversa combinazione di coloranti fluorescenti specifici per ciascun cromosoma. Per il rilevamento sonda e acquisizione delle immagini, il sistema di imaging spettrale consiste di interferometro Sagnac e una telecamera CCD. Questo permette misurazione dello spettro di luce visibile emessa dal campione e di acquisire una immagine spettrale frocromosomi m individuali. HiSKY, il software utilizzato per analizzare i risultati delle immagini catturate, fornisce una facile identificazione di anomalie cromosomiche. Il risultato finale è uno metafase e una immagine classificazione cariotipo, in cui ciascuna coppia di cromosomi ha un colore distinto (Figura 2). Questo permette una facile identificazione delle identità e le traslocazioni cromosomiche. Per ulteriori informazioni, visitare il sito Applied Spectral Imaging ( http://www.spectral-imaging.com/ ).
SKY è stato recentemente utilizzato per l'identificazione di difetti segregazione dei cromosomi e le anomalie cromosomiche negli esseri umani e nei topi con malattia autosomica dominante del rene policistico (ADPKD), una malattia genetica caratterizzata da una disfunzione in cilia primaria 11-13. Utilizzando questa tecnica, abbiamo dimostrato la presenza di segregazione dei cromosomi anormali e difetti cromosomici in pazienti ADPKD e modelli di topo 14. Ulteriori analisi basate SKY non solo ci ha permesso di identificare il numero cromosomico e identità, ma anche per rilevare con precisione le aberrazioni cromosomiche molto complessi come le delezioni e le traslocazioni cromosomiche (Figura 2).
Protocollo
1. Cella pretrattamento e la preparazione metafase
- Le cellule sono cresciute in modif Dulbeccòs di Medio di Eagle (DMEM) contenente 10-15% siero bovino fetale (FBS) e 1% penicillina / streptomicina a 37 ° C con 5% CO 2 incubatore, fino a raggiungere 70-80% confluenza.
- Trattare le cellule con soluzione Colcemid a 0,05 ug / ml per 30-60 min.
- Raccogliere il terreno contenente le cellule galleggianti da 50 ml sterili provette Falcon. Lavare le cellule sulla piastra con sterile1X-PBS. Dopo aver incubato con tripsina sterile per 1-2 min, raccolto e raccogliere le cellule rimanenti nel tubo stesso.
- Centrifugare le provette a 1000 rpm per 5 min, aspirare il surnatante lasciando 0,5 ml e allentare pellet da sfogliando con un dito solo.
- A seconda delle dimensioni pellet, aggiungere 5-10 ml di soluzione ipotonica dello 0,56% KCl in dH 2 O e incubare la sospensione a 37 ° C per 30-45 min.
- Aggiungere una goccia di metanolo / acido acetico(3:1 vol / vol) per ml di sospensione cellulare ipotonico e invertire delicatamente il tubo per la miscelazione.
- Centrifugare a 1200 rpm per 5 minuti e raccogliere il pellet come step 1.4, quindi aggiungere 5 ml di metanolo dolce / acido acetico (3:1 vol / vol) goccia a goccia la soluzione fissativa, mentre sfogliando il pellet continuamente. Questa procedura è critica così che i metafase non verrà intrappolato nella ciuffi delle cellule 'che comprometterebbero l'esperimento.
- Centrifugare nuovamente a 1200 rpm per 5 min e aggiungere 5-10 ml di ghiaccio-freddo metanolo / fissativo acido acetico lungo la parete del tubo. In questa fase, se necessario, le cellule possono essere conservate in soluzione fissativa in un tubo stretto e sigillato a -20 ° C per breve termine o -80 ° C per lungo termine (anni) per uso futuro.
- Vetrini Pulizia in etanolo assoluto, quindi immergerlo in dH 2 O per approximately10X per formare una guaina di acqua sulla superficie del vetrino. Porre il vetrino su una piastra di vetro e calo del 15-20μl di sospensione cellulare (dal punto 1,8) Da 10 "sopra il vetrino. Porre il vetrino in un bagnomaria regolato a 65-70 ° C per 1-2 minuti e lasciare asciugare.
- Controllare il vetrino sotto un microscopio ottico con obiettivi a secco 10X e 40X assicurandosi che vi sia cromosomi in metafase e gli spread sono distribuiti uniformemente. Verifica della presenza citoplasma che circonda i cromosomi. Se citoplasma è presente procedere con pretrattamento diapositiva (digestione pepsina), se non citoplasma è presente e le cromosomi hanno buona morfologia, allora non c'è bisogno di pretrattamento diapositiva.
2. Presentazione pretrattamento (pepsina trattamento)
- Applicare 120 ml di soluzione 1:200 RNasi (20 mg / ml) disciolto in 2X-SSC su un 24 mm coverglass microscopio x 60 mm e invertire la metafase faccia scivolare sul coverglass poi capovolgere delicatamente la faccia slitta metafase e incubare a 37 ° C per 45 min.
- Rimuovere con cautela il vetrino coprioggetti senza graffiare il vetrino e lavare in SSC 2X-buffer in una vaschetta Coplin ogni 5 min per 15 mincon agitazione.
- Aggiungere 5-15 pl di soluzione di archivio di pepsina (100 mg / ml in dH 2 O) in un bicchiere pulito e quindi aggiungere 100 ml di preriscaldato (37 ° C) 0.01M HCl. È importante che la pepsina viene aggiunto in un pulito becher prima e non direttamente nella soluzione di HCl, altrimenti non sarebbe sciogliere in soluzione. Incubare il vetrino in una vaschetta Coplin, contenente la HCl / pepsina soluzione a 37 ° C per 3-5 min. Questo passaggio è molto critica come digestione troppo causerà i cromosomi da overdigested e troppo digestione poco lascerà il citoplasma non digerito che potrebbe portare ad legame non specifico della sonda e interferire con il segnale di ibridazione.
- Lavare il vetrino in una vaschetta Coplin contenente 100 ml 1X-PBS per 5 min due volte a temperatura ambiente.
- Lavare il vetrino in una vaschetta Coplin contenente 100 ml 1X-PBS/MgCl 2 per 5 min a temperatura ambiente (50 ml di 1M MgCl 2 in 950 ml di 1X-PBS).
- Mettere il vetrino in una vaschetta Coplin containing 100 ml di formaldeide 1% per 10 min a temperatura ambiente (1,7 ml del 37% formaldeide in 100 ml di 1X-PBS/MgCl 2).
- Lavare il vetrino in una vaschetta Coplin contenente 100 ml 1X-PBS per 5 min.
- Osservare il vetrino sotto un microscopio ottico 40X con lenti a secco per assicurare che i vetrini vengono digeriti correttamente e non citoplasma è presente e la morfologia dei cromosomi è conservato. Selezionare un'area per l'ibridazione con una penna di diamante.
3. Cromosoma e sonda denaturazione e l'ibridazione
- Preparare la soluzione fresca denaturazione (70% formamide/2X SSC, pH 7.0) e preriscaldare a 70-80 ° C in una vaschetta Coplin posto in un luogo bagnomaria il vetrino nel vaso Coplin contenente la soluzione di denaturazione a bagnomaria a 70 ° C per i cromosomi del mouse e 80 ° C per i cromosomi umani per anni '30-1.5 min.
- Disporre il vetrino in ghiacciato etanolo al 70% per 3 min seguita da 80% e 100% di etanolo per 3 minuti ciascuna e lasciare asciugare all'aria. Esaminare il vetrinoper morfologia cromosoma. Buona la morfologia dei cromosomi si indica con cromosomi scure e non "phase-luce" o halo cromosomi.
- Riscaldare la sonda SkyPaint (vernice kit SKY; flaconcino # 1) a 37 ° C con agitazione per 20 min, vortex e centrifugare brevemente a 1000 rpm per pochi secondi.
- Denaturare il sonda in un termociclatore programmato per un due-step ciclo a 85 ° C per 5 ciclo min seguita da 37 ° C per 60 min per consentire marcato-sonda di DNA per preannealing.
- Applicare 10 pl della sonda denaturata sull'area di ibridazione e coprire con un mm 22 x 40 millimetri coprioggetti facendo attenzione a non intrappolare bolle d'aria. Sigillare i bordi del coverglass con cemento gomma e incubare in una camera umidificata a 37 ° C per 48-72 ore.
4. Sonda di rilevamento della fluorescenza
- Rimuovere il copri con cura e porre il vetrino in una vaschetta Coplin contenente preriscaldato (45 ° C) la soluzione di lavaggio I (preparata formamide 50% in 2X SSC). Lavare per 5 mintre volte a 45 ° C in un bagno d'acqua agitando a 45 rpm
- Lavare il vetrino nella soluzione di lavaggio II (1X SSC) a 45 ° C per 5 min due volte con agitazione.
- Lavare il vetrino nella soluzione di lavaggio III (4X SSC/0.1% Tween 20) per 5 min a 45 ° C con agitazione.
- Applicare 80 microlitri di reagente bloccante (paint kit SKY; flacone # 2), inserire un coprioggetto e incubare a 37 ° C per 30 min.
- Rimuovere il vetrino e consentire al fluido di drenare. Applicare 80 microlitri di reagente di colorazione Cy5 (Concentrated Ricerca anticorpi CAD kit; flacone # 3), applicare il coprioggetti e incubare a 37 ° C per 40 min.
- Lavare il vetrino con soluzione di lavaggio III a 45 ° C per 5 min tre volte con agitazione.
- Applicare 80 microlitri di reagente Cy5.5 colorazione (Concentrated Ricerca anticorpi CAD kit; fiala # 4), posizionare il coprioggetti e incubare a 37 ° C per 40 min.
- Lavare il vetrino con soluzione di lavaggio III a 45 ° C per 5 min tre volte con agitazione.
- Inclinare la diapositiva e consentire il fluido scolare. Applicare 20 l di anti-fade reattivo DAPI (paint kit SKY; flaconcino # 5) e mettere da 24 mm coverglass microscopio x 60 mm. Rimuovere con cura le bolle d'aria che potrebbero essere formate. Le diapositive possono essere ripreso immediatamente oppure conservati a 4 ° C al buio per non più di 1 settimana.
5. Acquisizione di immagini e analisi
- Acquisizione Immagine è compiuta da visualizzando vetrini metafase usando un microscopio Olympus equipaggiata con un obiettivo 60X immersione in olio, un cubo Spectral (progettato su misura tripla banda filtro passa), un filtro DAPI e un modulo interferometro Sagnac con una telecamera CCD.
- Spettrale cariotipi sono state effettuate utilizzando il software View SKY (Applied Version immagini spettrali 1,62) seguendo il manuale dell'utente.
- Dopo aver analizzato le immagini, i cromosomi possono essere visti come immagini a colori (con appositi colori fluorescenti), immagini a colori pseudo (con i colori per la classificazione) e invertiti immagini DAPI (specifico disegno a strisce).
Una procedura di SKY completa dura solitamente circa una volta alla settimana (Figura 1). Ciò include l'acquisizione delle immagini e l'analisi a condizione che le cellule in metafase sono in quantità adeguata. Analisi Karyotyping rivela cariotipo normale topo (40, XY) di cellule provenienti da topi wild type (Figura 2a). In contrasto, cellule da PKD1 - / - topo (modello topo PKD) mostra un significativo aumento in numero dei cromosomi e anomalie strutturali, quali delezioni cromosomiche (cromosoma # 8) e le traslocazioni (cromosomi # 11 e 19) (Figura 2b). Abbiamo anche analizzato i tessuti vascolari dai pazienti ADPKD. Uno studio semplice contando i numeri cromosomiche indicato che non-ADPKD e alcuni campioni ADPKD vascolari avuto normali numeri cromosomiche di 23 coppie (Figura 2c). In generale, però, abbiamo osservato fallimento di segregazione cromosomica, con conseguente in 46 coppie di cromosomi in ACampioni DPKD invece di 23 coppie (figura 2d).
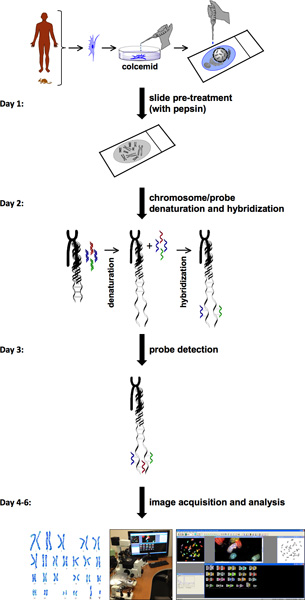
Figura 1. Diagramma di flusso protocollo di SKY. Diagramma di flusso del protocollo di SKY illustra i passaggi per completare un esperimento a partire dal pre-trattamento delle cellule in metafase e preparati per l'acquisizione delle immagini e analisi. Una approssimativa di una settimana timeline è presentato a sinistra con passo-passo le procedure per ogni giorno.

Figura 2. Cariotipo spettrale del mouse su linee cellulari e immortalati appena isolato le cellule primarie umane. Colori e immagini capovolte DAPI di cromosomi singoli mostrato prima l'ordinamento cromosomico. Dopo la cernita, i cromosomi sono presentati nella tabella "classificazione". A) l'immagine di SKY uno spread metafase dal topo wild-type contiene cariotipo normale (40, XY). B) SKY immagine di uno spread metafase da PKD1 - / -. del mouse mostra il numero dei cromosomi anormali (68 invece di 40) e le anomalie cromosomiche come le delezioni cromosomiche (cromosoma # 8) e le traslocazioni cromosomiche (cromosomi # 11 e 19) c) SKY immagine di uno spread metafase dal tessuto vascolare di un non-ADPKD paziente ha cariotipo normale (46, XX). d) immagine SKY di uno spread metafase dal tessuto vascolare di un paziente ADPKD contiene cariotipo anormale (92, XXXX). Parti dei dati sono stati riportati in precedenza e riutilizzato con autorizzazione 14.
Discussione
Cariotipo spettrale (SKY) è una tecnica utilizzata nello studio di citogenetica composizioni genomiche e cromosomiche. Questa tecnica si avvale di sonde cromosoma pittura, e il rilevamento di queste sonde sono stati acquisiti tramite un interferometro di Sagnac. Il processo di SKY completo dura di solito circa una settimana, e coinvolge diversi passaggi chiave (Figura 1). La SKY utilizza un protocollo standard, che è stato descritto da Padilla-Nash et al 3. Il protocollo è stato p...
Divulgazioni
Non ci sono conflitti di interesse dichiarati.
Riconoscimenti
Gli autori ringraziano Brian Muntean, Shao Lo, Maki Takahashi e Blair Mell per la loro assistenza tecnica. Questo lavoro è stato sostenuto da premi dal NIH (DK080640) e The University of Translational Research Award stimolazione Toledo & Promedica al dottor Surya Nauli.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reattivo | Azienda | Numero di catalogo | Comments |
| DMEM | Cellgro Mediatech Inc. | 10-013-CV | |
| Siero bovino fetale (FBS) | Hyclone | SH30088-03 | |
| Penecillin / streptomicina | Thermo Scientific | SV30010 | |
| Colcemid | Roche Diagnostics | 10 295 892 001 | 10 pg / ml |
| HCl | Fisher Scientific | A144-500 | |
| KCl | Fisher Scientific | S77375-1 | |
| Fosfato soluzione salina tamponata | Thermo Scientific | SH30256-01 | |
| SKY paint kit sonda (Human) | Applicata Imaging Spectral | SKY000028 | |
| SKY vernice sonda kit (mouse) | Applicata Imaging Spectral | SKY000030 | |
| Concentrato kit per il rilevamento degli anticorpi | Applicata Imaging Spectral | SKY000033 | |
| Tripsina | Thermo Scientific | SH30236.01 | |
| Metanolo | Fisher Scientific | A433P-4 | |
| Acido acetico | Fisher Scientific | A38-212 | |
| RNasi A | Roche Diagnostics | 10 109 169 001 | |
| Pepsina | Sigma-Aldrich | P6887-5G | |
| MgCl 2 | Fluka | 63069-500ML | |
| 37% Formaldeide | Mallinckrodt Baker | 2106-02 | |
| 20X SSC | Promega | V4261 | |
| Formammide | Fluka | 47671 | Preparare al momento dell'uso |
| Tween-20 | Fisher Scientific | BP337-500 | |
| Microscopio diapositive di vetro | Fisher Scientific | 12-549 | |
| Microscopio copertura in vetro 24x60mm | VWR Vista Vision | 16004-312 | |
| Mastice | Elmer (locali negozio al dettaglio) | ||
| Ibridizzazione / humidifiedchamber / Tray | Simport | M920-2 | mettere gli asciugamani di carta bagnati in fondo |
| Thermocycler | Eppendorf | Epgradient S | |
| Scuotendo la piattaforma / Agitatore orbitale | Bellco Biotecnologie | ||
| Scuotendo bagno / acqua | Rigore scientifico | ||
| DAPI filtro cubo | Tecnologia Chroma | ||
| SKY filtro cubo | Tecnologia Chroma | ||
| SpectraCube | Applicata Imaging Spectral | ||
| Inverted cella microscopio cultura | Nikon | Nikon Eclipse TS100 | |
| Microscopio a fluorescenza | Olimpo | IX70 | Olio 60X |
Riferimenti
- Caspersson, T., Farber, S., Foley, G. E., Kudynowski, J., Modest, E. J., Simonsson, E., Wagh, U., Zech, L. Chemical differentiation along metaphase chromosomes. Experimental Cell Research. 49, 219-222 (1968).
- Seabright, M. A rapid banding technique for human chromosomes. Lancet. 2, 971-972 (1971).
- Padilla-Nash, H. M., Barenboim-Stapleton, L., Difilippantonio, M. J., Ried, T. Spectral karyotyping analysis of human and mouse chromosomes. Nature Protocols. 1, 3129-3142 (2006).
- Schrock, E., Zschieschang, P., O'Brien, P., Helmrich, A., Hardt, T., Matthaei, A., Stout-Weider, K. Spectral karyotyping of human, mouse, rat and ape chromosomes--applications for genetic diagnostics and research. Cytogenetic and Genome Research. 114, 199-221 (2006).
- Padilla-Nash, H. M., Heselmeyer-Haddad, K., Wangsa, D., Zhang, H., Ghadimi, B. M., Macville, M., Augustus, M., Schrock, E., Hilgenfeld, E., Ried, T. Jumping translocations are common in solid tumor cell lines and result in recurrent fusions of whole chromosome arms. Genes, Chromosomes & Cancer. 30, 349-363 (2001).
- Veldman, T., Vignon, C., Schrock, E., Rowley, J. D., Ried, T. Hidden chromosome abnormalities in haematological malignancies detected by multicolour spectral karyotyping. Nature Genetics. 15, 406-410 (1997).
- Karpf, A. R., Matsui, S. Genetic disruption of cytosine DNA methyltransferase enzymes induces chromosomal instability in human cancer cells. Cancer Research. 65, 8635-8639 (2005).
- Matsui, S., Faitar, S. L., Rossi, M. R., Cowell, J. K. Application of spectral karyotyping to the analysis of the human chromosome complement of interspecies somatic cell hybrids. Cancer Genetics and Cytogenetics. 142, 30-35 (2003).
- Nestor, A. L., Hollopeter, S. L., Matsui, S. I., Allison, D. A model for genetic complementation controlling the chromosomal abnormalities and loss of heterozygosity formation in cancer. Cytogenetic and Genome Research. 116, 235-247 (2007).
- Ried, T., Liyanage, M., du Manoir, S., Heselmeyer, K., Auer, K., Macville, M., Schrock, E. Tumor cytogenetics revisited: comparative genomic hybridization and spectral karyotyping. Journal of Molecular Medicine (Berlin, Germany). 75, 801-814 (1997).
- AbouAlaiwi, W. A., Takahashi, M., Mell, B. R., Jones, T. J., Ratnam, S., Kolb, R. J., Nauli, S. M. Ciliary polycystin-2 is a mechanosensitive calcium channel involved in nitric oxide signaling cascades. Circulation Research. 104, 860-869 (2009).
- Nauli, S. M., Alenghat, F. J., Luo, Y., Williams, E., Vassilev, P., Li, X., Elia, A. E., Lu, W., Brown, E. M., Quinn, S. J., Ingber, D. E., Zhou, J. Polycystins 1 and 2 mediate mechanosensation in the primary cilium of kidney cells. Nature Genetics. 33, 129-137 (2003).
- Nauli, S. M., Kawanabe, Y., Kaminski, J. J., Pearce, W. J., Ingber, D. E., Zhou, J. Endothelial cilia are fluid shear sensors that regulate calcium signaling and nitric oxide production through polycystin-1. Circulation. 117, 1161-1171 (2008).
- AbouAlaiwi, W. A., Ratnam, S., Booth, R. L., Shah, J. V., Nauli, S. M. Endothelial cells from humans and mice with polycystic kidney disease are characterized by polyploidy and chromosome segregation defects through survivin down-regulation. Human Molecular Genetics. 20, 354-367 (2011).
- Padilla-Nash, H. M., Nash, W. G., Padilla, G. M., Roberson, K. M., Robertson, C. N., Macville, M., Schrock, E., Ried, T. Molecular cytogenetic analysis of the bladder carcinoma cell line BK-10 by spectral karyotyping. Genes, Chromosomes & Cancer. 25, 53-59 (1999).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon