Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Identification de lymphocytes T rares spécifiques de l’antigène dans des poumons de souris avec un peptide : tétramères du complexe majeur d’histocompatibilité
Dans cet article
Résumé
Nous fournissons un protocole détaillé pour isoler et identifier des populations rares de lymphocytes T spécifiques de l’antigène dans les poumons de souris grâce à l’enrichissement des lymphocytes T à base de billes magnétiques et aux tétramères du complexe majeur d’histocompatibilité (CMH).
Résumé
L’identification et la caractérisation des lymphocytes T spécifiques de l’antigène au cours de la santé et de la maladie restent essentielles pour améliorer notre compréhension de la physiopathologie immunitaire. Les défis techniques du suivi des populations de lymphocytes T spécifiques de l’antigène dans le répertoire des lymphocytes T endogènes ont été considérablement avancés par le développement de réactifs peptide :tétramère CMH. Ces multimères solubles marqués par fluorescence de molécules du CMH de classe I ou de classe II complexées à des épitopes peptidiques antigéniques se lient directement aux lymphocytes T avec la spécificité correspondante du récepteur des lymphocytes T (TCR) et peuvent donc identifier les populations de lymphocytes T spécifiques de l’antigène dans leur état natif sans qu’il soit nécessaire de disposer d’une réponse fonctionnelle induite par ex vivo stimulation. Pour les populations extrêmement rares, les lymphocytes T liés aux tétramères peuvent être enrichis magnétiquement pour augmenter la sensibilité et la fiabilité de la détection.
À mesure que l’étude de l’immunité des lymphocytes T résidents des tissus s’approfondit, il est urgent d’identifier les lymphocytes T spécifiques de l’antigène qui circulent vers les tissus non lymphoïdes et y résident. Dans ce protocole, nous présentons un ensemble détaillé d’instructions pour l’isolement et la caractérisation des lymphocytes T spécifiques de l’antigène présents dans les poumons de souris. Cela implique l’isolement des lymphocytes T à partir de tissus pulmonaires digérés, suivi d’une étape générale d’enrichissement magnétique des lymphocytes T et d’une coloration au tétramère pour l’analyse et le tri par cytométrie en flux. Les étapes mises en évidence dans ce protocole utilisent des techniques courantes et des réactifs facilement disponibles, ce qui le rend accessible à presque tous les chercheurs engagés dans l’immunologie des lymphocytes T de souris, et sont hautement adaptables pour une variété d’analyses en aval de toute population de lymphocytes T spécifiques à l’antigène à basse fréquence résidant dans les poumons.
Introduction
Au cœur du système immunitaire adaptatif se trouve la capacité d’un lymphocyte T à reconnaître et à répondre à un antigène spécifique. Le moment et l’endroit où un lymphocyte T répond à son antigène apparenté déterminent l’équilibre entre l’infection et l’auto-immunité, l’homéostasie et le cancer, la santé et la maladie1. Il s’ensuit que l’étude des lymphocytes T dans un contexte spécifique d’immunité devrait se concentrer sur les cellules présentant une spécificité pour un antigène pertinent d’intérêt. Parmi les avancées technologiques qui ont considérablement amélioré la capacité à caractériser les populations de lymphocytes T spécifiques de l’antigène, on trouve des multimères solubles marqués par fluorescence (généralement des tétramères) de molécules de classe I ou de classe II de complexes majeurs d’histocompatibilité (CMH) complexées à des épitopes de peptides antigéniques, mieux connus sous le nom de « tétramères peptides :CMH »2,3,4,5 . En représentant les ligands naturels des récepteurs de l’antigène des lymphocytes T (TCR), les tétramères peptide :CMH de classe I et de classe II permettent d’identifier directement les lymphocytes T CD8+ et CD4+ spécifiques de l’antigène, respectivement, dans le répertoire endogène des lymphocytes T du système immunitaire sans qu’il soit nécessaire de répondre à la stimulation de l’antigène dans un essai. Les tétramères représentent une approche plus élégante de l’étude des lymphocytes T spécifiques de l’antigène que les modèles de transfert adoptif de lymphocytes T transgéniques TCR6, et ont été de plus en plus utilisés pour identifier les populations de lymphocytes T étrangers et auto-spécifiques de l’antigène dans des modèles murins expérimentaux et des maladies humaines 4,5.
Alors que les tétramères peuvent facilement identifier les populations à haute fréquence de lymphocytes T qui se sont développées en réponse à la stimulation de l’antigène, leur utilisation pour les lymphocytes T naïfs, spécifiques de l’antigène ou mémoire est limitée par les très faibles fréquences de ces populations7. Notre groupe et d’autres ont développé et popularisé des stratégies d’enrichissement magnétique basées sur les tétramères qui augmentent la sensibilité de la détection pour permettre l’étude de ces populations cellulaires dans les tissus lymphoïdes de souris 8,9,10,11.
L’émergence des lymphocytes T résidents dans les tissus sur le terrain a mis l’accent sur le développement de nouvelles façons d’étudier les lymphocytes T dans l’espace non lymphoïde. Comme de nombreuses autres surfaces muqueuses, les lymphocytes T des poumons rencontrent une gamme d’antigènes propres et étrangers dérivés de l’épithélium de l’hôte, des microbes commensales et infectieux et des entités environnementales, y compris les allergènes. L’analyse transcriptionnelle des lymphocytes T prélevés dans des tissus non lymphoïdes (NLT) démontre un phénotype de type mémoire qui porte un destin et une fonction spécifiques aux tissus uniques, souvent dirigés vers le trafic et l’homéostasie tissulaire12. De plus, les lymphocytes T mémoires (Trms) résidents des tissus ont tendance à être plus limités par le clonage que ceux en circulation13. Il est essentiel de déterminer comment et pourquoi les antigènes entraînent la résidence des lymphocytes T dans la NLT pour comprendre comment le système immunitaire protège contre les infections, maintient l’homéostasie tissulaire et, parfois, se transforme en auto-immunité. Cependant, il semble y avoir une plus grande attrition parmi les lymphocytes T résidents des tissus des poumons par rapport aux autres NLT14. En conséquence, la capacité d’identifier et de caractériser les lymphocytes T endogènes du poumon avec une spécificité antigénique donnée est limitée par leur rareté inhérente.
En combinant l’utilisation de techniques d’enrichissement cellulaire à base de billes magnétiques et la coloration peptide :CMH tétramère, nous avons réussi à détecter des cellules T spécifiques de l’antigène du soi élargies mais rares dans les poumons de souris15,16. Nous présentons ici une description détaillée d’un protocole que nous avons optimisé pour isoler et caractériser de manière fiable toute population rare de lymphocytes T spécifiques de l’antigène présente dans les poumons de souris (Figure 1). Ce protocole comprend une étape de coloration des anticorps in vivo pour distinguer les cellules T résidentes des cellules T vasculaires17, suivie de deux méthodes différentes de traitement des tissus pulmonaires pour tenir compte de la disponibilité des ressources. S’ensuit une étape générale d’enrichissement magnétique des lymphocytes T, une coloration au tétramère et une analyse par cytométrie en flux. La viabilité cellulaire et la coloration des tétramères sont encore améliorées dans ce protocole par l’ajout d’aminoguanidine, qui bloque l’apoptose18 induite par l’activation des lymphocytes T induite par l’oxyde nitrique synthase (iNOS) et le dasatinib qui limite la régulation négative du TCR19. Les étapes mises en évidence dans ce protocole utilisent des techniques courantes et des réactifs facilement disponibles, ce qui le rend accessible à presque tous les chercheurs engagés dans l’immunologie des lymphocytes T de souris et est hautement adaptable à une variété d’analyses en aval. Bien qu’il soit peu probable que les lymphocytes T naïfs soient trouvés dans les poumons, nous pensons que ce protocole sera particulièrement utile pour l’étude des lymphocytes T spécifiques de l’antigène auto-spécifique et des Trms dans les poumons.
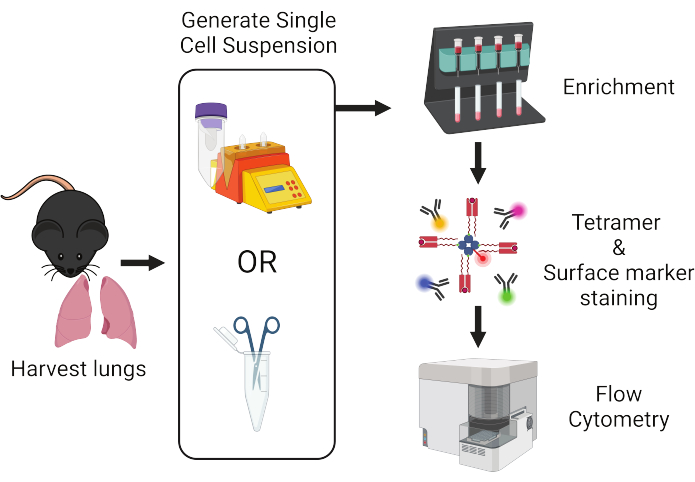
Figure 1 : Vue d’ensemble du flux de travail du protocole. Les poumons sont prélevés sur des souris et dissociés en cellules uniques. Les échantillons sont ensuite enrichis pour les lymphocytes T avant d’être colorés avec des tétramères peptide :CMH et des anticorps marqués par fluorescence pour l’analyse cytométrique en flux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Les procédures décrites dans ce protocole sont approuvées et élaborées conformément aux lignes directrices établies par l’Institutional Animal Care and Use Committee (IACUC) du Massachusetts General Hospital, un programme de gestion des animaux accrédité par l’American Association for the Accreditation of Laboratory Animal Care (AAALAC). Des expériences ont été réalisées sur des souris mâles et femelles âgées de 8 à 12 semaines sur un fond génétique C57BL/6 élevé et maintenu dans l’animalerie de l’HGM dans des conditions spécifiques exemptes d’agents pathogènes.
1. Préparation des solutions mères
- Préparez le tampon HEPES en dissolvant 10 mM de HEPES, 5 mM de KCl, 1,8 mM de CaCl2, 150 mM de NaCl et 1 mM de MgCl2, et ajustez à pH 7,4.
- Préparez 10 fois la solution mère de Liberase en dissolvant 700 μg/mL de Liberase Thermolysin Medium (TM) (voir le tableau des matériaux) dans la solution saline équilibrée de Hank avec du Ca++ et du Mg++ (voir le tableau des matériaux).
- Préparez une solution mère d’aminoguanidine 10x en dissolvant 100 mM de sel d’hémisulfate d’aminoguanidine (voir le tableau des matériaux) dans la solution saline équilibrée de Hank avec du Ca++ et du Mg++ (voir le tableau des matériaux).
- Préparez un milieu complet d’acides aminés de jambon d’Eagle (EHAA) en mélangeant un milieu EHAA (voir le tableau des matériaux) avec 10 % de sérum de veau fœtal, 100 μ/mL de pénicilline/streptomycine, 50 μg/mL de gentamycine, 2 mM de L-glutamine et 55 μM de 2-mercaptoéthanol.
REMARQUE : D’autres supports de cellules T courants, tels que RPMI ou DMEM, peuvent également être utilisés. - Préparez le tampon du trieur en mélangeant 1 PBS avec 2 % de FBS et 0,05 % d’azoture de sodium.
- Préparez le bloc Fc en diluant l’anticorps CD16/32 anti-souris purifié (voir la table des matériaux) à 1:100 v/v dans le tampon du trieur.
- Préparez la solution de digestion de Liberase TM/DNase en ajoutant 100 μg/mL de Liberase TM et 50 μg/mL de DNase I (voir le tableau des matériaux) dans un milieu RPMI 1640 sans L-glutamine (voir le tableau des matériaux).
- Préparez un mélange de kétamine et de xylazine en ajoutant 10 mg/mL de kétamine (voir le tableau des matériaux) et 1 mg/mL de xylazine (voir le tableau des matériaux) dans une solution saline normale (0,9 % de NaCl) (voir le tableau des matériaux).
2. Génération d’une suspension unicellulaire à partir de tissu pulmonaire via un dissociateur tissulaire automatisé
- Pour chaque souris, préparez 4 ml de tampon HEPES glacé dans un tube de dissociation tissulaire spécialisé (voir le tableau des matériaux) et conservez-le sur de la glace.
- Anesthésier les souris par injection intrapéritonéale de 150 à 200 μL de mélange de kétamine et de xylazine. Injecter 1 à 3 μg d’anticorps anti-souris CD45 conjugué au fluorophore (voir le tableau des matériaux) dilué dans 100 μL de solution saline normale par voie intraveineuse dans chaque souris anesthésiée. Euthanasier la souris 3 minutes après l’administration de l’anticorps avec une injection intrapéritonéale de 500 à 600 μL de mélange de kétamine et de xylazine.
- Réséquez les poumons et placez-les dans leur tube de dissociation tissulaire respectif refroidi sur de la glace. Placez les tubes sur le dissociateur tissulaire automatisé (voir tableau des matériaux) et exécutez le premier programme (programme prédéfini) pour le tissu pulmonaire.
- Ajouter 500 μL de solution mère de Liberase et 500 μL de solution mère d’aminoguanidine dans chaque tube contenant le tissu pulmonaire dissocié. Incuber sur un mélangeur à nutation pendant 30 min à 37 °C.
- Remettez les tubes sur le dissociateur et lancez le deuxième programme (programme prédéfini) pour le tissu pulmonaire.
- Verser la suspension unicellulaire du tube de dissociation tissulaire à travers une crépine cellulaire de 100 μm (voir le tableau des matériaux) dans un tube conique de 50 mL. Rincez le tube de dissociation tissulaire avec 5 ml d’EHAA complet (ou tout milieu de cellule T équivalent) et passez-le à travers la passoire dans le tube de 50 ml.
- Retirer la crépine et augmenter le volume de la suspension cellulaire à 50 mL avec l’EHAA complet. Centrifuger la suspension cellulaire à 400 x g pendant 5 min à 4 °C. Aspirez soigneusement le surnageant, en laissant derrière lui environ 100 μL de surnageant et de pastilles cellulaires.
- Ajouter environ 100 μL de solution Fc Block pour porter le volume total à 200 μL et agiter vigoureusement pour remettre la pastille en suspension.
3. Protocole alternatif : Génération d’une suspension unicellulaire à partir de tissu pulmonaire par dissociation manuelle
- Pour chaque souris, préparez 1 mL d’EHAA complet dans un tube de microfuge de 1,5 mL et conservez-le sur de la glace.
- Anesthésier les souris par injection intrapéritonéale de 150 à 200 μL de mélange de kétamine et de xylazine. Injecter 1 à 3 μg d’anticorps anti-souris CD45 conjugué au fluorophore dilué dans 100 μL de solution saline normale par voie intraveineuse dans chaque souris anesthésiée. Euthanasier la souris 3 minutes après l’administration de l’anticorps avec une injection intrapéritonéale de 500 à 600 μL de mélange de kétamine et de xylazine.
- Réséquez les poumons et placez-les dans leur tube microfuge respectif de 1,5 mL refroidi sur de la glace.
- Une fois que tous les poumons ont été prélevés, transférez chaque poumon dans un tube de microfuge frais sans aucun milieu cellulaire.
- Coupez les poumons en petits fragments (morceaux de ~2-3 mm) à l’aide de ciseaux à l’intérieur du tube de microfuge.
- Ajouter 1 mL de cocktail de digestion Liberase TM/DNase I dans chaque tube.
- Incuber 30 min à 37 °C au bain-marie. Remettez les échantillons dans la glace par la suite pour éviter une digestion excessive.
- Verser l’échantillon sur une passoire à cellules de 100 μm placée sur un tube conique de 50 mL. Écrasez le tissu contre le grillage avec l’extrémité en caoutchouc du piston à partir d’une seringue stérile de 1 ml.
- Rincer la passoire avec un tampon de tri à froid, en recueillant un volume final de 7 mL dans le tube. Centrifuger la suspension cellulaire à 400 x g pendant 5 min à 4 °C. Aspirez soigneusement le surnageant, en laissant environ 100 μL de surnageant et de pastille cellulaire.
- Ajouter 100 μL de solution Fc Block pour porter le volume total à 200 μL et agiter vigoureusement pour remettre la pastille en suspension.
4. Enrichissement de l’échantillon pulmonaire pour les lymphocytes T par enrichissement en billes magnétiques
- Ajouter 50 μL de microbilles CD90.2 anti-souris (voir le tableau des matériaux). Vortex et incuber pendant 10 min à 4 °C.
REMARQUE : CD90.2 correspond à l’allèle exprimé par les lymphocytes T chez les souris C57BL/6. Vérifiez le génotype de l’allèle si d’autres souches de souris sont utilisées. - Pendant ce temps, placez une colonne de séparation de cellules paramagnétique (voir Tableau des matériaux) sur un aimant de séparation de cellules à une ou quatre positions (voir Tableau des matériaux). Placez un tube conique ouvert de 15 mL sous chaque colonne pour capter le débit. Amorcez la colonne avec 3 ml de tampon de tri à froid, permettant à la colonne de s’écouler par gravité dans le tube conique situé en dessous.
- Une fois que les cellules ont incubé avec les microbilles pendant 10 min, ajoutez un tampon de tri à froid à un volume de 1 mL et transférez-les à travers une passoire cellulaire de 100 μm placée au sommet de la colonne. Recueillir le flux de la colonne dans un nouveau tube conique de 15 mL placé sous la colonne.
- Une fois que la suspension de la cellule s’est complètement écoulée à travers la colonne par gravité, lavez la colonne en rinçant le tube d’origine avec 3 ml supplémentaires de tampon de tri à froid et en l’appliquant sur la colonne à travers le grillage, rinçant ainsi la crépine. Retirez la passoire.
- Lorsque l’échantillon s’est de nouveau complètement écoulé dans la colonne et qu’aucun autre écoulement ne sort de la colonne, retirez la colonne de l’aimant et placez-la sur un tube conique neuf de 15 ml.
- Ajouter 5 mL de tampon de tri à froid dans la colonne.
- Éluez immédiatement les cellules liées à la colonne en poussant le piston de la colonne vers le haut en un mouvement continu et en forçant le tampon de tri hors du bas de la colonne dans le nouveau tube.
- Centrifuger les échantillons élués à 400 x g pendant 5 min à 4 °C. Aspirez soigneusement le surnageant, en laissant derrière lui environ 100 μL de surnageant et de pastilles cellulaires.
- Ajoutez 100 μL de tampon de tri à froid pour porter le volume total à 200 μL et agitez vigoureusement pour remettre la pastille en suspension.
- Analysez l’échantillon à flux continu contenant la fraction cellulaire non liée, si vous le souhaitez.
5. Coloration des lymphocytes T spécifiques de l’antigène avec des tétramères peptide :CMH
- Ajouter 1 μL de Dasatinib 10 μM (voir le Tableau des matières) à chaque échantillon, vortex et incuber pendant 5 min à RT.
- Ajouter un tétramère peptide :CMH conjugué PE ou APC à une concentration finale de 10 nM (ou une concentration optimisée empiriquement pour un tétramère donné).
- Vortex et incuber dans l’obscurité pendant 1 h à RT (ou temps et température optimisés empiriquement).
6. Analyse des lymphocytes T marqués au tétramère par cytométrie en flux
- Pendant que l’échantillon est coloré avec des tétramères peptide :CMH, préparez un mélange maître d’anticorps pour colorer les marqueurs de surface des cellules (tableau 1).
REMARQUE : Évitez les fluorophores qui entrent en conflit avec l’anticorps CD45 administré par voie intraveineuse ou les tétramères. - Avec 15 minutes restantes dans la période d’incubation pour la coloration au tétramère, ajoutez un mélange maître d’anticorps de surface à 1:100 (ou une concentration optimisée empiriquement) à chaque échantillon.
- Continuer à incuber dans l’obscurité à RT pour le reste de la période d’incubation de 1 h.
- Ajouter 50 000 billes de comptage par cytométrie en flux (voir le tableau des matériaux) et ajouter un tampon de tri à froid jusqu’à un volume final d’environ 5 ml. Centrifuger l’échantillon coloré à 400 x g pendant 5 min à 4 °C. Aspirer soigneusement le surnageant, en laissant derrière lui environ 100 μL de surnageant et de pastilles de cellules/billes.
- Remettez l’échantillon en suspension avec 200 μL supplémentaires de tampon de tri et transférez l’échantillon dans un tube de tri cellulaire activé par fluorescence (FACS) de 5 mL.
- Analysez l’échantillon sur un cytomètre en flux. Collectez autant de cellules que possible, jusqu’à un maximum de 2 500 000 événements au total. Assurez-vous qu’au moins 20 000 événements de cordon de comptage ont été collectés. Maintenez le taux d’acquisition à 5 000 événements par seconde ou moins.
- Enregistrez toutes les données sous forme de fichiers FCS.
| Fluorochrome | Anticorps |
| BUV395 | CD90.2 |
| Bleu Pacifique | CD45 (ajouté plus tôt par injection intraveineuse) |
| Orange du Pacifique | CD8 |
| Violet brillant 785 | CD4 |
| FITC | CD3 |
| PerCP-Cy5.5 | Vidage (B220, CD11b, CD11c, F4/80) |
| PE | Tétramère pMHC ou marqueur phénotypique |
| PE-Cy7 | Marqueur phénotypique (par exemple CD44,-1, CD69, etc.) |
| APC | Tétramère pMHC ou marqueur phénotypique |
| AlexaFluor 700 | Marqueur phénotypique (par exemple CD44,-1, CD69, etc.) |
| APC-Cy7 | Taches vivantes/mortes |
Tableau 1 : Matrice de coloration de l’échantillon. Un panel typique d’anticorps de cytométrie en flux conjugués aux fluorophores utilisés pour identifier et caractériser les lymphocytes T spécifiques de l’antigène.
7. Analyse des données
- Analyser les fichiers de données FCS à l’aide d’un logiciel de cytométrie en flux approprié.
- Mettre en place une séquence de portes d’inclusion successives pour identifier 1) les événements de type lymphocytaire, 2) uniques, 3) vivants, 4) extravasculaires, 5) négatifs et 6) CD90.2 positifs qui sont CD4 ou CD8 positifs (Figure 2).
- Identifier les lymphocytes T positifs aux tétramères au sein de ces populations.
- Déterminez le nombre absolu de cellules tétramères positives en calculant le rapport entre les billes ajoutées (50 000) et le nombre de billes collectées pendant l’échantillonnage et en multipliant la valeur par le nombre total de cellules tétramères positives collectées.
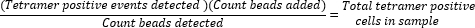
Résultats
La figure 2 illustre la stratégie de contrôle représentative utilisée dans l’identification des lymphocytes T CD4+ spécifiques de l’antigène rare dans les poumons avec des tétramères peptide :CMH de classe II. Le même processus peut être appliqué aux lymphocytes T CD8+ spécifiques de l’antigène avec des tétramères peptide :CMH de classe I (données non présentées).
En raison du nombre élevé de cellules non lymphoïd...
Discussion
Les caractérisations antérieures des lymphocytes T spécifiques de l’antigène dans les poumons ont bénéficié du nombre robuste de lymphocytes T spécifiques de l’antigène qui se développent à la suite d’un événement d’amorçage aigu tel qu’une immunisation intranasale ou une infection 20,21,22. Cependant, les populations de lymphocytes T plus rares dans les poumons, telles que les lymphocytes T spécifiques ...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions L. Kuhn pour son assistance technique pour le traitement des tissus et la production de tétramères. Ce travail a été financé par les National Institutes of Health (R01 AI107020 et P01 AI165072 à J.J.M., T32 AI007512 à D.S.S.), le Massachusetts Consortium on Pathogen Readiness (J.J.M) et le Massachusetts General Hospital Executive Committee on Research (J.J.M.).
matériels
| Name | Company | Catalog Number | Comments |
| 100 mm cell strainer | Fisher Scientific | 22-363-549 | |
| 10x PBS without Ca++ or Mg++ | Corning | 46-013-CM | |
| 1x PBS without Ca++ or Mg++ | Corning | 21-031-CV | |
| AccuCheck Counting Beads | Invitrogen | PCB100 | |
| Aminoguanidine Hemisulfate Salt | Sigma-Aldrich | A7009 | |
| CD90.2 microbeads, mouse | Miltenyi | 130-121-278 | |
| Cell separation magnet (MidiMACS Separator) | Miltenyi | 130-042-302 | Holds single LS column |
| Cell separation magnet (QuadroMACS Separator) | Miltenyi | 130-090-976 | Holds 4 LS columns |
| Dasatinib | Sigma-Aldrich | CDS023389 | |
| DNase I | Roche | 10104159001 | |
| Eagle’s Ham’s Amino Acids medium | Sigma-Aldrich | C5572 | |
| gentleMACS | Miltenyi | 130-093-235 | Automated tissue dissociator |
| gentleMACS C Tubes | Miltenyi | 130-093-237 | Automated tissue dissociator tubes |
| Hank's Balanced Salt Solution with Ca++ or Mg++ | Corning | 21-020-CM | |
| HEPES | Gibco | 15630080 | |
| Ketamine | Vedco | NDC 50989-996-06 | |
| Liberase TM | Roche | 5401119001 | |
| Pacific Blue anti-mouse CD45 antibody (Clone: 30-F11) | Biolegend | 103126 | |
| Paramagnetic cell separation columns (LS Columns) | Miltenyi | 130-042-401 | Comes with plunger |
| Purified anti mouse CD16/32 antibody (Clone: 93) | Biolegend | 101302 | |
| RPMI 1640 medium without L-glutamine | Corning | 15-040-CM | |
| Sodium Chloride 0.9% (Normal Saline) | Cytiva | Z1376 | |
| Xylazine | Pivetal | NDC 466066-750-02 |
Références
- Sun, L., Su, Y., Jiao, A., Wang, X., Zhang, B. T cells in health and disease. Signal Transduct Target Ther. 8 (1), 235 (2023).
- Altman, J. D., et al. Phenotypic analysis of antigen-specific T lymphocytes. Science. 274 (5284), 94-96 (1996).
- Crawford, F., Kozono, H., White, J., Marrack, P., Kappler, J. Detection of antigen-specific T cells with multivalent soluble class II MHC covalent peptide complexes. Immunity. 8 (6), 675-682 (1998).
- Nepom, G. T., et al. HLA class II tetramers: tools for direct analysis of antigen-specific CD4+ T cells. Arthritis Rheum. 46 (1), 5-12 (2002).
- Davis, M. M., Altman, J. D., Newell, E. W. Interrogating the repertoire: broadening the scope of peptide-MHC multimer analysis. Nat Rev Immunol. 11 (8), 551-558 (2011).
- Moon, J. J., et al. Tracking epitope-specific T cells. Nat Protoc. 4 (4), 565-581 (2009).
- Jenkins, M. K., Moon, J. J. The role of naive T cell precursor frequency and recruitment in dictating immune response magnitude. J Immunol. 188 (9), 4135-4140 (2012).
- Moon, J. J., et al. Naive CD4(+) T cell frequency varies for different epitopes and predicts repertoire diversity and response magnitude. Immunity. 27 (2), 203-213 (2007).
- Obar, J. J., Khanna, K. M., Lefrancois, L. Endogenous naive CD8+ T cell precursor frequency regulates primary and memory responses to infection. Immunity. 28 (6), 859-869 (2008).
- Kotturi, M. F., et al. Naive precursor frequencies and MHC binding rather than the degree of epitope diversity shape CD8+ T cell immunodominance. J Immunol. 181 (3), 2124-2133 (2008).
- Legoux, F. P., Moon, J. J. Peptide:MHC tetramer-based enrichment of epitope-specific T cells. J Vis Exp. (68), e4420 (2012).
- Szabo, P. A., Miron, M., Farber, D. L. Location, location, location: Tissue resident memory T cells in mice and humans. Sci Immunol. 4 (34), 9673 (2019).
- Poon, M. M. L., et al. Tissue adaptation and clonal segregation of human memory T cells in barrier sites. Nat Immunol. 24 (2), 309-319 (2023).
- Zheng, M. Z. M., Wakim, L. M. Tissue resident memory T cells in the respiratory tract. Mucosal Immunol. 15 (3), 379-388 (2022).
- Legoux, F. P., et al. CD4+ T cell tolerance to tissue-restricted self antigens is mediated by antigen-specific regulatory T Cells rather than deletion. Immunity. 43 (5), 896-908 (2015).
- Shin, D. S., et al. Lung injury induces a polarized immune response by self-antigen-specific CD4(+) Foxp3(+) regulatory T cells. Cell Rep. 42 (8), 112839 (2023).
- Anderson, K. G., et al. Intravascular staining for discrimination of vascular and tissue leukocytes. Nat Protoc. 9 (1), 209-222 (2014).
- Vig, M., et al. Inducible nitric oxide synthase in T cells regulates T cell death and immune memory. J Clin Invest. 113 (12), 1734-1742 (2004).
- Lissina, A., et al. Protein kinase inhibitors substantially improve the physical detection of T-cells with peptide-MHC tetramers. J Immunol Methods. 340 (1), 11-24 (2009).
- Hogan, R. J., et al. Protection from respiratory virus infections can be mediated by antigen-specific CD4(+) T cells that persist in the lungs. J Exp Med. 193 (8), 981-986 (2001).
- Hogan, R. J., et al. Activated antigen-specific CD8+ T cells persist in the lungs following recovery from respiratory virus infections. J Immunol. 166 (3), 1813-1822 (2001).
- Zhao, J., et al. Airway memory CD4(+) T cells mediate protective immunity against emerging respiratory coronaviruses. Immunity. 44 (6), 1379-1391 (2016).
- Hondowicz, B. D., et al. Interleukin-2-dependent allergen-specific tissue-resident memory cells drive asthma. Immunity. 44 (1), 155-166 (2016).
- Jungblut, M., Oeltze, K., Zehnter, I., Hasselmann, D., Bosio, A. Standardized preparation of single-cell suspensions from mouse lung tissue using the gentleMACS Dissociator. J Vis Exp. (29), e1266 (2009).
- Faustino, L. D., et al. Interleukin-33 activates regulatory T cells to suppress innate gammadelta T cell responses in the lung. Nat Immunol. 21 (11), 1371-1383 (2020).
- Atif, S. M., Gibbings, S. L., Jakubzick, C. V. Isolation and identification of interstitial macrophages from the lungs using different digestion enzymes and staining strategies. Methods Mol Biol. 1784, 69-76 (2018).
- Pape, K. A., Taylor, J. J., Maul, R. W., Gearhart, P. J., Jenkins, M. K. Different B cell populations mediate early and late memory during an endogenous immune response. Science. 331 (6021), 1203-1207 (2011).
- Naeher, D., et al. A constant affinity threshold for T cell tolerance. J Exp Med. 204 (11), 2553-2559 (2007).
- Moon, J. J., et al. Quantitative impact of thymic selection on Foxp3+ and Foxp3- subsets of self-peptide/MHC class II-specific CD4+ T cells. Proc Natl Acad Sci U S A. 108 (35), 14602-14607 (2011).
- Koehli, S., Naeher, D., Galati-Fournier, V., Zehn, D., Palmer, E. Optimal T-cell receptor affinity for inducing autoimmunity. Proc Natl Acad Sci U S A. 111 (48), 17248-17253 (2014).
- Zhang, Z., Legoux, F. P., Vaughan, S. W., Moon, J. J. Opposing peripheral fates of tissue-restricted self antigen-specific conventional and regulatory CD4(+) T cells. Eur J Immunol. 50 (1), 63-72 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon