Method Article
Cellules stromales hétéromulticellulaires dans des cultures 3D sans échafaudage de cellules cancéreuses épithéliales pour stimuler l’invasion
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Il existe un besoin critique de modèles de cancer 3D qui capturent la diaphonie hétérocellulaire pour étudier les métastases cancéreuses. Notre étude présente la génération d’épithélium stromal hétéromulticellulaire dans un environnement sans échafaudage qui peut être utilisé pour étudier l’invasion et les distributions spatiales cellulaires.
Résumé
Le cancer du sein est la deuxième cause de décès lié au cancer chez les femmes aux États-Unis. Il a été démontré que les modèles organoïdes de tumeurs solides récapitulent fidèlement les aspects de la progression du cancer tels que la prolifération et l’invasion. Bien que les organoïdes dérivés de patients et les organoïdes de xénogreffes dérivés de patients soient pertinents sur le plan physiopathologique, ils sont coûteux à propager, difficiles à manipuler et composés principalement des types de cellules les plus prolifératives au sein du microenvironnement tumoral (TME). Ces limitations empêchent leur utilisation pour élucider les mécanismes cellulaires de progression de la maladie qui dépendent des cellules stromales associées aux tumeurs qui se trouvent dans le TME et sont connues pour contribuer aux métastases et à la résistance au traitement.
Ici, nous rapportons des méthodes de culture de cultures 3D multicellulaires épithéliales-stromales. Les avantages de ces méthodes comprennent un système rentable permettant de générer rapidement des cultures 3D de type organoïde dans des environnements sans échafaudage qui peuvent être utilisés pour suivre l’invasion à une résolution unicellulaire dans des échafaudages d’hydrogel. Plus précisément, nous démontrons comment générer ces cultures 3D hétéromulticellulaires en utilisant des cellules cancéreuses du sein BT-474 en combinaison avec des fibroblastes (BJ-5ta), des cellules de type monocytes (THP-1) et/ou des cellules endothéliales (EA.hy926). De plus, le marquage fluorescent différentiel des populations cellulaires permet à la microscopie time-lapse de définir l’assemblage de cultures 3D et la dynamique d’invasion.
Notamment, l’ajout de deux combinaisons de cellules stromales aux cultures 3D de cellules BT-474 réduit considérablement la circularité des cultures 3D, ce qui correspond à la présence de structures sphéroïdes de type organoïde ou secondaire. Dans les expériences de colorant tracker, les fibroblastes et les cellules endothéliales co-localisent dans les protubérances périphériques de type organoïde et sont séparés spatialement du sphéroïde primaire BT-474. Enfin, les cultures 3D hétéromulticellulaires de cellules BT-474 ont augmenté la capacité d’invasion de l’hydrogel. Comme nous avons observé ces structures protrusives dans des cultures 3D hétéromulticellulaires de cellules épithéliales mammaires non tumorigènes et tumorigènes, ce travail fournit une méthode efficace et reproductible pour générer des cultures 3D de type organoïde dans un environnement sans échafaudage pour des analyses ultérieures des phénotypes associés à la progression tumorale solide.
Introduction
Il est maintenant reconnu que la progression du cancer dépend de deux facteurs majeurs : les changements génétiques/épigénétiques dans les cellules tumorales et une myriade d’interactions avec des cellules non tumorales dans le microenvironnement tumoral (TME)1. Bien que les modifications génétiques dans les cellules soient reconnues comme nécessaires à l’initiation de la tumeur, ces modifications seules ne sont pas suffisantes pour la progression tumorale et les métastases2. Les composants du TME, que l’on pensait à l’origine être des spectateurs silencieux, sont maintenant connus pour favoriser activement la progression du cancer via une diaphonie mutuelle et dynamique avec les cellules tumorales3. La composition des EUT diffère en fonction du tissu d’où provient la tumeur, du stade de la tumeur et des caractéristiques du patient, mais les caractéristiques caractéristiques comprennent les fibroblastes associés au cancer stromal (CAF), la matrice extracellulaire (ECM), les cellules endothéliales vasculaires, ainsi que les cellules immunitaires adaptatives et myéloïdes 1,4.
Les CAF stromales dans l’EUT sont composées de sous-types de fibroblastes d’origines et de fonctions diverses5. Ces CAF sont des composants clés de l’EUT car ils interagissent avec les cellules tumorales à plusieurs interfaces. Les CAF sécrètent des protéines ECM qui modifient la rigidité de la matrice, ce qui peut soit limiter l’administration de médicaments par un dépôt excessif de collagène, de protéoglycanes et de fibronectine, soit permettre aux cellules tumorales d’envahir le site tumoral primaire via la sécrétion de métalloprotéinases matricielles (MMP) dégradant la MEC6,7. De plus, les CAF favorisent la croissance, la migration et la vascularisation tumorales via la sécrétion d’une variété de facteurs de croissance, de cytokines et de facteurs angiogéniques tels que le facteur de croissance épidermique (EGF), le facteur de croissance transformant β (TGF-β) et le facteur de croissance de l’endothélium vasculaire (VEGF), respectivement 1,6. En parallèle, les cellules endothéliales, stimulées par l’EUT hypoxique, favorisent également la vascularisation tumorale et suppriment les fonctions des cellules immunitaires via l’augmentation de la sécrétion de facteurs angiogéniques et la diminution de la sécrétion de molécules d’adhésion leucocytaire 1,8.
Compte tenu de la complexité apparente de la progression du cancer, il est devenu essentiel d’intégrer les composantes stromales de l’EUT dans la recherche fondamentale sur le cancer. Cependant, la mise en place de modèles qui récapitulent fidèlement la physiopathologie tumorale connue reste un besoin non satisfait important 9,10. Alors que les modèles de culture cellulaire bidimensionnels (2D) traditionnels sont faciles à manipuler, rapidement cultivés et hautement reproductibles, ils ne sont composés que de clones de cellules cancéreuses à prolifération rapide et ne reflètent pas l’hétérogénéité cellulaire trouvée dans les tumeurs 10,11,12. De la même manière, les modèles de souris transgéniques ne capturent pas non plus la biologie des tumeurs humaines en raison de la faible hétérogénéité génétique due à la consanguinité, des différences significatives dans le système immunitaire et de la complexité histologique13,14. En raison de ces limites, les thérapies développées à partir de modèles classiques de cancer ne parviennent souvent pas à être appliquées en milieu clinique.
Les modèles de cancer dérivés de patients, tels que les xénogreffes dérivées de patients et les organoïdes dérivés de patients, peuvent remédier aux inconvénients des modèles de cancer conventionnels en capturant les caractéristiques moléculaires de la tumeur in situ, le patrimoine génétique et l’organisation cellulaire 10,11,15. Cependant, ces xénogreffes et organoïdes dérivés de patients nécessitent des procédures de greffe compliquées et un long temps de culture16,17. Combiné à la variation des sites d’acquisition et d’échantillonnage des tumeurs et à la faible efficacité de la cryoconservation, il est nécessaire de développer des modèles qui agissent comme un pont entre les cultures cellulaires 2D classiques et les modèles de cancer dérivés de patients11,18. À cet égard, les modèles 3D de culture cellulaire peuvent servir de modèles qui peuvent être cultivés rapidement et capturer des caractéristiques tumorales in vivo importantes telles que l’interaction cellule-cellule, l’interaction cellule-ECM, l’hypoxie, l’angiogenèse et la production d’ECM19,20.
Les modèles de culture cellulaire 3D sont classés en systèmes de modèles sans échafaudage et en systèmes basés sur des échafaudages. Dans les systèmes sans échafaudage, les cellules sont amenées à s’auto-agréger en une forme sphérique en utilisant des plaques de culture cellulaire spécifiques à faible attachement ou en manipulant les paramètres physiques des méthodes de culture. Les méthodes établies pour obtenir des sphéroïdes 3D sans échafaudage vont de la simple culture de granulés cellulaires par centrifugation aux chutes de microplaques suspendues, à la lévitation magnétique et aux bioréacteurs dynamiques et aux systèmes microfluidiques20,21. Les cultures cellulaires 3D basées sur des échafaudages sont établies par l’ajout d’échafaudages à base de polymères ou d’hydrogels pour imiter la matrice extracellulaire physiologique19,22. De tels modèles recèlent un immense potentiel pour modéliser l’organisation cellulaire in vivo, la topologie, l’attachement à la matrice, la migration et la réponse aux médicaments.
En plus de la manipulation d’échafaudages pour les modèles de composition ECM dans les états pathologiques, les cultures cellulaires 3D peuvent également être utilisées pour modéliser des populations cellulaires hétérogènes dans un TME. Des cultures cellulaires 3D composées de cellules cancéreuses et de fibroblastes stromaux ou de cellules endothéliales ont été utilisées pour étudier l’interaction entre le cancer et les lignées cellulaires individuelles non tumorales 23,24,25. Des méthodes reproductibles et rentables pour étendre de telles cultures cellulaires 3D composées de plusieurs lignées cellulaires hétérogènes aideraient les chercheurs à élucider la progression tumorale. Ici, nous rapportons des méthodes de culture de cultures 3D multicellulaires épithéliales-stromales pour étudier la prolifération, l’invasion et la plasticité de l’état cellulaire. Le protocole décrit des cultures 3D de cellules cancéreuses du sein basées sur des échafaudages sans échafaudage et à l’extrait de membrane basale, co-cultivées avec une combinaison de cellules stromales allant des fibroblastes (BJ-5ta), des cellules endothéliales (Ea.hy926) et des cellules de type monocytes (THP-1). Le cancer du sein est actuellement le deuxième cancer le plus fréquent dans le monde et le cancer le plus diagnostiqué chez les femmes aux États-Unis26. La mortalité par cancer du sein est en grande partie due à la nature métastatique et résistante au traitement de la maladie, car la survie globale et sans métastases est considérablement réduite chez les patientes diagnostiquées avec des sous-types de cancer du sein agressifs enrichis en HER2 et de type basal27. Nos protocoles de culture cellulaire 3D décrits peuvent aider à développer des méthodes de culture rentables, rapides et reproductibles qui peuvent être associées à des méthodes de préservation des tissus fixées au formol et à des applications ultérieures de biologie spatiale.
Protocole
1. Milieu de culture cellulaire
REMARQUE : Préparez tous les milieux à l’intérieur d’une enceinte de biosécurité.
- Pour préparer le milieu de culture cellulaire pour BJ-5ta, BT474, EA.hy926 et MDA-MB-468, complétez 500 ml de milieu à haute teneur en glucose Modified Eagle Medium (DMEM) de Dulbecco avec 10 % de sérum de veau fœtal inactivé par la chaleur (FBS) et 1 % de pénicilline-streptomycine à l’aide d’une pipette. Incorporer 0,1 % de gentamicine à l’aide d’une micropipette.
- Pour préparer le milieu de culture cellulaire pour MCF10A, compléter 500 mL de DMEM/F12 avec 5 % de sérum de cheval et 1 % de pénicilline-streptomycine à l’aide d’une pipette. Incorporer 0,1 % de gentamicine, 1 mL d’hydrocortisone de 1 μg/mL, 500 μL d’insuline de 10 μg/mL, 50 μL de toxine cholérique de 100 ng/mL et 10 μL de facteur de croissance épidermique de 20 ng/mL à l’aide d’une micropipette.
- Pour préparer le milieu de culture cellulaire pour MCF10Ca1h, compléter à l’aide d’une pipette 500 mL de DMEM/F12 avec 5 % de sérum de cheval et 1 % de pénicilline-streptomycine. Incorporer 0,1 % de gentamicine à l’aide d’une micropipette.
- Pour préparer le milieu de culture cellulaire pour THP-1, compléter à l’aide d’une pipette 500 mL de RPMI 1640 avec 10 % de FBS et 1 % de pénicilline-streptomycine. Incorporer 0,1 % de gentamicine à l’aide d’une micropipette.
- Changez le milieu de culture cellulaire tous les 2-3 jours pour BJ-5ta, BT474, EA.hy926, MDA-MB-468, MCF10A et MCF10Ca1h jusqu’à ce que les cellules atteignent 70-80 % de confluence en évaluant leur croissance quotidiennement avec un microscope à fond clair. Changez le milieu de culture cellulaire une fois par semaine pour les cellules THP-1.
- Cultivez des cellules BJ-5ta, BT474, EA.hy926, MDA-MB-468, MCF10A et MCF10Ca1h dans des boîtes de 100 mm avec des surfaces traitées pour culture cellulaire dans des incubateurs de culture cellulaire standard avec 5 % de CO2.
- Cultivez des cellules THP-1 dans des flacons traités pour culture cellulaire T75 dans des incubateurs de culture cellulaire standard avec 5 % de CO2.
2. Collecte de cellules
- Allumez la lumière UV pour désinfecter l’intérieur de l’enceinte de biosécurité pendant 15 min.
- Ouvrez le châssis de la fenêtre de l’enceinte de biosécurité pour stabiliser le flux d’air et allumez le système d’aspiration sous vide.
- Nettoyez la surface intérieure de la hotte et la tubulure du système d’aspiration sous vide avec de l’éthanol à 70 %.
- Préparez un milieu de culture cellulaire frais sans sérum à l’intérieur de l’enceinte de sécurité biologique : à l’aide d’une pipette, ajoutez 500 ml de milieu modifié à haute teneur en glucose (DMEM) de Dulbecco avec 1 % de pénicilline/streptomycine. Incorporer 0,1 % de gentamicine à l’aide d’une micropipette.
- Réchauffez le milieu de culture cellulaire, la solution saline tamponnée au phosphate (PBS) et la trypsine-EDTA (0,25 %) à 37 °C en plaçant les éléments dans un bain de billes avant de commencer l’expérience.
- Assurez-vous que les cellules sont confluentes à 70-80 % grâce à une inspection visuelle au microscope.
- Aspirez et jetez le milieu de culture des cellules plaquées avec l’aspirateur sous vide. Lavez une fois le milieu restant avec 2 ml de PBS à l’aide d’une pipette, puis aspirez et jetez le PBS avec l’aspirateur.
REMARQUE : Les cellules THP-1 sont cultivées en suspension ; Les étapes 2.7 à 2.9 peuvent être omises. - Ajouter 1 mL de trypsine dans la boîte de culture cellulaire à l’aide d’une micropipette et placer la plaque dans un incubateur à 5 % de CO2 à 37 °C pendant 5 min
- Inactiver la trypsine en ajoutant 1 mL de l’inhibiteur de la trypsine de soja (dans 1x PBS) dans la plaque à l’aide d’une micropipette. Dispersez les grappes de cellules en pipetant le mélange liquide à l’aide d’une micropipette P1000 et collectez la suspension cellulaire au bas de la plaque. Transvaser la suspension cellulaire à l’aide d’une micropipette dans un tube conique de 15 mL et centrifuger à 100 × g pendant 5 min à température ambiante. Jetez le surnageant avec l’aspirateur.
- Passez au comptage des cellules.
3. Préparation de la solution de colorant de travail et des cellules de coloration en suspension
- Avant d’ouvrir le flacon des sondes de fluorescence moléculaire (voir le tableau des matériaux), laissez le produit se réchauffer à température ambiante pendant 15 min dans un bain de billes réglé à 37 °C.
- Dissoudre le colorant bleu lyophilisé (masse = 5 mg, poids moléculaire = 209,6 g/mol) à une concentration finale de 10 mM avec 2,385 mL de DMSO à l’aide d’une micropipette.
- Dissoudre le colorant orange lyophilisé cell tracker (masse = 50 μg, poids moléculaire = 550,4 g/mol) à une concentration finale de 10 mM avec 9,084 μL de DMSO à l’aide d’une micropipette.
- Dissoudre le colorant lyophilisé cell tracker deep red (masse = 15 μg, poids moléculaire = 698,3 g/mol) à une concentration finale de 1 mM avec 20 μL de DMSO à l’aide d’une micropipette.
- Préparez la solution de colorant bleu pour traqueur de cellules de travail (5 μM) en diluant 1 μL de colorant dans 2 ml de milieu DMEM sans sérum à l’aide d’une micropipette.
- Préparez la solution de colorant orange (5 μM) en diluant 1 μL de colorant dans 2 ml de milieu DMEM sans sérum à l’aide d’une micropipette.
- Préparez la solution de colorant rouge foncé (1 μM) en diluant 2 μL de colorant dans 2 ml de milieu DMEM sans sérum à l’aide d’une micropipette.
- Remettre en suspension les cellules épithéliales, les fibroblastes BJ-5ta et les cellules endothéliales Ea.hy926 dans les solutions de colorant bleu, orange et rouge foncé préparées (2 ml), respectivement à l’aide d’une micropipette.
- Incuber les tubes à 37 °C dans l’incubateur à 5 % de CO2 pendant 30 min.
- Après 30 min d’incubation, centrifuger les tubes à 100 x g pendant 5 min à température ambiante.
- Aspirez et jetez le surnageant à l’aide de l’aspirateur et remettez complètement la pastille en suspension dans 1 mL de milieu DMEM contenant du sérum FBS à 10 % à l’aide d’une micropipette.
- Passez au comptage des cellules.
4. Comptage des cellules
- Prélever 10 μL de la suspension cellulaire et la transférer dans un microtube à l’aide d’une micropipette.
- Mélanger avec 10 μL de bleu de trypan et pipeter soigneusement.
- Transférez 20 μL de la solution de trypan cellulaire à l’aide d’une micropipette sur une lame de chambre de comptage cellulaire. Insérez et comptez les cellules à l’aide d’un compteur de cellules automatisé.
- Calculez le nombre total moyen de cellules vivantes à partir de deux lectures.
5. Calculs
- Préparez des stocks de cellules de travail pour chaque type de cellule à une concentration de 6,67 × 103 cellules/mL, ce qui équivaut à 2 000 cellules/300 μL.
REMARQUE : Le volume total du stock dépendra de la taille de l’échantillon expérimental. - Pour les sphéroïdes en monoculture, assurez-vous que chaque échantillon indépendant est constitué de 300 μL de la substance de travail des cellules épithéliales, soit l’équivalent de 2 000 cellules épithéliales.
- Pour les sphéroïdes en co-culture (deux types de cellules), assurez-vous que chaque échantillon indépendant est composé de 150 μL de cellules épithéliales et de 150 μL de cellules stromales. L’échantillon contiendra 1 000 cellules épithéliales et 1 000 cellules stromales.
- Pour les sphéroïdes en co-culture (trois types de cellules), assurez-vous que chaque échantillon indépendant est composé de 150 μL de cellule épithéliale, de 75 μL de cellule stromale #1 et de 75 μL de cellule stromale #2. L’échantillon contiendra 1 000 cellules épithéliales, 500 cellules stromales de stock de travail #1 et 500 cellules stromales de stock de travail #2.
6. Placage
- Transférez le volume requis à l’aide d’une micropipette pour trois réplicats techniques, plus une supplémentaire, dans un microtube. Mélangez soigneusement à l’aide d’une pipette, puis transférez 300 μL de l’échantillon dans un puits d’une microplaque de 96 puits à fond en U et à fixation ultra-basse.
- Répétez le processus pour chaque réplique technique supplémentaire.
- Placez la plaque à 96 puits dans un incubateur à 37 °C.
7. Imagerie en fond clair
- Observez la croissance et la morphologie des sphéroïdes toutes les 24 h jusqu’à 96 h à l’aide d’un microscope.
- Imagez les sphéroïdes à l’aide d’un microscope à contraste de phase.
8. Configuration du protocole d’imagerie à grand champ pour les sphéroïdes colorés avec des colorants de traqueur et les sphéroïdes recouverts d’extrait de membrane basale
- Allumez les appareils d’imagerie et d’incubation automatisés répertoriés dans la Table des matières, puis créez un nouveau protocole d’imagerie dans le gestionnaire de tâches du logiciel d’imagerie. Sous l’onglet Procédure , réglez le point de consigne de température de l’incubateur automatisé sur 37 °C et laissez l’incubateur équilibrer le CO2 % et la température avant de passer à l’étape suivante.
REMARQUE : Un autre imageur fluorescent peut être utilisé s’il dispose des filtres appropriés pour chacun des colorants (bleu, rouge foncé et orange) utilisés. - Réglez les paramètres de l’image sur les spécifications suivantes : Grossissement : 4X PL FL Phase, Champ de vision : 3185 x 3185 μm, WFOV complet.
- Utilisez les spécifications suivantes pour les canaux : DAPI : 377/447 nm, éclairage = 10, temps d’intégration = 107 ms, gain = 10, RFP : 531/593 nm, éclairage = 10, temps d’intégration = 137 ms, gain = 10, CY5 : 628/685 nm, éclairage = 10, temps d’intégration = 137 ms, gain = 10.
- Pour imager les sphéroïdes dans une superposition de solution d’extrait de membrane basale, utilisez les spécifications de fond clair suivantes : éclairage = 10, temps d’intégration = 5 ms et gain = 17,1.
- Sélectionnez les puits souhaités pour l’imagerie et approuvez les modifications de spécification en cliquant sur l’icône de sélection des puits .
- Accédez à l’onglet Réduction des données pour ajuster les paramètres d’analyse cellulaire.
- Définissez la valeur de seuil sur 19 500 avec un arrière-plan clair, puis sélectionnez Remplir les trous dans les masques.
- Pour la sélection d’objets, définissez la taille minimale de l’objet sur 100 μm et la taille maximale de l’objet sur 1 000 μm, puis sélectionnez Analyser l’image entière.
REMARQUE : Seuls un masque primaire et un nombre d’objets sont nécessaires pour cette analyse. - Enregistrez toutes les modifications et ouvrez l’application d’imagerie.
- Placez la microplaque expérimentale à fond en forme de U dans l’incubateur en ouvrant le tiroir, puis fermez-la à l’aide du logiciel d’imagerie.
- Pour exécuter le protocole, cliquez sur l’onglet Infos sur la procédure, ajoutez un utilisateur et choisissez le protocole.
- Assurez-vous que le type de plaque approprié est sélectionné et réglez la durée d’imagerie sur 30 min par plaque.
- Sélectionnez l’intervalle d’imagerie souhaité, indiquez si la plaque est équipée d’un couvercle et ajustez l’heure et la durée de début de l’imagerie.
- Cliquez sur Schedule Plate/Vessel pour démarrer le processus d’imagerie.
9. Rechargement de la solution d’extraction de la membrane basale (facultatif)
REMARQUE : La solution d’extrait de membrane basale peut être appliquée sur les sphéroïdes 24 heures après le placage.
- Remplissez un seau à glace de glace pour garder la solution d’extraction de la membrane du sous-sol froide et conservez-la à 4 °C lorsqu’elle n’est pas utilisée.
- Aspirez environ 170 μL de fluide à l’aide d’une pipette multicanaux.
- À l’aide d’une loupe et d’une mini boîte lumineuse, observez de près les petits sphéroïdes. Placez la plaque sphéroïde à 96 puits sur la boîte lumineuse et positionnez la loupe au-dessus de votre tête.
- Réglez la pipette P200 à 30 μL et collectez l’extrait de la membrane basale pour créer trois microgouttelettes.
- Ajoutez une microgouttelette sur chaque sphéroïde des trois réplicats techniques à l’aide d’une micropipette.
- Assurez-vous que la plaque à 96 puits est plate et positionnez la pipette verticalement au-dessus du sphéroïde. Libérez la gouttelette sans toucher le fond du puits.
- Placez la plaque dans un incubateur à 37 °C pendant 20 min.
- Recouvrir les sphéroïdes avec 50 μL supplémentaires de la solution d’extraction de la membrane basale par puits à l’aide d’une micropipette.
- Repositionnez les sphéroïdes au centre du puits à l’aide d’une pointe de pipette.
- Incuber à 37 °C pendant 30 min.
- Ajouter 100 μL de milieu de culture cellulaire dans chaque puits.
10. Quantification
- Téléchargez et ouvrez le logiciel ImageJ. Téléchargez les images sphéroïdes.
- Définissez les mesures en cliquant sur Analyser | Définissez les mesures et sélectionnez Zone | Centroïde. Mesurez les sphéroïdes à l’aide de l’outil lasso pour tracer le sphéroïde et mesurer l’objet.
- Enregistrez les valeurs de trois répétitions biologiques et calculez la surface moyenne et la circularité.
- Convertissez la surface de pixels en mètres carrés à l’aide de la formule suivante :
Aire (m2) = ((550/504) × √ (aire en pixels)) 2 - Analysez les données.
- Sélectionnez Entrer des valeurs de réplication, empilées dans des colonnes.
- Entrez les données dans les colonnes et sélectionnez les échantillons de sphéroïdes de contrôle en monoculture et de traitement en coculture.
- Effectuez une analyse de colonne ANOVA unidirectionnelle.
- Utilisez les paramètres ANOVA suivants :
- Évitez d’assortir ou d’apparier.
- Supposons une distribution gaussienne des résidus.
- Supposons des écarts-types égaux.
- Utilisez le test de comparaison multiple de Tukey pour comparer la moyenne de chaque colonne avec la moyenne de toutes les autres colonnes.
11. Traitement d’images immunofluorescentes à grand champ
- Enregistrez les images directement à partir du logiciel d’imagerie au format de fichier PNG ou Tiff.
- Sélectionnez chaque canal individuel ou les canaux qui se chevauchent et enregistrez-les au format PNG.
- Ajustez la luminosité et le contraste dans le logiciel de l’imageur ou dans ImageJ si les images sont exportées sous forme de fichiers Tiff.
Résultats
Dans cette étude, nous avons développé un système de culture cellulaire pour générer des sphéroïdes 3D hétéromulticellulaires composés de cellules épithéliales et stromales avec une morphologie de type organoïde. Les sphéroïdes ont été établis en plaquant 2 000 cellules épithéliales dans des conditions de monoculture. Dans des conditions de co-culture de deux types de cellules, les sphéroïdes ont été établis en plaquant 1 000 cellules épithéliales et 1 000 cellules stromales. Dans des conditions de co-culture de trois types de cellules, les sphéroïdes ont été établis en plaquant 1 000 cellules épithéliales et deux types de cellules stromales différentes de 500 cellules de chaque type de cellule. En amont de l’établissement des sphéroïdes, les cellules peuvent être colorées avec des colorants fluorescents qui permettent de surveiller l’organisation spatiale cellulaire. Après 24 h de formation initiale de sphéroïdes, les applications en aval comprennent les perturbations pharmacologiques, l’imagerie et la collecte d’échantillons. L’imagerie en accéléré est utile pour évaluer les changements dans le comportement et la morphologie des sphéroïdes, y compris l’aire et la circularité (Figure 1A). À 24 heures après le placage, les sphéroïdes peuvent être intégrés dans un environnement d’échafaudage, et l’imagerie en accéléré peut être utilisée pour évaluer l’apparition de structures invasives à partir du sphéroïde (figure 1B). La collecte d’échantillons de sphéroïdes hétéromulticellulaires a de nombreuses applications, notamment le profilage génomique et protéomique aux niveaux mondial et unicellulaire grâce à des techniques expérimentales telles que le séquençage de l’ARN, le séquençage de l’ARN unicellulaire, le séquençage protéomique et l’immunofluorescence cyclique.
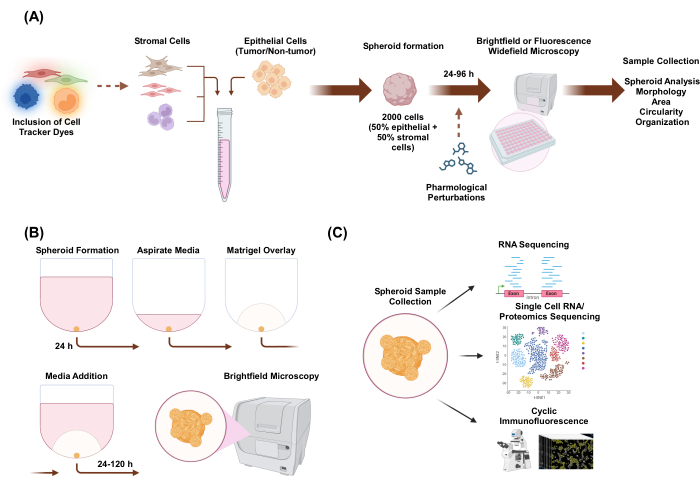
Figure 1 : Représentation schématique du processus de culture cellulaire 3D et des applications potentielles. (A) La suspension cellulaire de cellules épithéliales avec des cellules stromales non épithéliales est pipetée dans des plaques de fixation 3D ultra-basses pour former des sphéroïdes. Les sphéroïdes sont imagés par microscopie à fond clair toutes les 24 h pendant 96 h. Les lignées cellulaires peuvent être colorées avec des colorants fluorescents pour la microscopie à champ large en aval avant d’induire la formation de sphéroïdes ou peuvent être perturbées pharmacologiquement après la formation de sphéroïdes. Les paramètres sphéroïdes tels que la morphologie, la surface, la circularité et l’organisation peuvent être analysés. (B) Les sphéroïdes des cellules épithéliales et épithéliales avec des cellules stromales sont formés à l’aide du protocole de A. Les sphéroïdes ont été établis en plaquant 2 000 cellules épithéliales dans des conditions de monoculture. Dans des conditions de co-culture de deux types de cellules, les sphéroïdes ont été établis en plaquant 1 000 cellules épithéliales et 1 000 cellules stromales. Dans des conditions de co-culture de trois types de cellules, les sphéroïdes ont été établis en plaquant 1 000 cellules épithéliales et deux types de cellules stromales différentes de 500 cellules de chaque type de cellule. Après 24 h, une solution d’extrait de membrane basale semblable à un échafaudage est superposée et les images sont capturées en microscopie à fond clair toutes les 24 h pendant 120 h. (C) Les cultures 3D hétéro-multicellulaires établies sans échafaudage et basées sur un échafaudage peuvent être utilisées pour une variété d’applications en aval telles que l’immunofluorescence cyclique, le séquençage de l’ARN unicellulaire et la protéomique unicellulaire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les sphéroïdes de monoculture MCF10A, MCF10Ca1h et BT-474 conservent un phénotype sphérique compact jusqu’à 96 h après le placage. Lorsqu’ils sont co-cultivés avec des cellules endothéliales microvasculaires EA.hy926, des fibroblastes BJ-5ta et/ou des cellules monocytaires THP-1, les sphéroïdes ont développé des protubérances cellulaires à la périphérie, qui sont devenues plus prononcées à 96 h (Figure 2A-C). Il est important de noter que ces phénomènes de protubérance, de bourgeonnement et de compactage des sphéroïdes représentent des variations de l’organisation cellulaire du sphéroïde cancéreux et des cellules stromales co-cultivées qui sont corrélées avec l’invasivité 3D après l’ajout d’un hydrogel à base de membrane basale comme Matrigel. La morphologie bourgeonnante variait d’agrégats de cellules solides à des agrégats de cellules lâches, ressemblant à la morphologie des organoïdes. En revanche, les sphéroïdes de monoculture MDA-MB-468 sont apparus sous forme de gros agrégats de cellules lâches. Cependant, lorsque les cellules MDA-MB-468 ont été co-cultivées avec EA.hy926, BJ-5ta et/ou THP-1, elles ont formé des sphéroïdes compacts (Figure 2D).
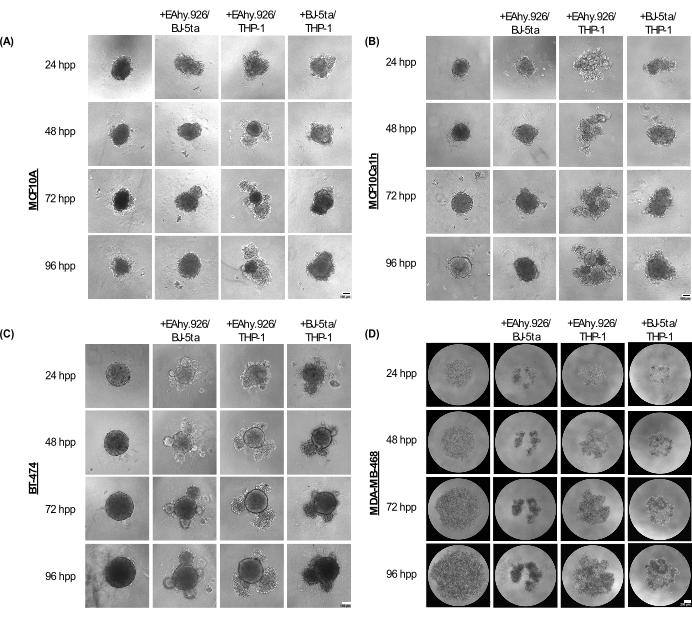
Figure 2 : La gamme d’architectures et de morphologies adoptées par des combinaisons de cellules stromales non épithéliales lorsqu’elles sont combinées avec des cellules tumorales/non tumorales épithéliales dans des cultures 3D sans échafaudage. Images représentatives en fond clair de (A) MCF10A, (B) MCF10Ca1h, (C) BT-474, (D) sphéroïdes MDA-MB-468 en monoculture ou en co-culture avec des fibroblastes stromaux BJ-5ta/cellules endothéliales microvasculaires Ea.hy926 ou des cellules monocytaires de type Ea.hy926/THP-1 ou des cellules BJ-5ta/THP-1 sur 96 h. Chaque sphéroïde a été formé en plaquant 2 000 cellules. Les sphéroïdes en monoculture ont été formés à l’aide de 2 000 cellules épithéliales. Les sphéroïdes en co-culture ont été formés à l’aide de 1 000 cellules épithéliales et de deux types de cellules stromales différentes de 500 cellules. La formation de structures bourgeonnantes ou agrégées de type organoïde a été initiée dans des conditions de co-culture sphéroïde 24 h après le placage dans les cellules épithéliales MCF10A, MCF10Ca1h et BT-474. Le compactage des cellules MDA-MB-468 a été observé dans des conditions de co-culture sphéroïde 25 h après le placage. Barres d’échelle = 100 μm. Abréviation : hpp = heures après le placage. Veuillez cliquer ici pour voir une version agrandie de cette figure.
72 h après le placage, les cellules MCF10A, MCF10Ca1h et BT-474 co-cultivées avec EA.hy926 et THP-1, ou avec BJ-5ta et THP-1, ont montré une augmentation significative de la surface des sphéroïdes épithéliales par rapport aux sphéroïdes épithéliales en monoculture. MCF10Ca1h a également montré une augmentation significative de la surface des sphéroïdes lorsqu’il est co-cultivé avec EA.hy926 et BJ-5ta. L’apparition de structures bourgeonnantes chez les sphéroïdes co-cultivés a conduit à une diminution significative de la circularité des sphéroïdes pour MCF10A, MCF10Ca1h et BT-474 co-cultivés avec EA.hy926 et THP-1, ou avec BJ-5ta et THP-1. Des effets similaires ont été observés pour le BT-474 co-cultivé avec EA.hy926 et BJ-5ta (figures 3A,B). En revanche, les cellules MDA-MB-468 co-cultivées avec EA.hy926 et BJ-5ta, EA.hy926 et THP-1, ou BJ-5ta et THP-1, ont montré une diminution significative de la surface des sphéroïdes par rapport aux sphéroïdes MDA-MB-468 en monoculture ; pourtant, il n’y a pas eu d’effet sur la circularité (Figure 3A,B).

Figure 3 : Graphiques d’analyse de zone et de circularité de cultures stromales 3D sans échafaudage mono-cultivées et hétéromulticellulaires 72 h après le placage. (A) Aire moyenne (cm 2) et (B) circularité moyenne de MCF10A, MCF10Ca1h, BT-474 et MDA-MB-468 dans des cultures de sphéroïdes stromaux mono-cultivés et hétéromulticellulaires 72 h après le placage. Les données rapportées sont représentatives d’au moins trois répétitions biologiques indépendantes et sont rapportées sous forme de moyennes techniques de répétitions ± MEB, sauf indication contraire. *, **, *** ou **** représentent des valeurs p < 0,05, 0,01, 0,001 ou 0,0001, respectivement, sauf indication contraire. Veuillez cliquer ici pour voir une version agrandie de cette figure.
L’application d’un colorant de suivi cellulaire sur les cellules épithéliales tumorigènes BT-474 et les cellules stromales avant l’établissement du sphéroïde a démontré que les cellules stromales, y compris EA.hy926 et BJ-5ta, formaient les structures bourgeonnantes au périmètre des sphéroïdes centraux BT474 (Figure 4, Vidéo supplémentaire S1, Vidéo supplémentaire S2, Vidéo supplémentaire S3 et Vidéo supplémentaire S4). 48 h après le placage, des images individuelles de fluorescence à grand champ de sphéroïdes co-cultivés avec des fibroblastes révèlent que les sphéroïdes de fibroblastes se sont co-localisés avec les cellules endothéliales, mais n’ont pas co-localisé avec les sphéroïdes BT-474. On a également constaté qu’une minorité de cellules endothéliales co-localisaient avec les sphéroïdes BT-474 dans des conditions de co-culture. Cela suggère que la disposition des cellules stromales à l’intérieur du sphéroïde est corrélée à une morphologie de type organoïde.
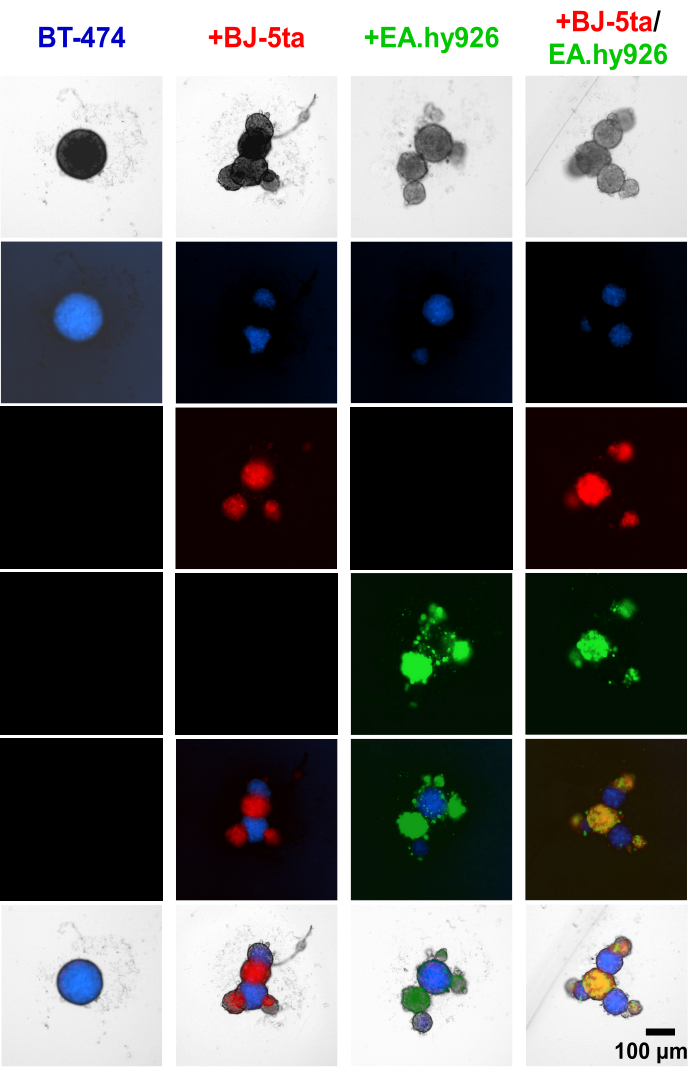
Figure 4 : Images fixes en fluorescence à grand champ de cellules stromales et BT-474 teintées différentiellement dans des cultures 3D hétéromulticellulaires à 48 h après le placage. Les sphéroïdes BT-474 sont colorés avec un colorant fluorescent blue cell tracker. Les fibroblastes BJ-5ta sont colorés avec un colorant de traqueur de cellules orange, représenté en rouge. Les cellules endothéliales Ea.hy926 sont colorées avec un colorant de suivi des globules rouges profonds, représenté en vert. Chaque sphéroïde a été formé en plaquant 2 000 cellules. Les sphéroïdes en monoculture ont été formés à l’aide de 2 000 cellules épithéliales. Les sphéroïdes en co-culture (BT-474/BJ-5ta, BT-474/Ea.hy926) ont été formés à l’aide de 1 000 cellules épithéliales et de 1 000 cellules stromales. Des sphéroïdes en double co-culture (BT-474/BJ-5ta/Ea.hy926) ont été formés à l’aide de 1 000 cellules épithéliales et de 500 cellules de chaque type de cellules stromales. Les figures sont représentatives d’au moins trois réplicats biologiques. Barre d’échelle = 100 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Pour évaluer la pertinence biologique de notre modèle de sphéroïde organoïde, les sphéroïdes ont été recouverts d’une solution d’extrait de membrane basale 24 h après le placage. Les sphéroïdes BT-474 en monoculture n’ont montré aucune propriété invasive 120 h après le placage. Cependant, les sphéroïdes BT-474 co-cultivés avec BJ-5ta ou EA.hy926 ont développé des structures à la périphérie du sphéroïde, qui ont envahi l’environnement de la solution d’extraction de membrane basale de l’échafaudage. Le nombre et la longueur de ces protubérances ont été considérablement augmentés chez les sphéroïdes BT-474 co-cultivés avec BJ-5ta et EA.hy926 de 48 h à 120 h après le placage (figure 5, vidéo supplémentaire S5, vidéo supplémentaire S6, vidéo supplémentaire S7 et vidéo supplémentaire S8).
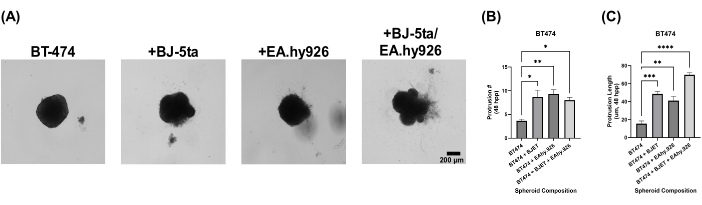
Figure 5 : Images fixes en fond clair de cellules BT-474 dans des cultures 3D hétéromulticellulaires avec superposition d’une solution d’extrait de membrane basale 5 jours après la superposition. Chaque sphéroïde a été formé en plaquant 2 000 cellules. La solution d’extrait de la membrane basale a été recouverte de sphéroïdes 24 h après le placage. Les sphéroïdes en monoculture ont été formés à l’aide de 2 000 cellules épithéliales. Des sphéroïdes en co-culture (BT-474/BJ-5ta et BT-474/Ea.hy926) ont été formés à partir de 1 000 cellules épithéliales et de 1 000 cellules stromales. Des sphéroïdes en double co-culture (BT-474/BJ-5ta/Ea.hy926) ont été formés à l’aide de 1 000 cellules épithéliales et de 500 cellules de chaque type de cellules stromales. (A) Des structures invasives dépassant du sphéroïde cancéreux noyé dans une solution d’extrait de membrane basale peuvent être observées dans des conditions de co-culture. (B) Qualification du nombre de protruations invasives à l’instant 48 hpp. (C) Qualification de la longueur de la saillie invasive au temps 48 hpp. Les figures sont représentatives d’au moins trois réplicats biologiques. Barre d’échelle = 200 μm. Abréviation : hpp = heures après le placage. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vidéo supplémentaire S1 : Sphéroïde de monoculture BT-474 avec colorant bleu de traçage cellulaire. Les cellules BT-474 (2 000) ont été remises en suspension dans une microplaque de 96 puits à fond en U et à fixation ultra-faible pour former des sphéroïdes. Un chevauchement d’images de fluorescence en fond clair et à champ large a été capturé sur 48 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S2 : Sphéroïdes BT-474 co-cultivés avec des fibroblastes BJ-5ta. Les fibroblastes BT-474 (1 000) et 1 000 fibroblastes BJ-5ta ont été remis en suspension dans des plaques ULA pour former des sphéroïdes. Les cellules BT-474 ont été incubées avec un colorant bleu et les fibroblastes BJ-5ta avec un colorant orange Cell Tracker. Un chevauchement d’images de fluorescence en fond clair et en champ large (bleu = BT-474, rouge = BJ-5ta) a été capturé sur 48 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S3 : Sphéroïdes BT-474 co-cultivés avec des cellules endothéliales Ea.hy926. Les cellules endothéliales BT-474 (1 000) et 1 000 Ea.hy926 ont été remises en suspension dans des plaques ULA pour former des sphéroïdes. Les cellules BT-474 ont été incubées avec le colorant bleu Cell Tracker et les cellules endothéliales Ea.hy926 avec le colorant Cell Tracker Deep Red. Un chevauchement d’images fluorescentes en fond clair et en champ large (bleu = BT-474, vert = Ea.hy926) a été capturé sur 48 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S4 : Sphéroïdes BT-474 co-cultivés avec des fibroblastes BJ-5ta et des cellules endothéliales Ea.hy926. BT-474 (1 000), 500 fibroblastes BJ-5ta et 500 cellules endothéliales Ea.hy926 ont été remis en suspension dans des plaques ULA pour former des sphéroïdes. Les cellules BT-474 ont été incubées avec un colorant bleu de traçage cellulaire. Les fibroblastes BJ-5ta et les cellules endothéliales Ea.hy926 ont été incubés avec un colorant orange et un colorant rouge foncé, respectivement. Un chevauchement d’images fluorescentes en fond clair et en champ large (bleu = BT-474, rouge = BJ-5ta, vert = Ea.hy926) a été capturé sur 48 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S5 : Sphéroïde de monoculture BT-474 dans une solution d’extrait de membrane basale. Les cellules BT-474 (2 000) ont été remises en suspension dans des plaques ULA pour former des sphéroïdes, et les sphéroïdes ont été noyés dans une solution de membrane basale 24 heures après le placage. Les images en fond clair ont été prises sur une période de 60 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S6 : Sphéroïdes BT-474 co-cultivés avec des fibroblastes BJ-5ta dans une solution d’extrait de membrane basale. Les fibroblastes BT-474 (1 000) et 1 000 BJ-5ta ont été remis en suspension dans des plaques ULA pour former des sphéroïdes, et les sphéroïdes ont été noyés dans une solution d’extrait de membrane basale 24 heures après le placage. Les images en fond clair ont été prises sur une période de 60 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S7 : Sphéroïdes BT-474 co-cultivés avec des cellules endothéliales Ea.hy926 dans une solution d’extrait de membrane basale. Les cellules endothéliales BT-474 (1 000) et 1 000 Ea.hy926 ont été remises en suspension dans des plaques ULA pour former des sphéroïdes, et les sphéroïdes ont été incorporés dans une solution d’extraction de membrane basale 24 heures après le placage. Les images en fond clair ont été prises sur une période de 60 h. Abréviation : ULA = attachement ultrafaible. Veuillez cliquer ici pour télécharger cette vidéo.
Vidéo supplémentaire S8 : Sphéroïdes BT-474 co-cultivés avec des fibroblastes BJ-5ta et des cellules endothéliales Ea.hy926 dans une solution d’extrait de membrane basale. BT-474 (1 000), 500 fibroblastes BJ-5ta et 500 cellules endothéliales Ea.hy926 ont été remis en suspension dans des plaques ULA pour former des sphéroïdes, et les sphéroïdes ont été incorporés dans une solution d’extraction de membrane basale 24 h après le placage. Les images en fond clair ont été prises sur une période de 60 h. Veuillez cliquer ici pour télécharger cette vidéo.
Discussion
Notre modèle sphéroïde hétéro-multicellulaire démontre que les interactions entre les cellules épithéliales et stromales entraînent le bourgeonnement des cellules stromales dans des conditions de culture 3D sans échafaudage et la formation de structures invasives dans des conditions de culture 3D basées sur des échafaudages. Nous avons observé une formation cohérente de structure bourgeonnante dans les lignées cellulaires tumorigènes (MCF10Ca1h et BT-474) et la lignée cellulaire épithéliale non tumorigène MCF10A (Figure 2A-C). Il est intéressant de noter que si les sphéroïdes des lignées cellulaires MCF10A, MCF10Ca1h et BT-474, que ce soit en monoculture ou en co-culture avec des fibroblastes et des cellules endothéliales, ont formé des structures circulaires et compactes dans des environnements sans échafaudage, les sphéroïdes de la lignée cellulaire MDA-MB-468 ont montré moins de compactage (Figure 2D). C’était inattendu, car les cellules MDA-MB-468 devaient former des sphéroïdes circulaires, comme les autres lignées cellulaires épithéliales testées. La lignée cellulaire SUM-149, une lignée cellulaire primaire hybride épithélio-mésenchymateuse du cancer du sein, forme des sphéroïdes compacts comme MCF10A, MCF10Ca1h et BT-474 (données non présentées)28. Les recherches futures devraient porter sur les voies de signalisation médiant la formation de sphéroïdes moins compacts dans la lignée cellulaire MDA-MB-468, car de telles anomalies peuvent indiquer des vulnérabilités cellulaires qui pourraient être exploitées pour le développement de nouvelles stratégies thérapeutiques.
En ce qui concerne la combinaison de différentes lignées cellulaires, les cellules de type monocyte THP-1 induisent la formation de structures d’agrégats lâches dans toutes les conditions de co-culture de sphéroïdes. On observe moins de compactage dans les structures en bourgeonnement dans des conditions de co-culture avec des cellules THP-1/Ea.hy926 par rapport à celles dans des conditions de co-culture avec des cellules THP-1/BJ-5ta (Figure 2A-D). Par conséquent, le compactage des sphéroïdes est influencé par la composition des cellules stromales dans le modèle29,30.
Un placage incohérent des combinaisons hétérocellulaires peut entraîner un développement incomplet des sphéroïdes ou une variabilité de la taille des sphéroïdes, par rapport à leurs réplicats techniques. Il est crucial d’assurer la cohérence du placage cellulaire, car la disposition spatiale des cellules qui détermine le phénotype et le développement des sphéroïdes se produit principalement dans les 48 premières heures31. Au cours de cette période critique, les rapports cellulaires jouent un rôle crucial, la proportion de cellules stromales affectant à la fois la surface sphéroïde et la circularité.
Les agrégats sphéroïdes en vrac, qui dépendent du type de cellule, peuvent facilement se démonter lors d’une manipulation manuelle. Cependant, une manipulation manuelle est nécessaire lors de la superposition de sphéroïdes avec une solution d’extraction de membrane basale semblable à un échafaudage ou lors de la collecte d’échantillons entiers de sphéroïdes. Pour atténuer ce problème, il est conseillé de travailler à un rythme plus lent lors de la collecte des sphéroïdes par aspiration à pipette et de minimiser la manipulation manuelle dans la mesure du possible. Pendant la superposition de la solution d’extrait de membrane basale, les sphéroïdes ont tendance à se déplacer à l’intérieur du puits, se déposant souvent au bord du puits. Un rapport solution/média d’extrait de membrane basale est important pour maintenir les propriétés matricielles du système 3D ; Cependant, les sphéroïdes ne peuvent pas être complètement enveloppés en raison de la viscosité de la solution d’extrait de la membrane basale, de sorte qu’une intervention manuelle est nécessaire32. Le positionnement non centré des sphéroïdes pose un défi pour l’imagerie, car la réfraction de la lumière près du bord en plastique du puits peut interférer avec la clarté de l’image.
Les sphéroïdes utilisés dans cette étude étaient composés de lignées cellulaires cancéreuses humaines ; Les méthodes décrites ici n’ont pas encore été appliquées aux lignées cellulaires primaires. Nos résultats suggèrent que des interactions cellule-cellule spécifiques peuvent conduire à la formation de structures de type organoïde, indiquant l’applicabilité potentielle de cette méthode à un large éventail de lignées cellulaires tumorigènes. Cependant, alors que notre modèle tente d’imiter la complexité du microenvironnement tumoral, les différents taux de prolifération cellulaire présentent un défi pour les études à long terme33. Les cellules à prolifération rapide ont tendance à dépasser celles qui prolifèrent plus lentement, ce qui rend ce modèle moins adapté aux expériences prolongées. De plus, les différentes exigences en matière de types de milieux de croissance pour les lignées cellulaires peuvent introduire des facteurs supplémentaires qui peuvent conduire à des phénotypes sphéroïdes. La plupart des lignées cellulaires de cette étude ont été cultivées dans des milieux DMEM. Les études avec des lignées cellulaires cultivées dans différents milieux peuvent nécessiter différentes conditions de culture, en testant une variété de ratios de milieux ; Pourtant, cet effet est observé dans l’expérimentation à long terme30.
Notre modèle offre plusieurs avantages par rapport aux autres modèles 3D de cancer. Les sphéroïdes que nous générons ont une morphologie et une taille initiales uniformes et reproductibles, ce qui facilite la comparaison des résultats du traitement avec les témoins. En revanche, certains modèles utilisant des cellules uniques suspendues dans des milieux ou des échafaudages peuvent entraîner des distributions spatiales cellulaires variables, ce qui entraîne des incohérences. Ce modèle démontre que les interactions entre des populations cellulaires distinctes peuvent être étudiées efficacement.
Traditionnellement, les modèles organoïdes et sphéroïdes sont composés de cellules cancéreuses en monoculture ou de co-cultures de cellules cancéreuses pour étudier les gradients de nutriments, l’hypoxie ou l’arrangement cellulaire34. De plus, ce modèle peut être étudié dans un environnement d’échafaudage dans les 24 heures suivant le placage, ce qui permet d’explorer les événements d’invasion précoces induits par diverses interactions cellule-cellule. Alors que les approches alternatives du cancer en 3D utilisent différents composants de matrice d’échafaudage pour étudier les changements de morphologie des sphéroïdes et de caractère invasif, ce modèle les complète en permettant d’étudier ces changements dans le contexte de la dynamique des populations cellulaires et de la composition de l’échafaudage35.
La plupart des sphéroïdes produits à l’aide de nos méthodes conservent leur intégrité structurelle après une manipulation manuelle, ce qui permet la collecte d’échantillons dans les applications en aval. Des modifications génétiques conventionnelles telles que CRISPR-Cas9, la transduction lentivirale de shRNA et l’interférence siRNA peuvent être introduites en amont de la formation des sphéroïdes. Les comportements complexes observés chez ces sphéroïdes suggèrent des altérations dynamiques de l’expression des gènes, qui peuvent être étudiées plus en détail à l’aide du RNA-seq. Des techniques avancées telles que CycIF, scRNA-seq, CosMx et Visium peuvent également être utilisées pour étudier la génomique et la protéomique à des niveaux spatiaux ou unicellulaires. Ce modèle sphéroïde a le potentiel d’imiter les études in vivo de thérapies tumorales ciblées en capturant les interactions entre les bourgeons cellulaires stromaux et les agents thérapeutiques avant qu’ils n’atteignent les cellules cancéreuses. Ceci est important car cela peut aider à déterminer si les cellules stromales influencent la puissance du médicament, protégeant potentiellement les cellules cancéreuses des effets thérapeutiques.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Nous remercions les membres du Laboratoire d’oncogène du développement de l’Université Baylor pour leurs remarques et commentaires utiles lors de la préparation de ce manuscrit. Le soutien financier a été fourni par le Département de biologie de l’Université Baylor et le Collège des arts et des sciences, NIH-NIGMS 2SC1GM121182 (à J.A.K.).
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL Polypropylene Centrifuge Tubes | Falcon | 14-959-53A | |
| 96-Well, Treated, U-Shaped-Bottom Microplate | Thermo Scientific | 12566432 | |
| 100 mm Dish Cell Culture Treated Surface | Thermo Scientific | 130182 | |
| 170 L CO2 Incubators | VWR | VWR51014991(10810902) | |
| 200 Self-Sealing Barrier Pipet Tips | Fisher Scientific | 02-682-255 | |
| 500 mL DMEM/Hams F-12, [+] L-glutamine | Corning | 10-090-CV | |
| 1000 Self-Sealing Barrier Pipet Tips | Molecular Biology Products Inc. | 2179HR | |
| Animal-Free Recombinant Human EGF | PeproTech | AF-100-15 | |
| Automated Pipette Repeater E3x | Eppendorf | 4987000410 | |
| Basement membrane solution | Huntsman Cancer Institute | https://healthcare.utah.edu/huntsmancancerinstitute/ | |
| Bead Bath | Lab Armor | 74300706 | |
| Biorender | Biorender | Biorender.com | |
| Biosafety Laminar Flow Cabinets | Thermo Scientific | 300590389 | |
| BioSpa 8 Automated Incubator | Agilent | 23082120 | |
| Cell Counter | Invitrogen | AMQAF2000 | |
| Cell Counting Chamber Slides | Thermo Scientific | C10283 | |
| CellTracker Deep Red | Thermo Fisher Scientific | C34565 | |
| Centrifuge | Thermo Scientific | 13100675 | |
| Cholera Toxin B subunit | Sigma-Aldrich | C9903-1MG | |
| Combitips advanced, Sterile, 5.0 mL | Fisher Scientific | 13-683-714 | |
| Cytation 10 Reader Imager | Agilent | 2105245 | |
| Defined Trypsin Inhibitor | Gibco | R007100 | soybean trypsin inhibitor in 1x PBS |
| Donor Horse Serum, 500 mL, United States Origin | Corning | 35-030-CV | |
| Dulbecco's Modified Eagle's Medium | Corning | 10-013-CV | |
| Excel Sofware | Microsoft office 365 | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Fetal Bovine Serum | Thermo Scientific | 26140079 | |
| Fetal Bovine Serum, qualified, US origin | Thermo Scientific | 26140079 | |
| Gentamicin Sulfate, Liquid | Corning | 30-005-CR | |
| GraphPad Prism | |||
| Hydrocortisone solution,50 µM, sterile-filtered, BioXtra | Sigma-Aldrich | H6909-10ML | |
| Image J Software | Fiji | https://imagej.net/ij/ | |
| Insulin solution human | Sigma-Aldrich | I9278-5ML | |
| Magnifying Glass | Fisher Scientific | 01-182-392 | |
| Microcentrifuge Tube | Costar | 3621 | |
| Microscope | Olympus | CKX53SF | |
| Mini Lightbox | Fisher Scientific | 361044708 | |
| Molecular Probes CellTracker Blue CMAC Dye | Fisher Scientific | C2110 | |
| Molecular Probes CellTracker Orange CMRA Dye | Fisher Scientific | C34551 | |
| Penicillin-Streptomycin Solution | Cytiva | SV30010 | |
| Penicillin-Streptomycin Solution | Cytiva | SV30010 | |
| Phase-Contrast Microscope | Echo | RVSF1000 | |
| Phosphate Buffered Saline | Fisher Scientific | SH3025601 | |
| Pipet Controller | Corning | 4099 | |
| Pipette 100 – 1,000 µL | Eppendorf | 3123000063 | |
| Pipette 20 – 200 µL | Eppendorf | 3123000055 | |
| Prism | GraphPad Software | https://www.graphpad.com/features | |
| RPMI 1640 with L-glutamine and 25 mM HEPES | Corning | 10-041-CV | |
| Serological Pipette, 10 mL | Thermo Scientific | 170356N | |
| Serological Pipette, 25 mL | Thermo Scientific | 170357N | |
| Serological Pipette, 5 mL | Thermo Scientific | 170355N | |
| Trypan Blue Solution, 0.4% | Corning | 25900CI | |
| Trypsin-EDTA, 0.25% | Corning | 25053CI |
Références
- de Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer Cell. 41 (3), 374-403 (2023).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: The next generation. Cell. 144 (5), 646-674 (2011).
- Anderson, N. M., Simon, M. C. The tumor microenvironment. Curr Biol. 30 (16), R921-R925 (2020).
- Baghban, R., et al. Tumor microenvironment complexity and therapeutic implications at a glance. Cell Commun Signal. 18 (1), 59 (2020).
- Sahai, E., et al. A framework for advancing our understanding of cancer-associated fibroblasts. Nat Rev Cancer. 20 (3), 174-186 (2020).
- Erdogan, B., Webb, D. J. Cancer-associated fibroblasts modulate growth factor signaling and extracellular matrix remodeling to regulate tumor metastasis. Biochem Soc Trans. 45 (1), 229-236 (2017).
- Iozzo, R. V., Sanderson, R. D. Proteoglycans in cancer biology, tumour microenvironment and angiogenesis. J Cell Mol Med. 15 (5), 1013-1031 (2011).
- Amersfoort, J., Eelen, G. Immunomodulation by endothelial cells - partnering up with the immune system. Nat Rev Immunol. 22 (9), 576-588 (2022).
- Balani, S., Nguyen, L. V., Eaves, C. J. Modeling the process of human tumorigenesis. Nat Commun. 8 (1), 15422 (2017).
- Drost, J., Clevers, H. Organoids in cancer research. Nat Rev Cancer. 18 (7), 407-418 (2018).
- Wang, E., Xiang, K., Zhang, Y., Wang, X. -. F. Patient-derived organoids (PDOs) and PDO-derived xenografts (PDOXs): New opportunities in establishing faithful pre-clinical cancer models. J Natl Cancer Cent. 2 (4), 263-276 (2022).
- Kamb, A. What's wrong with our cancer models. Nat Rev Drug Discov. 4 (2), 161-165 (2005).
- Tuveson, D., Clevers, H. Cancer modeling meets human organoid technology. Science. 364 (6444), 952-955 (2019).
- Zitvogel, L., Pitt, J. M., Daillère, R., Smyth, M. J. Mouse models in oncoimmunology. Nat Rev Cancer. 16 (12), 759-773 (2016).
- Liu, Y., et al. Patient-derived xenograft models in cancer therapy: technologies and applications. Signal Transduct Target Ther. 8 (1), 160 (2023).
- Ben-David, U., et al. Patient-derived xenografts undergo mouse-specific tumor evolution. Nat Genet. 49 (11), 1567-1575 (2017).
- Byrne, A. T., et al. Interrogating open issues in cancer precision medicine with patient-derived xenografts. Nat Rev Cancer. 17 (4), 254-268 (2017).
- Ivanics, T., et al. Patient-derived xenograft cryopreservation and reanimation outcomes are dependent on cryoprotectant type. Lab Invest. 98 (7), 947-956 (2018).
- Biju, T. S., Priya, V. V., Francis, A. P. Role of three-dimensional cell culture in therapeutics and diagnostics: an updated review. Drug Deliv and Transl Res. 13 (9), 2239-2253 (2023).
- Urzì, O., et al. Three-dimensional cell cultures: The bridge between in vitro and in vivo models. Int J Mol Sci. 24 (15), 12046 (2023).
- Cacciamali, A., Villa, R., Dotti, S. 3D cell cultures: Evolution of an ancient tool for new applications. Front Physiol. 13, 836480 (2022).
- Abuwatfa, W. H., Pitt, W. G. Scaffold-based 3D cell culture models in cancer research. J Biomed Sci. 31 (1), 7 (2024).
- Yakavets, I., et al. Advanced co-culture 3D breast cancer model for investigation of fibrosis induced by external stimuli: optimization study. Sci Rep. 10 (1), 21273 (2020).
- Mohammadrezaei, D., et al. Predicting and elucidating the post-printing behavior of 3D printed cancer cells in hydrogel structures by integrating in-vitro and in-silico experiments. Sci Rep. 13 (1), 1211 (2023).
- Kuen, J., Darowski, D., Kluge, T., Majety, M. Pancreatic cancer cell/fibroblast co-culture induces M2 like macrophages that influence therapeutic response in a 3D model. PLOS ONE. 12 (7), e0182039 (2017).
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Kennecke, H., et al. Metastatic behavior of breast cancer subtypes. J Clin Oncol. 28 (20), 3271-3277 (2010).
- Neve, R. M., et al. A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes. Cancer Cell. 10 (6), 515-527 (2006).
- Pingitore, P., et al. Human multilineage 3D spheroids as a model of liver steatosis and fibrosis. Int J Mol Sci. 20 (7), 1629 (2019).
- Bar-Ephraim, Y. E., Kretzschmar, K. Organoids in immunological research. Nat Rev Immunol. 20 (5), 279-293 (2020).
- Beerkens, A. P. M., et al. Characterizing OXPHOS inhibitor-mediated alleviation of hypoxia using high-throughput live cell-imaging. Cancer Metab. 12 (1), 13 (2024).
- Feaster, T. K., et al. Matrigel mattress. Cir Res. 117 (12), 995-1000 (2015).
- Huang, Z., Yu, P., Tang, J. Characterization of triple-negative breast cancer MDA-MB-231 cell spheroid model. Onco Targets Ther. 13, 5395-5405 (2020).
- Nam, S., Khawar, I. A., Park, J. K., Chang, S., Kuh, H. -. J. Cellular context-dependent interaction between cancer and stellate cells in hetero-type multicellular spheroids of pancreatic tumor. Biochem Biophys Res Commun. 515 (1), 183-189 (2019).
- Monteiro, M. V., Gaspar, V. M., Ferreira, L. P. Hydrogel 3D in vitro tumor models for screening cell aggregation mediated drug response. Biomater Sci. 8 (7), 1855-1864 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon