Method Article
Cellule stromali eteromulticellulari in colture 3D prive di scaffold di cellule tumorali epiteliali per guidare l'invasione
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
C'è un bisogno critico di modelli di cancro 3D che catturino il crosstalk eterocellulare per studiare le metastasi del cancro. Il nostro studio presenta la generazione di epiteliali stromali eteromulticellulari in un ambiente privo di scaffold e scaffold che può essere utilizzato per studiare l'invasione e le distribuzioni spaziali cellulari.
Abstract
Il cancro al seno è la seconda causa di morte correlata al cancro tra le donne negli Stati Uniti È stato dimostrato che i modelli organoidi di tumori solidi ricapitolano fedelmente aspetti della progressione del cancro come la proliferazione e l'invasione. Sebbene gli organoidi derivati da pazienti e gli organoidi xenotrapianto derivati da pazienti siano fisiopatologicamente rilevanti, sono costosi da propagare, difficili da manipolare e comprendono principalmente i tipi di cellule più proliferative all'interno del microambiente tumorale (TME). Queste limitazioni ne impediscono l'uso per chiarire i meccanismi cellulari di progressione della malattia che dipendono dalle cellule stromali associate al tumore, che si trovano all'interno del TME e che sono note per contribuire alle metastasi e alla resistenza alla terapia.
Qui, riportiamo i metodi per coltivare colture 3D multicellulari epiteliali-stromali. I vantaggi di questi metodi includono un sistema economico per la generazione rapida di colture 3D simili a organoidi all'interno di ambienti privi di scaffold che possono essere utilizzati per tracciare l'invasione con risoluzione di una singola cellula all'interno di scaffold di idrogel. In particolare, dimostriamo come generare queste colture 3D eteromulticellulari utilizzando cellule di carcinoma mammario BT-474 in combinazione con fibroblasti (BJ-5ta), cellule simili a monociti (THP-1) e/o cellule endoteliali (EA.hy926). Inoltre, la marcatura fluorescente differenziale delle popolazioni cellulari consente la microscopia time-lapse per definire l'assemblaggio di colture 3D e le dinamiche di invasione.
In particolare, l'aggiunta di due combinazioni di cellule stromali alle colture 3D di cellule BT-474 riduce significativamente la circolarità delle colture 3D, coerentemente con la presenza di strutture sferoidi secondarie o simili a organoidi. Negli esperimenti con colorante tracker, i fibroblasti e le cellule endoteliali co-localizzano nelle sporgenze periferiche simili a organoidi e sono segregati spazialmente dallo sferoide primario BT-474. Infine, le colture 3D eteromulticellulari di cellule BT-474 hanno una maggiore capacità di invasione dell'idrogel. Poiché abbiamo osservato queste strutture protrusive in colture 3D eteromulticellulari di cellule epiteliali mammarie sia non tumorigeniche che tumorigeniche, questo lavoro fornisce un metodo efficiente e riproducibile per generare colture 3D organoidi simili in un ambiente privo di scaffold per successive analisi dei fenotipi associati alla progressione del tumore solido.
Introduzione
È ora riconosciuto che la progressione del cancro dipende da due fattori principali: i cambiamenti genetici/epigenetici nelle cellule tumorali e una miriade di interazioni con cellule non tumorali nel microambiente tumorale (TME)1. Sebbene i cambiamenti genetici nelle cellule siano riconosciuti come necessari per l'inizio del tumore, tali alterazioni da sole non sono sufficienti per la progressione del tumore e le metastasi2. I componenti della TME, originariamente ritenuti spettatori silenziosi, sono ora noti per promuovere attivamente la progressione del cancro attraverso un crosstalk reciproco e dinamico con le cellule tumorali3. La composizione della TME varia a seconda del tessuto da cui ha origine il tumore, dello stadio del tumore e delle caratteristiche del paziente, ma le caratteristiche distintive includono i fibroblasti associati al cancro stromale (CAF), la matrice extracellulare (ECM), le cellule endoteliali vascolari e le cellule immunitarie adattative e mieloidi 1,4.
I CAF stromali contenuti nel TME sono composti da sottotipi di fibroblasti di diversa origine e funzione5. Tali CAF sono componenti chiave del TME in quanto interagiscono con le cellule tumorali in diverse interfacce. I CAF secernono proteine della ECM che alterano la rigidità della matrice, il che può limitare la somministrazione di farmaci attraverso un'eccessiva deposizione di collagene, proteoglicani e fibronectina o consentire alle cellule tumorali di invadere il sito tumorale primario attraverso la secrezione di metalloproteinasi della matrice che degradano la MEC (MMP)6,7. Inoltre, i CAF promuovono la crescita, la migrazione e la vascolarizzazione del tumore attraverso la secrezione di una varietà di fattori di crescita, citochine e fattori angiogenici come il fattore di crescita epidermico (EGF), il fattore di crescita trasformante β (TGF-β) e il fattore di crescita dell'endotelio vascolare (VEGF), rispettivamente 1,6. Parallelamente, le cellule endoteliali, indotte dall'ipossica TME, promuovono anche la vascolarizzazione tumorale e sopprimono le funzioni delle cellule immunitarie attraverso l'aumento della secrezione di fattori angiogenici e la riduzione della secrezione di molecole di adesione leucocitaria 1,8.
Con l'apparente complessità della progressione del cancro, è diventato essenziale incorporare i componenti stromali della TME nella ricerca di base sul cancro. Tuttavia, la creazione di modelli che ricapitolino fedelmente la fisiopatologia tumorale nota è ancora un significativo bisogno insoddisfatto 9,10. Mentre i tradizionali modelli di coltura cellulare bidimensionale (2D) sono facili da maneggiare, rapidamente coltivati e sono altamente riproducibili, sono costituiti solo da cloni di cellule tumorali a rapida proliferazione e non riflettono l'eterogeneità cellulare riscontrata nei tumori 10,11,12. Allo stesso modo, anche i modelli murini transgenici non catturano la biologia dei tumori umani a causa della bassa eterogeneità genetica dovuta alla consanguineità, alle differenze significative nel sistema immunitario e alla complessità istologica13,14. A causa di tali limitazioni, le terapie sviluppate a partire da modelli di cancro classici spesso non riescono a tradursi in contesti clinici.
I modelli tumorali derivati da pazienti, come gli xenotrapianti derivati da pazienti e gli organoidi derivati da pazienti, possono affrontare gli svantaggi dei modelli tumorali convenzionali catturando le caratteristiche molecolari del tumore in situ, il background genetico e l'organizzazione cellulare 10,11,15. Tuttavia, tali xenotrapianti e organoidi derivati da pazienti richiedono complicate procedure di attecchimento e un lungo tempo di coltura16,17. In combinazione con la variazione nei siti di acquisizione e campionamento del tumore e la scarsa efficienza nella crioconservazione, è necessario sviluppare modelli che fungano da ponte tra le classiche colture cellulari 2D e i modelli di cancro derivati da pazienti11,18. A questo proposito, i modelli 3D di colture cellulari possono fungere da modelli che possono essere coltivati rapidamente e catturare importanti caratteristiche tumorali in vivo come l'interazione cellula-cellula, l'interazione cellula-ECM, l'ipossia, l'angiogenesi e la produzione di ECM19,20.
I modelli 3D di colture cellulari sono classificati in sistemi di modelli senza scaffold e basati su scaffold. Nei sistemi privi di scaffold le cellule sono indotte ad autoaggregarsi in una forma sferica utilizzando specifiche piastre di coltura cellulare a basso attaccamento o manipolando i parametri fisici dei metodi di coltura. I metodi consolidati per ottenere sferoidi 3D privi di scaffold vanno da semplici colture di pellet cellulari mediante centrifugazione a gocce di micropiastre sospese, levitazione magnetica e bioreattori dinamici e sistemi microfluidici20,21. Le colture cellulari 3D basate su scaffold vengono stabilite mediante l'aggiunta di scaffold a base di polimeri o idrogel per imitare la matrice extracellulare fisiologica19,22. Tali modelli hanno un immenso potenziale per modellare in vivo l'organizzazione cellulare, la topologia, l'attaccamento alla matrice, la migrazione e la risposta ai farmaci.
Oltre alla manipolazione di scaffold per modelli di composizione della ECM in stati patologici, le colture cellulari 3D possono essere utilizzate anche per modellare popolazioni cellulari eterogenee in un TME. Le colture cellulari 3D composte da cellule tumorali e fibroblasti stromali o cellule endoteliali sono state utilizzate per studiare l'interazione tra il cancro e le singole linee cellulari non tumorali 23,24,25. Metodi riproducibili ed economici per espandere tali colture cellulari 3D composte da più linee cellulari eterogenee aiuterebbero i ricercatori a chiarire la progressione del tumore. Qui, riportiamo i metodi per coltivare colture 3D multicellulari epiteliali-stromali per studiare la proliferazione, l'invasione e la plasticità dello stato cellulare. Il protocollo descrive colture 3D basate su scaffold ed estratti di membrana basale di cellule di cancro al seno co-coltivate con una combinazione di cellule stromali che vanno da fibroblasti (BJ-5ta), cellule endoteliali (Ea.hy926) e cellule simili a monociti (THP-1). Il cancro al seno è attualmente il secondo tumore più comune al mondo e il cancro più diagnosticato nelle donne negli Stati Uniti26. La morte per carcinoma mammario è in gran parte dovuta alla natura metastatica e resistente alla terapia della malattia, poiché la sopravvivenza globale e libera da metastasi è significativamente ridotta nelle pazienti con diagnosi di carcinoma mammario aggressivo arricchito con HER2 e sottotipi basali27. I nostri protocolli di coltura cellulare 3D descritti possono aiutare a sviluppare metodi di coltura convenienti, rapidi e riproducibili che possono essere abbinati a metodi di conservazione dei tessuti inclusi in paraffina fissati in formalina e successive applicazioni di biologia spaziale.
Protocollo
1. Terreno di coltura cellulare
NOTA: Preparare tutti i terreni all'interno di una cappa di biosicurezza.
- Per preparare il terreno di coltura cellulare per BJ-5ta, BT474, EA.hy926 e MDA-MB-468, integrare 500 ml di Dulbecco's Modified Eagle Medium (DMEM) ad alto contenuto di glucosio con il 10% di siero fetale bovino inattivato termicamente (FBS) e l'1% di penicillina-streptomicina con una pipetta. Incorporare lo 0,1% di gentamicina con una micropipetta.
- Per preparare il terreno di coltura cellulare per MCF10A, integrare 500 ml di DMEM/F12 con il 5% di siero di cavallo e l'1% di penicillina-streptomicina con una pipetta. Incorporare lo 0,1% di gentamicina, 1 mL di 1 μg/mL di idrocortisone, 500 μL di 10 μg/mL di insulina, 50 μL di tossina del colera da 100 ng/mL e 10 μL di fattore di crescita epidermico da 20 ng/mL con una micropipetta.
- Per preparare il terreno di coltura cellulare per MCF10Ca1h, integrare 500 mL di DMEM/F12 con il 5% di siero di cavallo e l'1% di penicillina-streptomicina con una pipetta. Incorporare lo 0,1% di gentamicina con una micropipetta.
- Per preparare il terreno di coltura cellulare per THP-1, integrare 500 mL di RPMI 1640 con il 10% di FBS e l'1% di penicillina-streptomicina con una pipetta. Incorporare lo 0,1% di gentamicina con una micropipetta.
- Cambiare il terreno di coltura cellulare ogni 2-3 giorni per BJ-5ta, BT474, EA.hy926, MDA-MB-468, MCF10A e MCF10Ca1h fino a quando le cellule non raggiungono il 70-80% di confluenza valutando la loro crescita quotidianamente con un microscopio a campo chiaro. Cambiare il terreno di coltura cellulare una volta alla settimana per le cellule THP-1.
- Coltiva cellule BJ-5ta, BT474, EA.hy926, MDA-MB-468, MCF10A e MCF10Ca1h in piastre da 100 mm con superfici trattate per colture cellulari in incubatori per colture cellulari standard con CO2 al 5%.
- Coltivare cellule THP-1 in fiasche trattate con colture cellulari T75 in incubatori per colture cellulari standard con il 5% di CO2.
2. Raccolta di cellule
- Accendi la luce UV per disinfettare l'interno della cabina di biosicurezza per 15 minuti.
- Aprire l'anta della finestra dell'armadio di biosicurezza per stabilizzare il flusso d'aria e accendere il sistema di aspirazione del vuoto.
- Pulire la superficie interna della cappa e i tubi del sistema di aspirazione del vuoto con etanolo al 70%.
- Preparare un terreno di coltura cellulare fresco privo di siero all'interno della cabina di biosicurezza: integrare 500 ml di terreno di coltura cellulare modificato di Dulbecco (DMEM) ad alto contenuto di glucosio con l'1% di penicillina/streptomicina utilizzando una pipetta. Incorporare lo 0,1% di gentamicina con una micropipetta.
- Riscaldare il terreno di coltura cellulare, la soluzione salina tamponata con fosfato (PBS) e la tripsina-EDTA (0,25%) a 37 °C ponendo gli elementi in un bagno di perline prima di iniziare l'esperimento.
- Assicurarsi che le cellule siano confluenti al 70-80% attraverso l'ispezione visiva al microscopio.
- Aspirare ed eliminare il terreno di coltura dalle celle piastrate con l'aspiratore a vuoto. Lavare il terreno rimanente una volta con 2 ml di PBS con una pipetta, quindi aspirare ed eliminare il PBS con l'aspiratore a vuoto.
NOTA: Le cellule THP-1 vengono coltivate in sospensione; I passaggi 2.7-2.9 possono essere omessi. - Aggiungere 1 mL di tripsina alla piastra di coltura cellulare utilizzando una micropipetta e posizionare la piastra all'interno di un incubatore al 5% di CO2 a 37 °C per 5 minuti.
- Inattivare la tripsina aggiungendo 1 mL di inibitore della tripsina di soia (in 1x PBS) alla piastra con una micropipetta. Disperdere i cluster cellulari pipettando la miscela liquida con una micropipetta P1000 e raccogliere la sospensione cellulare dal fondo della piastra. Trasferire la sospensione cellulare con una micropipetta in una provetta conica da 15 mL e centrifugare a 100 × g per 5 minuti a temperatura ambiente. Eliminare il surnatante con l'aspiratore a vuoto.
- Continuare con il conteggio delle cellule.
3. Preparazione della soluzione colorante di lavoro e delle celle di colorazione in sospensione
- Prima di aprire il flaconcino delle sonde fluorescenti molecolari per tracciatori cellulari (vedere la Tabella dei materiali), lasciare riscaldare il prodotto a temperatura ambiente per 15 minuti in un bagno di perle impostato a 37 °C.
- Sciogliere il colorante blu liofilizzato (massa = 5 mg, peso molecolare = 209,6 g/mol) a una concentrazione finale di 10 mM con 2,385 mL di DMSO utilizzando una micropipetta.
- Sciogliere il colorante arancione liofilizzato (massa = 50 μg, peso molecolare = 550,4 g/mol) a una concentrazione finale di 10 mM con 9,084 μl di DMSO utilizzando una micropipetta.
- Sciogliere il colorante rosso intenso per il tracciatore cellulare liofilizzato (massa = 15 μg, peso molecolare = 698,3 g/mol) a una concentrazione finale di 1 mM con 20 μl di DMSO utilizzando una micropipetta.
- Preparare la soluzione di terreno colorante blu per il tracciante della cella di lavoro (5 μM) diluendo 1 μL di colorante in 2 mL di terreno DMEM privo di siero con una micropipetta.
- Preparare la soluzione di terreno colorante arancione per il tracciante a celle di lavoro (5 μM) diluendo 1 μL di colorante in 2 mL di terreno DMEM privo di siero con una micropipetta.
- Preparare la soluzione di terreno colorante rosso intenso per il tracker delle celle di lavoro (1 μM) diluendo 2 μL di colorante in 2 mL di terreno DMEM privo di siero con una micropipetta.
- Risospendere le cellule epiteliali, i fibroblasti BJ-5ta e le cellule endoteliali Ea.hy926 nelle soluzioni di coloranti blu, arancioni e rosso intenso preparate per il tracker cellulare di lavoro (2 mL), rispettivamente con una micropipetta.
- Incubare le provette a 37 °C nell'incubatore al 5% di CO2 per 30 minuti.
- Dopo 30 minuti di incubazione, centrifugare le provette a 100 x g per 5 minuti a temperatura ambiente.
- Aspirare ed eliminare il surnatante con l'aspiratore a vuoto e risospendere accuratamente il pellet in 1 mL di terreno DMEM contenente siero FBS al 10% con una micropipetta.
- Continuare con il conteggio delle cellule.
4. Conteggio delle cellule
- Raccogliere 10 μl della sospensione cellulare e trasferirla in una microprovetta con una micropipetta.
- Miscelare con 10 μl di blu di tripano e pipettare accuratamente.
- Trasferire 20 μl della soluzione di tripano cellulare con una micropipetta in un vetrino della camera di conteggio delle cellule. Inserisci e conta le celle utilizzando un contatore di celle automatizzato.
- Calcola il numero medio totale di cellule vive da due letture.
5. Calcoli
- Preparare le scorte di cellule di lavoro per ogni tipo di cellula a una concentrazione di 6,67 × 103 cellule/mL equivalenti a 2.000 cellule/300 μL.
NOTA: Il volume totale delle scorte dipenderà dalla dimensione del campione sperimentale. - Per gli sferoidi monocolturali, assicurarsi che ogni campione indipendente sia costituito da 300 μl di materiale di lavoro delle cellule epiteliali equivalenti a 2.000 cellule epiteliali.
- Per gli sferoidi in co-coltura (due tipi di cellule), assicurarsi che ogni campione indipendente sia costituito da 150 μl di materiale di lavoro di cellule epiteliali e 150 μl di materiale di lavoro di cellule stromali. Il campione conterrà 1.000 cellule epiteliali e 1.000 cellule stromali.
- Per gli sferoidi in co-coltura (tre tipi di cellule), assicurarsi che ogni campione indipendente sia costituito da 150 μL di materiale di lavoro di cellule epiteliali, 75 μL di materiale di lavoro di cellule stromali #1 e 75 μL di materiale di lavoro di cellule stromali #2. Il campione conterrà 1.000 cellule epiteliali, 500 cellule stromali del working stock #1 e 500 cellule stromali del working stock #2.
6. Placcatura
- Trasferire il volume richiesto con una micropipetta per tre repliche tecniche, più una extra, in una microprovetta. Miscelare accuratamente con una pipetta, quindi trasferire 300 μl di campione in un pozzetto di una micropiastra a 96 pozzetti con fondo a U e attacco ultrabasso.
- Ripetere il processo per ogni replica tecnica aggiuntiva.
- Collocare la piastra a 96 pozzetti in un incubatore a 37 °C.
7. Imaging in campo chiaro
- Osservare la crescita e la morfologia degli sferoidi ogni 24 ore fino a 96 ore utilizzando un microscopio.
- Immagine di sferoidi utilizzando un microscopio a contrasto di fase.
8. Configurazione del protocollo di imaging a campo largo per sferoidi colorati con coloranti tracker e sferoidi sovrapposti con estratto di membrana basale
- Accendere i dispositivi di imaging e l'incubatore automatizzato elencati nella Tabella dei materiali e creare un nuovo protocollo di imaging nel task manager del software di imaging. Nella scheda Procedura , impostare il set point di temperatura dell'incubatore automatico su 37 °C e attendere che l'incubatore equilibri il2% di CO e la temperatura prima di continuare con il passaggio successivo.
NOTA: È possibile utilizzare un imager fluorescente alternativo se dispone dei filtri appropriati per ciascuno dei coloranti (blu, rosso intenso e arancione) utilizzati. - Impostare le impostazioni dell'immagine sulle seguenti specifiche: Ingrandimento: 4X PL FL Phase, Campo visivo: 3185 X 3185 μm, Full WFOV.
- Utilizzare le seguenti specifiche per i canali: DAPI: 377/447 nm, illuminazione = 10, tempo di integrazione = 107 ms, guadagno = 10, RFP: 531/593 nm, illuminazione = 10, tempo di integrazione = 137 ms, guadagno = 10, CY5: 628/685 nm, illuminazione = 10, tempo di integrazione = 137 ms, guadagno = 10.
- Per l'imaging degli sferoidi in una sovrapposizione di soluzione di estratto dalla membrana basale, utilizzare le seguenti specifiche di campo chiaro: illuminazione = 10, tempo di integrazione = 5 ms e guadagno = 17,1.
- Selezionare i pozzetti desiderati per l'imaging e approvare le modifiche alle specifiche facendo clic sull'icona di selezione dei pozzetti .
- Passare alla scheda Riduzione dati per regolare le impostazioni dell'analisi cellulare.
- Impostate il valore di soglia su 19.500 con uno sfondo chiaro e selezionate Riempi buchi nelle maschere.
- Per la selezione degli oggetti, impostare la dimensione minima dell'oggetto su 100 μm e la dimensione massima dell'oggetto su 1.000 μm, quindi selezionare Analizza l'intera immagine.
NOTA: per questa analisi sono necessari solo una maschera primaria e il conteggio degli oggetti. - Salvare tutte le modifiche e aprire l'applicazione imager.
- Posizionare la micropiastra sperimentale con fondo a forma di U nell'incubatore aprendo il cassetto, quindi chiuderla utilizzando il software dell'imager.
- Per eseguire il protocollo, fare clic sulla scheda Informazioni sulla procedura , aggiungere un utente e scegliere il protocollo.
- Assicurarsi che sia selezionato il tipo di lastra corretto e impostare il tempo di imaging su 30 minuti per piastra.
- Selezionare l'intervallo di imaging desiderato, indicare se la lastra è dotata di coperchio e regolare l'ora di inizio e la durata dell'imaging.
- Fare clic su Pianifica piastra/recipiente per avviare il processo di imaging.
9. Sovrapposizione della soluzione di estratto della membrana basale (opzionale)
NOTA: La soluzione di estratto della membrana basale può essere applicata agli sferoidi 24 ore dopo la placcatura.
- Riempi un secchiello del ghiaccio con ghiaccio per mantenere fredda la soluzione di estratto della membrana del seminterrato e conservala a 4 °C quando non viene utilizzata.
- Aspirare circa 170 μl di terreno con una pipetta multicanale.
- Usa una lente d'ingrandimento e una mini lightbox per osservare da vicino i piccoli sferoidi. Posiziona la piastra sferoidale a 96 pozzetti sopra la lightbox e posiziona la lente d'ingrandimento sopra la testa.
- Impostare la pipetta P200 a 30 μl e raccogliere l'estratto della membrana basale per creare tre microgoccioline.
- Aggiungere una microgoccia su ogni sferoide delle tre repliche tecniche con una micropipetta.
- Assicurarsi che la piastra a 96 pozzetti sia piana e posizionare la pipetta verticalmente sopra lo sferoide. Rilasciare la gocciolina senza toccare il fondo del pozzetto.
- Porre la piastra in un'incubatrice a 37 °C per 20 minuti.
- Sovrapporre gli sferoidi con altri 50 μl di soluzione di estratto della membrana basale per pozzetto con una micropipetta.
- Riposizionare gli sferoidi al centro del pozzetto utilizzando la punta di una pipetta.
- Incubare a 37 °C per 30 min.
- Aggiungere 100 μL di terreno di coltura cellulare a ciascun pozzetto.
10. Quantificazione
- Scaricare e aprire il software ImageJ. Carica le immagini sferoidi.
- Impostare le misure facendo clic su Analizza | Imposta le misure e seleziona l'area | Baricentro. Misura gli sferoidi utilizzando lo strumento lazo per tracciare lo sferoide e misurare l'oggetto.
- Registra i valori per tre repliche biologiche e calcola l'area media e la circolarità.
- Converti l'area da pixel a metri quadrati utilizzando la seguente formula:
Area (m2) = ((550/504) × √ (area in pixel)) 2 - Analizza i dati.
- Selezionare Immetti i valori di replica, impilati in colonne.
- Inserire i dati nelle colonne e selezionare il controllo degli sferoidi in monocoltura e i campioni di sferoidi per il trattamento di co-coltura.
- Eseguire un'analisi della colonna ANOVA unidirezionale.
- Utilizzare i seguenti parametri ANOVA:
- Evita l'abbinamento o l'accoppiamento.
- Supponiamo una distribuzione gaussiana dei residui.
- Supponiamo deviazioni standard uguali.
- Usa il test di confronto multiplo di Tukey per confrontare la media di ogni colonna con la media di ogni altra colonna.
11. Elaborazione di immagini in immunofluorescenza a campo largo
- Salva le immagini direttamente dal software imager in formato PNG o Tiff.
- Seleziona ogni singolo canale o i canali sovrapposti e salvali come file PNG.
- Regolare la luminosità e il contrasto nel software imager o in ImageJ se le immagini vengono esportate come file Tiff.
Risultati
In questo studio, abbiamo sviluppato un sistema di coltura cellulare per generare sferoidi 3D eteromulticellulari costituiti da cellule epiteliali e stromali con morfologia organoide. Gli sferoidi sono stati stabiliti mediante placcatura di 2.000 cellule epiteliali in condizioni di monocoltura. In condizioni di co-coltura di due tipi di cellule, gli sferoidi sono stati stabiliti mediante placcatura di 1.000 cellule epiteliali e 1.000 cellule stromali. In condizioni di co-coltura di tre tipi di cellule, gli sferoidi sono stati stabiliti mediante placcatura di 1.000 cellule epiteliali e due diversi tipi di cellule stromali di 500 cellule di ciascun tipo di cellula. A monte dell'insediamento degli sferoidi, le cellule possono essere colorate con coloranti fluorescenti che consentono di monitorare l'organizzazione spaziale cellulare. Dopo 24 ore dalla formazione iniziale degli sferoidi, le applicazioni a valle includono perturbazioni farmacologiche, imaging e raccolta di campioni. L'imaging time-lapse è utile per valutare i cambiamenti nel comportamento e nella morfologia degli sferoidi, compresa l'area e la circolarità (Figura 1A). A 24 ore dopo la placcatura, gli sferoidi possono essere incorporati in un ambiente di scaffold e l'imaging time-lapse può essere utilizzato per valutare l'insorgenza di strutture invasive dallo sferoide (Figura 1B). La raccolta di campioni di sferoidi eteromulticellulari ha molte applicazioni, tra cui la profilazione genomica e proteomica a livello globale e di singola cellula attraverso tecniche sperimentali come il sequenziamento dell'RNA, il sequenziamento dell'RNA a singola cellula, il sequenziamento proteomico e l'immunofluorescenza ciclica.
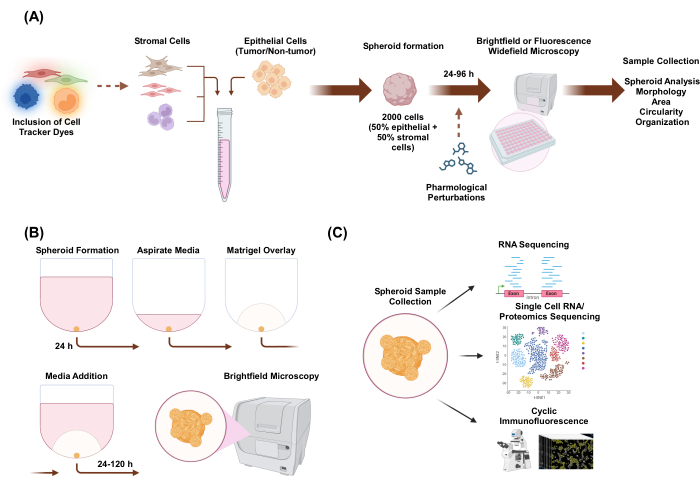
Figura 1: Rappresentazione schematica del processo di coltura cellulare 3D e potenziali applicazioni. (A) La sospensione cellulare di cellule epiteliali con cellule stromali non epiteliali viene pipettata in piastre di attacco 3D ultrabasse per formare sferoidi. Gli sferoidi vengono riprodotti mediante microscopia a campo chiaro ogni 24 ore per 96 ore. Le linee cellulari possono essere colorate con coloranti fluorescenti per la microscopia a campo largo a valle prima di indurre la formazione di sferoidi o possono essere perturbate farmacologicamente dopo la formazione di sferoidi. È possibile analizzare parametri sferoidi come la morfologia, l'area, la circolarità e l'organizzazione. (B) Gli sferoidi delle cellule epiteliali ed epiteliali con cellule stromali si formano utilizzando il protocollo di A. Gli sferoidi sono stati stabiliti mediante placcatura di 2.000 cellule epiteliali in condizioni di monocoltura. In condizioni di co-coltura di due tipi di cellule, gli sferoidi sono stati stabiliti mediante placcatura di 1.000 cellule epiteliali e 1.000 cellule stromali. In condizioni di co-coltura di tre tipi di cellule, gli sferoidi sono stati stabiliti mediante placcatura di 1.000 cellule epiteliali e due diversi tipi di cellule stromali di 500 cellule di ciascun tipo di cellula. Dopo 24 ore, viene sovrapposta una soluzione di estratto di membrana basale simile a uno scaffold e le immagini vengono acquisite in microscopia a campo chiaro ogni 24 ore per 120 ore. (C) Le colture 3D etero-multicellulari consolidate prive di scaffold e basate su scaffold possono essere utilizzate per una varietà di applicazioni a valle come l'immunofluorescenza ciclica, il sequenziamento dell'RNA a singola cellula e la proteomica a singola cellula. Clicca qui per visualizzare una versione più grande di questa figura.
Gli sferoidi monocoltura MCF10A, MCF10Ca1h e BT-474 mantengono un fenotipo sferico compatto per un massimo di 96 ore di post-placcatura. Quando sono stati co-coltivati con cellule endoteliali microvascolari EA.hy926, fibroblasti BJ-5ta e/o cellule monocitarie THP-1, gli sferoidi hanno sviluppato protrusioni cellulari alla periferia, che sono diventate più pronunciate a 96 ore (Figura 2A-C). È importante sottolineare che queste sporgenze, gemmazione e fenomeni di compattazione degli sferoidi rappresentano variazioni dell'organizzazione cellulare dello sferoide tumorale e delle cellule stromali co-coltivate che sono correlate con l'invasività 3D in seguito all'aggiunta di un idrogel basato sulla membrana basale come Matrigel. La morfologia delle gemme variava da aggregati di cellule solide a quelle di cellule sciolte, simili alla morfologia degli organoidi. Al contrario, gli sferoidi di monocoltura MDA-MB-468 apparivano come grandi aggregati di cellule sciolte. Tuttavia, quando le cellule MDA-MB-468 sono state co-coltivate con EA.hy926, BJ-5ta e/o THP-1, hanno formato sferoidi compatti (Figura 2D).
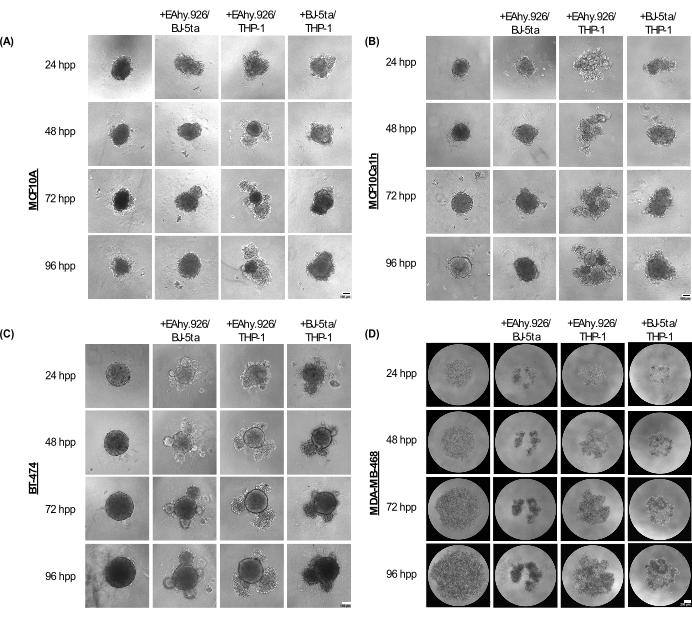
Figura 2: La gamma di architetture e morfologie adottate da combinazioni di cellule stromali non epiteliali in combinazione quando combinate con cellule tumorali/non tumorali epiteliali in colture 3D prive di scaffold. Immagini rappresentative in campo chiaro di (A) MCF10A, (B) MCF10Ca1h, (C) BT-474, (D) sferoidi MDA-MB-468 in monocoltura o in condizioni di co-coltura con fibroblasti stromali BJ-5ta/cellule endoteliali microvascolari Ea.hy926 o cella monocitaria Ea.hy926/THP-1 o cellule BJ-5ta/THP-1 in 96 ore. Ogni sferoide è stato formato placcando 2.000 cellule. Gli sferoidi in condizioni di monocoltura sono stati formati utilizzando 2.000 cellule epiteliali. Gli sferoidi in condizioni di co-coltura sono stati formati utilizzando 1.000 cellule epiteliali e due diversi tipi di cellule stromali da 500 cellule. La formazione di strutture organoidi in gemmazione o aggregate è stata avviata in condizioni di co-coltura di sferoidi 24 ore dopo la placcatura in cellule epiteliali MCF10A, MCF10Ca1h e BT-474. La compattazione delle cellule MDA-MB-468 è stata osservata in condizioni di co-coltura di sferoidi 25 ore dopo la placcatura. Barre di scala = 100 μm. Abbreviazione: hpp = ore dopo la placcatura. Clicca qui per visualizzare una versione più grande di questa figura.
A 72 ore dalla placcatura, le cellule MCF10A, MCF10Ca1h e BT-474 co-coltivate con EA.hy926 e THP-1, o con BJ-5ta e THP-1, hanno mostrato un aumento significativo dell'area sferoidale rispetto agli sferoidi epiteliali in monocoltura. MCF10Ca1h ha anche mostrato un aumento significativo dell'area sferoidale quando co-coltivato con EA.hy926 e BJ-5ta. L'insorgenza di strutture gemmative in sferoidi co-coltivati ha portato a una significativa diminuzione della circolarità sferoidale per MCF10A, MCF10Ca1h e BT-474 co-coltivati con EA.hy926 e THP-1, o con BJ-5ta e THP-1. Effetti simili sono stati osservati per BT-474 co-coltivato sia con EA.hy926 che con BJ-5ta (Figura 3A, B). Al contrario, le cellule MDA-MB-468 co-coltivate con EA.hy926 e BJ-5ta, EA.hy926 e THP-1, o BJ-5ta e THP-1, hanno mostrato una significativa diminuzione dell'area sferoidale rispetto agli sferoidi MDA-MB-468 in monocoltura; tuttavia, non c'è stato alcun effetto sulla circolarità (Figura 3A, B).

Figura 3: Grafici di analisi dell'area e della circolarità di colture 3D senza scaffold stromali monocolturali ed eteromulticellulari a 72 ore dopo la placcatura. (A) Area media (cm2) e (B) circolarità media di MCF10A, MCF10Ca1h, BT-474 e MDA-MB-468 in colture di sferoidi stromali monocolturali ed eteromulticellulari 72 ore dopo la placcatura. I dati riportati sono rappresentativi di almeno tre repliche biologiche indipendenti e sono riportati come medie tecniche di replica ± SEM, se non diversamente indicato. *, **, *** o **** rappresentano i valori p < rispettivamente 0,05, 0,01, 0,001 o 0,0001, se non diversamente specificato. Clicca qui per visualizzare una versione più grande di questa figura.
L'applicazione del colorante cell tracker alle cellule epiteliali tumorigeniche BT-474 e alle cellule stromali prima dell'insediamento degli sferoidi ha dimostrato che le cellule stromali, tra cui EA.hy926 e BJ-5ta, formavano le strutture in gemmazione sul perimetro degli sferoidi centrali BT474 (Figura 4, Video supplementare S1, Video supplementare S2, Video supplementare S3 e Video supplementare S4). A 48 ore dopo la placcatura, le singole immagini di fluorescenza a campo largo di sferoidi co-coltivati con fibroblasti rivelano che gli sferoidi dei fibroblasti co-localizzati insieme alle cellule endoteliali ma non co-localizzati con gli sferoidi BT-474. È stato anche scoperto che una minoranza di cellule endoteliali co-localizza con gli sferoidi BT-474 in condizioni di co-coltura. Ciò suggerisce che la disposizione delle cellule stromali all'interno dello sferoide è correlata con una morfologia simile a quella degli organoidi.
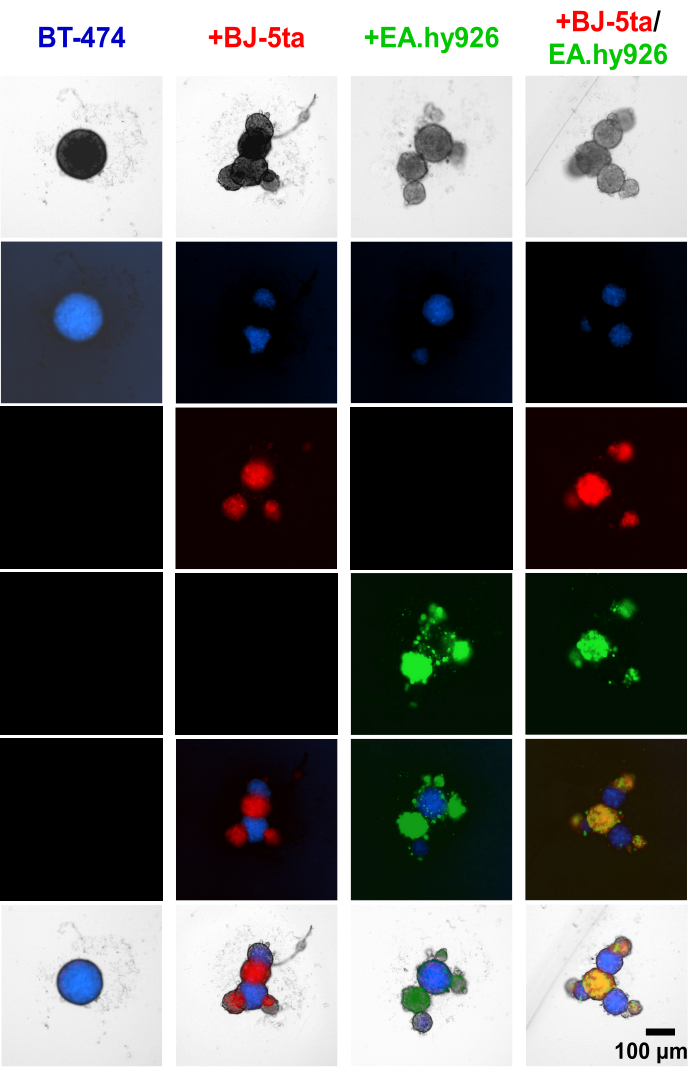
Figura 4: Immagini fisse a fluorescenza ad ampio campo di cellule stromali e BT-474 colorate in modo differenziale in colture 3D eteromulticellulari a 48 ore dopo la placcatura. Gli sferoidi BT-474 sono colorati con colorante fluorescente blue cell tracker. I fibroblasti BJ-5ta sono colorati con colorante tracciante cellulare arancione, rappresentato in colore rosso. Le cellule endoteliali Ea.hy926 sono colorate con un colorante tracciante di cellule rosso intenso, rappresentato in colore verde. Ogni sferoide è stato formato placcando 2.000 cellule. Gli sferoidi in condizioni di monocoltura sono stati formati utilizzando 2.000 cellule epiteliali. Gli sferoidi in condizioni di co-coltura (BT-474/BJ-5ta, BT-474/Ea.hy926) sono stati formati utilizzando 1.000 cellule epiteliali e 1.000 cellule stromali. Gli sferoidi in condizioni di doppia co-coltura (BT-474/BJ-5ta/Ea.hy926) sono stati formati utilizzando 1.000 cellule epiteliali e 500 cellule di ciascun tipo di cellula stromale. Le cifre sono rappresentative di almeno tre repliche biologiche. Barra della scala = 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Per valutare la rilevanza biologica del nostro modello di sferoide organoide, gli sferoidi sono stati sovrapposti con una soluzione di estratto di membrana basale 24 ore dopo la piastratura. Gli sferoidi BT-474 in monocoltura non hanno mostrato proprietà invasive 120 ore dopo la placcatura. Tuttavia, gli sferoidi BT-474 co-coltivati con BJ-5ta o EA.hy926 hanno sviluppato strutture alla periferia dello sferoide, che hanno invaso l'ambiente della soluzione di estratto della membrana basale dell'impalcatura. Il numero e la lunghezza di queste sporgenze sono stati significativamente aumentati negli sferoidi BT-474 co-coltivati sia con BJ-5ta che con EA.hy926 da 48 ore a 120 ore di post-placcatura (Figura 5, Video supplementare S5, Video supplementare S6, Video supplementare S7 e Video supplementare S8).
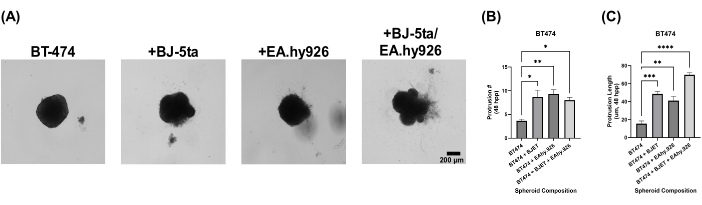
Figura 5: Immagini fisse in campo chiaro di cellule BT-474 in colture 3D eteromulticellulari con sovrapposizione della soluzione di estratto della membrana basale a 5 giorni dopo la sovrapposizione. Ogni sferoide è stato formato placcando 2.000 cellule. La soluzione di estratto della membrana basale è stata sovrapposta agli sferoidi 24 ore dopo la placcatura. Gli sferoidi in condizioni di monocoltura sono stati formati utilizzando 2.000 cellule epiteliali. Gli sferoidi in condizioni di co-coltura (BT-474/BJ-5ta e BT-474/Ea.hy926) sono stati formati utilizzando 1.000 cellule epiteliali e 1.000 cellule stromali. Gli sferoidi in condizioni di doppia co-coltura (BT-474/BJ-5ta/Ea.hy926) sono stati formati utilizzando 1.000 cellule epiteliali e 500 cellule di ciascun tipo di cellula stromale. (A) In condizioni di co-coltura si possono osservare strutture invasive che sporgono dallo sferoide tumorale incorporato nella soluzione di estratto della membrana basale. (B) Qualificazione della conta delle protrusioni invasive al tempo 48 hpp. (C) Qualificazione della lunghezza della protrusione invasiva al tempo 48 hpp. Le cifre sono rappresentative di almeno tre repliche biologiche. Barra della scala = 200 μm. Abbreviazione: hpp = ore dopo la placcatura. Clicca qui per visualizzare una versione più grande di questa figura.
Video supplementare S1: Sferoide di monocoltura BT-474 con colorante blu cell tracker. Le cellule BT-474 (2.000) sono state risospese in una micropiastra a 96 pozzetti con fondo a forma di U e attacco ultrabasso per formare sferoidi. Una sovrapposizione di immagini a fluorescenza in campo chiaro e a campo largo è stata catturata nell'arco di 48 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S2: Sferoidi BT-474 co-coltivati con fibroblasti BJ-5ta. BT-474 (1.000) e 1.000 fibroblasti BJ-5ta sono stati risospesi in piastre ULA per formare sferoidi. Le cellule BT-474 sono state incubate con colorante blu cell tracker e fibroblasti BJ-5ta con colorante arancione cell tracker. Una sovrapposizione di immagini in campo chiaro e a fluorescenza in campo largo (blu = BT-474, rosso = BJ-5ta) è stata catturata nell'arco di 48 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S3: Sferoidi BT-474 co-coltivati con cellule endoteliali Ea.hy926. BT-474 (1.000) e 1.000 cellule endoteliali Ea.hy926 sono state risospese in piastre ULA per formare sferoidi. Le cellule BT-474 sono state incubate con il colorante blu cell tracker e le cellule endoteliali Ea.hy926 con il colorante rosso intenso cell tracker. Una sovrapposizione di immagini fluorescenti in campo chiaro e a campo largo (blu = BT-474, verde = Ea.hy926) è stata catturata nell'arco di 48 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S4: Sferoidi BT-474 co-coltivati con fibroblasti BJ-5ta e cellule endoteliali Ea.hy926. BT-474 (1.000), 500 fibroblasti BJ-5ta e 500 cellule endoteliali Ea.hy926 sono stati risospesi in piastre ULA per formare sferoidi. Le cellule BT-474 sono state incubate con colorante blu cell tracker. I fibroblasti BJ-5ta e le cellule endoteliali Ea.hy926 sono stati incubati rispettivamente con il colorante arancione e il colorante rosso intenso. Una sovrapposizione di immagini fluorescenti in campo chiaro e a campo largo (blu = BT-474, rosso = BJ-5ta, verde = Ea.hy926) è stata catturata nell'arco di 48 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S5: Sferoide di monocoltura BT-474 in soluzione di estratto di membrana basale. Le cellule BT-474 (2.000) sono state risospese in piastre ULA per formare sferoidi e gli sferoidi sono stati incorporati in una soluzione di membrana basale 24 ore dopo la placcatura. Le immagini in campo chiaro sono state scattate nell'arco di 60 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S6: Sferoidi BT-474 co-coltivati con fibroblasti BJ-5ta in una soluzione di estratto di membrana basale. I fibroblasti BT-474 (1.000) e 1.000 BJ-5ta sono stati risospesi in piastre ULA per formare sferoidi e gli sferoidi sono stati incorporati in una soluzione di estratto di membrana basale 24 ore dopo la placcatura. Le immagini in campo chiaro sono state scattate nell'arco di 60 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S7: Sferoidi BT-474 co-coltivati con cellule endoteliali Ea.hy926 in una soluzione di estratto di membrana basale. BT-474 (1.000) e 1.000 cellule endoteliali Ea.hy926 sono state risospese in piastre ULA per formare sferoidi e gli sferoidi sono stati incorporati in una soluzione di estratto di membrana basale 24 ore dopo la placcatura. Le immagini in campo chiaro sono state scattate nell'arco di 60 ore. Abbreviazione: ULA = attacco ultrabasso. Clicca qui per scaricare questo video.
Video supplementare S8: Sferoidi BT-474 co-coltivati con fibroblasti BJ-5ta e cellule endoteliali Ea.hy926 in soluzione di estratto di membrana basale. BT-474 (1.000), 500 fibroblasti BJ-5ta e 500 cellule endoteliali Ea.hy926 sono stati risospesi in piastre ULA per formare sferoidi e gli sferoidi sono stati incorporati in una soluzione di estratto di membrana basale 24 ore dopo la placcatura. Le immagini in campo chiaro sono state scattate nell'arco di 60 ore. Clicca qui per scaricare questo video.
Discussione
Il nostro modello di sferoide etero-multicellulare dimostra che le interazioni tra cellule epiteliali e stromali guidano la gemmazione delle cellule stromali in condizioni di coltura 3D senza scaffold e la formazione di strutture invasive in condizioni di coltura 3D basate su scaffold. Abbiamo osservato una consistente formazione di strutture gemmative sia nelle linee cellulari tumorigeniche (MCF10Ca1h e BT-474) che nella linea cellulare epiteliale non tumorigenica MCF10A (Figura 2A-C). È interessante notare che, mentre gli sferoidi delle linee cellulari MCF10A, MCF10Ca1h e BT-474, sia in monocoltura che in co-coltura con fibroblasti e cellule endoteliali, hanno formato strutture circolari e compatte in gemmazione in ambienti privi di scaffold, gli sferoidi della linea cellulare MDA-MB-468 hanno mostrato una minore compattazione (Figura 2D). Questo era inaspettato, poiché si prevedeva che le cellule MDA-MB-468 formassero sferoidi circolari, come le altre linee cellulari epiteliali testate. Si è scoperto che la linea cellulare SUM-149, una linea cellulare primaria ibrida epiteliale-mesenchimale per il carcinoma mammario, forma sferoidi compatti come MCF10A, MCF10Ca1h e BT-474 (dati non mostrati)28. La ricerca futura dovrebbe indagare le vie di segnalazione che mediano la formazione di sferoidi meno compatti nella linea cellulare MDA-MB-468 poiché tali anomalie possono indicare vulnerabilità cellulari che possono essere sfruttate per lo sviluppo di nuove strategie terapeutiche.
Per quanto riguarda la combinazione di diverse linee cellulari, le cellule monocitarie THP-1 inducono la formazione di strutture di aggregati sciolti in tutte le condizioni di co-coltura di sferoidi. Si osserva una minore compattazione nelle strutture in gemmazione in condizioni di co-coltura con cellule THP-1/Ea.hy926 rispetto a quelle in condizioni di co-coltura con cellule THP-1/BJ-5ta (Figura 2A-D). Pertanto, la compattazione degli sferoidi è influenzata dalla composizione delle cellule stromali nel modello 29,30.
Una placcatura incoerente delle combinazioni eterocellulari può portare a uno sviluppo incompleto degli sferoidi o alla variabilità delle dimensioni degli sferoidi, rispetto alle loro repliche tecniche. Garantire la coerenza nella placcatura cellulare è fondamentale, poiché la disposizione spaziale delle cellule che determina il fenotipo e lo sviluppo degli sferoidi avviene principalmente entro le prime 48 ore31. Durante questo periodo critico, i rapporti cellulari svolgono un ruolo cruciale, con la proporzione di cellule stromali che influenza sia l'area sferoidale che la circolarità.
Gli aggregati sferoidi sciolti, che dipendono dal tipo di cella, possono essere facilmente smontati durante la movimentazione manuale. Tuttavia, la manipolazione manuale è necessaria quando si sovrappongono sferoidi con una soluzione di estratto della membrana basale simile a uno scaffold o quando si raccolgono campioni di sferoidi interi. Per mitigare questo problema, è consigliabile lavorare a un ritmo più lento durante la raccolta degli sferoidi tramite aspirazione con pipetta e ridurre al minimo la manipolazione manuale quando possibile. Durante la sovrapposizione della soluzione di estrazione della membrana basale, gli sferoidi tendono a spostarsi all'interno del pozzo, spesso depositandosi sul bordo del pozzo. Un rapporto soluzione/media dell'estratto della membrana basale è importante per mantenere le proprietà della matrice del sistema 3D; Tuttavia, gli sferoidi non possono essere completamente avvolti a causa della viscosità della soluzione di estratto della membrana basale, quindi è necessario un intervento manuale32. Il posizionamento non centrato degli sferoidi rappresenta una sfida per l'imaging, poiché la rifrazione della luce vicino al bordo plastico del pozzetto può interferire con la nitidezza dell'immagine.
Gli sferoidi utilizzati in questo studio erano composti da linee cellulari tumorali umane; I metodi qui descritti non sono ancora stati applicati alle linee cellulari primarie. I nostri risultati suggeriscono che specifiche interazioni cellula-cellula possono guidare la formazione di strutture simili a organoidi, indicando la potenziale applicabilità di questo metodo a un'ampia gamma di linee cellulari tumorigeniche. Tuttavia, mentre il nostro modello tenta di imitare la complessità del microambiente tumorale, i diversi tassi di proliferazione cellulare rappresentano una sfida per gli studi a lungo termine33. Le cellule a proliferazione rapida tendono a crescere più di quelle a proliferazione più lenta, rendendo questo modello meno adatto per esperimenti prolungati. Inoltre, i diversi requisiti del tipo di terreno di crescita per le linee cellulari possono introdurre ulteriori fattori che possono portare a fenotipi sferoidi. La maggior parte delle linee cellulari in questo studio sono state coltivate in terreni DMEM. Gli studi con linee cellulari coltivate in terreni diversi possono richiedere condizioni di coltura diverse per testare una varietà di rapporti di terreni; Tuttavia, questo effetto è osservato nella sperimentazione a lungo termine30.
Il nostro modello offre numerosi vantaggi rispetto ad altri modelli 3D di cancro. Gli sferoidi che generiamo hanno una morfologia e una dimensione iniziali uniformi e riproducibili, rendendo più facile confrontare i risultati del trattamento con i controlli. Al contrario, alcuni modelli che utilizzano singole cellule sospese in terreni o scaffold possono portare a distribuzioni spaziali cellulari variabili, portando a incoerenza. Questo modello dimostra che le interazioni tra popolazioni cellulari distinte possono essere studiate in modo efficace.
Tradizionalmente, i modelli di organoidi e sferoidi sono stati composti da cellule tumorali monocolturali o co-colture di cellule tumorali per studiare i gradienti di nutrienti, l'ipossia o la disposizione cellulare34. Inoltre, questo modello può essere studiato in un ambiente di scaffold entro 24 ore dalla placcatura, offrendo l'opportunità di esplorare gli eventi di invasione precoce guidati da diverse interazioni cellula-cellula. Mentre gli approcci alternativi al cancro 3D impiegano diversi componenti della matrice di scaffold per studiare i cambiamenti nella morfologia e nell'invasività degli sferoidi, questo modello li integra consentendo lo studio di questi cambiamenti nel contesto sia della dinamica della popolazione cellulare che della composizione dello scaffold35.
La maggior parte degli sferoidi prodotti con i nostri metodi mantiene la propria integrità strutturale dopo la manipolazione manuale, consentendo la raccolta dei campioni nelle applicazioni a valle. Modificazioni genetiche convenzionali come CRISPR-Cas9, la trasduzione lentivirale dello shRNA e l'interferenza del siRNA possono essere introdotte a monte della formazione degli sferoidi. I comportamenti complessi osservati in questi sferoidi suggeriscono alterazioni dinamiche nell'espressione genica, che possono essere ulteriormente studiate utilizzando l'RNA-seq. Tecniche avanzate come CycIF, scRNA-seq, CosMx e Visium possono anche essere impiegate per studiare la genomica e la proteomica a livello spaziale o di singola cellula. Questo modello di sferoide ha il potenziale per imitare gli studi in vivo di terapie tumorali mirate, catturando le interazioni tra le gemme delle cellule stromali e gli agenti terapeutici prima che raggiungano le cellule tumorali. Questo è significativo perché può aiutare a determinare se le cellule stromali influenzano la potenza del farmaco, proteggendo potenzialmente le cellule tumorali dagli effetti terapeutici.
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Ringraziamo i membri del Baylor University Developmental Oncogene Laboratory per le loro utili osservazioni e feedback durante la preparazione di questo manoscritto. Il sostegno finanziario è stato fornito dal Dipartimento di Biologia della Baylor University e dal College of Arts and Sciences, NIH-NIGMS 2SC1GM121182 (a J.A.K.).
Materiali
| Name | Company | Catalog Number | Comments |
| 15 mL Polypropylene Centrifuge Tubes | Falcon | 14-959-53A | |
| 96-Well, Treated, U-Shaped-Bottom Microplate | Thermo Scientific | 12566432 | |
| 100 mm Dish Cell Culture Treated Surface | Thermo Scientific | 130182 | |
| 170 L CO2 Incubators | VWR | VWR51014991(10810902) | |
| 200 Self-Sealing Barrier Pipet Tips | Fisher Scientific | 02-682-255 | |
| 500 mL DMEM/Hams F-12, [+] L-glutamine | Corning | 10-090-CV | |
| 1000 Self-Sealing Barrier Pipet Tips | Molecular Biology Products Inc. | 2179HR | |
| Animal-Free Recombinant Human EGF | PeproTech | AF-100-15 | |
| Automated Pipette Repeater E3x | Eppendorf | 4987000410 | |
| Basement membrane solution | Huntsman Cancer Institute | https://healthcare.utah.edu/huntsmancancerinstitute/ | |
| Bead Bath | Lab Armor | 74300706 | |
| Biorender | Biorender | Biorender.com | |
| Biosafety Laminar Flow Cabinets | Thermo Scientific | 300590389 | |
| BioSpa 8 Automated Incubator | Agilent | 23082120 | |
| Cell Counter | Invitrogen | AMQAF2000 | |
| Cell Counting Chamber Slides | Thermo Scientific | C10283 | |
| CellTracker Deep Red | Thermo Fisher Scientific | C34565 | |
| Centrifuge | Thermo Scientific | 13100675 | |
| Cholera Toxin B subunit | Sigma-Aldrich | C9903-1MG | |
| Combitips advanced, Sterile, 5.0 mL | Fisher Scientific | 13-683-714 | |
| Cytation 10 Reader Imager | Agilent | 2105245 | |
| Defined Trypsin Inhibitor | Gibco | R007100 | soybean trypsin inhibitor in 1x PBS |
| Donor Horse Serum, 500 mL, United States Origin | Corning | 35-030-CV | |
| Dulbecco's Modified Eagle's Medium | Corning | 10-013-CV | |
| Excel Sofware | Microsoft office 365 | https://www.microsoft.com/en-us/microsoft-365/excel | |
| Fetal Bovine Serum | Thermo Scientific | 26140079 | |
| Fetal Bovine Serum, qualified, US origin | Thermo Scientific | 26140079 | |
| Gentamicin Sulfate, Liquid | Corning | 30-005-CR | |
| GraphPad Prism | |||
| Hydrocortisone solution,50 µM, sterile-filtered, BioXtra | Sigma-Aldrich | H6909-10ML | |
| Image J Software | Fiji | https://imagej.net/ij/ | |
| Insulin solution human | Sigma-Aldrich | I9278-5ML | |
| Magnifying Glass | Fisher Scientific | 01-182-392 | |
| Microcentrifuge Tube | Costar | 3621 | |
| Microscope | Olympus | CKX53SF | |
| Mini Lightbox | Fisher Scientific | 361044708 | |
| Molecular Probes CellTracker Blue CMAC Dye | Fisher Scientific | C2110 | |
| Molecular Probes CellTracker Orange CMRA Dye | Fisher Scientific | C34551 | |
| Penicillin-Streptomycin Solution | Cytiva | SV30010 | |
| Penicillin-Streptomycin Solution | Cytiva | SV30010 | |
| Phase-Contrast Microscope | Echo | RVSF1000 | |
| Phosphate Buffered Saline | Fisher Scientific | SH3025601 | |
| Pipet Controller | Corning | 4099 | |
| Pipette 100 – 1,000 µL | Eppendorf | 3123000063 | |
| Pipette 20 – 200 µL | Eppendorf | 3123000055 | |
| Prism | GraphPad Software | https://www.graphpad.com/features | |
| RPMI 1640 with L-glutamine and 25 mM HEPES | Corning | 10-041-CV | |
| Serological Pipette, 10 mL | Thermo Scientific | 170356N | |
| Serological Pipette, 25 mL | Thermo Scientific | 170357N | |
| Serological Pipette, 5 mL | Thermo Scientific | 170355N | |
| Trypan Blue Solution, 0.4% | Corning | 25900CI | |
| Trypsin-EDTA, 0.25% | Corning | 25053CI |
Riferimenti
- de Visser, K. E., Joyce, J. A. The evolving tumor microenvironment: From cancer initiation to metastatic outgrowth. Cancer Cell. 41 (3), 374-403 (2023).
- Hanahan, D., Weinberg, R. A. Hallmarks of cancer: The next generation. Cell. 144 (5), 646-674 (2011).
- Anderson, N. M., Simon, M. C. The tumor microenvironment. Curr Biol. 30 (16), R921-R925 (2020).
- Baghban, R., et al. Tumor microenvironment complexity and therapeutic implications at a glance. Cell Commun Signal. 18 (1), 59 (2020).
- Sahai, E., et al. A framework for advancing our understanding of cancer-associated fibroblasts. Nat Rev Cancer. 20 (3), 174-186 (2020).
- Erdogan, B., Webb, D. J. Cancer-associated fibroblasts modulate growth factor signaling and extracellular matrix remodeling to regulate tumor metastasis. Biochem Soc Trans. 45 (1), 229-236 (2017).
- Iozzo, R. V., Sanderson, R. D. Proteoglycans in cancer biology, tumour microenvironment and angiogenesis. J Cell Mol Med. 15 (5), 1013-1031 (2011).
- Amersfoort, J., Eelen, G. Immunomodulation by endothelial cells - partnering up with the immune system. Nat Rev Immunol. 22 (9), 576-588 (2022).
- Balani, S., Nguyen, L. V., Eaves, C. J. Modeling the process of human tumorigenesis. Nat Commun. 8 (1), 15422 (2017).
- Drost, J., Clevers, H. Organoids in cancer research. Nat Rev Cancer. 18 (7), 407-418 (2018).
- Wang, E., Xiang, K., Zhang, Y., Wang, X. -. F. Patient-derived organoids (PDOs) and PDO-derived xenografts (PDOXs): New opportunities in establishing faithful pre-clinical cancer models. J Natl Cancer Cent. 2 (4), 263-276 (2022).
- Kamb, A. What's wrong with our cancer models. Nat Rev Drug Discov. 4 (2), 161-165 (2005).
- Tuveson, D., Clevers, H. Cancer modeling meets human organoid technology. Science. 364 (6444), 952-955 (2019).
- Zitvogel, L., Pitt, J. M., Daillère, R., Smyth, M. J. Mouse models in oncoimmunology. Nat Rev Cancer. 16 (12), 759-773 (2016).
- Liu, Y., et al. Patient-derived xenograft models in cancer therapy: technologies and applications. Signal Transduct Target Ther. 8 (1), 160 (2023).
- Ben-David, U., et al. Patient-derived xenografts undergo mouse-specific tumor evolution. Nat Genet. 49 (11), 1567-1575 (2017).
- Byrne, A. T., et al. Interrogating open issues in cancer precision medicine with patient-derived xenografts. Nat Rev Cancer. 17 (4), 254-268 (2017).
- Ivanics, T., et al. Patient-derived xenograft cryopreservation and reanimation outcomes are dependent on cryoprotectant type. Lab Invest. 98 (7), 947-956 (2018).
- Biju, T. S., Priya, V. V., Francis, A. P. Role of three-dimensional cell culture in therapeutics and diagnostics: an updated review. Drug Deliv and Transl Res. 13 (9), 2239-2253 (2023).
- Urzì, O., et al. Three-dimensional cell cultures: The bridge between in vitro and in vivo models. Int J Mol Sci. 24 (15), 12046 (2023).
- Cacciamali, A., Villa, R., Dotti, S. 3D cell cultures: Evolution of an ancient tool for new applications. Front Physiol. 13, 836480 (2022).
- Abuwatfa, W. H., Pitt, W. G. Scaffold-based 3D cell culture models in cancer research. J Biomed Sci. 31 (1), 7 (2024).
- Yakavets, I., et al. Advanced co-culture 3D breast cancer model for investigation of fibrosis induced by external stimuli: optimization study. Sci Rep. 10 (1), 21273 (2020).
- Mohammadrezaei, D., et al. Predicting and elucidating the post-printing behavior of 3D printed cancer cells in hydrogel structures by integrating in-vitro and in-silico experiments. Sci Rep. 13 (1), 1211 (2023).
- Kuen, J., Darowski, D., Kluge, T., Majety, M. Pancreatic cancer cell/fibroblast co-culture induces M2 like macrophages that influence therapeutic response in a 3D model. PLOS ONE. 12 (7), e0182039 (2017).
- Bray, F., et al. Global cancer statistics 2022: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin. 74 (3), 229-263 (2024).
- Kennecke, H., et al. Metastatic behavior of breast cancer subtypes. J Clin Oncol. 28 (20), 3271-3277 (2010).
- Neve, R. M., et al. A collection of breast cancer cell lines for the study of functionally distinct cancer subtypes. Cancer Cell. 10 (6), 515-527 (2006).
- Pingitore, P., et al. Human multilineage 3D spheroids as a model of liver steatosis and fibrosis. Int J Mol Sci. 20 (7), 1629 (2019).
- Bar-Ephraim, Y. E., Kretzschmar, K. Organoids in immunological research. Nat Rev Immunol. 20 (5), 279-293 (2020).
- Beerkens, A. P. M., et al. Characterizing OXPHOS inhibitor-mediated alleviation of hypoxia using high-throughput live cell-imaging. Cancer Metab. 12 (1), 13 (2024).
- Feaster, T. K., et al. Matrigel mattress. Cir Res. 117 (12), 995-1000 (2015).
- Huang, Z., Yu, P., Tang, J. Characterization of triple-negative breast cancer MDA-MB-231 cell spheroid model. Onco Targets Ther. 13, 5395-5405 (2020).
- Nam, S., Khawar, I. A., Park, J. K., Chang, S., Kuh, H. -. J. Cellular context-dependent interaction between cancer and stellate cells in hetero-type multicellular spheroids of pancreatic tumor. Biochem Biophys Res Commun. 515 (1), 183-189 (2019).
- Monteiro, M. V., Gaspar, V. M., Ferreira, L. P. Hydrogel 3D in vitro tumor models for screening cell aggregation mediated drug response. Biomater Sci. 8 (7), 1855-1864 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon